Method Article
Medición de la longitud de los telómeros de PCR cuantitativa multiplex monocromática
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí presentamos un protocolo para la medición de la longitud relativa de los telómeros (TL) utilizando el ensayo monocromo multiplex cuantitativo de reacción en cadena de la polimerasa (MMqPCR). El ensayo MMqPCR es un método repetible, eficiente y rentable para medir la TL del ADN humano en estudios poblacionales.
Resumen
Los telómeros son estructuras de ribonucleoproteínas en el extremo de todos los cromosomas eucariotas que protegen el ADN del daño y preservan la estabilidad cromosómica. La longitud de los telómeros (TL) se ha asociado con diversas exposiciones, procesos biológicos y resultados de salud. Este artículo describe el protocolo de ensayo monocromo multiplex cuantitativo de reacción en cadena de la polimerasa (MMqPCR) que se lleva a cabo de forma rutinaria en nuestro laboratorio para medir la LT media relativa del ADN humano. Existen varios métodos diferentes de medición de TL basados en PCR, pero el protocolo específico para el método MMqPCR presentado en esta publicación es repetible, eficiente, rentable y adecuado para estudios poblacionales. Este protocolo detallado describe toda la información necesaria para que los investigadores establezcan este ensayo en su laboratorio. Además, este protocolo proporciona pasos específicos para aumentar la reproducibilidad de la medición de TL mediante este ensayo, definida por el coeficiente de correlación intraclase (ICC) a través de mediciones repetidas de la misma muestra. El ICC es un factor crítico en la evaluación de la potencia esperada para una población de estudio específica; por lo tanto, la notificación de los ICC específicos de la cohorte para cualquier ensayo de TL es un paso necesario para mejorar el rigor general de los estudios poblacionales de TL. Los resultados de ejemplo que utilizan muestras de ADN extraídas de células mononucleares de sangre periférica demuestran la viabilidad de generar datos de TL altamente repetibles utilizando este protocolo MMqPCR.
Introducción
Los telómeros son complejos protectores que se encuentran al final de todos los cromosomas eucariotas, compuestos por secuencias de ADN repetitivas altamente conservadas y proteínas asociadas. El telómero protege la integridad del ADN, preservando la estabilidad cromosómica. El acortamiento progresivo de los telómeros ocurre en las células en división como resultado de la síntesis incompleta de ADN de hebra rezagada, daño en el ADN y otros factores 1,2. El aumento de la evidencia que respalda la longitud de los telómeros (TL) como biomarcador del envejecimiento y las enfermedades relacionadas con la edad a lo largo de la vida humana ha ido acompañado de un aumento en los tipos de ensayos de medición de TL utilizados para evaluar el papel de la LT en estudios de exposición, enfermedad y salud humana 3,4,5. Los metanálisis han reportado asociaciones de la LT con la mortalidad general, las exposiciones ambientales y los resultados de salud, incluyendo cáncer, enfermedad cardiovascular y diabetes 6,7,8,9,10,11,12,13 . Estas metaasociaciones derivan de estudios que utilizan una de más de dos docenas de diferentes metodologías de medición de TL donde las fortalezas de las asociaciones tienden a variar entre diferentes metodologías 14,15,16. La selección del método óptimo de medición de TL para un estudio de investigación es un paso crucial para garantizar resultados precisos, ya que cada método posee sus propias ventajas y desventajas 5,17.
Debido al costo relativamente bajo de los reactivos, la rápida entrega del ensayo, la escalabilidad y el menor requisito inicial de ADN, las técnicas de medición de TL basadas en PCR a menudo se utilizan preferentemente cuando se realizan estudios con grandes poblaciones de muestras, estudios con acceso limitado a muestras con altas concentraciones de ADN o estudios que priorizan el alto rendimiento. El primer método de medición de TL basado en PCR, la reacción en cadena de la polimerasa cuantitativa singleplex (qPCR) desarrollada originalmente por Richard Cawthon, utiliza la relación de las señales de fluorescencia de la amplificación de los telómeros (T) y del gen de copia única (S), ejecutadas en placas de PCR separadas18. En este enfoque, se utilizan cebadores complementarios a las secuencias repetidas de ADN de telómeros (T) para amplificar el contenido total de ADN telomérico en la muestra y se cuantifican mediante la detección del indicador fluorescente SYBR green. De manera similar, los cebadores complementarios a una región intergénica de un gen de copia única (S) conservado se utilizan para cuantificar el número de copias del genoma. Estas dos estimaciones se cuantifican en relación con una curva estándar de ADN genómico utilizada en todos los ensayos de un proyecto para controlar la variación de placa a placa. La división del ADN telomérico total (T) por el número de copias del genoma único (S) produce la relación T/S, una medida relativa sin unidades que representa el contenido telomérico promedio por célula para una muestra de ADN individual18,19. Por lo tanto, la relación T/S no es una medida específica de la longitud funcional; sin embargo, de acuerdo con las normas de la literatura, utilizamos el término TL promedio por muestra en todo este protocolo.
Este método se avanzó en 2009 cuando Richard Cawthon describió el ensayo de qPCR multiplex monocromo (MMqPCR) como un enfoque para reducir potencialmente la variabilidad en la relación T/S en relación con el método original de qPCR singleplex19. El ensayo MMqPCR posee los beneficios del ensayo qPCR con la ventaja adicional de medir las señales T y S dentro del mismo pozo de reacción utilizando un fluoróforo de informe, disminuyendo así el error en relación con la qPCR singleplex y dando como resultado una mayor precisión y reproducibilidad19. Además, este método multiplex reduce potencialmente los costos y mejora el rendimiento, ya que se requieren la mitad de reacciones en comparación con el ensayo singleplex19.
Dadas las ventajas de la medición de la TL con MMqPCR, este método es adecuado para los estudios poblacionales de las asociaciones de la TL con las exposiciones, los resultados de salud y los procesos biológicos. Sin embargo, iniciar el método puede ser un desafío. Para abordar estos desafíos, describimos, en detalle, el protocolo de medición MMqPCR TL utilizado en nuestro laboratorio, destacando los pasos clave implementados para aumentar la precisión del ensayo, disminuir el riesgo de contaminación y mejorar la repetibilidad.
Además, este protocolo describe los pasos para la limpieza de los datos y el cálculo del coeficiente de correlación intraclase (ICC), una medida estadística importante de la reproducibilidad de las mediciones de TL2. Con nuestros resultados representativos, demostramos la capacidad de generar altos ICCs utilizando este protocolo. Además, identificamos el control de calidad (QC) y los pasos de resolución de problemas que se espera que disminuyan la variación en las mediciones de TL y aumenten el ICC resultante. Debido a la alta repetibilidad, eficiencia y rentabilidad de este método, la medición de TL MMqPCR es ideal para la investigación epidemiológica de TL. La Figura 1 proporciona una descripción general visual del método MMqPCR tal como se describe en este protocolo.
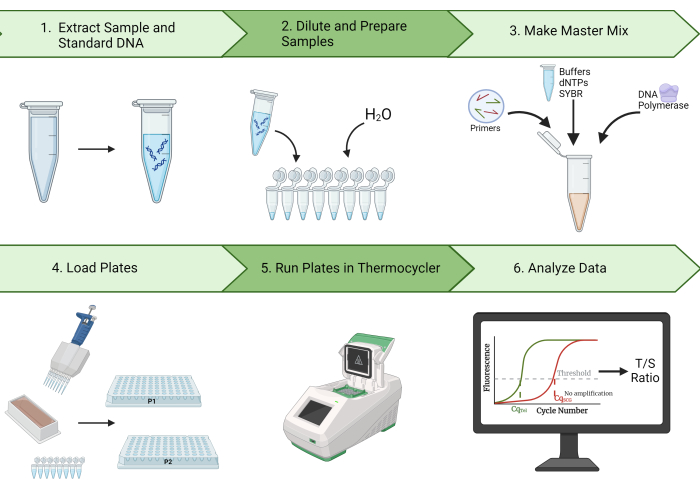
Figura 1: Descripción general del método. Una amplia visión general del método de reacción en cadena de la polimerasa cuantitativa multicromática monocromática para medir la longitud de los telómeros. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
Esta investigación se realizó de acuerdo con los lineamientos institucionales. Este protocolo describe el ensayo MMqPCR realizado a una profundidad de medición de triplicados duplicados, es decir, mediciones triplicadas de cada muestra repetidas a través de placas duplicadas, implementadas utilizando dos termocicladores simultáneamente para mejorar el rendimiento. El uso de placas duplicadas es una de las principales consideraciones que se tienen en cuenta en la implementación de este protocolo para lograr una alta reproducibilidad, como lo indican los altos ICC. A pesar de que es posible utilizar menos repeticiones, el impacto en el ICC y, posteriormente, en la potencia y el tamaño de muestra necesario, deben ser cuidadosamente considerados y medidos con cada cohorte por cada laboratorio20,21. Si solo hay un termociclador disponible, sugerimos mantener la profundidad de medición (es decir, duplicar triplicados) y hacer funcionar 2 placas secuencialmente.
1. Condiciones de preparación y almacenamiento de las existencias
- Para conocer todos los materiales y equipos utilizados para este protocolo, consulte la Tabla de materiales. Además, para conocer los reactivos y las cantidades específicos de la mezcla maestra utilizados para el ensayo MMqPCR, consulte la Tabla 1. Si se ejecutan dos placas secuencialmente, reduzca a la mitad el volumen de la mezcla maestra mientras se mantiene la concentración, lo que garantiza que se utilicen las mismas alícuotas de reactivo en las placas secuenciales del mismo ensayo.
NOTA: Todos los reactivos madre deben prepararse con anticipación y los reactivos alícuotas deben descongelarse por completo antes de su uso. - Alícuota y almacene 1x Tris-EDTA, pH 7,6 (TE) en tubos de 5 mL en volúmenes de 3 mL a temperatura ambiente durante un máximo de 2 años.
- Alicube y almacene betaína 5 M en tubos de 5 mL en volúmenes de 1.280 μL a -20 °C durante un máximo de 1 año.
- Alicuota y almacene SYBR Green en volúmenes de 3 μL en tubos individuales de tiras de PCR a -20 °C durante un máximo de 2 años, cubiertos con papel de aluminio y mínimamente expuestos a la luz.
- Almacene la ADN polimerasa en los tubos originales a -20 °C durante un máximo de 1 año.
- Alicuone y almacene tampón polimerasa 10x en tubos de 1,5 ml en volúmenes de 660 μl a -20 °C durante un máximo de 1 año.
- Para diluciones de tampón 1x de tampón polimerasa 10x, agregue 9,9 μL de tampón 10x a 89,1 μL de PCR grado H2O en tubos de 0,5 mL; almacenar a -20 °C hasta por 1 año.
- Alícuota y almacene 1 M de MgCl2 en tubos de 0,5 mL en volúmenes de 70 μL a 4 °C lejos de la luz durante un máximo de 1 año.
- Almacene los cebadores liofilizados con oligonucleótidos de genes de copia directa e inversa (T) y de un solo gen (S) a temperatura ambiente, lejos de la luz. Las cuatro secuencias específicas de cebadores de oligonucleótidos se presentan en la Tabla 2.
NOTA: El gen de copia única en este protocolo es la albúmina; si se elige un gen de copia única diferente, es posible que sea necesario ajustar las concentraciones del cebador para garantizar una eficiencia de PCR aceptable.- Reconstitución del cebador: Agite cada tubo de cebador liofilizado durante 10 s y luego coloque los tubos en una mini centrífuga durante 5 s para asegurarse de que el pellet liofilizado esté en el fondo del tubo antes de agregar 1x tampón TE.
- Verifique la concentración de nM en cada tubo de cebador individual ([nM]= alfa) y rehidrate cada tubo de cebador agregando 1x TE igual a 10x del valor alfa en μL (por ejemplo, si la concentración de nM en el tubo es de 24.6 nM, luego agregue 246 μL de 1x TE al tubo de cebador) para crear una solución de 100 μM de cada cebador.
- Cebadores de telómeros hacia adelante y hacia atrás alícuotas por separado en volúmenes de 16 μL y cebadores de albúmina hacia adelante e hacia atrás por separado en volúmenes de 11 μL. Almacene los cuatro tipos de alícuotas de imprimación a -20 °C durante un máximo de 6 meses.
- Mezcla de dNTP: Vórtice de 4 a 8 tubos de cada uno de los cuatro tipos de dNTPS de soluciones madre de 25 mM (es decir, 16 a 32 tubos en total) durante 10 s seguido de un centrifugado rápido a temperatura ambiente utilizando una mini centrífuga. Coloque los tubos en una distribución uniforme en las posiciones de los tubos en la mini centrífuga y luego cierre la tapa para permitir que la máquina alcance la velocidad máxima, girando durante unos 5 s.
- En un tubo de 5 mL, agregue 200-250 μL de cada tubo, asegurándose de que se agreguen partes iguales de cada uno de los cuatro tipos de dNTP y agite la mezcla de dNTP.
- Alícuota 210 μL de mezcla en tubos de 0,5 mL y almacenar a -20 °C hasta por 1 año.
- Almacene el DTT a -20 °C y el acetato de sodio 3 M a temperatura ambiente.
- Solución DTT: Mida 0,1545 g de DTT en un bote de pesaje tarado en una báscula con una espátula de acero inoxidable.
PRECAUCIÓN: El ditiotreitol (DTT) es un reactivo peligroso. La TDT es dañina si se ingiere, causa irritación de la piel y puede causar daños oculares graves. Las personas deben usar guantes protectores, protección para los ojos y protección facial cuando manipulen DTT. Además, las personas deben evitar respirar los vapores de DTT, trabajar en una campana de PCR cuando manipulen DTT y evitar la exposición prolongada o repetida. - Prepare 10 mL de acetato de sodio 0.01 M agregando 33.33 μL de acetato de sodio 3 M y 9,967 μL de PCR grado H2O en un tubo de 15 mL y pocillo de vórtice. Añada el DTT medido en el tubo de 15 mL de acetato de sodio 0,01 M. Transfiera aproximadamente 100 μL de la solución de acetato de sodio 0,01 M al recipiente de pesaje para recoger el DTT residual. Pipetee los 100 μL de nuevo en el tubo de 15 mL. Una vez que todo el DTT esté en la solución, vórtice el tubo hasta que se disuelva por completo.
- Vierta el contenido del tubo de 15 mL en un canal de carga, aspire toda la solución en una jeringa de plástico de 10 mL desde un extremo del canal de carga. Coloque un filtro de jeringa estéril de acetato de celulosa de 0,2 μm de 25 mm en el extremo de la jeringa llena, deje que se sature completamente durante 1 minuto y gotee lentamente la solución en un nuevo tubo de 15 ml empujando ligeramente el émbolo de la jeringa hacia abajo, asegurándose de que toda la solución gotee a través del filtro hacia el nuevo tubo de 15 mL.
- Solución alícuota de TDT en volúmenes de 200 μL en tubos de 0,5 mL y almacenamiento a -20 °C durante un máximo de 6 semanas.
- Solución DTT: Mida 0,1545 g de DTT en un bote de pesaje tarado en una báscula con una espátula de acero inoxidable.
Tabla 1: Volúmenes finales y concentraciones de reactivos. Volúmenes y concentraciones de reactivos en alícuotas individuales, mezcla maestra y en pocillos de PCR. Haga clic aquí para descargar esta tabla.
Tabla 2: Secuencias de telómeros y oligonucleótidos de genes de copia única. Lista de las secuencias de cebadores de genes de copia única de telómeros y albúmina utilizadas en la metodología. Haga clic aquí para descargar esta tabla.
2. Extracción de ADN genómico y preparación de muestras
- Extracción de muestras analíticas: Realice una extracción de ADN genómico para muestras de acuerdo con las pautas del fabricante utilizando kits o métodos establecidos dentro del laboratorio.
- Compruebe la calidad de la muestra de ADN con un espectrofotómetro y el ADN bicatenario (dsDNA) con un fluorómetro. Las muestras con proporciones medias inaceptables de 260/280 y 260/230 o una concentración de dsDNA por debajo de la detección (<0,20 ng/μL) no deben analizarse para TL 22,23. Además, si la concentración de dsDNA es demasiado alta para generar una lectura utilizando el fluorímetro (>1000 ng/μL), diluya la muestra y repita el ensayo.
NOTA: Para los resultados presentados aquí, los valores aceptables para la pureza de los ácidos nucleicos fueron una relación de 260/280 entre 1,6 y 2,0 y una relación de 260/230 entre 2,0 y 2,2.
- Compruebe la calidad de la muestra de ADN con un espectrofotómetro y el ADN bicatenario (dsDNA) con un fluorómetro. Las muestras con proporciones medias inaceptables de 260/280 y 260/230 o una concentración de dsDNA por debajo de la detección (<0,20 ng/μL) no deben analizarse para TL 22,23. Además, si la concentración de dsDNA es demasiado alta para generar una lectura utilizando el fluorímetro (>1000 ng/μL), diluya la muestra y repita el ensayo.
- Extracción de muestras de control: Realizar una extracción de ADN genómico en una muestra de control derivada del mismo material biológico que las muestras objeto de ensayo para un proyecto determinado. Asegúrese de extraer suficiente ADN de control para todas las placas que se espera que se ejecuten para toda la cohorte, de modo que se utilice un único estándar de ADN para todos los ensayos.
- Verifique la calidad de la muestra de ADN de control mediante espectrofotómetro y fluorómetro. La muestra de control debe tener proporciones medias aceptables de 260/280 y 260/230 y concentraciones detectables de dsDNA 22,23.
- ADN de control alícuota a una concentración de 2 ng/μL en cantidades de 150 μL en tubos de 0,5 mL marcados con el tipo de muestra y la fecha. Almacene las alícuotas de ADN de control a -20 °C durante un máximo de 5 años. Utilice una muestra de ADN de control para todas las placas de todas las muestras de la misma cohorte o proyecto de investigación. Prepare estas alícuotas de stock con anticipación.
- Preparación de muestras analíticas: Para muestras que tienen una concentración inicial de >5 ng/μL, cree alícuotas de dilución utilizando un factor de dilución de número entero, agregando la cantidad adecuada de muestra de ADN extraída (Archivo Suplementario 1 Hoja de Configuración de MMqPCR, Columna F) y grado de PCR H2O (Archivo Suplementario 1 Hoja de Configuración de MMqPCR, Columna G) para diluir la concentración entre 2,5 y 5,0 ng/μL (Archivo Suplementario 1 Hoja de configuración de MMqPCR, columna K). No diluya muestras con una concentración <5 ng/μL, sin embargo, alícuota un volumen suficiente para analizar la muestra 3 veces utilizando este protocolo.
NOTA: Se deben realizar alícuotas de dilución durante la preparación de la muestra para que se puedan agregar 15 ng de ADN al tubo de tira de PCR correspondiente a cada muestra. Las alícuotas de la muestra permiten a los técnicos evitar que los ciclos innecesarios de congelación y descongelación afecten la integridad del ADN de la muestra en stock si la muestra no cumple con los criterios de calidad y debe repetirse. La plantilla de MMqPCR de muestra que se encuentra en el Archivo Complementario 1 puede ayudar a identificar el factor de dilución y los volúmenes correctos para preparar alícuotas de dilución (Archivo Suplementario 1, Hoja de configuración de MMqPCR, Columnas E-I).- Llene las tiras de PCR con las muestras A1-8 en la tira de PCR A, las muestras B1-8 en la tira de PCR B y las muestras C1-8 en la tira C de PRC, como se detalla en la hoja de configuración de MMqPCR del archivo complementario 1 y se enumeran en la tabla 3 con cantidades de muestra adecuadamente diluidas (hoja de configuración del archivo complementario 1 MMqPCR, columna L) y cantidades de PCR de grado H2O (archivo complementario 1 Hoja de configuración de MMqPCR, columna M) para una cantidad total de 15 ng de ADN por tubo de PCR y un volumen total de 75 μL. Déjelo a un lado.
NOTA: Las muestras en tiras de PCR se pueden almacenar durante la noche a 4 °C para ser sembradas al día siguiente.
- Llene las tiras de PCR con las muestras A1-8 en la tira de PCR A, las muestras B1-8 en la tira de PCR B y las muestras C1-8 en la tira C de PRC, como se detalla en la hoja de configuración de MMqPCR del archivo complementario 1 y se enumeran en la tabla 3 con cantidades de muestra adecuadamente diluidas (hoja de configuración del archivo complementario 1 MMqPCR, columna L) y cantidades de PCR de grado H2O (archivo complementario 1 Hoja de configuración de MMqPCR, columna M) para una cantidad total de 15 ng de ADN por tubo de PCR y un volumen total de 75 μL. Déjelo a un lado.
- Preparación de la curva estándar: descongele una alícuota de ADN de control, un vórtice durante 30 s, una centrífuga durante 5 s y aspire el volumen completo (150 μL) en el primer tubo de la tira de tubo de PCR de curva estándar (SC).
- Pipetear 70 μL de PCR grado H2O en los tubos segundo a octavo de la tira de PCR SC. Vuelva a suspender completamente el ADN de control en el primer tubo antes de aspirar 70 μL y dispensarlo en el segundo tubo de PCR. Espere 30 s y, a continuación, vuelva a suspender completamente la solución en el segundo tubo de PCR antes de aspirar 70 μL y dispensarla en el tercer tubo de PCR. Repita esto para los tubos tres a siete para crear una dilución en serie de 2 veces de siete estándares. El tubo final solo debe contener PCR grado H2O para funcionar como un control sin plantilla (NTC). Deje la tira de PCR SC a un lado.
Tabla 3: Organización de las muestras y patrón de control en placa. Ubicación de todas las muestras y patrones en una placa de PCR de 96 pocillos. Haga clic aquí para descargar esta tabla.
3. Preparación de la mezcla maestra MMqPCR
- Reúna las alícuotas de los reactivos de la mezcla maestra enumeradas en la Tabla 1, excepto la ADN polimerasa, y deje que alcancen la temperatura ambiente dentro de la campana de PCR. Añada 1 μL de la alícuota SYBR Green a la alícuota tampón 1x.
NOTA: Esta cantidad exacta es necesaria para generar la concentración correcta de SYBR debido a las limitaciones de la precisión de la pipeta durante el ensayo, por lo que incluso si solo se ejecuta una placa a la vez, esta cantidad debe permanecer sin cambios. - Agite todas las alícuotas durante 10 s, centrifugue durante 5 s, luego agregue las siguientes cantidades de reactivos al tubo de 5 mL que contiene la betaína 5 M (ahora el tubo de mezcla maestra): 1,235.2 μL de PCR grado H2O, 640 μL del tampón 10x, 204.8 μL de los dNTP, 192 μL de la TDT, 64 μL de MgCl2, 48 μL de SYBR Green / 1x tampón (del paso 3.1), 14,4 μL de ambos cebadores de telómeros y 9,6 μL de ambos cebadores de albúmina, como se indica en la Tabla 1.
- Saque la ADN polimerasa del almacenamiento a -20 °C, vórtice durante 10 s, centrifugue durante 5 s, luego agregue lentamente 128 μL a la mezcla maestra; mover inmediatamente la ADN polimerasa de vuelta al almacenamiento de -20 °C.
- Agite el tubo de mezcla maestra de 5 mL durante 30 s.
4. Preparación de la placa de 96 pocillos
- Coloque dos placas de 96 pocillos, un canal de carga y puntas de pipeta multicanal dentro de la cubierta de PCR. Etiquete cada plato con el número de placa (1 o 2). Gire la placa uno (la placa P1) 180° para que los números de las columnas queden al revés. Deje la placa dos (P2) mirando hacia el técnico.
- Vierta el contenido del tubo de mezcla maestra de 5 ml en el canal de carga. Utilice una punta de pipeta para recoger y dispensar cualquier solución que quede en el tubo.
- Cargue las puntas en la pipeta multicanal, empujando la base de cada punta de pipeta para garantizar un sellado hermético. Compruebe que las puntas no tengan ningún filtro dentro de la región de salida donde se aspirará la mezcla maestra; Si hay un trozo de filtro, reemplace la punta.
- Utilice el pipeteo inverso para llenar las placas con la mezcla maestra viscosa, gire el canal de carga y, a continuación, presione el émbolo de la pipeta más allá del primer tope, aspirando más de 15 μL en las puntas de la pipeta, asegurándose de que las puntas se llenen al mismo tiempo con el mismo volumen. Al expulsar la mezcla maestra en una columna, presione el émbolo hasta el primer tope, dejando la mezcla maestra adicional en las puntas de pipeta. Dejando el émbolo presionado, sumerja las puntas de nuevo en el canal de carga y suelte el émbolo para llenar las puntas con otros 15 μL. Utilice las mismas puntas para llenar ambas placas.
- Llene las placas expulsando primero la mezcla maestra en todas las columnas pares o en todas las impares, alternando entre placas (es decir, si el técnico elige comenzar con números de columna impares, la columna 1 en P2 se llenará primero, seguida de la columna 11 en P1, luego la columna 3 en P2, etc.). Después de que el técnico haya llenado todos los pozos de la serie (pares o impares) trabajando de izquierda a derecha, haga las otras series (pares o impares) de columnas de la misma manera. Consulte la Figura 2 para ver un gráfico de este proceso.
NOTA: Esta forma de llenado de placas funciona para controlar los efectos de posición a través de las placas.
- Llene las placas expulsando primero la mezcla maestra en todas las columnas pares o en todas las impares, alternando entre placas (es decir, si el técnico elige comenzar con números de columna impares, la columna 1 en P2 se llenará primero, seguida de la columna 11 en P1, luego la columna 3 en P2, etc.). Después de que el técnico haya llenado todos los pozos de la serie (pares o impares) trabajando de izquierda a derecha, haga las otras series (pares o impares) de columnas de la misma manera. Consulte la Figura 2 para ver un gráfico de este proceso.
- Agite las cuatro tiras de PCR cerradas que contienen las muestras y la curva estándar y, a continuación, haga girar cada una en una mini centrífuga durante 5 s.
- Alinee las tiras de PCR en la gradilla de tubos de PCR en el orden en que se colocarán en la placa. Para P1, las primeras 3 columnas son muestras (tira de PCR A), las columnas 4-6 son el estándar (tira de PCR S), las columnas 7-9 son muestras (tira de PCR B) y las columnas 10-12 son muestras (tira de PCR C; ver Figura 3 y Tabla 3). Dado que P2 se llena mientras se gira 180° con respecto al técnico, las columnas serán lo contrario.
- Gire ambas placas 180° para que los números de las columnas se inviertan en relación con el técnico de cada placa (es decir, si el técnico miraba hacia la etiqueta para P2 anteriormente, ahora mirará hacia la etiqueta para P1 en el borde de la placa).
- Ajuste la pipeta multicanal a 10 μL y cargue las puntas de pipeta en la pipeta multicanal como se describe en el paso 4.3.
- Utilice la pipeta multicanal para resuspender las soluciones y, a continuación, realice el pipeteo inverso como se describe en el paso 4.4 para aspirar y dispensar 10 μL de las muestras y soluciones estándar, empezando por la tira de PCR A.
- Llene las primeras 3 columnas de cada placa con la tira de PCR A alternando las columnas de manera similar a como se colocó la mezcla maestra en la placa (es decir, par o impar).
- Rellene las tres columnas siguientes con la tira de PCR de curva estándar. Para esta tira, vuelva a suspender el7º tubo de dilución estándar (penúltimo), utilizando una pipeta de 200 μL ajustada a ~90 μL, a fondo antes de volver a suspender toda la tira con la pipeta multicanal.
NOTA: El mayor volumen y la menor concentración de ADN en este tubo con frecuencia dan como resultado mediciones variadas en los pocillos de la placa de PCR debido a una mezcla deficiente. El uso de un volumen mayor para mezclar este tubo homogeneiza mejor la solución de ADN. - Llene las tres columnas siguientes con la tira de PCR B y las tres últimas con la tira de PCR C. Reemplace las puntas de pipeta entre cada tira de PCR, llenando los 96 pocillos de ambas placas con las muestras correspondientes y el estándar de control como se describe en la Figura 3.
- Una vez que las placas estén llenas, golpee suavemente la placa en la parte superior de la mesa para asegurarse de que el líquido en los lados de los pocillos corra hasta el fondo y luego cubra la parte superior de las placas con películas de sellado. Use las yemas de los dedos para presionar todos los bordes de la película para asegurar un sellado hermético.
- Mezcle las placas girándolas en la parte superior de la campana durante 30 s, luego coloque las placas selladas en la rueda giratoria de placas durante 2 minutos con las aberturas del pozo hacia el centro.
- Coloque las placas en un termociclador, con los números de las columnas de las placas en orden legible. Use un pañuelo de papel limpio para limpiar la parte superior de cada placa antes de cerrar la tapa del termociclador.
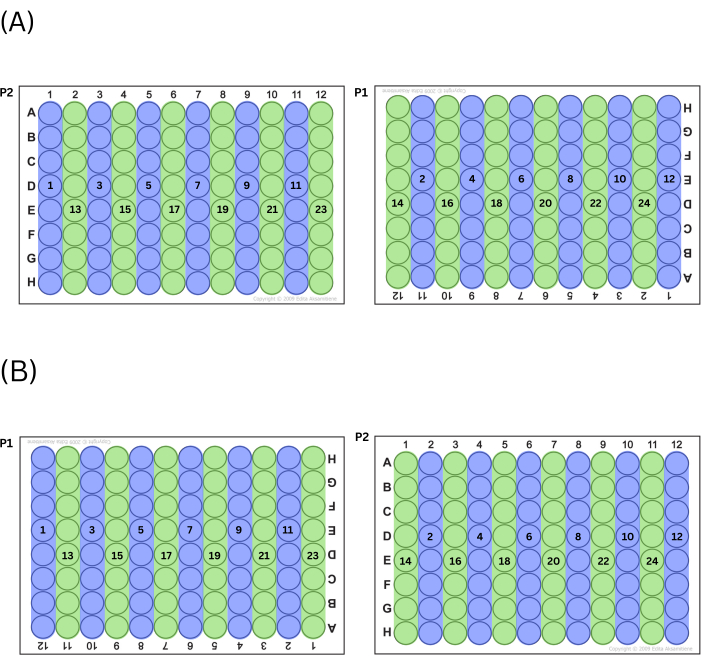
Figura 2: Proceso de llenado de placas. (A) Si elige llenar primero las columnas impares, este es el orden en que se llenan los pozos. (B) Si elige llenar primero las columnas pares, este es el orden en que se llenan los pozos. Haga clic aquí para ver una versión más grande de esta figura.
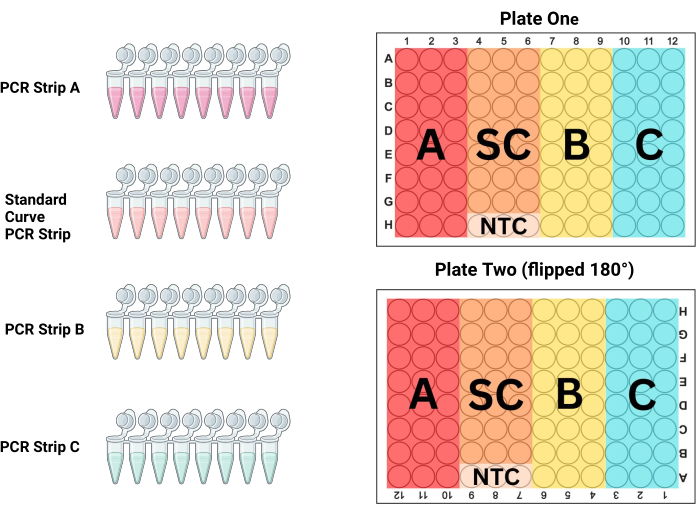
Figura 3: Disposición de las placas. La tira de PCR A, la tira B, la tira C y la tira de curva estándar (SC) deben usarse para llenar tres columnas en cada placa para producir triplicados duplicados de cada muestra y dilución estándar. Este diagrama muestra cuál de las columnas debe llenarse con cada tira. La placa dos se gira 180° (tenga en cuenta que los encabezados de columna y fila están al revés) antes de cargarla, pero la placa se llena de manera idéntica a la placa uno, lo que elimina posibles errores de pipeteo y al mismo tiempo controla los efectos de posición en las placas. Haga clic aquí para ver una versión más grande de esta figura.
5. Termociclado MMqPCR
- Mientras las placas giran en el paso 4.8.1, encienda la computadora y los termociclistas. Abra el software del termociclador. Este protocolo describe el uso del software CFX Maestro.
- Cree el protocolo de termociclado TL de acuerdo con el protocolo de termociclado MMqPCR19 (Figura 4).
- Añadir un paso de incubación para activar la ADN polimerasa a 95 °C durante 15 min.
- Para evitar la unión del cebador y el dímero, ejecute dos ciclos de 94 °C durante 15 s, 49 °C durante 1 min, luego 3 ciclos de 94 °C durante 15 s, 59 °C durante 15 s.
- Para la amplificación de los telómeros, agregue 27 ciclos de 85 °C durante 15 s, 74 °C durante 30 s y luego la adquisición de la señal.
- Para la amplificación de la albúmina, agregue 31 ciclos de 94 °C durante 15 s, 84 °C durante 30 s y luego la adquisición de la señal.
- Incluya una curva de fusión de 59 °C a 95 °C a intervalos de 5 s por cada grado creciente en el protocolo de ciclo térmico.
- Haga clic en Iniciar ejecución para ambos termociclistas. Cuando se le solicite, proporcione un título para los archivos de análisis.
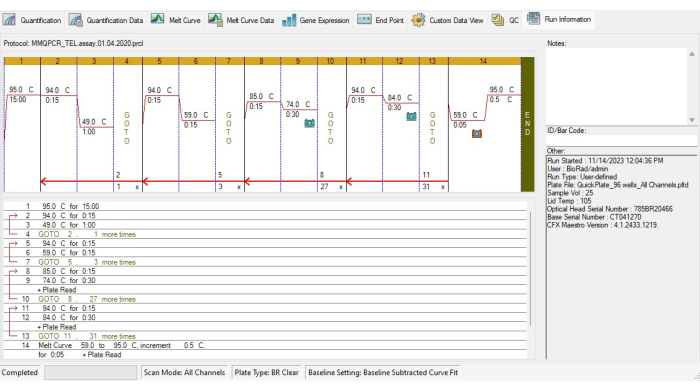
Figura 4: Perfil de termociclado del ensayo MMqPCR. Protocolo MMqPCR creado en el software de acuerdo con el protocolo de termociclado original19. Haga clic aquí para ver una versión más grande de esta figura.
6. Análisis de datos MMqPCR
- Una vez finalizado el termociclado, analice los datos de la siguiente manera para producir valores TL para las muestras que se han procesado.
- En el software, seleccione la función Configuración de placas . En el menú desplegable, haga clic en Ver/Editar placa.
- Resalte todos los pocillos y haga clic en Seleccionar fluoróforos, luego marque la casilla etiquetada SYBR y desmarque todas las demás casillas. Haga clic en Aceptar.
- Mientras todos los pozos siguen resaltados, junto a la palabra Cargar, marque la casilla junto a SYBR. Ahora todos los pozos deben tener SYBR escrito en ellos.
- A continuación, resalte los tres pocillos NTC en la parte superior o inferior de la dilución en serie de control estándar y seleccione NTC en el menú de tipo de muestra a la derecha. Los tres pozos ahora deberían ser amarillos y llamarse NTC. La Figura 5A muestra qué pozos deben seleccionarse para P1 y la Figura 5B muestra qué pozos deben seleccionarse para P2.
NOTA: P2 se invertirá de P1, por lo que el NTC estará en la parte inferior de las columnas 4-6 para P1 y en la parte superior de las columnas 7-9 para P2. - Resalte los 21 pocillos de la dilución en serie de control estándar y seleccione Estándar en el menú de tipo de muestra. Estos pozos deben ser verdes. Mientras estos pozos aún están resaltados, haga clic en Replicación técnica. Seleccione 3 en el menú Tamaño de réplica y, a continuación, seleccione Horizontal > Aplicar. Estos pozos deben estar etiquetados en grupos de tres desde Std-1 hasta Std-7.
- Mientras el estándar aún está resaltado, desplácese hacia abajo y seleccione Serie de dilución. En el campo Factor de dilución, introduzca 2 y, a continuación, escriba la concentración inicial de dilución y la direccionalidad según el número de placa, tal como se describe en los pasos siguientes.
- Para la placa P1, ingrese 2.00E-03 en el campo de concentración inicial y marque la casilla de Disminución y luego seleccione Aplicar. Los valores en los 21 pozos deben tener los valores de concentración escritos en cada pozo que van desde 2.00E-03 hasta 3.13E-05 de arriba a abajo.
- Para la placa P2, introduzca 3.13E-5 en el campo de concentración inicial y marque la casilla Creciente y, a continuación, seleccione Aplicar. Los valores en los 21 pozos deben tener los valores de concentración escritos en cada pozo que van desde 3.13E-05 a 2.00E-03 de arriba a abajo.
- Para las muestras, resalte las columnas de la tira de PCR A (1-3 para P1 y P2), seleccione Desconocido en el menú de tipo de muestra y, a continuación, seleccione Réplica técnica. Seleccione 3 en el menú Tamaño de réplica y, a continuación, seleccione Horizontal > Aplicar. Los pozos para estas columnas deben ser azules y etiquetados por fila en grupos de 3 desde Unk-1 hasta Unk-8.
- Repita el paso 6.3 para las columnas de la tira de PCR B (columnas 7-9 para P1 y 4-6 para P2). Deben estar etiquetados como Unk-9 a Unk-16.
- Repita el paso 6.3 para las columnas de la tira de PCR C (columnas 10-12 para P1 y P2). Deben estar etiquetados como Unk-17 a Unk-24. Cuando se completan los pasos 6.2-6.3.2, las ventanas de configuración de la placa deben parecerse a la Figura 5A,B.
- Seleccione Aceptar en la parte inferior derecha de la ventana del editor de placas. Haga clic en Sí para aplicar los cambios cuando se le solicite.
- Para el control de calidad a nivel de ensayo, asegúrese de que las curvas de la pestaña de cuantificación sean adecuadas (por ejemplo, sin curvas de amplificación invertidas) para el paso 9 y el paso 12 del perfil de termociclado, que corresponden a los amplicones de telómero y albúmina respectivamente. Se accede a las curvas mediante el menú desplegable en el lado central derecho de la ventana del software. La figura 6A,B muestra las curvas apropiadas.
- Asegúrese de que las eficiencias de PCR informadas tanto en el Paso 9 como en el Paso 12 estén entre el 90 % y el 110 % y que estas dos eficiencias no difieran entre sí más del 10 % (es decir, 92,4 % para el Paso 9 y 98 % para el Paso 12 es apropiado, pero 92,4 % para el Paso 9 y 108 % para el Paso 12 no es apropiado). Además, asegúrese de que la curva R2 estándar sea de >0,995. En caso de que P1 o P2 no cumplan con estos criterios, es necesario volver a ejecutar las placas.
- Para P1, seleccione el Paso 9 y resalte los 21 pozos correspondientes a la curva estándar como se ve en la Figura 6A,B. Copie los valores de Cq en la esquina inferior derecha de la ventana del software. Abra la plantilla de hoja de datos de telómeros disponible como Archivo complementario 1 y pegue estos valores en la hoja estándar 1, columna B. Seleccione el paso 12 y, a continuación, copie estos valores Cq. Pegue estos valores en la hoja estándar 1, columna I.
- Identifique el valor de la pendiente en el paso 9 y escríbalo en la celda C4 de la hoja estándar 1. Identifique el valor de la pendiente en el paso 12 y escríbalo en la celda J4 de la hoja estándar 1.
NOTA: El sistema calcula el porcentaje de eficiencia de los cebadores utilizando las pendientes de la curva estándar identificada en el paso 6.6.1. El porcentaje de eficiencia se puede calcular manualmente utilizando la siguiente fórmula: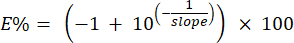
- Asegúrese de que el coeficiente de variación (CV) para cada triplicado de dilución estándar sea inferior a 0,1. Si es mayor o igual a 0.1, excluya hasta tres pozos individuales en total de todas las concentraciones de muestras de control incluidas en la curva de dilución serial estándar de 7 puntos en la placa. Solo se debe excluir un pocillo de cualquier conjunto por triplicado.
NOTA: La exclusión de puntos estándar del análisis en el software cambiará la pendiente informada, R, la intersección con el eje y y la eficiencia de ambos cebadores. Por lo tanto, después de que cualquier pozo estándar se elimina del análisis, se deben repetir los pasos 6.5.1-6.6.1. - Repita los pasos 6.6.1 - 6.6.2 para el archivo de análisis P2 y la hoja estándar 2 correspondiente.
- Solo cuando las hojas estándar 1 y estándar 2 se completen con CV aceptables, recoja datos de muestra individuales de los archivos de análisis. Asegúrese de que los ajustes de control de calidad se informen en la columna L de la hoja P1 v P2, por ejemplo, cuántos pozos se excluyeron en el archivo de análisis P1.
- Identifique el valor de la pendiente en el paso 9 y escríbalo en la celda C4 de la hoja estándar 1. Identifique el valor de la pendiente en el paso 12 y escríbalo en la celda J4 de la hoja estándar 1.
- Asegúrese de que solo los 72 pozos de muestra en el archivo de análisis P1 estén resaltados en azul en la pestaña Cuantificación, como se ve en Figura 7A,B. Escoger Paso 9. Copie los valores Cq y SQ en la esquina inferior derecha de la ventana del software. Pegue estos valores en la hoja P1 de muestras, comenzando en la celda D3. Escoger Paso 12 a continuación, copie estos valores Cq y SQ, y péguelos en la hoja P1 de muestras, empezando por la celda F3.
- Repita este paso para las muestras del archivo de análisis P2 y la hoja de muestras P2 correspondiente. Las proporciones de telómeros a copia única (T/S) (columna H) se calculan automáticamente, pero se pueden calcular manualmente utilizando la siguiente fórmula:
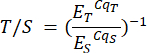
Donde ET/S es la eficiencia de la amplificación exponencial para reacciones dirigidas al telómero o al gen de copia única, respectivamente, y CqT/S es el ciclo en el que un contenido telomérico dirigido a una réplica dada o al gen de copia única alcanza el umbral crítico de cuantificación de fluorescencia. Las relaciones T/S promedio (Columna I) se calculan a través de seis mediciones por muestra.
NOTA: Los números de identificación de la muestra en CFX no corresponden a la misma muestra biológica en las dos placas. La plantilla de la hoja de datos de los telómeros tiene en cuenta esta diferencia y alineará los triplicados duplicados adecuados. - En CFX Maestro, verifique que los valores de Cq para todas las muestras analíticas se encuentren entre el valor de Cq más bajo y el más alto de la curva estándar, como se ve en la Figura 7B. Si una muestra está fuera del rango como se ve en la Figura 7A, será necesario volver a ejecutarla después del ajuste de concentración. Una muestra con un valor de Cq inferior al estándar de concentración más alto debe diluirse por un factor aproximado al número de ciclos de amplificación que está fuera del rango, y una muestra con un valor de Cq superior al estándar de concentración más bajo tendría que concentrarse por un factor aproximado al número de ciclos de amplificación más allá del umbral.
NOTA: La amplia variación en la concentración de las muestras analíticas introduce un nivel potencial adicional de variación y debe hacerse con precaución. - Asegúrese de que la concentración inicial en el NTC sea inferior al 5% de la cantidad promedio de ADN presente en los pocillos de muestra. Esto se puede comprobar visualizando el % de contaminación del agua en las hojas de las muestras P1 y P2 de la MMqPCR. Si el NTC indica contaminación, vuelva a encender las placas.
NOTA: Debido a la sensibilidad del ensayo MMqPCR, una contaminación incontrolable menor puede ocasionalmente hacer que los pocillos de NTC se amplifiquen. Sin embargo, esta amplificación debe ser menor y ocurrir varios ciclos después de la amplificación del gen de copia única en el Paso 12 del protocolo de termociclado. - Para el control de calidad a nivel de muestra, revise las desviaciones estándar (DE) (columna J) y las CV (columna K) en las muestras P1 y las hojas de muestras P2. Asegúrese de que los CV intraplaca a través de las relaciones T/S triplicadas de la muestra sean inferiores a 0,10 (10%). Para corregir cualquier CV intraplaca superior a 0,1, se puede excluir una relación T/S de un conjunto de triplicados.
NOTA: Solo se puede excluir una réplica total en las seis mediciones por muestra, es decir, no se puede excluir una réplica para la misma muestra tanto en P1 como en P2. - Compruebe que las CV de la placa interplaca de muestra individual en la hoja P1 v P2, columna G, sean inferiores a 0,05 (5%). Para corregir cualquier CV mayor que 0,05, realice la exclusión de una relación T/S de las seis mediciones por muestra en ambas placas. Excluya las muestras después de la inspección visual a través de los triplicados intraplaca y las seis mediciones según sea necesario.
NOTA: Solo las muestras que superen estos criterios de control de calidad, CV intraplaca < 10% y CV entre placas < 5%, pueden incluirse en los resultados finales de TL. De lo contrario, la muestra debe excluirse de las hojas de datos P1 v P2 e ICC para esta ejecución, y la muestra debe volver a evaluarse en un segundo ensayo MMqPCR. - Para el control de calidad a nivel de placa, compruebe que la variación media dentro de la placa en todas las muestras de cada placa, situada en la parte inferior de las muestras P1 y las hojas de las muestras P2, sea inferior a 0,05 (5%). Para el control de calidad adicional a nivel de placa, compruebe que la variación general entre placas en la hoja P1 v P2, celda G29, sea inferior a 0,06 (6%). Asegúrese de que las muestras que no pasaron el control de calidad se eliminen del cálculo de CV entre placas en la celda G30.
NOTA: Cuando se trabaja con grupos de muestras relacionados (por ejemplo, miembros de la familia, diferentes puntos de tiempo, etc.), todas las muestras de un grupo deben ejecutarse en la misma placa. Por lo tanto, si una muestra del grupo no supera los criterios de control de calidad, se deben volver a ejecutar todas las muestras del grupo. - En un archivo de datos de cohorte o experimento general separado, registre los datos finales de TL para cada muestra que pasó el control de calidad (hoja P1 v P2, columna I), los cálculos de ICC para cada muestra que pasó el control de calidad (hoja de datos ICC, columnas E-K) y los datos de control de calidad de la ejecución del ensayo final de la hoja P1 v P2 (columnas J-L)
- Repita este paso para las muestras del archivo de análisis P2 y la hoja de muestras P2 correspondiente. Las proporciones de telómeros a copia única (T/S) (columna H) se calculan automáticamente, pero se pueden calcular manualmente utilizando la siguiente fórmula:
- Utilice el Archivo Suplementario 2 creado por la Red de Investigación de Telómeros (TRN) para calcular el ICC para el proyecto20.
- Por último, para mejorar la comparabilidad entre los estudios de TL, transforme los datos finales de TL para cada muestra en el archivo de datos de la cohorte general o del experimento por separado en puntuaciones Z utilizando la siguiente ecuación en la que la relación T/S media de todas las muestras de la cohorte se resta de la relación T/S de la muestra individual y luego se divide por la DE en todas las muestras de la cohorte.
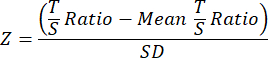
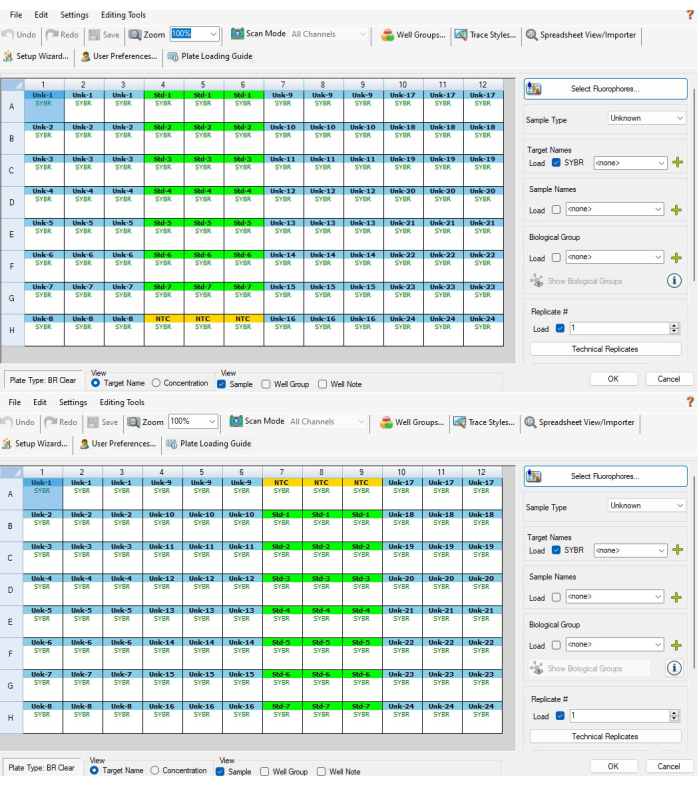
Figura 5: Configuración de placas basada en software. (A) Configuración de placa basada en software para una placa P1 después de completar los pasos 6.2-6.4. (B) Configuración de placa basada en software para una placa P2 después de completar los pasos 6.2-6.4. Los ID de muestra y estándar no están alineados entre las dos placas CFX debido a la forma en que el software asigna los ID de muestra en función de la posición del pozo (es decir, la muestra CFX 1 en P1 es la muestra CFX 24 en P2). La plantilla de Excel proporcionada en el Archivo Suplementario 1 da cuenta de esto, asegurando que las mediciones entre placas realizadas en la misma muestra biológica estén correctamente alineadas entre sí. Haga clic aquí para ver una versión más grande de esta figura.
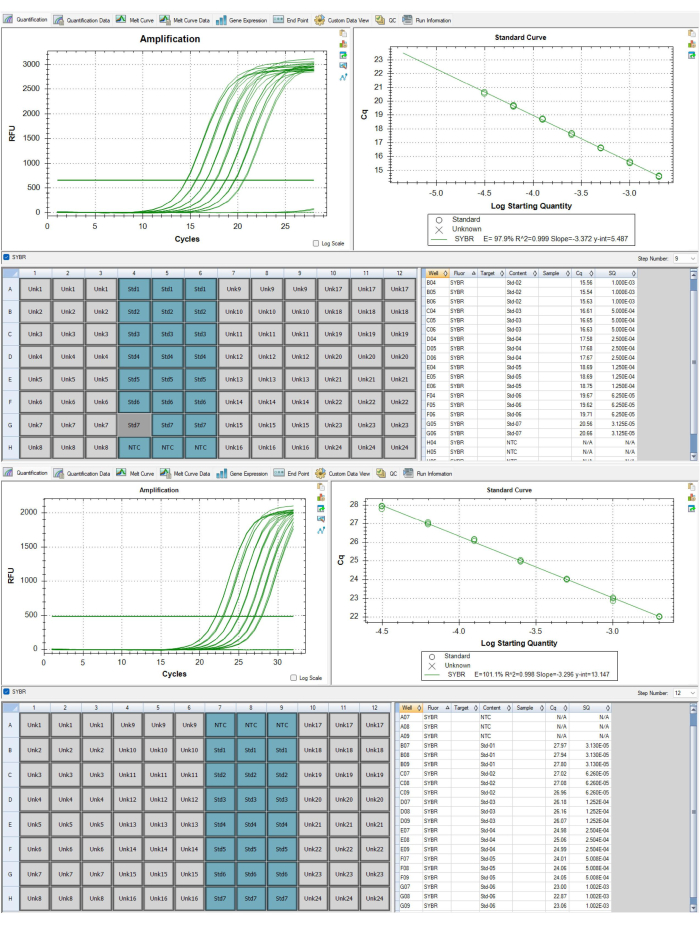
Figura 6: Curvas estándar paraamplicones de telómero y albúmina. (A) Esta curva estándar proviene del amplicón de telómeros P1 del conjunto de datos de resultados representativos. Se eliminó una norma porque no cumplía con los criterios de control de calidad. (B) Esta curva estándar proviene del amplicón de albúmina P2 del conjunto de datos de resultados representativos. Haga clic aquí para ver una versión más grande de esta figura.
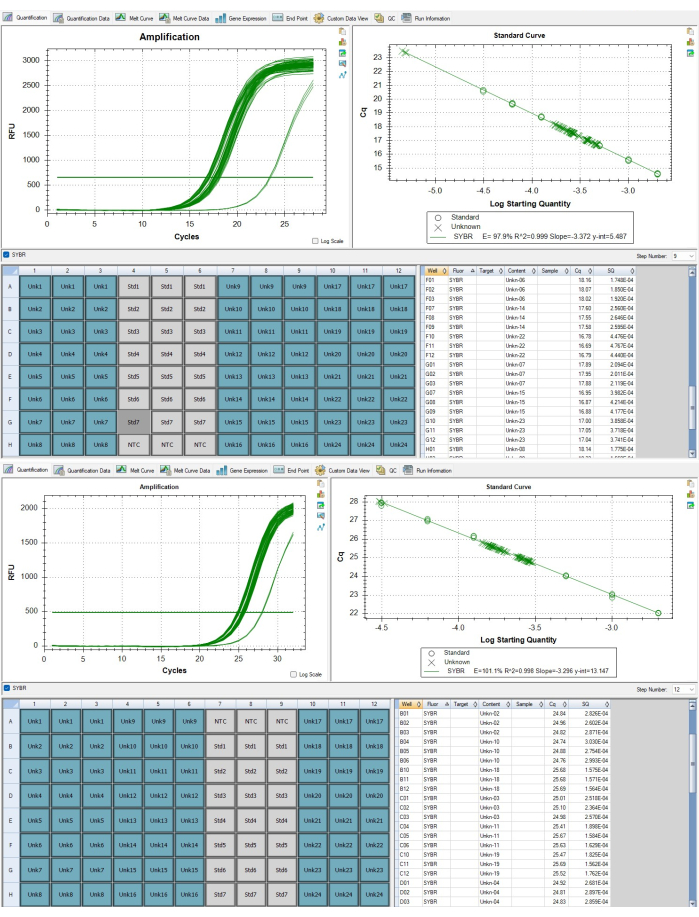
Figura 7: Telómeros y amplicones de genes de copia única. (A) Amplificación de telómeros y (B) amplificación del gen de la albúmina de muestras reportadas en resultados representativos mostrados en el software. Haga clic aquí para ver una versión más grande de esta figura.
7. Informes de datos MMqPCR
- Al informar los resultados de la investigación utilizando los datos de TL creados a partir de la MMqPCR, asegúrese de informar los elementos descritos en las Pautas mínimas de informes de TRN. Estas directrices se pueden encontrar en el sitio web de TRN (https://trn.tulane.edu/resources/reporting-guidelines/) y en el Archivo Complementario 3 se proporciona una plantilla para presentar esta información. Cite este protocolo y otras directrices y recursos de TRN cuando corresponda.
Resultados
Los resultados presentados en la Tabla 4 y la Tabla 5 ofrecen un ejemplo de mediciones de TL altamente repetibles obtenidas siguiendo el protocolo. Para estos resultados, se extrajo ADN de 24 muestras de células mononucleares de sangre periférica (PBMC) utilizando un kit comercial según las pautas de los fabricantes. Estas 24 muestras se analizaron a través de dos placas de 96 pocillos. La calidad de todas las muestras de ADN se verificó mediante espectrofotómetro y fluorómetro, utilizando la relación promedio de 260/280, la relación promedio de 260/230 y la concentración de dsDNA para determinar la elegibilidad del ensayo y el factor de dilución de la muestra (Tabla 6). La Tabla 6 también destaca la importancia de cuantificar la concentración de dsDNA, que puede diferir de la concentración de ADN medida con un espectrofotómetro. Esta variabilidad es el resultado de diferentes enfoques de cuantificación. Específicamente, el espectrofotómetro deriva la concentración de ADN en función de la absorción a 280 nm y es susceptible a las fluctuaciones debidas a contaminantes (por ejemplo, proteínas, sal, etc.) que afectan las lecturas de absorbancia. Por el contrario, las concentraciones de dsDNA medidas con un fluorómetro se determinan por la fluorescencia de un colorante que se une específicamente al dsDNA y, como tal, se supone que es un reflejo más preciso del contenido de ADN. Las muestras de ADN experimentaron hasta tres ciclos de congelación-descongelación antes del análisis de MMqPCR TL. El ADN de control se creó a partir de extracciones de ADN agrupadas de PBMC de un individuo, que se utilizó para crear una dilución serial de siete puntos de 2 ng/μL a 0,0313 ng/μL de ADN. Las curvas estándar independientes creadas para la PCR Paso 9 (amplicón de telómeros) y Paso 12 (amplicón de gen de copia única, albúmina en este protocolo) se presentan en la Figura 6A,B. Los ensayos se ejecutaron en un sistema comercial de detección de PCR en tiempo real que generó una curva de fusión que muestra los productos de amplicón individuales que se producen a distintas temperaturas, como se ve en la Figura 8.
Tabla 4: Datos de salida de la medición de MMqPCR para obtener resultados representativos óptimos. Promedio de las relaciones T/S, SD, CV y TL con puntuación Z para 24 muestras. Haga clic aquí para descargar esta tabla.
Tabla 5: Salida de datos de longitud de los telómeros. Los datos brutos del ensayo MMqPCR se analizan en el software y luego se agregan a la plantilla de hoja de cálculo adjunta como Archivo complementario 1 para su posterior análisis y control de calidad. Esta hoja de salida de la plantilla TL presenta un resumen de los datos, mostrando los ID de muestra, los TL promedio, los SD y los CV de cada muestra en ambas placas. Haga clic aquí para descargar esta tabla.
Tabla 6: Métricas de calidad del ADN del espectrofotómetro y del fluorómetro para los resultados de las muestras. Datos de control de calidad del análisis de espectrofotómetro duplicado y medición de fluorómetro singular de dsDNA y cualquier contaminante por muestra. Haga clic aquí para descargar esta tabla.
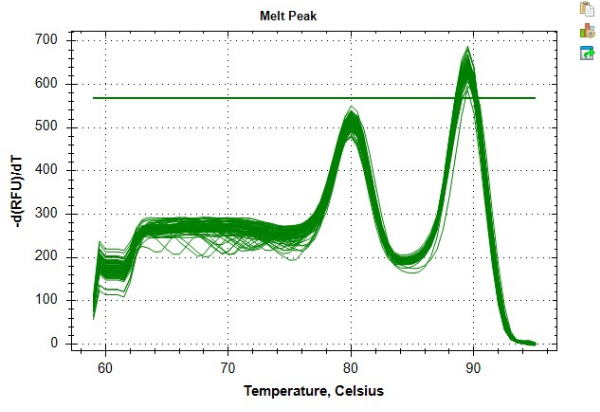
Figura 8: Ejemplo de curva de fusión. Curva de fusión para los datos de muestra tal como se generan en el software. El pico anterior ~80 °C representa el amplicón de los telómeros y el pico secundario a ~89 °C representa el amplicón de la albúmina. Haga clic aquí para ver una versión más grande de esta figura.
Para este proyecto, la eficiencia promedio de los telómeros fue del 98,6% con un rango de 90,7 a 102,1 y la eficiencia promedio de la albúmina fue del 102,3% con un rango de 93,6 a 108,2 en todas las corridas. Se eliminó un promedio de 0,97 réplicas de las curvas estándar, lo que cumple con los criterios de control de calidad de este protocolo. El CV medio entre placas fue del 1,83% con una DE de 0,00616 y el CV medio entre placas fue del 3,78% con una DE de 0,00658. En la Tabla 4 y la Tabla 5 se presentan los TL promedio para estas muestras representativas.La media de LT fue de 1,37 con una DE de 0,24 y un rango de 0,84 a 2,32. En la Tabla 4 se enumeran la relación T/S, SD, CV y TL con puntuación Z por muestra. Dado que la relación T/S del ensayo MMqPCR es una medida relativa de TL, las proporciones se transformaron en esta puntuación Z para permitir la comparación entre estudios. El ICC para este proyecto se calculó utilizando el script R como se describe en las pautas de TRN que se encuentran en el Archivo Suplementario 2, teniendo en cuenta los efectos de lote y ejecución20. Con el fin de calcular el ICC dentro del proyecto, volvimos a ejecutar el 10% de las muestras que pasaron, asegurándonos de llenar las placas ICC con al menos una muestra de cada placa de la cohorte. El ICC global del proyecto de 0,801 [IC: 0,703, 0,86] indica la alta reproducibilidad de los resultados de TL.
No todos los resultados serán óptimos. Los resultados de la Figura 9 y la Tabla 7 muestran resultados subóptimos del ensayo MMqPCR. La Figura 9 muestra una curva estándar con una eficiencia de los telómeros inferior al 90%, que está por debajo de los estándares de control de calidad, lo que requiere que se repita toda la placa. Los problemas con la eficiencia de los cebadores generalmente se deben a un problema con los reactivos, por lo que es importante realizar un seguimiento de las fechas en que los reactivos se alícuota y cuándo caducan como primer paso para determinar qué reactivo es responsable de la baja eficiencia. Los reactivos caducados deben reemplazarse antes de volver a ejecutar la placa. La Tabla 7 muestra una placa que pasó los criterios iniciales de control de calidad a nivel de muestra, pero tiene un alto nivel de variabilidad entre las placas, lo que lleva a una falla de control de calidad a nivel de placa. La variación entre placas suele deberse a errores en las técnicas de pipeteo y llenado de placas. En este caso, el técnico debe evaluar cualquier problema que haya ocurrido durante la configuración de la placa y asegurarse de que las pipetas estén calibradas.
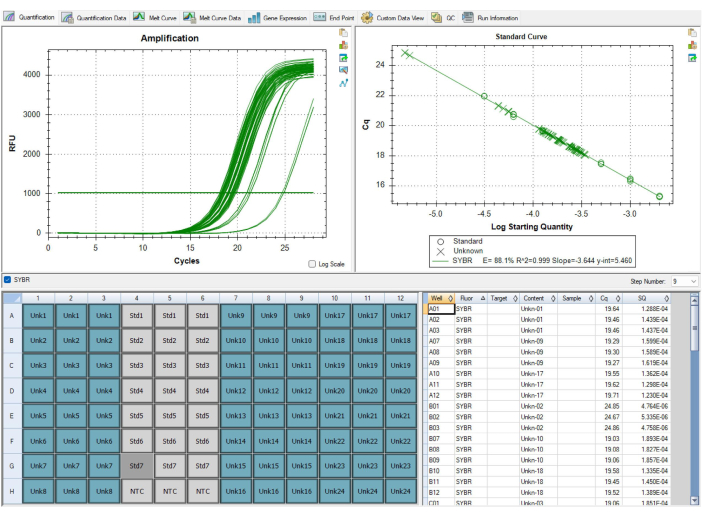
Figura 9: Resultados subóptimos. Esta curva estándar mostrada en el software no logró pasar el control de calidad, ya que la eficiencia de amplificación para el cebador de telómeros fue inferior al 90%. La imagen es de P1, pero P2 tuvo eficiencias igualmente bajas. Los datos de esta ejecución no se pudieron utilizar y todas las muestras tuvieron que volver a ejecutarse después de reemplazar el reactivo causal que había caducado. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 7: Resultados subóptimos. La tabla muestra la plantilla de datos de telómeros para una ejecución en la que muchas de las muestras no cumplieron con los estándares de control de calidad. Las muestras que debían repetirse se determinaron en función de los valores de CV, luego el nombre de la muestra se cambió a una fuente roja para facilitar su identificación. Haga clic aquí para descargar esta tabla.
Legajo Complementario 1: Plantilla de hoja de cálculo de datos de telómeros. Haga clic aquí para descargar este archivo.
Legajo Complementario 2: Cálculo de ICC. Este protocolo ha sido creado por la Red de Investigación de Telómeros (TRN). Este archivo ha sido modificado de20. Haga clic aquí para descargar este archivo.
Legajo Complementario 3: Directrices para la presentación de informes de TRN. Haga clic aquí para descargar este archivo.
Discusión
El método más utilizado en la medición de TL en estudios poblacionales más amplios, antes de 2002, fue el análisis de Southern blot de longitudes de fragmentos de restricción terminales (TRF)24,25. TRF, a pesar de proporcionar una excelente precisión y reproducibilidad en laboratorios especializados, tiene una aplicabilidad limitada debido a la cantidad y calidad del ADN requerido y al rendimiento limitado, lo que proporciona el telón de fondo para una mayor utilización de los ensayos de TL basados en qPCR y, posteriormente, el ensayo MMqPCR. El método MMqPCR TL proporciona una medición de TL repetible cuando se configura, optimiza y mantiene con especial atención a cada criterio de control de calidad. El cálculo y la notificación del ICC específico para cada cohorte analizada son necesarios para garantizar la fiabilidad del ensayo. Si bien está sujeto a la calidad del ADN y a la experiencia técnica, el método MMqPCR es adecuado para grandes estudios poblacionales que investigan la TL porque requiere pequeñas cantidades de ADN, es más confiable que la PCR singleplex y es más eficiente en costos de reactivos y tiempo técnico que otros métodos. La capacidad de generar ICC altos proporciona datos adicionales que respaldan el uso de MMqPCR para estudios poblacionales grandes de LT. La medición de la TL con MMqPCR se puede aplicar a una amplia gama de estudios que buscan definir el papel de la TL como biomarcador de la mortalidad por todas las causas, el envejecimiento, el estrés a lo largo de la vida, las exposiciones ambientales y los resultados de salud física, como las enfermedades cardiovasculares y el cáncer 4,7,8,10,11,12,13,26,27,28, 29,30,31.
Una limitación del método MMqPCR es que informa de TL como una relación T/S, una estimación relativa de la longitud que varía en función de la selección del gen de copia única, la composición de la mezcla maestra y los parámetros de ciclo de PCR32. La relación T/S no tiene unidades. Por lo tanto, sin combinarse con otras metodologías de medición de TL, este método es incapaz de informar estimaciones en valores de par de bases 18,33,34. Como resultado, la relación T/S debe transformarse en una puntuación Z para que tenga relevancia en todos los estudios35. Se debe tener mucho cuidado al hacer esto en laboratorios, métodos y ensayos. Además, este método, al igual que con los ensayos de hibridación basados en gel, puede incluir la cuantificación de los telómeros intersticiales. Sin embargo, estas secuencias comprenden una proporción muy pequeña del contenido total de ADN de los telómeros por genoma. Además, es más probable que las secuencias teloméricas intersticiales contengan secuencias de pares de bases no coincidentes que se desvían de las repeticiones de telómeros canónicas, lo que disminuye la probabilidad de unión y amplificación de cebadores. Además, si bien la cantidad mínima de ADN necesaria para el ensayo MMqPCR es ventajosa, es importante tener en cuenta que las mediciones de TL basadas en qPCR están influenciadas por factores preanalíticos que afectan la calidad e integridad del ADN, incluidas las condiciones de almacenamiento de la muestra, la metodología de extracción de ADN y el tejido biológico36,37. Se ha demostrado que el control analítico de estos factores puede mejorar la validez externa de las mediciones de TL generadas mediante qPCR38. Aun así, el impacto de las diferencias en la calidad del ADN en la TL generadas por el ensayo MMqPCR específicamente debe evaluarse sistemáticamente, ya que no existe una guía actual basada en datos para determinar si una muestra es de calidad suficiente para generar una estimación precisa de la TL utilizando este enfoque. A pesar de estas limitaciones, las aplicaciones de este ensayo para estudios sobre resultados de salud a nivel poblacional son considerables.
Cuando se utiliza el ensayo MMqPCR, se necesita una evaluación continua de la precisión y el rendimiento. Tal como está diseñado actualmente, los técnicos ejecutan triplicados de muestras simultáneamente en placas duplicadas utilizando dos termociclistas. En ausencia de varios termocicladores, recomendamos conservar las mediciones duplicadas por triplicado y las placas de funcionamiento secuencial para mejorar la precisión, incluso a costa de un rendimiento disminuido. Cualquier decisión de priorizar el rendimiento sobre la precisión, por ejemplo, mediante el empleo de mediciones por triplicado único, debe ir acompañada de pruebas y evaluaciones exhaustivas de los ICC resultantes antes de proceder con el análisis de las muestras analíticas. A la hora de tomar decisiones sobre el rendimiento y la precisión, hay que tener en cuenta el tamaño de la muestra y la calidad del ADN de la muestra. Un tamaño de muestra más pequeño o muestras de ADN de mala calidad requieren priorizar una mayor precisión23. Esto es aún más importante cuando se trabaja con muestras de grupos relacionados (por ejemplo, miembros de la familia, sujetos con múltiples puntos de tiempo). En estos casos, una forma de prevenir la pérdida de poder estadístico a través de la confusión inadvertida de grupo por placa es una planificación cuidadosa, por ejemplo, asignando muestras relacionadas a la misma placa antes de comenzar el experimento.
Para este ensayo, se utilizó un espectrofotómetro para evaluar la calidad de la muestra de ADN: las muestras dentro del rango de 1,6-2,0 para relaciones 260/280 y 2,0-2,2 para relaciones 260/230 se consideraron aceptables. Esta evaluación de la calidad y la evaluación precisa del ADN bicatenario a través de un fluorómetro son pasos críticos en este protocolo para obtener datos de TL repetibles. Otras medidas más descriptivas de la integridad del ADN, como el tamaño de los fragmentos y/o las medidas resumidas de la calidad del ADN determinadas mediante gel de agarosa (por ejemplo, el número de integridad del ADN) también pueden utilizarse para determinar la calidad de la muestra38. También recomendamos que la dilución de las muestras solo se produzca en el momento de la preparación de la muestra para realizar el ensayo MMqPCR. Esto garantiza que las alícuotas de ADN se sometan a la menor cantidad posible de manipulación después de la extracción, disminuyendo la variabilidad en el manejo previo al ensayo. En caso de que sea necesario transportar muestras de ADN, deben enviarse en hielo seco a la concentración más alta posible para mitigar la degradación que se produce en las muestras de ADN a concentraciones más bajas39,40. Debido a la degradación del ADN que se produce con los ciclos de congelación y descongelación, el número de congelaciones y descongelaciones para almacenar ADN debe minimizarse41. Las alícuotas del ADN de control agrupado deben crearse antes de ejecutar la cohorte y las alícuotas de las muestras analíticas individuales de ADN deben crearse antes de ejecutar el ensayo.
Las métricas clave del rendimiento del ensayo y el control de calidad incluyen la señal NTC, los CV entre placas e intraplacas, y la curva estándar R2. El incumplimiento de los criterios de control de calidad se puede mitigar de varias maneras. Cambiar regularmente las existencias de PCR de grado H2O y alicuota de las subexistencias de PCR de grado H2O para cada par de placas ejecutadas minimizará las fuentes de contaminación, así como la amplificación de NTC. Los pasos adicionales para reducir la contaminación incluyen los siguientes: designar una cubierta de PCR específica dedicada solo al ensayo MMqPCR; limpiar la campana y el equipo de PCR con una solución descontaminante de ADN; irradiar la habitación con luz ultravioleta; y practicar la técnica estéril cuando se está en la campana de PCR. Para mejorar la repetibilidad del ensayo y disminuir los CV, se recomienda tomar muestras de vórtice, diluciones y las tiras de PCR enérgicamente en sus respectivos pasos de vórtice y resuspender completamente al pipetear muestras de ADN. Lo más probable es que una curva R2 estándar por debajo del umbral (<0,995) se pueda atribuir a errores de pipeteo durante la carga de la placa. Para evitar esto, preste mucha atención al pipeteo preciso y calibre las pipetas anualmente, mezcle enérgicamente las tiras de PCR estándar antes de cargarlas y organice cuidadosamente los suministros para promover un flujo de trabajo eficiente. Si se observa que el uso de dos máquinas y una placa produce CV consistentemente más altos, la máquina debe ser reparada como una forma potencial de mejorar el problema. Las placas de control de calidad del fabricante deben ejecutarse en las placas con regularidad para evaluar el rendimiento del termociclador.
Si los problemas persisten incluso después de la aplicación de los pasos recomendados anteriormente, se pueden usar los siguientes pasos para solucionar problemas del protocolo. Mantener un registro de cuándo se alicaron todos los reactivos y las fechas de caducidad pertinentes puede ayudar a agilizar el proceso de resolución de problemas cuando inevitablemente surjan dificultades. Una parte importante de cualquier proceso de solución de problemas es ajustar solo un reactivo a la vez para determinar la causa específica de las placas que no pasan los criterios de control de calidad, comenzando con el reactivo menos costoso en cuestión. Por ejemplo, si tanto el telómero como el gen de copia única tienen eficiencias bajas, es más probable que los reactivos compartidos, como la TDT, los dNTP o la alícuota SYBR, sean la causa que los cebadores específicos de amplicones. Al precio de cotización, las nuevas alícuotas deben probarse en orden de TDT, luego dNTP y, finalmente, si el problema persiste, nuevas alícuotas SYBR. Por el contrario, si solo uno de los amplicones (telómero o gen de copia única) tiene una eficiencia baja, es más probable que la causa de la dificultad sea uno de los cebadores. La interpretación de los picos de la curva de fusión presentados en la Figura 8 puede servir como fuente de información clave para la resolución de problemas. La visualización de los dos picos de la curva de fusión se puede utilizar para identificar problemas potenciales con una muestra en particular, ya que una muestra de problema individual se destacará de la tendencia general de picos exhibidos por los estándares o las muestras analíticas restantes. La curva de fusión también se puede usar para diagnosticar problemas con un cebador en particular si los picos de un amplicón dado son sistemáticamente menos nítidos que el otro.
Este manuscrito detalla cómo configurar con éxito el ensayo MMqPCR para medir la LT con una amplia aplicabilidad a la investigación en salud pública e introduce recomendaciones clave para el control de calidad y la resolución de problemas con el objetivo de aumentar la accesibilidad y la fiabilidad de este método eficiente y rentable.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores desean agradecer al Comité Asesor de la Red de Investigación de Telómeros y a la financiación del Instituto Nacional sobre el Envejecimiento / Instituto Nacional de Ciencias de la Salud Ambiental (U24 AG066528 y U24 AG066528-S1) que han hecho posible este trabajo.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.5mL Tubes | USA Scientific | 1605-0099 | Seal-Rite 0.5mL Microcentrifuge Tubes, Sterile Storage Temperature and Conditions: Room temperature |
| 1.5mL Tubes | USA Scientific | 1615-5599 | Seal-Rite 1.5mL Microcentrifuge Tubes, Sterile Storage Temperature and Conditions: Room temperature |
| 100mM DTT | In House | Not Applicable | Made with stock DTT, diluted sodium acetate, and PCR Grade H2O Storage Temperature and Conditions: minus 20 °C |
| 15mL Tubes | Thermo Fisher | 14-959-53A | Corning 352196 Falcon 15mL Conical Centrifuge Tubes Storage Temperature and Conditions: Room temperature |
| 1M MgCl2 | Thermo Fisher | 50152107 | Biotang Inc 1M MgCl2 1M Magnesium Chloride Solution, Prepared in 18.2 Megohms Water and Filtered through 0.22 Micron Filter Storage Temperature and Conditions: 4 °C |
| 1x Gold Buffer | In House | Not Applicable | 10X Gold Buffer diluted with PCR Grade H2O Storage Temperature and Conditions: minus 20 °C |
| 25mM dNTPs | New England BioLabs | N0446S | Deoxynucleotide Solution Set Storage Temperature and Conditions: minus 20 °C |
| 5mL Tubes | Thermo Fisher | 3391276 | Argos Technologies Microcentrifuge Tubes – 5mL Storage Temperature and Conditions: Room temperature |
| 96 Well Plate | Bio-Rad | HSP9601 | Hard-Shell 96-Well PCR Plates, Low Profile, Thin Wall, Skirted, White / Clear Storage Temperature and Conditions: Room temperature |
| Aluminum Foil | Office Depot | 3489072 | Reynolds Wrap Stanard Aluminum Foil Roll, 12" x 75', Silver Storage Temperature and Conditions: Room temperature |
| AmpliTaq Gold Kit – Polymerase and Buffer | Thermo Fisher | 4311806 | AmpliTaq Gold DNA Polymerase with Gold Buffer and MgCl2 (MgCl2 in this kit is not used), 10X Gold Buffer, 2.5U AmpliTaq Gold Polymerase Storage Temperature and Conditions: minus 20 °C |
| Betaine | Thermo Fisher | AAJ77507AB | Betaine, 5M Solution, Molecular Biology Grade, Ultrapure, 10mL Storage Temperature and Conditions: minus 20 °C |
| Big Tube Rack | Thermo Fisher | 344817 | Fisherbrand 4-Way Tube Rack Storage Temperature and Conditions: Room temperature |
| CFX Maestro Software | Bio-Rad | 12004110 | Software for real-time PCR plate setup, data collection, statistics, and graphiing of results Storage Temperature and Conditions: Room temperature |
| CFX96 Optical Reaction Module for Real-Time PCR Systems with Starter Package | Bio-Rad | 1845096 | 96-well optical module for real-time PCR Storage Temperature and Conditions: Room temperature |
| DTT | Fisher Scientific | AAJ1539706 | Dithiothreitol, >99.5+ Molecular Biology Grade, 5 g Storage Temperature and Conditions: minus 20 °C |
| ELIMINase | Fisher Scientific | 04-355-32 | ELIMINase Laboratory Decontaminant Storage Temperature and Conditions: Room temperature |
| HEPA Filter | USA Scientific | Replacement Filters | High-Efficiency Particulate Air Filter for AirClean Workstations Storage Temperature and Conditions: Room temperature |
| Kimwipes | Thermo Fisher | 06666A | Kimberly-Clark Professional Kimtech Science Kimwipes Delicate Task Wipers, 1-Ply Storage Temperature and Conditions: Room temperature |
| Loading Trough | Thermo Fisher | 14387069 | Thermo Scientific Matrix Reagent Reservoirs Storage Temperature and Conditions: Room temperature |
| Microsoft Excel | Microsoft | Not Applicable | Microsoft 365 package, Excel software application Storage Temperature and Conditions: Room temperature |
| Mini Centrifuge | Genesee Scientific | 31-500B | Poseidon 31-500B Mini Centrifuge, Blue Lid Storage Temperature and Conditions: Room temperature |
| PCR Grade H2O | Thermo Fisher | AM9937 | Nuclease-Free Water (not DEPC-Treated) Storage Temperature and Conditions: Room temperature |
| PCR Hood | USA Scientific | 4263-2588 | Nucleic Acid Workstation with HEPA Filtration, AirClean Systems Combination PCR Workstation Storage Temperature and Conditions: Room temperature |
| PCR Strips | Thermo Fisher | AB0776 | Low Profile Tubes and Flat Caps, Strips of 8 Storage Temperature and Conditions: Room temperature |
| PCR Tube Rack | Thermo Fisher | 344820 | Fisherbrand 96-Well PCR Tube Rack Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Multichannel) | Ranin | 17005860 | Pipette Tips SR LTS 20µL F 960A/5, 20µL Maximum Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1181-3850 | 10µL Graduated TipOne RPT Filter Tips Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1180-1850 | 20µL Beveled TipOne RPT Filter Tips Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1111-0880 | 200µL Natural TipOne Pipette Tips in Racks Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1111-2890 | 1000µL Natural Graduated TipOne Pipette Tips in Racks Storage Temperature and Conditions: Room temperature |
| Pipettors (Multichannel) | Ranin | 17013802 | Pipet-Lite Multi Pipette L8-10XLS, 0.5 to 10µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Multichannel) | Ranin | 17013803 | Pipet-Lite Multi Pipette L8-20LS+, 2 to 20µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F144802G | Gilson Pipetman Classic Pipets, 1 to 10µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F123600 | Gilson Pipetman Classic Pipets, 2 to 20µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F123601 | Gilson Pipetman Classic Pipets, 20 to 200µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F123602 | Gilson Pipetman Classic Pipets, 200 to 1000µL Storage Temperature and Conditions: Room temperature |
| Plate Sealing Film | Bio-Rad | MSB1001 | Microseal “B” PCR Plate Sealing Film, Adhesive, Optical Storage Temperature and Conditions: Room temperature |
| Plate Spinner | Thermo Fisher | 14-100-141 | Fisherbrand Mini Plate Spinner Centrifuge, 230 V Storage Temperature and Conditions: Room temperature |
| Pre-Hood Filter | USA Scientific | 4235-3724 | Prefilter for AirClean Systems Workstations Storage Temperature and Conditions: Room temperature |
| R Software | The R Project for Statistical Computing | Not Applicable | R version 4.2.2 Storage Temperature and Conditions: Room temperature |
| Scale | Thermo Fisher | 01-922-329 | OHAUS 30430060 PR Series Analytical Balance, 62g Capacity Storage Temperature and Conditions: Room temperature |
| Scissors | Office Depot | 458612 | Office Depot Brand Scissors, 8”, Straight, Black, Pack of 2 Storage Temperature and Conditions: Room temperature |
| Sharpies | Sharpie | 2151734 | Brush Twin Permanent Markers, Black Storage Temperature and Conditions: Room temperature |
| Single Copy Gene Forward Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Single Copy Gene Reverse Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Small Tube Rack | Thermo Fisher | 21-402-17 | Thermo Fisher 8601 Reversible Microtube Racks with Lid Storage Temperature and Conditions: Room temperature |
| Sodium Acetate | Thermo Fisher | J63560.EQE | 3M NaOAc pH 5.2 Storage Temperature and Conditions: Room temperature |
| Stainless Steel Spatula | Thermo Fisher | 3990240 | Bel-Art SP Scienceware Stainless-Steel Sampling Spoon and Spatula Storage Temperature and Conditions: Room temperature |
| SYBR Green | Thermo Fisher | S7563 | SYBR Green I Nucleic Acid Gel Stain – 10,000X Concentrate in DMSO Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Syringe Filters | Fisher Scientific | 09-927-55A | GD/X 25 mm Sterile Syringe Filter, cellulose acetate filtration medium, 0.2 μm Storage Temperature and Conditions: Room temperature |
| Syringes | Thermo Fisher | 148232A | BD Luer-Lok Disposable Syringes without Needles, 10mL Storage Temperature and Conditions: Room temperature |
| TE Buffer | Fisher Scientific | BP2474100 | TE Buffer, Tris-EDTA, 1X Solution, pH 7.6, Molecular Biology, Fisher BioReagents Storage Temperature and Conditions: Room temperature |
| Telomere Forward Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Telomere Reverse Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| UV Light | USA Scientific | 4288-2540 | UV Light Bulb for Workstations Storage Temperature and Conditions: Room temperature |
| Vortex | Thermo Fisher | 14-955-151 | Fisherbrand Mini Vortex Mixer, 115 V, 50/60 Hz Storage Temperature and Conditions: Room temperature |
| Weigh Boat | Thermo Fisher | 01-549-752 | Fisherbrand Sterile Hexagonal Weighing Boat, 10mL Storage Temperature and Conditions: Room temperature |
Referencias
- Hemann, M., Strong, M., Hao, L., Greider, C. The shortest telomere, not average telomere length, is critical for cell viability and chromosome stability. Cell. 107 (1), 67-77 (2001).
- Xu, L., Li, S., Stohr, B. A. The role of telomere biology in cancer. Ann Rev Pathol: Mech Dis. 8, 49-78 (2013).
- Lindrose, A., Drury, S. Minimum reporting recommendations for pcr-based telomere length measurement. Telomere Research Network. , (2020).
- Verhulst, S., et al. Commentary: The reliability of telomere length measurements. Int J Epidemiol. 44 (5), 1683-1686 (2015).
- Aubert, G., Hills, M., Lansdorp, P. M. Telomere length measurement-caveats and a critical assessment of the available technologies and tools. Mutat Res. 730 (1-2), 59-67 (2012).
- Mundstock, E., et al. Effect of obesity on telomere length: Systematic review and meta-analysis. Obesity. 23 (11), 2165-2174 (2015).
- Haycock, P. C., et al. Leucocyte telomere length and risk of cardiovascular disease: Systematic review and meta-analysis. BMJ. 349, 4227 (2014).
- Astuti, Y., Wardhana, A., Watkins, J., Wulaningsih, W. Cigarette smoking and telomere length: A systematic review of 84 studies and meta-analysis. Environ Res. 158, 480-489 (2017).
- Ridout, K. K., Ridout, S. J., Price, L. H., Sen, S., Tyrka, A. R. Depression and telomere length: A meta-analysis. J Affect Disord. 191, 237-247 (2016).
- Wang, Q., Zhan, Y., Pedersen, N. L., Fang, F., Hägg, S. Telomere length and all-cause mortality: A meta-analysis. Ageing Res Rev. 48, 11-20 (2018).
- Hu, R., Hua, X. G., Jiang, Q. C. Associations of telomere length in risk and recurrence of prostate cancer: A meta-analysis. Andrologia. 51 (7), e13304 (2019).
- Schneider, C. V., et al. Association of telomere length with risk of disease and mortality. JAMA Inter Med. 182 (3), 291-300 (2022).
- Deng, Y., et al. Telomere length and the risk of cardiovascular diseases: A mendelian randomization study. Front Cardiovasc Med. 9, 1012615 (2022).
- D'mello, M. J., et al. Association between shortened leukocyte telomere length and cardiometabolic outcomes: Systematic review and meta-analysis. Circulation: Cardiovasc Gene. 8 (1), 82-90 (2015).
- Wilbourn, R. V., et al. The relationship between telomere length and mortality risk in non-model vertebrate systems: A meta-analysis. Philos Trans R Soc Lond B Biol Sci. 373 (1741), 20160447 (2018).
- Gardner, M., et al. Gender and telomere length: Systematic review and meta-analysis. Exp Gerontol. 51, 15-27 (2014).
- Lai, T. -. P., Wright, W. E., Shay, J. W. Comparison of telomere length measurement methods. Philos Trans R Soc Lond B Biol Sci. 373 (1741), 20160451 (2018).
- Cawthon, R. Telomere measurement by quatitative pcr. Nuc Acid Res. 30 (10), e47-e53 (2002).
- Cawthon, R. M. Telomere length measurement by a novel monochrome multiplex quantitative pcr method. Nuc Acid Res. 37 (3), e21-e21 (2009).
- Lindrose, A. R., et al. Method comparison studies of telomere length measurement using qpcr approaches: A critical appraisal of the literature. PLoS One. 16 (1), e0245582 (2021).
- Lin, J., et al. Effects of DNA extraction, DNA integrity, and laboratory on the precision of qpcr-based telomere length measurement - a multi-lab impartial study. bioRxiv. , (2022).
- Wilfinger, W. W., Mackey, K., Chomczynski, P. Effect of ph and ionic strength on the spectrophotometric assessment of nucleic acid purity. Biotechniques. 22 (3), 474-481 (1997).
- Boesenberg-Smith, K. A., Pessarakli, M. M., Wolk, D. M. Assessment of DNA yield and purity: An overlooked detail of pcr troubleshooting. Clin Microbiol Newsletter. 34 (1), 1-6 (2012).
- Harley, C. B., Futcher, A. B., Greider, C. W. Telomeres shorten during ageing of human fibroblasts. Nature. 345 (6274), 458-460 (1990).
- Kimura, M., et al. Measurement of telomere length by the southern blot analysis of terminal restriction fragment lengths. Nat Protoc. 5 (9), 1596-1607 (2010).
- Ye, Q., et al. Telomere length and chronological age across the human lifespan: A systematic review and meta-analysis of 414 study samples including 743,019 individuals. Ageing Res Rev. 90, 102031 (2023).
- Axelrad, M. D., Budagov, T., Atzmon, G. Telomere length and telomerase activity; a yin and yang of cell senescence. J Vis Exp. (75), e50246 (2013).
- Liu, M., et al. Immune-mediated inflammatory diseases and leukocyte telomere length: A mendelian randomization study. Front Genetics. 14, 1129247 (2023).
- Van Ockenburg, S., et al. Stressful life events and leukocyte telomere attrition in adulthood: A prospective population-based cohort study. Psychol Med. 45 (14), 2975-2984 (2015).
- Zong, Z. Q., et al. Ambient air pollution exposure and telomere length: A systematic review and meta-analysis. Public Health. 215, 42-55 (2023).
- Tang, L., Li, D., Wang, J., Su, B., Tian, Y. Ambient air pollution, genetic risk and telomere length in uk biobank. J Expo Sci Environ Epidemiol. , (2023).
- Aubert, G., Hills, M., Lansdorp, P. Telomere length measurement-caveats and a critical assessment of the available technologies and tools. Mutat Res. 730 (1), 59-67 (2012).
- Lin, J., et al. Systematic and cell type-specific telomere length changes in subsets of lymphocytes. J Immunol Res. 2016, 5371050 (2016).
- Needham, B. L., et al. Socioeconomic status, health behavior, and leukocyte telomere length in the national health and nutrition examination survey, 1999-2002. Soc Sci Med. 85, 1-8 (2013).
- Verhulst, S. Improving comparability between qpcr-based telomere studies. Mol Ecol Res. 20 (1), 11-13 (2020).
- Dagnall, C. L., et al. Effect of pre-analytic variables on the reproducibility of qpcr relative telomere length measurement. PloS one. 12 (9), e0184098 (2017).
- Lin, J., Smith, D. L., Esteves, K., Drury, S. Telomere length measurement by qpcr-summary of critical factors and recommendations for assay design. Psychoneuroendocrinology. 99, 271-278 (2019).
- Wolf, S. E., et al. Cross-tissue comparison of telomere length and quality metrics of DNA among individuals aged 8 to 70 years. PLOS One. 19 (2), e0290918 (2024).
- RoDer, B., FruHwirth, K., Vogl, C., Wagner, M., Rossmanith, P. Impact of long-term storage on stability of standard DNA for nucleic acid-based methods. J Clin Microbiol. 48 (11), 4260-4262 (2010).
- Dagnall, C., et al. Effect of pre-analytic variables on the reproducibility of qpcr relative telomere length measurement. PLoS One. 12 (9), e0184098 (2017).
- Shao, W., Khin, S., Kopp, W. C. Characterization of effect of repeated freeze and thaw cycles on stability of genomic DNA using pulsed field gel electrophoresis. Biopreserv Biobank. 10 (1), 4-11 (2012).
- Institute for Statistics and Mathematics of WU. The Comprehensive R Archive Network Available from: https://cran.r-project.org/ (2024)
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biol Rev. 85 (4), 935-956 (2010).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados