Method Article
Monochrome Multiplex Quantitative PCR 텔로미어 길이 측정
* 이 저자들은 동등하게 기여했습니다
요약
여기에서는 MMqPCR(monochrome multiplex quantitative polymerase chain reaction) 분석을 사용하여 상대 텔로미어 길이(TL)를 측정하기 위한 프로토콜을 제시합니다. MMqPCR 분석은 인구 기반 연구에서 인간 DNA의 TL을 측정하기 위한 반복 가능하고 효율적이며 비용 효율적인 방법입니다.
초록
텔로미어는 모든 진핵 염색체의 끝에 있는 리보핵단백질 구조로, DNA가 손상되지 않도록 보호하고 염색체 안정성을 보존합니다. 텔로미어 길이(TL)는 다양한 노출, 생물학적 과정 및 건강 결과와 관련이 있습니다. 이 기사에서는 인간 DNA의 상대 평균 TL을 측정하기 위해 당사 실험실에서 일상적으로 수행되는 단색 다중 정량 중합효소 연쇄 반응(MMqPCR) 분석 프로토콜에 대해 설명합니다. 여러 가지 다른 PCR 기반 TL 측정 방법이 있지만, 이 간행물에 제시된 MMqPCR 방법에 대한 특정 프로토콜은 반복 가능하고 효율적이며 비용 효율적이며 인구 기반 연구에 적합합니다. 이 상세한 프로토콜은 연구자가 실험실에서 이 분석을 확립하는 데 필요한 모든 정보를 간략하게 설명합니다. 또한 이 프로토콜은 동일한 샘플의 반복 측정에서 클래스 내 상관 계수(ICC)로 정의되는 이 분석을 통해 TL 측정의 재현성을 높이기 위한 특정 단계를 제공합니다. ICC는 특정 연구 모집단에 대한 기대 전력을 평가하는 데 중요한 요소입니다. 따라서 모든 TL 분석에 대해 코호트별 ICC를 보고하는 것은 TL에 대한 인구 기반 연구의 전반적인 엄격성을 향상시키는 데 필요한 단계입니다. 말초 혈액 단핵 세포에서 추출한 DNA 샘플을 활용한 예시 결과는 이 MMqPCR 프로토콜을 사용하여 반복성이 높은 TL 데이터를 생성할 수 있음을 보여줍니다.
서문
텔로미어는 모든 진핵 염색체의 끝에서 발견되는 보호 복합체로, 고도로 보존되고 반복적인 DNA 염기서열 및 관련 단백질로 구성됩니다. 텔로미어는 DNA의 무결성을 보호하여 염색체 안정성을 보존합니다. 텔로미어의 점진적인 단축은 불완전한 지연 가닥 DNA 합성, DNA 손상 및 기타 요인의 결과로 세포 분열에서 발생합니다 1,2. 인간의 수명에 걸친 노화 및 노화 관련 질병의 바이오마커로서 텔로미어 길이(TL)를 뒷받침하는 증거가 증가함에 따라 인체 노출, 질병 및 건강 연구에서 TL의 역할을 평가하는 데 사용되는 TL 측정 분석 유형이 증가했습니다 3,4,5. 메타 분석에서는 암, 심혈관 질환 및 당뇨병을 포함한 전체 사망률, 환경 노출 및 건강 결과와 TL의 연관성을 보고했습니다 6,7,8,9,10,11,12,13 . 이러한 메타-연관성은 20개 이상의 서로 다른 TL 측정 방법론 중 하나를 사용하는 연구에서 파생되며, 연관성의 강도는 서로 다른 방법론 간에 다른 경향이 있습니다 14,15,16. 연구 연구를 위한 최적의 TL 측정 방법을 선택하는 것은 각 방법마다 고유한 장점과 단점이 있기 때문에 정확한 결과를 보장하기 위한 중요한 단계입니다 5,17.
상대적으로 저렴한 시약 비용, 빠른 분석 턴어라운드, 확장성 및 낮은 초기 DNA 요구 사항으로 인해 PCR 기반 TL 측정 기술은 대규모 시료 집단을 대상으로 한 연구, 높은 DNA 농도의 시료에 대한 접근이 제한된 연구 또는 높은 처리량을 우선시하는 연구를 수행할 때 우선적으로 활용되는 경우가 많습니다. TL 측정의 첫 번째 PCR 기반 방법인 단일 정량 중합효소 연쇄 반응(qPCR)은 원래 Richard Cawthon에 의해 개발되었으며, 텔로미어(T) 및 단일 복제 유전자(S) 증폭의 형광 신호의 비율을 활용하며, 별도의 PCR 플레이트18에서 실행됩니다. 이 접근법에서는 반복되는 텔로미어 DNA 염기서열(T)을 보완하는 프라이머를 사용하여 샘플의 총 텔로머 DNA 함량을 증폭하고 형광 리포터 SYBR green을 검출하여 정량화합니다. 유사하게, 보존된 단일 복제(S) 유전자의 유전자 간 영역에 상보적인 프라이머는 게놈 복제 수를 정량화하는 데 사용됩니다. 이 두 추정치는 플레이트 간 변동을 제어하기 위한 프로젝트의 모든 분석에서 사용되는 게놈 DNA 표준 곡선을 기준으로 정량화됩니다. 총 텔로머 DNA(T)를 단일 게놈 복제 수(S)로 나누면 개별 DNA 샘플에 대한 세포당 평균 텔로머 함량을 나타내는 단위 없는 상대 측정치인 T/S 비율이 생성됩니다(18,19). 따라서 T/S 비율은 기능적 길이의 특정 측정이 아닙니다. 그러나 문헌 규범에 따라 이 프로토콜 전체에서 샘플당 평균 TL이라는 용어를 사용합니다.
이 방법은 2009년 Richard Cawthon이 원래의 singleplex qPCR방법 19에 비해 T/S 비율의 변동성을 잠재적으로 줄이기 위한 접근 방식으로 단색 다중 다중 qPCR(MMqPCR) 분석을 설명하면서 발전했습니다. MMqPCR 분석은 qPCR 분석의 장점과 함께 하나의 보고 형광단을 사용하여 동일한 반응 웰 내에서 T 및 S 신호를 측정할 수 있는 추가적인 이점을 가지고 있으며, 이를 통해 singleplex qPCR에 비해 오류를 줄이고 정밀도와 재현성을 높일 수 있습니다19. 또한, 이 멀티플렉스 분석법은 singleplex assay19에 비해 절반의 반응이 필요하기 때문에 잠재적으로 비용을 절감하고 처리량을 향상시킬 수 있습니다.
MMqPCR TL 측정의 장점을 감안할 때 이 방법은 노출, 건강 결과 및 생물학적 과정과의 TL 연관성에 대한 인구 기반 연구에 매우 적합합니다. 그러나 방법을 시작하는 것은 어려울 수 있습니다. 이러한 문제를 해결하기 위해 실험실에서 사용되는 MMqPCR TL 측정 프로토콜을 자세히 설명하고 분석 정밀도를 높이고 오염 위험을 줄이며 반복성을 향상시키기 위해 구현된 주요 단계를 강조합니다.
또한 이 프로토콜은 데이터를 정리하고 TL 측정2의 재현성에 대한 중요한 통계적 척도인 ICC(intraclass correlation coefficient)를 계산하는 단계를 간략하게 설명합니다. 우리의 대표적인 결과를 통해 이 프로토콜을 사용하여 높은 ICC를 생성할 수 있는 능력을 보여줍니다. 또한 TL 측정의 변동을 줄이고 결과 ICC를 증가시킬 것으로 예상되는 품질 관리(QC) 및 문제 해결 단계를 식별합니다. 이 방법의 높은 반복성, 효율성 및 비용 효율성으로 인해 MMqPCR TL 측정은 역학 TL 연구에 이상적입니다. 그림 1은 이 프로토콜에 설명된 MMqPCR 방법의 시각적 개요를 제공합니다.
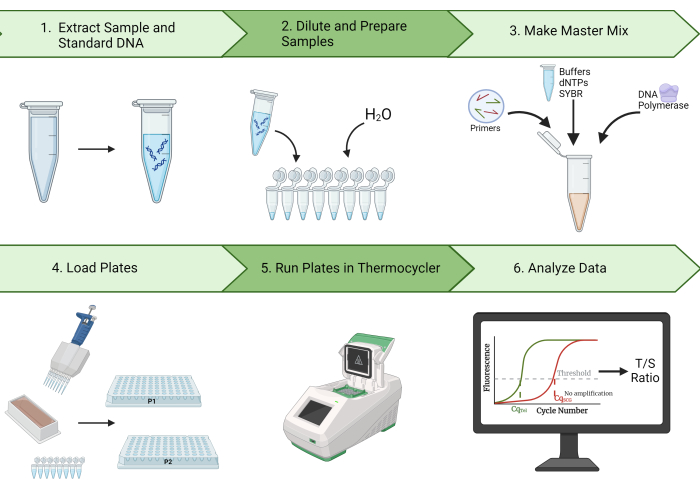
그림 1: 분석법 개요. 텔로미어 길이를 측정하기 위한 monochrome multiplex quantitative polymerase chain reaction method에 대한 광범위한 개요. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
프로토콜
이 연구는 기관 지침에 따라 수행되었습니다. 이 프로토콜은 중복 삼중의 측정 깊이, 즉 중복 플레이트에서 반복되는 각 샘플의 삼중 측정에서 수행된 MMqPCR 분석을 설명하며, 처리량을 향상시키기 위해 두 개의 열순환기를 동시에 사용하여 구현됩니다. 중복 플레이트의 사용은 높은 ICC에서 알 수 있듯이 높은 재현성을 달성하기 위해 이 프로토콜을 구현할 때 가장 먼저 고려해야 할 사항입니다. 비록 더 적은 반복실험을 사용할 수 있지만, ICC에 미치는 영향, 그에 따른 검정력 및 필요한 표본 크기는 각 실험실의 모든 코호트와 함께 신중하게 고려하고 측정해야 합니다20,21. Thermocycler를 하나만 사용할 수 있는 경우 측정 깊이를 유지하고(즉, 중복 삼중) 2개의 플레이트를 순차적으로 실행하는 것이 좋습니다.
1. 재고 준비 및 보관 조건
- 이 프로토콜에 사용되는 모든 재료 및 장비에 대해서는 재료 표를 참조하십시오. 또한, MMqPCR 분석에 사용되는 특정 마스터 믹스 시약 및 양에 대해서는 표 1을 참조하십시오. 두 개의 플레이트를 순차적으로 실행하는 경우 농도를 유지하면서 마스터 믹스 부피의 절반을 사용하여 동일한 분석의 순차 플레이트에 동일한 시약 부분 표본이 사용되도록 합니다.
알림: 모든 스톡 시약은 미리 준비해야 하며 분취된 시약은 사용하기 전에 완전히 해동해야 합니다. - 1x Tris-EDTA, pH 7.6(TE)을 5mL 튜브에 담아 3mL 용량의 3mL 용량으로 최대 2년 동안 분취 및 보관합니다.
- -20°C에서 1,280μL 용량의 5mL 튜브에 담긴 5M 베타인을 최대 1년 동안 분취 및 보관합니다.
- -20°C에서 최대 2년 동안 개별 PCR 스트립 튜브에 3μL 용량의 SYBR Green을 분취하여 보관하고 호일로 덮고 빛에 최소한으로 노출시킵니다.
- DNA 중합효소를 -20°C의 원래 튜브에 최대 1년 동안 보관합니다.
- -20°C에서 1.5 mL 용량의 1.5 mL tube에 담긴 10x polymerase buffer를 최대 1년 동안 분취 및 보관합니다.
- 10x 중합효소 완충액의 1x 완충액 희석의 경우, 0.5mL 튜브에서 9.9μL의 10x 완충액을 PCR 등급 H2O 89.1μL에 추가합니다. -20 °C에서 최대 1년 동안 보관하십시오.
- 0.5 mL 튜브에 담긴 1 M MgCl2 를 4 °C에서 빛으로부터 떨어진 70 μL 용량의 분취하여 최대 1년 동안 보관할 수 있습니다.
- 정방향 및 역방향 텔로미어(T) 및 단일(S) 복제 유전자 올리고뉴클레오티드 동결건조 프라이머를 빛으로부터 멀리 떨어진 실온에 보관합니다. 4개의 특정 올리고뉴클레오티드 프라이머 서열이 표 2에 제시되어 있습니다.
참고: 이 프로토콜의 단일 복제 유전자는 알부민입니다. 다른 단일 복제 유전자를 선택하는 경우, 허용 가능한 PCR 효율을 보장하기 위해 프라이머 농도를 조정해야 할 수 있습니다.- 프라이머 재구성: 각 동결건조된 프라이머 튜브를 10초 동안 소용돌이친 다음 튜브를 미니 원심분리기에 5초 동안 넣어 1x TE 버퍼를 추가하기 전에 동결건조된 펠릿이 튜브 바닥에 있는지 확인합니다.
- 각 개별 프라이머 튜브에서 nM 농도([nM]= 알파)를 확인하고 알파 값(μL)의 10배에 해당하는 1x TE를 추가하여 각 프라이머 튜브를 재수화하여(예: 튜브의 nM 농도가 24.6nM인 경우 프라이머 튜브에 246μL의 1x TE를 추가) 각 프라이머의 100μM 용액을 생성합니다.
- 전방 및 역방향 텔로미어 프라이머는 16 μL 부피로 따로 추출하고 정방향 및 역방향 알부민 프라이머는 11 μL 부피로 별도로 추출합니다. 4가지 유형의 프라이머 부분 표본을 모두 -20°C에서 최대 6개월 동안 보관하십시오.
- dNTP 혼합물: 25mM 원액의 4가지 dNTPS 유형 각각(즉, 총 16-32개의 튜브)의 4-8개 튜브를 10초 동안 소용돌이친 다음 미니 원심분리기를 사용하여 실온에서 빠르게 스핀다운합니다. 튜브를 미니 원심분리기의 튜브 위치에 균일한 분포로 배치한 다음 뚜껑을 닫아 기계가 최대 속도에 도달하도록 약 5초 동안 회전합니다.
- 5mL 튜브에 각 튜브에서 200-250μL를 추가하여 4가지 dNTP 유형 각각의 동일한 부분이 추가되도록 하고 dNTP 혼합물을 소용돌이칩니다.
- 0.5 mL 튜브에 210 μL의 혼합물을 분취하고 -20 °C에서 최대 1년 동안 보관합니다.
- 재고 DTT는 -20 °C에서 보관하고 3M 아세트산 나트륨은 실온에서 보관하십시오.
- DTT 솔루션: 스테인리스강 주걱을 사용하여 가늘게 진 계량 보트에서 0.1545g DTT를 측정합니다.
주의: 디티오트레이톨(DTT)은 위험한 시약입니다. DTT는 삼키면 해롭고, 피부 자극을 일으키며, 눈에 심각한 손상을 줄 수 있습니다. 개인은 DTT를 다룰 때 보호 장갑, 보안경 및 안면 보호구를 착용해야 합니다. 또한 개인은 DTT 증기를 흡입하지 않아야 하고, DTT를 다룰 때 PCR 후드에서 작업해야 하며, 장기간 또는 반복적인 노출을 피해야 합니다. - 15mL 튜브와 와류 웰에 33.33 μL의 3 M 아세트산 나트륨과 9,967 μL의 PCR 등급 H2O를 첨가하여 10 mL의 0.01 M 아세트산 나트륨을 만듭니다. 측정된 DTT를 0.01M 아세트산나트륨이 든 15mL 튜브에 넣습니다. 약 100μL의 0.01M 아세트산나트륨 용액을 계량 보트에 전달하여 잔류 DTT를 수집합니다. 100 μL를 15 mL 튜브에 다시 피펫팅합니다. 모든 DTT가 용액에 들어가면 완전히 용해될 때까지 튜브를 소용돌이칩니다.
- 15mL 튜브의 내용물을 로딩 트로프에 붓고 로딩 트로프의 한쪽 끝에서 전체 용액을 10mL 플라스틱 주사기로 흡입합니다. 제균 0.2μm 셀룰로오스 아세테이트 주사기 필터를 주입된 주사기 끝에 25mm 부착하고 1분 동안 완전히 포화시킨 다음 주사기 플런저를 가볍게 밀어 용액을 새 15mL 튜브에 천천히 떨어뜨려 전체 용액이 필터를 통해 새 15mL 튜브로 떨어지도록 합니다.
- DTT 용액을 200μL 용량으로 0.5mL 튜브에 분취하고 -20°C에서 최대 6주 동안 보관합니다.
- DTT 솔루션: 스테인리스강 주걱을 사용하여 가늘게 진 계량 보트에서 0.1545g DTT를 측정합니다.
표 1: 시약의 최종 부피 및 농도. 개별 부분 표본, 마스터 믹스 및 PCR 웰에 있는 시약의 부피 및 농도. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 2: 텔로미어 및 단일 복제 유전자 올리고뉴클레오티드 프라이머 염기서열. 방법론에 사용된 텔로미어 및 알부민 단일 복제 유전자 프라이머 염기서열 목록. 이 표를 다운로드하려면 여기를 클릭하십시오.
2. 게놈 DNA 추출 및 시료 전처리
- 분석 시료 추출: 키트 또는 실험실 내에서 확립된 방법을 사용하여 제조업체의 지침에 따라 시료에 대한 게놈 DNA 추출을 수행합니다.
- 형광계를 사용하여 분광광도계와 이중 가닥 DNA(dsDNA)로 DNA 샘플 품질을 확인합니다. 평균 260/280 및 260/230 비율이 허용되지 않거나 dsDNA 농도가 검출 미만(<0.20ng/μL)인 샘플은 TL22,23에 대해 분석해서는 안 됩니다. 또한 dsDNA 농도가 너무 높아 형광측정기(>1000ng/μL)를 사용하여 판독값을 생성할 수 없는 경우 샘플을 희석하고 분석을 반복합니다.
참고: 여기에 제시된 결과의 경우, 핵산의 순도에 대한 허용 가능한 값은 1.6 - 2.0 사이의 260/280 비율과 2.0 - 2.2 사이의 260/230 비율이었습니다.
- 형광계를 사용하여 분광광도계와 이중 가닥 DNA(dsDNA)로 DNA 샘플 품질을 확인합니다. 평균 260/280 및 260/230 비율이 허용되지 않거나 dsDNA 농도가 검출 미만(<0.20ng/μL)인 샘플은 TL22,23에 대해 분석해서는 안 됩니다. 또한 dsDNA 농도가 너무 높아 형광측정기(>1000ng/μL)를 사용하여 판독값을 생성할 수 없는 경우 샘플을 희석하고 분석을 반복합니다.
- 대조군 시료 추출: 주어진 프로젝트에 대해 분석되는 대상 시료와 동일한 생물학적 물질에서 파생된 대조군 시료에 대해 게놈 DNA 추출을 수행합니다. 모든 분석에 단일 DNA 표준물질이 활용될 수 있도록 전체 코호트에 대해 실행될 것으로 예상되는 모든 플레이트에 대해 충분한 대조군 DNA를 추출해야 합니다.
- 분광광도계와 형광측정기로 대조군 DNA 시료 품질을 확인합니다. 대조군 샘플은 허용 가능한 평균 260/280 및 260/230 비율과 dsDNA22,23의 검출 가능한 농도를 가져야 합니다.
- 0.5mL 튜브에 150μL 양으로 2ng/μL 농도의 분취 대조 DNA를 넣고 시료 유형 및 날짜로 라벨링합니다. 대조군 DNA 부분 표본을 -20 °C에서 최대 5년 동안 보관하십시오. 동일한 코호트 또는 연구 프로젝트의 모든 샘플의 모든 플레이트에 대해 하나의 대조군 DNA 샘플을 사용합니다. 이러한 재고 부분 표본을 미리 준비하십시오.
- 분석 시료 준비: 시작 농도가 >5ng/μL인 시료의 경우 정수 희석 인자를 사용하여 희석 분취액을 생성하고 추출된 DNA 시료(보충 파일 1 MMqPCR 설정 시트, F열) 및 PCR 등급 H2O(보충 파일 1 MMqPCR 설정 시트, 열 G)를 추가하여 농도를 2.5 - 5.0 ng/μL 사이로 희석합니다(보충 파일 1 MMqPCR 설정 시트, 열 K). <5ng/μL 농도의 시료를 희석하지 말고, 이 프로토콜을 사용하여 시료를 3배 분석할 수 있을 만큼 충분한 부피를 분주하십시오.
참고: 각 샘플의 해당 PCR 스트립 튜브에 15ng의 DNA를 추가할 수 있도록 샘플 준비 중에 희석 부분 표본을 만들어야 합니다. 시료 부분 표본을 통해 기술자는 시료가 품질 기준을 통과하지 못해 재실행해야 하는 경우 불필요한 동결-해동 주기가 스톡 시료 DNA 무결성에 영향을 미치는 것을 방지할 수 있습니다. 보충 파일 1에 있는 샘플 MMqPCR 템플릿은 희석 부분 표본을 준비하기 위한 올바른 희석 계수 및 부피를 식별하는 데 도움이 될 수 있습니다(보충 파일 1, MMqPCR 설정 시트, 컬럼 E-I).- 보충 파일 1 MMqPCR 셋업 시트에 자세히 설명되어 있고 표 3에 나열된 대로 PCR 스트립 A의 샘플 A1-8, PCR 스트립 B의 샘플 B1-8, PRC 스트립 C의 샘플 C1-8을 적절하게 희석된 샘플 양(보충 파일 1 MMqPCR 설정 시트, 열 L) 및 PCR 등급 H2O 양(보충 파일 1)으로 채웁니다. PCR 튜브당 총 15ng의 DNA 양과 75μL의 총 부피에 대한 MMqPCR 설정 시트, 컬럼 M). 따로 보관하십시오.
참고: PCR 스트립의 샘플은 4°C에서 밤새 보관하여 다음날 도금할 수 있습니다.
- 보충 파일 1 MMqPCR 셋업 시트에 자세히 설명되어 있고 표 3에 나열된 대로 PCR 스트립 A의 샘플 A1-8, PCR 스트립 B의 샘플 B1-8, PRC 스트립 C의 샘플 C1-8을 적절하게 희석된 샘플 양(보충 파일 1 MMqPCR 설정 시트, 열 L) 및 PCR 등급 H2O 양(보충 파일 1)으로 채웁니다. PCR 튜브당 총 15ng의 DNA 양과 75μL의 총 부피에 대한 MMqPCR 설정 시트, 컬럼 M). 따로 보관하십시오.
- 표준 곡선 준비: 대조군 DNA 부분 표본을 해동하고 30초 동안 와류를 일으키고 5초 동안 원심분리를 하고 표준 곡선(SC) PCR 튜브 스트립의 첫 번째 튜브에 전체 부피(150μL)를 흡입합니다.
- 70 μL의 PCR 등급 H2O를 SC PCR 스트립의 두 번째에서 여덟 번째 튜브에 피펫팅합니다. 70 μL를 흡인하여 두 번째 PCR 튜브에 분주하기 전에 첫 번째 튜브에서 control DNA를 완전히 재현탁시킵니다. 30초 동안 기다린 다음 70μL를 흡인하여 세 번째 PCR 튜브에 분배하기 전에 두 번째 PCR 튜브에 용액을 완전히 재현탁합니다. 튜브 3에서 7에 대해 이 작업을 반복하여 7가지 표준물질의 2배 연속 희석을 생성합니다. 최종 튜브는 NTC(Non-Template Control)로 기능하기 위해 PCR 등급 H2O만 포함해야 합니다. SC PCR 스트립을 따로 보관하십시오.
표 3: 플레이트의 샘플 구성 및 제어 표준. 96웰 PCR 플레이트에서 모든 시료 및 표준물질의 위치. 이 표를 다운로드하려면 여기를 클릭하십시오.
3. MMqPCR 마스터 믹스 준비
- DNA 중합효소를 제외하고 표 1에 나열된 마스터 믹스 시약 부분 표본을 수집하고 PCR 후드 내부의 실온에 두십시오. 1x 완충액 부분 표본에 SYBR Green 부분 표본 1μL를 추가합니다.
참고: 이 정확한 양은 분석 중 피펫 정확도의 한계로 인해 정확한 농도의 SYBR을 생성하는 데 필요하므로 한 번에 하나의 플레이트만 실행하더라도 이 양은 변경되지 않아야 합니다. - 10초 동안 모든 부분 표본을 소용돌이치고, 5초 동안 원심분리기를 한 다음, 5M 베타인(현재 마스터 믹스 튜브)을 보유하는 5mL 튜브에 다음 양의 시약을 추가합니다: PCR 등급 H2O1,235.2 μL, 10x 완충액 640 μL, dNTP 204.8 μL, DTT 192 μL, 표 1에 나열된 바와 같이 MgCl2 64 μL, SYBR Green / 1x 완충액 48 μL(단계 3.1에서), 두 텔로미어 프라이머 14.4 μL, 및 두 알부민 프라이머 9.6 μL.
- -20 °C 보관에서 DNA 중합효소를 꺼내고, 10초 동안 와류하고, 5초 동안 원심분리기를 제거한 다음 마스터 믹스에 128μL를 천천히 추가합니다. DNA 중합효소를 즉시 -20°C 보관으로 다시 이동합니다.
- 5mL 마스터 믹스 튜브를 30초 동안 소용돌이칩니다.
4. 96웰 플레이트의 준비
- 2개의 96웰 플레이트, 로딩 트러프 및 멀티채널 피펫 팁을 PCR 후드 내부에 배치합니다. 각 플레이트에 플레이트 번호(1 또는 2)를 표시합니다. 플레이트 1(P1 플레이트)을 180° 돌려 기둥 번호가 거꾸로 되도록 합니다. 플레이트 2(P2)가 기술자를 향하도록 둡니다.
- 5mL 마스터 믹스 튜브의 내용물을 로딩 트러프에 붓습니다. 피펫 팁을 사용하여 튜브에 남아 있는 용액을 수집하고 분주합니다.
- 팁을 멀티채널 피펫에 로드하고 각 피펫 팁의 바닥을 밀어 단단히 밀봉합니다. 팁의 마스터 믹스가 흡입될 출력 영역 내에 필터가 없는지 확인하십시오. 필터 조각이 있으면 팁을 교체하십시오.
- 역 피펫팅을 사용하여 플레이트에 점성 마스터 믹스를 채우고, 로딩 트로프를 소용돌이치게 한 다음 첫 번째 스톱을 지나 피펫 플런저를 눌러 피펫 팁에 15μL 이상을 흡인하여 팁이 동일한 부피로 동시에 채워지도록 합니다. 마스터 믹스를 컬럼으로 배출할 때 플런저를 첫 번째 스톱까지 누르고 여분의 마스터 믹스는 피펫 팁에 남겨 둡니다. 플런저를 누른 상태에서 팁을 로딩 트러프에 다시 담그고 플런저를 풀어 팁에 15μL를 더 채웁니다. 동일한 팁을 사용하여 두 플레이트를 모두 채웁니다.
- 마스터 믹스를 먼저 모든 짝수 또는 모든 홀수 열로 배출하고 플레이트 사이를 번갈아 가며 플레이트를 채웁니다(즉, 기술자가 홀수 열 번호로 시작하기로 선택한 경우 P2의 열 1이 먼저 채워진 다음 P1의 열 11, P2의 열 3 등). 기술자가 왼쪽에서 오른쪽으로 작업하면서 시리즈(홀수 또는 짝수)의 모든 웰을 채운 후 동일한 방식으로 다른 시리즈(짝수 또는 홀수)의 열을 수행합니다. 이 프로세스의 차트는 그림 2 를 참조하십시오.
참고: 이러한 플레이트 충전 방식은 플레이트 전체의 위치 효과를 제어하는 기능을 합니다.
- 마스터 믹스를 먼저 모든 짝수 또는 모든 홀수 열로 배출하고 플레이트 사이를 번갈아 가며 플레이트를 채웁니다(즉, 기술자가 홀수 열 번호로 시작하기로 선택한 경우 P2의 열 1이 먼저 채워진 다음 P1의 열 11, P2의 열 3 등). 기술자가 왼쪽에서 오른쪽으로 작업하면서 시리즈(홀수 또는 짝수)의 모든 웰을 채운 후 동일한 방식으로 다른 시리즈(짝수 또는 홀수)의 열을 수행합니다. 이 프로세스의 차트는 그림 2 를 참조하십시오.
- 샘플과 표준 곡선을 포함하는 4개의 닫힌 PCR 스트립을 소용돌이친 다음 미니 원심분리기에서 각각 5초 동안 회전시킵니다.
- PCR 튜브 랙의 PCR 스트립을 플레이트에 놓는 순서대로 정렬합니다. P1의 경우 처음 3개의 열은 샘플(PCR 스트립 A)이고, 4-6열은 표준(PCR 스트립 S)이고, 7-9열은 샘플(PCR 스트립 B)이고, 10-12열은 샘플(PCR 스트립 C, 그림 3 및 표 3 참조)입니다. P2는 기술자에 대해 180° 회전하는 동안 채워지기 때문에 기둥은 그 반대가 됩니다.
- 두 플레이트를 모두 180° 돌려 각 플레이트의 기술자를 기준으로 기둥 번호가 뒤집히도록 합니다(즉, 기술자가 이전에 P2 레이블을 향했던 경우 이제 플레이트 가장자리에서 P1 레이블을 향하게 됨).
- 멀티채널 피펫을 10 μL로 설정하고 단계 4.3에 설명된 대로 피펫 팁을 멀티채널 피펫에 로드합니다.
- 멀티채널 피펫을 사용하여 용액을 재현탁한 다음 4.4단계에 설명된 대로 역 피펫팅을 수행하여 PCR 스트립 A부터 시작하여 10μL의 검체 및 표준 용액을 흡입 및 분주합니다.
- 마스터 믹스가 플레이트에 배치된 것과 유사한 방식(즉, 짝수 또는 홀수)으로 컬럼을 번갈아 가며 PCR 스트립 A를 사용하여 각 플레이트의 처음 3개 열을 채웁니다.
- 표준 곡선 PCR 스트립을 사용하여 다음 세 열을 채웁니다. 이 스트립의 경우 ~90μL로 설정된 200μL 피펫을 사용하여 7번째 표준 희석 튜브(끝에서 두 번째)를 철저히 재현탁한 후 다중 채널 피펫을 사용하여 전체 스트립을 재현탁합니다.
참고: 이 튜브의 부피가 크고 DNA 농도가 작으면 혼합 불량으로 인해 PCR 플레이트 웰 전체에서 다양한 측정이 발생하는 경우가 많습니다. 더 큰 부피를 사용하여 이 튜브를 혼합하면 DNA 용액이 더 잘 균질화됩니다. - 다음 3개의 열을 PCR 스트립 B로 채우고 마지막 3개의 열을 PCR 스트립 C로 채웁니다. 각 PCR 스트립 사이의 피펫 팁을 교체하고 그림 3에 설명된 대로 두 플레이트의 모든 96-well을 해당 샘플 및 대조 표준물질로 채웁니다.
- 플레이트가 채워지면 벤치 상단의 플레이트를 부드럽게 두드려 웰 측면의 액체가 바닥으로 내려가도록 한 다음 밀봉 필름을 사용하여 플레이트 상단을 덮습니다. 손가락 끝으로 필름의 모든 가장자리를 눌러 단단히 밀봉하십시오.
- 후드 상단을 30초 동안 소용돌이쳐 플레이트를 섞은 다음 웰 개구부가 중앙을 향하도록 하여 밀봉된 플레이트를 플레이트 스피너에 2분 동안 놓습니다.
- 플레이트를 thermocycler에 넣고 플레이트의 열 번호를 읽을 수 있는 순서로 놓습니다. thermocycler 상단을 닫기 전에 깨끗한 티슈를 사용하여 각 플레이트의 상단을 청소하십시오.
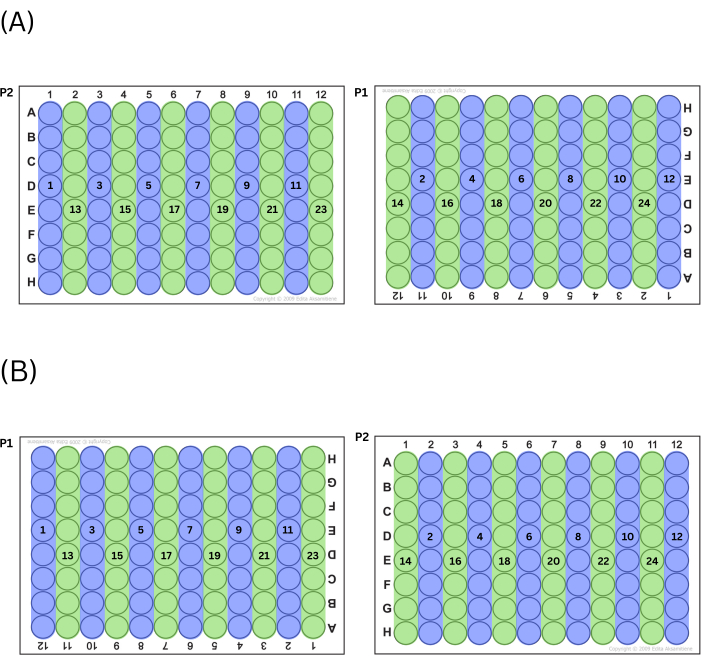
그림 2: 플레이트 충전 공정. (A) 홀수 열을 먼저 채우기로 선택한 경우 이것이 우물이 채워지는 순서입니다. (B) 짝수 열을 먼저 채우기로 선택한 경우 우물이 채워지는 순서입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
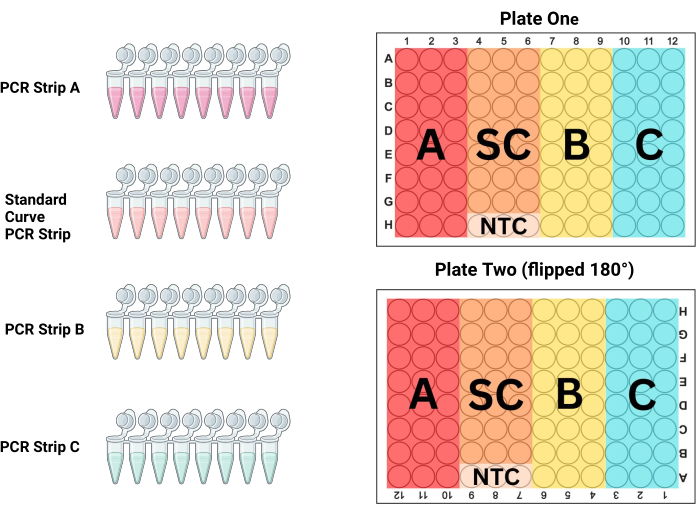
그림 3: 플레이트 레이아웃. PCR 스트립 A, 스트립 B, 스트립 C 및 표준 곡선(SC) 스트립을 모두 사용하여 각 플레이트의 3개 열을 채워 각 샘플 및 표준물질 희석의 중복 삼중을 생성해야 합니다. 이 다이어그램은 각 스트립으로 채워야 하는 열을 보여줍니다. 플레이트 2는 로딩 전에 180° 뒤집히지만(열 및 행 헤더가 거꾸로 되어 있음) 플레이트는 플레이트 1과 동일하게 충전되어 잠재적인 피펫팅 오류를 제거하는 동시에 플레이트 전체의 위치 효과를 제어합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
5. MMqPCR 열실실
- 4.8.1단계에서 플레이트가 회전하는 동안 컴퓨터와 열순환기를 켭니다. thermocycler 소프트웨어를 엽니다. 이 프로토콜은 CFX Maestro 소프트웨어의 사용에 대해 설명합니다.
- MMqPCR 열순환 프로토콜19(그림 4)에 따라 TL 열순환 프로토콜을 생성합니다.
- DNA 중합효소를 95°C에서 15분 동안 활성화하기 위해 배양 단계를 추가합니다.
- 프라이머-이량체 결합을 피하려면 15초 동안 94°C, 1분 동안 49°C의 두 사이클을 실행한 다음 15초 동안 94°C, 15초 동안 59°C의 3 사이클을 실행합니다.
- 텔로미어 증폭을 위해 15초 동안 85°C, 30초 동안 74°C의 27 사이클을 추가한 다음 신호 수집을 추가합니다.
- 알부민 증폭을 위해 15초 동안 94°C, 30초 동안 84°C의 31주기 주기를 추가한 다음 신호 획득을 추가합니다.
- 열 순환 프로토콜에서 각 증가 정도에 대해 5초 간격으로 59°C에서 95°C까지의 용융 곡선을 포함합니다.
- 두 thermocycler에 대해 Start Run(실행 시작 )을 클릭합니다. 메시지가 표시되면 해석 파일의 제목을 제공합니다.
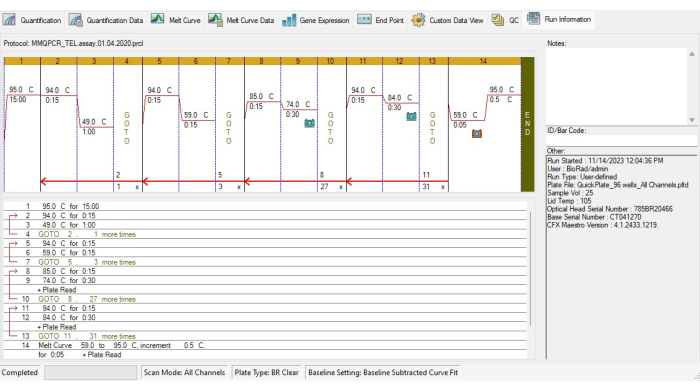
그림 4: MMqPCR 분석의 열순환 프로파일. 원래 thermocycling 프로토콜19에 따라 소프트웨어에서 생성된 MMqPCR 프로토콜. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
6. MMqPCR 데이터 분석
- 열순환이 완료되면 다음과 같은 방식으로 데이터를 분석하여 처리된 샘플에 대한 TL 값을 생성합니다.
- 소프트웨어에서 플레이트 설정 피쳐를 선택합니다. 드롭다운 메뉴에서 View/Edit Plate를 클릭합니다.
- 모든 well을 강조 표시하고 Select Fluorophores(형광단 선택)를 클릭한 다음 SYBR 레이블이 지정된 상자를 선택하고 다른 모든 상자의 선택을 취소합니다. 확인을 클릭합니다.
- 모든 웰이 여전히 강조 표시되어 있지만 로드라는 단어 옆의 SYBR 옆의 상자를 선택합니다. 이제 모든 우물에는 SYBR이 기록되어 있어야 합니다.
- 그런 다음 표준 제어 직렬 희석의 상단 또는 하단에 있는 3개의 NTC 웰을 강조 표시하고 오른쪽의 샘플 유형 메뉴에서 NTC 를 선택합니다. 세 개의 우물은 이제 노란색으로 표시되어야 하며 NTC라고 불려야 합니다. 그림 5A 는 P1에 대해 선택해야 하는 웰을 보여주고, 그림 5B 는 P2에 대해 선택해야 하는 웰을 보여줍니다.
알림: P2는 P1에서 반전되므로 NTC는 P4의 경우 6-1열의 하단에 있고 P7의 경우 9-2열의 상단에 있습니다. - 표준 제어 직렬 희석의 21개 웰을 강조 표시하고 시료 유형 메뉴에서 Standard 를 선택합니다. 이 우물은 녹색이어야 합니다. 이러한 웰이 계속 강조 표시되어 있는 동안 기술 복제(Technical Replicate)를 클릭합니다. 리플리케이트 크기(Replicate Size) 메뉴에서 3 을 선택한 다음 수평(Horizontal) > 적용(Apply) 을 선택합니다. 이 우물은 Std-1에서 Std-7까지 3개 세트로 표시해야 합니다.
- 표준이 여전히 강조 표시된 상태에서 아래로 스크롤하여 Dilution Series를 선택합니다. Dilution Factor(희석 계수) 필드에 2를 입력한 후 다음 단계에 설명된 대로 플레이트 번호에 따라 희석 시작 농도와 방향성을 입력합니다.
- P1 플레이트의 경우 시작 농도 필드에 2.00E-03을 입력하고 감소 상자를 선택한 다음 적용을 선택합니다. 21개 웰의 값에는 각 웰에 위에서 아래로 2.00E-03에서 3.13E-05 사이의 농도 값이 표시되어야 합니다.
- P2 플레이트의 경우 시작 농도 필드에 3.13E-5를 입력하고 증가 확인란을 선택한 다음 적용을 선택합니다. 21개 웰의 값에는 각 웰에 위에서 아래로 3.13E-05에서 2.00E-03 사이의 농도 값이 표시되어야 합니다.
- 샘플의 경우 PCR 스트립 A(P1 및 P2의 경우 1-3)에 대한 열을 강조 표시하고 샘플 유형 메뉴에서 알 수 없음을 선택한 다음 Technical Replicate를 선택합니다. 리플리케이트 크기(Replicate Size) 메뉴에서 3을 선택한 다음 수평(Horizontal) > 적용(Apply) 을 선택합니다. 이 열의 웰은 파란색이어야 하며 Unk-1에서 Unk-8까지 3개 세트로 행으로 레이블이 지정되어야 합니다.
- PCR 스트립 B의 열에 대해 6.3단계를 반복합니다(P1의 경우 7-9열, P2의 경우 4-6열). Unk-9에서 Unk-16까지 레이블이 지정되어야 합니다.
- PCR 스트립 C의 컬럼에 대해 6.3단계를 반복합니다(P1 및 P2의 경우 10-12열). Unk-17에서 Unk-24까지 레이블이 지정되어야 합니다. 6.2-6.3.2 단계가 완료되면 플레이트 설정 창이 그림 5A, B와 유사해야 합니다.
- 플레이트 편집기 창의 오른쪽 아래에서 확인을 선택합니다. Yes(예 )를 클릭하여 메시지가 표시될 때 변경 사항을 적용합니다.
- 분석 수준 QC의 경우, 정량화 탭의 곡선이 각각 텔로미어 및 알부민 앰플리콘에 해당하는 열순환 프로파일의 9단계 및 12단계 모두에 적합한지(예: 역 증폭 곡선 없음) 확인하십시오. 곡선은 소프트웨어 창의 오른쪽 중간에 있는 드롭다운 메뉴를 통해 액세스할 수 있습니다. 그림 6A,B는 적절한 곡선을 표시합니다.
- 9단계 및 12단계 모두에서 보고된 PCR 효율이 90%에서 110% 사이이고 이 두 가지 효율이 서로 10% 이상 다르지 않은지 확인하십시오(즉, 9단계의 경우 92.4%, 12단계의 경우 98%가 적절하지만 9단계의 경우 92.4%, 12단계의 경우 108%는 적절하지 않음). 또한 표준 R2 곡선이 >0.995인지 확인합니다. P1 또는 P2가 이러한 기준을 충족하지 못하면 플레이트를 다시 실행해야 합니다.
- P1의 경우 9단계를 선택하고 그림 6A,B와 같이 표준 곡선에 해당하는 21개의 웰을 강조 표시합니다. 소프트웨어 창의 오른쪽 하단 모서리에 있는 Cq 값을 복사합니다. 보충 파일 1로 사용할 수 있는 텔로미어 데이터 시트 템플릿을 열고 이 값을 표준 1 시트, B 열에 붙여넣습니다. 12단계를 선택한 다음 이 Cq 값을 복사합니다. 이 값을 표준 1 시트, I 열에 붙여 넣습니다.
- 9단계에서 기울기 값을 식별하고 표준 1 시트의 C4 셀에 입력합니다. 12단계에서 경사 값을 식별하고 표준 1 시트의 J4 셀에 입력합니다.
참고: 시스템은 6.6.1단계에서 식별된 표준 곡선의 기울기를 사용하여 프라이머의 효율 백분율을 계산합니다. 효율성 백분율은 다음 공식을 사용하여 수동으로 계산할 수 있습니다.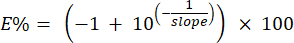
- 각 표준 희석 삼중 항목에 대한 변동 계수(CV)가 0.1 미만인지 확인합니다. 0.1보다 크거나 같으면 플레이트의 7포인트 표준 직렬 희석 곡선에 포함된 모든 농도의 대조군에서 총 3개의 개별 웰을 제외합니다. 하나의 웰만 트리플리케이트 세트에서 제외해야 합니다.
참고: 소프트웨어의 분석에서 표준 점을 제외하면 두 프라이머에 대해 보고된 기울기, R, y절편 및 효율성이 변경됩니다. 따라서 분석에서 표준 웰을 제거한 후에는 6.5.1-6.6.1단계를 반복해야 합니다. - P2 해석 파일 및 해당 표준 2 시트에 대해 6.6.1 - 6.6.2단계를 반복합니다.
- 표준 1 및 표준 2 시트가 모두 허용 가능한 CV와 함께 완료된 경우에만 분석 파일에서 개별 샘플 데이터를 수집합니다. QC 조정이 P1 v P2 시트 열 L에 보고되어 있는지 확인합니다(예: P1 분석 파일에서 제외된 웰 수).
- 9단계에서 기울기 값을 식별하고 표준 1 시트의 C4 셀에 입력합니다. 12단계에서 경사 값을 식별하고 표준 1 시트의 J4 셀에 입력합니다.
- P1 분석 파일에 있는 72개의 시료 웰만 다음과 같이 Quantification 탭에서 파란색으로 강조 표시되어 있는지 확인합니다. 그림 7A,ᄂ. 고르다 단계 9. 소프트웨어 창의 오른쪽 하단 모서리에 있는 Cq 및 SQ 값을 복사합니다. 이러한 값을 D3 셀에서 시작하여 샘플 P1 시트에 붙여넣습니다. 고르다 단계 12 그런 다음 이러한 Cq 및 SQ 값을 복사하여 F3 셀에서 시작하여 샘플 P1 시트에 붙여넣습니다.
- P2 해석 파일의 샘플과 해당 샘플 P2 시트에 대해 이 단계를 반복합니다. 텔로미어 대 단일 복사본(T/S) 비율(H열)은 자동으로 계산되지만 다음 공식을 사용하여 수동으로 계산할 수 있습니다.
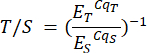
여기서 ET/S 는 텔로미어 또는 단일 복제 유전자를 각각 표적으로 하는 반응에 대한 지수 증폭의 효율성이고, CqT/S 는 텔로메릭 함량 또는 단일 복제 유전자를 표적으로 하는 주어진 복제가 형광 정량화의 임계 임계값에 도달하는 주기입니다. 평균 T/S 비율(I열)은 샘플당 6개의 측정값에 걸쳐 계산됩니다.
참고: CFX의 샘플 ID 번호는 두 플레이트의 동일한 생물학적 샘플과 일치하지 않습니다. 텔로미어 데이터 시트 템플릿은 이러한 차이를 설명하고 적절한 중복 삼중을 정렬합니다. - CFX Maestro에서 그림 7B와 같이 모든 분석 샘플의 Cq 값이 표준 곡선의 가장 낮은 Cq 값과 가장 높은 Cq 값 사이에 있는지 확인합니다. 샘플이 그림 7A에서 볼 수 있듯이 범위를 벗어난 경우 농도 조정 후 다시 실행해야 합니다. 최고 농도 표준물질보다 낮은 Cq 값을 가진 시료는 범위를 벗어난 증폭 주기 수에 가까운 인자로 희석해야 하며, Cq 값이 가장 낮은 농도 표준물질보다 높은 시료는 임계값을 초과하는 증폭 주기 수에 가까운 인자로 농축해야 합니다.
참고: 분석 샘플의 농도 변동이 크면 추가적인 잠재적 변동 수준이 발생하므로 주의해서 수행해야 합니다. - NTC의 시작 농도가 샘플 웰에 존재하는 평균 DNA 양의 5% 미만인지 확인합니다. 이는 MMqPCR의 시료 P1 및 시료 P2 시트에서 수질 오염률(%)을 확인하여 확인할 수 있습니다. NTC가 오염을 나타내면 플레이트를 다시 실행하십시오.
참고: MMqPCR 분석의 민감도로 인해 제어할 수 없는 경미한 오염으로 인해 때때로 NTC 웰이 증폭될 수 있습니다. 그러나, 이 증폭은 미미해야 하며, 열순환 프로토콜의 단계 12에서 단일 복제 유전자 증폭 후 여러 주기에 걸쳐 발생해야 합니다. - 시료 수준 QC의 경우 시료 P1 및 시료 P2 시트에서 표준 편차(SD)(J열) 및 CV(K열)를 검토합니다. 샘플 삼중 T/S 비율에서 intraplate CV가 0.10(10%) 미만인지 확인합니다. 0.1보다 큰 intraplate CV를 수정하기 위해 삼중 집합에서 하나의 T/S 비율을 제외할 수 있습니다.
참고: 샘플당 6개의 측정값에서 하나의 총 반복실험만 제외될 수 있으며, 즉, 동일한 샘플에 대한 반복실험은 P1과 P2 모두에서 제외될 수 없습니다. - P1 v P2 시트, G 열의 개별 샘플 플레이트 간 CV가 0.05(5%) 미만인지 확인합니다. 0.05보다 큰 CV를 수정하려면 두 플레이트에서 샘플당 6개의 측정에서 하나의 T/S 비율을 제외합니다. intraplate triplicates에 대한 육안 검사 후 샘플을 제외하고 필요에 따라 6개의 측정 모두를 수행합니다.
참고: 이러한 QC 기준을 통과하는 샘플, 즉 판내 CV < 10% 및 판간 CV < 5%만 최종 TL 결과에 포함될 수 있습니다. 그렇지 않으면 이 실행에 대한 P1 v P2 및 ICC 데이터 시트에서 샘플을 제외해야 하며 두 번째 MMqPCR 분석에서 샘플을 다시 평가해야 합니다. - 플레이트 수준 QC의 경우 샘플 P1 및 샘플 P2 시트의 하단에 위치한 각 플레이트의 모든 샘플에 대한 평균 플레이트 내 변동이 0.05(5%) 미만인지 확인합니다. 추가 플레이트 수준 QC의 경우 P1 v P2 시트, 셀 G29의 전체 플레이트 간 변동이 0.06(6%) 미만인지 확인합니다. QC를 통과하지 못한 샘플이 G30 셀의 플레이트 간 CV 계산에서 제거되었는지 확인합니다.
알림: 관련 샘플 그룹(예: 가족 구성원, 다른 시점 등)으로 작업할 때 그룹의 모든 샘플을 동일한 플레이트에서 실행해야 합니다. 따라서 그룹의 한 샘플이 QC 기준을 통과하지 못하면 그룹의 모든 샘플을 다시 실행해야 합니다. - 별도의 전체 코호트 또는 실험 데이터 파일에 QC를 통과한 각 샘플(P1 v P2 시트, 열 I)에 대한 최종 TL 데이터, QC를 통과한 각 샘플에 대한 ICC 계산(ICC 데이터 시트, E-K열) 및 P1 v P2 시트(J-L열)의 최종 분석 실행 QC 데이터를 기록합니다.
- P2 해석 파일의 샘플과 해당 샘플 P2 시트에 대해 이 단계를 반복합니다. 텔로미어 대 단일 복사본(T/S) 비율(H열)은 자동으로 계산되지만 다음 공식을 사용하여 수동으로 계산할 수 있습니다.
- TRN(Telomere Research Network)에서 만든 보충 파일 2 를 사용하여 프로젝트20에 대한 ICC를 계산합니다.
- 마지막으로, TL 연구 간의 비교성을 높이기 위해 코호트의 모든 샘플에 대한 평균 T/S 비율을 개별 샘플 T/S 비율에서 뺀 다음 코호트의 모든 샘플에 대한 SD로 나눈 다음 코호트의 모든 샘플에 대한 SD로 나누는 다음 방정식을 사용하여 별도의 전체 코호트 또는 실험 데이터 파일의 각 샘플에 대한 최종 TL 데이터를 Z-점수로 변환합니다.
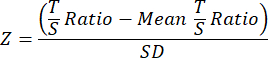
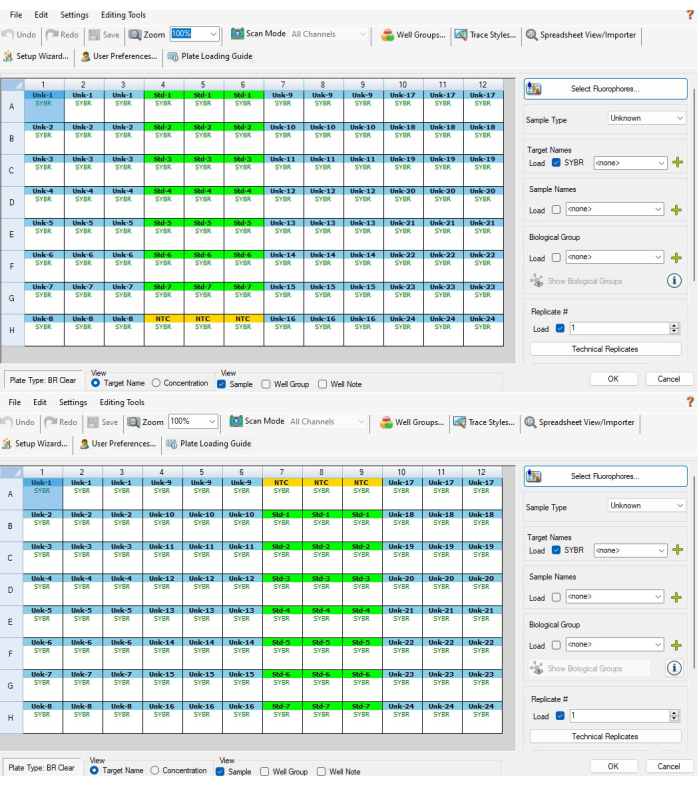
그림 5: 소프트웨어 기반 플레이트 설정. (A) 1-6.2단계를 완료한 후 P6.4 플레이트에 대한 소프트웨어 기반 플레이트 설정. (B) 2-6.2단계를 완료한 후 P6.4 플레이트에 대한 소프트웨어 기반 플레이트 설정. 소프트웨어가 웰 위치를 기반으로 샘플 ID를 할당하는 방식(즉, P1의 CFX 샘플 1은 P2의 CFX 샘플 24임)으로 인해 샘플 및 표준 ID가 두 CFX 플레이트 간에 정렬되지 않습니다. 보충 파일 1에 제공된 엑셀 템플릿이 이를 설명하여 동일한 생물학적 샘플에 대한 판간 측정이 서로 올바르게 정렬되도록 합니다 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
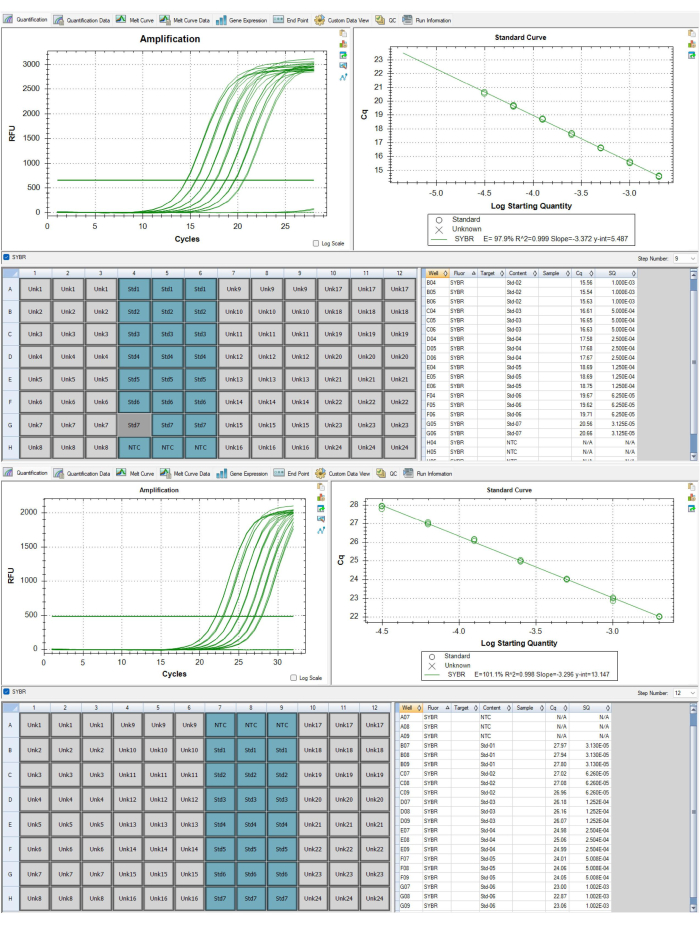
그림 6: 엘로미어와 알부민 앰플리콘에 대한 표준 곡선. (A) 이 표준 곡선은 대표 결과 데이터 세트의 P1 텔로미어 앰플리콘에서 가져온 것입니다. 한 표준물질은 QC 기준을 충족하지 못하여 제거되었습니다. (B) 이 표준 곡선은 대표 결과 데이터 세트의 P2 알부민 앰플리콘에서 가져온 것입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
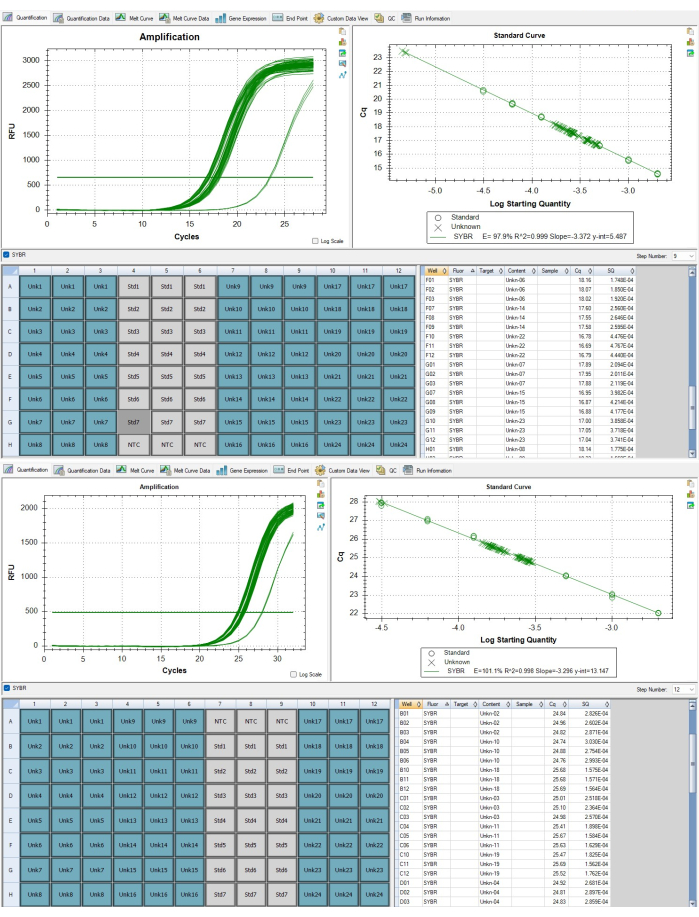
그림 7: 텔로미어 및 단일 복제 유전자 증폭체. (A) 텔로미어 증폭 및 (B) 소프트웨어에 표시된 대표 결과에 보고된 샘플의 알부민 유전자 증폭. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
7. MMqPCR 데이터 보고
- MMqPCR에서 생성된 TL 데이터를 사용하여 연구 결과를 보고할 때 TRN 최소 보고 지침에 설명된 항목의 보고를 확인하십시오. 이러한 지침은 TRN 웹사이트(https://trn.tulane.edu/resources/reporting-guidelines/)에서 찾을 수 있으며 이 정보를 보고하기 위한 템플릿은 보충 파일 3에 제공됩니다. 적절한 경우 이 프로토콜과 다른 TRN 지침 및 리소스를 인용합니다.
결과
표 4 및 표 5에 제시된 결과는 프로토콜에 따라 얻은 매우 반복 가능한 TL 측정의 예를 제공합니다. 이러한 결과를 위해 제조업체 지침에 따라 상용 키트를 사용하여 24개의 말초 혈액 단핵 세포(PBMC) 샘플에서 DNA를 추출했습니다. 이 24개의 샘플은 두 개의 96웰 플레이트에 걸쳐 실행되었습니다. 모든 DNA 샘플은 평균 260/280 비율, 평균 260/230 비율 및 dsDNA 농도를 사용하여 분광 광도계 및 형광계를 통해 품질을 확인하여 분석 적합성 및 샘플 희석 계수를 결정했습니다(표 6). 표 6은 또한 분광광도계를 사용하여 측정한 DNA 농도와 다를 수 있는 dsDNA 농도를 정량화하는 것의 중요성을 강조합니다. 이러한 변동성은 정량화에 대한 다양한 접근 방식의 결과입니다. 특히, 분광광도계는 280nm에서의 흡수를 기반으로 DNA 농도를 도출하며 흡광도 판독값에 영향을 미치는 오염 물질(예: 단백질, 염분 등)로 인한 변동에 취약합니다. 이와 대조적으로, 형광측정기를 사용하여 측정한 dsDNA 농도는 dsDNA에 특이적으로 결합하는 염료의 형광에 의해 측정되며, 따라서 DNA 함량을 보다 정확하게 반영하는 것으로 간주됩니다. DNA 샘플은 MMqPCR TL 분석 전에 최대 3번의 동결-해동 주기를 경험했습니다. 대조군 DNA는 한 개인으로부터 PBMC의 통합 DNA 추출을 통해 생성되었으며, 이를 사용하여 DNA를 2ng/μL에서 0.0313ng/μL까지 7점 연속 희석했습니다. PCR 9단계(텔로미어 앰플리콘) 및 12단계(이 프로토콜의 단일 복제 유전자 앰플리콘, 알부민)에 대해 생성된 독립적인 표준 곡선은 그림 6A,B에 나와 있습니다. 이 분석은 그림 8에서 볼 수 있듯이 개별 앰플리콘 제품이 고유한 온도에서 생산되는 것을 보여주는 용융 곡선을 생성한 상용 Real-Time PCR 검출 시스템에서 실행되었습니다.
표 4: 최적의 대표 결과를 위한 MMqPCR 측정의 출력 데이터. 24개 샘플에 대한 평균 T/S 비율, SD, CV 및 Z-점수 TL. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 5: 텔로미어 길이 데이터 출력. MMqPCR 분석의 원시 데이터는 소프트웨어에서 분석된 다음 추가 분석 및 QC를 위해 보충 파일 1로 첨부된 스프레드시트 템플릿에 추가됩니다. 이 TL 템플릿 출력 시트는 두 플레이트의 각 샘플에 대한 샘플 ID, 평균 TL, SD 및 CV를 표시하는 데이터 요약을 제공합니다. 이 표를 다운로드하려면 여기를 클릭하십시오.
표 6: 시료 결과에 대한 분광광도계 및 형광광계 DNA 품질 지표. dsDNA 및 시료당 모든 오염 물질에 대한 중복 분광 광도계 분석 및 단일 형광계 측정의 QC 데이터. 이 표를 다운로드하려면 여기를 클릭하십시오.
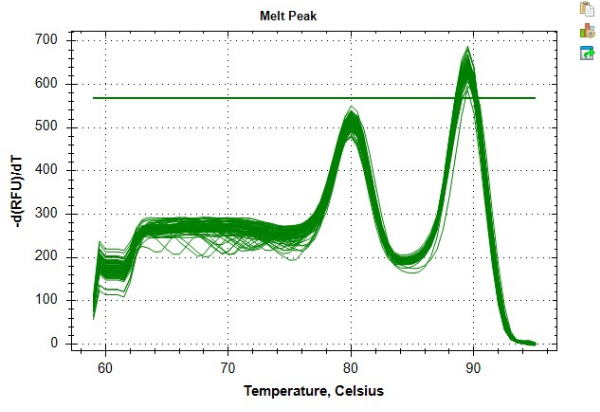
그림 8: 용융 곡선의 예. 소프트웨어에서 생성된 샘플 데이터에 대한 용융 곡선. 초기 피크 ~80°C는 텔로미어 앰플리콘을 나타내고 ~89°C의 두 번째 피크는 알부민 앰플리콘을 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
이 프로젝트의 경우, 평균 텔로미어 효율은 90.7 - 102.1 범위에서 98.6%였으며, 모든 실행에서 평균 알부민 효율은 93.6 - 108.2 범위에서 102.3%였습니다. 표준 곡선에서 평균 0.97번의 반복이 제거되었으며, 이는 이 프로토콜의 QC 기준을 충족합니다. 평균 판간 CV는 SD가 0.00616으로 1.83%였고 평균 판내 CV는 SD가 0.00658로 3.78%였습니다. 이러한 대표 샘플에 대한 평균 TL은 표 4 및 표 5 에 제시되어 있습니다.평균 TL은 1.37이었고 SD는 0.24이고 범위는 0.84에서 2.32였습니다. 샘플당 T/S 비율, SD, CV 및 Z-scored TL은 표 4에 나열되어 있습니다. MMqPCR 분석의 T/S 비율 출력은 TL의 상대적 측정이기 때문에 비율은 연구 간 비교를 허용하기 위해 이 Z-점수로 변환되었습니다. 이 프로젝트의 ICC는 배치 및 실행 효과20을 고려하여 보충 파일 2에 있는 TRN 지침에 설명된 대로 R 스크립트를 사용하여 계산되었습니다. 프로젝트 내 ICC를 계산하기 위해 통과 샘플의 10%를 다시 실행하여 코호트의 각 플레이트에서 하나 이상의 샘플로 ICC 플레이트를 채우도록 했습니다. 전체 프로젝트 ICC가 0.801[CI: 0.703, 0.86]이라는 것은 TL 결과의 재현성이 높다는 것을 나타냅니다.
모든 결과가 최적은 아닙니다. 그림 9 및 표 7의 결과는 MMqPCR 분석의 최적이 아닌 결과를 보여줍니다. 그림 9는 텔로미어 효율이 QC 표준보다 낮은 90% 미만의 표준 곡선을 보여주며, 이는 전체 플레이트를 반복해야 합니다. 프라이머 효율성 문제는 일반적으로 시약 문제로 인해 발생하므로 어떤 시약이 낮은 효율성의 원인이 되는지 확인하는 첫 번째 단계로 시약이 분량된 날짜와 만료 시기를 추적하는 것이 중요합니다. 만료된 시약은 플레이트를 다시 실행하기 전에 교체해야 합니다. 표 7은 초기 샘플 수준 QC 기준을 통과했지만 플레이트 간의 변동성이 높아 플레이트 수준 QC 실패로 이어진 플레이트를 표시합니다. 플레이트 간의 차이는 일반적으로 피펫팅 및 플레이트 충진 기술의 오류로 인해 발생합니다. 이 경우 기술자는 플레이트 설정 중에 발생한 모든 문제를 평가하고 파이펫이 교정되었는지 확인해야 합니다.
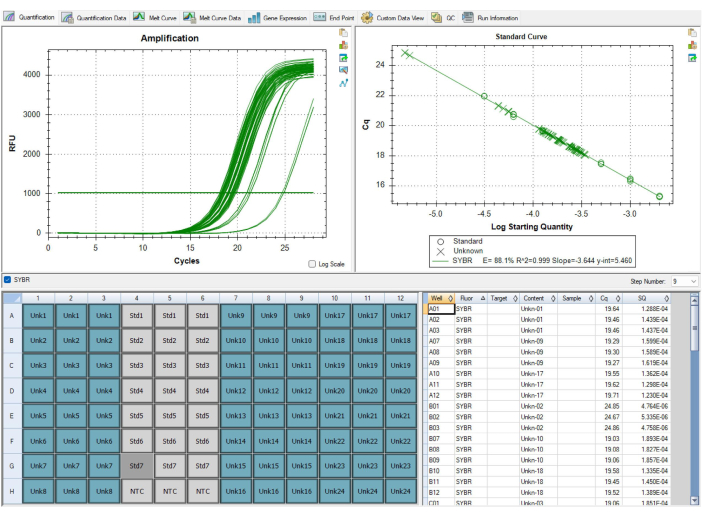
그림 9: 차선의 결과. 소프트웨어에 표시된 이 표준 곡선은 텔로미어 프라이머의 증폭 효율이 90% 미만이었기 때문에 QC를 통과하지 못했습니다. 이미지는 P1에서 가져온 것이지만 P2도 마찬가지로 효율성이 낮았습니다. 이 실행의 데이터는 사용할 수 없었으며 만료된 원인 시약을 교체한 후 모든 샘플을 다시 실행해야 했습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
표 7: 최적이 아닌 결과. 이 표에는 많은 샘플이 QC 표준을 충족하지 못한 실행에 대한 텔로미어 데이터 템플릿이 표시됩니다. 다시 실행해야 하는 샘플은 CV 값을 기반으로 결정된 다음 쉽게 식별할 수 있도록 샘플 이름을 빨간색 글꼴로 변경했습니다. 이 표를 다운로드하려면 여기를 클릭하십시오.
보충 파일 1: Telomere 데이터 스프레드시트 템플릿입니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 2: ICC 계산. 이 프로토콜은 텔로미어 연구 네트워크(TRN)에 의해 만들어졌습니다. 이 파일은20에서 수정되었습니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 3: TRN 보고 지침. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
2002년 이전에 대규모 인구 기반 연구에서 가장 일반적으로 사용된 TL 측정 방법은 말단 제한 단편 길이(TRF)24,25의 서던 블롯 분석이었습니다. TRF는 전문 실험실에서 우수한 정밀도와 재현성을 제공함에도 불구하고 필요한 DNA의 양과 품질 및 제한된 처리량으로 인해 적용 가능성이 제한되어 qPCR 기반 TL 분석의 활용도를 높이고 이후 MMqPCR 분석을 위한 배경을 제공합니다. MMqPCR TL 분석법은 각 QC 기준에 세심한 주의를 기울여 설정, 최적화 및 유지 관리할 때 반복 가능한 TL 측정을 제공합니다. 분석된 각 코호트에 대한 특정 ICC의 계산 및 보고는 분석 신뢰성을 보장하기 위해 필요합니다. MMqPCR 방법은 DNA 품질 및 기술 전문 지식에 따라 달라지지만, 소량의 DNA가 필요하고 singleplex PCR보다 신뢰성이 높으며 다른 방법보다 시약 비용과 기술자 시간이 더 효율적이기 때문에 TL을 조사하는 대규모 인구 기반 연구에 매우 적합합니다. 높은 ICC를 생성할 수 있는 능력은 TL의 대규모 인구 기반 연구를 위한 MMqPCR의 사용을 지원하는 추가 데이터를 제공합니다. MMqPCR TL 측정은 모든 원인으로 인한 사망률, 노화, 평생 스트레스, 환경 노출 및 심혈관 질환 및 암과 같은 신체 건강 결과의 바이오마커로서 TL의 역할을 정의하려는 광범위한 연구에 적용될 수 있습니다 4,7,8,10,11,12,13,26,27,28, 29,30,31.
MMqPCR 방법의 한 가지 한계는 TL을 T/S 비율로 보고한다는 것인데, 이는 단일 복제 유전자, 마스터 믹스 조성 및 PCR 순환 매개변수32의 선택에 따라 달라지는 길이의 상대적 추정치입니다. T/S 비율은 단위가 없습니다. 따라서, 다른 TL 측정 방법론과 결합하지 않고, 이 방법은 염기쌍 값 18,33,34에서 추정치를 보고할 수 없다. 결과적으로, T/S 비율은 연구 전반에 걸쳐 관련성을 갖기 위해 Z-점수로 변환되어야 한다35. 실험실, 방법 및 분석에서 이 작업을 수행할 때 상당한 주의를 기울여야 합니다. 또한, 이 방법은 겔 기반 혼성화 분석과 마찬가지로 간질 텔로미어의 정량화를 포함할 수 있습니다. 그러나 이러한 염기서열은 게놈당 총 텔로미어 DNA 함량에서 매우 작은 비율을 차지합니다. 또한, 간질 텔로미어 서열은 표준 텔로미어 반복에서 벗어나는 불일치 염기쌍 서열을 포함할 가능성이 더 높기 때문에 프라이머 결합 및 증폭의 가능성이 감소합니다. 또한 MMqPCR 분석에 필요한 최소한의 DNA가 유리하지만 TL의 qPCR 기반 측정은 샘플 보관 조건, DNA 추출 방법론 및 생물학적 조직 36,37을 포함하여 DNA 품질 및 무결성에 영향을 미치는 사전 분석 요인의 영향을 받는다는 점에 유의해야 합니다36,37. 이러한 요인에 대한 분석적 제어는 qPCR38을 사용하여 생성된 TL 측정의 외부 타당성을 향상시킬 수 있는 것으로 나타났습니다. 그럼에도 불구하고, 이 접근법을 사용하여 샘플의 품질이 TL의 정확한 추정치를 생성하기에 충분한 품질인지 결정하기 위한 현재 데이터 기반 지침이 없기 때문에 MMqPCR 분석에 의해 생성된 TL에 대한 DNA 품질 차이의 영향은 특히 체계적으로 평가되어야 합니다. 이러한 한계에도 불구하고 인구 수준 건강 결과에 대한 연구에 대한 이 분석의 적용은 상당합니다.
MMqPCR 분석을 사용할 때 정밀도와 처리량에 대한 지속적인 평가가 필요합니다. 현재 설계된 대로, 기술자는 두 개의 열순환기를 사용하여 중복 플레이트에서 동시에 3개의 시료를 실행합니다. 여러 thermocycler가 없는 경우, 처리량이 감소하는 경우에도 정밀도를 높이기 위해 중복 삼중 측정을 유지하고 플레이트를 순차적으로 실행하는 것이 좋습니다. 예를 들어 단일 삼중 측정 사용과 같이 정밀도보다 처리량을 우선시하는 모든 결정은 분석 샘플 분석을 진행하기 전에 결과 ICC에 대한 철저한 테스트 및 평가를 수반해야 합니다. 처리량 및 정밀도에 관한 결정을 내릴 때는 시료 크기와 시료 DNA의 품질을 고려해야 합니다. 표본 크기가 작거나 DNA 표본의 품질이 낮으면 더 높은 정밀도를 우선시해야 한다23. 이는 관련 그룹(예: 가족 구성원, 여러 시점을 가진 피험자)의 샘플로 작업할 때 훨씬 더 중요합니다. 이러한 경우, 예를 들어 실험을 시작하기 전에 관련 샘플을 동일한 플레이트에 할당하는 것과 같은 신중한 계획은 의도하지 않은 플레이트별 그룹 교란으로 인한 통계적 검증력의 손실을 방지하는 한 가지 방법입니다.
이 분석을 위해 분광 광도계를 사용하여 DNA 샘플 품질을 평가했습니다: 260/280 비율의 경우 1.6-2.0, 260/230 비율의 경우 2.0-2.2 범위 내의 샘플이 허용되는 것으로 간주되었습니다. 이 품질 평가와 형광측정기를 통한 이중 가닥 DNA의 정확한 평가는 반복 가능한 TL 데이터를 얻기 위한 이 프로토콜에서 중요한 단계입니다. DNA 무결성에 대한 다른, 보다 기술적인 측정치들, 예를 들면, 단편 크기 및/또는 아가로스 겔을 통해 결정된 DNA 품질의 요약 측정치들(예를 들어, DNA 무결성 수)이 시료 품질을 결정하는 데 이용될 수 있다(38). 또한 MMqPCR 분석을 실행하기 위한 시료 전처리 시에만 시료 희석을 수행하는 것이 좋습니다. 이를 통해 DNA 부분 표본이 추출 후 조작을 최소화하여 사전 분석 처리의 변동성을 줄일 수 있습니다. DNA 샘플을 운송해야 하는 경우, 낮은 농도39,40에서 DNA 샘플에서 발생하는 분해를 완화하기 위해 가능한 가장 높은 농도의 드라이아이스로 운송해야 합니다. 동결-해동 주기에서 발생하는 DNA의 분해로 인해 DNA를 비축하기 위한 동결-해동의 횟수를 최소화해야 합니다41. 코호트를 실행하기 전에 풀링된 대조군 DNA의 부분 표본을 생성해야 하며 개별 분석 DNA 샘플의 부분 표본은 분석을 실행하기 전에 생성해야 합니다.
분석 성능 및 QC의 주요 지표에는 NTC 신호, 판간 및 판내 CV, 표준 곡선 R2가 포함됩니다. QC 기준을 충족하지 못하는 경우 여러 가지 방법으로 완화할 수 있습니다. PCR 등급 H2O 스톡을 정기적으로 변경하고 실행되는 각 플레이트 쌍에 대해 PCR 등급 H2O 하위 스톡을 분주하면 오염 원인과 NTC 증폭을 최소화할 수 있습니다. 오염을 줄이기 위한 추가 단계는 다음과 같습니다: MMqPCR 분석 전용 특정 PCR 후드 지정; DNA 오염 제거 용액으로 PCR 후드 및 장비를 닦아내는 단계; 자외선으로 방을 조사하는 것; PCR 후드에 있을 때 멸균 기술을 연습합니다. 분석 반복성을 높이고 CV를 줄이려면 샘플, 희석액 및 PCR 스트립을 각각의 소용돌이 단계에서 세게 소용돌이치고 DNA 샘플을 피펫팅할 때 완전히 재현탁하는 것이 좋습니다. 임계값 미만의 표준 R2 곡선(<0.995)은 플레이트 로딩 중 피펫팅 오류에 기인할 가능성이 높습니다. 이를 방지하려면 정밀한 피펫팅에 세심한 주의를 기울이고 매년 피펫을 교정하고, 로딩 전에 표준 PCR 스트립을 적극적으로 혼합하고, 효율적인 워크플로우를 촉진하기 위해 소모품을 신중하게 구성하십시오. 두 대의 기계를 사용하고 한 플레이트를 사용하면 지속적으로 더 높은 CV를 출력하는 것으로 관찰되면 문제를 개선할 수 있는 잠재적인 방법으로 기계를 서비스해야 합니다. 제조업체의 QC 플레이트는 Thermocycler의 성능을 평가하기 위해 플레이트에서 정기적으로 실행되어야 합니다.
위에서 권장하는 단계를 적용한 후에도 문제가 지속되면 다음 단계를 사용하여 프로토콜을 문제 해결할 수 있습니다. 모든 시약이 분량된 시기와 관련 만료 날짜를 기록하면 불가피하게 문제가 발생할 때 문제 해결 프로세스를 간소화하는 데 도움이 될 수 있습니다. 문제 해결 프로세스의 중요한 부분은 한 번에 하나의 시약만 조정하여 플레이트가 QC 기준을 통과하지 못하는 구체적인 원인을 파악하고 문제의 가장 저렴한 시약부터 시작하는 것입니다. 예를 들어, 텔로미어와 단일 복제 유전자의 효율이 모두 낮은 경우 DTT, dNTP 또는 SYBR 분취액과 같은 공유 시약이 앰플리콘 특이적 프라이머보다 원인일 가능성이 더 높습니다. 표시된 가격에서 DTT, dNTP 순으로 새로운 분취액을 테스트해야 하며, 마지막으로 문제가 지속되면 새로운 SYBR 분취액을 테스트해야 합니다. 반대로, 앰플리콘(텔로미어 또는 단일 복제 유전자) 중 하나만 효율이 낮은 경우 어려움의 원인은 프라이머 중 하나일 가능성이 더 높습니다. 그림 8에 제시된 용융 곡선 피크의 해석은 문제 해결을 위한 핵심 정보의 원천이 될 수 있습니다. 두 개의 용융 곡선 피크를 시각화하면 특정 샘플의 잠재적 문제를 식별하는 데 사용할 수 있는데, 이는 개별 문제 샘플이 표준물질 또는 나머지 분석 샘플에서 나타나는 피크의 일반적인 추세와 차별화되기 때문입니다. 용융 곡선은 주어진 앰플리콘의 피크가 다른 앰플리콘보다 체계적으로 덜 선명한 경우 특정 프라이머의 문제를 진단하는 데에도 사용할 수 있습니다.
이 원고는 공중 보건 연구에 광범위하게 적용할 수 있는 TL 측정을 위한 MMqPCR 분석을 성공적으로 설정하는 방법을 자세히 설명하고, 이 효율적이고 비용 효율적인 방법의 접근성과 신뢰성을 높이기 위해 QC 및 문제 해결을 위한 주요 권장 사항을 소개합니다.
공개
저자는 밝힐 것이 없습니다.
감사의 말
저자들은 이 연구를 가능하게 해준 텔로미어 연구 네트워크 자문 위원회(Telomere Research Network Advisory Committee)와 국립 노화 연구소(National Institute on Aging)/국립 환경 보건 과학 연구소(National Institute of Environmental Health Sciences)의 자금 지원(U24 AG066528 및 U24 AG066528-S1)에 감사를 표합니다.
자료
| Name | Company | Catalog Number | Comments |
| 0.5mL Tubes | USA Scientific | 1605-0099 | Seal-Rite 0.5mL Microcentrifuge Tubes, Sterile Storage Temperature and Conditions: Room temperature |
| 1.5mL Tubes | USA Scientific | 1615-5599 | Seal-Rite 1.5mL Microcentrifuge Tubes, Sterile Storage Temperature and Conditions: Room temperature |
| 100mM DTT | In House | Not Applicable | Made with stock DTT, diluted sodium acetate, and PCR Grade H2O Storage Temperature and Conditions: minus 20 °C |
| 15mL Tubes | Thermo Fisher | 14-959-53A | Corning 352196 Falcon 15mL Conical Centrifuge Tubes Storage Temperature and Conditions: Room temperature |
| 1M MgCl2 | Thermo Fisher | 50152107 | Biotang Inc 1M MgCl2 1M Magnesium Chloride Solution, Prepared in 18.2 Megohms Water and Filtered through 0.22 Micron Filter Storage Temperature and Conditions: 4 °C |
| 1x Gold Buffer | In House | Not Applicable | 10X Gold Buffer diluted with PCR Grade H2O Storage Temperature and Conditions: minus 20 °C |
| 25mM dNTPs | New England BioLabs | N0446S | Deoxynucleotide Solution Set Storage Temperature and Conditions: minus 20 °C |
| 5mL Tubes | Thermo Fisher | 3391276 | Argos Technologies Microcentrifuge Tubes – 5mL Storage Temperature and Conditions: Room temperature |
| 96 Well Plate | Bio-Rad | HSP9601 | Hard-Shell 96-Well PCR Plates, Low Profile, Thin Wall, Skirted, White / Clear Storage Temperature and Conditions: Room temperature |
| Aluminum Foil | Office Depot | 3489072 | Reynolds Wrap Stanard Aluminum Foil Roll, 12" x 75', Silver Storage Temperature and Conditions: Room temperature |
| AmpliTaq Gold Kit – Polymerase and Buffer | Thermo Fisher | 4311806 | AmpliTaq Gold DNA Polymerase with Gold Buffer and MgCl2 (MgCl2 in this kit is not used), 10X Gold Buffer, 2.5U AmpliTaq Gold Polymerase Storage Temperature and Conditions: minus 20 °C |
| Betaine | Thermo Fisher | AAJ77507AB | Betaine, 5M Solution, Molecular Biology Grade, Ultrapure, 10mL Storage Temperature and Conditions: minus 20 °C |
| Big Tube Rack | Thermo Fisher | 344817 | Fisherbrand 4-Way Tube Rack Storage Temperature and Conditions: Room temperature |
| CFX Maestro Software | Bio-Rad | 12004110 | Software for real-time PCR plate setup, data collection, statistics, and graphiing of results Storage Temperature and Conditions: Room temperature |
| CFX96 Optical Reaction Module for Real-Time PCR Systems with Starter Package | Bio-Rad | 1845096 | 96-well optical module for real-time PCR Storage Temperature and Conditions: Room temperature |
| DTT | Fisher Scientific | AAJ1539706 | Dithiothreitol, >99.5+ Molecular Biology Grade, 5 g Storage Temperature and Conditions: minus 20 °C |
| ELIMINase | Fisher Scientific | 04-355-32 | ELIMINase Laboratory Decontaminant Storage Temperature and Conditions: Room temperature |
| HEPA Filter | USA Scientific | Replacement Filters | High-Efficiency Particulate Air Filter for AirClean Workstations Storage Temperature and Conditions: Room temperature |
| Kimwipes | Thermo Fisher | 06666A | Kimberly-Clark Professional Kimtech Science Kimwipes Delicate Task Wipers, 1-Ply Storage Temperature and Conditions: Room temperature |
| Loading Trough | Thermo Fisher | 14387069 | Thermo Scientific Matrix Reagent Reservoirs Storage Temperature and Conditions: Room temperature |
| Microsoft Excel | Microsoft | Not Applicable | Microsoft 365 package, Excel software application Storage Temperature and Conditions: Room temperature |
| Mini Centrifuge | Genesee Scientific | 31-500B | Poseidon 31-500B Mini Centrifuge, Blue Lid Storage Temperature and Conditions: Room temperature |
| PCR Grade H2O | Thermo Fisher | AM9937 | Nuclease-Free Water (not DEPC-Treated) Storage Temperature and Conditions: Room temperature |
| PCR Hood | USA Scientific | 4263-2588 | Nucleic Acid Workstation with HEPA Filtration, AirClean Systems Combination PCR Workstation Storage Temperature and Conditions: Room temperature |
| PCR Strips | Thermo Fisher | AB0776 | Low Profile Tubes and Flat Caps, Strips of 8 Storage Temperature and Conditions: Room temperature |
| PCR Tube Rack | Thermo Fisher | 344820 | Fisherbrand 96-Well PCR Tube Rack Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Multichannel) | Ranin | 17005860 | Pipette Tips SR LTS 20µL F 960A/5, 20µL Maximum Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1181-3850 | 10µL Graduated TipOne RPT Filter Tips Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1180-1850 | 20µL Beveled TipOne RPT Filter Tips Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1111-0880 | 200µL Natural TipOne Pipette Tips in Racks Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1111-2890 | 1000µL Natural Graduated TipOne Pipette Tips in Racks Storage Temperature and Conditions: Room temperature |
| Pipettors (Multichannel) | Ranin | 17013802 | Pipet-Lite Multi Pipette L8-10XLS, 0.5 to 10µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Multichannel) | Ranin | 17013803 | Pipet-Lite Multi Pipette L8-20LS+, 2 to 20µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F144802G | Gilson Pipetman Classic Pipets, 1 to 10µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F123600 | Gilson Pipetman Classic Pipets, 2 to 20µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F123601 | Gilson Pipetman Classic Pipets, 20 to 200µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F123602 | Gilson Pipetman Classic Pipets, 200 to 1000µL Storage Temperature and Conditions: Room temperature |
| Plate Sealing Film | Bio-Rad | MSB1001 | Microseal “B” PCR Plate Sealing Film, Adhesive, Optical Storage Temperature and Conditions: Room temperature |
| Plate Spinner | Thermo Fisher | 14-100-141 | Fisherbrand Mini Plate Spinner Centrifuge, 230 V Storage Temperature and Conditions: Room temperature |
| Pre-Hood Filter | USA Scientific | 4235-3724 | Prefilter for AirClean Systems Workstations Storage Temperature and Conditions: Room temperature |
| R Software | The R Project for Statistical Computing | Not Applicable | R version 4.2.2 Storage Temperature and Conditions: Room temperature |
| Scale | Thermo Fisher | 01-922-329 | OHAUS 30430060 PR Series Analytical Balance, 62g Capacity Storage Temperature and Conditions: Room temperature |
| Scissors | Office Depot | 458612 | Office Depot Brand Scissors, 8”, Straight, Black, Pack of 2 Storage Temperature and Conditions: Room temperature |
| Sharpies | Sharpie | 2151734 | Brush Twin Permanent Markers, Black Storage Temperature and Conditions: Room temperature |
| Single Copy Gene Forward Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Single Copy Gene Reverse Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Small Tube Rack | Thermo Fisher | 21-402-17 | Thermo Fisher 8601 Reversible Microtube Racks with Lid Storage Temperature and Conditions: Room temperature |
| Sodium Acetate | Thermo Fisher | J63560.EQE | 3M NaOAc pH 5.2 Storage Temperature and Conditions: Room temperature |
| Stainless Steel Spatula | Thermo Fisher | 3990240 | Bel-Art SP Scienceware Stainless-Steel Sampling Spoon and Spatula Storage Temperature and Conditions: Room temperature |
| SYBR Green | Thermo Fisher | S7563 | SYBR Green I Nucleic Acid Gel Stain – 10,000X Concentrate in DMSO Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Syringe Filters | Fisher Scientific | 09-927-55A | GD/X 25 mm Sterile Syringe Filter, cellulose acetate filtration medium, 0.2 μm Storage Temperature and Conditions: Room temperature |
| Syringes | Thermo Fisher | 148232A | BD Luer-Lok Disposable Syringes without Needles, 10mL Storage Temperature and Conditions: Room temperature |
| TE Buffer | Fisher Scientific | BP2474100 | TE Buffer, Tris-EDTA, 1X Solution, pH 7.6, Molecular Biology, Fisher BioReagents Storage Temperature and Conditions: Room temperature |
| Telomere Forward Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Telomere Reverse Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| UV Light | USA Scientific | 4288-2540 | UV Light Bulb for Workstations Storage Temperature and Conditions: Room temperature |
| Vortex | Thermo Fisher | 14-955-151 | Fisherbrand Mini Vortex Mixer, 115 V, 50/60 Hz Storage Temperature and Conditions: Room temperature |
| Weigh Boat | Thermo Fisher | 01-549-752 | Fisherbrand Sterile Hexagonal Weighing Boat, 10mL Storage Temperature and Conditions: Room temperature |
참고문헌
- Hemann, M., Strong, M., Hao, L., Greider, C. The shortest telomere, not average telomere length, is critical for cell viability and chromosome stability. Cell. 107 (1), 67-77 (2001).
- Xu, L., Li, S., Stohr, B. A. The role of telomere biology in cancer. Ann Rev Pathol: Mech Dis. 8, 49-78 (2013).
- Lindrose, A., Drury, S. Minimum reporting recommendations for pcr-based telomere length measurement. Telomere Research Network. , (2020).
- Verhulst, S., et al. Commentary: The reliability of telomere length measurements. Int J Epidemiol. 44 (5), 1683-1686 (2015).
- Aubert, G., Hills, M., Lansdorp, P. M. Telomere length measurement-caveats and a critical assessment of the available technologies and tools. Mutat Res. 730 (1-2), 59-67 (2012).
- Mundstock, E., et al. Effect of obesity on telomere length: Systematic review and meta-analysis. Obesity. 23 (11), 2165-2174 (2015).
- Haycock, P. C., et al. Leucocyte telomere length and risk of cardiovascular disease: Systematic review and meta-analysis. BMJ. 349, 4227(2014).
- Astuti, Y., Wardhana, A., Watkins, J., Wulaningsih, W. Cigarette smoking and telomere length: A systematic review of 84 studies and meta-analysis. Environ Res. 158, 480-489 (2017).
- Ridout, K. K., Ridout, S. J., Price, L. H., Sen, S., Tyrka, A. R. Depression and telomere length: A meta-analysis. J Affect Disord. 191, 237-247 (2016).
- Wang, Q., Zhan, Y., Pedersen, N. L., Fang, F., Hägg, S. Telomere length and all-cause mortality: A meta-analysis. Ageing Res Rev. 48, 11-20 (2018).
- Hu, R., Hua, X. G., Jiang, Q. C. Associations of telomere length in risk and recurrence of prostate cancer: A meta-analysis. Andrologia. 51 (7), e13304(2019).
- Schneider, C. V., et al. Association of telomere length with risk of disease and mortality. JAMA Inter Med. 182 (3), 291-300 (2022).
- Deng, Y., et al. Telomere length and the risk of cardiovascular diseases: A mendelian randomization study. Front Cardiovasc Med. 9, 1012615(2022).
- D'mello, M. J., et al. Association between shortened leukocyte telomere length and cardiometabolic outcomes: Systematic review and meta-analysis. Circulation: Cardiovasc Gene. 8 (1), 82-90 (2015).
- Wilbourn, R. V., et al. The relationship between telomere length and mortality risk in non-model vertebrate systems: A meta-analysis. Philos Trans R Soc Lond B Biol Sci. 373 (1741), 20160447(2018).
- Gardner, M., et al. Gender and telomere length: Systematic review and meta-analysis. Exp Gerontol. 51, 15-27 (2014).
- Lai, T. -P., Wright, W. E., Shay, J. W. Comparison of telomere length measurement methods. Philos Trans R Soc Lond B Biol Sci. 373 (1741), 20160451(2018).
- Cawthon, R. Telomere measurement by quatitative pcr. Nuc Acid Res. 30 (10), e47-e53 (2002).
- Cawthon, R. M. Telomere length measurement by a novel monochrome multiplex quantitative pcr method. Nuc Acid Res. 37 (3), e21-e21 (2009).
- Lindrose, A. R., et al. Method comparison studies of telomere length measurement using qpcr approaches: A critical appraisal of the literature. PLoS One. 16 (1), e0245582(2021).
- Lin, J., et al. Effects of DNA extraction, DNA integrity, and laboratory on the precision of qpcr-based telomere length measurement - a multi-lab impartial study. bioRxiv. , (2022).
- Wilfinger, W. W., Mackey, K., Chomczynski, P. Effect of ph and ionic strength on the spectrophotometric assessment of nucleic acid purity. Biotechniques. 22 (3), 474-481 (1997).
- Boesenberg-Smith, K. A., Pessarakli, M. M., Wolk, D. M. Assessment of DNA yield and purity: An overlooked detail of pcr troubleshooting. Clin Microbiol Newsletter. 34 (1), 1-6 (2012).
- Harley, C. B., Futcher, A. B., Greider, C. W. Telomeres shorten during ageing of human fibroblasts. Nature. 345 (6274), 458-460 (1990).
- Kimura, M., et al. Measurement of telomere length by the southern blot analysis of terminal restriction fragment lengths. Nat Protoc. 5 (9), 1596-1607 (2010).
- Ye, Q., et al. Telomere length and chronological age across the human lifespan: A systematic review and meta-analysis of 414 study samples including 743,019 individuals. Ageing Res Rev. 90, 102031(2023).
- Axelrad, M. D., Budagov, T., Atzmon, G. Telomere length and telomerase activity; a yin and yang of cell senescence. J Vis Exp. (75), e50246(2013).
- Liu, M., et al. Immune-mediated inflammatory diseases and leukocyte telomere length: A mendelian randomization study. Front Genetics. 14, 1129247(2023).
- Van Ockenburg, S., et al. Stressful life events and leukocyte telomere attrition in adulthood: A prospective population-based cohort study. Psychol Med. 45 (14), 2975-2984 (2015).
- Zong, Z. Q., et al. Ambient air pollution exposure and telomere length: A systematic review and meta-analysis. Public Health. 215, 42-55 (2023).
- Tang, L., Li, D., Wang, J., Su, B., Tian, Y. Ambient air pollution, genetic risk and telomere length in uk biobank. J Expo Sci Environ Epidemiol. , (2023).
- Aubert, G., Hills, M., Lansdorp, P. Telomere length measurement-caveats and a critical assessment of the available technologies and tools. Mutat Res. 730 (1), 59-67 (2012).
- Lin, J., et al. Systematic and cell type-specific telomere length changes in subsets of lymphocytes. J Immunol Res. 2016, 5371050(2016).
- Needham, B. L., et al. Socioeconomic status, health behavior, and leukocyte telomere length in the national health and nutrition examination survey, 1999-2002. Soc Sci Med. 85, 1-8 (2013).
- Verhulst, S. Improving comparability between qpcr-based telomere studies. Mol Ecol Res. 20 (1), 11-13 (2020).
- Dagnall, C. L., et al. Effect of pre-analytic variables on the reproducibility of qpcr relative telomere length measurement. PloS one. 12 (9), e0184098(2017).
- Lin, J., Smith, D. L., Esteves, K., Drury, S. Telomere length measurement by qpcr-summary of critical factors and recommendations for assay design. Psychoneuroendocrinology. 99, 271-278 (2019).
- Wolf, S. E., et al. Cross-tissue comparison of telomere length and quality metrics of DNA among individuals aged 8 to 70 years. PLOS One. 19 (2), e0290918(2024).
- RoDer, B., FruHwirth, K., Vogl, C., Wagner, M., Rossmanith, P. Impact of long-term storage on stability of standard DNA for nucleic acid-based methods. J Clin Microbiol. 48 (11), 4260-4262 (2010).
- Dagnall, C., et al. Effect of pre-analytic variables on the reproducibility of qpcr relative telomere length measurement. PLoS One. 12 (9), e0184098(2017).
- Shao, W., Khin, S., Kopp, W. C. Characterization of effect of repeated freeze and thaw cycles on stability of genomic DNA using pulsed field gel electrophoresis. Biopreserv Biobank. 10 (1), 4-11 (2012).
- Institute for Statistics and Mathematics of WU. The Comprehensive R Archive Network. , Available from: https://cran.r-project.org/ (2024).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biol Rev. 85 (4), 935-956 (2010).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유