Method Article
Misurazione quantitativa della lunghezza dei telomeri PCR multiplex monocromatica
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui presentiamo un protocollo per la misura della lunghezza relativa dei telomeri (TL) utilizzando il saggio di reazione a catena della polimerasi quantitativa multiplex monocromatica (MMqPCR). Il test MMqPCR è un metodo ripetibile, efficiente ed economico per misurare la TL dal DNA umano in studi basati sulla popolazione.
Abstract
I telomeri sono strutture ribonucleoproteiche all'estremità di tutti i cromosomi eucariotici che proteggono il DNA dai danni e preservano la stabilità dei cromosomi. La lunghezza dei telomeri (TL) è stata associata a varie esposizioni, processi biologici e risultati di salute. Questo articolo descrive il protocollo di saggio della reazione a catena della polimerasi quantitativa multiplex monocromatica (MMqPCR) condotto di routine nel nostro laboratorio per misurare la TL media relativa dal DNA umano. Esistono diversi metodi di misurazione TL basati sulla PCR, ma il protocollo specifico per il metodo MMqPCR presentato in questa pubblicazione è ripetibile, efficiente, economico e adatto per studi basati sulla popolazione. Questo protocollo dettagliato delinea tutte le informazioni necessarie agli investigatori per stabilire questo test nel loro laboratorio. Inoltre, questo protocollo fornisce passaggi specifici per aumentare la riproducibilità della misurazione della TL con questo test, definita dal coefficiente di correlazione intraclasse (ICC) su misurazioni ripetute dello stesso campione. L'ICC è un fattore critico nella valutazione della potenza attesa per una specifica popolazione di studio; pertanto, la segnalazione di ICC specifici per coorte per qualsiasi test di TL è un passo necessario per migliorare il rigore complessivo degli studi di TL basati sulla popolazione. I risultati di esempio che utilizzano campioni di DNA estratti da cellule mononucleate del sangue periferico dimostrano la fattibilità della generazione di dati TL altamente ripetibili utilizzando questo protocollo MMqPCR.
Introduzione
I telomeri sono complessi protettivi che si trovano all'estremità di tutti i cromosomi eucariotici, composti da sequenze di DNA ripetitive altamente conservate e proteine associate. Il telomero protegge l'integrità del DNA, preservando la stabilità cromosomica. L'accorciamento progressivo dei telomeri si verifica nelle cellule in divisione a causa di una sintesi incompleta del DNA a filamento ritardato, danni al DNA e altri fattori 1,2. L'aumento delle prove a sostegno della lunghezza dei telomeri (TL) come biomarcatore dell'invecchiamento e delle malattie legate all'età nel corso della vita umana è stato accompagnato da un aumento dei tipi di saggi di misurazione della TL utilizzati per valutare il ruolo della TL negli studi sull'esposizione, la malattia e la salute umana 3,4,5. Le meta-analisi hanno riportato associazioni di TL con la mortalità complessiva, le esposizioni ambientali e gli esiti di salute, tra cui cancro, malattie cardiovascolari e diabete 6,7,8,9,10,11,12,13 . Queste meta-associazioni derivano da studi che utilizzano una delle oltre due dozzine di diverse metodologie di misurazione del TL in cui i punti di forza delle associazioni tendono a variare tra le diverse metodologie 14,15,16. La selezione del metodo di misurazione TL ottimale per uno studio di ricerca è un passo cruciale per garantire risultati accurati, poiché ogni metodo possiede i propri vantaggi e svantaggi 5,17.
A causa del costo relativamente basso dei reagenti, della rapida inversione di tendenza dei saggi, della scalabilità e del minore fabbisogno iniziale di DNA, le tecniche di misurazione TL basate sulla PCR sono spesso utilizzate preferenzialmente quando si conducono studi con grandi popolazioni di campioni, studi con accesso limitato a campioni con alte concentrazioni di DNA o studi che danno priorità a un'elevata produttività. Il primo metodo di misurazione della TL basato sulla PCR, la reazione a catena della polimerasi quantitativa singleplex (qPCR) originariamente sviluppata da Richard Cawthon, utilizza il rapporto tra i segnali di fluorescenza del telomero (T) e l'amplificazione del gene a copia singola (S), eseguita su piastre PCR separate18. In questo approccio, primer complementari alle sequenze ripetute di DNA dei telomeri (T) vengono utilizzati per amplificare il contenuto totale di DNA telomerico nel campione e quantificato mediante rilevamento del reporter fluorescente SYBR green. Allo stesso modo, per quantificare il numero di copie del genoma vengono utilizzati primer complementari a una regione intergenica di un gene a copia singola (S) conservato. Queste due stime sono quantificate rispetto a una curva standard del DNA genomico utilizzata in tutti i saggi in un progetto per controllare la variazione da piastra a piastra. Dividendo il DNA telomerico totale (T) per il numero di copie del genoma singolo (S) si ottiene il rapporto T/S, una misura relativa senza unità che rappresenta il contenuto telomerico medio per cellula per un singolo campione di DNA18,19. Pertanto, il rapporto T/S non è una misura specifica della lunghezza funzionale; tuttavia, coerentemente con le norme della letteratura, utilizziamo il termine TL medio per campione in tutto questo protocollo.
Questo metodo è stato avanzato nel 2009 quando Richard Cawthon ha descritto il test qPCR multiplex monocromatico (MMqPCR) come un approccio per ridurre potenzialmente la variabilità del rapporto T/S rispetto al metodo qPCR singleplex originale19. Il test MMqPCR possiede i vantaggi del test qPCR con l'ulteriore vantaggio di misurare i segnali T e S all'interno dello stesso pozzetto di reazione utilizzando un fluoroforo di segnalazione, riducendo così l'errore relativo alla qPCR singleplex e ottenendo una maggiore precisione e riproducibilità19. Inoltre, questo metodo multiplex riduce potenzialmente i costi e migliora la produttività, poiché è necessaria la metà delle reazioni rispetto al saggio singleplex19.
Dati i vantaggi della misurazione MMqPCR TL, questo metodo è adatto per studi basati sulla popolazione delle associazioni di TL con esposizioni, esiti sanitari e processi biologici. Tuttavia, l'avvio del metodo può essere impegnativo. Per affrontare queste sfide, descriviamo in dettaglio il protocollo di misura MMqPCR TL utilizzato nel nostro laboratorio, evidenziando i passaggi chiave implementati per aumentare la precisione del saggio, ridurre il rischio di contaminazione e migliorare la ripetibilità.
Inoltre, questo protocollo delinea i passaggi per la pulizia dei dati e il calcolo del coefficiente di correlazione intraclasse (ICC), un'importante misura statistica della riproducibilità delle misurazioni TL2. Con i nostri risultati rappresentativi, dimostriamo la capacità di generare ICC elevati utilizzando questo protocollo. Inoltre, identifichiamo le fasi di controllo qualità (QC) e di risoluzione dei problemi che dovrebbero ridurre la variazione nelle misurazioni TL e aumentare l'ICC risultante. Grazie all'elevata ripetibilità, efficienza ed economicità di questo metodo, la misurazione MMqPCR TL è ideale per la ricerca epidemiologica TL. La Figura 1 fornisce una panoramica visiva del metodo MMqPCR come descritto in questo protocollo.
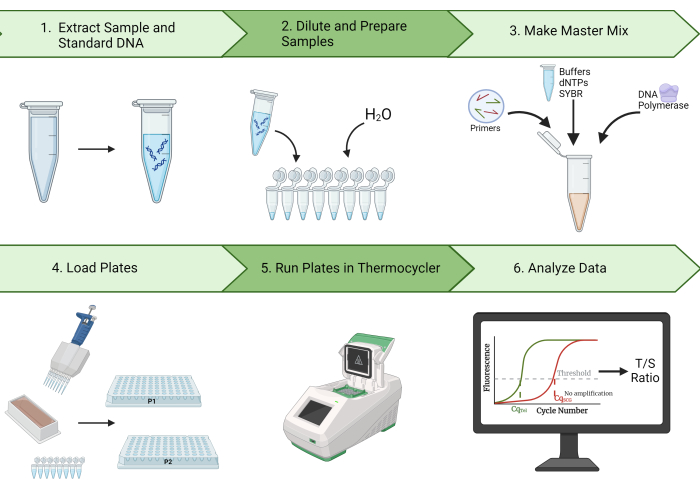
Figura 1: Panoramica del metodo. Un'ampia panoramica del metodo di reazione a catena quantitativa della polimerasi multiplex monocromatica per misurare la lunghezza dei telomeri. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
Questa ricerca è stata condotta nel rispetto delle linee guida istituzionali. Questo protocollo descrive il test MMqPCR condotto a una profondità di misurazione di triplicati duplicati, ovvero misurazioni triplicate di ciascun campione ripetute su piastre duplicate, implementate utilizzando due termociclatori contemporaneamente per aumentare la produttività. L'uso di lastre duplicate è una considerazione fondamentale nell'implementazione di questo protocollo per ottenere un'elevata riproducibilità, come indicato da ICC elevati. Sebbene sia possibile utilizzare meno repliche, l'impatto sull'ICC, e di conseguenza sulla potenza e sulla dimensione del campione necessaria, deve essere attentamente considerato e misurato con ogni singola coorte da ogni laboratorio20,21. Se è disponibile un solo termociclatore, si consiglia di mantenere la profondità di misurazione (ad esempio, triplicare i duplicati) e di eseguire 2 piastre in sequenza.
1. Condizioni di preparazione e conservazione delle scorte
- Per tutti i materiali e le attrezzature utilizzate per questo protocollo, vedere la Tabella dei materiali. Inoltre, per i reagenti specifici della master mix e le quantità utilizzate per il test MMqPCR, vedere la Tabella 1. Se si eseguono due piastre in sequenza, dimezzare il volume della miscela master mantenendo la concentrazione, assicurandosi che le stesse aliquote di reagenti vengano utilizzate su piastre sequenziali dello stesso saggio.
NOTA: Tutti i reagenti stock devono essere preparati in anticipo e i reagenti aliquotati devono essere completamente scongelati prima dell'uso. - Aliquotare e conservare 1x Tris-EDTA, pH 7,6 (TE) in provette da 5 mL in volumi da 3 mL a temperatura ambiente per un massimo di 2 anni.
- Aliquotare e conservare 5 M di betaina in provette da 5 mL in volumi da 1.280 μL a -20 °C per un massimo di 1 anno.
- Aliquotare e conservare SYBR Green in volumi da 3 μL in singole provette per PCR a -20 °C per un massimo di 2 anni, ricoperte da un foglio e minimamente esposte alla luce.
- Conservare la DNA polimerasi nelle provette originali a -20 °C per un massimo di 1 anno.
- Aliquotare e conservare 10 tamponi polimerasi in provette da 1,5 mL in volumi da 660 μL a -20 °C per un massimo di 1 anno.
- Per diluizioni 1x del tampone 10x della polimerasi, aggiungere 9,9 μL di tampone 10x a 89,1 μL di PCR grado H2O in provette da 0,5 mL; conservare a -20 °C per un massimo di 1 anno.
- Aliquotare e conservare 1 M di MgCl2 in provette da 0,5 mL in volumi da 70 μL a 4 °C al riparo dalla luce per un massimo di 1 anno.
- Conservare i primer liofilizzati a copia diretta e inversa del telomero (T) e dell'oligonucleotide a copia singola (S) a temperatura ambiente, lontano dalla luce. Le quattro sequenze specifiche di primer oligonucleotidici sono presentate nella Tabella 2.
NOTA: Il gene a copia singola in questo protocollo è l'albumina; se si sceglie un gene a copia singola diverso, potrebbe essere necessario regolare le concentrazioni del primer per garantire un'efficienza PCR accettabile.- Ricostituzione del primer: Agitare ogni provetta di primer liofilizzata per 10 s e poi posizionare le provette in una mini centrifuga per 5 s per assicurarsi che il pellet liofilizzato si trovi sul fondo della provetta prima di aggiungere 1x tampone TE.
- Controllare ogni singola provetta per verificare la concentrazione di nM ([nM]= alfa) e reidratare ogni provetta aggiungendo 1x TE pari a 10x del valore alfa in μL (ad esempio, se la concentrazione di nM sulla provetta è di 24,6 nM, aggiungere 246 μL di 1x TE alla provetta di innesco) per creare una soluzione da 100 μM di ciascun primer.
- Aliquotare separatamente i primer dei telomeri diretti e inversi in volumi da 16 μl e i primer dell'albumina diretta e inversa separatamente in volumi da 11 μl. Conservare tutti e quattro i tipi di aliquote di primer a -20 °C per un massimo di 6 mesi.
- Miscela dNTP: Vorticare 4-8 provette di ciascuno dei quattro tipi di dNTPS di soluzioni stock da 25 mM (cioè 16-32 provette in totale) per 10 s seguite da una rapida centrifuga a temperatura ambiente. Posizionare le provette in una distribuzione uniforme nelle posizioni delle provette nella mini centrifuga, quindi chiudere il coperchio per consentire alla macchina di raggiungere la massima velocità, girando per circa 5 s.
- In una provetta da 5 mL, aggiungere 200-250 μL da ciascuna provetta, assicurandosi che vengano aggiunte parti uguali di ciascuno dei quattro tipi di dNTP, e agitare la miscela di dNTP.
- Aliquotare 210 μL di miscela in provette da 0,5 mL e conservare a -20 °C per un massimo di 1 anno.
- Conservare il DTT a -20 °C e l'acetato di sodio 3 M a temperatura ambiente.
- Soluzione DTT: Misurare 0,1545 g DTT in una pesa tarata su una bilancia utilizzando una spatola in acciaio inossidabile.
ATTENZIONE: Il ditiotreitolo (DTT) è un reagente pericoloso. Il DTT è dannoso se ingerito, provoca irritazione della pelle e può causare gravi danni agli occhi. Le persone devono indossare guanti protettivi, protezione per gli occhi e protezione per il viso durante la manipolazione della DTT. Inoltre, le persone dovrebbero evitare di respirare i vapori del DTT, lavorare nel cappuccio della PCR quando maneggiano il DTT ed evitare l'esposizione prolungata o ripetuta. - Preparare 10 mL di acetato di sodio 0,01 M aggiungendo 33,33 μL di acetato di sodio 3 M e 9,967 μL di grado PCR H2O in una provetta da 15 mL e pozzetto vortex. Aggiungere il DTT misurato nella provetta da 15 ml di acetato di sodio 0,01 M. Trasferire circa 100 μl della soluzione di acetato di sodio 0,01 M nella navicella di pesa per raccogliere il DTT residuo. Pipettare nuovamente i 100 μl nella provetta da 15 mL. Una volta che tutto il DTT è nella soluzione, agitare il tubo fino a quando non si dissolve completamente.
- Versare il contenuto della provetta da 15 mL in una mangiatoia di carico, aspirare l'intera soluzione in una siringa di plastica da 10 mL da un'estremità della mangiatoia di carico. Collegare un filtro sterile per siringa in acetato di cellulosa da 0,2 μm da 25 mm all'estremità della siringa riempita, lasciare saturare completamente per 1 minuto e gocciolare lentamente la soluzione in una nuova provetta da 15 ml spingendo leggermente lo stantuffo della siringa verso il basso, assicurandosi che l'intera soluzione goccioli attraverso il filtro nella nuova provetta da 15 ml.
- Aliquotare la soluzione DTT in volumi da 200 μL in provette da 0,5 mL e conservare a -20 °C per un massimo di 6 settimane.
- Soluzione DTT: Misurare 0,1545 g DTT in una pesa tarata su una bilancia utilizzando una spatola in acciaio inossidabile.
Tabella 1: Volumi finali e concentrazioni dei reagenti. Volumi e concentrazioni dei reagenti nelle singole aliquote, nella master mix e nei pozzetti di PCR. Clicca qui per scaricare questa tabella.
Tabella 2: Sequenze di primer di telomeri e oligonucleotidi genici a copia singola. Elenco delle sequenze di primer geniche a copia singola dei telomeri e delle albumine utilizzate nella metodologia. Clicca qui per scaricare questa tabella.
2. Estrazione del DNA genomico e preparazione del campione
- Estrazione di campioni analitici: eseguire un'estrazione genomica del DNA per i campioni in conformità con le linee guida del produttore utilizzando kit o metodi consolidati all'interno del laboratorio.
- Controllare la qualità del campione di DNA con uno spettrofotometro e il DNA a doppio filamento (dsDNA) utilizzando un fluorimetro. I campioni con rapporti medi 260/280 e 260/230 inaccettabili o una concentrazione di dsDNA inferiore alla rilevazione (<0,20 ng/μL) non devono essere analizzati per TL22,23. Inoltre, se la concentrazione di dsDNA è troppo alta per generare una lettura utilizzando il fluorimetro (>1000 ng/μL), diluire il campione e ripetere il test.
NOTA: Per i risultati qui presentati, i valori accettabili per la purezza degli acidi nucleici erano un rapporto 260/280 tra 1,6 e 2,0 e un rapporto 260/230 tra 2,0 e 2,2.
- Controllare la qualità del campione di DNA con uno spettrofotometro e il DNA a doppio filamento (dsDNA) utilizzando un fluorimetro. I campioni con rapporti medi 260/280 e 260/230 inaccettabili o una concentrazione di dsDNA inferiore alla rilevazione (<0,20 ng/μL) non devono essere analizzati per TL22,23. Inoltre, se la concentrazione di dsDNA è troppo alta per generare una lettura utilizzando il fluorimetro (>1000 ng/μL), diluire il campione e ripetere il test.
- Estrazione di campioni di controllo: eseguire un'estrazione di DNA genomico su un campione di controllo derivato dallo stesso materiale biologico dei campioni oggetto di analisi per un determinato progetto. Assicurarsi di estrarre una quantità sufficiente di DNA di controllo per tutte le piastre che si prevede di eseguire per l'intera coorte, in modo da utilizzare un unico standard di DNA per tutti i saggi.
- Controllare la qualità del campione di DNA di controllo mediante spettrofotometro e fluorimetro. Il campione di controllo deve avere rapporti medi accettabili di 260/280 e 260/230 e concentrazioni rilevabili di dsDNA22,23.
- Aliquotare il DNA di controllo a una concentrazione di 2 ng/μL in quantità da 150 μL in provette da 0,5 mL etichettate con il tipo e la data del campione. Conservare le aliquote di DNA di controllo a -20 °C per un massimo di 5 anni. Utilizzare un campione di DNA di controllo per tutte le piastre di tutti i campioni della stessa coorte o progetto di ricerca. Prepara queste aliquote di stock in anticipo.
- Preparazione dei campioni analitici: per i campioni che hanno una concentrazione iniziale di >5 ng/μL, creare aliquote di diluizione utilizzando un fattore di diluizione a numero intero, aggiungendo la quantità appropriata di campione di DNA estratto (File supplementare 1 MMqPCR Set Up Sheet, colonna F) e grado PCR H2O (File supplementare 1 MMqPCR Set Up Sheet, colonna G) per diluire la concentrazione compresa tra 2,5 e 5,0 ng/μL (File supplementare 1 Foglio di impostazione MMqPCR, colonna K). Non diluire i campioni con una concentrazione <5 ng/μL, tuttavia, aliquotare un volume sufficiente per analizzare il campione 3 volte utilizzando questo protocollo.
NOTA: Le aliquote di diluizione devono essere effettuate durante la preparazione del campione in modo da poter aggiungere 15 ng di DNA alla provetta per PCR corrispondente di ciascun campione. Le aliquote dei campioni consentono ai tecnici di evitare che inutili cicli di congelamento-scongelamento influiscano sull'integrità del DNA del campione di serie se il campione non supera i criteri di qualità e deve essere ripetuto. Il modello MMqPCR di esempio che si trova nel file supplementare 1 può aiutare a identificare il fattore di diluizione e i volumi corretti per preparare le aliquote di diluizione (file supplementare 1, foglio di impostazione MMqPCR, colonne E-I).- Riempire le strisce PCR con i campioni A1-8 nella striscia PCR A, i campioni B1-8 nella striscia PCR B e i campioni C1-8 nella striscia PRC C, come descritto nel foglio di configurazione MMqPCR del file supplementare 1 ed elencato nella tabella 3 con quantità di campione adeguatamente diluite (file supplementare 1 , foglio di configurazione MMqPCR, colonna L) e quantità di grado PCR H2O (file supplementare 1 MMqPCR Set Up Sheet, Colonna M) per una quantità totale di 15 ng di DNA per provetta PCR e un volume totale di 75 μL. Metterlo da parte.
NOTA: I campioni in strisce PCR possono essere conservati durante la notte a 4 °C per essere piastrati il giorno successivo.
- Riempire le strisce PCR con i campioni A1-8 nella striscia PCR A, i campioni B1-8 nella striscia PCR B e i campioni C1-8 nella striscia PRC C, come descritto nel foglio di configurazione MMqPCR del file supplementare 1 ed elencato nella tabella 3 con quantità di campione adeguatamente diluite (file supplementare 1 , foglio di configurazione MMqPCR, colonna L) e quantità di grado PCR H2O (file supplementare 1 MMqPCR Set Up Sheet, Colonna M) per una quantità totale di 15 ng di DNA per provetta PCR e un volume totale di 75 μL. Metterlo da parte.
- Preparazione della curva standard: scongelare un'aliquota di DNA di controllo, agitare per 30 s, centrifugare per 5 s e aspirare l'intero volume (150 μl) nella prima provetta della striscia di provette PCR con curva standard (SC).
- Pipettare 70 μl di grado PCR H2O nella seconda e nell'ottava provetta della striscia SC PCR. Risospendere accuratamente il DNA di controllo nella prima provetta prima di aspirare 70 μl e dispensarlo nella seconda provetta per PCR. Attendere 30 s, quindi risospendere accuratamente la soluzione nella seconda provetta per PCR prima di aspirare 70 μl e dispensarla nella terza provetta per PCR. Ripetere l'operazione per le provette da tre a sette per creare una diluizione seriale di 2 volte di sette standard. La provetta finale deve contenere solo PCR di grado H2O per funzionare come controllo senza stampo (NTC). Mettere da parte la striscia SC PCR.
Tabella 3: Organizzazione dei campioni e standard di controllo in piastra. Posizione di tutti i campioni e gli standard su una piastra PCR a 96 pozzetti. Clicca qui per scaricare questa tabella.
3. Preparazione della miscela master MMqPCR
- Raccogliere le aliquote del reagente della miscela master elencate nella Tabella 1, ad eccezione della DNA polimerasi, e lasciarle raggiungere la temperatura ambiente all'interno della cappa per PCR. Aggiungere 1 μl dell'aliquota SYBR Green all'aliquota tampone 1x.
NOTA: Questa quantità esatta è necessaria per generare la corretta concentrazione di SYBR a causa delle limitazioni dell'accuratezza della pipetta durante il test, quindi anche se viene eseguita una sola piastra alla volta, questa quantità dovrebbe rimanere invariata. - Agitare tutte le aliquote per 10 s, centrifugare per 5 s, quindi aggiungere le seguenti quantità di reagenti alla provetta da 5 mL che contiene la betaina 5 M (ora la provetta di miscelazione master): 1.235,2 μL di grado PCR H2O, 640 μL del tampone 10x, 204,8 μL dei dNTP, 192 μL del DTT, 64 μL di MgCl2, 48 μL di SYBR Green / 1x tampone (dal passaggio 3.1), 14,4 μL di entrambi i primer dei telomeri e 9,6 μL di entrambi i primer di albumina, come elencato nella Tabella 1.
- Estrarre la DNA polimerasi dalla conservazione a -20 °C, agitare per 10 s, centrifugare per 5 s, quindi aggiungere lentamente 128 μl alla miscela master; riportare immediatamente la DNA polimerasi a -20 °C.
- Agitare la provetta master mix da 5 ml per 30 s.
4. Preparazione della piastra a 96 pozzetti
- Posizionare due piastre da 96 pozzetti, una vasca di caricamento e puntali per pipette multicanale all'interno della cappa per PCR. Etichettare ogni targa con il numero di targa (1 o 2). Ruotare la piastra uno (la piastra P1) di 180° in modo che i numeri delle colonne siano capovolti. Lasciare la piastra due (P2) rivolta verso il tecnico.
- Versare il contenuto della provetta di miscelazione master da 5 ml nella mangiatoia di caricamento. Utilizzare una punta per pipetta per raccogliere ed erogare la soluzione rimasta nella provetta.
- Caricare i puntali sulla pipetta multicanale, spingendo alla base di ciascun puntale per garantire una tenuta ermetica. Verificare che i puntali non abbiano alcun filtro all'interno della regione di uscita in cui verrà aspirata la miscela master; Se c'è un pezzo di filtro, sostituire la punta.
- Utilizzare il pipettaggio inverso per riempire le piastre con la miscela master viscosa, agitare la vaschetta di caricamento, quindi premere lo stantuffo della pipetta oltre il primo arresto, aspirando una quantità superiore a 15 μl nei puntali della pipetta, assicurandosi che i puntali si riempiano contemporaneamente con lo stesso volume. Quando si espelle la miscela master in una colonna, premere lo stantuffo fino al primo arresto, lasciando la miscela master in eccesso nei puntali delle pipette. Lasciando premuto lo stantuffo, immergere nuovamente i puntali nella vasca di caricamento e rilasciare lo stantuffo per riempire i puntali con altri 15 μl. Utilizzare gli stessi puntali per riempire entrambe le piastre.
- Riempire le piastre espellendo prima la miscela master in tutte le colonne pari o dispari, alternando le piastre (ad esempio, se il tecnico sceglie di iniziare con numeri di colonna dispari, la colonna 1 su P2 verrà riempita per prima, seguita dalla colonna 11 su P1, quindi dalla colonna 3 su P2, ecc.). Dopo che il tecnico ha riempito tutti i pozzetti della serie (pari o dispari) lavorando da sinistra a destra, fare le altre serie (pari o dispari) di colonne allo stesso modo. Vedere la Figura 2 per un grafico di questo processo.
NOTA: Questo tipo di riempimento delle piastre funziona per controllare gli effetti di posizione tra le piastre.
- Riempire le piastre espellendo prima la miscela master in tutte le colonne pari o dispari, alternando le piastre (ad esempio, se il tecnico sceglie di iniziare con numeri di colonna dispari, la colonna 1 su P2 verrà riempita per prima, seguita dalla colonna 11 su P1, quindi dalla colonna 3 su P2, ecc.). Dopo che il tecnico ha riempito tutti i pozzetti della serie (pari o dispari) lavorando da sinistra a destra, fare le altre serie (pari o dispari) di colonne allo stesso modo. Vedere la Figura 2 per un grafico di questo processo.
- Agitare le quattro strisce PCR chiuse contenenti i campioni e la curva standard, quindi farle girare ciascuna in una mini centrifuga per 5 s.
- Allineare le strisce PCR nel rack per provette PCR nell'ordine in cui verranno posizionate sulla piastra. Per P1 le prime 3 colonne sono campioni (striscia PCR A), le colonne 4-6 sono lo standard (striscia PCR S), le colonne 7-9 sono campioni (striscia PCR B) e le colonne 10-12 sono campioni (striscia PCR C; vedi Figura 3 e Tabella 3). Poiché P2 viene riempito ruotato di 180° rispetto al tecnico, le colonne saranno l'opposto.
- Ruotare entrambe le piastre di 180° in modo che i numeri delle colonne siano invertiti rispetto al tecnico per ciascuna piastra (ad esempio, se il tecnico ha affrontato l'etichetta per P2 in precedenza, ora sarà rivolto verso l'etichetta per P1 sul bordo della piastra).
- Impostare la pipetta multicanale su 10 μl e caricare i puntali della pipetta sulla pipetta multicanale come descritto al punto 4.3.
- Utilizzare la pipetta multicanale per risospendere le soluzioni, quindi eseguire il pipettaggio inverso come descritto nella fase 4.4 per aspirare ed erogare 10 μl dei campioni e delle soluzioni standard, iniziando con la striscia PCR A.
- Riempire le prime 3 colonne di ciascuna piastra utilizzando la striscia PCR A alternando le colonne in modo simile a quando la miscela master è stata posizionata nella piastra (cioè, pari o dispari).
- Riempi le tre colonne successive utilizzando la striscia PCR a curva standard. Per questa striscia, risospendere la 7aprovetta di diluizione standard (penultima), utilizzando una pipetta da 200 μl impostata a ~90 μl, prima di risospendere l'intera striscia utilizzando la pipetta multicanale.
NOTA: Il volume maggiore e la minore concentrazione di DNA in questa provetta spesso determinano misurazioni diverse tra i pozzetti della piastra PCR a causa della scarsa miscelazione. L'uso di un volume maggiore per miscelare questa provetta omogeneizza meglio la soluzione di DNA. - Riempire le tre colonne successive con la striscia per PCR B e le ultime tre con la striscia per PCR C. Riposizionare i puntali delle pipette tra una striscia per PCR e l'altra, riempiendo tutti i 96 pozzetti di entrambe le piastre con i campioni e lo standard di controllo corrispondenti, come descritto nella Figura 3.
- Una volta riempite le piastre, picchiettare delicatamente la piastra sul piano di lavoro per assicurarsi che il liquido sui lati dei pozzetti scorra verso il basso, quindi coprire le piastre con pellicole sigillanti. Usa la punta delle dita per premere su tutti i bordi della pellicola per garantire una tenuta ermetica.
- Mescolare le piastre facendo roteare sulla parte superiore della cappa per 30 s, quindi posizionare le piastre sigillate nella rotella per 2 minuti con le aperture del pozzetto rivolte verso il centro.
- Posizionare le piastre in un termociclatore, con i numeri delle colonne delle piastre in ordine leggibile. Utilizzare un fazzoletto pulito per pulire la parte superiore di ogni piastra prima di chiudere la parte superiore del termociclatore.
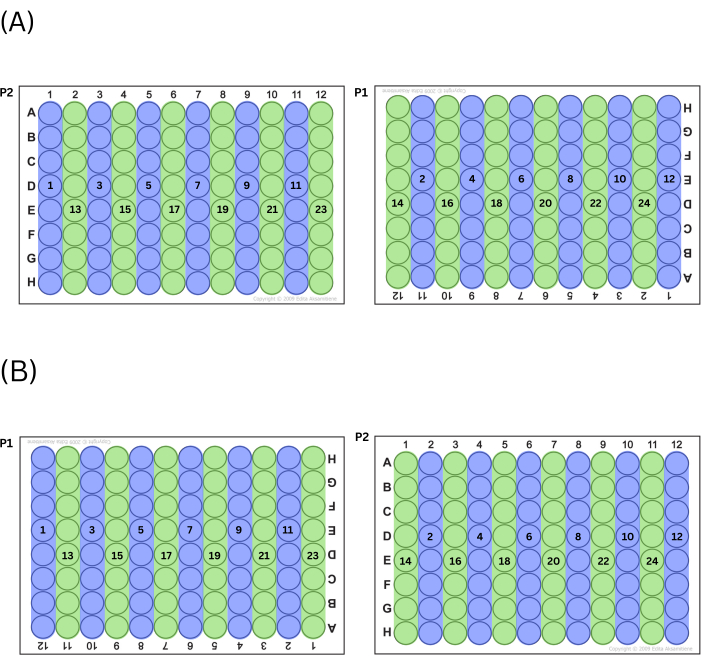
Figura 2: Processo di riempimento delle piastre. (A) Se si sceglie di riempire prima le colonne dispari, questo è l'ordine in cui vengono riempiti i pozzetti. (B) Se si sceglie di riempire prima le colonne pari, questo è l'ordine in cui i pozzetti vengono riempiti. Clicca qui per visualizzare una versione più grande di questa figura.
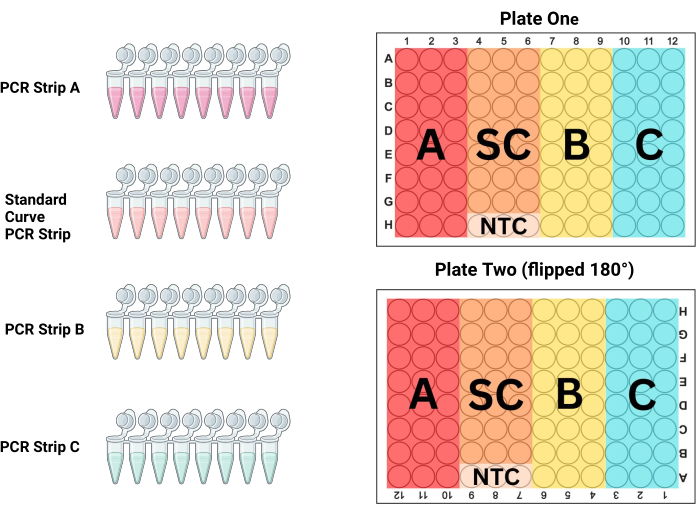
Figura 3: Layout della targa. Le strisce PCR A, B, C e la curva standard (SC) devono essere utilizzate per riempire tre colonne su ciascuna piastra per produrre triplicati duplicati di ciascun campione e diluizione standard. Questo diagramma mostra quale delle colonne deve essere riempita con ogni striscia. La piastra due viene capovolta di 180° (si noti che le intestazioni della colonna e della riga sono capovolte) prima del caricamento, ma la piastra viene riempita in modo identico alla piastra uno, eliminando potenziali errori di pipettaggio e controllando comunque gli effetti di posizione tra le piastre. Clicca qui per visualizzare una versione più grande di questa figura.
5. Termocilatura MMqPCR
- Mentre le piastre girano al punto 4.8.1, accendere il computer e i termociclatori. Aprire il software del termociclatore. Questo protocollo descrive l'uso del software CFX Maestro.
- Creare il protocollo di termociclo TL in conformità con il protocollo di termociclo MMqPCR19 (Figura 4).
- Aggiungere una fase di incubazione per attivare la DNA polimerasi a 95 °C per 15 minuti.
- Per evitare il legame innesco-dimero, eseguire due cicli a 94 °C per 15 s, 49 °C per 1 min, quindi 3 cicli a 94 °C per 15 s, 59 °C per 15 s.
- Per l'amplificazione dei telomeri, aggiungere 27 cicli di 85 °C per 15 s, 74 °C per 30 s, quindi l'acquisizione del segnale.
- Per l'amplificazione dell'albumina, aggiungere 31 cicli di 94 °C per 15 s, 84 °C per 30 s, quindi l'acquisizione del segnale.
- Includere una curva di fusione da 59 °C a 95 °C a intervalli di 5 s per ogni grado crescente nel protocollo dei cicli termici.
- Fare clic su Avvia esecuzione per entrambi i termociclatori. Quando richiesto, specificare un titolo per i file di analisi.
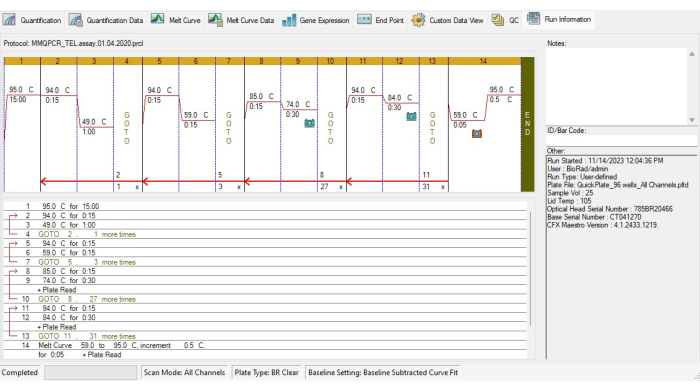
Figura 4: Profilo di termociclo del saggio MMqPCR. Protocollo MMqPCR creato nel software in conformità con il protocollo di termociclo originale19. Clicca qui per visualizzare una versione più grande di questa figura.
6. Analisi dei dati MMqPCR
- Al termine del ciclo termico, analizzare i dati nel modo seguente per produrre valori TL per i campioni che sono stati elaborati.
- Nel software, selezionare la funzione Impostazione piastra . Nel menu a discesa, fare clic su Visualizza/Modifica targa.
- Evidenzia tutti i pozzetti e fai clic su Seleziona fluorofori, quindi seleziona la casella SYBR e deseleziona tutte le altre caselle. Fare clic su OK.
- Mentre tutti i pozzetti sono ancora evidenziati, accanto alla parola Carica, seleziona la casella accanto a SYBR. Ora tutti i pozzi dovrebbero avere la scritta SYBR su di essi.
- Quindi, evidenziare i tre pozzetti NTC nella parte superiore o inferiore della diluizione seriale di controllo standard e selezionare NTC dal menu del tipo di campione a destra. I tre pozzi dovrebbero ora essere gialli e chiamarsi NTC. La Figura 5A mostra quali pozzetti devono essere selezionati per P1 e la Figura 5B mostra quali pozzi devono essere selezionati per P2.
NOTA: P2 sarà invertito da P1, quindi l'NTC sarà nella parte inferiore delle colonne 4-6 per P1 e nella parte superiore delle colonne 7-9 per P2. - Evidenziare i 21 pozzetti della diluizione seriale di controllo standard e selezionare Standard dal menu del tipo di campione. Questi pozzetti dovrebbero essere verdi. Mentre questi pozzetti sono ancora evidenziati, fare clic su Replica tecnica. Seleziona 3 dal menu Replica dimensioni, quindi seleziona Orizzontale > Applica. Questi pozzetti dovrebbero essere etichettati in gruppi di tre da Std-1 a Std-7.
- Mentre lo standard è ancora evidenziato, scorrere verso il basso e selezionare Serie di diluizione. Nel campo Fattore di diluizione, immettere 2, quindi digitare la concentrazione iniziale di diluizione e la direzionalità in base al numero della piastra, come descritto nei passaggi seguenti.
- Per la targa P1, inserisci 2.00E-03 nel campo di concentrazione iniziale e seleziona la casella Decrescente , quindi seleziona Applica. I valori nei 21 pozzi dovrebbero avere i valori di concentrazione scritti in ciascun pozzo che vanno da 2.00E-03 a 3.13E-05 dall'alto verso il basso.
- Per la targa P2, inserisci 3.13E-5 nel campo di concentrazione iniziale e seleziona la casella per Crescente , quindi seleziona Applica. I valori nei 21 pozzi dovrebbero avere i valori di concentrazione scritti in ciascun pozzo che vanno da 3.13E-05 a 2.00E-03 dall'alto verso il basso.
- Per i campioni, evidenziare le colonne per la striscia PCR A (1-3 per P1 e P2), selezionare Sconosciuto dal menu del tipo di campione, quindi selezionare Replica tecnica. Seleziona 3 dal menu Replica dimensioni , quindi seleziona Orizzontale > Applica. I pozzetti per queste colonne dovrebbero essere blu ed etichettati per riga in gruppi di 3 da Unk-1 a Unk-8.
- Ripetere il passaggio 6.3 per le colonne per la striscia PCR B (colonne 7-9 per P1 e 4-6 per P2). Dovrebbero essere etichettati da Unk-9 a Unk-16.
- Ripetere il passaggio 6.3 per le colonne per la striscia PCR C (colonne 10-12 per P1 e P2). Dovrebbero essere etichettati da Unk-17 a Unk-24. Quando i passaggi 6.2-6.3.2 sono completati, le finestre di configurazione della piastra dovrebbero assomigliare alla Figura 5A, B.
- Selezionare OK in basso a destra nella finestra dell'editor delle lastre. Fare clic su Sì per applicare le modifiche quando richiesto.
- Per il controllo qualità a livello di test, assicurarsi che le curve nella scheda di quantificazione siano appropriate (ad esempio, nessuna curva di amplificazione invertita) sia per il passaggio 9 che per il passaggio 12 del profilo di termociclo, che corrispondono rispettivamente agli ampliconi dei telomeri e delle albumine. Le curve sono accessibili dal menu a discesa sul lato centrale destro della finestra del software. Le figure 6A, B mostrano le curve appropriate.
- Assicurarsi che le efficienze PCR riportate sia nella fase 9 che nella fase 12 siano comprese tra il 90% e il 110% e che queste due efficienze non differiscano l'una dall'altra più del 10% (ad esempio, il 92,4% per la fase 9 e il 98% per la fase 12 è appropriato, ma il 92,4% per la fase 9 e il 108% per la fase 12 non sono appropriati). Inoltre, assicurarsi che la curva R2 standard sia >0,995. Se P1 o P2 non soddisfano questi criteri, le piastre devono essere ripetute.
- Per P1, selezionare il passaggio 9 ed evidenziare i 21 pozzetti corrispondenti alla curva standard come mostrato nella Figura 6A, B. Copiare i valori Cq nell'angolo in basso a destra della finestra del software. Aprire il modello di foglio dati dei telomeri disponibile come file supplementare 1 e incollare questi valori nel foglio standard 1, colonna B. Selezionare il passaggio 12 , quindi copiare questi valori Cq. Incolla questi valori nel foglio 1 standard, colonna I.
- Identificare il valore della pendenza nel passaggio 9 e digitarlo nella cella C4 del foglio 1 standard. Identificare il valore della pendenza nel passaggio 12 e digitarlo nella cella J4 del foglio standard 1.
NOTA: Il sistema calcola l'efficienza percentuale degli inneschi utilizzando le pendenze della curva standard identificate al punto 6.6.1. La percentuale di efficienza può essere calcolata manualmente utilizzando la seguente formula: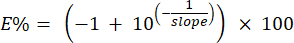
- Assicurarsi che il coefficiente di variazione (CV) per ogni triplicato di diluizione standard sia inferiore a 0,1. Se maggiore o uguale a 0,1, escludere fino a tre singoli pozzetti in totale da tutte le concentrazioni dei campioni di controllo inclusi nella curva di diluizione seriale standard a 7 punti sulla piastra. Solo un pozzo dovrebbe essere escluso da ogni set triplicato.
NOTA: L'esclusione dei punti standard dall'analisi sul software modificherà la pendenza riportata, R, l'intercetta y e l'efficienza per entrambi i primer. Pertanto, dopo che qualsiasi pozzetto standard è stato rimosso dall'analisi, i passaggi 6.5.1-6.6.1 devono essere ripetuti. - Ripetere i passaggi 6.6.1 - 6.6.2 per il file di analisi P2 e il foglio standard 2 corrispondente.
- Solo quando sia i fogli standard 1 che standard 2 sono completati con CV accettabili, raccogliere i dati dei singoli campioni dai file di analisi. Assicurarsi che le regolazioni del controllo qualità siano riportate nella colonna L del foglio P1 e P2, ad esempio il numero di pozzetti esclusi nel file di analisi P1.
- Identificare il valore della pendenza nel passaggio 9 e digitarlo nella cella C4 del foglio 1 standard. Identificare il valore della pendenza nel passaggio 12 e digitarlo nella cella J4 del foglio standard 1.
- Assicurarsi che solo i 72 pozzetti del campione nel file di analisi P1 siano evidenziati in blu nella scheda Quantificazione, come mostrato in Figura 7A,B. Selezionare Passaggio 9. Copiare i valori Cq e SQ nell'angolo in basso a destra della finestra del software. Incolla questi valori nel foglio P1 degli esempi, a partire dalla cella D3. Selezionare Passaggio 12 quindi copiare questi valori Cq e SQ e incollarli nel foglio P1 degli esempi, a partire dalla cella F3.
- Ripetere questo passaggio per i campioni nel file di analisi P2 e nel foglio P2 dei campioni corrispondenti. I rapporti tra telomeri e singola copia (T/S) (colonna H) vengono calcolati automaticamente, ma possono essere calcolati manualmente utilizzando la seguente formula:
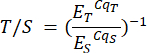
Dove ET/S è l'efficienza dell'amplificazione esponenziale per reazioni che hanno come bersaglio rispettivamente il telomero o il gene a copia singola, e CqT/S è il ciclo in cui una data replica che ha come bersaglio il contenuto telomerico o il gene a copia singola raggiunge la soglia critica della quantificazione della fluorescenza. I rapporti T/S medi (colonna I) sono calcolati su sei misurazioni per campione.
NOTA: I numeri ID del campione in CFX non corrispondono allo stesso campione biologico tra le due piastre. Il modello di foglio dati dei telomeri tiene conto di questa differenza e allineerà le triplicate duplicate appropriate. - In CFX Maestro, verificare che i valori Cq per tutti i campioni analitici siano compresi tra il valore Cq più basso e quello più alto della curva standard, come mostrato nella Figura 7B. Se un campione è al di fuori dell'intervallo, come mostrato nella Figura 7A, dovrà essere ripetuto dopo l'aggiustamento della concentrazione. Un campione con un valore Cq inferiore allo standard di concentrazione più elevato deve essere diluito di un fattore approssimativo al numero di cicli di amplificazione che è fuori intervallo, mentre un campione con un valore Cq superiore allo standard di concentrazione più basso dovrebbe essere concentrato di un fattore approssimativo al numero di cicli di amplificazione oltre la soglia.
NOTA: Un'ampia variazione nella concentrazione dei campioni analitici introduce un ulteriore livello di variazione potenziale e deve essere eseguita con cautela. - Assicurarsi che la concentrazione iniziale nel NTC fosse inferiore al 5% della quantità media di DNA presente nei pozzetti del campione. Questo può essere verificato visualizzando la percentuale di contaminazione dell'acqua sui fogli dei campioni P1 e P2 della MMqPCR. Se l'NTC indica una contaminazione, eseguire nuovamente le piastre.
NOTA: A causa della sensibilità del test MMqPCR, una contaminazione incontrollabile può occasionalmente causare l'amplificazione dei pozzetti NTC. Tuttavia, questa amplificazione dovrebbe essere minore e verificarsi diversi cicli dopo l'amplificazione genica a copia singola nella fase 12 del protocollo di termociclo. - Per il controllo qualità a livello di campione, esaminare le deviazioni standard (SD) (colonna J) e i CV (colonna K) nei campioni P1 e nei fogli P2 dei campioni. Assicurarsi che i CV intraplacca nei rapporti T/S triplicati del campione siano inferiori a 0,10 (10%). Per correggere eventuali CV intraplacca superiori a 0,1, può essere escluso un rapporto T/S da una serie di triplicati.
NOTA: Può essere esclusa una sola replica totale tra le sei misurazioni per campione, ovvero non è possibile escludere una replica per lo stesso campione sia su P1 che su P2. - Verificare che i CV interpiatto del singolo campione nel foglio P1 v P2, colonna G, siano inferiori a 0,05 (5%). Per correggere eventuali CV superiori a 0,05, eseguire l'esclusione di un rapporto T/S dalle sei misurazioni per campione su entrambe le piastre. Escludere i campioni dopo l'ispezione visiva attraverso i triplicati intrapiastra e tutte e sei le misurazioni, se necessario.
NOTA: Solo i campioni che soddisfano questi criteri QC, CV intraplacca < 10% e CV interplacca < 5%, possono essere inclusi nei risultati finali TL. In caso contrario, il campione deve essere escluso dalle schede tecniche P1 v P2 e ICC per questa corsa e il campione deve essere rivalutato in un secondo test MMqPCR. - Per il controllo qualità a livello di piastra, verificare che la variazione media all'interno della piastra tra tutti i campioni su ciascuna piastra, situata nella parte inferiore dei fogli P1 e P2, sia inferiore a 0,05 (5%). Per un ulteriore controllo qualità a livello di piastra, verificare che la variazione complessiva tra le piastre nel foglio P1 rispetto a P2, cella G29, sia inferiore a 0,06 (6%). Assicurarsi che i campioni che non hanno superato il controllo qualità vengano rimossi dal calcolo del CV interpiatto nella cella G30.
NOTA: Quando si lavora con gruppi di campioni correlati (ad esempio, membri della famiglia, punti temporali diversi, ecc.), tutti i campioni di un gruppo devono essere eseguiti sulla stessa piastra. Pertanto, se un campione del gruppo non supera i criteri di controllo qualità, tutti i campioni del gruppo devono essere rieseguiti. - In una coorte globale separata o in un file di dati dell'esperimento, registrare i dati TL finali per ogni campione che ha superato il QC (foglio P1 v P2, colonna I), i calcoli ICC per ogni campione che ha superato il QC (foglio dati ICC, colonne E-K) e i dati QC dell'esecuzione del test finale dal foglio P1 v P2 (colonne J-L)
- Ripetere questo passaggio per i campioni nel file di analisi P2 e nel foglio P2 dei campioni corrispondenti. I rapporti tra telomeri e singola copia (T/S) (colonna H) vengono calcolati automaticamente, ma possono essere calcolati manualmente utilizzando la seguente formula:
- Utilizzare il file supplementare 2 creato dalla rete di ricerca sui telomeri (TRN) per calcolare l'ICC per il progetto20.
- Infine, per migliorare la comparabilità tra gli studi di TL, trasformare i dati finali di TL per ciascun campione nella coorte complessiva separata o nel file di dati dell'esperimento in punteggi Z utilizzando la seguente equazione in cui il rapporto T/S medio per tutti i campioni della coorte viene sottratto dal rapporto T/S del campione individuale e diviso per la SD tra tutti i campioni della coorte.
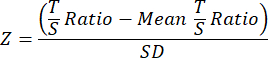
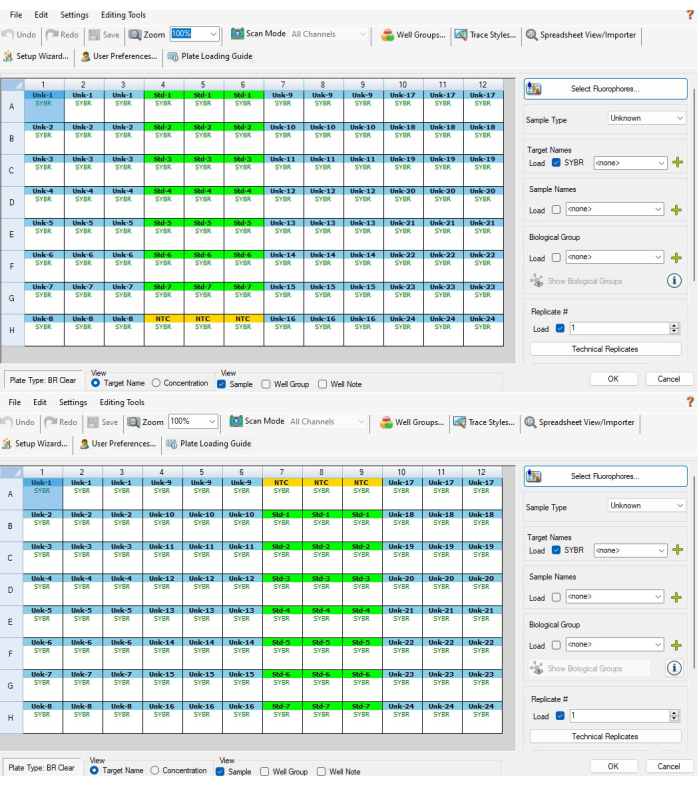
Figura 5: Configurazione della piastra basata su software. (A) Configurazione della piastra basata su software per una piastra P1 dopo aver completato i passaggi 6.2-6.4. (B) Configurazione della piastra basata su software per una piastra P2 dopo aver completato i passaggi 6.2-6.4. Gli ID del campione e dello standard non sono allineati tra le due piastre CFX a causa del modo in cui il software assegna gli ID del campione in base alla posizione del pozzetto (ad esempio, il campione CFX 1 su P1 è il campione CFX 24 su P2). Il modello excel fornito nel file supplementare 1 tiene conto di ciò, garantendo che le misurazioni interpiastra effettuate sullo stesso campione biologico siano correttamente allineate l'una all'altra Fare clic qui per visualizzare una versione più grande di questa figura.
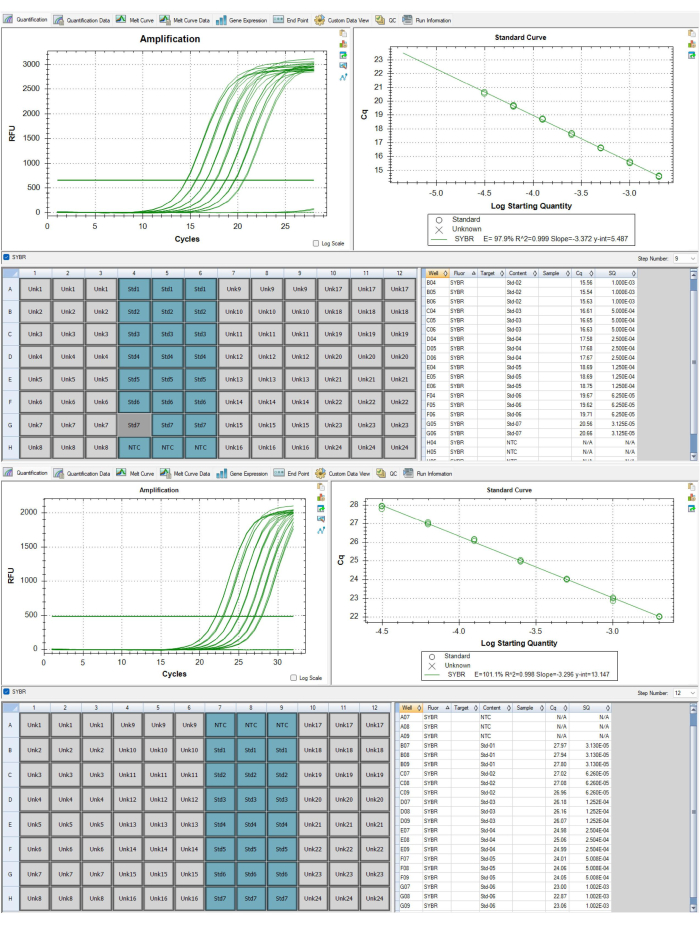
Figura 6: Curve standard perampliconi di elomero e albumina. (A) Questa curva standard proviene dall'amplicone dei telomeri P1 del set di dati rappresentativi dei risultati. Uno standard è stato rimosso perché non soddisfaceva i criteri di controllo qualità. (B) Questa curva standard proviene dall'amplicone di albumina P2 del set di dati rappresentativo dei risultati. Clicca qui per visualizzare una versione più grande di questa figura.
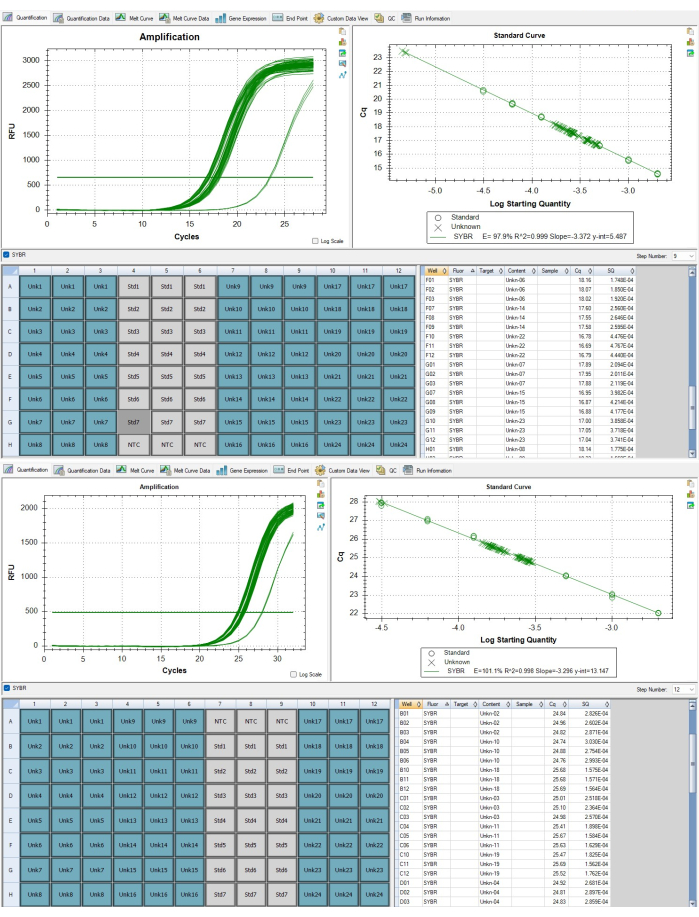
Figura 7: Amplicone dei telomeri e dei geni a copia singola. (A) Amplificazione dei telomeri e (B) amplificazione genica dell'albumina di campioni riportati nei risultati rappresentativi visualizzati nel software. Clicca qui per visualizzare una versione più grande di questa figura.
7. Segnalazione dei dati MMqPCR
- Quando si riportano i risultati della ricerca utilizzando i dati TL creati dalla MMqPCR, assicurarsi di riportare gli elementi descritti nelle linee guida per la segnalazione minima TRN. Queste linee guida sono disponibili sul sito web di TRN (https://trn.tulane.edu/resources/reporting-guidelines/) e un modello per la segnalazione di queste informazioni è fornito nel File supplementare 3. Citare questo protocollo e altre linee guida e risorse TRN, se del caso.
Risultati
I risultati presentati nella Tabella 4 e nella Tabella 5 offrono un esempio di misure TL altamente ripetibili ottenute seguendo il protocollo. Per questi risultati, il DNA è stato estratto da 24 campioni di cellule mononucleate del sangue periferico (PBMC) utilizzando un kit commerciale secondo le linee guida dei produttori. Questi 24 campioni sono stati analizzati su due piastre da 96 pozzetti. Tutti i campioni di DNA sono stati controllati per verificarne la qualità tramite spettrofotometro e fluorimetro, utilizzando il rapporto medio 260/280, il rapporto medio 260/230 e la concentrazione di dsDNA per determinare l'idoneità al test e il fattore di diluizione del campione (Tabella 6). La Tabella 6 evidenzia anche l'importanza di quantificare la concentrazione di dsDNA, che può differire dalla concentrazione di DNA misurata utilizzando uno spettrofotometro. Questa variabilità è il risultato di diversi approcci alla quantificazione. In particolare, lo spettrofotometro ricava la concentrazione di DNA in base all'assorbimento a 280 nm ed è suscettibile alle fluttuazioni dovute a contaminanti (ad esempio, proteine, sale, ecc.) che influiscono sulle letture dell'assorbanza. Al contrario, le concentrazioni di dsDNA misurate utilizzando un fluorimetro sono determinate dalla fluorescenza di un colorante che si lega specificamente al dsDNA e, come tale, si presume che rifletta più accuratamente il contenuto di DNA. I campioni di DNA hanno subito fino a tre cicli di congelamento-scongelamento prima dell'analisi MMqPCR TL. Il DNA di controllo è stato creato da estrazioni di DNA raggruppate di PBMC da un individuo, che è stato utilizzato per creare una diluizione seriale a sette punti da 2 ng/μL a 0,0313 ng/μL di DNA. Le curve standard indipendenti create per la PCR Step 9 (amplicone dei telomeri) e il Step 12 (amplicone genico a copia singola, albumina in questo protocollo) sono presentate nella Figura 6A,B. I saggi sono stati eseguiti su un sistema commerciale di rilevamento PCR in tempo reale che ha generato una curva di fusione che mostra i singoli prodotti ampliconi prodotti a temperature distinte, come mostrato nella Figura 8.
Tabella 4: Dati di output dalla misurazione MMqPCR per risultati rappresentativi ottimali. Rapporti T/S medi, SD, CV e TL con punteggio Z per 24 campioni. Clicca qui per scaricare questa tabella.
Tabella 5: Output dei dati sulla lunghezza dei telomeri. I dati grezzi del test MMqPCR vengono analizzati nel software, quindi aggiunti al modello di foglio di calcolo allegato come file supplementare 1 per ulteriori analisi e controllo qualità. Questo foglio di output del modello TL presenta un riepilogo dei dati, visualizzando gli ID dei campioni, i TL medi, i SD e i CV per ciascun campione su entrambe le lastre. Clicca qui per scaricare questa tabella.
Tabella 6: Spettrofotometro e fluorimetro Metriche di qualità del DNA per i risultati dei campioni. Dati di controllo qualità derivanti dall'analisi dello spettrofotometro duplicato e dalla misurazione con fluorimetro singolare del dsDNA e di eventuali contaminanti per campione. Clicca qui per scaricare questa tabella.
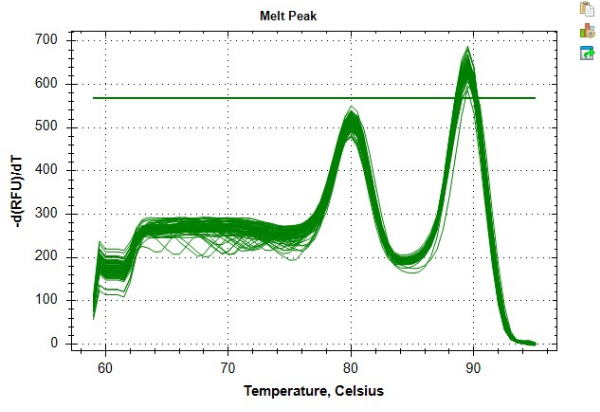
Figura 8: Esempio di curva di fusione. Curva di fusione per i dati del campione generati nel software. Il picco precedente di ~80 °C rappresenta l'amplicone dei telomeri e il picco secondario a ~89 °C rappresenta l'amplicone dell'albumina. Clicca qui per visualizzare una versione più grande di questa figura.
Per questo progetto, l'efficienza media dei telomeri è stata del 98,6% con un intervallo da 90,7 a 102,1 e l'efficienza media dell'albumina è stata del 102,3% con un intervallo da 93,6 a 108,2 in tutte le esecuzioni. C'è stata una media di 0,97 repliche rimosse dalle curve standard, che soddisfa i criteri QC di questo protocollo. Il CV medio interplacca è stato dell'1,83% con una SD di 0,00616 e il CV medio intraplate è stato del 3,78% con una SD di 0,00658. I TL medi per questi campioni rappresentativi sono presentati nella Tabella 4 e nella Tabella 5. La TL media era 1,37 con una SD di 0,24 e un intervallo da 0,84 a 2,32. Il rapporto T/S, SD, CV e TL con punteggio Z per campione sono elencati nella Tabella 4. Poiché l'output del rapporto T/S del test MMqPCR è una misura relativa di TL, i rapporti sono stati trasformati in questo punteggio Z per consentire il confronto tra studi. L'ICC per questo progetto è stato calcolato utilizzando lo script R come descritto dalle linee guida TRN presenti nel file supplementare 2, tenendo conto degli effetti batch ed run20. Al fine di calcolare l'ICC intra-progetto, abbiamo riesaminato il 10% dei campioni superati, assicurandoci di popolare le piastre ICC con almeno un campione da ciascuna piastra della coorte. L'ICC complessivo del progetto di 0,801 [CI: 0,703, 0,86] indica l'elevata riproducibilità dei risultati TL.
Non tutti i risultati saranno ottimali. I risultati nella Figura 9 e nella Tabella 7 mostrano risultati non ottimali dal test MMqPCR. La Figura 9 mostra una curva standard con un'efficienza dei telomeri inferiore al 90%, che è inferiore agli standard QC, che richiede la ripetizione dell'intera piastra. I problemi con l'efficienza del primer sono solitamente dovuti a un problema con i reagenti, quindi è importante tenere traccia delle date in cui i reagenti vengono aliquotati e quando scadono come primo passo per determinare quale reagente è responsabile della bassa efficienza. I reagenti scaduti devono essere sostituiti prima di riavviare la piastra. La tabella 7 mostra una piastra che ha superato i criteri di controllo qualità iniziali a livello di campione, ma presenta un alto livello di variabilità tra le piastre, con conseguente fallimento del controllo qualità a livello di piastra. La variazione tra le piastre è solitamente dovuta a errori nelle tecniche di pipettaggio e riempimento delle piastre. In questo caso, il tecnico deve valutare eventuali problemi verificatisi durante l'impostazione della piastra e assicurarsi che le pipette siano calibrate.
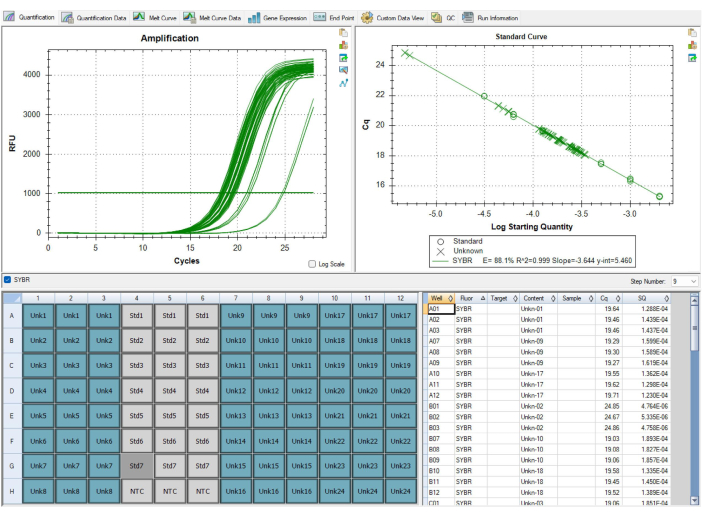
Figura 9: Risultati non ottimali. Questa curva standard visualizzata nel software non è riuscita a superare il QC, poiché l'efficienza di amplificazione per l'innesco dei telomeri era inferiore al 90%. L'immagine è di P1, ma P2 aveva efficienze altrettanto basse. Non è stato possibile utilizzare i dati di questa corsa e tutti i campioni hanno dovuto essere rianalizzati dopo aver sostituito il reagente causale scaduto. Clicca qui per visualizzare una versione più grande di questa figura.
Tabella 7: Risultati non ottimali. Nella tabella viene visualizzato il modello di dati dei telomeri per un'esecuzione in cui molti dei campioni non sono riusciti a soddisfare gli standard QC. I campioni che dovevano essere rieseguiti sono stati determinati in base ai valori CV, quindi il nome del campione è stato modificato in un carattere rosso per una facile identificazione. Clicca qui per scaricare questa tabella.
Fascicolo supplementare 1: Modello di foglio di calcolo dei dati dei telomeri. Clicca qui per scaricare questo file.
Fascicolo supplementare 2: Calcolo ICC. Questo protocollo è stato creato dalla Telomere Research Network (TRN). Questo file è stato modificato da20. Clicca qui per scaricare questo file.
Fascicolo supplementare 3: Linee guida per la segnalazione TRN. Clicca qui per scaricare questo file.
Discussione
Il metodo più comunemente utilizzato per la misurazione della TL in studi più ampi basati sulla popolazione, prima del 2002, era l'analisi Southern blot delle lunghezze dei frammenti di restrizione terminale (TRF)24,25. La TRF, nonostante fornisca un'eccellente precisione e riproducibilità in laboratori specializzati, è limitata nell'applicabilità a causa della quantità e della qualità del DNA richiesto e della produttività limitata, fornendo così lo sfondo per un maggiore utilizzo dei test TL basati su qPCR e, successivamente, del test MMqPCR. Il metodo MMqPCR TL fornisce una misurazione ripetibile del TL se impostato, ottimizzato e mantenuto con particolare attenzione a ciascun criterio di controllo qualità. Il calcolo e la segnalazione dell'ICC specifico per ciascuna coorte analizzata sono necessari per garantire l'affidabilità del test. Sebbene sia soggetto alla qualità del DNA e alla competenza tecnica, il metodo MMqPCR è adatto per studi su larga scala basati sulla popolazione che studiano la TL perché richiede piccole quantità di DNA, è più affidabile della PCR singleplex ed è più efficiente nei costi dei reagenti e nel tempo dei tecnici rispetto ad altri metodi. La capacità di generare ICC elevati fornisce dati aggiuntivi a sostegno dell'uso della MMqPCR per ampi studi di TL basati sulla popolazione. La misurazione della TL MMqPCR può essere applicata a un'ampia gamma di studi che cercano di definire il ruolo della TL come biomarcatore della mortalità per tutte le cause, dell'invecchiamento, dello stress nel corso della vita, delle esposizioni ambientali e degli esiti di salute fisica come malattie cardiovascolari e cancro 4,7,8,10,11,12,13,26,27,28, 29,30,31.
Una limitazione del metodo MMqPCR è che riporta TL come un rapporto T/S, una stima relativa della lunghezza che varia a seconda della selezione del gene a copia singola, della composizione della miscela master e dei parametri del ciclo PCR32. Il rapporto T/S è privo di unità. Pertanto, senza combinarsi con altre metodologie di misurazione del TL, questo metodo non è in grado di riportare stime nei valori delle coppie di basi 18,33,34. Di conseguenza, il rapporto T/S deve essere trasformato in un punteggio Z per avere rilevanza in tutti gli studi35. È necessario prestare molta attenzione quando si esegue questa operazione in laboratori, metodi e saggi. Inoltre, questo metodo, come i saggi di ibridazione basati su gel, può includere la quantificazione dei telomeri interstiziali. Tuttavia, queste sequenze comprendono una percentuale molto piccola del contenuto totale di DNA dei telomeri per genoma. Inoltre, è più probabile che le sequenze telomeriche interstiziali contengano sequenze di coppie di basi non corrispondenti che si discostano dalle ripetizioni canoniche dei telomeri, diminuendo la probabilità di legame e amplificazione del primer. Inoltre, sebbene la quantità minima di DNA necessaria per il test MMqPCR sia vantaggiosa, è importante notare che le misurazioni di TL basate sulla qPCR sono influenzate da fattori pre-analitici che influiscono sulla qualità e sull'integrità del DNA, tra cui le condizioni di conservazione del campione, la metodologia di estrazione del DNA e il tessuto biologico36,37. È stato dimostrato che il controllo analitico di questi fattori può migliorare la validità esterna delle misure di TL generate utilizzando la qPCR38. Ciononostante, l'impatto delle differenze nella qualità del DNA sulla TL generata dal test MMqPCR deve essere valutato sistematicamente, poiché non esistono attualmente linee guida basate sui dati per determinare se un campione è di qualità sufficiente per generare una stima accurata della TL utilizzando questo approccio. Nonostante queste limitazioni, le applicazioni di questo test per gli studi sugli esiti di salute a livello di popolazione sono considerevoli.
Quando si utilizza il test MMqPCR, è necessaria una valutazione continua della precisione e della produttività. Come attualmente progettato, i tecnici eseguono triplicati di campioni contemporaneamente su piastre duplicate utilizzando due termociclatori. In assenza di più termociclatori, si consiglia di conservare le misurazioni in doppio-triplicato e di eseguire le piastre in sequenza per una maggiore precisione anche a costo di una minore produttività. Qualsiasi decisione di dare priorità alla produttività rispetto alla precisione, ad esempio impiegando misure a triplicato singolo, dovrebbe essere accompagnata da test e valutazioni approfonditi degli ICC risultanti prima di procedere con l'analisi dei campioni analitici. Quando si prendono decisioni in merito alla produttività e alla precisione, è necessario prendere in considerazione la dimensione del campione e la qualità del DNA del campione. Una dimensione del campione più piccola o campioni di DNA di scarsa qualità richiedono la priorità di una maggiore precisione23. Questo è ancora più importante quando si lavora con campioni di gruppi correlati (ad esempio, membri della famiglia, soggetti con più punti temporali). In questi casi, un'attenta pianificazione, ad esempio l'assegnazione di campioni correlati alla stessa lastra prima di iniziare l'esperimento, è un modo per prevenire la perdita di potenza statistica dovuta alla confusione involontaria gruppo per piastra.
Per questo test, è stato utilizzato uno spettrofotometro per valutare la qualità del campione di DNA: campioni compresi tra 1,6 e 2,0 per i rapporti 260/280 e 2,0-2,2 per i rapporti 260/230 sono stati considerati accettabili. Questa valutazione della qualità e la valutazione accurata del DNA a doppio filamento tramite fluorimetro sono passaggi fondamentali in questo protocollo per ottenere dati TL ripetibili. Altre misure più descrittive dell'integrità del DNA, come la dimensione del frammento e/o le misure riassuntive della qualità del DNA determinate tramite gel di agarosio (ad esempio, il numero di integrità del DNA) possono essere utilizzate per determinare la qualità del campione38. Si raccomanda inoltre che la diluizione dei campioni avvenga solo al momento della preparazione del campione per l'esecuzione del test MMqPCR. Ciò garantisce che le aliquote di DNA subiscano la minor quantità possibile di manipolazione dopo l'estrazione, diminuendo la variabilità nella manipolazione pre-test. Nel caso in cui i campioni di DNA debbano essere trasportati, devono essere spediti su ghiaccio secco alla massima concentrazione possibile per mitigare la degradazione che si verifica nei campioni di DNA a concentrazioni più basse39,40. A causa della degradazione del DNA che si verifica con i cicli di congelamento-scongelamento, il numero di congelamento-scongelamento per il DNA di riserva dovrebbe essere ridotto al minimo41. Le aliquote del DNA di controllo aggregato devono essere create prima di eseguire la coorte e le aliquote dei singoli campioni di DNA analitico devono essere create prima di eseguire il saggio.
Le metriche chiave delle prestazioni del saggio e del controllo qualità includono il segnale NTC, i CV interpiatto e intrapiatto e la curva standard R2. Il mancato rispetto dei criteri di controllo qualità può essere mitigato in diversi modi. La sostituzione regolare delle scorte di PCR di grado H2O e l'aliquotazione di sottoceppi di PCR di grado H2O per ogni coppia di piastre analizzate ridurrà al minimo le fonti di contaminazione e l'amplificazione NTC. Ulteriori passaggi per ridurre la contaminazione includono quanto segue: designare una cappa PCR specifica dedicata solo al test MMqPCR; pulire la cappa PCR e l'apparecchiatura con una soluzione decontaminante del DNA; irradiare la stanza con luce ultravioletta; e praticare la tecnica sterile quando si è nel cappuccio della PCR. Per migliorare la ripetibilità del test e ridurre i CV, si consiglia di agitare vigorosamente i campioni, le diluizioni e le strisce PCR nelle rispettive fasi di vortice e di risospendere accuratamente durante il pipettaggio dei campioni di DNA. Una curva R2 standard inferiore alla soglia (<0,995) è molto probabilmente attribuibile a errori di pipettaggio durante il caricamento della piastra. Per evitare ciò, prestare particolare attenzione al pipettaggio preciso e calibrare le pipette ogni anno, miscelare energicamente le strisce PCR standard prima del caricamento e organizzare attentamente le forniture per promuovere un flusso di lavoro efficiente. Se si osserva che l'utilizzo di due macchine e una lastra produce CV costantemente più elevati, la macchina deve essere riparata come un potenziale modo per migliorare il problema. Le piastre QC del produttore devono essere eseguite regolarmente sulle piastre per valutare le prestazioni del termociclatore.
Se i problemi persistono anche dopo l'applicazione dei passaggi consigliati in precedenza, è possibile utilizzare i passaggi seguenti per risolvere i problemi relativi al protocollo. Tenere un registro di quando tutti i reagenti sono stati aliquotati e di eventuali date di scadenza pertinenti può aiutare a semplificare il processo di risoluzione dei problemi quando inevitabilmente sorgono difficoltà. Una parte importante di qualsiasi processo di risoluzione dei problemi consiste nel regolare un solo reagente alla volta per determinare la causa specifica delle piastre che non superano i criteri QC, a partire dal reagente meno costoso in questione. Ad esempio, se sia il telomero che il gene a copia singola hanno basse efficienze, i reagenti condivisi come il DTT, i dNTP o l'aliquota SYBR sono più probabilmente la causa rispetto ai primer specifici per gli ampliconi. Al prezzo di listino, le nuove aliquote dovrebbero essere testate in ordine di DTT, poi dNTP e infine, se il problema persiste, nuove aliquote SYBR. Al contrario, se solo uno degli ampliconi (telomero o gene a copia singola) ha una bassa efficienza, la causa della difficoltà è più probabilmente uno dei primer. L'interpretazione dei picchi della curva di fusione presentata nella Figura 8 può servire come fonte di informazioni chiave per la risoluzione dei problemi. La visualizzazione dei due picchi della curva di fusione può essere utilizzata per identificare potenziali problemi con un particolare campione, poiché un singolo campione problematico si distinguerà dall'andamento generale dei picchi esibiti dagli standard o dai campioni analitici rimanenti. La curva di fusione può anche essere utilizzata per diagnosticare problemi con un particolare innesco se i picchi per un dato amplicone sono sistematicamente meno acuti dell'altro.
Questo manoscritto descrive in dettaglio come impostare con successo il test MMqPCR per misurare la TL con un'ampia applicabilità alla ricerca sulla salute pubblica e introduce raccomandazioni chiave per il controllo qualità e la risoluzione dei problemi con l'obiettivo di aumentare l'accessibilità e l'affidabilità di questo metodo efficiente ed economico.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori desiderano ringraziare il Comitato consultivo della rete di ricerca sui telomeri e il National Institute on Aging / National Institute of Environmental Health Sciences (U24 AG066528 e U24 AG066528-S1) che hanno reso possibile questo lavoro.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.5mL Tubes | USA Scientific | 1605-0099 | Seal-Rite 0.5mL Microcentrifuge Tubes, Sterile Storage Temperature and Conditions: Room temperature |
| 1.5mL Tubes | USA Scientific | 1615-5599 | Seal-Rite 1.5mL Microcentrifuge Tubes, Sterile Storage Temperature and Conditions: Room temperature |
| 100mM DTT | In House | Not Applicable | Made with stock DTT, diluted sodium acetate, and PCR Grade H2O Storage Temperature and Conditions: minus 20 °C |
| 15mL Tubes | Thermo Fisher | 14-959-53A | Corning 352196 Falcon 15mL Conical Centrifuge Tubes Storage Temperature and Conditions: Room temperature |
| 1M MgCl2 | Thermo Fisher | 50152107 | Biotang Inc 1M MgCl2 1M Magnesium Chloride Solution, Prepared in 18.2 Megohms Water and Filtered through 0.22 Micron Filter Storage Temperature and Conditions: 4 °C |
| 1x Gold Buffer | In House | Not Applicable | 10X Gold Buffer diluted with PCR Grade H2O Storage Temperature and Conditions: minus 20 °C |
| 25mM dNTPs | New England BioLabs | N0446S | Deoxynucleotide Solution Set Storage Temperature and Conditions: minus 20 °C |
| 5mL Tubes | Thermo Fisher | 3391276 | Argos Technologies Microcentrifuge Tubes – 5mL Storage Temperature and Conditions: Room temperature |
| 96 Well Plate | Bio-Rad | HSP9601 | Hard-Shell 96-Well PCR Plates, Low Profile, Thin Wall, Skirted, White / Clear Storage Temperature and Conditions: Room temperature |
| Aluminum Foil | Office Depot | 3489072 | Reynolds Wrap Stanard Aluminum Foil Roll, 12" x 75', Silver Storage Temperature and Conditions: Room temperature |
| AmpliTaq Gold Kit – Polymerase and Buffer | Thermo Fisher | 4311806 | AmpliTaq Gold DNA Polymerase with Gold Buffer and MgCl2 (MgCl2 in this kit is not used), 10X Gold Buffer, 2.5U AmpliTaq Gold Polymerase Storage Temperature and Conditions: minus 20 °C |
| Betaine | Thermo Fisher | AAJ77507AB | Betaine, 5M Solution, Molecular Biology Grade, Ultrapure, 10mL Storage Temperature and Conditions: minus 20 °C |
| Big Tube Rack | Thermo Fisher | 344817 | Fisherbrand 4-Way Tube Rack Storage Temperature and Conditions: Room temperature |
| CFX Maestro Software | Bio-Rad | 12004110 | Software for real-time PCR plate setup, data collection, statistics, and graphiing of results Storage Temperature and Conditions: Room temperature |
| CFX96 Optical Reaction Module for Real-Time PCR Systems with Starter Package | Bio-Rad | 1845096 | 96-well optical module for real-time PCR Storage Temperature and Conditions: Room temperature |
| DTT | Fisher Scientific | AAJ1539706 | Dithiothreitol, >99.5+ Molecular Biology Grade, 5 g Storage Temperature and Conditions: minus 20 °C |
| ELIMINase | Fisher Scientific | 04-355-32 | ELIMINase Laboratory Decontaminant Storage Temperature and Conditions: Room temperature |
| HEPA Filter | USA Scientific | Replacement Filters | High-Efficiency Particulate Air Filter for AirClean Workstations Storage Temperature and Conditions: Room temperature |
| Kimwipes | Thermo Fisher | 06666A | Kimberly-Clark Professional Kimtech Science Kimwipes Delicate Task Wipers, 1-Ply Storage Temperature and Conditions: Room temperature |
| Loading Trough | Thermo Fisher | 14387069 | Thermo Scientific Matrix Reagent Reservoirs Storage Temperature and Conditions: Room temperature |
| Microsoft Excel | Microsoft | Not Applicable | Microsoft 365 package, Excel software application Storage Temperature and Conditions: Room temperature |
| Mini Centrifuge | Genesee Scientific | 31-500B | Poseidon 31-500B Mini Centrifuge, Blue Lid Storage Temperature and Conditions: Room temperature |
| PCR Grade H2O | Thermo Fisher | AM9937 | Nuclease-Free Water (not DEPC-Treated) Storage Temperature and Conditions: Room temperature |
| PCR Hood | USA Scientific | 4263-2588 | Nucleic Acid Workstation with HEPA Filtration, AirClean Systems Combination PCR Workstation Storage Temperature and Conditions: Room temperature |
| PCR Strips | Thermo Fisher | AB0776 | Low Profile Tubes and Flat Caps, Strips of 8 Storage Temperature and Conditions: Room temperature |
| PCR Tube Rack | Thermo Fisher | 344820 | Fisherbrand 96-Well PCR Tube Rack Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Multichannel) | Ranin | 17005860 | Pipette Tips SR LTS 20µL F 960A/5, 20µL Maximum Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1181-3850 | 10µL Graduated TipOne RPT Filter Tips Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1180-1850 | 20µL Beveled TipOne RPT Filter Tips Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1111-0880 | 200µL Natural TipOne Pipette Tips in Racks Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1111-2890 | 1000µL Natural Graduated TipOne Pipette Tips in Racks Storage Temperature and Conditions: Room temperature |
| Pipettors (Multichannel) | Ranin | 17013802 | Pipet-Lite Multi Pipette L8-10XLS, 0.5 to 10µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Multichannel) | Ranin | 17013803 | Pipet-Lite Multi Pipette L8-20LS+, 2 to 20µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F144802G | Gilson Pipetman Classic Pipets, 1 to 10µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F123600 | Gilson Pipetman Classic Pipets, 2 to 20µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F123601 | Gilson Pipetman Classic Pipets, 20 to 200µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F123602 | Gilson Pipetman Classic Pipets, 200 to 1000µL Storage Temperature and Conditions: Room temperature |
| Plate Sealing Film | Bio-Rad | MSB1001 | Microseal “B” PCR Plate Sealing Film, Adhesive, Optical Storage Temperature and Conditions: Room temperature |
| Plate Spinner | Thermo Fisher | 14-100-141 | Fisherbrand Mini Plate Spinner Centrifuge, 230 V Storage Temperature and Conditions: Room temperature |
| Pre-Hood Filter | USA Scientific | 4235-3724 | Prefilter for AirClean Systems Workstations Storage Temperature and Conditions: Room temperature |
| R Software | The R Project for Statistical Computing | Not Applicable | R version 4.2.2 Storage Temperature and Conditions: Room temperature |
| Scale | Thermo Fisher | 01-922-329 | OHAUS 30430060 PR Series Analytical Balance, 62g Capacity Storage Temperature and Conditions: Room temperature |
| Scissors | Office Depot | 458612 | Office Depot Brand Scissors, 8”, Straight, Black, Pack of 2 Storage Temperature and Conditions: Room temperature |
| Sharpies | Sharpie | 2151734 | Brush Twin Permanent Markers, Black Storage Temperature and Conditions: Room temperature |
| Single Copy Gene Forward Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Single Copy Gene Reverse Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Small Tube Rack | Thermo Fisher | 21-402-17 | Thermo Fisher 8601 Reversible Microtube Racks with Lid Storage Temperature and Conditions: Room temperature |
| Sodium Acetate | Thermo Fisher | J63560.EQE | 3M NaOAc pH 5.2 Storage Temperature and Conditions: Room temperature |
| Stainless Steel Spatula | Thermo Fisher | 3990240 | Bel-Art SP Scienceware Stainless-Steel Sampling Spoon and Spatula Storage Temperature and Conditions: Room temperature |
| SYBR Green | Thermo Fisher | S7563 | SYBR Green I Nucleic Acid Gel Stain – 10,000X Concentrate in DMSO Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Syringe Filters | Fisher Scientific | 09-927-55A | GD/X 25 mm Sterile Syringe Filter, cellulose acetate filtration medium, 0.2 μm Storage Temperature and Conditions: Room temperature |
| Syringes | Thermo Fisher | 148232A | BD Luer-Lok Disposable Syringes without Needles, 10mL Storage Temperature and Conditions: Room temperature |
| TE Buffer | Fisher Scientific | BP2474100 | TE Buffer, Tris-EDTA, 1X Solution, pH 7.6, Molecular Biology, Fisher BioReagents Storage Temperature and Conditions: Room temperature |
| Telomere Forward Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Telomere Reverse Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| UV Light | USA Scientific | 4288-2540 | UV Light Bulb for Workstations Storage Temperature and Conditions: Room temperature |
| Vortex | Thermo Fisher | 14-955-151 | Fisherbrand Mini Vortex Mixer, 115 V, 50/60 Hz Storage Temperature and Conditions: Room temperature |
| Weigh Boat | Thermo Fisher | 01-549-752 | Fisherbrand Sterile Hexagonal Weighing Boat, 10mL Storage Temperature and Conditions: Room temperature |
Riferimenti
- Hemann, M., Strong, M., Hao, L., Greider, C. The shortest telomere, not average telomere length, is critical for cell viability and chromosome stability. Cell. 107 (1), 67-77 (2001).
- Xu, L., Li, S., Stohr, B. A. The role of telomere biology in cancer. Ann Rev Pathol: Mech Dis. 8, 49-78 (2013).
- Lindrose, A., Drury, S. Minimum reporting recommendations for pcr-based telomere length measurement. Telomere Research Network. , (2020).
- Verhulst, S., et al. Commentary: The reliability of telomere length measurements. Int J Epidemiol. 44 (5), 1683-1686 (2015).
- Aubert, G., Hills, M., Lansdorp, P. M. Telomere length measurement-caveats and a critical assessment of the available technologies and tools. Mutat Res. 730 (1-2), 59-67 (2012).
- Mundstock, E., et al. Effect of obesity on telomere length: Systematic review and meta-analysis. Obesity. 23 (11), 2165-2174 (2015).
- Haycock, P. C., et al. Leucocyte telomere length and risk of cardiovascular disease: Systematic review and meta-analysis. BMJ. 349, 4227(2014).
- Astuti, Y., Wardhana, A., Watkins, J., Wulaningsih, W. Cigarette smoking and telomere length: A systematic review of 84 studies and meta-analysis. Environ Res. 158, 480-489 (2017).
- Ridout, K. K., Ridout, S. J., Price, L. H., Sen, S., Tyrka, A. R. Depression and telomere length: A meta-analysis. J Affect Disord. 191, 237-247 (2016).
- Wang, Q., Zhan, Y., Pedersen, N. L., Fang, F., Hägg, S. Telomere length and all-cause mortality: A meta-analysis. Ageing Res Rev. 48, 11-20 (2018).
- Hu, R., Hua, X. G., Jiang, Q. C. Associations of telomere length in risk and recurrence of prostate cancer: A meta-analysis. Andrologia. 51 (7), e13304(2019).
- Schneider, C. V., et al. Association of telomere length with risk of disease and mortality. JAMA Inter Med. 182 (3), 291-300 (2022).
- Deng, Y., et al. Telomere length and the risk of cardiovascular diseases: A mendelian randomization study. Front Cardiovasc Med. 9, 1012615(2022).
- D'mello, M. J., et al. Association between shortened leukocyte telomere length and cardiometabolic outcomes: Systematic review and meta-analysis. Circulation: Cardiovasc Gene. 8 (1), 82-90 (2015).
- Wilbourn, R. V., et al. The relationship between telomere length and mortality risk in non-model vertebrate systems: A meta-analysis. Philos Trans R Soc Lond B Biol Sci. 373 (1741), 20160447(2018).
- Gardner, M., et al. Gender and telomere length: Systematic review and meta-analysis. Exp Gerontol. 51, 15-27 (2014).
- Lai, T. -P., Wright, W. E., Shay, J. W. Comparison of telomere length measurement methods. Philos Trans R Soc Lond B Biol Sci. 373 (1741), 20160451(2018).
- Cawthon, R. Telomere measurement by quatitative pcr. Nuc Acid Res. 30 (10), e47-e53 (2002).
- Cawthon, R. M. Telomere length measurement by a novel monochrome multiplex quantitative pcr method. Nuc Acid Res. 37 (3), e21-e21 (2009).
- Lindrose, A. R., et al. Method comparison studies of telomere length measurement using qpcr approaches: A critical appraisal of the literature. PLoS One. 16 (1), e0245582(2021).
- Lin, J., et al. Effects of DNA extraction, DNA integrity, and laboratory on the precision of qpcr-based telomere length measurement - a multi-lab impartial study. bioRxiv. , (2022).
- Wilfinger, W. W., Mackey, K., Chomczynski, P. Effect of ph and ionic strength on the spectrophotometric assessment of nucleic acid purity. Biotechniques. 22 (3), 474-481 (1997).
- Boesenberg-Smith, K. A., Pessarakli, M. M., Wolk, D. M. Assessment of DNA yield and purity: An overlooked detail of pcr troubleshooting. Clin Microbiol Newsletter. 34 (1), 1-6 (2012).
- Harley, C. B., Futcher, A. B., Greider, C. W. Telomeres shorten during ageing of human fibroblasts. Nature. 345 (6274), 458-460 (1990).
- Kimura, M., et al. Measurement of telomere length by the southern blot analysis of terminal restriction fragment lengths. Nat Protoc. 5 (9), 1596-1607 (2010).
- Ye, Q., et al. Telomere length and chronological age across the human lifespan: A systematic review and meta-analysis of 414 study samples including 743,019 individuals. Ageing Res Rev. 90, 102031(2023).
- Axelrad, M. D., Budagov, T., Atzmon, G. Telomere length and telomerase activity; a yin and yang of cell senescence. J Vis Exp. (75), e50246(2013).
- Liu, M., et al. Immune-mediated inflammatory diseases and leukocyte telomere length: A mendelian randomization study. Front Genetics. 14, 1129247(2023).
- Van Ockenburg, S., et al. Stressful life events and leukocyte telomere attrition in adulthood: A prospective population-based cohort study. Psychol Med. 45 (14), 2975-2984 (2015).
- Zong, Z. Q., et al. Ambient air pollution exposure and telomere length: A systematic review and meta-analysis. Public Health. 215, 42-55 (2023).
- Tang, L., Li, D., Wang, J., Su, B., Tian, Y. Ambient air pollution, genetic risk and telomere length in uk biobank. J Expo Sci Environ Epidemiol. , (2023).
- Aubert, G., Hills, M., Lansdorp, P. Telomere length measurement-caveats and a critical assessment of the available technologies and tools. Mutat Res. 730 (1), 59-67 (2012).
- Lin, J., et al. Systematic and cell type-specific telomere length changes in subsets of lymphocytes. J Immunol Res. 2016, 5371050(2016).
- Needham, B. L., et al. Socioeconomic status, health behavior, and leukocyte telomere length in the national health and nutrition examination survey, 1999-2002. Soc Sci Med. 85, 1-8 (2013).
- Verhulst, S. Improving comparability between qpcr-based telomere studies. Mol Ecol Res. 20 (1), 11-13 (2020).
- Dagnall, C. L., et al. Effect of pre-analytic variables on the reproducibility of qpcr relative telomere length measurement. PloS one. 12 (9), e0184098(2017).
- Lin, J., Smith, D. L., Esteves, K., Drury, S. Telomere length measurement by qpcr-summary of critical factors and recommendations for assay design. Psychoneuroendocrinology. 99, 271-278 (2019).
- Wolf, S. E., et al. Cross-tissue comparison of telomere length and quality metrics of DNA among individuals aged 8 to 70 years. PLOS One. 19 (2), e0290918(2024).
- RoDer, B., FruHwirth, K., Vogl, C., Wagner, M., Rossmanith, P. Impact of long-term storage on stability of standard DNA for nucleic acid-based methods. J Clin Microbiol. 48 (11), 4260-4262 (2010).
- Dagnall, C., et al. Effect of pre-analytic variables on the reproducibility of qpcr relative telomere length measurement. PLoS One. 12 (9), e0184098(2017).
- Shao, W., Khin, S., Kopp, W. C. Characterization of effect of repeated freeze and thaw cycles on stability of genomic DNA using pulsed field gel electrophoresis. Biopreserv Biobank. 10 (1), 4-11 (2012).
- Institute for Statistics and Mathematics of WU. The Comprehensive R Archive Network. , Available from: https://cran.r-project.org/ (2024).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biol Rev. 85 (4), 935-956 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon