Method Article
Monochrome Multiplex-Quantitative PCR-Telomerlängenmessung
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll zur Messung der relativen Telomerlänge (TL) mit dem monochromen Multiplex-Assay der quantitativen Polymerase-Kettenreaktion (MMqPCR) vor. Der MMqPCR-Assay ist eine wiederholbare, effiziente und kostengünstige Methode zur Messung von TL aus menschlicher DNA in populationsbasierten Studien.
Zusammenfassung
Telomere sind Ribonukleoproteinstrukturen am Ende aller eukaryotischen Chromosomen, die die DNA vor Schäden schützen und die Chromosomenstabilität erhalten. Die Telomerlänge (TL) wurde mit verschiedenen Expositionen, biologischen Prozessen und gesundheitlichen Folgen in Verbindung gebracht. Dieser Artikel beschreibt das monochrome Multiplex-Assay-Protokoll für die quantitative Polymerase-Kettenreaktion (MMqPCR), das routinemäßig in unserem Labor zur Messung der relativen mittleren TL aus menschlicher DNA durchgeführt wird. Es gibt mehrere verschiedene PCR-basierte TL-Messmethoden, aber das in dieser Veröffentlichung vorgestellte spezifische Protokoll für die MMqPCR-Methode ist wiederholbar, effizient, kostengünstig und für populationsbasierte Studien geeignet. Dieses detaillierte Protokoll enthält alle Informationen, die die Prüfärzte benötigen, um diesen Assay in ihrem Labor zu etablieren. Darüber hinaus bietet dieses Protokoll spezifische Schritte zur Erhöhung der Reproduzierbarkeit der TL-Messung durch diesen Assay, definiert durch den Intraklassen-Korrelationskoeffizienten (ICC) bei wiederholten Messungen derselben Probe. Der ICC ist ein kritischer Faktor bei der Bewertung der erwarteten Aussagekraft für eine bestimmte Studienpopulation; Daher ist die Berichterstattung über kohortenspezifische ICCs für jeden TL-Assay ein notwendiger Schritt, um die allgemeine Strenge populationsbasierter Studien zu TL zu verbessern. Beispielergebnisse mit DNA-Proben, die aus mononukleären Zellen des peripheren Blutes extrahiert wurden, zeigen die Machbarkeit der Generierung hochgradig wiederholbarer TL-Daten mit diesem MMqPCR-Protokoll.
Einleitung
Telomere sind schützende Komplexe, die sich am Ende aller eukaryotischen Chromosomen befinden und aus hochkonservierten, sich wiederholenden DNA-Sequenzen und assoziierten Proteinen bestehen. Das Telomer schützt die Integrität der DNA und bewahrt die Stabilität der Chromosomen. Eine fortschreitende Verkürzung der Telomere tritt in sich teilenden Zellen als Folge einer unvollständigen DNA-Synthese im Nachlaufstrang, DNA-Schäden und anderer Faktoren auf 1,2. Die zunehmende Evidenz für die Telomerlänge (TL) als Biomarker für das Altern und altersbedingte Krankheiten über die gesamte menschliche Lebensspanne hinweg ging einher mit einer Zunahme der Arten von TL-Messassays, die zur Bewertung der Rolle von TL in Studien zur Exposition, Krankheit und Gesundheit des Menschen verwendet wurden 3,4,5. Metaanalysen haben Assoziationen von TL mit der Gesamtmortalität, Umwelteinflüssen und gesundheitlichen Folgen, einschließlich Krebs, Herz-Kreislauf-Erkrankungen und Diabetes, berichtet 6,7,8,9,10,11,12,13 . Diese Meta-Assoziationen stammen aus Studien, die eine von mehr als zwei Dutzend verschiedenen TL-Messmethoden verwenden, wobei die Stärken der Assoziationen tendenziell zwischen verschiedenen Methoden variieren 14,15,16. Die Auswahl der optimalen TL-Messmethode für eine Forschungsstudie ist ein entscheidender Schritt, um genaue Ergebnisse zu gewährleisten, da jede Methode ihre eigenen Vor- und Nachteile hat 5,17.
Aufgrund der relativ niedrigen Kosten für Reagenzien, der schnellen Assay-Durchlaufzeit, der Skalierbarkeit und des geringeren anfänglichen DNA-Bedarfs werden PCR-basierte TL-Messtechniken häufig bevorzugt bei der Durchführung von Studien mit großen Probenpopulationen, Studien mit begrenztem Zugang zu Proben mit hohen DNA-Konzentrationen oder bei Studien mit hohem Durchsatz eingesetzt. Die erste PCR-basierte Methode zur TL-Messung, die ursprünglich von Richard Cawthon entwickelte quantitative Singleplex-Polymerase-Kettenreaktion (qPCR), nutzt das Verhältnis der Fluoreszenzsignale der Telomer- (T) und Einzelkopiegen-Amplifikation (S), die auf separaten PCR-Platten durchgeführt werden18. Bei diesem Ansatz werden Primer, die zu den wiederholten Telomer-DNA-Sequenzen (T) komplementär sind, verwendet, um den Gesamttelomer-DNA-Gehalt in der Probe zu amplifizieren und durch Detektion des fluoreszierenden Reporters SYBR grün zu quantifizieren. In ähnlicher Weise werden Primer, die zu einer intergenen Region eines konservierten Einzelkopie-Gens (S) komplementär sind, verwendet, um die Anzahl der Genomkopien zu quantifizieren. Diese beiden Schätzungen werden relativ zu einer genomischen DNA-Standardkurve quantifiziert, die für alle Assays in einem Projekt verwendet wird, um die Variation von Platte zu Platte zu kontrollieren. Dividiert man die gesamte Telomer-DNA (T) durch die Kopienzahl (S) des einzelnen Genoms, erhält man das T/S-Verhältnis, eine einheitslose, relative Messung, die den durchschnittlichen Telomergehalt pro Zelle für eine einzelne DNA-Probe darstellt18,19. Das T/S-Verhältnis ist also kein spezifisches Maß für die Funktionslänge; In Übereinstimmung mit den Literaturnormen verwenden wir jedoch in diesem Protokoll den Begriff durchschnittliche TL pro Probe.
Diese Methode wurde 2009 weiterentwickelt, als Richard Cawthon den monochromen Multiplex-qPCR-Assay (MMqPCR) als einen Ansatz beschrieb, um die Variabilität des T/S-Verhältnisses im Vergleich zur ursprünglichen Singleplex-qPCR-Methode möglicherweise zu reduzieren19. Der MMqPCR-Assay besitzt die Vorteile des qPCR-Assays mit dem zusätzlichen Vorteil, dass T- und S-Signale innerhalb derselben Reaktionsvertiefung unter Verwendung eines Berichtsfluorophors gemessen werden, wodurch der Fehler im Vergleich zur Singleplex-qPCR verringert und eine höhere Präzision und Reproduzierbarkeit erreichtwird 19. Darüber hinaus senkt diese Multiplex-Methode potenziell die Kosten und erhöht den Durchsatz, da im Vergleich zum Singleplex-Assay19 nur halb so viele Reaktionen erforderlich sind.
Angesichts der Vorteile der MMqPCR-TL-Messung eignet sich diese Methode gut für populationsbasierte Studien zu TL-Assoziationen mit Expositionen, gesundheitlichen Ergebnissen und biologischen Prozessen. Die Einführung der Methode kann jedoch eine Herausforderung darstellen. Um diesen Herausforderungen zu begegnen, beschreiben wir detailliert das in unserem Labor verwendete MMqPCR TL-Messprotokoll und heben die wichtigsten Schritte hervor, die implementiert wurden, um die Assay-Präzision zu erhöhen, das Kontaminationsrisiko zu verringern und die Wiederholbarkeit zu verbessern.
Darüber hinaus beschreibt dieses Protokoll die Schritte zur Bereinigung der Daten und zur Berechnung des Intraklassen-Korrelationskoeffizienten (ICC), einem wichtigen statistischen Maß für die Reproduzierbarkeit von TL-Messungen2. Mit unseren repräsentativen Ergebnissen demonstrieren wir, dass wir in der Lage sind, mit diesem Protokoll hohe ICCs zu generieren. Darüber hinaus identifizieren wir Schritte zur Qualitätskontrolle (QC) und Fehlerbehebung, von denen erwartet wird, dass sie die Variation in den TL-Messungen verringern und die resultierende ICC erhöhen. Aufgrund der hohen Wiederholbarkeit, Effizienz und Kosteneffizienz dieser Methode ist die MMqPCR TL-Messung ideal für die epidemiologische TL-Forschung. Abbildung 1 zeigt einen visuellen Überblick über die MMqPCR-Methode, wie sie in diesem Protokoll beschrieben wird.
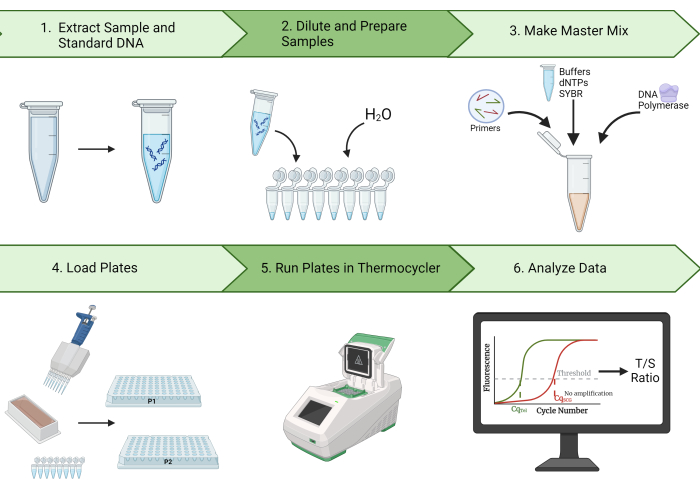
Abbildung 1: Übersicht über die Methode. Ein breiter Überblick über die monochrome Multiplex-Methode der quantitativen Polymerase-Kettenreaktion zur Messung der Telomerlänge. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protokoll
Diese Forschung wurde in Übereinstimmung mit den institutionellen Richtlinien durchgeführt. Dieses Protokoll beschreibt den MMqPCR-Assay, der in einer Messtiefe von doppelten Triplikaten durchgeführt wird, d. h. dreifache Messungen jeder Probe werden über doppelte Platten wiederholt, wobei zwei Thermocycler gleichzeitig durchgeführt werden, um den Durchsatz zu erhöhen. Die Verwendung von Duplikatplatten ist eine der wichtigsten Überlegungen bei der Implementierung dieses Protokolls, um eine hohe Reproduzierbarkeit zu erreichen, die durch hohe ICCs angezeigt wird. Obwohl es möglich ist, weniger Replikate zu verwenden, müssen die Auswirkungen auf die ICC und damit auf die Leistung und die erforderliche Probengröße sorgfältig abgewogen und mit jeder einzelnen Kohorte von jedem Labor gemessen werden20,21. Wenn nur ein Thermocycler verfügbar ist, empfehlen wir, die Messtiefe beizubehalten (d. h. Triplikate zu duplizieren) und 2 Platten nacheinander laufen zu lassen.
1. Vorbereitung und Lagerbedingungen
- Informationen zu allen Materialien und Geräten, die für dieses Protokoll verwendet werden, finden Sie in der Materialtabelle. Weitere Informationen zu den spezifischen Mastermix-Reagenzien und den für den MMqPCR-Assay verwendeten Mengen finden Sie in Tabelle 1. Wenn zwei Platten nacheinander laufen, halbieren Sie das Mastermix-Volumen bei gleichbleibender Konzentration, wobei sichergestellt wird, dass dieselben Reagenzien-Aliquots auf aufeinanderfolgenden Platten desselben Assays verwendet werden.
HINWEIS: Alle Stammreagenzien sollten im Voraus vorbereitet werden und aliquotierte Reagenzien sollten vor der Verwendung vollständig aufgetaut werden. - Aliquotieren und lagern Sie 1x Tris-EDTA, pH 7,6 (TE) in 5 mL Röhrchen in 3 mL Volumen bei Raumtemperatur für bis zu 2 Jahre.
- Aliquotieren und lagern Sie 5 M Betain in 5-ml-Röhrchen in einem Volumen von 1.280 μl bei -20 °C für bis zu 1 Jahr.
- Aliquotieren und lagern Sie SYBR Green in 3 μL-Volumina in einzelnen PCR-Streifenröhrchen bei -20 °C für bis zu 2 Jahre, mit Folie bedeckt und minimal Lichteinwirkung.
- Lagern Sie die DNA-Polymerase in Originalröhrchen bei -20 °C für bis zu 1 Jahr.
- Aliquotieren und lagern Sie 10x Polymerase-Puffer in 1,5 mL Röhrchen in 660 μl Volumen bei -20 °C für bis zu 1 Jahr.
- Für 1x-Pufferverdünnungen von 10x Polymerase-Puffer 9,9 μl 10x Puffer zu 89,1 μl PCR-Qualität H2O in 0,5-ml-Röhrchen hinzufügen; Bei -20 °C bis zu 1 Jahr lagern.
- Aliquotieren und lagern Sie 1 M MgCl2 in 0,5 mL Röhrchen in 70 μl Volumen bei 4 °C Lichtschutz für bis zu 1 Jahr.
- Lagern Sie die lyophilisierten Primer für das Vorwärts- und Rückwärts-Telomer (T) und das Einzelkopie-Gen-Oligonukleotid bei Raumtemperatur ohne Licht. Die vier spezifischen Oligonukleotid-Primer-Sequenzen sind in Tabelle 2 dargestellt.
HINWEIS: Das Einzelkopie-Gen in diesem Protokoll ist Albumin; Wenn Sie ein anderes Einzelkopie-Gen wählen, müssen die Primer-Konzentrationen möglicherweise angepasst werden, um eine akzeptable PCR-Effizienz zu gewährleisten.- Rekonstitution des Primers: Jedes lyophilisierte Primerröhrchen 10 s lang vortexen und dann die Röhrchen 5 s lang in eine Minizentrifuge legen, um sicherzustellen, dass sich das lyophilisierte Pellet am Boden des Röhrchens befindet, bevor 1x TE Puffer hinzugefügt wird.
- Überprüfen Sie jedes einzelne Primerrohr auf nM-Konzentration ([nM]= alpha) und rehydrieren Sie jedes Primerröhrchen, indem Sie 1x TE entsprechend dem 10-fachen des Alpha-Wertes in μL hinzufügen (z. B. wenn die nM-Konzentration auf dem Röhrchen 24,6 nM beträgt, fügen Sie 246 μl 1x TE zum Primerröhrchen hinzu), um eine 100 μM-Lösung jedes Primers herzustellen.
- Aliquot Vorwärts- und Rückwärts-Telomer-Primer getrennt in 16 μL-Volumina und Vorwärts- und Rückwärts-Albumin-Primer getrennt in 11 μL-Volumina. Lagern Sie alle vier Arten von Primer-Aliquoten bis zu 6 Monate bei -20 °C.
- dNTP-Mischung: 4-8 Röhrchen von jedem der vier dNTPS-Typen von 25 mM-Stammlösungen (d. h. insgesamt 16-32 Röhrchen) für 10 s vortexen, gefolgt von einem schnellen Schleudern bei Raumtemperatur unter Verwendung einer Minizentrifuge. Platzieren Sie die Röhrchen in einer gleichmäßigen Verteilung in den Röhrchenpositionen in der Minizentrifuge und schließen Sie dann den Deckel, damit die Maschine die volle Geschwindigkeit erreichen und sich etwa 5 s lang drehen kann.
- In ein 5-ml-Röhrchen geben Sie 200-250 μl aus jedem Röhrchen und stellen Sie sicher, dass jeder der vier dNTP-Typen zu gleichen Teilen hinzugefügt wird, und wirbeln Sie das dNTP-Gemisch ein.
- 210 μl des Gemisches in 0,5 ml-Röhrchen aliquotieren und bis zu 1 Jahr bei -20 °C lagern.
- Lagern Sie den Vorrat an DTT bei -20 °C und lagern Sie 3 M Natriumacetat bei Raumtemperatur.
- DVB-T-Lösung: Messen Sie 0,1545 g DVB-T in einem tarierten Waagschiffchen auf einer Waage mit einem Edelstahlspatel.
ACHTUNG: Dithiothreitol (DTT) ist ein gefährliches Reagenz. DVB-T ist schädlich, wenn es verschluckt wird, verursacht Hautreizungen und kann schwere Augenschäden verursachen. Einzelpersonen sollten beim Umgang mit DVB-T Schutzhandschuhe, Augenschutz und Gesichtsschutz tragen. Darüber hinaus sollten Personen das Einatmen von DTT-Dämpfen vermeiden, beim Umgang mit DTT in einer PCR-Haube arbeiten und eine längere oder wiederholte Exposition vermeiden. - Stellen Sie 10 ml 0,01 M Natriumacetat her, indem Sie 33,33 μl 3 M Natriumacetat und 9.967 μl PCR-Qualität H2O in ein 15-ml-Röhrchen und eine Wirbelvertiefung geben. Geben Sie das gemessene DTT in das 15-ml-Röhrchen mit 0,01 M Natriumacetat. Etwa 100 μl der 0,01 M Natriumacetatlösung werden in das Wägeschiffchen gegeben, um das restliche DVB-t aufzufangen. Pipettieren Sie die 100 μl zurück in das 15 ml-Röhrchen. Sobald sich das gesamte DTT in der Lösung befindet, wirbeln Sie das Röhrchen ein, bis es sich vollständig aufgelöst hat.
- Gießen Sie den Inhalt des 15-ml-Röhrchens in eine Ladewanne und saugen Sie die gesamte Lösung aus einem Ende der Ladewanne in eine 10-ml-Kunststoffspritze ab. Befestigen Sie einen sterilen 0,2 μm Celluloseacetat-Spritzenvorsatzfilter 25 mm am Ende der gefüllten Spritze, lassen Sie ihn 1 Minute lang vollständig sättigen und tropfen Sie die Lösung langsam in ein neues 15-ml-Röhrchen, indem Sie den Spritzenkolben leicht nach unten drücken, wobei Sie sicherstellen, dass die gesamte Lösung durch den Filter in das neue 15-ml-Röhrchen tropft.
- Die DTT-Lösung in einem Volumen von 200 μl in 0,5 ml-Röhrchen aliquotieren und bis zu 6 Wochen bei -20 °C lagern.
- DVB-T-Lösung: Messen Sie 0,1545 g DVB-T in einem tarierten Waagschiffchen auf einer Waage mit einem Edelstahlspatel.
Tabelle 1: Endvolumina und Konzentrationen der Reagenzien. Volumina und Konzentrationen von Reagenzien in einzelnen Aliquoten, Mastermixen und in PCR-Wells. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 2: Telomer- und Einzelkopie-Gen-Oligonukleotid-Primersequenzen. Liste der Telomer- und Albumin-Einzelkopien-Genprimersequenzen, die in der Methodik verwendet werden. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
2. Genomische DNA-Extraktion und Probenvorbereitung
- Extraktion von analytischen Proben: Führen Sie eine genomische DNA-Extraktion für Proben gemäß den Richtlinien des Herstellers unter Verwendung von Kits oder etablierten Methoden im Labor durch.
- Überprüfen Sie die Qualität der DNA-Probe mit einem Spektralphotometer und doppelsträngige DNA (dsDNA) mit einem Fluorometer. Proben mit einem inakzeptablen durchschnittlichen Verhältnis von 260/280 und 260/230 oder einer dsDNA-Konzentration unterhalb des Nachweises (<0,20 ng/μl) sollten nicht auf TL22,23 analysiert werden. Wenn die dsDNA-Konzentration zu hoch ist, um mit dem Fluorometer einen Messwert zu erzeugen (>1000 ng/μl), verdünnen Sie die Probe und wiederholen Sie den Assay.
HINWEIS: Für die hier vorgestellten Ergebnisse waren die akzeptablen Werte für die Reinheit von Nukleinsäuren ein 260/280-Verhältnis zwischen 1,6 und 2,0 und ein 260/230-Verhältnis zwischen 2,0 und 2,2.
- Überprüfen Sie die Qualität der DNA-Probe mit einem Spektralphotometer und doppelsträngige DNA (dsDNA) mit einem Fluorometer. Proben mit einem inakzeptablen durchschnittlichen Verhältnis von 260/280 und 260/230 oder einer dsDNA-Konzentration unterhalb des Nachweises (<0,20 ng/μl) sollten nicht auf TL22,23 analysiert werden. Wenn die dsDNA-Konzentration zu hoch ist, um mit dem Fluorometer einen Messwert zu erzeugen (>1000 ng/μl), verdünnen Sie die Probe und wiederholen Sie den Assay.
- Extraktion von Kontrollproben: Führen Sie eine genomische DNA-Extraktion an einer Kontrollprobe durch, die aus demselben biologischen Material stammt wie die Probandenproben, die für ein bestimmtes Projekt untersucht werden. Stellen Sie sicher, dass ausreichend Kontroll-DNA für alle Platten extrahiert wird, die voraussichtlich für die gesamte Kohorte ausgeführt werden, so dass ein einziger DNA-Standard für alle Assays verwendet wird.
- Überprüfen Sie die Qualität der Kontroll-DNA-Probe mit Spektralphotometer und Fluorometer. Die Kontrollprobe sollte ein akzeptables durchschnittliches Verhältnis von 260/280 und 260/230 sowie nachweisbare Konzentrationen von dsDNA 22,23 aufweisen.
- Aliquot kontrolliert die DNA in einer Konzentration von 2 ng/μl in 150 μl Mengen in 0,5 mL Röhrchen, die mit dem Probentyp und dem Datum markiert sind. Lagern Sie die Kontroll-DNA-Aliquoten bis zu 5 Jahre lang bei -20 °C. Verwenden Sie eine Kontroll-DNA-Probe für alle Platten aller Proben aus derselben Kohorte oder demselben Forschungsprojekt. Bereiten Sie diese Stammaliquote im Voraus vor.
- Vorbereitung von analytischen Proben: Für Proben mit einer Ausgangskonzentration von >5 ng/μl erstellen Sie Verdünnungsaliquote unter Verwendung eines ganzzahligen Verdünnungsfaktors, indem Sie die entsprechende Menge der extrahierten DNA-Probe (Supplementary File 1 MMqPCR Set Up Sheet, Spalte F) und der PCR-Klasse H2O (Supplementary File 1 MMqPCR Set Up Sheet, Spalte G) hinzufügen, um die Konzentration auf 2,5 - 5,0 ng/μL zu verdünnen (Supplementary File 1 MMqPCR-Einrichtungsblatt, Spalte K). Verdünnen Sie keine Proben mit einer Konzentration <5 ng/μl, aliquotieren Sie jedoch genügend Volumen, um die Probe 3x mit diesem Protokoll zu testen.
HINWEIS: Verdünnungsaliquote müssen während der Probenvorbereitung hergestellt werden, so dass 15 ng DNA in das entsprechende PCR-Streifenröhrchen jeder Probe gegeben werden können. Die Probenaliquoten ermöglichen es Technikern, unnötige Gefrier-Tau-Zyklen zu vermeiden, die die Integrität der Stammprobe beeinträchtigen, wenn die Probe die Qualitätskriterien nicht erfüllt und erneut durchgeführt werden muss. Das MMqPCR-Proben-Template in der Ergänzungsdatei 1 kann dabei helfen, den korrekten Verdünnungsfaktor und die richtigen Volumina für die Herstellung von Verdünnungsaliquoten zu identifizieren (Ergänzende Datei 1, MMqPCR Setup Sheet, Spalten E-I).- Füllen Sie die PCR-Streifen mit den Proben A1-8 im PCR-Streifen A, den Proben B1-8 im PCR-Streifen B und den Proben C1-8 im PRC-Streifen C, wie im MMqPCR-Einrichtungsblatt für die Zusatzdatei 1 beschrieben und in Tabelle 3 aufgeführt, mit entsprechend verdünnten Probenmengen (Zusatzdatei 1 MMqPCR-Einrichtungsblatt, Spalte L) und Mengen der PCR-Klasse H2O (Zusatzdatei 1 ) MMqPCR Setup Up Sheet, Spalte M) für eine Gesamtmenge von 15 ng DNA pro PCR-Röhrchen und ein Gesamtvolumen von 75 μl. Beiseite stellen.
HINWEIS: Proben in PCR-Streifen können über Nacht bei 4 °C gelagert werden, um am nächsten Tag plattiert zu werden.
- Füllen Sie die PCR-Streifen mit den Proben A1-8 im PCR-Streifen A, den Proben B1-8 im PCR-Streifen B und den Proben C1-8 im PRC-Streifen C, wie im MMqPCR-Einrichtungsblatt für die Zusatzdatei 1 beschrieben und in Tabelle 3 aufgeführt, mit entsprechend verdünnten Probenmengen (Zusatzdatei 1 MMqPCR-Einrichtungsblatt, Spalte L) und Mengen der PCR-Klasse H2O (Zusatzdatei 1 ) MMqPCR Setup Up Sheet, Spalte M) für eine Gesamtmenge von 15 ng DNA pro PCR-Röhrchen und ein Gesamtvolumen von 75 μl. Beiseite stellen.
- Vorbereitung der Standardkurve: Tauen Sie ein Kontroll-DNA-Aliquot auf, wirbeln Sie es 30 s lang, zentrifugieren Sie es 5 s lang und aspirieren Sie das volle Volumen (150 μl) in das erste Röhrchen des PCR-Röhrchenstreifens (SC).
- Pipettieren Sie 70 μl PCR-Grade H2O in das zweite bis achte Röhrchen des SC-PCR-Streifens. Resuspendieren Sie die Kontroll-DNA gründlich im ersten Röhrchen, bevor Sie 70 μl aspirieren und in das zweite PCR-Röhrchen abgeben. Warten Sie 30 s und resuspendieren Sie die Lösung dann gründlich im zweiten PCR-Röhrchen, bevor Sie 70 μl aspirieren und in das dritte PCR-Röhrchen abgeben. Wiederholen Sie dies für die Röhrchen drei bis sieben, um eine 2-fache serielle Verdünnung von sieben Standards zu erzeugen. Das endgültige Röhrchen sollte nur PCR-Qualität H2O enthalten, um als Nicht-Template-Kontrolle (NTC) zu fungieren. Legen Sie den SC-PCR-Streifen beiseite.
Tabelle 3: Organisation der Proben und Kontrollstandard in der Platte. Position aller Proben und Standards auf einer 96-Well-PCR-Platte. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
3. MMqPCR-Mastermix-Zubereitung
- Sammeln Sie die in Tabelle 1 aufgeführten Mastermix-Reagenzienaliquoten, mit Ausnahme der DNA-Polymerase, und lassen Sie sie in der PCR-Haube auf Raumtemperatur kommen. Fügen Sie 1 μl des SYBR Green Aliquot zum 1x Puffer Aliquot hinzu.
HINWEIS: Diese genaue Menge wird benötigt, um die korrekte Konzentration von SYBR zu erzeugen, da die Pipettengenauigkeit während des Assays eingeschränkt ist, so dass diese Menge auch dann unverändert bleiben sollte, wenn jeweils nur eine Platte ausgeführt wird. - Alle Aliquoten 10 s lang vortexen, 5 s zentrifugieren, dann die folgenden Mengen an Reagenzien in das 5-ml-Röhrchen geben, das das 5-M-Betain enthält (jetzt das Mastermix-Röhrchen): 1.235,2 μl PCR-Grade H2O, 640 μl des 10x-Puffers, 204,8 μl der dNTPs, 192 μl des DTT, 64 μl des MgCl2, 48 μl SYBR Green / 1x Puffer (aus Schritt 3.1), 14,4 μl beider Telomerprimer und 9,6 μl beider Albuminprimer, wie in Tabelle 1 aufgeführt.
- Nehmen Sie die DNA-Polymerase aus der -20 °C-Lagerung, wirbeln Sie sie 10 s lang, zentrifugieren Sie sie 5 s lang und fügen Sie dann langsam 128 μl zum Mastermix hinzu. Bringen Sie die DNA-Polymerase sofort wieder in die -20 °C-Lagerung.
- Vortexen Sie das 5 mL Mastermix-Röhrchen für 30 s.
4. Vorbereitung der 96-Well-Platte
- Platzieren Sie zwei 96-Well-Platten, eine Laderinne und Mehrkanal-Pipettenspitzen in der PCR-Haube. Beschriften Sie jedes Schild mit der Plattennummer (1 oder 2). Drehen Sie die erste Platte (die P1-Platte) um 180°, so dass die Nummern der Spalten auf dem Kopf stehen. Lassen Sie die zweite Platte (P2) in Richtung des Technikers.
- Gießen Sie den Inhalt des 5-mL-Mastermix-Röhrchens in die Ladewanne. Verwenden Sie eine Pipettenspitze, um die im Röhrchen verbleibende Lösung aufzufangen und abzugeben.
- Legen Sie die Spitzen auf die Mehrkanalpipette und drücken Sie sie an die Basis jeder Pipettenspitze, um eine dichte Versiegelung zu gewährleisten. Stellen Sie sicher, dass die Spitzen keinen Filter innerhalb des Ausgangsbereichs haben, in dem der Mastermix aspiriert wird. Wenn ein Stück Filter vorhanden ist, ersetzen Sie die Spitze.
- Verwenden Sie das Rückwärtspipettieren, um die Platten mit dem viskosen Mastermix zu füllen, schwenken Sie die Ladewanne und drücken Sie dann den Pipettenkolben über den ersten Anschlag hinaus, saugen Sie mehr als 15 μl in die Pipettenspitzen an und stellen Sie sicher, dass sich die Spitzen gleichzeitig mit dem gleichen Volumen füllen. Wenn Sie den Mastermix in eine Säule ausstoßen, drücken Sie den Kolben bis zum ersten Anschlag und lassen Sie den zusätzlichen Mastermix in den Pipettenspitzen. Lassen Sie den Kolben gedrückt, tauchen Sie die Spitzen wieder in die Ladewanne und lassen Sie den Kolben los, um die Spitzen mit weiteren 15 μl zu füllen. Verwenden Sie die gleichen Spitzen, um beide Platten zu füllen.
- Füllen Sie die Platten, indem Sie den Mastermix zuerst in alle geraden oder alle ungeraden Spalten auswerfen, abwechselnd (d. h. wenn der Techniker mit ungeraden Spaltennummern beginnt, wird zuerst Spalte 1 auf P2 gefüllt, gefolgt von Spalte 11 auf P1, dann Spalte 3 auf P2 usw.). Nachdem der Techniker alle Vertiefungen der Reihe (ungerade oder gerade) von links nach rechts gefüllt hat, führen Sie die anderen Reihen (gerade oder ungerade) von Säulen auf die gleiche Weise aus. In Abbildung 2 finden Sie ein Diagramm dieses Prozesses.
HINWEIS: Diese Art der Plattenfüllung dient zur Steuerung von Positionseffekten auf den Platten.
- Füllen Sie die Platten, indem Sie den Mastermix zuerst in alle geraden oder alle ungeraden Spalten auswerfen, abwechselnd (d. h. wenn der Techniker mit ungeraden Spaltennummern beginnt, wird zuerst Spalte 1 auf P2 gefüllt, gefolgt von Spalte 11 auf P1, dann Spalte 3 auf P2 usw.). Nachdem der Techniker alle Vertiefungen der Reihe (ungerade oder gerade) von links nach rechts gefüllt hat, führen Sie die anderen Reihen (gerade oder ungerade) von Säulen auf die gleiche Weise aus. In Abbildung 2 finden Sie ein Diagramm dieses Prozesses.
- Die vier geschlossenen PCR-Streifen, die die Proben und die Standardkurve enthalten, werden dann jeweils 5 s lang in einer Mini-Zentrifuge gedreht.
- Richten Sie die PCR-Streifen im PCR-Röhrchengestell in der Reihenfolge aus, in der sie auf der Platte platziert werden. Für P1 sind die ersten 3 Spalten Proben (PCR-Streifen A), die Spalten 4-6 sind der Standard (PCR-Streifen S), die Spalten 7-9 sind Proben (PCR-Streifen B) und die Spalten 10-12 sind Proben (PCR-Streifen C; siehe Abbildung 3 und Tabelle 3). Da P2 gefüllt wird, wenn er um 180° in Bezug auf den Techniker gedreht wird, sind die Säulen umgekehrt.
- Drehen Sie beide Platten um 180°, sodass die Nummern der Spalten in Bezug auf den Techniker für jede Platte umgedreht werden (d. h. wenn der Techniker zuvor auf die Beschriftung für P2 gerichtet war, zeigt er jetzt auf die Beschriftung für P1 an der Plattenkante).
- Stellen Sie die Mehrkanalpipette auf 10 μl ein und laden Sie die Pipettenspitzen auf die Mehrkanalpipette, wie in Schritt 4.3 beschrieben.
- Verwenden Sie die Mehrkanalpipette, um die Lösungen zu resuspendieren, und führen Sie dann das Rückpipettieren wie in Schritt 4.4 beschrieben durch, um 10 μl der Proben und Standardlösungen abzusaugen und zu dispensieren, beginnend mit dem PCR-Streifen A.
- Füllen Sie die ersten 3 Spalten jeder Platte mit PCR-Streifen A, indem Sie die Säulen auf ähnliche Weise abwechseln, wie der Mastermix in die Platte gelegt wurde (d. h. gerade oder ungerade).
- Füllen Sie die nächsten drei Spalten mit dem Standard-Kurven-PCR-Streifen. Für diesen Streifen resuspendieren Sie das 7. Standardverdünnungsröhrchen (vorletztes) mit einer 200-μl-Pipette auf ~90 μl gründlich, bevor Sie den gesamten Streifen mit der Mehrkanalpipette resuspendieren.
HINWEIS: Das größere Volumen und die geringere DNA-Konzentration in diesem Röhrchen führen aufgrund schlechter Durchmischung häufig zu unterschiedlichen Messungen in den PCR-Plattenvertiefungen. Durch die Verwendung eines größeren Volumens zum Mischen dieses Röhrchens wird die DNA-Lösung besser homogenisiert. - Füllen Sie die nächsten drei Säulen mit PCR-Streifen B und die letzten drei mit PCR-Streifen C. Setzen Sie die Pipettenspitzen zwischen den einzelnen PCR-Streifen wieder ein und füllen Sie alle 96-Wells auf beiden Platten mit den entsprechenden Proben und dem Kontrollstandard, wie in Abbildung 3 dargestellt.
- Sobald die Platten gefüllt sind, klopfen Sie vorsichtig auf die Platte auf der Tischplatte, um sicherzustellen, dass die Flüssigkeit an den Seiten der Vertiefungen nach unten abläuft, und decken Sie dann die Plattenoberseiten mit Siegelfolien ab. Drücken Sie mit den Fingerspitzen auf alle Kanten der Folie, um eine dichte Versiegelung zu gewährleisten.
- Mischen Sie die Platten, indem Sie sie 30 s lang auf der Oberseite der Haube schwenken, und legen Sie dann die versiegelten Platten für 2 Minuten mit den Vertiefungsöffnungen zur Mitte in den Plattenschleuder.
- Legen Sie die Platten in einen Thermocycler, wobei die Nummern der Spalten der Platten in lesbarer Reihenfolge angezeigt werden. Reinigen Sie die Oberseite jeder Platte mit einem sauberen Tuch, bevor Sie die Oberseite des Thermocyclers schließen.
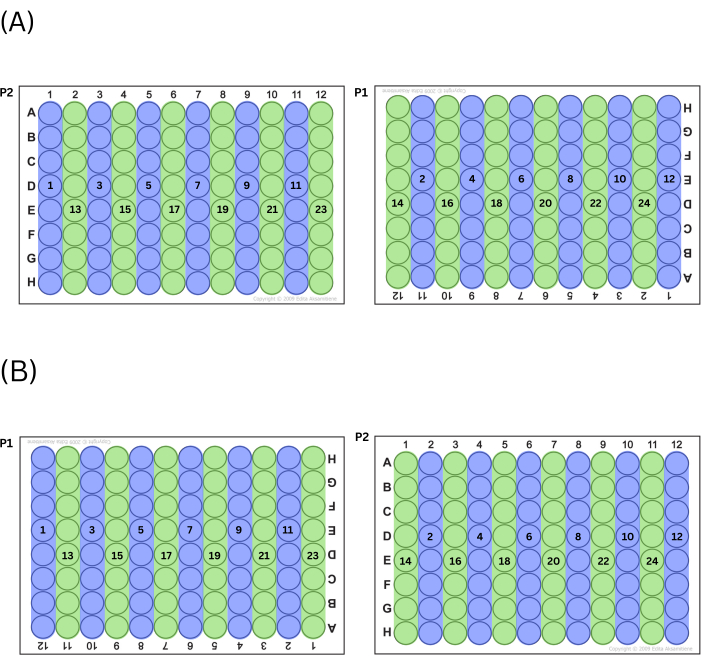
Abbildung 2: Prozess zum Befüllen von Platten. (A) Wenn Sie sich dafür entscheiden, ungerade Spalten zuerst zu füllen, ist dies die Reihenfolge, in der die Vertiefungen gefüllt werden. (B) Wenn Sie sich dafür entscheiden, gerade Spalten zuerst zu füllen, ist dies die Reihenfolge, in der die Vertiefungen gefüllt werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
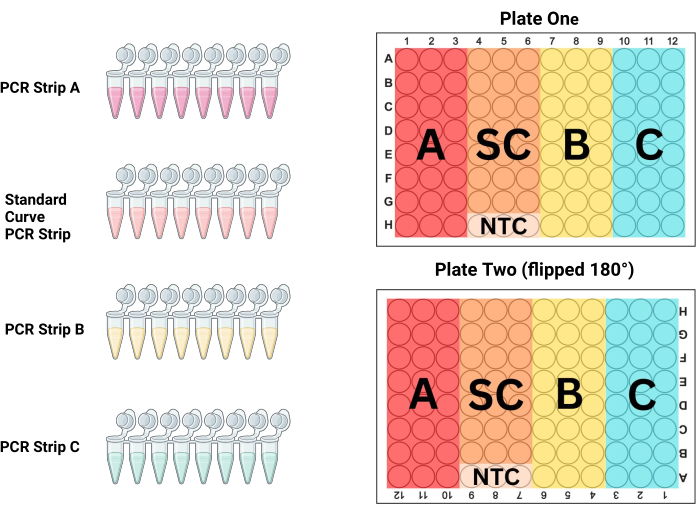
Abbildung 3: Plattenlayout. PCR-Streifen A, Streifen B, Streifen C und der Standardkurvenstreifen (SC) sollten alle verwendet werden, um drei Spalten auf jeder Platte zu füllen, um doppelte Triplikate jeder Probe und Standardverdünnung zu erzeugen. Dieses Diagramm zeigt, welche der Spalten mit welchen Streifen gefüllt werden sollen. Die zweite Platte wird vor dem Laden um 180° gedreht (beachten Sie, dass die Spalten- und Zeilenüberschriften auf dem Kopf stehen), aber die Platte wird identisch mit der ersten Platte gefüllt, wodurch potenzielle Pipettierfehler vermieden werden, während die Positionseffekte auf den Platten weiterhin kontrolliert werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
5. MMqPCR-Thermozylierung
- Während sich die Platten in Schritt 4.8.1 drehen, schalten Sie den Computer und die Thermocycler ein. Öffnen Sie die Thermocycler-Software. Dieses Protokoll beschreibt die Verwendung der CFX Maestro-Software.
- Erstellen Sie das TL-Thermocycling-Protokoll in Übereinstimmung mit dem MMqPCR-Thermocycling-Protokoll19 (Abbildung 4).
- Fügen Sie einen Inkubationsschritt hinzu, um die DNA-Polymerase bei 95 °C für 15 Minuten zu aktivieren.
- Um eine Primer-Dimer-Bindung zu vermeiden, führen Sie zwei Zyklen mit 94 °C für 15 s, 49 °C für 1 Minute und dann 3 Zyklen mit 94 °C für 15 s und 59 °C für 15 s durch.
- Für die Telomerverstärkung fügen Sie 27 Zyklen mit 85 °C für 15 s, 74 °C für 30 s hinzu und dann die Signalerfassung.
- Für die Albuminverstärkung fügen Sie 31 Zyklen mit 94 °C für 15 s, 84 °C für 30 s hinzu und dann die Signalerfassung.
- Nehmen Sie eine Schmelzkurve von 59 °C bis 95 °C in Intervallen von 5 s für jedes zunehmende Grad in das Temperaturwechselprotokoll auf.
- Klicken Sie für beide Thermocycler auf Start Run . Wenn Sie dazu aufgefordert werden, geben Sie einen Titel für die Analysedateien an.
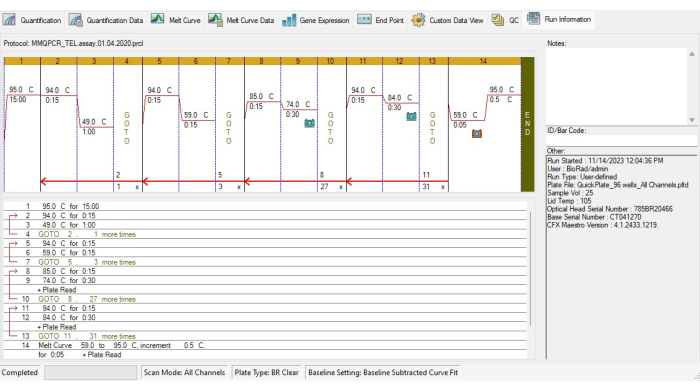
Abbildung 4: Thermocycling-Profil des MMqPCR-Assays. Das MMqPCR-Protokoll, das in der Software in Übereinstimmung mit dem ursprünglichen Thermocycling-Protokoll19 erstellt wurde. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
6. MMqPCR-Datenanalyse
- Wenn der Thermocycling abgeschlossen ist, analysieren Sie die Daten auf folgende Weise, um TL-Werte für die verarbeiteten Proben zu erzeugen.
- Wählen Sie in der Software die Funktion Plate Setup aus. Klicken Sie im Dropdown-Menü auf Platte anzeigen/bearbeiten.
- Markieren Sie alle Vertiefungen und klicken Sie auf Fluorophore auswählen, aktivieren Sie dann das Kontrollkästchen mit der Bezeichnung SYBR und deaktivieren Sie alle anderen Kontrollkästchen. Klicken Sie auf OK.
- Während alle Wells noch hervorgehoben sind, aktivieren Sie neben dem Wort Load das Kontrollkästchen neben SYBR. Jetzt sollten alle Bohrlöcher mit SYBR beschriftet sein.
- Markieren Sie als Nächstes die drei NTC-Wells oben oder unten in der seriellen Standardverdünnung und wählen Sie NTC aus dem Menü für den Probentyp auf der rechten Seite. Die drei Vertiefungen sollten nun gelb sein und NTC heißen. Abbildung 5A zeigt, welche Wells für P1 ausgewählt werden sollten, und Abbildung 5B zeigt, welche Wells für P2 ausgewählt werden sollten.
HINWEIS: P2 wird von P1 invertiert, so dass sich der NTC am unteren Rand der Spalten 4-6 für P1 und am oberen Rand der Spalten 7-9 für P2 befindet. - Markieren Sie die 21 Vertiefungen der seriellen Standardverdünnung und wählen Sie Standard aus dem Menü "Probentyp". Diese Brunnen sollten grün sein. Während diese Wells noch hervorgehoben sind, klicken Sie auf Technisches Replizieren. Wählen Sie im Menü "Größe replizieren" die Option " 3" und dann "Horizontal" > "Anwenden" aus. Diese Vertiefungen sollten in Dreiergruppen von Std-1 bis Std-7 beschriftet werden.
- Während der Standard noch markiert ist, scrollen Sie nach unten, und wählen Sie Verdünnungsreihe aus. Geben Sie im Feld Verdünnungsfaktor den Wert 2 ein und geben Sie dann die Anfangskonzentration der Verdünnung und die Richtungsabhängigkeit entsprechend der Plattennummer ein, wie in den folgenden Schritten beschrieben.
- Geben Sie für die P1-Platte 2.00E-03 in das Feld für die Anfangskonzentration ein, aktivieren Sie das Kontrollkästchen für Absteigend und wählen Sie dann Anwenden aus. Die Werte in den 21 Vertiefungen sollten mit den Konzentrationswerten im Bereich von 2,00E-03 bis 3,13E-05 von oben nach unten gekennzeichnet sein.
- Geben Sie für die P2-Platte 3.13E-5 in das Feld für die Anfangskonzentration ein, aktivieren Sie das Kontrollkästchen für Erhöhen , und wählen Sie dann Anwenden aus. Die Werte in den 21 Vertiefungen sollten die Konzentrationswerte im Bereich von 3,13E-05 bis 2,00E-03 von oben nach unten enthalten.
- Markieren Sie für die Proben die Spalten für PCR-Streifen A (1-3 für P1 und P2), wählen Sie Unbekannt aus dem Menü für den Probentyp und dann Technisches Replizieren aus. Wählen Sie im Menü "Größe replizieren" die Option "3" und dann "Horizontal" > "Anwenden" aus. Die Vertiefungen für diese Spalten sollten blau sein und in 3er-Sätzen von Unk-1 bis Unk-8 zeilenweise beschriftet sein.
- Wiederholen Sie Schritt 6.3 für die Spalten für PCR-Streifen B (Spalten 7-9 für P1 und 4-6 für P2). Sie sollten mit Unk-9 bis Unk-16 beschriftet sein.
- Wiederholen Sie Schritt 6.3 für die Spalten für den PCR-Streifen C (Spalten 10-12 für P1 und P2). Sie sollten mit Unk-17 bis Unk-24 beschriftet sein. Wenn die Schritte 6.2 bis 6.3.2 abgeschlossen sind, sollten die Fenster für die Platteneinrichtung in Abbildung 5A, B aussehen.
- Wählen Sie unten rechts im Fenster des Platteneditors die Option OK aus. Klicken Sie auf Ja , um die Änderungen zu übernehmen, wenn Sie dazu aufgefordert werden.
- Für die Qualitätskontrolle auf Assay-Ebene ist sicherzustellen, dass die Kurven auf der Registerkarte "Quantifizierung" sowohl für Schritt 9 als auch für Schritt 12 des Thermocycling-Profils, die den Telomer- bzw. Albumin-Amplikons entsprechen, geeignet sind (z. B. keine invertierten Amplifikationskurven). Der Zugriff auf Kurven erfolgt über das Dropdown-Menü in der Mitte rechts des Softwarefensters. Abbildung 6A,B zeigt entsprechende Kurven.
- Stellen Sie sicher, dass die PCR-Effizienzen, die sowohl in Schritt 9 als auch in Schritt 12 angegeben werden, zwischen 90 % und 110 % liegen und dass sich diese beiden Effizienzen nicht um mehr als 10 % voneinander unterscheiden (d. h. 92,4 % für Schritt 9 und 98 % für Schritt 12 sind angemessen, aber 92,4 % für Schritt 9 und 108 % für Schritt 12 sind nicht angemessen). Stellen Sie außerdem sicher, dass dieStandard-R2-Kurve >0,995 beträgt. Sollte entweder P1 oder P2 diese Kriterien nicht erfüllen, müssen die Platten erneut durchlaufen werden.
- Wählen Sie für P1 Schritt 9 aus, und markieren Sie die 21 Vertiefungen, die der Standardkurve entsprechen, wie in Abbildung 6A, B dargestellt. Kopieren Sie die Cq-Werte in der unteren rechten Ecke des Softwarefensters. Öffnen Sie die Telomer-Datenblattvorlage, die als Zusatzdatei 1 verfügbar ist, und fügen Sie diese Werte in das Standardblatt 1, Spalte B, ein. Wählen Sie Schritt 12 aus, und kopieren Sie dann diese Cq-Werte. Fügen Sie diese Werte in das Standardblatt 1, Spalte I, ein.
- Identifizieren Sie den Neigungswert in Schritt 9, und geben Sie ihn in Zelle C4 im Standardblatt 1 ein. Identifizieren Sie den Steigungswert in Schritt 12, und geben Sie ihn in Zelle J4 im Standardblatt 1 ein.
HINWEIS: Das System berechnet den prozentualen Wirkungsgrad der Grundierungen anhand der Steigungen der Standardkurve, die in Schritt 6.6.1 identifiziert wurde. Der prozentuale Wirkungsgrad kann manuell mit der folgenden Formel berechnet werden: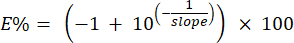
- Es ist sicherzustellen, dass der Variationskoeffizient (CV) für jedes Standardverdünnungsdreifachverfahren kleiner als 0,1 ist. Wenn größer oder gleich 0,1, sind insgesamt bis zu drei einzelne Vertiefungen von allen Konzentrationen von Kontrollproben auszuschließen, die in der 7-Punkt-Standard-Serienverdünnungskurve auf der Platte enthalten sind. Nur eine Vertiefung sollte aus einem Dreifachsatz ausgeschlossen werden.
HINWEIS: Wenn Sie Standardpunkte aus der Analyse in der Software ausschließen, ändern sich die gemeldete Steigung, R, der Y-Achsenabschnitt und die Effizienz für beide Primer. Nachdem ein Standard-Well aus der Analyse entfernt wurde, müssen die Schritte 6.5.1 bis 6.6.1 wiederholt werden. - Wiederholen Sie die Schritte 6.6.1 - 6.6.2 für die P2-Analysedatei und das entsprechende Standard-2-Blatt.
- Erst wenn sowohl die Blätter Standard 1 als auch Standard 2 mit akzeptablen Lebensläufen ausgefüllt sind, entnehmen Sie die Daten der einzelnen Proben aus den Analysedateien. Stellen Sie sicher, dass QC-Anpassungen in der Spalte L des P1- vs. P2-Blatts gemeldet werden, z. B. wie viele Wells in der P1-Analysedatei ausgeschlossen wurden.
- Identifizieren Sie den Neigungswert in Schritt 9, und geben Sie ihn in Zelle C4 im Standardblatt 1 ein. Identifizieren Sie den Steigungswert in Schritt 12, und geben Sie ihn in Zelle J4 im Standardblatt 1 ein.
- Stellen Sie sicher, dass nur die 72 Probenvertiefungen in der P1-Analysedatei auf der Registerkarte "Quantifizierung" blau hervorgehoben sind, wie in Abbildung 7A,B. Auswählen Schritt 9. Kopieren Sie die Cq- und SQ-Werte in der unteren rechten Ecke des Softwarefensters. Fügen Sie diese Werte in das Blatt P1 der Beispiele ein, beginnend in Zelle D3. Auswählen Schritt 12 Kopieren Sie dann diese Cq- und SQ-Werte, und fügen Sie sie in das Beispielblatt P1 ein, beginnend in Zelle F3.
- Wiederholen Sie diesen Schritt für die Proben in der P2-Analysedatei und dem entsprechenden P2-Blatt für Proben. Das Verhältnis von Telomern zu Einzelkopien (T/S) (Spalte H) wird automatisch berechnet, kann aber auch manuell mit der folgenden Formel berechnet werden:
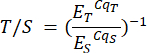
Dabei ist ET/S die Effizienz der exponentiellen Amplifikation für Reaktionen, die auf das Telomer- bzw. das Einzelkopie-Gen abzielen, und CqT/S ist der Zyklus, bei dem ein gegebenes Replikat, das auf den Telomergehalt abzielt, oder das Einzelkopie-Gen die kritische Schwelle der Fluoreszenzquantifizierung erreicht. Die durchschnittlichen T/S-Verhältnisse (Spalte I) werden anhand von sechs Messungen pro Probe berechnet.
HINWEIS: Die Proben-ID-Nummern in CFX entsprechen nicht derselben biologischen Probe auf den beiden Platten. Die Telomer-Datenblattvorlage berücksichtigt diesen Unterschied und gleicht die entsprechenden doppelten Triplikate aus. - Überprüfen Sie in CFX Maestro, ob die Cq-Werte für alle analytischen Proben zwischen dem niedrigsten und dem höchsten Cq-Wert der Standardkurve liegen, wie in Abbildung 7B zu sehen ist. Liegt eine Probe außerhalb des Bereichs, wie in Abbildung 7A gezeigt, muss sie nach der Konzentrationsanpassung erneut durchgeführt werden. Eine Probe mit einem Cq-Wert, der niedriger als der höchste Konzentrationsstandard ist, muss um einen Faktor verdünnt werden, der ungefähr der Anzahl der Amplifikationszyklen entspricht, die außerhalb des Bereichs liegt, und eine Probe mit einem Cq-Wert, der höher als der niedrigste Konzentrationsstandard ist, müsste um einen Faktor konzentriert werden, der ungefähr der Anzahl der Amplifikationszyklen jenseits des Schwellenwerts entspricht.
HINWEIS: Große Schwankungen in der Konzentration von Analyseproben führen zu einer zusätzlichen potenziellen Abweichung und sollten mit Vorsicht durchgeführt werden. - Stellen Sie sicher, dass die Anfangskonzentration im NTC weniger als 5 % der durchschnittlichen DNA-Menge in den Probenvertiefungen betrug. Dies kann überprüft werden, indem man sich die prozentuale Wasserkontamination auf den Proben P1 und P2 der Proben P2 der MMqPCR ansieht. Wenn der NTC eine Kontamination anzeigt, führen Sie die Platten erneut aus.
HINWEIS: Aufgrund der Empfindlichkeit des MMqPCR-Assays kann eine geringfügige, unkontrollierbare Kontamination gelegentlich dazu führen, dass sich die NTC-Wells amplifizieren. Diese Amplifikation sollte jedoch gering sein und mehrere Zyklen nach der Einzelkopie-Genamplifikation in Schritt 12 des Thermocycling-Protokolls auftreten. - Für die Qualitätskontrolle auf Probenebene sind die Standardabweichungen (SD) (Spalte J) und CVs (Spalte K) in den Proben P1 und P2 der Proben zu überprüfen. Stellen Sie sicher, dass die Intraplatten-CVs über die dreifachen T/S-Verhältnisse der Probe weniger als 0,10 (10 %) betragen. Zur Korrektur von Intraplatten-CVs von mehr als 0,1 kann ein T/S-Verhältnis aus einem Satz von Triplikaten ausgeschlossen werden.
HINWEIS: Es kann nur ein Gesamtreplikat über die sechs Messungen pro Probe ausgeschlossen werden, d. h. ein Replikat für dieselbe Probe kann nicht sowohl auf P1 als auch auf P2 ausgeschlossen werden. - Vergewissern Sie sich, dass die CVs der einzelnen Proben zwischen den Platten im P1 v P2-Blatt, Spalte G, kleiner als 0,05 (5 %) sind. Um CVs über 0,05 zu korrigieren, schließen Sie ein T/S-Verhältnis aus den sechs Messungen pro Probe auf beiden Platten aus. Schließen Sie Proben nach der visuellen Inspektion über Intraplatten-Tripplikate und bei Bedarf alle sechs Messungen aus.
HINWEIS: Nur Proben, die diese QC-Kriterien erfüllen, Intraplatten-CV < 10 % und Interplatten-CV < 5 %, dürfen in die endgültigen TL-Ergebnisse einbezogen werden. Andernfalls sollte die Probe aus den P1-, V-, P2- und ICC-Datenblättern für diesen Lauf ausgeschlossen werden, und die Probe sollte in einem zweiten MMqPCR-Assay erneut bewertet werden. - Bei der Qualitätskontrolle auf Plattenebene ist zu überprüfen, ob die durchschnittliche Abweichung innerhalb der Platte über alle Proben auf jeder Platte, die sich am unteren Rand der P1- und P2-Probenblätter befindet, weniger als 0,05 (5 %) beträgt. Für zusätzliche QK auf Plattenebene überprüfen Sie, ob die Gesamtstreuung zwischen den Platten im P1 vs. P2-Blech, Zelle G29, weniger als 0,06 (6 %) beträgt. Stellen Sie sicher, dass Proben, die die Qualitätskontrolle nicht bestanden haben, aus der Berechnung der Interplatten-CV in Zelle G30 entfernt werden.
HINWEIS: Wenn Sie mit verwandten Probengruppen (z. B. Familienmitglieder, unterschiedliche Zeitpunkte usw.) arbeiten, müssen alle Proben in einer Gruppe auf derselben Platte ausgeführt werden. Wenn eine Stichprobe der Gruppe die QC-Kriterien nicht erfüllt, müssen daher alle Stichproben in der Gruppe erneut ausgeführt werden. - Notieren Sie in einer separaten Gesamtkohorten- oder Experimentdatendatei die endgültigen TL-Daten für jede Probe, die die Qualitätskontrolle bestanden hat (P1 v P2 Blatt, Spalte I), ICC-Berechnungen für jede Probe, die die Qualitätskontrolle bestanden hat (ICC-Datenblatt, Spalten E-K) und die QC-Daten des endgültigen Assay-Laufs aus P1 v P2 (Spalten J-L)
- Wiederholen Sie diesen Schritt für die Proben in der P2-Analysedatei und dem entsprechenden P2-Blatt für Proben. Das Verhältnis von Telomern zu Einzelkopien (T/S) (Spalte H) wird automatisch berechnet, kann aber auch manuell mit der folgenden Formel berechnet werden:
- Verwenden Sie die vom Telomere Research Network (TRN) erstellte Zusatzdatei 2 , um die ICC für das Projekt20 zu berechnen.
- Um die Vergleichbarkeit zwischen TL-Studien zu verbessern, transformieren Sie schließlich die endgültigen TL-Daten für jede Stichprobe in der separaten Gesamtkohorte oder Experimentdatendatei in Z-Scores unter Verwendung der folgenden Gleichung, bei der das mittlere T/S-Verhältnis für alle Stichproben in der Kohorte vom T/S-Verhältnis der einzelnen Stichproben subtrahiert und dann durch die SD für alle Stichproben in der Kohorte dividiert wird.
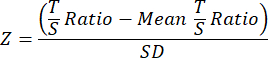
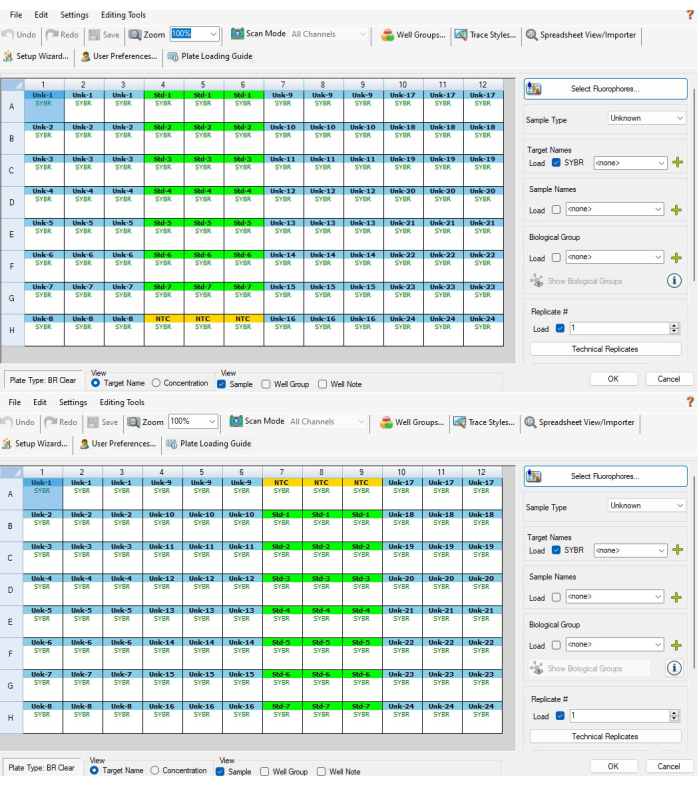
Abbildung 5: Softwarebasierte Platteneinrichtung. (A) Softwarebasierte Platteneinrichtung für eine P1-Platte nach Abschluss der Schritte 6.2-6.4. (B) Softwarebasierte Platteneinrichtung für eine P2-Platte nach Abschluss der Schritte 6.2-6.4. Proben- und Standard-IDs sind zwischen den beiden CFX-Platten nicht ausgerichtet, da die Software die Proben-IDs basierend auf der Well-Position zuweist (d. h. CFX-Probe 1 auf P1 ist CFX-Probe 24 auf P2). Die Excel-Vorlage, die in der ergänzenden Datei 1 bereitgestellt wird, berücksichtigt dies und stellt sicher, dass die Messungen zwischen den Platten, die an derselben biologischen Probe durchgeführt werden, korrekt aufeinander ausgerichtet sind. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
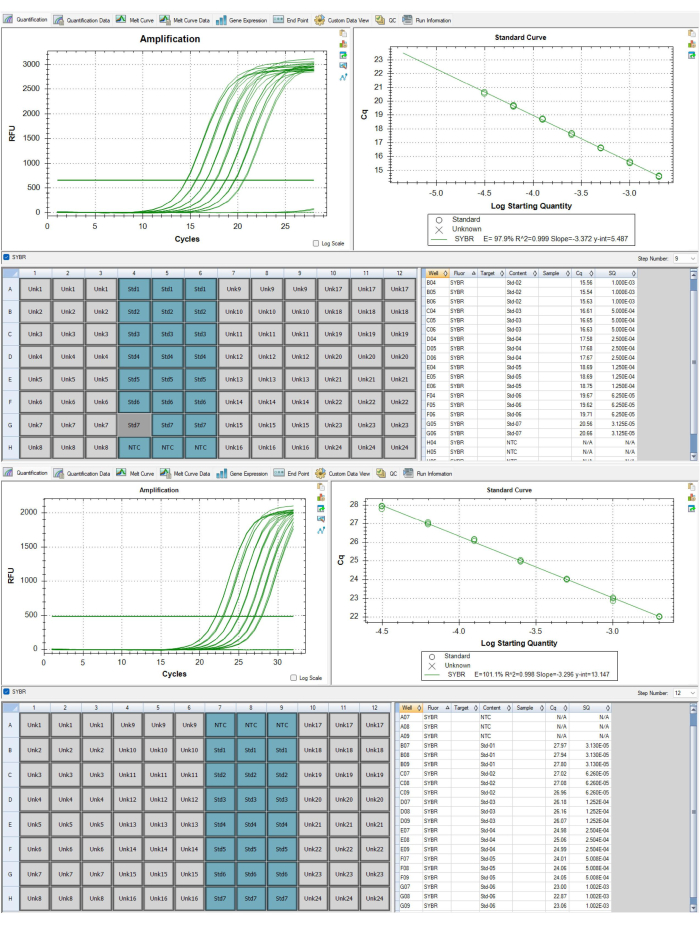
Abbildung 6: Standardkurven fürT-Elomer- und Albumin-Amplikone. (A) Diese Standardkurve stammt aus dem P1-Telomer-Amplikon des repräsentativen Ergebnisdatensatzes. Ein Standard wurde entfernt, weil er die QC-Kriterien nicht erfüllte. (B) Diese Standardkurve stammt aus dem P2-Albumin-Amplikon des repräsentativen Ergebnisdatensatzes. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
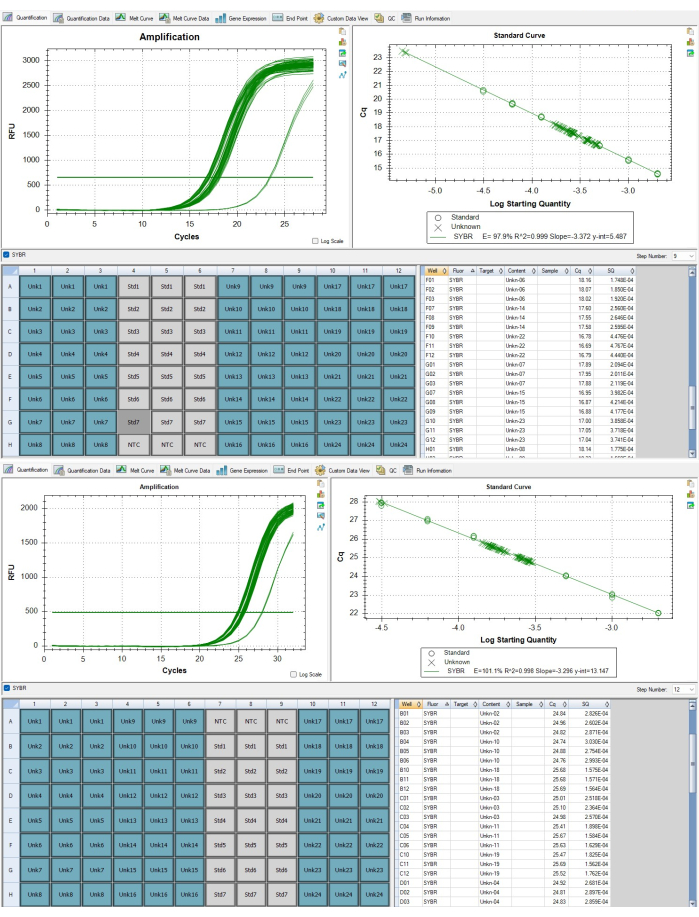
Abbildung 7: Telomer- und Einzelkopie-Genamplikone. (A) Telomeramplifikation und (B) Albumin-Genamplifikation von Proben, die in repräsentativen Ergebnissen in der Software angezeigt werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
7. MMqPCR-Datenübermittlung
- Wenn Sie die Forschungsergebnisse unter Verwendung der TL-Daten melden, die aus der MMqPCR erstellt wurden, stellen Sie sicher, dass die in den TRN-Mindestmelderichtlinien beschriebenen Punkte gemeldet werden. Diese Leitlinien sind auf der TRN-Website (https://trn.tulane.edu/resources/reporting-guidelines/) zu finden, und eine Vorlage für die Meldung dieser Informationen finden Sie in der Zusatzdatei 3. Zitieren Sie dieses Protokoll und andere TRN-Richtlinien und -Ressourcen, wo es angebracht ist.
Ergebnisse
Die in Tabelle 4 und Tabelle 5 dargestellten Ergebnisse sind ein Beispiel für hochgradig wiederholbare TL-Messungen, die durch Befolgen des Protokolls erzielt wurden. Für diese Ergebnisse wurde die DNA aus 24 Proben von mononukleären Blutzellen (PBMC) mit einem kommerziellen Kit gemäß den Herstellerrichtlinien extrahiert. Diese 24 Proben wurden über zwei 96-Well-Platten geleitet. Alle DNA-Proben wurden mit einem Spektralphotometer und einem Fluorometer auf ihre Qualität geprüft, wobei das durchschnittliche Verhältnis von 260/280, das durchschnittliche Verhältnis von 260/230 und die dsDNA-Konzentration verwendet wurden, um die Eignung des Assays und den Verdünnungsfaktor der Probe zu bestimmen (Tabelle 6). Tabelle 6 unterstreicht auch die Bedeutung der Quantifizierung der dsDNA-Konzentration, die sich von der mit einem Spektralphotometer gemessenen DNA-Konzentration unterscheiden kann. Diese Variabilität ist das Ergebnis unterschiedlicher Ansätze zur Quantifizierung. Insbesondere leitet das Spektralphotometer die DNA-Konzentration basierend auf der Absorption bei 280 nm ab und ist anfällig für Schwankungen aufgrund von Verunreinigungen (z. B. Protein, Salz usw.), die sich auf die Absorptionswerte auswirken. Im Gegensatz dazu werden dsDNA-Konzentrationen, die mit einem Fluorometer gemessen werden, durch die Fluoreszenz eines Farbstoffs bestimmt, der spezifisch an dsDNA bindet, und es wird davon ausgegangen, dass sie den DNA-Gehalt genauer widerspiegeln. Die DNA-Proben erlebten vor der MMqPCR TL-Analyse bis zu drei Gefrier-Auftau-Zyklen. Die Kontroll-DNA wurde aus gepoolten DNA-Extraktionen von PBMCs von einer Person hergestellt, die verwendet wurde, um eine siebenstufige serielle Verdünnung von 2 ng/μl auf 0,0313 ng/μl DNA zu erzeugen. Die unabhängigen Standardkurven, die für PCR-Schritt 9 (Telomer-Amplicon) und Schritt 12 (Einzelkopie-Gen Amplicon, Albumin in diesem Protokoll) erstellt wurden, sind in Abbildung 6A, B dargestellt. Die Assays wurden auf einem kommerziellen Real-Time-PCR-Nachweissystem durchgeführt, das eine Schmelzkurve erzeugte, die zeigt, wie die einzelnen Amplikonprodukte bei unterschiedlichen Temperaturen hergestellt werden, wie in Abbildung 8 zu sehen ist.
Tabelle 4: Ausgabedaten aus der MMqPCR-Messung für optimale repräsentative Ergebnisse. Mittlere T/S-Verhältnisse, SDs, CVs und Z-bewertete TL für 24 Stichproben. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 5: Ausgabe von Daten zur Telomerlänge. Die Rohdaten des MMqPCR-Assays werden in der Software analysiert und dann zur weiteren Analyse und Qualitätskontrolle in die Tabellenkalkulationsvorlage eingefügt, die als ergänzende Datei 1 angehängt ist. Dieses Ausgabeblatt für TL-Vorlagen enthält eine Zusammenfassung der Daten mit Proben-IDs, durchschnittlichen TLs, SDs und CVs für jede Probe auf beiden Platten. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 6: Spektralphotometer und Fluorometer DNA-Qualitätsmetriken für Probenergebnisse. QC-Daten aus der Duplikat-Spektralphotometer-Analyse und der singulären Fluorometermessung von dsDNA und etwaigen Kontaminaten pro Probe. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
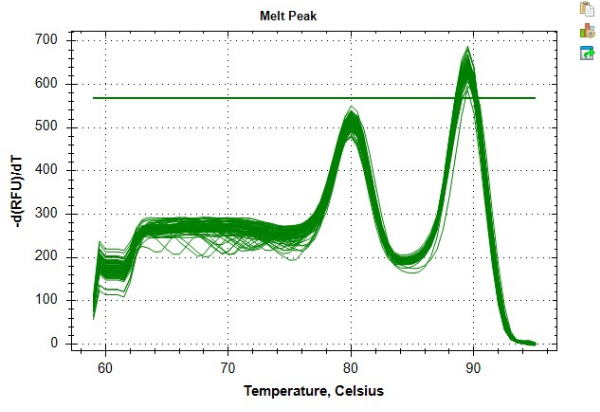
Abbildung 8: Beispiel für eine Schmelzkurve. Schmelzekurve für Probendaten, wie sie in der Software generiert werden. Der frühere Peak ~80 °C repräsentiert das Telomer-Amplikon und der sekundäre Peak bei ~89 °C repräsentiert das Albumin-Amplicon. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Bei diesem Projekt betrug der durchschnittliche Telomerwirkungsgrad 98,6 % mit einem Bereich von 90,7 bis 102,1 und der durchschnittliche Albuminwirkungsgrad 102,3 % mit einem Bereich von 93,6 bis 108,2 über alle Durchläufe. Es wurden durchschnittlich 0,97 Replikate aus den Standardkurven entfernt, was den QC-Kriterien dieses Protokolls entspricht. Die durchschnittliche Interplatten-CV betrug 1,83 % mit einer SD von 0,00616 und die durchschnittliche Intraplatten-CV 3,78 % mit einer SD von 0,00658. Die durchschnittlichen TL für diese repräsentativen Stichproben sind in Tabelle 4 und Tabelle 5 dargestellt. Der mittlere TL betrug 1,37 mit einem SD von 0,24 und einer Spanne von 0,84 bis 2,32. Das T/S-Verhältnis, SD, CV und Z-bewertete TL pro Probe sind in Tabelle 4 aufgeführt. Da es sich bei der Ausgabe des T/S-Verhältnisses des MMqPCR-Assays um ein relatives Maß für TL handelt, wurden die Verhältnisse in diesen Z-Score umgewandelt, um einen studienübergreifenden Vergleich zu ermöglichen. Die ICC für dieses Projekt wurde unter Verwendung des R-Skripts berechnet, wie es in den TRN-Richtlinien in der Ergänzungsdatei 2 beschrieben ist, unter Berücksichtigung von Batch- und Run-Effekten20. Um die projektinterne ICC zu berechnen, haben wir 10 % der durchlaufenden Proben erneut durchgeführt, wobei wir darauf geachtet haben, dass die ICC-Platten mit mindestens einer Probe aus jeder Platte der Kohorte gefüllt wurden. Die Gesamtprojekt-ICC von 0,801 [KI: 0,703; 0,86] deutet auf die hohe Reproduzierbarkeit der TL-Ergebnisse hin.
Nicht alle Ergebnisse werden optimal sein. Die Ergebnisse in Abbildung 9 und Tabelle 7 zeigen suboptimale Ergebnisse des MMqPCR-Assays. Abbildung 9 zeigt eine Standardkurve mit einem Telomerwirkungsgrad von unter 90 %, was unter den QC-Standards liegt und eine Wiederholung der gesamten Platte erfordert. Probleme mit der Primereffizienz sind in der Regel auf ein Problem mit Reagenzien zurückzuführen, daher ist es wichtig, die Daten der Aliquotierung der Reagenzien und deren Ablauf als ersten Schritt zu verfolgen, um festzustellen, welches Reagenz für die geringe Effizienz verantwortlich ist. Abgelaufene Reagenzien sollten ausgetauscht werden, bevor die Platte erneut ausgeführt wird. Tabelle 7 zeigt eine Platte, die die anfänglichen QK-Kriterien auf Probenebene erfüllte, aber ein hohes Maß an Variabilität zwischen den Platten aufweist, was zu einem Versagen der QK auf Plattenebene führte. Schwankungen zwischen den Platten sind in der Regel auf Fehler beim Pipettieren und beim Befüllen der Platten zurückzuführen. In diesem Fall sollte der Techniker alle Probleme bewerten, die beim Einrichten der Platte aufgetreten sind, und sicherstellen, dass die Pipetten kalibriert sind.
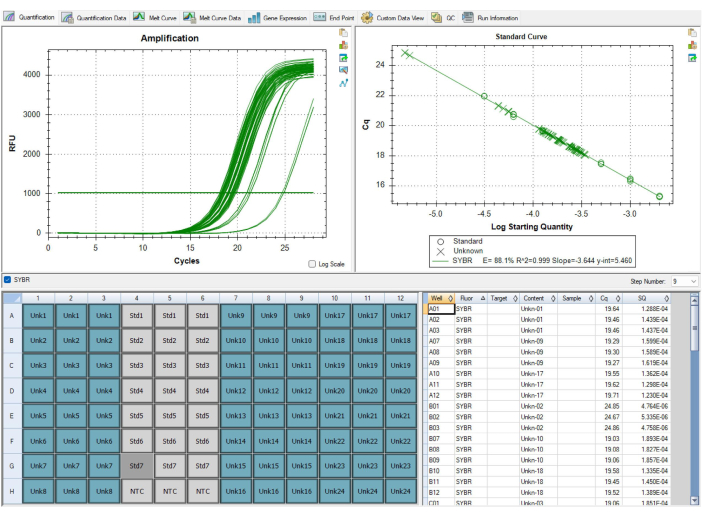
Abbildung 9: Suboptimale Ergebnisse. Diese in der Software angezeigte Standardkurve bestand die Qualitätskontrolle nicht, da die Effizienz der Amplifikation für den Telomer-Primer weniger als 90 % betrug. Das Bild stammt von P1, aber P2 hatte ähnlich niedrige Wirkungsgrade. Die Daten aus diesem Lauf konnten nicht verwendet werden, und alle Proben mussten nach dem Austausch des abgelaufenen ursächlichen Reagenzes erneut durchgeführt werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 7: Suboptimale Ergebnisse. In der Tabelle wird die Telomer-Datenvorlage für einen Lauf angezeigt, bei dem viele der Stichproben die QC-Standards nicht erfüllten. Samples, die erneut ausgeführt werden mussten, wurden auf der Grundlage von CV-Werten bestimmt, dann wurde der Samplename zur leichteren Identifizierung in eine rote Schrift geändert. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Datei 1: Vorlage für eine Telomer-Datentabelle. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 2: ICC-Berechnung. Dieses Protokoll wurde vom Telomere Research Network (TRN) entwickelt. Diese Datei wurde von20 geändert. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 3: TRN-Melderichtlinien. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Die vor 2002 in größeren populationsbasierten Studien am häufigsten verwendete Methode zur TL-Messung war die Southern-Blot-Analyse der terminalen Restriktionsfragmentlängen (TRF)24,25. Obwohl TRF in spezialisierten Labors eine hervorragende Präzision und Reproduzierbarkeit bietet, ist seine Anwendbarkeit aufgrund der Menge und Qualität der erforderlichen DNA und des begrenzten Durchsatzes begrenzt, was den Hintergrund für die zunehmende Verwendung von qPCR-basierten TL-Assays und anschließend des MMqPCR-Assays bildet. Die MMqPCR TL-Methode bietet eine wiederholbare TL-Messung, wenn sie unter sorgfältiger Beachtung der einzelnen QC-Kriterien eingerichtet, optimiert und gewartet wird. Die Berechnung und Berichterstattung des spezifischen ICC für jede analysierte Kohorte ist erforderlich, um die Zuverlässigkeit des Assays zu gewährleisten. Die MMqPCR-Methode unterliegt zwar der DNA-Qualität und dem technischen Fachwissen, eignet sich aber gut für große populationsbasierte Studien, in denen TL untersucht wird, da sie kleine DNA-Mengen erfordert, zuverlässiger ist als die Singleplex-PCR und in Bezug auf Reagenzienkosten und Technikerzeit effizienter ist als andere Methoden. Die Fähigkeit, hohe ICCs zu generieren, liefert zusätzliche Daten zur Unterstützung der Verwendung von MMqPCR für große populationsbasierte Studien mit TL. Die MMqPCR-TL-Messung kann auf eine breite Palette von Studien angewendet werden, die versuchen, die Rolle von TL als Biomarker für Gesamtmortalität, Alterung, lebenslangen Stress, Umwelteinflüsse und körperliche Gesundheitsfolgen wie Herz-Kreislauf-Erkrankungen und Krebszu definieren 4,7,8,10,11,12,13,26,27,28, 29,30,31.
Eine Einschränkung der MMqPCR-Methode besteht darin, dass sie TL als T/S-Verhältnis angibt, eine relative Schätzung der Länge, die je nach Auswahl des Einzelkopiegens, der Mastermix-Zusammensetzung und den PCR-Cycling-Parametern variiert32. Das T/S-Verhältnis ist einheitenlos. Ohne Kombination mit anderen TL-Messmethoden ist diese Methode daher nicht in der Lage, Schätzungen in den Basispaarwerten 18,33,34 zu berichten. Infolgedessen muss das T/S-Verhältnis in einen Z-Score umgewandelt werden, um in allen Studien relevant zu sein35. Dabei ist erhebliche Vorsicht geboten, wenn dies über Labore, Methoden und Assays hinweg geschieht. Darüber hinaus kann dieses Verfahren, wie bei gelbasierten Hybridisierungsassays, die Quantifizierung interstitieller Telomere umfassen. Diese Sequenzen machen jedoch nur einen sehr kleinen Teil des gesamten Telomer-DNA-Gehalts pro Genom aus. Darüber hinaus ist es wahrscheinlicher, dass interstitielle Telomersequenzen nicht übereinstimmende Basenpaarsequenzen enthalten, die von kanonischen Telomerwiederholungen abweichen, was die Wahrscheinlichkeit einer Primerbindung und Amplifikation verringert. Darüber hinaus ist die minimale Menge an DNA, die für den MMqPCR-Assay benötigt wird, zwar von Vorteil, aber es ist wichtig zu beachten, dass qPCR-basierte Messungen von TL von präanalytischen Faktoren beeinflusst werden, die sich auf die DNA-Qualität und -Integrität auswirken, einschließlich der Lagerbedingungen der Proben, der DNA-Extraktionsmethode und des biologischen Gewebes36,37. Es wurde gezeigt, dass die analytische Kontrolle dieser Faktoren die externe Validität von TL-Messungen verbessern kann, die mit qPCR38 erzeugt wurden. Dennoch müssen die Auswirkungen von Unterschieden in der DNA-Qualität auf die TL, die durch den MMqPCR-Assay erzeugt werden, systematisch bewertet werden, da es derzeit keine datengestützten Leitlinien gibt, um festzustellen, ob eine Probe von ausreichender Qualität ist, um mit diesem Ansatz eine genaue Schätzung der TL zu erstellen. Trotz dieser Einschränkungen sind die Anwendungen dieses Assays für Studien zu gesundheitlichen Ergebnissen auf Bevölkerungsebene beträchtlich.
Bei der Verwendung des MMqPCR-Assays ist eine kontinuierliche Bewertung der Präzision und des Durchsatzes erforderlich. In der derzeitigen Form führen Techniker mit zwei Thermocyclern gleichzeitig Dreifachproben auf doppelten Platten durch. In Ermangelung mehrerer Thermocycler empfehlen wir, doppelte dreifache Messungen beizubehalten und Platten sequentiell laufen zu lassen, um die Präzision zu erhöhen, auch auf Kosten eines verringerten Durchsatzes. Jede Entscheidung, dem Durchsatz Vorrang vor der Präzision zu geben, z. B. durch die Verwendung einzelner dreifacher Messungen, sollte von einer gründlichen Prüfung und Bewertung der resultierenden ICCs begleitet werden, bevor mit der Analyse der analytischen Proben fortgefahren wird. Bei Entscheidungen über Durchsatz und Präzision muss man die Probengröße und die Qualität der Proben-DNA berücksichtigen. Eine kleinere Probengröße oder DNA-Proben von schlechter Qualität erfordern eine höhere Präzision23. Dies ist von umso größerer Bedeutung, wenn mit Proben aus verwandten Gruppen (z. B. Familienmitgliedern, Probanden mit mehreren Zeitpunkten) gearbeitet wird. In diesen Fällen ist eine sorgfältige Planung, z. B. die Zuweisung verwandter Proben zur gleichen Platte vor Beginn des Experiments, eine Möglichkeit, den Verlust der statistischen Aussagekraft durch versehentliche Gruppierung durch Plattenverwechslung zu verhindern.
Für diesen Assay wurde ein Spektralphotometer verwendet, um die Qualität der DNA-Proben zu beurteilen: Proben im Bereich von 1,6-2,0 für 260/280-Verhältnisse und 2,0-2,2 für 260/230-Verhältnisse wurden als akzeptabel angesehen. Diese Qualitätsbewertung und die genaue Beurteilung der doppelsträngigen DNA mittels Fluorometer sind entscheidende Schritte in diesem Protokoll, um wiederholbare TL-Daten zu erhalten. Andere, beschreibendere Messungen der DNA-Integrität, wie z. B. die Fragmentgröße und/oder zusammenfassende Messungen der DNA-Qualität, die über Agarosegel bestimmt werden (z. B. die DNA-Integritätszahl), können ebenfalls zur Bestimmung der Probenqualitätverwendet werden 38. Wir empfehlen außerdem, dass die Verdünnung der Proben nur zum Zeitpunkt der Probenvorbereitung für die Durchführung des MMqPCR-Assays erfolgt. Dadurch wird sichergestellt, dass DNA-Aliquote nach der Extraktion so wenig wie möglich manipuliert werden, wodurch die Variabilität bei der Handhabung vor dem Assay verringert wird. Sollten DNA-Proben transportiert werden müssen, sollten sie auf Trockeneis in der höchstmöglichen Konzentration transportiert werden, um den Abbau zu mildern, der bei DNA-Proben bei niedrigeren Konzentrationen auftritt39,40. Aufgrund des DNA-Abbaus, der bei Frost-Tau-Zyklen auftritt, sollte die Anzahl der Gefrier-Tau-Zyklen zur Lager-DNA minimiert werden41. Aliquots der gepoolten Kontroll-DNA sollten vor der Durchführung der Kohorte erstellt werden, und Aliquots einzelner analytischer DNA-Proben sollten vor der Durchführung des Assays erstellt werden.
Zu den wichtigsten Kennzahlen der Assay-Leistung und der Qualitätskontrolle gehören das NTC-Signal, Interplatten- und Intraplatten-CVs sowie die Standardkurve R2. Die Nichteinhaltung der QC-Kriterien kann auf verschiedene Weise gemildert werden. Durch den regelmäßigen Wechsel der PCR-StämmederH2O-Sorte und die Aliquotierung der PCR-Grade H2O-Unterbestände für jedes Plattenpaar werden die Kontaminationsquellen sowie die NTC-Amplifikation minimiert. Zu den zusätzlichen Schritten zur Verringerung der Kontamination gehören die folgenden: Benennung einer spezifischen PCR-Haube, die nur für MMqPCR-Assays bestimmt ist; Abwischen der PCR-Haube und des Geräts mit einer DNA-Dekontaminationslösung; Bestrahlung des Raumes mit ultraviolettem Licht; und das Üben steriler Techniken in der PCR-Haube. Um die Wiederholbarkeit des Assays zu verbessern und CVs zu verringern, wird empfohlen, Proben, Verdünnungen und PCR-Streifen in ihren jeweiligen Vortex-Schritten kräftig zu wirbeln und beim Pipettieren von DNA-Proben gründlich zu resuspendieren. Eine Unterhalb des Schwellenwerts derStandard-R2-Kurve (<0,995) ist höchstwahrscheinlich auf Pipettierfehler beim Laden der Platte zurückzuführen. Um dies zu vermeiden, achten Sie sorgfältig auf präzises Pipettieren und kalibrieren Sie die Pipetten jährlich, mischen Sie Standard-PCR-Streifen vor dem Laden kräftig und organisieren Sie die Vorräte sorgfältig, um einen effizienten Arbeitsablauf zu fördern. Wenn bei der Verwendung von zwei Maschinen und einer Platte beobachtet wird, dass sie konstant höhere CVs ausgibt, sollte die Maschine gewartet werden, um das Problem zu beheben. QC-Platten des Herstellers sollten regelmäßig auf die Platten aufgebracht werden, um die Leistung des Thermocyclers zu beurteilen.
Wenn die Probleme auch nach Anwendung der oben empfohlenen Schritte weiterhin bestehen, können die folgenden Schritte zur Fehlerbehebung des Protokolls verwendet werden. Das Führen von Aufzeichnungen darüber, wann alle Reagenzien aliquotiert wurden und welche Verfallsdaten relevant sind, kann dazu beitragen, den Fehlerbehebungsprozess zu rationalisieren, wenn unweigerlich Schwierigkeiten auftreten. Ein wichtiger Teil jeder Fehlerbehebung besteht darin, jeweils nur ein Reagenz anzupassen, um die spezifische Ursache dafür zu ermitteln, dass die Platten die QC-Kriterien nicht erfüllen, beginnend mit dem fraglichsten Reagenz. Wenn beispielsweise sowohl das Telomer- als auch das Einzelkopien-Gen eine geringe Effizienz aufweisen, sind gemeinsame Reagenzien wie das DTT, dNTPs oder SYBR-Aliquot wahrscheinlicher die Ursache als amplikonspezifische Primer. Zum angegebenen Preis sollten neue Aliquots in der Reihenfolge von DTT, dann dNTPs und schließlich, wenn das Problem weiterhin besteht, neue SYBR-Aliquots getestet werden. Umgekehrt, wenn nur eines der Amplikons (Telomer oder Einzelkopiengen) eine geringe Effizienz aufweist, ist die Ursache der Schwierigkeit eher einer der Primer. Die Interpretation der in Abbildung 8 dargestellten Schmelzkurvenpeaks kann als Quelle für wichtige Informationen für die Fehlerbehebung dienen. Die Visualisierung der beiden Schmelzkurven-Peaks kann verwendet werden, um potenzielle Probleme mit einer bestimmten Probe zu identifizieren, da sich eine einzelne Problemprobe vom allgemeinen Trend der Peaks abhebt, die durch Standards oder verbleibende analytische Proben angezeigt werden. Die Schmelzekurve kann auch verwendet werden, um Probleme mit einem bestimmten Primer zu diagnostizieren, wenn die Peaks für ein bestimmtes Amplikon systematisch weniger scharf sind als für das andere.
Dieses Manuskript beschreibt, wie der MMqPCR-Assay zur Messung von TL mit breiter Anwendbarkeit in der Forschung im Bereich der öffentlichen Gesundheit erfolgreich eingerichtet werden kann, und stellt wichtige Empfehlungen für die Qualitätskontrolle und Fehlerbehebung vor, mit dem Ziel, die Zugänglichkeit und Zuverlässigkeit dieser effizienten und kostengünstigen Methode zu erhöhen.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Die Autoren danken dem Telomere Research Network Advisory Committee und der Finanzierung durch das National Institute on Aging / National Institute of Environmental Health Sciences (U24 AG066528 und U24 AG066528-S1), die diese Arbeit ermöglicht haben.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.5mL Tubes | USA Scientific | 1605-0099 | Seal-Rite 0.5mL Microcentrifuge Tubes, Sterile Storage Temperature and Conditions: Room temperature |
| 1.5mL Tubes | USA Scientific | 1615-5599 | Seal-Rite 1.5mL Microcentrifuge Tubes, Sterile Storage Temperature and Conditions: Room temperature |
| 100mM DTT | In House | Not Applicable | Made with stock DTT, diluted sodium acetate, and PCR Grade H2O Storage Temperature and Conditions: minus 20 °C |
| 15mL Tubes | Thermo Fisher | 14-959-53A | Corning 352196 Falcon 15mL Conical Centrifuge Tubes Storage Temperature and Conditions: Room temperature |
| 1M MgCl2 | Thermo Fisher | 50152107 | Biotang Inc 1M MgCl2 1M Magnesium Chloride Solution, Prepared in 18.2 Megohms Water and Filtered through 0.22 Micron Filter Storage Temperature and Conditions: 4 °C |
| 1x Gold Buffer | In House | Not Applicable | 10X Gold Buffer diluted with PCR Grade H2O Storage Temperature and Conditions: minus 20 °C |
| 25mM dNTPs | New England BioLabs | N0446S | Deoxynucleotide Solution Set Storage Temperature and Conditions: minus 20 °C |
| 5mL Tubes | Thermo Fisher | 3391276 | Argos Technologies Microcentrifuge Tubes – 5mL Storage Temperature and Conditions: Room temperature |
| 96 Well Plate | Bio-Rad | HSP9601 | Hard-Shell 96-Well PCR Plates, Low Profile, Thin Wall, Skirted, White / Clear Storage Temperature and Conditions: Room temperature |
| Aluminum Foil | Office Depot | 3489072 | Reynolds Wrap Stanard Aluminum Foil Roll, 12" x 75', Silver Storage Temperature and Conditions: Room temperature |
| AmpliTaq Gold Kit – Polymerase and Buffer | Thermo Fisher | 4311806 | AmpliTaq Gold DNA Polymerase with Gold Buffer and MgCl2 (MgCl2 in this kit is not used), 10X Gold Buffer, 2.5U AmpliTaq Gold Polymerase Storage Temperature and Conditions: minus 20 °C |
| Betaine | Thermo Fisher | AAJ77507AB | Betaine, 5M Solution, Molecular Biology Grade, Ultrapure, 10mL Storage Temperature and Conditions: minus 20 °C |
| Big Tube Rack | Thermo Fisher | 344817 | Fisherbrand 4-Way Tube Rack Storage Temperature and Conditions: Room temperature |
| CFX Maestro Software | Bio-Rad | 12004110 | Software for real-time PCR plate setup, data collection, statistics, and graphiing of results Storage Temperature and Conditions: Room temperature |
| CFX96 Optical Reaction Module for Real-Time PCR Systems with Starter Package | Bio-Rad | 1845096 | 96-well optical module for real-time PCR Storage Temperature and Conditions: Room temperature |
| DTT | Fisher Scientific | AAJ1539706 | Dithiothreitol, >99.5+ Molecular Biology Grade, 5 g Storage Temperature and Conditions: minus 20 °C |
| ELIMINase | Fisher Scientific | 04-355-32 | ELIMINase Laboratory Decontaminant Storage Temperature and Conditions: Room temperature |
| HEPA Filter | USA Scientific | Replacement Filters | High-Efficiency Particulate Air Filter for AirClean Workstations Storage Temperature and Conditions: Room temperature |
| Kimwipes | Thermo Fisher | 06666A | Kimberly-Clark Professional Kimtech Science Kimwipes Delicate Task Wipers, 1-Ply Storage Temperature and Conditions: Room temperature |
| Loading Trough | Thermo Fisher | 14387069 | Thermo Scientific Matrix Reagent Reservoirs Storage Temperature and Conditions: Room temperature |
| Microsoft Excel | Microsoft | Not Applicable | Microsoft 365 package, Excel software application Storage Temperature and Conditions: Room temperature |
| Mini Centrifuge | Genesee Scientific | 31-500B | Poseidon 31-500B Mini Centrifuge, Blue Lid Storage Temperature and Conditions: Room temperature |
| PCR Grade H2O | Thermo Fisher | AM9937 | Nuclease-Free Water (not DEPC-Treated) Storage Temperature and Conditions: Room temperature |
| PCR Hood | USA Scientific | 4263-2588 | Nucleic Acid Workstation with HEPA Filtration, AirClean Systems Combination PCR Workstation Storage Temperature and Conditions: Room temperature |
| PCR Strips | Thermo Fisher | AB0776 | Low Profile Tubes and Flat Caps, Strips of 8 Storage Temperature and Conditions: Room temperature |
| PCR Tube Rack | Thermo Fisher | 344820 | Fisherbrand 96-Well PCR Tube Rack Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Multichannel) | Ranin | 17005860 | Pipette Tips SR LTS 20µL F 960A/5, 20µL Maximum Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1181-3850 | 10µL Graduated TipOne RPT Filter Tips Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1180-1850 | 20µL Beveled TipOne RPT Filter Tips Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1111-0880 | 200µL Natural TipOne Pipette Tips in Racks Storage Temperature and Conditions: Room temperature |
| Pipette Tips (Single Channel) | USA Scientific | 1111-2890 | 1000µL Natural Graduated TipOne Pipette Tips in Racks Storage Temperature and Conditions: Room temperature |
| Pipettors (Multichannel) | Ranin | 17013802 | Pipet-Lite Multi Pipette L8-10XLS, 0.5 to 10µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Multichannel) | Ranin | 17013803 | Pipet-Lite Multi Pipette L8-20LS+, 2 to 20µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F144802G | Gilson Pipetman Classic Pipets, 1 to 10µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F123600 | Gilson Pipetman Classic Pipets, 2 to 20µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F123601 | Gilson Pipetman Classic Pipets, 20 to 200µL Storage Temperature and Conditions: Room temperature |
| Pipettors (Single Channel) | Thermo Fisher | F123602 | Gilson Pipetman Classic Pipets, 200 to 1000µL Storage Temperature and Conditions: Room temperature |
| Plate Sealing Film | Bio-Rad | MSB1001 | Microseal “B” PCR Plate Sealing Film, Adhesive, Optical Storage Temperature and Conditions: Room temperature |
| Plate Spinner | Thermo Fisher | 14-100-141 | Fisherbrand Mini Plate Spinner Centrifuge, 230 V Storage Temperature and Conditions: Room temperature |
| Pre-Hood Filter | USA Scientific | 4235-3724 | Prefilter for AirClean Systems Workstations Storage Temperature and Conditions: Room temperature |
| R Software | The R Project for Statistical Computing | Not Applicable | R version 4.2.2 Storage Temperature and Conditions: Room temperature |
| Scale | Thermo Fisher | 01-922-329 | OHAUS 30430060 PR Series Analytical Balance, 62g Capacity Storage Temperature and Conditions: Room temperature |
| Scissors | Office Depot | 458612 | Office Depot Brand Scissors, 8”, Straight, Black, Pack of 2 Storage Temperature and Conditions: Room temperature |
| Sharpies | Sharpie | 2151734 | Brush Twin Permanent Markers, Black Storage Temperature and Conditions: Room temperature |
| Single Copy Gene Forward Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Single Copy Gene Reverse Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Small Tube Rack | Thermo Fisher | 21-402-17 | Thermo Fisher 8601 Reversible Microtube Racks with Lid Storage Temperature and Conditions: Room temperature |
| Sodium Acetate | Thermo Fisher | J63560.EQE | 3M NaOAc pH 5.2 Storage Temperature and Conditions: Room temperature |
| Stainless Steel Spatula | Thermo Fisher | 3990240 | Bel-Art SP Scienceware Stainless-Steel Sampling Spoon and Spatula Storage Temperature and Conditions: Room temperature |
| SYBR Green | Thermo Fisher | S7563 | SYBR Green I Nucleic Acid Gel Stain – 10,000X Concentrate in DMSO Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Syringe Filters | Fisher Scientific | 09-927-55A | GD/X 25 mm Sterile Syringe Filter, cellulose acetate filtration medium, 0.2 μm Storage Temperature and Conditions: Room temperature |
| Syringes | Thermo Fisher | 148232A | BD Luer-Lok Disposable Syringes without Needles, 10mL Storage Temperature and Conditions: Room temperature |
| TE Buffer | Fisher Scientific | BP2474100 | TE Buffer, Tris-EDTA, 1X Solution, pH 7.6, Molecular Biology, Fisher BioReagents Storage Temperature and Conditions: Room temperature |
| Telomere Forward Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| Telomere Reverse Primer | Integrated DNA Technologies | Custom | See separate table Storage Temperature and Conditions: minus 20 °C when rehydrated |
| UV Light | USA Scientific | 4288-2540 | UV Light Bulb for Workstations Storage Temperature and Conditions: Room temperature |
| Vortex | Thermo Fisher | 14-955-151 | Fisherbrand Mini Vortex Mixer, 115 V, 50/60 Hz Storage Temperature and Conditions: Room temperature |
| Weigh Boat | Thermo Fisher | 01-549-752 | Fisherbrand Sterile Hexagonal Weighing Boat, 10mL Storage Temperature and Conditions: Room temperature |
Referenzen
- Hemann, M., Strong, M., Hao, L., Greider, C. The shortest telomere, not average telomere length, is critical for cell viability and chromosome stability. Cell. 107 (1), 67-77 (2001).
- Xu, L., Li, S., Stohr, B. A. The role of telomere biology in cancer. Ann Rev Pathol: Mech Dis. 8, 49-78 (2013).
- Lindrose, A., Drury, S. Minimum reporting recommendations for pcr-based telomere length measurement. Telomere Research Network. , (2020).
- Verhulst, S., et al. Commentary: The reliability of telomere length measurements. Int J Epidemiol. 44 (5), 1683-1686 (2015).
- Aubert, G., Hills, M., Lansdorp, P. M. Telomere length measurement-caveats and a critical assessment of the available technologies and tools. Mutat Res. 730 (1-2), 59-67 (2012).
- Mundstock, E., et al. Effect of obesity on telomere length: Systematic review and meta-analysis. Obesity. 23 (11), 2165-2174 (2015).
- Haycock, P. C., et al. Leucocyte telomere length and risk of cardiovascular disease: Systematic review and meta-analysis. BMJ. 349, 4227 (2014).
- Astuti, Y., Wardhana, A., Watkins, J., Wulaningsih, W. Cigarette smoking and telomere length: A systematic review of 84 studies and meta-analysis. Environ Res. 158, 480-489 (2017).
- Ridout, K. K., Ridout, S. J., Price, L. H., Sen, S., Tyrka, A. R. Depression and telomere length: A meta-analysis. J Affect Disord. 191, 237-247 (2016).
- Wang, Q., Zhan, Y., Pedersen, N. L., Fang, F., Hägg, S. Telomere length and all-cause mortality: A meta-analysis. Ageing Res Rev. 48, 11-20 (2018).
- Hu, R., Hua, X. G., Jiang, Q. C. Associations of telomere length in risk and recurrence of prostate cancer: A meta-analysis. Andrologia. 51 (7), e13304 (2019).
- Schneider, C. V., et al. Association of telomere length with risk of disease and mortality. JAMA Inter Med. 182 (3), 291-300 (2022).
- Deng, Y., et al. Telomere length and the risk of cardiovascular diseases: A mendelian randomization study. Front Cardiovasc Med. 9, 1012615 (2022).
- D'mello, M. J., et al. Association between shortened leukocyte telomere length and cardiometabolic outcomes: Systematic review and meta-analysis. Circulation: Cardiovasc Gene. 8 (1), 82-90 (2015).
- Wilbourn, R. V., et al. The relationship between telomere length and mortality risk in non-model vertebrate systems: A meta-analysis. Philos Trans R Soc Lond B Biol Sci. 373 (1741), 20160447 (2018).
- Gardner, M., et al. Gender and telomere length: Systematic review and meta-analysis. Exp Gerontol. 51, 15-27 (2014).
- Lai, T. -. P., Wright, W. E., Shay, J. W. Comparison of telomere length measurement methods. Philos Trans R Soc Lond B Biol Sci. 373 (1741), 20160451 (2018).
- Cawthon, R. Telomere measurement by quatitative pcr. Nuc Acid Res. 30 (10), e47-e53 (2002).
- Cawthon, R. M. Telomere length measurement by a novel monochrome multiplex quantitative pcr method. Nuc Acid Res. 37 (3), e21-e21 (2009).
- Lindrose, A. R., et al. Method comparison studies of telomere length measurement using qpcr approaches: A critical appraisal of the literature. PLoS One. 16 (1), e0245582 (2021).
- Lin, J., et al. Effects of DNA extraction, DNA integrity, and laboratory on the precision of qpcr-based telomere length measurement - a multi-lab impartial study. bioRxiv. , (2022).
- Wilfinger, W. W., Mackey, K., Chomczynski, P. Effect of ph and ionic strength on the spectrophotometric assessment of nucleic acid purity. Biotechniques. 22 (3), 474-481 (1997).
- Boesenberg-Smith, K. A., Pessarakli, M. M., Wolk, D. M. Assessment of DNA yield and purity: An overlooked detail of pcr troubleshooting. Clin Microbiol Newsletter. 34 (1), 1-6 (2012).
- Harley, C. B., Futcher, A. B., Greider, C. W. Telomeres shorten during ageing of human fibroblasts. Nature. 345 (6274), 458-460 (1990).
- Kimura, M., et al. Measurement of telomere length by the southern blot analysis of terminal restriction fragment lengths. Nat Protoc. 5 (9), 1596-1607 (2010).
- Ye, Q., et al. Telomere length and chronological age across the human lifespan: A systematic review and meta-analysis of 414 study samples including 743,019 individuals. Ageing Res Rev. 90, 102031 (2023).
- Axelrad, M. D., Budagov, T., Atzmon, G. Telomere length and telomerase activity; a yin and yang of cell senescence. J Vis Exp. (75), e50246 (2013).
- Liu, M., et al. Immune-mediated inflammatory diseases and leukocyte telomere length: A mendelian randomization study. Front Genetics. 14, 1129247 (2023).
- Van Ockenburg, S., et al. Stressful life events and leukocyte telomere attrition in adulthood: A prospective population-based cohort study. Psychol Med. 45 (14), 2975-2984 (2015).
- Zong, Z. Q., et al. Ambient air pollution exposure and telomere length: A systematic review and meta-analysis. Public Health. 215, 42-55 (2023).
- Tang, L., Li, D., Wang, J., Su, B., Tian, Y. Ambient air pollution, genetic risk and telomere length in uk biobank. J Expo Sci Environ Epidemiol. , (2023).
- Aubert, G., Hills, M., Lansdorp, P. Telomere length measurement-caveats and a critical assessment of the available technologies and tools. Mutat Res. 730 (1), 59-67 (2012).
- Lin, J., et al. Systematic and cell type-specific telomere length changes in subsets of lymphocytes. J Immunol Res. 2016, 5371050 (2016).
- Needham, B. L., et al. Socioeconomic status, health behavior, and leukocyte telomere length in the national health and nutrition examination survey, 1999-2002. Soc Sci Med. 85, 1-8 (2013).
- Verhulst, S. Improving comparability between qpcr-based telomere studies. Mol Ecol Res. 20 (1), 11-13 (2020).
- Dagnall, C. L., et al. Effect of pre-analytic variables on the reproducibility of qpcr relative telomere length measurement. PloS one. 12 (9), e0184098 (2017).
- Lin, J., Smith, D. L., Esteves, K., Drury, S. Telomere length measurement by qpcr-summary of critical factors and recommendations for assay design. Psychoneuroendocrinology. 99, 271-278 (2019).
- Wolf, S. E., et al. Cross-tissue comparison of telomere length and quality metrics of DNA among individuals aged 8 to 70 years. PLOS One. 19 (2), e0290918 (2024).
- RoDer, B., FruHwirth, K., Vogl, C., Wagner, M., Rossmanith, P. Impact of long-term storage on stability of standard DNA for nucleic acid-based methods. J Clin Microbiol. 48 (11), 4260-4262 (2010).
- Dagnall, C., et al. Effect of pre-analytic variables on the reproducibility of qpcr relative telomere length measurement. PLoS One. 12 (9), e0184098 (2017).
- Shao, W., Khin, S., Kopp, W. C. Characterization of effect of repeated freeze and thaw cycles on stability of genomic DNA using pulsed field gel electrophoresis. Biopreserv Biobank. 10 (1), 4-11 (2012).
- Institute for Statistics and Mathematics of WU. The Comprehensive R Archive Network Available from: https://cran.r-project.org/ (2024)
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biol Rev. 85 (4), 935-956 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten