Method Article
Investigación de las interacciones entre bacterias y hongos utilizando columnas de carreteras fúngicas en diversos entornos y sustratos
En este artículo
Resumen
Este protocolo proporciona instrucciones detalladas sobre cómo construir, esterilizar, ensamblar, utilizar y reutilizar las columnas de autopistas fúngicas para enriquecer los pares de bacterias y hongos que interactúan a través de las autopistas fúngicas de diversos sustratos ambientales.
Resumen
Las interacciones bacteriano-fúngicas (BFI) desempeñan un papel integral en la configuración de la composición de la comunidad microbiana, las funciones biogeoquímicas, la dinámica espacial y la dispersión microbiana. Las redes miceliales creadas por hongos filamentosos u otros microorganismos filamentosos (por ejemplo, oomicetos) actúan como "autopistas fúngicas" que pueden ser utilizadas por las bacterias para su transporte a través de entornos heterogéneos, lo que facilita en gran medida su movilidad y les otorga acceso a regiones que pueden ser difíciles o imposibles de alcanzar por sí solas (por ejemplo, debido a las bolsas de aire dentro del suelo). Se han creado varios dispositivos y protocolos experimentales para estudiar estas autopistas fúngicas, incluidas las columnas de autopistas fúngicas. La columna de autopista fúngica diseñada por nuestro grupo se puede utilizar para una variedad de aplicaciones in situ o in vitro , así como con diversos tipos de muestras ambientales y asociadas al huésped. En este artículo, describimos los métodos para realizar experimentos con estas columnas, incluido el diseño, la impresión, la esterilización y la preparación de los dispositivos. Las opciones para analizar los datos obtenidos del uso de estos dispositivos también se discuten aquí, y se ofrecen consejos para la solución de problemas con respecto a las posibles trampas asociadas con los experimentos con columnas de autopistas fúngicas. Estos dispositivos se pueden utilizar para obtener una comprensión más completa de la diversidad, los mecanismos y la dinámica de los BFI de las carreteras fúngicas para proporcionar información valiosa sobre la dinámica estructural y funcional dentro de entornos complejos (por ejemplo, suelos) y en diversos hábitats en los que coexisten bacterias y hongos.
Introducción
Las interacciones bacteriano-fúngicas (BFI) son extremadamente importantes para dar forma a las propiedades estructurales, espaciales y funcionales de los microbiomas ambientales. Por ejemplo, el crecimiento y la expansión de hongos filamentosos u otros microorganismos filamentosos similares a los hongos genera una red biológica que puede funcionar como una "autopista" para facilitar el movimiento de otros microorganismos, como las bacterias. La heterogeneidad y la saturación inconsistente dentro de los sustratos ambientales pueden dificultar la motilidad bacteriana; Sin embargo, las bacterias pueden utilizar estas carreteras para facilitar el acceso a áreas adicionales del medio ambiente 1,2. Estas interacciones son fundamentales para comprender la dinámica espacial de las comunidades microbianas. Se han utilizado varias técnicas y métodos para examinar las carreteras fúngicas, sin embargo, se limitan en gran medida a investigaciones de laboratorio 3,4.
En un método basado en placas, se extrae una gran sección de agar del centro de la placa de Petri, creando un espacio entre dos islas de agar. Las hifas fúngicas pueden atravesar esta brecha, proporcionando los medios para que las bacterias compatibles crucen de una isla de agar a la otra5. Otros métodos modificados de placas de Petri incluyen placas invertidas donde la tierra se coloca en la tapa para que las hifas fúngicas puedan crecer verticalmente y colonizar el medio sin contacto directo, proporcionando los medios para el transporte bacteriano 5,6. Un método basado en gotitas de medios de crecimiento que se ha desarrollado recientemente se puede utilizar para evaluar el transporte selectivo de hifas de bacterias hacia ciertos perfiles de nutrientes7. También se han utilizado dispositivos de puente y rastro bacteriano para investigar el efecto de los factores abióticos sobre el movimiento bacteriano8. Aunque se han utilizado varios métodos y técnicas para investigar las carreteras fúngicas, sigue siendo necesario contar con dispositivos estandarizados que mantengan un microambiente estéril y promuevan el establecimiento de autopistas fúngicas a partir de sustratos ambientales complejos como el estiércol, el suelo y las rizosferas.
Nuestro grupo diseñó una versión impresa en 3D de columnas de autopistas fúngicas donde los hongos pueden transportar bacterias de un extremo al otro9. Estos dispositivos se ensamblan a partir de cuatro componentes impresos: la columna en sí con forma de reloj de arena y una estructura de celosía interna compleja, un anillo roscado y dos tapas (una tapa grande y otra pequeña), así como una pieza de malla de nailon esterilizada (Figura 1). La columna ensamblada se agrega directamente al sustrato ambiental deseado. Luego, la columna permite que los microbios colonicen un tapón de medio de crecimiento de agar conocido como tapón de medio "cebo" que se encuentra en la parte inferior de la columna y en contacto con el sustrato ambiental a través de la malla. Esta pieza de malla de nailon excluye a otros habitantes del suelo que pueden transportar bacterias, limitando así el movimiento bacteriano dentro de las columnas a las carreteras de hongos. Una vez que este tapón de cebo ha sido colonizado, los hongos filamentosos pueden extenderse y crecer a través de la red interna dentro del centro de la columna que está diseñada para crear un sistema insaturado que se asemeje al suelo (u otros medios no saturados) y minimizar la contaminación potencial del medio de cebo. Luego, los hongos crecen y colonizan el tapón de medio objetivo en la parte superior de la columna. Las columnas se pueden inocular con aislados de hongos específicos para probar su capacidad de transportar bacterias, o se pueden dejar sin inocular para identificar qué hongos del sustrato son capaces de transportar bacterias. Los organismos que alcanzan el medio objetivo pueden ser cultivados, aislados y sometidos a análisis de secuenciación (ya sea a partir de cultivos puros o de comunidades mixtas utilizando enfoques de secuenciación de amplicones o metagenómica). En general, las columnas proporcionan un método estandarizado, reproducible, reutilizable e intuitivo para interrogar las autopistas de hongos en diversos sustratos. Estos dispositivos se pueden utilizar para la investigación y como herramienta de enseñanza en el aula, y en este documento, proporcionamos pasos instructivos para usarlos en función de los experimentos que se han realizado en el pasado. Aunque este método facilita la estandarización del protocolo, el diseño y la construcción de los dispositivos se pueden modificar para otras aplicaciones y sustratos adicionales.
Protocolo
Los detalles de los reactivos y el equipo utilizado en el estudio se enumeran en la Tabla de Materiales.
1. Modificación del diseño de la columna, los materiales y los parámetros
- Descargue los diseños de pilaresdisponibles públicamente 9 y utilícelos tal cual o modifique los diseños de pilares en software de diseño asistido por ordenador (CAD) compatible.
- Obtenga resina de guía quirúrgica dental o seleccione un material de impresión 3D alternativo, como otras resinas transparentes fotosensibles.
- Ajuste las especificaciones de la columna según sea necesario si la impresora 3D, la tecnología de impresión 3D o el material de impresión 3D elegidos se cambian con respecto a lo que se utilizó anteriormente9.
2. 3D impresión de las columnas
- Configure los parámetros de impresión para utilizar un grosor de corte de 0,05 mm y un tiempo de exposición de 0,8 s, o ajuste los parámetros de impresión en función de la impresora, el material de impresión y el software de impresión seleccionados.
- Imprima las columnas de la carretera fúngica, los anillos roscados y las tapas con una impresora 3D compatible (Figura 1).

Figura 1: Componentes de la columna de la carretera fúngica. (A,B) Vistas superior y lateral de la tapa pequeña, el anillo roscado y la tapa grande (de izquierda a derecha). (C,D) Vista superior y lateral de la tapa pequeña. (E,F) Vista superior y lateral del anillo roscado. (G,H) Vista superior y lateral de la tapa grande. (I) Una pieza de filtro de malla de nylon (25 μm) colocada en el extremo de la columna, e insertada en el sustrato ambiental para evitar que la microfauna ingrese a la columna. (J) Columna sin montar. (K) Columna ensamblada: el extremo 'Cebo' entra en el sustrato y el extremo 'Objetivo' permanece descubierto y fuera del sustrato. Haga clic aquí para ver una versión más grande de esta figura.
3. Limpieza de los componentes de la columna impresa en 3D
- Sumerja las columnas, los tapones y los anillos roscados impresos en alcohol isopropílico al 99% a temperatura ambiente durante 15 minutos y agite moviendo el baño de un lado a otro con la mano durante 10-15 s cada minuto para eliminar el exceso de resina.
- Transfiera los componentes a un baño fresco de alcohol isopropílico al 99%. Sumerja durante 5 minutos y agite con la mano de la misma manera que en el paso 3.1.
- Transfiera los componentes a un dispositivo limpiador ultrasónico lleno de agua pura, sumerja los componentes y agite durante 2 minutos a velocidad media. No caliente el agua. Retire los componentes.
- Seque al aire todos los componentes durante al menos 30 minutos.
- Para realizar el poscurado de la resina, exponga todos los componentes impresos en 3D a una luz de 405 nm durante 30 minutos a 60 °C.
4. Esterilización de las columnas
- Si el material elegido es esterilizable en autoclave, autoclave las columnas, los anillos roscados, los tapones y las láminas de filtro de malla de nailon de 25 μm individualmente o dentro de un vaso de precipitados más grande durante 20-30 min a 121 °C, 1 atm.
NOTA: Los componentes de la columna pueden cambiar de forma y color después del autoclave, pero mantendrán las propiedades de material deseadas. El tamaño, la forma y el color finales de los componentes de la columna después del autoclave se muestran en la Figura 1.
5. Preparación de los medios para las columnas
- Prepare placas de Petri de 90 mm de medios esterilizados a base de agar: medio de carboximetilcelulosa de sodio (CMC), agar extracto de malta (MEA), agar papa dextrosa (PDA) o agar Reasoner 2A (R2A)9,10. Prepare el medio siguiendo las instrucciones del fabricante.
- Esterilice el medio esterilizándolo en autoclave durante 21 minutos o el tiempo recomendado por el fabricante a 121 °C, 1 atm, y vierta en placas de Petri de 90 mm hasta que el agar esté cerca de la parte superior de los lados de las placas de Petri (Figura 2). Realice este paso en una cabina de seguridad biológica para aumentar la esterilidad.
- Deje que el agar se solidifique y se seque según las instrucciones del fabricante.

Figura 2: Proceso de montaje de las columnas de la carretera fúngica. Usando un extremo abierto de la columna misma, se corta e inserta un tapón, y el investigador gira la columna a medida que se retira del medio para asegurarse de que el tapón permanezca dentro del extremo de la columna. Ese extremo está tapado con la pequeña pieza de tapa. A continuación, se añade un conector de medios en el otro extremo de la columna de la misma manera. A continuación, la pieza de malla se coloca sobre este extremo y se asegura con el anillo roscado. A continuación, se utiliza el tapón grande en este extremo de "cebo" sobre el anillo roscado. El lado con la malla se colocará en el sustrato ambiental. Haga clic aquí para ver una versión más grande de esta figura.
6. Preparación de las columnas de la carretera de hongos
NOTA: Este paso debe realizarse en una cabina de seguridad biológica para mantener la esterilidad de los componentes de la columna y los medios. La Figura 2 ilustra el proceso de montaje de la columna de la carretera fúngica.
- Utilice el propio extremo de la columna (sin tapas) para extraer un conector de medios que encaje firmemente en un extremo de la columna. Utilice un movimiento de torsión a medida que la columna se levanta del medio para que el agar permanezca dentro del extremo de la columna. También puede utilizar el extremo de la columna como plantilla, tallar el medio y transferirlo a la columna con la punta de una pipeta.
- Después de agregar el primer tapón al final de la columna, agregue la tapa pequeña a este extremo para mantener un microambiente estéril para los medios objetivo.
- Dale la vuelta a la columna y repite el paso 6.1 para el otro extremo de la columna.
- Corte una pieza circular de ~2 cm de diámetro de la malla de nailon esterilizada en autoclave (tamaño de poro de 25 μm) con tijeras esterilizadas (Figura 1). Coloca la malla sobre el extremo expuesto de la columna. Gire el anillo roscado en este extremo del medio de cebo de la columna mientras asegura la malla dentro de los hilos.
- Coloque la tapa grande en la parte inferior de la columna sobre la malla y el otro extremo del anillo roscado, y manténgala puesta cuando almacene o transporte la columna.
7. Inocular previamente el sustrato o el medio de cebo con un hongo de interés
NOTA: Este paso es opcional.
- Inocular un hongo de interés (por ejemplo, un hongo que se sabe que crea autopistas fúngicas) en el sustrato deseado cultivando primero el hongo en un medio sólido de crecimiento fúngico (p. ej., MEA, PDA; preparado como se describe en el paso 5) dentro de una placa de Petri y transfiriendo una pequeña sección (~ 1 cm) de agar con crecimiento fúngico visible al sustrato.
NOTA: En experimentos previos9 se ha utilizado sustrato de estiércol precolonizado con Coprinopsis cinerea durante 10 días antes de añadir las columnas al sustrato precolonizado. - En lugar de colonizar previamente el sustrato, agregue un hongo de cebo a una placa de Petri o directamente al fondo del medio de cebo dentro de la columna usando una pequeña cantidad de micelio fúngico (por ejemplo, deslizando un bucle estéril).
- Para la placa de Petri, espere hasta que haya un crecimiento visible que cubra una gran parte de la placa de Petri (alrededor del 50%-75% de la placa cubierta), luego elimine la sección del medio de cebo directamente desde la placa de Petri colonizada más cercana al borde exterior del crecimiento fúngico (como se describe en el paso 6.1).
- Al precolonizar directamente el medio de cebo, espere hasta que haya un crecimiento claro en todo el medio de cebo antes de agregar la columna al sustrato (esto probablemente llevará varios días).
NOTA: En experimentos previos9 se han utilizado medios de 14 días de edad precolonizados con C. cinerea como medio de cebo; Los tiempos de precolonización variarán según la tasa de crecimiento del hongo, el tipo de medio y las condiciones de incubación.
8. Preparación de los tratamientos de control y réplicas
- Prepare columnas de control negativo (como se describe en los pasos 1 a 6) y no las inocule ni coloque estas columnas en el sustrato. Utilícelos para proporcionar una línea de base para análisis posteriores y para evaluar cualquier contaminación en el proceso de preparación.
- Incluya al menos tres réplicas de columnas para cualquier experimento.
9. Añadir la columna al sustrato
- Establezca un microcosmos de laboratorio (por ejemplo, tierra dentro de una caja, maceta o tubo; Figura 3A) o lleve las columnas a un sitio de campo de interés.
- Retire la tapa grande de la parte inferior de la columna para exponer el anillo roscado y la malla.
- Agregue la columna al sustrato de interés (Figura 3) siguiendo las consideraciones específicas para cada tipo de sustrato que se describen a continuación. Si es necesario, y para minimizar el daño a la malla, haga una depresión en el sustrato con una mano enguantada o una herramienta pequeña (por ejemplo, una paleta pequeña) antes de insertar la columna en el sustrato.
- Estiércol: Las columnas se utilizaban anteriormente con éxito en el estiércol fresco de caballo. Almacene el estiércol a 4 °C antes de usarlo y añádalo a las cámaras, como las cajas magenta, antes de agregar las columnas. Inserte la columna en el sustrato para cubrir 1-2 cm de la altura total de la columna.
- Suelo: Inserte las columnas de modo que toda la sección inferior quede dentro del suelo (~1-2 cm de profundidad del suelo) (Figura 3B). Las columnas pueden descansar en la parte superior del suelo o enterrarse hasta justo debajo de la pequeña tapa en el lado objetivo.
- Rizosfera: Inserte las columnas en el suelo alrededor de las raíces de las plantas como se indica en el paso 9.3.2 mientras inclina y coloca la columna cerca de ciertas raíces para aumentar las posibilidades de capturar microorganismos de la rizosfera (Figura 3C).
- Sustratos dentro de tubos: Agregue ~ 10 mL de un sustrato de interés a un tubo cónico de 50 mL o similar y humedezca el sustrato si lo desea (por ejemplo, en ambientes de muy baja humedad) con agua pura. Inserte la columna en el tubo de modo que la parte inferior quede enterrada y no se vean partes del anillo roscado o la malla (Figura 3A). Atornille la tapa del tubo de 50 ml para cerrarlo y colóquelo en una rejilla de tubo de 50 ml para mantenerlo en posición vertical.
10. Dejar la columna en el sustrato
- Deje la columna en el sustrato durante 3-21 días.
NOTA: La cantidad de tiempo que las columnas deben permanecer en sus sustratos depende de la tasa de crecimiento de los hongos de cebo utilizados, la tasa de crecimiento de los hongos que colonizan el medio de cebo y las condiciones ambientales (por ejemplo, las condiciones secas conducen a una desecación más rápida del medio, por lo que no se pueden usar durante tanto tiempo). - Establecer un ciclo de luz/oscuridad deseado para el experimento; Experimentos previos han mantenido las columnas dentro del laboratorio en la oscuridad, mientras que las columnas en el campo han sido sometidas a las condiciones locales de día y noche9.
11. Retirar la columna del sustrato
- Después de que la columna haya estado en contacto con el sustrato durante el tiempo deseado, retire la columna, sacuda con cuidado cualquier exceso de sustrato y vuelva a agregar la tapa grande en la parte inferior de la columna debajo de la malla para transportar la columna.
- Coloque la columna en un vaso de precipitados estéril o en un tubo de 50 ml para su transporte. Mantenga la columna en posición vertical durante el transporte.
12. Cultivo de aislados del medio objetivo de la columna
- En un entorno estéril, como una cabina de seguridad biológica, retire la tapa grande del medio de cebo, el anillo roscado y la malla. Deseche la malla.
NOTA: Si un investigador está interesado en saber qué organismos colonizaron el medio de cebo, siga los pasos 12.3-12.4 con el tapón del medio de cebo además del tapón del medio objetivo. - Retire la tapa pequeña del extremo de la columna que contiene el medio de destino.
- Extraiga el tapón medio objetivo volteando la columna para permitir que el tapón objetivo se salga de la columna, o utilizando pinzas esterilizadas o puntas de pipeta para extraer el tapón. Coloque el tapón de medio objetivo directamente en el centro de una placa de Petri de 90 mm llena de un medio de agar de su elección (como se preparó en el paso 5).
NOTA: Aquí se pueden usar medios específicos para hongos o bacterias para mejorar el crecimiento de los miembros de cualquiera de los reinos; el tapón también se puede dividir en dos o más piezas con tijeras esterilizadas o una punta de pipeta, lo que permite el recubrimiento en múltiples tipos de medios y/o la conservación de los medios objetivo para las extracciones de ADN (consulte el paso 14). - Incubar la placa de Petri inoculada con el tapón de medio objetivo durante al menos 72 h a 25 °C en la oscuridad.
13. Subcultivo para aislar microorganismos
NOTA: Este paso es opcional.
- Para bacterias: Usando un asa de inoculación estéril, deslice el dedo por una sola colonia de interés mientras minimiza el contacto con otras áreas de la placa de Petri. Coloque la colonia en una placa de Petri fresca de 90 mm que contenga el medio bacteriano preferido, como R2A (como se preparó en el paso 5), utilizando cualquier método que permita la formación de colonias individuales (por ejemplo, rayando en cuatro zonas). Incubar la placa de Petri durante 24-72 h a 25-37 °C, comprobando el crecimiento de la colonia.
- Para hongos: Usando un asa de inoculación estéril, una navaja de afeitar estéril o una punta de pipeta estéril, corte una sección mínima (1 mm2) de agar que contenga crecimiento de hifas. Coloque el pequeño trozo de agar en una placa de Petri fresca de 90 mm que contenga el medio fúngico preferido, como MEA o PDA, tal como se preparó en el paso 5. Incubar la placa de Petri durante un máximo de una semana a 25 °C en la oscuridad, comprobando diariamente para evitar el crecimiento excesivo.
- Repita el paso 1 o el paso 2 según sea necesario hasta que las morfologías del organismo sean uniformes.
14. Extracción de ADN de la placa o directamente del medio objetivo
- Congele todos los tapones de agar (blanco y/o cebo) o las piezas seleccionadas de las columnas en tubos de centrífuga de 1,5 mL a -20 °C o sumerja las piezas de los tapones en un conservante en un tubo de centrífuga de 1,5 mL antes de la extracción si las extracciones no se van a realizar inmediatamente.
- Si se realizó el cultivo del objetivo y/o del medio de cebo, y si se tomaron medidas de subcultivo posteriores, estampe o talle un trozo de agar de ~ 1 cm con crecimiento de hongos o bacterias (o una mezcla de ambos si no se tomaron medidas de aislamiento posteriores).
- Para extracciones de colonias bacterianas aisladas, deslice una colonia de la placa con un asa de inoculación estéril y gírela directamente en el tampón de extracción asociado con el kit comercial de extracción de ADN (paso 14.4).
- Muela las piezas de agar por separado en nitrógeno líquido utilizando un mortero y transfiere el tejido molido a los tubos de extracción.
- Utilice un kit comercial de extracción de ADN optimizado para tierra o bacterias y hongos, y siga las instrucciones del fabricante para extraer el ADN (consulte la Tabla de materiales).
- Cuantifique el ADN resultante utilizando un fluorómetro o un sistema comparable.
15. Evaluación de la diversidad taxonómica microbiana en el medio objetivo y/o de cebo utilizando enfoques de secuenciación de amplicones o metagenómica
- Realice la secuenciación de amplicones (16S y/o espaciador interno transcrito [ITS]) o la secuenciación metagenómica siguiendo los pasos que se indican a continuación:
- Secuenciación de amplicones: Amplifique la región del gen rRNA V3-V4 16S utilizando los cebadores Bakt 341F (5′-CCT ACG GGN GGC WGC AG-3′) y Bakt 805R (5′-GAC TAC HVG GGT ATC TAA TCC-3′). Amplifique la región ITS2 fúngica utilizando los cebadores ITS3 KYO2 (5′-GAT GAA GAA CGY AGY RAA-3′) e ITS4 (5′-TCC TCC GCT TAT TGA TAT GC-3′)9.
- Prepare bibliotecas de amplicones utilizando kits de preparación de bibliotecas comerciales compatibles con el secuenciador elegido. Secuencie los amplicones utilizando un secuenciador de lectura corta para generar 150 o 250 lecturas de extremos emparejados de pares de bases con suficiente cobertura siguiendo las instrucciones del fabricante de la plataforma de secuenciación para la concentración de carga.
- Secuenciación metagenómica: Cree una biblioteca metagenómica a partir del ADN extraído utilizando un kit de preparación de bibliotecas metagenómicas disponible en el mercado y compatible con el secuenciador elegido. Segreñe la biblioteca del metagenoma utilizando un secuenciador de lectura corta configurado para generar 150 o 250 lecturas de pares de bases con suficiente cobertura (~10-20 Gb por metagenoma) siguiendo las instrucciones del fabricante de la plataforma de secuenciación para la concentración de carga.
16. Análisis de los datos de secuenciación
- Analice los datos del amplicón: Utilice la plataforma QIIME2 11 con DADA212 para analizar los datos del amplicón antes de visualizar los resultados13. Siga los pasos que se indican a continuación para ejecutar QIIME2 en la plataforma bioinformática basada en la web Empowering the Development of Genomics Expertise (EDGE)14. QIIME2 también se puede ejecutar utilizando tutoriales disponibles públicamente, como la guía "Moving Pictures" que se proporciona en línea (https://docs.qiime2.org/2024.2/tutorials/moving-pictures/).
- Vaya a https://edgebioinformatics.org/ e inicie sesión o cree una cuenta.
- Seleccione EJECUTAR QIIME2 en la página de inicio. Seleccione Cargar archivos en la barra de menú de la izquierda para cargar archivos de las ejecuciones de secuenciación de amplicón.
- Cree un archivo de asignación de metadatos siguiendo las instrucciones proporcionadas cuando se pasa el cursor sobre la 'i' junto a 'Archivo de asignación de metadatos'.
- Agregue toda la información requerida (Nombre del proyecto/ejecución, tipo de lectura, parámetros, etc.) y seleccione los datos de entrada cargados correctos. Asegúrese de que DADA2 esté seleccionado como método de control de calidad12; Otros parámetros se pueden dejar como sus valores predeterminados a menos que se deseen otras modificaciones.
- Seleccione el tipo de amplicón como 16S Greengenes (http://greengenes.lbl.gov) para los amplicones bacterianos o Fungal ITS para los amplicones fúngicos. Analice los datos bacterianos (16S) y fúngicos (ITS) de forma independiente.
- Presione enviar y espere a que finalice la ejecución para ver los resultados.
- Generar visualizaciones de datos de amplicon (opcional): Realice análisis de datos de comunidad adicionales en R utilizando paquetes comunes como phyloseq (https://github.com/joey711/phyloseq), VEGAN (https://github.com/vegandevs/vegan) y APE (https://emmanuelparadis.github.io/) siguiendo las instrucciones proporcionadas en GitHub y a través de la Ayuda del paquete de R disponible en R Studio navegando hasta el menú Paquetes , haciendo clic en el paquete descargado y viendo la documentación.
- Análisis de datos metagenómicos
- Navegue hasta el sitio NMDC EDGE 15 (https://nmdc-edge.org/home) e inicie sesión con una cuenta ORCiD (https://orcid.org/).
- Seleccione Cargar archivos en la barra de menú en el lado izquierdo de la pantalla y arrastre y suelte o busque los archivos FASTQ de entrada correctos.
- Seleccione Metagenómica y, a continuación, la opción Ejecutar varios flujos de trabajo en la barra de menú de la parte izquierda de la pantalla y establezca todos los flujos de trabajo en Activado. Agregue un nombre de proyecto y una descripción opcional.
- Seleccione los archivos de lectura sin procesar (fastq) cargados y seleccione el formato de archivo apropiado (intercalado o emparejado).
- Inicie la ejecución haciendo clic en Enviar y vea las tablas de resumen y las visualizaciones cuando se haya completado la ejecución seleccionando el proyecto dentro de la pestaña Mis proyectos en la parte superior de la pantalla.
17. Creación de visualizaciones adicionales de datos taxonómicos a partir de resultados ampliconarios y/o metagenómicos
- Genere cladogramas circulares utilizando el paquete GraPhlAn (https://github.com/biobakery/graphlan) siguiendo las instrucciones proporcionadas en GitHub.
NOTA: Los identificadores de taxonomía (https://www.ncbi.nlm.nih.gov/taxonomy) del Centro Nacional de Información Biotecnológica (NCBI) se pueden obtener a partir de asignaciones de secuencia representativas y pasar al programa 'eftech' de Entrez Direct E-utilities para recopilar información de rollup de taxonomía requerida por GraPhlAn16.
18. Reutilización de las columnas
- Si las columnas aún están ensambladas, desatornille y retire las tapas, el anillo roscado y la malla. Deseche la malla. Retire los tapones de agar restantes, lave las columnas con alcohol isopropílico al 99% y agua purificada, y limpie y seque los componentes como se describe en los pasos 3.1-3.4.
- Prepare nuevas láminas de malla de nailon para esterilizarlas en autoclave.
- Esterilizar los componentes en autoclave siguiendo las instrucciones proporcionadas en el paso 4.1.
Resultados
La columna de la carretera fúngica completamente ensamblada tiene aproximadamente 5 cm de largo (Figura 1). La columna no debe romperse en ningún área, y las tapas y el anillo roscado deben encajar fácil y firmemente para crear microambientes dentro de la columna. La malla del filtro puede extenderse más allá del anillo roscado (como se muestra en la Figura 1 y la Figura 2), o se puede recortar con tijeras esterilizadas. Los tapones de agar deben encajar perfectamente en cada extremo de la columna. Cuando se coloca en el sustrato, la malla del filtro debe entrar en contacto con el sustrato y la columna no debe estar completamente enterrada.
Las columnas fueron previamente probadas en estiércol de caballo9. Las columnas también se colocaron en suelo a granel y de rizosfera en un sitio de campo de investigación, así como en pequeños volúmenes de suelo en tubos de 50 mL en el laboratorio (Figura 3). Después de que las columnas de la carretera fúngica se retiraron del sustrato y se desmontaron, el crecimiento microbiano fue visible tanto en el cebo como en los tapones de los medios objetivo (ejemplos mostrados en la Figura 4A). Las bacterias y los hongos se aislaron de los medios objetivo y de cebo mediante técnicas de subcultivo (Figura 4B), y los microbios presentes en los tapones de los medios se identificaron taxonómicamente mediante secuenciación de amplicones (Figura 4C, D). La Figura 4C, D muestra los resultados combinados de la secuenciación de amplicones a través de múltiples experimentos, mostrando qué microbios pudieron alcanzar el tapón de medios objetivo desde las columnas agregadas al estiércol de caballo9. Las visualizaciones de estos datos bacterianos y fúngicos se generaron como se describe en el paso 17. Los resultados también pueden mostrarse como abundancias relativas de taxones.
Se han obtenido resultados subóptimos en los casos en los que las columnas se añadieron a ambientes de humedad extremadamente baja, y los tapones de los medios se secaron por completo en cuestión de días, lo que provocó que no se recuperaran los microbios colonizados (Figura 5A). También hemos visto casos en los que los microbios simplemente no crecen a partir del conector de medios objetivo (Figura 5B), y casos en los que no recuperamos suficientes datos de secuenciación del conector de medios objetivo para realizar análisis significativos. Otros casos en los que los hongos crecen demasiado fuera de las columnas también han dado lugar a la necesidad de repetir los experimentos (Figura 5C).

Figura 3: Ejemplos de las columnas colocadas en muestras ambientales en laboratorio y campo. (A) Columna colocada dentro de un tubo de 50 mL con tierra humedecida en un entorno de laboratorio. También se muestra con una regla para la escala. (B) Columna colocada en el suelo en el campo. (C) Columna colocada en la red de raíces de una planta en el campo. Haga clic aquí para ver una versión más grande de esta figura.
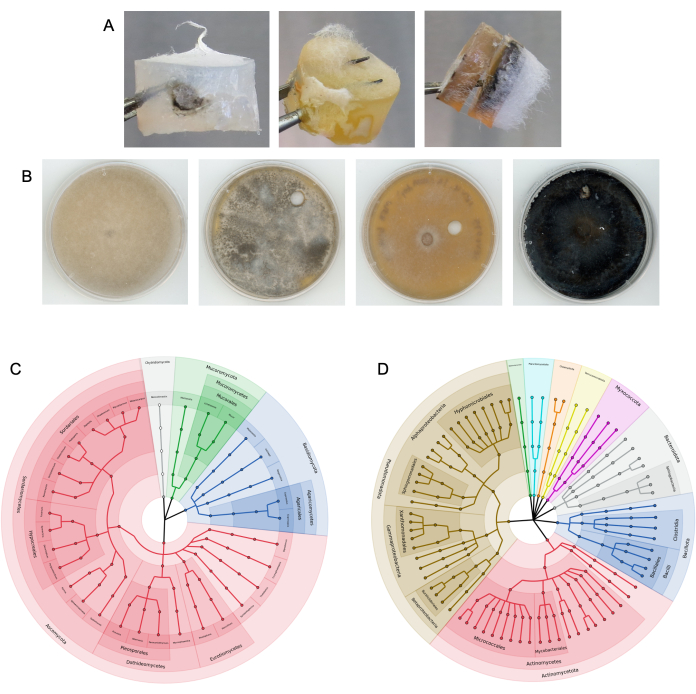
Figura 4: Resultados representativos de experimentos exitosos con columnas. (A) Ejemplos de tapones de medios colonizados extraídos de columnas. (B) Ejemplos de aislados de hongos subcultivados a partir de medios objetivo. El sustrato era tierra. Secuencia ITS superior Identidad de NCBI BLAST de izquierda a derecha: Rhizopus azygosporous, Aspergillus novofumigatus, Curvularia subpapendorfii y Phaeomycocentrospora cantuariensis. (C,D) Cladogramas circulares que muestran la diversidad filogenética de (C) secuencias de ITS fúngicos y (D) 16S bacterianas recuperadas de los medios objetivo después de múltiples experimentos de carreteras fúngicas utilizando estiércol de caballo. Las secciones están coloreadas y etiquetadas por filo, con nodos finales que representan géneros únicos. Haga clic aquí para ver una versión más grande de esta figura.
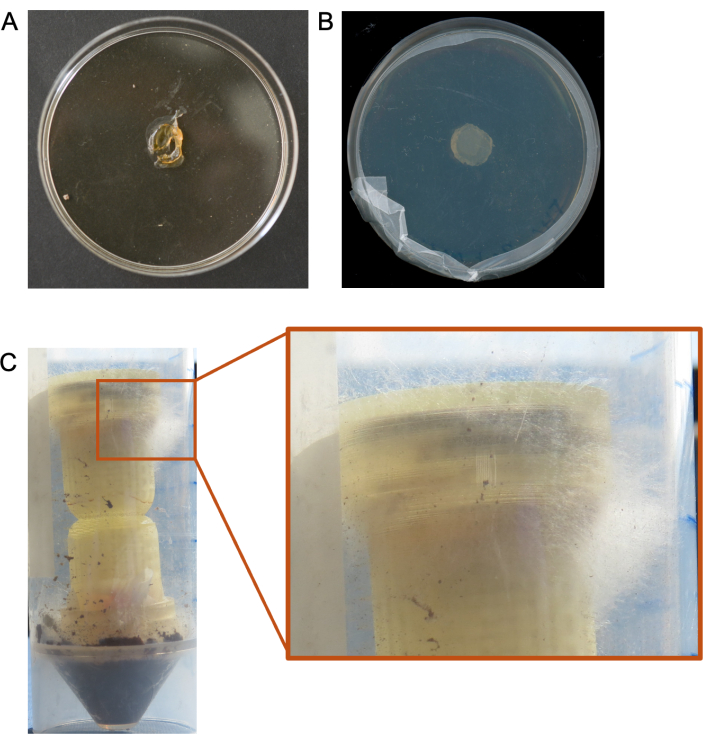
Figura 5. Resultados subóptimos de experimentos en columna. (A) Un ejemplo de un tapón de medio desecado resultante de condiciones ambientales de baja humedad. (B) Ejemplo de ausencia de crecimiento microbiano a partir de un tapón de medios de columna. (C) Ejemplo de crecimiento excesivo del hongo a través de la parte superior (medio objetivo) de la columna. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Al generar los componentes de la columna, la selección de una impresora 3D y el material de impresión se puede modificar en función de la disponibilidad y las propiedades deseadas del material17,18. La biocompatibilidad, la textura de la superficie, la esterilización en autoclave, la capacidad de imprimir detalles a escala fina y la transparencia relativa se tuvieron en cuenta en la selección de materiales de nuestro grupo. También hay que tener en cuenta otras características, como la porosidad, la hidrofobicidad, los parámetros de impresión, etc. Se probaron varias resinas (ver Tabla de Materiales) antes de la selección final, y muchos materiales biocompatibles funcionarán para la impresión de estas columnas. El material elegido para la construcción de los componentes de la columna determinará qué enfoques de limpieza, poscurado y esterilización se deben utilizar. No todos los materiales serán esterilizables en autoclave, y es posible que se requiera luz ultravioleta, lejía u otras técnicas de esterilización, según las instrucciones del fabricante del material. Algunas técnicas de esterilización o limpieza también pueden dañar o no ser compatibles con el material elegido, por lo que se debe prestar especial atención a esta información del fabricante del material. En el caso de las impresoras 3D, algunas consideraciones son el tiempo de impresión, la compatibilidad de los materiales, el tamaño de la plataforma de construcción, la tecnología de impresión y el coste19. Los componentes impresos en 3D de las columnas pueden ser frágiles y pueden romperse si se manipulan con demasiada fuerza. Es posible que el roscado del anillo y las tapas no siempre coincidan exactamente, por lo tanto, recomendamos que se impriman y sterilicen los componentes adicionales antes del paso de ensamblaje, o que se realice una impresión preliminar para probar cómo los parámetros y el material elegido afectan el roscado. Es posible que sea necesario ajustar las especificaciones de diseño para el roscado dentro de las tapas y el anillo en función del material de impresión 3D elegido. Las dimensiones, la complejidad de la celosía y otras características físicas se pueden modificar en el software de diseño CAD antes de la impresión. Según el diseño, la columna en sí tiene 4 cm de altura, y la estructura de celosía dentro del centro de la columna tiene una celda unitaria de 2 mm, un diámetro de puntal de 0,5 mm y la altura total de la celosía es de 22 mm9. Estos parámetros se pueden ajustar si un investigador desea, por ejemplo, una estructura de red más grande o más compleja. En general, la fabricación impresa en 3D de estos dispositivos permite flexibilidad en el diseño y, al mismo tiempo, garantiza que un solo diseño se pueda utilizar de manera estandarizada en todas las organizaciones y grupos, e incluso como herramientas de enseñanza en el aula9.
Varios pasos del protocolo pueden requerir la solución de problemas en función del entorno o de la configuración experimental. Las columnas de autopistas fúngicas no son muy efectivas en condiciones de baja humedad, ya que los tapones de los medios se secan rápidamente antes de facilitar el crecimiento de hongos, lo que puede limitar la duración de los experimentos en estos entornos (Figura 5A). Las técnicas que han mejorado la efectividad de las columnas en ambientes de baja humedad incluyen el aumento artificial de la humedad mediante la adición de humedad al sustrato y/o el sellado de la columna y el sustrato en un recipiente secundario con una fuente de agua (por ejemplo, un pequeño recipiente de agua pura). La forma de reloj de arena y la estructura de celosía se incorporaron para evitar el movimiento bacteriano solo (sin el establecimiento de una autopista de hongos) si se formara condensación en ambientes de alta humedad. Los hongos de crecimiento rápido pueden crecer demasiado en el área de superficie del medio objetivo y del cebo y extenderse fuera de la parte superior o inferior de la columna (Figura 5C). Disminuir el tiempo de incubación del hongo del cebo o la duración del experimento puede minimizar o eliminar este crecimiento excesivo. Además, una limitación de estos dispositivos es que los hongos de rápido crecimiento en el sustrato de interés pueden limitar la colonización del cebo y los medios objetivo por hongos de crecimiento lento, lo que podría sesgar las interacciones en las carreteras que se observan. Es posible que algunos hongos, especialmente los hongos de crecimiento más lento, no colonicen el medio de cebo de una manera que les permita crecer a través del tapón de agar y en la estructura de la red. Si hay suficiente humedad en el ambiente, se pueden usar tapones de agar más delgados para estimular el crecimiento en la red después de la colonización del tapón de agar cebo. Los medios se pueden elegir en función de si un investigador desea seleccionar el crecimiento de hongos o bacterias, pero esto también puede limitar el subcultivo a los organismos que prefieren ese tipo de medio20. Si no se observa crecimiento en el medio objetivo, puede ser necesario inocular el medio de cebo o el sustrato con un hongo que se sabe que crea autopistas fúngicas.
La secuenciación metagenómica o de amplicones puede realizarse como parte de estos experimentos, y ambas estrategias imparten sus propias limitaciones y fortalezas21. La secuenciación metagenómica es ideal para obtener información genómica adicional sobre los microbios. Sin embargo, la cantidad recuperable de ácidos nucleicos directamente del medio objetivo puede ser muy baja, lo que puede requerir la utilización de secuenciación de amplicones u otros métodos de amplificación antes de la secuenciación. Las bibliotecas de secuenciación de amplicones deben prepararse por separado (16S e ITS), y este método carece de resolución taxonómica y limita cualquier evaluación sobre las características del genoma o el potencial funcional que se pueda lograr mediante la secuenciación metagenómica. Es posible que se prefieran los métodos de secuenciación directa de los tapones en los casos en que los microbios no puedan subcultivarse. Se recomienda que los tapones se dividan en varias secciones para permitir los enfoques de cultivo y secuenciación.
Un beneficio de estos dispositivos es que se pueden utilizar tanto en el laboratorio como en el campo. Se debe tener especial cuidado para garantizar que las columnas en el campo puedan permanecer en posición vertical y estén protegidas de animales y perturbaciones ambientales que puedan perturbar su colocación. Las columnas aún no se han probado en posición horizontal, en una posición en la que estén completamente cubiertas por un sustrato, y no se han probado en entornos que estén expuestos a lluvias o nieve sustanciales. Como se indicó anteriormente, la estructura de la red se diseñó para minimizar la probabilidad de que las bacterias puedan moverse al medio objetivo en ambientes de alta humedad. Sin embargo, es posible que si la columna se expusiera a mayores volúmenes de agua y esta agua saturara completamente la columna, el movimiento bacteriano se facilitaría a través de la columna independientemente de cualquier autopistas de hongos presentes. Para experimentos de laboratorio, las columnas se pueden usar dentro de tubos cónicos de 50 mL, pequeños microcosmos de sustratos, en el suelo que rodea las plantas en macetas, en cajas o dentro de otros sistemas experimentales controlados. Las columnas se han utilizado con éxito en suelos, rizosferas y estiércol, y su utilidad se puede ampliar a otros sustratos, como hojarasca, lodo, arena, nieve, compost, etc.
Las columnas de autopistas fúngicas permiten una serie de comparaciones para comprender este fenotipo BFI dentro de diversos tipos de muestras. La comparación de la composición de la comunidad entre el cebo y los medios objetivo puede indicar qué bacterias pueden utilizar las autopistas fúngicas y qué hongos pueden servir como autopistaspotenciales. Si se utiliza la secuenciación del metagenoma, también se pueden examinar las características genómicas que distinguen a los organismos del cebo frente a los medios objetivo. También es posible comparar los medios objetivo de las columnas colocadas en diferentes sustratos (por ejemplo, tierra frente a estiércol) o colocados en el mismo sustrato en diferentes condiciones (por ejemplo, temperatura o humedad). En general, las columnas de autopistas fúngicas amplían las capacidades de los métodos anteriores para interrogar esta forma de BFI y permiten exámenes exhaustivos de estas interacciones que dan forma a la dinámica espacial de los microbiomas ambientales complejos.
Divulgaciones
Los autores no tienen ningún conflicto de intereses que revelar.
Agradecimientos
Esta investigación fue apoyada por una subvención del Área de Enfoque Científico del Departamento de Energía de los Estados Unidos (DOE), Investigación Biológica y Ambiental (BER), División de Ciencias del Sistema Biológico (BSSD) bajo la subvención número LANLF59T.
Materiales
| Name | Company | Catalog Number | Comments |
| 50 mL tubes | Greiner BIO-ONE | 5622-7261 | 50 mL tubes for performing column experiments in the lab |
| 90 mm Petri dishes | Thermo Scientific Nunc | 08-757-099 | Petri dishes for preparation of agar and for microbial growth |
| Asiga Freeform Pico Plus 39 digital light processing (DLP) 3D printer | Asiga Germany | Freeform Pico Plus 39 | 3D printer used to generate batches of the columns; other 3D printers can be used |
| Autoclave | Fisher Scientific | LS40F20 | Benchtop autoclave to sterilize the column components |
| Beaker | Fisher Scientific | FB100600 | 600 mL beaker for various uses throughout the protocol |
| Dental LT Clear Resin V2 | Formlabs | RS-F2-DLCL-02 | Alternative resin for 3D printing that was tested |
| Dental Surgical Guide Resin | Formlabs | RS-F2-SGAM-01 | Was used to generate the columns discussed in manuscript; Other photosensitive resins can be used in place of this material |
| DNA Low Bind 1.5 mL tubes | Eppendorf | 13-698-791 | Tubes used for various preparations including nucleic acid extractions |
| DNA/RNA shield preservative | Zymo Research | R1100-50 | Preservative used prior to nucleic acid extractions |
| EDGE Bioinformatics | Open source; Developed by the Los Alamos National Laboratory (LANL) | n/a | Bioinformatics platform for processing amplicon data |
| FastDNA spin kit for soil | MP Biomedicals LLC | 116560200-CF | DNA extraction kit option for soil |
| Forceps | Fisher Scientific | 10-300 | Forceps that can be sterilized |
| Formlabs BioMed Clear Resin | Formlabs | RS-F2-BMCL-01 | Alternative resin for 3D printing that was tested |
| Formlabs Form 3B+ stereolithography (SLA) 3D printer | Formlabs | Form 3B+ | Alternative 3D printer |
| Formlabs IBT Resin | Formlabs | RS-F2-IBCL-01 | Alternative resin for 3D printing that was tested |
| Inoculating Loops | Fisher Scientific | 22-363-598 | Used to isolate/transfer microbes |
| Malt Extract Agar (MEA) | Criterion | 89405-654 | A media type used in columns |
| MiSeq sequencer + MiSeq sequencing kit | Illumina | SY-410-1003 | Can use other sequencers |
| Mortar & Pestle | Fisher Scientific | FB961K; FB961A | Can use any common mortar & pestle that can be sterilized between uses |
| NEBNext Ultra II DNA Library Prep Kit for Illumina | New England Biolabs | E7805S | Library prep kit for metagenomic sequencing |
| Nextera XT DNA Library Preparation Kit (24 samples) | Illumina | FC-131-1024 | Library prep kit for amplicon sequencing |
| NMDC EDGE | Open source: Developed by the National Microbiome Data Collaborative (NMDC) | n/a | Bioinformatics platform for processing metagenomic data |
| Nylon mesh | Sefar | 03-25/19 | The mesh used as part of the column construction |
| Pipette tips | Rainin | 30807966 | Can use many different sterilized pipette tips for the protocol steps |
| Potato Dextrose Agar | Cole Parmer | EW-14200-28 | A media type used in columns |
| QIIME2 | Open source | n/a | Software for processing amplicon data |
| Qubit dsDNA HS assay kit | Thermo Fisher Scientific | Q32851 | Used to quantify DNA after extractions |
| Qubit Fluorometer | Thermo Fisher Scientific | Q33238 | Used to quantify DNA after extractions |
| Quick-DNA Fungal/Bacterial Miniprep Kit | Zymo Research | D6005 | DNA extraction kit option that works with both bacteria and fungi |
| R2A agar | BD Difco | 218263 | A media type used in columns (bacterial media) |
| Rack for 50 mL tubes | Fisher Scientific | 03-448-11 | Rack to hold 50 mL tubes upright |
| Scissors | Fisher Scientific | 12-000-155 | Fine precision scissors that can be sterilized |
| Sodium carboxymethyl cellulose medium | Aldrich | 419273-100G | A media type used in columns |
| SolidWorks CAD software | SolidWorks | n/a | Software used to design the columns |
| Trowel scoop | Fisher Scientific | S41701 | To make a depression in the substrate prior to adding the column |
| UltraPure DNase/RNase-Free Distilled Water | Invitrogen: ThermoFisher Scientific | 10977015 | Water for the ultrasonicator water bath |
| Ultrasonicator | Fisher Scientific | FB-11201 | Ultrasonicator for cleaning the columns |
Referencias
- Or, D., Smets, B. F., Wraith, J. M., Dechesne, A., Friedman, S. P. Physical constraints affecting bacterial habitats and activity in unsaturated porous media - A review. Adv Water Resour. 30 (6), 1505-1527 (2007).
- Kohlmeier, S., et al. Taking the fungal highway: Mobilization of pollutant-degrading bacteria by fungi. Environ Sci Technol. 39 (12), 4640-4646 (2005).
- Simon, A., Hervé, V., Al-Dourobi, A., Verrecchia, E., Junier, P. An in situ inventory of fungi and their associated migrating bacteria in forest soils using fungal highway columns. FEMS Microbiol Ecol. 93 (1), 217 (2017).
- Wick, L. Y., et al. Effect of fungal hyphae on the access of bacteria to phenanthrene in soil. Environ Sci Technol. 41 (2), 500-505 (2007).
- Bravo, D., et al. Isolation of oxalotrophic bacteria able to disperse on fungal mycelium. FEMS Microbiol Lett. 348 (2), 157-166 (2013).
- Furuno, S., Remer, R., Chatzinotas, A., Harms, H., Wick, L. Y. Use of mycelia as paths for the isolation of contaminant-degrading bacteria from soil. Microb Biotechnol. 5 (1), 142-148 (2012).
- Buffi, M., et al. Fungal drops: A novel approach for macro- and microscopic analyses of fungal mycelial growth. Microlife. 4, 042 (2023).
- Kuhn, T., et al. Design and construction of 3D printed devices to investigate active and passive bacterial dispersal on hydrated surfaces. BMC Biol. 20 (1), 203 (2022).
- Junier, P., et al. Democratization of fungal highway columns as a tool to investigate bacteria associated with soil fungi. FEMS Microbiol Ecol. 97 (2), 003 (2021).
- Reasoner, D. J., Geldreich, E. E. A new medium for the enumeration and subculture of bacteria from potable water. Appl Environ Microbiol. 49 (1), 1-7 (1985).
- Bolyen, E., et al. Reproducible, interactive, scalable, and extensible microbiome data science using QIIME 2. Nat Biotechnol. 37 (8), 852-857 (2019).
- Callahan, B. J., et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nat Methods. 13 (7), 581-583 (2016).
- Vázquez-Baeza, Y., Pirrung, M., Gonzalez, A., Knight, R. EMPeror: A tool for visualizing high-throughput microbial community data. Gigascience. 2 (1), 16 (2013).
- Li, P. -. E., et al. Enabling the democratization of the genomics revolution with a fully integrated web-based bioinformatics platform. Nucleic Acids Res. 45 (1), 67-80 (2017).
- Eloe-Fadrosh, E. A., et al. The National Microbiome Data Collaborative Data Portal: An integrated multi-omics microbiome data resource. Nucleic Acids Res. 50 (1), D828-D836 (2022).
- Entrez Direct: E-utilities on the Unix Command Line. Entrez Programming Utilities Help Available from: https://www.ncbi.nlm.nih.gov/books/NBK179288/ (2024)
- Palmara, G., Frascella, F., Roppolo, I., Chiappone, A., Chiadò, A. Functional 3D printing: Approaches and bioapplications. Biosens Bioelectron. 175, 112849 (2021).
- Guttridge, C., Shannon, A., O'Sullivan, A., O'Sullivan, K. J., O'Sullivan, L. W. Biocompatible 3D printing resins for medical applications: A review of marketed intended use, biocompatibility certification, and post-processing guidance. Ann 3D Print Med. 5, 100044 (2022).
- Yao, L., et al. Comparison of accuracy and precision of various types of photo-curing printing technology. J Phys Conf Ser. 1549 (3), 032151 (2020).
- Basu, S., et al. Evolution of bacterial and fungal growth media. Bioinformation. 11 (4), 182-184 (2015).
- Liu, Y. -. X., et al. A practical guide to amplicon and metagenomic analysis of microbiome data. Protein Cell. 12 (5), 315-330 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados