Method Article
Untersuchung bakteriell-pilzlicher Wechselwirkungen mit Hilfe von Pilzautobahnsäulen in verschiedenen Umgebungen und Substraten
In diesem Artikel
Zusammenfassung
Dieses Protokoll enthält detaillierte Anweisungen zum Bauen, Sterilisieren, Zusammenbauen, Verwenden und Wiederverwenden der Pilzautobahnsäulen, um Bakterien-Pilz-Paare anzureichern, die über Pilzautobahnen aus verschiedenen Umweltsubstraten interagieren.
Zusammenfassung
Bakterien-Pilz-Interaktionen (BFIs) spielen eine wesentliche Rolle bei der Gestaltung der Zusammensetzung der mikrobiellen Gemeinschaft, der biogeochemischen Funktionen, der räumlichen Dynamik und der mikrobiellen Ausbreitung. Myzelnetzwerke, die von filamentösen Pilzen oder anderen filamentösen Mikroorganismen (z. B. Oomyceten) gebildet werden, fungieren als "Pilzautobahnen", die von Bakterien für den Transport in heterogenen Umgebungen genutzt werden können, was ihre Mobilität erheblich erleichtert und ihnen Zugang zu Regionen gewährt, die allein schwer oder unmöglich zu erreichen sind (z. B. aufgrund von Lufteinschlüssen im Boden). Es wurden mehrere Geräte und experimentelle Protokolle entwickelt, um diese Pilzautobahnen zu untersuchen, einschließlich der Säulen von Pilzautobahnen. Die von unserer Gruppe entwickelte Pilzautobahnsäule kann für eine Vielzahl von in situ oder in vitro Anwendungen sowie für verschiedene Umwelt- und wirtsassoziierte Probentypen verwendet werden. Im Folgenden beschreiben wir die Methoden zur Durchführung von Experimenten mit diesen Säulen, einschließlich des Entwerfens, Druckens, Sterilisierens und Vorbereitens der Geräte. Die Optionen zur Analyse von Daten, die durch die Verwendung dieser Geräte gewonnen werden, werden hier ebenfalls diskutiert, und es werden Ratschläge zur Fehlerbehebung in Bezug auf potenzielle Fallstricke im Zusammenhang mit Experimenten mit Pilzautobahnsäulen angeboten. Diese Geräte können verwendet werden, um ein umfassenderes Verständnis der Vielfalt, der Mechanismen und der Dynamik von Pilzautobahn-BFIs zu erlangen und wertvolle Einblicke in die strukturelle und funktionelle Dynamik in komplexen Umgebungen (z. B. Böden) und in verschiedenen Lebensräumen zu erhalten, in denen Bakterien und Pilze nebeneinander existieren.
Einleitung
Bakterien-Pilz-Interaktionen (BFIs) sind äußerst wichtig für die Gestaltung der strukturellen, räumlichen und funktionellen Eigenschaften von Umweltmikrobiomen. Zum Beispiel erzeugt das Wachstum und die Ausbreitung von filamentösen Pilzen oder anderen pilzähnlichen filamentösen Mikroorganismen ein biologisches Netzwerk, das als "Autobahn" fungieren kann, um die Bewegung anderer Mikroorganismen, wie z. B. Bakterien, zu erleichtern. Heterogenität und inkonsistente Sättigung innerhalb von Umweltsubstraten können die bakterielle Motilität behindern; Bakterien können diese Autobahnen jedoch nutzen, um den Zugang zu weiteren Bereichen der Umwelt zu erleichtern 1,2. Diese Wechselwirkungen sind entscheidend für das Verständnis der räumlichen Dynamik mikrobieller Gemeinschaften. Zur Untersuchung von Pilzautobahnen wurden verschiedene Techniken und Methoden eingesetzt, die sich jedoch weitgehend auf laborbasierte Untersuchungen beschränken 3,4.
Bei einer plattenbasierten Methode wird ein großer Teil des Agars aus der Mitte der Petrischale entfernt, wodurch eine Lücke zwischen zwei Agarinseln entsteht. Pilzhyphen können diese Lücke durchqueren und bieten kompatiblen Bakterien die Möglichkeit, von einer Agarinsel zur anderen zu gelangen5. Andere modifizierte Petrischale-Verfahren umfassen umgekehrte Platten, bei denen Erde in den Deckel eingebracht wird, so dass Pilzhyphen vertikal wachsen und das Medium ohne direkten Kontakt besiedeln können, wodurch die Mittel für den Bakterientransport bereitgestellt werden 5,6. Eine kürzlich entwickelte Methode auf Basis von Wachstumsmedien kann verwendet werden, um den selektiven Hyphentransport von Bakterien zu bestimmten Nährstoffprofilen zu bewerten7. Bakterielle Brücken- und Trail-Geräte wurden auch verwendet, um die Wirkung abiotischer Faktoren auf die bakterielle Bewegung zu untersuchen8. Obwohl verschiedene Methoden und Techniken zur Untersuchung von Pilzautobahnen eingesetzt wurden, besteht nach wie vor ein Bedarf an standardisierten Geräten, die eine sterile Mikroumgebung aufrechterhalten und gleichzeitig die Etablierung von Pilzautobahnen aus komplexen Umweltsubstraten wie Mist, Boden und Rhizosphären fördern.
Unsere Gruppe entwarf eine 3D-gedruckte Version von Pilz-Autobahnsäulen, in denen Pilze Bakterien von einem Ende zum anderen transportieren können9. Diese Geräte bestehen aus vier gedruckten Komponenten: der Säule selbst mit Sanduhrform und einer komplexen inneren Gitterstruktur, einem Gewindering und zwei Kappen (einer großen und einer kleinen Kappe) sowie einem Stück sterilisiertem Nylonnetz (Abbildung 1). Die zusammengebaute Säule wird direkt auf das gewünschte Umgebungssubstrat aufgebracht. Die Säule ermöglicht es Mikroben dann, einen Agar-Wachstumsmedium-Pfropfen zu besiedeln, der als "Köder"-Medienpfropfen bekannt ist und sich am Boden der Säule befindet und durch das Netz mit dem Umgebungssubstrat in Kontakt steht. Dieses Stück Nylonmaschenweite schließt andere Bodenbewohner aus, die Bakterien transportieren können, wodurch die Bewegung der Bakterien innerhalb der Säulen auf Pilzautobahnen begrenzt wird. Sobald dieser Köderwopfen besiedelt ist, können sich filamentöse Pilze durch das innere Gitter in der Mitte der Säule ausdehnen und wachsen, das so konzipiert ist, dass ein ungesättigtes System entsteht, das Erde (oder anderen ungesättigten Medien) ähnelt und eine mögliche Kontamination durch das Ködermedium minimiert. Die Pilze wachsen dann auf den Zielmedium-Pfropfen am oberen Ende der Säule zu und besiedeln ihn. Die Säulen können entweder mit bestimmten Pilzisolaten beimpft werden, um ihre Fähigkeit zum Transport von Bakterien zu testen, oder sie können nicht geimpft werden, um zu identifizieren, welche Pilze aus dem Substrat in der Lage sind, Bakterien zu transportieren. Organismen, die das Zielmedium erreichen, können weiter kultiviert, isoliert und Sequenzierungsanalysen unterzogen werden (entweder aus Reinkulturen oder aus gemischten Gemeinschaften unter Verwendung von Amplikon- oder metagenomischen Sequenzierungsansätzen). Insgesamt bieten die Säulen eine standardisierte, reproduzierbare, wiederverwendbare und intuitive Methode zur Untersuchung von Pilzautobahnen in verschiedenen Substraten. Diese Geräte können für die Forschung und als Lehrmittel im Klassenzimmer verwendet werden, und hier bieten wir Unterrichtsschritte für ihre Verwendung an, die auf den in der Vergangenheit durchgeführten Experimenten basieren. Obwohl diese Methode die Standardisierung von Protokollen erleichtert, können das Design und die Konstruktion der Bauelemente für andere Anwendungen und zusätzliche Substrate modifiziert werden.
Protokoll
Die Einzelheiten zu den Reagenzien und der in der Studie verwendeten Ausrüstung sind in der Materialtabelle aufgeführt.
1. Ändern des Säulendesigns, der Materialien und Parameter
- Laden Sie die öffentlich zugänglichen Stützenkonstruktionen9 herunter und verwenden Sie sie unverändert oder ändern Sie die Stützenkonstruktionen in kompatibler CAD-Software (Computer-Aided Design).
- Besorgen Sie sich Harz für zahnchirurgische Schablonen oder wählen Sie ein alternatives 3D-Druckmaterial, z. B. andere lichtempfindliche klare Harze.
- Passen Sie die Spezifikationen der Spalte nach Bedarf an, wenn der ausgewählte 3D-Drucker, die gewählte 3D-Drucktechnologie oder das ausgewählte 3D-Druckmaterial gegenüber dem zuvor verwendeten Material geändert wird9.
2. 3D Bedrucken der Spalten
- Stellen Sie die Druckparameter so ein, dass eine Schichtdicke von 0,05 mm und eine Belichtungszeit von 0,8 s verwendet wird, oder passen Sie die Druckparameter je nach ausgewähltem Drucker, Druckmaterial und Drucksoftware an.
- Drucken Sie die Autobahnsäulen für Pilze, die Gewinderinge und die Kappen mit einem kompatiblen 3D-Drucker (Abbildung 1).

Abbildung 1: Komponenten der Spalte "Fungal Highway". (A,B) Draufsicht und Seitenansicht der Small Cap, des Gewinderings und der Large Cap (von links nach rechts). (C,D) Draufsicht und Seitenansicht des Small Caps. (E,F) Draufsicht und Seitenansicht des Gewinderings. (G,H) Draufsicht und Seitenansicht der großen Kappe. (I) Ein Nylonnetz-Filterstück (25 μm), das am Ende der Säule platziert und in das Umgebungssubstrat eingesetzt wird, um das Eindringen von Mikrofauna in die Säule zu verhindern. (J) Nicht zusammengesetzte Säule. (K) Zusammengebaute Säule: Das "Köder"-Ende geht in das Substrat, und das "Ziel"-Ende bleibt unbedeckt und außerhalb des Substrats. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
3. Reinigung der 3D-gedruckten Säulenkomponenten
- Tauchen Sie die bedruckten Säulen, Kappen und Gewinderinge 15 Minuten lang bei Raumtemperatur in 99%igen Isopropylalkohol und rühren Sie, indem Sie das Bad jede Minute 10-15 s lang von Hand hin und her bewegen, um überschüssiges Harz zu entfernen.
- Übertragen Sie die Komponenten in ein frisches 99%iges Isopropylalkoholbad. 5 min eintauchen und mit der Hand auf die gleiche Weise wie in Schritt 3.1 rühren.
- Übertragen Sie die Komponenten in ein mit reinem Wasser gefülltes Ultraschallreinigungsgerät, tauchen Sie die Komponenten ein und rühren Sie sie 2 Minuten lang auf mittlerer Stufe. Erhitzen Sie das Wasser nicht. Entfernen Sie die Komponenten.
- Trocknen Sie alle Komponenten mindestens 30 Minuten lang an der Luft.
- Um das Harz nachzuhärten, setzen Sie alle 3D-gedruckten Komponenten 30 Minuten lang 405 nm Licht bei 60 °C aus.
4. Sterilisieren der Säulen
- Wenn das gewählte Material autoklavierbar ist, autoklavieren Sie die Säulen, Gewinderinge, Kappen und Folien des 25-μm-Nylongewebefilters einzeln oder in einem größeren Becherglas für 20-30 min bei 121 °C, 1 atm.
HINWEIS: Komponenten der Säule können nach dem Autoklavieren Form und Farbe ändern, behalten jedoch die gewünschten Materialeigenschaften bei. Die endgültige Größe, Form und Farbe der Säulenkomponenten nach dem Autoklavieren sind in Abbildung 1 dargestellt.
5. Vorbereiten der Medien für die Spalten
- Bereiten Sie 90-mm-Petrischalen mit sterilisierten Medien auf Agarbasis vor: entweder Natriumcarboxymethylcellulosemedium (CMC), Malzextrakt-Agar (MEA), Kartoffel-Dextrose-Agar (PDA) oder Reasoner's 2A-Agar (R2A)9,10. Bereiten Sie das Medium gemäß den Anweisungen des Herstellers vor.
- Sterilisieren Sie das Medium, indem Sie es 21 Minuten lang oder die vom Hersteller empfohlene Zeit bei 121 °C und 1 atm autoklavieren und in 90-mm-Petrischalen gießen, bis sich der Agar nahe der Oberseite der Seiten der Petrischalen befindet (Abbildung 2). Führen Sie diesen Schritt in einer biologischen Sicherheitswerkbank durch, um die Sterilität zu erhöhen.
- Lassen Sie das Agar gemäß den Anweisungen des Herstellers fest werden und trocknen.

Abbildung 2: Montageprozess für die Pilz-Autobahnsäulen. Mit einem offenen Ende der Säule selbst wird ein Stopfen herausgeschnitten und eingeführt, und der Forscher dreht die Säule, während sie vom Medium entfernt wird, um sicherzustellen, dass der Stopfen am Ende der Säule bleibt. Dieses Ende wird mit dem kleinen Kappenstück verschlossen. Auf die gleiche Weise wird dann am anderen Ende der Spalte ein Medienstecker hinzugefügt. Über dieses Ende wird dann das Netzstück gelegt und mit dem Gewindering gesichert. Die große Kappe wird dann an diesem "Köder"-Ende über dem Gewindering verwendet. Die Seite mit dem Netz wird in das Umgebungssubstrat eingelegt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
6. Vorbereitung der Pilz-Autobahnsäulen
HINWEIS: Dieser Schritt ist in einer biologischen Sicherheitswerkbank durchzuführen, um die Sterilität der Säulenkomponenten und des Mediums zu erhalten. Abbildung 2 veranschaulicht den Assemblierungsprozess der Pilzautobahn-Säule.
- Verwenden Sie das Säulenende selbst (ohne Kappen), um einen Medienstopfen herauszuziehen, der fest in ein Ende der Säule passt. Verwenden Sie eine Drehbewegung, während die Säule vom Medium angehoben wird, so dass der Agar am Ende der Säule bleibt. Alternativ können Sie das Ende der Säule als Schablone verwenden, das Medium ausschneiden und mit einer Pipettenspitze in die Säule übertragen.
- Nachdem der erste Stopfen am Ende der Säule angebracht wurde, fügen Sie die kleine Kappe an diesem Ende hinzu, um eine sterile Mikroumgebung für das Zielmedium zu erhalten.
- Drehen Sie die Spalte um und wiederholen Sie Schritt 6.1 für das andere Ende der Spalte.
- Schneiden Sie mit einer sterilisierten Schere ein kreisförmiges Stück aus dem autoklavierten Nylonnetz mit einem Durchmesser von ~2 cm (25 μm Porengröße) ab (Abbildung 1). Legen Sie das Netz über das freiliegende Ende der Säule. Drehen Sie den Gewindering auf das Ende des Ködermediums der Säule, während Sie das Netz in den Fäden befestigen.
- Setzen Sie die große Kappe auf die Unterseite der Säule über das Netz und das andere Ende des Gewinderings und behalten Sie diese Kappe auf, wenn Sie die Säule lagern oder transportieren.
7. Vorinokulieren des Substrats oder des Ködermediums mit einem Pilz von Interesse
HINWEIS: Dieser Schritt ist optional.
- Imimpieren Sie einen Pilz von Interesse (z. B. einen Pilz, von dem bekannt ist, dass er Pilzautobahnen bildet) auf das gewünschte Substrat, indem Sie den Pilz zuerst auf einem festen Pilzwachstumsmedium (z. B. MEA, PDA; hergestellt wie in Schritt 5 beschrieben) in einer Petrischale züchten und einen kleinen (~1 cm) Abschnitt Agar mit sichtbarem Pilzwachstum auf das Substrat übertragen.
HINWEIS: In früheren Experimenten9 wurde 10 Tage lang vorkolonisiertes Mistsubstrat mit Coprinopsis cinerea verwendet, bevor die Säulen zum vorkolonisierten Substrat hinzugefügt wurden. - Anstatt das Substrat vorzubesiedeln, geben Sie einen Köderpilz in eine Petrischale oder direkt auf den Boden des Ködermediums innerhalb der Säule mit einer kleinen Menge Pilzmyzel (z. B. aus dem Durchzug einer sterilen Schlaufe).
- Warten Sie bei der Petrischale, bis ein großer Teil der Petrischale (etwa 50 % bis 75 % der bedeckten Platte) sichtbares Wachstum aufweist, und stanzen Sie dann den Ködermedienabschnitt direkt aus der kolonisierten Petrischale heraus, der dem äußeren Rand des Pilzbewuchses am nächsten liegt (wie in Schritt 6.1 beschrieben).
- Wenn Sie das Ködermedium direkt vorbesiedeln, warten Sie, bis das Ködermedium deutlich wächst, bevor Sie die Säule auf das Substrat setzen (dies wird wahrscheinlich mehrere Tage dauern).
HINWEIS: In früheren Experimenten9 wurden 14 Tage alte Medien verwendet, die mit C. cinerea als Ködermedium vorbesiedelt waren; Die Zeit vor der Besiedlung hängt von der Wachstumsrate des Pilzes, dem Medientyp und den Inkubationsbedingungen ab.
8. Vorbereitung der Kontrollbehandlungen und Wiederholungen
- Bereiten Sie Negativkontrollsäulen vor (wie in den Schritten 1-6 beschrieben) und inokulieren Sie sie nicht und legen Sie diese Säulen nicht in das Substrat. Verwenden Sie sie, um eine Grundlage für nachfolgende Analysen zu schaffen und eine eventuelle Kontamination im Vorbereitungsprozess zu beurteilen.
- Fügen Sie mindestens drei Replikate von Spalten für jedes Experiment hinzu.
9. Hinzufügen der Säule zum Substrat
- Richten Sie einen Labor-Mikrokosmos ein (z. B. Erde in einer Kiste, einem Topf oder einer Tube; Abbildung 3A) Oder bringen Sie die Spalten zu einer interessanten Feldwebsite.
- Entfernen Sie die große Kappe von der Unterseite der Säule, um den Gewindering und das Netz freizulegen.
- Fügen Sie die Spalte dem gewünschten Substrat hinzu (Abbildung 3), indem Sie die spezifischen Überlegungen für jeden unten beschriebenen Substrattyp befolgen. Falls erforderlich und um eine Beschädigung des Netzes zu minimieren, machen Sie mit einer behandschuhten Hand oder einem kleinen Werkzeug (z. B. einer kleinen Kelle) eine Vertiefung in das Substrat, bevor Sie die Säule in das Substrat einführen.
- Mist: Die Säulen wurden zuvor erfolgreich in frischem Pferdemist eingesetzt. Lagern Sie den Mist vor der Verwendung bei 4 °C und geben Sie ihn in Kammern, wie z. B. Magentaboxen, bevor Sie die Säulen hinzufügen. Setzen Sie die Säule in das Substrat ein, um 1-2 cm der Gesamtsäulenhöhe abzudecken.
- Boden: Setzen Sie die Säulen so ein, dass sich der gesamte untere Teil im Boden befindet (~1-2 cm Bodentiefe) (Abbildung 3B). Die Säulen können auf der Oberseite des Bodens aufliegen oder bis direkt unter die kleine Kappe auf der Zielseite eingegraben werden.
- Rhizosphäre: Führen Sie die Säulen wie in Schritt 9.3.2 beschrieben in den Boden ein, während Sie die Säule in der Nähe bestimmter Wurzeln anwinkeln und platzieren, um die Chancen zu erhöhen, Rhizosphären-Mikroorganismen einzufangen (Abbildung 3C).
- Substrate in Röhrchen: Geben Sie ~10 mL eines interessanten Substrats in ein konisches 50 mL-Röhrchen oder ähnliches und befeuchten Sie das Substrat bei Bedarf (z. B. in Umgebungen mit sehr niedriger Luftfeuchtigkeit) mit reinem Wasser. Führen Sie die Säule so in das Rohr ein, dass der Boden vergraben ist und keine Teile des Gewinderings oder -netzes sichtbar sind (Abbildung 3A). Schrauben Sie den Deckel des 50-ml-Röhrchens zu und legen Sie es in ein 50-ml-Röhrchengestell, um es aufrecht zu halten.
10. Säule im Substrat belassen
- Lassen Sie die Säule 3-21 Tage im Substrat.
HINWEIS: Die Zeit, die die Säulen in ihren Substraten verbleiben müssen, hängt von der Wachstumsrate der verwendeten Köderpilze, der Wachstumsrate der Pilze, die das Ködermedium besiedeln, und den Umweltbedingungen ab (z. B. führen trockene Bedingungen zu einer schnelleren Austrocknung des Mediums und können daher nicht so lange verwendet werden). - Legen Sie einen gewünschten Hell-Dunkel-Zyklus für das Experiment fest; Frühere Experimente haben die Säulen im Labor im Dunkeln gehalten, während die Säulen im Feld den lokalen Tag-/Nachtbedingungen ausgesetzt wurden9.
11. Entfernen der Säule vom Substrat
- Nachdem die Säule für die gewünschte Zeit mit dem Substrat in Kontakt war, entfernen Sie die Säule, schütteln Sie das überschüssige Substrat vorsichtig ab und setzen Sie die große Kappe wieder auf den Boden der Säule unter dem Netz, um die Säule zu transportieren.
- Legen Sie die Säule für den Transport in ein steriles Becherglas oder ein 50-ml-Röhrchen. Halten Sie die Säule während des Transports aufrecht.
12. Kultivierung von Isolaten aus dem Zielmedium der Säule
- Entfernen Sie in einer sterilen Umgebung, wie z. B. einer biologischen Sicherheitswerkbank, die große Kappe des Ködermediums, den Gewindering und das Netz. Verwerfen Sie das Netz.
HINWEIS: Wenn ein Forscher daran interessiert ist, welche Organismen das Ködermedium besiedelt haben, befolgen Sie die Schritte 12.3-12.4 mit dem Ködermedium-Plug zusätzlich zum Zielmedium-Plug. - Entfernen Sie die kleine Kappe vom Säulenende mit dem Zielmedium.
- Nehmen Sie den Stopfen des Zielmediums heraus, indem Sie die Säule umdrehen, damit der Zielstopfen aus der Säule fallen kann, oder indem Sie eine sterilisierte Pinzette oder Pipettenspitzen verwenden, um den Stopfen herauszuziehen. Platzieren Sie den Stopfen des Zielmediums direkt in der Mitte einer 90-mm-Petrischale, die mit einem Agarmedium Ihrer Wahl gefüllt ist (wie in Schritt 5 vorbereitet).
HINWEIS: Pilz- oder bakterienspezifische Medien können hier verwendet werden, um das Wachstum von Mitgliedern aus beiden Königreichen zu fördern; Der Stopfen kann auch mit einer sterilisierten Schere oder einer Pipettenspitze in zwei oder mehr Teile geteilt werden, was die Beschichtung auf mehreren Medientypen und/oder die Konservierung des Zielmediums für DNA-Extraktionen ermöglicht (siehe Schritt 14). - Die mit dem Zielmedium beimpfte Petrischale wird mindestens 72 h bei 25 °C im Dunkeln inkubiert.
13. Subkultivierung zur Isolierung von Mikroorganismen
HINWEIS: Dieser Schritt ist optional.
- Für Bakterien: Streichen Sie mit einer sterilen Impfschlaufe über eine einzelne Kolonie von Interesse und minimieren Sie dabei den Kontakt mit anderen Bereichen der Petrischale. Streifen Sie die Kolonie auf eine frische 90-mm-Petrischale, die das bevorzugte Bakterienmedium enthält, wie z. B. R2A (wie in Schritt 5 vorbereitet), mit einer Methode, die die Bildung einzelner Kolonien ermöglicht (z. B. Streifen in vier Zonen). Inkubieren Sie die Petrischale 24-72 Stunden lang bei 25-37 °C und überprüfen Sie das Koloniewachstum.
- Bei Pilzen: Mit einer sterilen Impfschlaufe, einem sterilen Rasierer oder einer sterilen Pipettenspitze einen minimalen (1 mm2) Abschnitt des Agars herausschneiden, der Hyphenwachstum enthält. Legen Sie das kleine Stück Agar auf eine frische 90-mm-Petrischale, die das bevorzugte Pilzmedium wie MEA oder PDA enthält, wie in Schritt 5 vorbereitet. Inkubieren Sie die Petrischale bis zu einer Woche lang bei 25 °C im Dunkeln und kontrollieren Sie sie täglich, um ein Überwachsen zu verhindern.
- Wiederholen Sie Schritt 1 oder Schritt 2 nach Bedarf, bis die Morphologien des Organismus einheitlich sind.
14. Extraktion von DNA aus der Platte oder direkt aus dem Zielmedium
- Frieren Sie die gesamten Agarpfropfen (Ziel und/oder Köder) oder die ausgewählten Stücke aus den Säulen in 1,5 mL Zentrifugenröhrchen bei -20 °C ein oder tauchen Sie Teile der Pfropfen vor der Extraktion in ein Konservierungsmittel in ein Konservierungsmittel in einem 1,5 mL Zentrifugenröhrchen, wenn die Extraktion nicht sofort erfolgt.
- Wenn eine Kultivierung aus dem Ziel- und/oder Ködermedium durchgeführt wurde und nachfolgende Subkultivierungsschritte unternommen wurden, stanzen oder schnitzen Sie ein ~1 cm großes Stück Agar mit Pilz- oder Bakterienwachstum aus (oder eine Mischung aus beidem, wenn keine nachfolgenden Isolierungsschritte unternommen wurden).
- Für die Extraktion isolierter Bakterienkolonien wischen Sie eine Kolonie mit einer sterilen Impfschlaufe von der Platte und schwenken Sie sie direkt in den Extraktionspuffer, der mit dem kommerziellen DNA-Extraktionskit verbunden ist (Schritt 14.4).
- Mahlen Sie die Agarstücke separat in flüssigem Stickstoff mit einem Mörser und Stößel und geben Sie das gemahlene Gewebe in Extraktionsröhrchen.
- Verwenden Sie ein kommerzielles DNA-Extraktionskit, das für Erde oder Bakterien und Pilze optimiert ist, und befolgen Sie die Anweisungen des Herstellers, um die DNA zu extrahieren (siehe Materialtabelle).
- Quantifizieren Sie die resultierende DNA mit einem Fluorometer oder einem vergleichbaren System.
15. Bewertung der mikrobiellen taxonomischen Diversität im Ziel- und/oder Ködermedium unter Verwendung von Amplikon- oder metagenomischen Sequenzierungsansätzen
- Führen Sie entweder eine Amplikon-Sequenzierung (16S und/oder interner transkribierter Spacer [ITS]) oder eine metagenomische Sequenzierung mit den folgenden Schritten durch:
- Amplikon-Sequenzierung: Amplifizieren Sie die V3-V4 16S rRNA-Genregion mit den Primern Bakt 341F (5′-CCT ACG GGN GGC WGC AG-3′) und Bakt 805R (5′-GAC TAC HVG GGT ATC TAA TCC-3′). Amplifizieren Sie die ITS2-Region des Pilzes mit den Primern ITS3, KYO2 (5′-GAT GAA, GAA CGY, AGY RAA-3′) und ITS4 (5′-TCC, TCC, GCT, TAT TGA, TAT GC-3′)9.
- Bereiten Sie Amplikonbibliotheken mit kommerziellen Library-Vorbereitungskits vor, die mit dem gewählten Sequenzer kompatibel sind. Sequenzieren Sie die Amplikons mit einem Short-Read-Sequenzer, um 150 oder 250 Basenpaar-Paired-End-Reads mit ausreichender Abdeckung zu erzeugen, indem Sie die Anweisungen des Herstellers der Sequenzierplattform für die Ladekonzentration befolgen.
- Metagenomische Sequenzierung: Erstellen Sie eine metagenomische Bibliothek aus der extrahierten DNA unter Verwendung eines kommerziell erhältlichen Vorbereitungskits für metagenomische Bibliotheken, das mit dem gewählten Sequenzierer kompatibel ist. Sequenzieren Sie die Metagenombibliothek mit einem Short-Read-Sequenzierer, der so eingestellt ist, dass 150 oder 250 Basenpaar-Paired-End-Reads mit ausreichender Abdeckung (~10-20 Gb pro Metagenom) generiert werden, indem Sie den Anweisungen des Herstellers der Sequenzierungsplattform für die Ladekonzentration folgen.
16. Analyse der Sequenzierungsdaten
17. Erstellung zusätzlicher Visualisierungen von Taxonomiedaten aus Amplikon- und/oder Metagenom-Ergebnissen
- Generieren Sie kreisförmige Kladogramme mit dem GraPhlAn (https://github.com/biobakery/graphlan)-Paket, indem Sie den Anweisungen in GitHub folgen.
HINWEIS: Taxonomie-Identifikatoren (https://www.ncbi.nlm.nih.gov/taxonomy) des National Center for Biotechnology Information (NCBI) können aus repräsentativen Sequenzzuweisungen abgerufen und an das "eftech"-Programm der Entrez Direct E-Utilities übergeben werden, um die von GraPhlAn16 geforderten Taxonomie-Rollup-Informationen zu sammeln.
18. Wiederverwendung der Spalten
- Wenn die Säulen noch montiert sind, schrauben Sie die Kappen, den Gewindering und das Netz ab und entfernen Sie sie. Verwerfen Sie das Netz. Entfernen Sie alle verbleibenden Agar-Pfropfen, waschen Sie die Säulen mit 99%igem Isopropylalkohol und gereinigtem Wasser und reinigen und trocknen Sie die Komponenten wie in den Schritten 3.1-3.4 beschrieben.
- Bereiten Sie neue Nylonnetze vor, die autoklaviert werden sollen.
- Autoklavieren Sie die Komponenten gemäß den Anweisungen in Schritt 4.1.
Ergebnisse
Die fertig montierte Pilzautobahnsäule ist ca. 5 cm lang (Abbildung 1). Die Säule sollte an keiner Stelle gebrochen sein, und die Kappen und der Gewindering sollten leicht und fest zusammenpassen, um Mikroumgebungen innerhalb der Säule zu schaffen. Das Filtergewebe kann über den Gewindering hinausragen (wie in Abbildung 1 und Abbildung 2 gezeigt) oder es kann mit einer sterilisierten Schere gekürzt werden. Die Agar-Stopfen sollten an jedem Ende der Säule eng anliegen. Beim Einsetzen in das Substrat sollte das Filtergewebe mit dem Substrat in Kontakt kommen und die Säule sollte nicht vollständig vergraben werden.
Säulen wurden zuvor in Pferdemist9 getestet. Die Säulen wurden auch in Schütt- und Rhizosphärenboden an einem Forschungsstandort sowie in kleine Bodenmengen in 50-ml-Röhrchen im Labor eingesetzt (Abbildung 3). Nachdem die Säulen der Pilzautobahn aus dem Substrat entfernt und zerlegt worden waren, war mikrobielles Wachstum sowohl auf dem Köder als auch auf den Stopfen des Zielmediums sichtbar (Beispiele in Abbildung 4A). Bakterien und Pilze wurden mittels Subkulturtechniken aus Ziel- und Ködermedien isoliert (Abbildung 4B), und die auf den Medienpfropfen vorhandenen Mikroben wurden taxonomisch mittels Amplikon-Sequenzierung identifiziert (Abbildung 4C,D). Abbildung 4C,D zeigt die kombinierten Ergebnisse der Amplikon-Sequenzierung über mehrere Experimente hinweg und zeigt, welche Mikroben in der Lage waren, den Zielmedienpfropfen aus Säulen zu erreichen, die dem Pferdemist9 hinzugefügt wurden. Visualisierungen dieser Bakterien- und Pilzdaten wurden generiert, wie in Schritt 17 beschrieben. Die Ergebnisse können auch als relative Abundanzen von Taxa dargestellt werden.
Suboptimale Ergebnisse wurden in Fällen erzielt, in denen die Säulen in Umgebungen mit extrem niedriger Luftfeuchtigkeit hinzugefügt wurden und die Medienstopfen innerhalb weniger Tage vollständig getrocknet wurden, was zu keiner Wiederherstellung besiedelter Mikroben führte (Abbildung 5A). Wir haben auch Fälle gesehen, in denen Mikroben einfach nicht aus dem Zielmedienstecker wachsen (Abbildung 5B), und Fälle, in denen wir nicht genügend Sequenzierungsdaten aus dem Zielmedienstecker für aussagekräftige Analysen gewinnen konnten. Andere Fälle, in denen Pilze aus den Säulen herauswachsen, haben ebenfalls dazu geführt, dass die Experimente wiederholt werden mussten (Abbildung 5C).

Abbildung 3: Beispiele für Säulen, die in Umweltproben in Labor- und Feldumgebungen platziert wurden. (A) Säule in einem 50-ml-Röhrchen mit angefeuchteter Erde in einer Laborumgebung. Auch mit einem Lineal für die Skalierung dargestellt. (B) Säule in den Boden auf dem Feld eingesetzt. (C) Säule, die in das Wurzelnetz einer Pflanze auf dem Feld eingesetzt wird. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
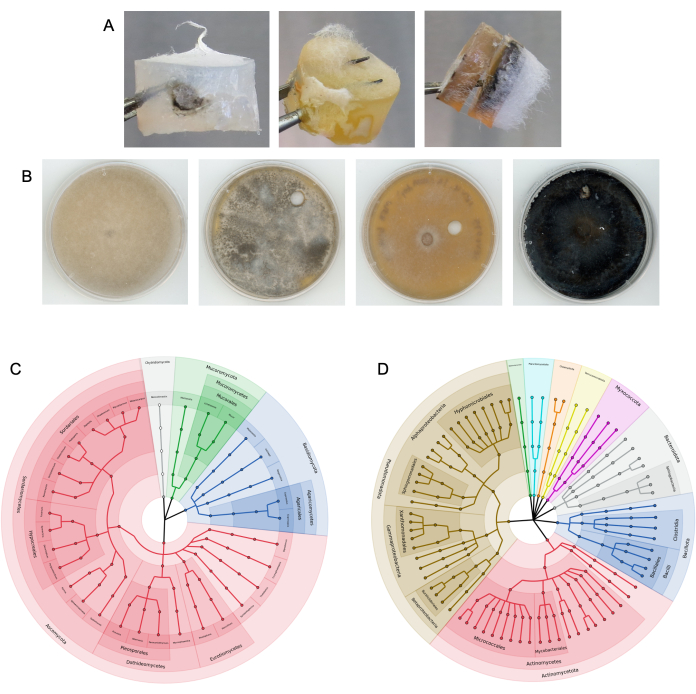
Abbildung 4: Repräsentative Ergebnisse erfolgreicher Säulenexperimente. (A) Beispiele für kolonisierte Medienstecker, die aus Säulen extrahiert wurden. (B) Beispiele für Pilzisolate, die aus Zielmedien subkultiviert wurden. Das Substrat war Erde. Top ITS-Sequenz NCBI BLAST-Identität von links nach rechts: Rhizopus azygosporous, Aspergillus novofumigatus, Curvularia subpapendorfii und Phaeomycocentrospora cantuariensis. (C,D) Zirkuläre Kladogramme, die die phylogenetische Vielfalt von (C) Pilz-ITS und (D) bakteriellen 16S-Sequenzen zeigen, die nach mehreren Pilzautobahnexperimenten mit Pferdemist aus dem Zielmedium gewonnen wurden. Die Abschnitte sind farbig und nach Stamm beschriftet, wobei die Endknoten einzigartige Gattungen repräsentieren. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
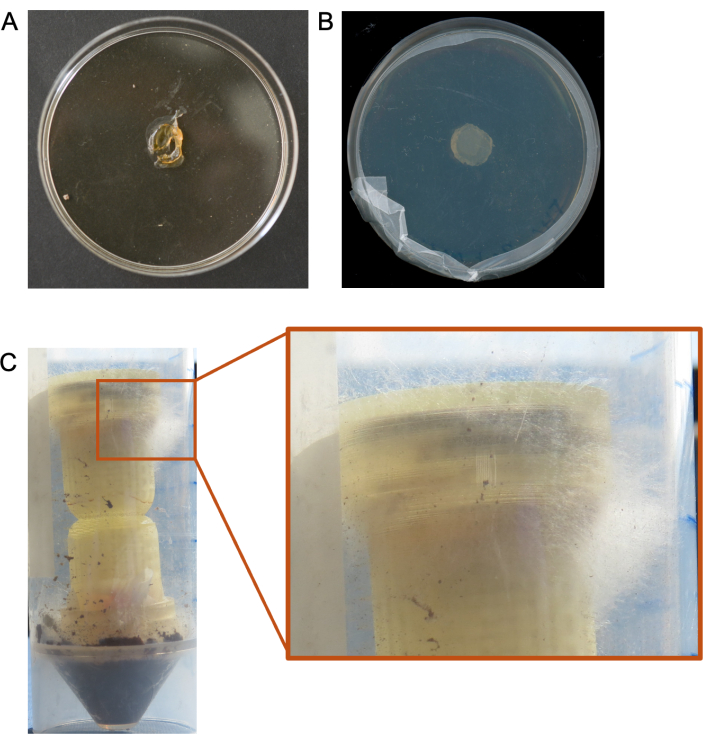
Abbildung 5. Suboptimale Ergebnisse aus Säulenexperimenten. (A) Ein Beispiel für einen ausgetrockneten Medienstopfen, der auf Umgebungsbedingungen mit niedriger Luftfeuchtigkeit zurückzuführen ist. (B) Beispiel für kein mikrobielles Wachstum aus einem Säulenmedienstopfen. (C) Beispiel für ein übermäßiges Wachstum des Pilzes durch die Oberseite (Zielmedium) der Säule. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Bei der Generierung der Säulenkomponenten kann die Auswahl eines 3D-Druckers und des Druckmaterials je nach Verfügbarkeit und gewünschten Materialeigenschaften modifiziert werden17,18. Die Biokompatibilität, die Oberflächenbeschaffenheit, die Autoklavierbarkeit, die Fähigkeit, feinskalige Details zu drucken, und die relative Transparenz wurden bei der Materialauswahl unserer Gruppe berücksichtigt. Andere Merkmale wie Porosität, Hydrophobie, Druckparameter usw. sollten ebenfalls berücksichtigt werden. Vor der endgültigen Auswahl wurden verschiedene Harze getestet (siehe Materialtabelle), und viele biokompatible Materialien eignen sich für den Druck dieser Säulen. Das Material, das für die Konstruktion der Säulenkomponenten gewählt wird, bestimmt, welche Reinigungs-, Nachhärtungs- und Sterilisationsansätze verwendet werden sollten. Nicht alle Materialien sind autoklavierbar, und je nach den Anweisungen des Materialherstellers können ultraviolettes Licht, Bleiche oder andere Sterilisationstechniken erforderlich sein. Einige Sterilisations- oder Reinigungstechniken können auch das gewählte Material beschädigen oder nicht mit ihm kompatibel sein, daher sollte diesen Informationen des Materialherstellers besondere Aufmerksamkeit geschenkt werden. Bei 3D-Druckern sind unter anderem die Druckzeit, die Materialkompatibilität, die Größe der Bauplattform, die Drucktechnologie und die Kosten19 zu berücksichtigen. Die 3D-gedruckten Komponenten der Säulen können zerbrechlich sein und bei zu starker Handhabung brechen. Das Gewinde für den Ring und die Kappen kann nicht immer genau übereinstimmen, daher empfehlen wir, zusätzliche Komponenten vor dem Montageschritt zu drucken und zu sterilisieren oder einen Vordruck durchzuführen, um zu testen, wie sich die gewählten Parameter und das gewählte Material auf das Gewindeschneiden auswirken. Die Konstruktionsspezifikationen für das Gewinde innerhalb der Kappen und des Rings müssen je nach gewähltem 3D-Druckmaterial möglicherweise angepasst werden. Die Abmessungen, die Komplexität des Gitters und andere physikalische Merkmale können vor dem Drucken in der CAD-Konstruktionssoftware geändert werden. Die Säule selbst ist 4 cm hoch, und die Gitterstruktur in der Mitte der Säule hat eine 2 mm große Elementarzelle, einen Strebendurchmesser von 0,5 mm und die gesamte Gitterhöhe beträgt 22 mm9. Diese Parameter können angepasst werden, wenn ein Forscher beispielsweise eine größere oder komplexere Gitterstruktur wünscht. Insgesamt ermöglicht die 3D-gedruckte Fertigung dieser Geräte Designflexibilität und stellt gleichzeitig sicher, dass ein einzelnes Design auf standardisierte Weise über Organisationen und Gruppen hinweg verwendet und sogar als Unterrichtsmittel im Klassenzimmer verwendet werden kann9.
Mehrere Schritte im Protokoll können je nach Umgebung oder Versuchsaufbau eine Fehlerbehebung erfordern. Die Autobahnsäulen von Pilzen sind bei niedriger Luftfeuchtigkeit nicht sehr effektiv, da die Medienstopfen schnell austrocknen, bevor sie das Pilzwachstum fördern, was die Dauer der Experimente in diesen Umgebungen begrenzen kann (Abbildung 5A). Zu den Techniken, die die Wirksamkeit der Säulen in Umgebungen mit niedriger Luftfeuchtigkeit verbessert haben, gehören die künstliche Erhöhung der Luftfeuchtigkeit durch Zugabe von Feuchtigkeit zum Substrat und/oder das Versiegeln der Säule und des Substrats in einem sekundären Behälter mit einer Wasserquelle (z. B. einem kleinen Behälter mit reinem Wasser). Die Sanduhrform und die Gitterstruktur wurden integriert, um die Bewegung von Bakterien allein (ohne die Einrichtung einer Pilzautobahn) zu verhindern, falls sich in Umgebungen mit hoher Luftfeuchtigkeit Kondenswasser bilden sollte. Schnell wachsende Pilze können die Oberfläche des Ziel- und Ködermediums überwuchern und sich aus dem oberen oder unteren Teil der Säule herausstrecken (Abbildung 5C). Durch die Verkürzung der Inkubationszeit des Köderpilzes oder der Dauer des Experiments kann diese Überwucherung minimiert oder beseitigt werden. Darüber hinaus besteht eine Einschränkung dieser Geräte darin, dass schnell wachsende Pilze im interessierenden Substrat die Besiedlung von Ködern und Zielmedien durch langsam wachsende Pilze einschränken können, was möglicherweise die beobachteten Wechselwirkungen mit der Autobahn beeinflusst. Einige Pilze, insbesondere langsamer wachsende Pilze, besiedeln das Ködermedium möglicherweise nicht so, dass sie durch den Agar-Pfropfen in die Gitterstruktur hineinwachsen können. Wenn genügend Feuchtigkeit in der Umgebung vorhanden ist, können dünnere Agar-Plugs verwendet werden, um das Wachstum in das Gitter nach der Besiedlung der Köder-Agar-Plugs zu fördern. Das Medium kann danach ausgewählt werden, ob ein Forscher nach Pilz- oder Bakterienwachstum selektieren möchte, aber dies kann die Subkultivierung auch auf Organismen beschränken, die diesen Medientypbevorzugen 20. Wenn im Zielmedium kein Wachstum zu sehen ist, kann es notwendig sein, das Ködermedium oder das Substrat mit einem Pilz zu beimpfen, von dem bekannt ist, dass er Pilzautobahnen bildet.
Im Rahmen dieser Experimente kann eine Metagenom- oder Amplikon-Sequenzierung durchgeführt werden, und beide Strategien vermitteln ihre eigenen Grenzen und Stärken21. Die metagenomische Sequenzierung ist ideal, um zusätzliche genomische Informationen über die Mikroben zu erhalten. Die rückgewinnbare Menge an Nukleinsäuren direkt aus dem Zielmedium kann jedoch sehr gering sein, was die Verwendung von Amplikon-Sequenzierung oder anderen Amplifikationsmethoden vor der Sequenzierung erforderlich machen kann. Amplikon-Sequenzierungsbibliotheken müssen separat erstellt werden (16S und ITS), und diese Methode hat keine taxonomische Auflösung und schränkt die Bewertung von Genommerkmalen oder funktionellem Potenzial ein, die mit metagenomischer Sequenzierung erreicht werden können. Direkte Sequenzierungsmethoden aus den Pfropfen können in Fällen bevorzugt werden, in denen Mikroben nicht subkultiviert werden können. Es wird empfohlen, die Plugs in mehrere Abschnitte aufzuteilen, um sowohl Kultivierungs- als auch Sequenzierungsansätze zu ermöglichen.
Ein Vorteil dieser Geräte ist, dass sie sowohl im Labor als auch im Feld eingesetzt werden können. Es muss besonders darauf geachtet werden, dass die Säulen auf dem Feld aufrecht stehen können und vor Tieren und Umwelteinflüssen geschützt sind, die ihre Platzierung stören könnten. Die Säulen wurden noch nicht in horizontaler Position getestet, d. h. in einer Position, in der sie vollständig von einem Substrat bedeckt sind, und sie wurden nicht in Umgebungen getestet, die starken Regenfällen oder Schnee ausgesetzt sind. Wie oben erwähnt, wurde die Gitterstruktur so konzipiert, dass die Wahrscheinlichkeit, dass Bakterien in Umgebungen mit hoher Luftfeuchtigkeit in das Zielmedium gelangen können, minimiert wird. Es ist jedoch möglich, dass, wenn die Säule größeren Wassermengen ausgesetzt wäre und dieses Wasser die Säule vollständig sättigt, die bakterielle Bewegung in der gesamten Säule unabhängig von vorhandenen Pilzautobahnen erleichtert würde. Für Laborexperimente können die Säulen in konischen 50-ml-Röhrchen, kleinen Mikrokosmen von Substraten, in der Erde um Topfpflanzen, in Boxen oder in anderen kontrollierten Versuchssystemen verwendet werden. Die Säulen wurden erfolgreich in Böden, Rhizosphären und Mist eingesetzt, und ihr Nutzen kann auf andere Substrate wie Laubstreu, Schlamm, Sand, Schnee, Kompost usw. ausgeweitet werden.
Die Spalten der Pilzautobahn ermöglichen eine Reihe von Vergleichen, um diesen BFI-Phänotyp innerhalb verschiedener Probentypen zu verstehen. Der Vergleich der Zusammensetzung der Gemeinschaft zwischen dem Köder und dem Zielmedium kann Aufschluss darüber geben, welche Bakterien Pilzautobahnen nutzen können und welche Pilze als potenzielle Autobahnen dienen können9. Wird die Metagenomsequenzierung eingesetzt, können auch genomische Merkmale untersucht werden, die Organismen vom Köder- vom Zielmedium unterscheiden. Es ist auch möglich, Zielmedien von Säulen zu vergleichen, die in unterschiedlichen Substraten (z. B. Erde versus Mist) oder unter verschiedenen Bedingungen (z. B. Temperatur oder Luftfeuchtigkeit) im selben Substrat platziert wurden. Insgesamt erweitern die Pilzautobahnsäulen die Fähigkeiten früherer Methoden zur Untersuchung dieser Form von BFI und ermöglichen umfangreiche Untersuchungen dieser Interaktionen, die die räumliche Dynamik komplexer Umweltmikrobiome prägen.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Diese Forschung wurde durch einen Science Focus Area Grant des U.S. Department of Energy (DOE), Biological and Environmental Research (BER), Biological System Science Division (BSSD) unter der Fördernummer LANLF59T unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 50 mL tubes | Greiner BIO-ONE | 5622-7261 | 50 mL tubes for performing column experiments in the lab |
| 90 mm Petri dishes | Thermo Scientific Nunc | 08-757-099 | Petri dishes for preparation of agar and for microbial growth |
| Asiga Freeform Pico Plus 39 digital light processing (DLP) 3D printer | Asiga Germany | Freeform Pico Plus 39 | 3D printer used to generate batches of the columns; other 3D printers can be used |
| Autoclave | Fisher Scientific | LS40F20 | Benchtop autoclave to sterilize the column components |
| Beaker | Fisher Scientific | FB100600 | 600 mL beaker for various uses throughout the protocol |
| Dental LT Clear Resin V2 | Formlabs | RS-F2-DLCL-02 | Alternative resin for 3D printing that was tested |
| Dental Surgical Guide Resin | Formlabs | RS-F2-SGAM-01 | Was used to generate the columns discussed in manuscript; Other photosensitive resins can be used in place of this material |
| DNA Low Bind 1.5 mL tubes | Eppendorf | 13-698-791 | Tubes used for various preparations including nucleic acid extractions |
| DNA/RNA shield preservative | Zymo Research | R1100-50 | Preservative used prior to nucleic acid extractions |
| EDGE Bioinformatics | Open source; Developed by the Los Alamos National Laboratory (LANL) | n/a | Bioinformatics platform for processing amplicon data |
| FastDNA spin kit for soil | MP Biomedicals LLC | 116560200-CF | DNA extraction kit option for soil |
| Forceps | Fisher Scientific | 10-300 | Forceps that can be sterilized |
| Formlabs BioMed Clear Resin | Formlabs | RS-F2-BMCL-01 | Alternative resin for 3D printing that was tested |
| Formlabs Form 3B+ stereolithography (SLA) 3D printer | Formlabs | Form 3B+ | Alternative 3D printer |
| Formlabs IBT Resin | Formlabs | RS-F2-IBCL-01 | Alternative resin for 3D printing that was tested |
| Inoculating Loops | Fisher Scientific | 22-363-598 | Used to isolate/transfer microbes |
| Malt Extract Agar (MEA) | Criterion | 89405-654 | A media type used in columns |
| MiSeq sequencer + MiSeq sequencing kit | Illumina | SY-410-1003 | Can use other sequencers |
| Mortar & Pestle | Fisher Scientific | FB961K; FB961A | Can use any common mortar & pestle that can be sterilized between uses |
| NEBNext Ultra II DNA Library Prep Kit for Illumina | New England Biolabs | E7805S | Library prep kit for metagenomic sequencing |
| Nextera XT DNA Library Preparation Kit (24 samples) | Illumina | FC-131-1024 | Library prep kit for amplicon sequencing |
| NMDC EDGE | Open source: Developed by the National Microbiome Data Collaborative (NMDC) | n/a | Bioinformatics platform for processing metagenomic data |
| Nylon mesh | Sefar | 03-25/19 | The mesh used as part of the column construction |
| Pipette tips | Rainin | 30807966 | Can use many different sterilized pipette tips for the protocol steps |
| Potato Dextrose Agar | Cole Parmer | EW-14200-28 | A media type used in columns |
| QIIME2 | Open source | n/a | Software for processing amplicon data |
| Qubit dsDNA HS assay kit | Thermo Fisher Scientific | Q32851 | Used to quantify DNA after extractions |
| Qubit Fluorometer | Thermo Fisher Scientific | Q33238 | Used to quantify DNA after extractions |
| Quick-DNA Fungal/Bacterial Miniprep Kit | Zymo Research | D6005 | DNA extraction kit option that works with both bacteria and fungi |
| R2A agar | BD Difco | 218263 | A media type used in columns (bacterial media) |
| Rack for 50 mL tubes | Fisher Scientific | 03-448-11 | Rack to hold 50 mL tubes upright |
| Scissors | Fisher Scientific | 12-000-155 | Fine precision scissors that can be sterilized |
| Sodium carboxymethyl cellulose medium | Aldrich | 419273-100G | A media type used in columns |
| SolidWorks CAD software | SolidWorks | n/a | Software used to design the columns |
| Trowel scoop | Fisher Scientific | S41701 | To make a depression in the substrate prior to adding the column |
| UltraPure DNase/RNase-Free Distilled Water | Invitrogen: ThermoFisher Scientific | 10977015 | Water for the ultrasonicator water bath |
| Ultrasonicator | Fisher Scientific | FB-11201 | Ultrasonicator for cleaning the columns |
Referenzen
- Or, D., Smets, B. F., Wraith, J. M., Dechesne, A., Friedman, S. P. Physical constraints affecting bacterial habitats and activity in unsaturated porous media - A review. Adv Water Resour. 30 (6), 1505-1527 (2007).
- Kohlmeier, S., et al. Taking the fungal highway: Mobilization of pollutant-degrading bacteria by fungi. Environ Sci Technol. 39 (12), 4640-4646 (2005).
- Simon, A., Hervé, V., Al-Dourobi, A., Verrecchia, E., Junier, P. An in situ inventory of fungi and their associated migrating bacteria in forest soils using fungal highway columns. FEMS Microbiol Ecol. 93 (1), 217 (2017).
- Wick, L. Y., et al. Effect of fungal hyphae on the access of bacteria to phenanthrene in soil. Environ Sci Technol. 41 (2), 500-505 (2007).
- Bravo, D., et al. Isolation of oxalotrophic bacteria able to disperse on fungal mycelium. FEMS Microbiol Lett. 348 (2), 157-166 (2013).
- Furuno, S., Remer, R., Chatzinotas, A., Harms, H., Wick, L. Y. Use of mycelia as paths for the isolation of contaminant-degrading bacteria from soil. Microb Biotechnol. 5 (1), 142-148 (2012).
- Buffi, M., et al. Fungal drops: A novel approach for macro- and microscopic analyses of fungal mycelial growth. Microlife. 4, 042 (2023).
- Kuhn, T., et al. Design and construction of 3D printed devices to investigate active and passive bacterial dispersal on hydrated surfaces. BMC Biol. 20 (1), 203 (2022).
- Junier, P., et al. Democratization of fungal highway columns as a tool to investigate bacteria associated with soil fungi. FEMS Microbiol Ecol. 97 (2), 003 (2021).
- Reasoner, D. J., Geldreich, E. E. A new medium for the enumeration and subculture of bacteria from potable water. Appl Environ Microbiol. 49 (1), 1-7 (1985).
- Bolyen, E., et al. Reproducible, interactive, scalable, and extensible microbiome data science using QIIME 2. Nat Biotechnol. 37 (8), 852-857 (2019).
- Callahan, B. J., et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nat Methods. 13 (7), 581-583 (2016).
- Vázquez-Baeza, Y., Pirrung, M., Gonzalez, A., Knight, R. EMPeror: A tool for visualizing high-throughput microbial community data. Gigascience. 2 (1), 16 (2013).
- Li, P. -. E., et al. Enabling the democratization of the genomics revolution with a fully integrated web-based bioinformatics platform. Nucleic Acids Res. 45 (1), 67-80 (2017).
- Eloe-Fadrosh, E. A., et al. The National Microbiome Data Collaborative Data Portal: An integrated multi-omics microbiome data resource. Nucleic Acids Res. 50 (1), D828-D836 (2022).
- Entrez Direct: E-utilities on the Unix Command Line. Entrez Programming Utilities Help Available from: https://www.ncbi.nlm.nih.gov/books/NBK179288/ (2024)
- Palmara, G., Frascella, F., Roppolo, I., Chiappone, A., Chiadò, A. Functional 3D printing: Approaches and bioapplications. Biosens Bioelectron. 175, 112849 (2021).
- Guttridge, C., Shannon, A., O'Sullivan, A., O'Sullivan, K. J., O'Sullivan, L. W. Biocompatible 3D printing resins for medical applications: A review of marketed intended use, biocompatibility certification, and post-processing guidance. Ann 3D Print Med. 5, 100044 (2022).
- Yao, L., et al. Comparison of accuracy and precision of various types of photo-curing printing technology. J Phys Conf Ser. 1549 (3), 032151 (2020).
- Basu, S., et al. Evolution of bacterial and fungal growth media. Bioinformation. 11 (4), 182-184 (2015).
- Liu, Y. -. X., et al. A practical guide to amplicon and metagenomic analysis of microbiome data. Protein Cell. 12 (5), 315-330 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten