Method Article
Investigando interações bactérias-fungos usando colunas de rodovias fúngicas em diversos ambientes e substratos
Neste Artigo
Resumo
Este protocolo fornece instruções detalhadas sobre como construir, esterilizar, montar, utilizar e reutilizar as colunas de rodovias fúngicas para enriquecer pares de bactérias-fungos que interagem por meio de rodovias fúngicas de diversos substratos ambientais.
Resumo
As interações bactérias-fungos (BFIs) desempenham um papel fundamental na formação da composição da comunidade microbiana, funções biogeoquímicas, dinâmica espacial e dispersão microbiana. As redes miceliais criadas por fungos filamentosos ou outros microrganismos filamentosos (por exemplo, oomicetos) atuam como 'rodovias fúngicas' que podem ser utilizadas por bactérias para transporte em ambientes heterogêneos, facilitando muito sua mobilidade e concedendo-lhes acesso a regiões que podem ser desafiadoras ou impossíveis de alcançar por conta própria (por exemplo, devido a bolsas de ar no solo). Vários dispositivos e protocolos experimentais foram criados para estudar essas rodovias fúngicas, incluindo colunas de rodovias fúngicas. A coluna de rodovia fúngica projetada por nosso grupo pode ser usada para uma variedade de aplicações in situ ou in vitro , bem como com diversos tipos de amostras ambientais e associadas ao hospedeiro. Aqui, descrevemos os métodos para realizar experimentos com essas colunas, incluindo projetar, imprimir, esterilizar e preparar os dispositivos. As opções para analisar os dados obtidos com o uso desses dispositivos também são discutidas aqui, e são oferecidos conselhos de solução de problemas sobre possíveis armadilhas associadas a experimentos usando colunas de rodovias fúngicas. Esses dispositivos podem ser usados para obter uma compreensão mais abrangente da diversidade, mecanismos e dinâmica dos BFIs de rodovias fúngicas para fornecer informações valiosas sobre a dinâmica estrutural e funcional em ambientes complexos (por exemplo, solos) e em diversos habitats nos quais bactérias e fungos coexistem.
Introdução
As interações bactérias-fungos (BFIs) são extremamente importantes na formação das propriedades estruturais, espaciais e funcionais dos microbiomas ambientais. Por exemplo, o crescimento e a expansão de fungos filamentosos ou outros microrganismos filamentosos semelhantes a fungos geram uma rede biológica que pode funcionar como uma 'rodovia' para facilitar o movimento de outros microrganismos, como bactérias. A heterogeneidade e a saturação inconsistente dentro dos substratos ambientais podem dificultar a motilidade bacteriana; no entanto, as bactérias podem usar essas rodovias para facilitar o acesso a áreas adicionais do ambiente 1,2. Essas interações são críticas para entender a dinâmica espacial das comunidades microbianas. Várias técnicas e métodos têm sido usados para examinar rodovias fúngicas, no entanto, eles são amplamente limitados a investigações laboratoriais 3,4.
Em um método baseado em placa, uma grande seção de ágar é removida do meio da placa de Petri, criando uma lacuna entre duas ilhas de ágar. As hifas fúngicas podem atravessar essa lacuna, fornecendo os meios para bactérias compatíveis cruzarem de uma ilha de ágar para outra5. Outros métodos modificados de placa de Petri incluem placas invertidas onde o solo é colocado na tampa para que as hifas fúngicas possam crescer verticalmente e colonizar o meio sem contato direto, fornecendo os meios para o transporte bacteriano 5,6. Um método baseado em gotículas de meio de crescimento que foi desenvolvido recentemente pode ser usado para avaliar o transporte seletivo de hifas de bactérias para certos perfis nutricionais7. Dispositivos de ponte bacteriana e trilha também têm sido usados para investigar o efeito de fatores abióticos no movimento bacteriano8. Embora vários métodos e técnicas tenham sido utilizados para investigar rodovias fúngicas, ainda há necessidade de dispositivos padronizados que mantenham um microambiente estéril enquanto promovem o estabelecimento de rodovias fúngicas a partir de substratos ambientais complexos, como esterco, solo e rizosferas.
Nosso grupo projetou uma versão impressa em 3D de colunas de rodovias fúngicas onde os fungos podem transportar bactérias de uma ponta a outra9. Esses dispositivos são montados a partir de quatro componentes impressos: a própria coluna com formato de ampulheta e uma complexa estrutura de treliça interna, um anel rosqueado e duas tampas (uma tampa grande e uma pequena tampa), além de um pedaço de malha de náilon esterilizada (Figura 1). A coluna montada é adicionada diretamente ao substrato ambiental desejado. A coluna então permite que os micróbios colonizem um tampão de meio de crescimento de ágar conhecido como tampão de mídia de 'isca' que está na parte inferior da coluna e em contato com o substrato ambiental através da malha. Este pedaço de malha de nylon exclui outros habitantes do solo que podem transportar bactérias, limitando assim o movimento bacteriano dentro das colunas para rodovias fúngicas. Uma vez que esse tampão de isca tenha sido colonizado, os fungos filamentosos podem se estender e crescer através da rede interna dentro do centro da coluna que é projetada para criar um sistema insaturado que se assemelha ao solo (ou outro meio insaturado) e minimizar a contaminação potencial do meio de isca. Os fungos então crescem e colonizam o tampão médio alvo no topo da coluna. As colunas podem ser inoculadas com isolados de fungos específicos para testar sua capacidade de transportar bactérias ou podem ser deixadas sem inoculação para identificar quais fungos do substrato são capazes de transportar bactérias. Os organismos que atingem o meio alvo podem ser cultivados, isolados e submetidos a análises de sequenciamento (seja de culturas puras ou de comunidades mistas usando abordagens de sequenciamento amplicon ou metagenômico). No geral, as colunas fornecem um método padronizado, reprodutível, reutilizável e intuitivo para interrogar rodovias fúngicas em diversos substratos. Esses dispositivos podem ser usados para pesquisa e como ferramenta de ensino em sala de aula e, aqui, fornecemos etapas instrucionais para usá-los com base nos experimentos que foram realizados no passado. Embora esse método facilite a padronização do protocolo, o projeto e a construção dos dispositivos podem ser modificados para outras aplicações e substratos adicionais.
Protocolo
Os detalhes dos reagentes e do equipamento utilizado no estudo estão listados na Tabela de Materiais.
1. Modificando o design da coluna, materiais e parâmetros
- Baixe os projetos de colunas disponíveis publicamente9 e use-os como estão ou modifique os projetos de colunas em software CAD (Computer-Aided Design) compatível.
- Obtenha resina de guia cirúrgico odontológico ou selecione um material de impressão 3D alternativo, como outras resinas transparentes fotossensíveis.
- Ajuste as especificações da coluna conforme necessário se a impressora 3D escolhida, a tecnologia de impressão 3D ou o material de impressão 3D forem alterados em relação ao que era usado anteriormente9.
2. 3D impressão das colunas
- Defina os parâmetros de impressão para usar uma espessura de fatia de 0,05 mm e um tempo de exposição de 0,8 s ou ajuste os parâmetros de impressão dependendo da impressora, do material de impressão e do software de impressão selecionados.
- Imprima as colunas de rodovias fúngicas, os anéis roscados e as tampas usando uma impressora 3D compatível (Figura 1).

Figura 1: Componentes da coluna da rodovia fúngica. (A, B) Vista superior e lateral da tampa pequena, anel rosqueado e tampa grande (da esquerda para a direita). (C, D) Vista superior e lateral da pequena tampa. (E, F) Vista superior e lateral do anel roscado. (G, H) Vista superior e lateral da grande tampa. (I) Uma peça de filtro de malha de nylon (25 μm) colocada na extremidade da coluna e inserida no substrato ambiental para evitar a entrada da microfauna na coluna. (J) Coluna desmontada. (K) Coluna montada: a extremidade 'Bait' vai para o substrato e a extremidade 'Target' permanece descoberta e fora do substrato. Clique aqui para ver uma versão maior desta figura.
3. Limpeza dos componentes da coluna impressa em 3D
- Mergulhe as colunas, tampas e anéis roscados impressos em álcool isopropílico 99% em temperatura ambiente por 15 min e agite movendo o banho para frente e para trás com a mão por 10-15 s a cada minuto para remover o excesso de resina.
- Transfira os componentes para um banho novo de álcool isopropílico a 99%. Mergulhe por 5 min e agite manualmente da mesma maneira que na etapa 3.1.
- Transfira os componentes para um dispositivo de limpeza ultrassônico cheio de água pura, mergulhe os componentes e agite por 2 min em uma configuração de velocidade média. Não aqueça a água. Remova os componentes.
- Seque ao ar todos os componentes por pelo menos 30 min.
- Para realizar a pós-cura da resina, exponha todos os componentes impressos em 3D à luz de 405 nm por 30 min a 60 °C.
4. Esterilização das colunas
- Se o material escolhido for autoclavável, autoclave as colunas, anéis roscados, tampas e folhas de filtro de malha de nylon de 25 μm individualmente ou dentro de um béquer maior por 20-30 min a 121 °C, 1 atm.
NOTA: Os componentes da coluna podem mudar de forma e cor após a autoclavagem, mas manterão as propriedades desejadas do material. O tamanho, a forma e a cor finais dos componentes da coluna após a autoclavagem são mostrados na Figura 1.
5. Preparando mídia para as colunas
- Prepare placas de Petri de 90 mm de meio esterilizado à base de ágar: meio carboximetilcelulose de sódio (CMC), ágar extrato de malte (MEA), ágar batata dextrose (PDA) ou ágar 2A de Reasoner (R2A) 9 , 10 . Prepare a mídia seguindo as instruções do fabricante.
- Esterilize o meio em autoclave por 21 min ou o tempo recomendado pelo fabricante a 121 ° C, 1 atm, e despeje em placas de Petri de 90 mm até que o ágar esteja próximo ao topo das laterais das placas de Petri (Figura 2). Execute esta etapa em um gabinete de segurança biológica para aumentar a esterilidade.
- Deixe o ágar-ágar solidificar e secar com base nas instruções do fabricante.

Figura 2: Processo de montagem das colunas de fungos da rodovia. Usando uma extremidade aberta da própria coluna, um plugue é cortado e inserido, e o pesquisador torce a coluna à medida que é removida da mídia para garantir que o plugue permaneça dentro da extremidade da coluna. Essa extremidade é tampada com a pequena peça de tampa. Um plugue de mídia é então adicionado à outra extremidade da coluna da mesma maneira. A peça de malha é então colocada sobre esta extremidade e fixada com o anel roscado. A tampa grande é então usada nesta extremidade 'Bait' sobre o anel rosqueado. O lado com a malha será colocado no substrato ambiental. Clique aqui para ver uma versão maior desta figura.
6. Preparando as colunas de rodovias fúngicas
NOTA: Esta etapa deve ser realizada em um gabinete de segurança biológica para manter a esterilidade dos componentes da coluna e do meio. A Figura 2 ilustra o processo de montagem da coluna de fungos da rodovia.
- Use a própria extremidade da coluna (sem tampas) para extrair um plugue de mídia que se encaixe firmemente em uma extremidade da coluna. Use um movimento de torção enquanto a coluna é levantada da mídia para que o ágar permaneça dentro da extremidade da coluna. Como alternativa, use o final da coluna como modelo, corte a mídia e transfira-a para a coluna usando uma ponta de pipeta.
- Depois que o primeiro plugue for adicionado ao final da coluna, adicione a tampa pequena a essa extremidade para manter um microambiente estéril para o meio de destino.
- Vire a coluna e repita a etapa 6.1 para a outra extremidade da coluna.
- Corte um pedaço circular de ~ 2 cm de diâmetro da malha de náilon autoclavada (tamanho de poro de 25 μm) usando uma tesoura esterilizada (Figura 1). Coloque a malha sobre a extremidade exposta da coluna. Torça o anel rosqueado nesta extremidade da coluna enquanto prende a malha dentro das roscas.
- Coloque a tampa grande na parte inferior da coluna sobre a malha e a outra extremidade do anel rosqueado e mantenha essa tampa ao armazenar ou transportar a coluna.
7. Pré-inoculação do substrato ou meio de isca com um fungo de interesse
NOTA: Esta etapa é opcional.
- Inocule um fungo de interesse (por exemplo, um fungo que é conhecido por criar rodovias fúngicas) no substrato desejado, primeiro cultivando o fungo em um meio sólido de crescimento fúngico (por exemplo, MEA, PDA; preparado conforme descrito na etapa 5) dentro de uma placa de Petri e transferindo uma pequena seção (~ 1 cm) de ágar com crescimento fúngico visível para o substrato.
NOTA: Experimentos anteriores9 usaram substrato de esterco pré-colonizado com Coprinopsis cinerea por 10 dias antes de adicionar as colunas ao substrato pré-colonizado. - Em vez de pré-colonizar o substrato, adicione um fungo de isca a uma placa de Petri ou diretamente ao fundo do meio de isca dentro da coluna usando uma pequena quantidade de micélios fúngicos (por exemplo, do toque de um laço estéril).
- Para a placa de Petri, espere até que haja crescimento visível cobrindo uma grande parte da placa de Petri (cerca de 50% -75% da placa coberta) e, em seguida, retire a seção do meio de isca diretamente da placa de Petri colonizada mais próxima da borda externa do crescimento fúngico (conforme descrito na etapa 6.1).
- Ao pré-colonizar diretamente o meio de isca, espere até que haja um crescimento claro em todo o meio de isca antes de adicionar a coluna ao substrato (isso provavelmente levará vários dias).
NOTA: Experimentos anteriores9 usaram meios de 14 dias pré-colonizados com C. cinerea como meio de isca; Os tempos de pré-colonização variam dependendo da taxa de crescimento do fungo, tipo de meio e condições de incubação.
8. Preparação dos tratamentos de controlo e dos replicados
- Prepare colunas de controle negativo (conforme descrito nas etapas 1 a 6) e não as inocule nem coloque essas colunas no substrato. Use-os para fornecer uma linha de base para análises subsequentes e para avaliar qualquer contaminação no processo de preparação.
- Inclua pelo menos três réplicas de colunas para qualquer experimento.
9. Adicionando a coluna ao substrato
- Configure um microcosmo de laboratório (por exemplo, solo dentro de uma caixa, vaso ou tubo; Figura 3A) ou traga as colunas para um local de campo de interesse.
- Remova a tampa grande da parte inferior da coluna para expor o anel rosqueado e a malha.
- Adicione a coluna ao substrato de interesse (Figura 3) seguindo as considerações específicas para cada tipo de substrato descrito abaixo. Se necessário, e para minimizar os danos à malha, faça uma depressão no substrato usando uma mão enluvada ou uma pequena ferramenta (por exemplo, uma pequena espátula) antes de inserir a coluna no substrato.
- Esterco: As colunas foram usadas anteriormente com sucesso em esterco de cavalo fresco. Conservar o estrume a 4 °C antes da utilização e adicionar em câmaras, como caixas magentas, antes de adicionar as colunas. Insira a coluna no substrato para cobrir 1-2 cm da altura total da coluna.
- Solo: Insira as colunas de forma que toda a seção inferior fique dentro do solo (~ 1-2 cm de profundidade do solo) (Figura 3B). As colunas podem repousar no topo do solo ou ser enterradas até logo abaixo da pequena tampa no lado alvo.
- Rizosfera: Insira as colunas no solo ao redor das raízes das plantas conforme instruído na etapa 9.3.2 enquanto inclina e coloca a coluna perto de certas raízes para aumentar as chances de capturar microrganismos da rizosfera (Figura 3C).
- Substratos dentro de tubos: Adicione ~ 10 mL de um substrato de interesse a um tubo cônico de 50 mL ou similar e molhe o substrato, se desejar (por exemplo, em ambientes de umidade muito baixa) usando água pura. Insira a coluna no tubo de forma que o fundo fique enterrado e nenhuma parte do anel ou malha rosqueada fique visível (Figura 3A). Feche a tampa do tubo de 50 mL e coloque-o em um suporte para tubos de 50 mL para mantê-lo na posição vertical.
10. Deixando a coluna no substrato
- Deixe a coluna no substrato por 3-21 dias.
NOTA: A quantidade de tempo que as colunas precisam ser deixadas em seus substratos depende da taxa de crescimento de qualquer fungo de isca usado, da taxa de crescimento dos fungos que colonizam o meio de isca e das condições ambientais (por exemplo, condições secas levam a uma dessecação mais rápida do meio, portanto, não podem ser usadas por tanto tempo). - Estabeleça um ciclo claro/escuro desejado para o experimento; Experimentos anteriores mantiveram as colunas dentro do laboratório no escuro, enquanto as colunas no campo foram submetidas às condições locais de dia / noite9.
11. Remoção da coluna do substrato
- Depois que a coluna estiver em contato com o substrato pelo tempo desejado, remova a coluna, sacuda cuidadosamente qualquer excesso de substrato e adicione a tampa grande de volta na parte inferior da coluna abaixo da malha para transportar a coluna.
- Coloque a coluna em um béquer estéril ou tubo de 50 mL para transporte. Mantenha a coluna na posição vertical durante o transporte.
12. Cultura de isolados do meio alvo da coluna
- Em um ambiente estéril, como um armário de segurança biológica, remova a tampa grande da mídia de isca, o anel rosqueado e a malha. Descarte a malha.
NOTA: Se um pesquisador estiver interessado em quais organismos colonizaram o meio de isca, siga as etapas 12.3-12.4 com o plugue do meio de isca, além do plugue do meio alvo. - Remova a tampa pequena da extremidade da coluna que contém o meio alvo.
- Retire o tampão médio alvo virando a coluna para permitir que o tampão alvo caia da coluna ou usando uma pinça esterilizada ou pontas de pipeta para extrair o plugue. Coloque o tampão do meio alvo diretamente no centro de uma placa de Petri de 90 mm cheia de um meio de ágar de sua escolha (conforme preparado na etapa 5).
NOTA: Meios específicos para fungos ou bactérias podem ser usados aqui para aumentar o crescimento de membros de qualquer reino; o plugue também pode ser dividido em duas ou mais partes usando uma tesoura esterilizada ou uma ponta de pipeta, permitindo o revestimento em vários tipos de mídia e/ou preservando a mídia alvo para extrações de DNA (consulte a etapa 14). - Incubar a placa de Petri inoculada com o tampão do meio-alvo durante, pelo menos, 72 h a 25 °C na obscuridade.
13. Subcultura para isolar microrganismos
NOTA: Esta etapa é opcional.
- Para bactérias: Usando uma alça de inoculação estéril, passe por uma única colônia de interesse, minimizando o contato com outras áreas da placa de Petri. Listre a colônia em uma placa de Petri fresca de 90 mm contendo o meio bacteriano preferido, como R2A (conforme preparado na etapa 5), usando qualquer método que permita a formação de colônias únicas (por exemplo, listras em quatro zonas). Incubar a placa de Petri durante 24-72 h a 25-37 °C, verificando o crescimento das colónias.
- Para fungos: Usando uma alça de inoculação estéril, lâmina esterilizada ou ponta de pipeta estéril, corte uma seção mínima (1 mm2) de ágar contendo crescimento de hifas. Coloque o pequeno pedaço de ágar em uma placa de Petri fresca de 90 mm contendo o meio fúngico preferido, como MEA ou PDA, conforme preparado na etapa 5. Incubar a placa de Petri por até uma semana a 25 °C no escuro, verificando diariamente para evitar o crescimento excessivo.
- Repita a etapa 1 ou a etapa 2 conforme necessário até que as morfologias dos organismos sejam uniformes.
14. Extração de DNA da placa ou diretamente do meio alvo
- Congele todos os tampões de ágar (alvo e/ou isca) ou as peças selecionadas das colunas em tubos de centrífuga de 1,5 mL a -20 °C ou mergulhe os pedaços dos tampões em um conservante em um tubo de centrífuga de 1,5 mL antes da extração, se as extrações não ocorrerem imediatamente.
- Se a cultura do alvo e / ou meio de isca foi realizada, e se as etapas subsequentes de subcultura foram tomadas, carimbe ou corte um pedaço de ágar de ~ 1 cm com crescimento fúngico ou bacteriano (ou uma mistura de ambos se as etapas de isolamento subsequentes não foram tomadas).
- Para extrações de colônias bacterianas isoladas, passe uma colônia da placa usando uma alça de inoculação estéril e gire diretamente no tampão de extração associado ao kit comercial de extração de DNA (etapa 14.4).
- Moer os pedaços de ágar separadamente em nitrogênio líquido usando um almofariz e pilão e transferir o tecido moído para tubos de extração.
- Use um kit comercial de extração de DNA otimizado para solo ou bactérias e fungos e siga as instruções do fabricante para extrair o DNA (consulte a Tabela de Materiais).
- Quantifique o DNA resultante usando um fluorômetro ou sistema comparável.
15. Avaliação da diversidade taxonômica microbiana no meio alvo e/ou isca usando abordagens de sequenciamento amplicon ou metagenômico
- Execute o sequenciamento de amplicon (16S e/ou espaçador interno transcrito [ITS]) ou o sequenciamento metagenômico seguindo as etapas abaixo:
- Sequenciamento de amplicon: Amplificar a região do gene V3-V4 16S rRNA usando os primers Bakt 341F (5′-CCT ACG GGN GGC WGC AG-3′) e Bakt 805R (5′-GAC TAC HVG GGT ATC TAA TCC-3′). Amplifique a região fúngica ITS2 usando os primers ITS3 KYO2 (5′-GAT GAA GAA CGY AGY RAA-3′) e ITS4 (5′-TCC TCC GCT TAT TGA TAT GC-3′)9.
- Prepare bibliotecas amplicon usando kits de preparação de bibliotecas comerciais compatíveis com o sequenciador escolhido. Sequencie os amplicons usando um sequenciador de leitura curta para gerar 150 ou 250 leituras de extremidade emparelhada de pares de pares de bases com cobertura suficiente, seguindo as instruções do fabricante da plataforma de sequenciamento para a concentração de carga.
- Sequenciamento metagenômico: Crie uma biblioteca metagenômica a partir do DNA extraído usando um kit de preparação de biblioteca metagenômica disponível comercialmente compatível com o sequenciador escolhido. Sequencie a biblioteca de metagenoma usando um sequenciador de leitura curta definido para gerar leituras de extremidade emparelhada de 150 ou 250 pares de bases com cobertura suficiente (~ 10-20 Gb por metagenoma) seguindo as instruções do fabricante da plataforma de sequenciamento para a concentração de carga.
16. Analisando os dados de sequenciamento
- Analise os dados do amplicon: Utilize a plataforma QIIME2 11 com DADA212 para analisar os dados do amplicon antes de visualizar os resultados13. Siga as etapas abaixo para executar o QIIME2 na plataforma de bioinformática baseada na web Empowering the Development of Genomics Expertise (EDGE)14. O QIIME2 também pode ser executado usando tutoriais disponíveis publicamente, como o guia "Imagens em movimento" fornecido online (https://docs.qiime2.org/2024.2/tutorials/moving-pictures/).
- Navegue até https://edgebioinformatics.org/ e faça login ou crie uma conta.
- Selecione RUN QIIME2 na página inicial. Selecione Upload Files na barra de menu à esquerda para fazer upload de arquivos das execuções de sequenciamento do amplicon.
- Crie um arquivo de mapeamento de metadados usando as instruções fornecidas quando o 'i' ao lado de 'Arquivo de mapeamento de metadados' é focalizado.
- Adicione todas as informações necessárias (nome do projeto/execução, tipo de leitura, parâmetros, etc.) e selecione os dados de entrada carregados corretos. Certifique-se de que DADA2 seja selecionado como método de controle de qualidade12; Outros parâmetros podem ser deixados como seus valores padrão, a menos que outras modificações sejam desejadas.
- Selecione o tipo de amplicon como 16S Greengenes (http://greengenes.lbl.gov) para os amplicons bacterianos ou ITS fúngico para os amplicons fúngicos. Analise dados bacterianos (16S) e fúngicos (ITS) de forma independente.
- Pressione enviar e aguarde a conclusão da execução para exibir os resultados.
- Gerar visualizações de dados do amplicon (opcional): execute análises adicionais de dados da comunidade no R usando pacotes comuns, como phyloseq (https://github.com/joey711/phyloseq), VEGAN (https://github.com/vegandevs/vegan) e APE (https://emmanuelparadis.github.io/), seguindo as diretrizes fornecidas no GitHub e por meio da Ajuda do pacote R disponível no R Studio navegando até o menu Pacotes , clicando no pacote baixado e exibindo a documentação.
- Analisar dados metagenômicos
- Navegue até o site NMDC EDGE 15 (https://nmdc-edge.org/home) e faça login usando uma conta ORCiD (https://orcid.org/).
- Selecione Carregar arquivos na barra de menus no lado esquerdo da tela e arraste e solte ou procure o(s) arquivo(s) FASTQ de entrada correto(s).
- Selecione Metagenômica e, em seguida, a opção Executar vários fluxos de trabalho na barra de menus no lado esquerdo da tela e defina todos os fluxos de trabalho como Ativado. Adicione um nome de projeto e uma descrição opcional.
- Selecione o(s) arquivo(s) de leitura bruta (fastq) carregado e selecione o formato de arquivo apropriado (intercalado ou emparelhado).
- Inicie a execução clicando em Enviar e visualizando as tabelas de resumo e visualizações quando a execução for concluída, selecionando o projeto na guia Meus Projetos na parte superior da tela.
17. Criação de visualizações adicionais de dados de taxonomia a partir de resultados amplicon e/ou metagenômicos
- Gere cladogramas circulares usando o pacote GraPhlAn (https://github.com/biobakery/graphlan) seguindo as instruções fornecidas no GitHub.
NOTA: Os identificadores de taxonomia (https://www.ncbi.nlm.nih.gov/taxonomy) do National Center for Biotechnology Information (NCBI) podem ser obtidos a partir de atribuições de sequência representativas e passados para o programa 'eftech' do Entrez Direct E-utilities para coletar informações de acúmulo de taxonomia exigidas pelo GraPhlAn16.
18. Reutilizando as colunas
- Se as colunas ainda estiverem montadas, desparafuse e remova as tampas, o anel rosqueado e a malha. Descarte a malha. Remova todos os tampões de ágar restantes, lave as colunas com álcool isopropílico 99% e água purificada e limpe e seque os componentes conforme descrito nas etapas 3.1-3.4.
- Prepare novas folhas de malha de nylon para serem autoclavadas.
- Autoclave os componentes seguindo as orientações fornecidas na etapa 4.1.
Resultados
A coluna da rodovia fúngica totalmente montada tem aproximadamente 5 cm de comprimento (Figura 1). A coluna não deve ser quebrada em nenhuma área, e as tampas e o anel rosqueado devem se encaixar com facilidade e firmeza para criar microambientes dentro da coluna. A malha do filtro pode se estender além do anel rosqueado (como mostrado na Figura 1 e na Figura 2) ou pode ser aparada com tesouras esterilizadas. Os plugues de ágar devem se encaixar perfeitamente em cada extremidade da coluna. Quando colocada no substrato, a malha do filtro deve entrar em contato com o substrato e a coluna não deve ser completamente enterrada.
As colunas foram previamente testadas em esterco de cavalo9. As colunas também foram colocadas em solo a granel e rizosfera em um local de campo de pesquisa, bem como em pequenos volumes de solo em tubos de 50 mL no laboratório (Figura 3). Depois que as colunas da rodovia fúngica foram removidas do substrato e desmontadas, o crescimento microbiano foi visível nos plugues de isca e meio alvo (exemplos mostrados na Figura 4A). Bactérias e fungos foram isolados do meio alvo e isca por meio de técnicas de subcultura (Figura 4B), e os micróbios presentes nos plugues do meio foram identificados taxonomicamente usando sequenciamento de amplicon (Figura 4C, D). A Figura 4C, D mostra os resultados combinados do sequenciamento do amplicon em vários experimentos, mostrando quais micróbios foram capazes de alcançar o plugue de mídia alvo a partir de colunas adicionadas ao estercode cavalo 9. As visualizações desses dados bacterianos e fúngicos foram geradas conforme descrito na etapa 17. Os resultados também podem ser exibidos como abundâncias relativas de táxons.
Resultados abaixo do ideal foram obtidos nos casos em que as colunas foram adicionadas a ambientes de umidade extremamente baixa e os plugues de mídia foram completamente dessecados em questão de dias, levando a nenhuma recuperação de micróbios colonizados (Figura 5A). Também vimos casos em que os micróbios simplesmente não crescem a partir do plugue de mídia alvo (Figura 5B) e casos em que não recuperamos dados de sequenciamento suficientes do plugue de mídia alvo para análises significativas. Outros casos em que os fungos crescem demais nas colunas também resultaram na necessidade de refazer os experimentos (Figura 5C).

Figura 3: Exemplos das colunas colocadas em amostras ambientais em laboratório e campo. (A) Coluna colocada dentro de um tubo de 50 mL com solo umedecido em um ambiente de laboratório. Também mostrado com uma régua para escala. (B) Coluna colocada no solo no campo. (C) Coluna colocada na rede radicular de uma planta no campo. Clique aqui para ver uma versão maior desta figura.
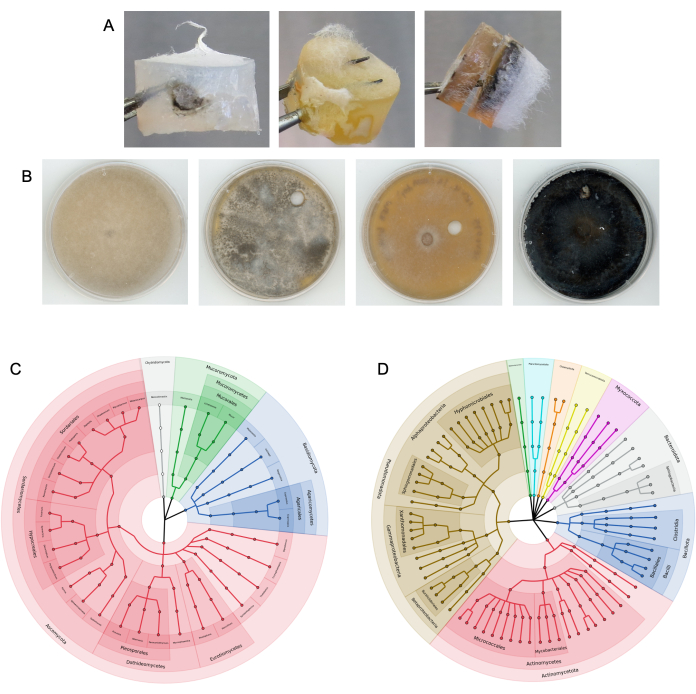
Figura 4: Resultados representativos de experimentos de coluna bem-sucedidos. (A) Exemplos de plugues de mídia colonizados extraídos de colunas. (B) Exemplos de isolados de fungos subcultivados a partir de meios alvo. O substrato foi solo. Sequência superior ITS Identidade NCBI BLAST da esquerda para a direita: Rhizopus azygosporous, Aspergillus novofumigatus, Curvularia subpapendorfii e Phaeomycocentrospora cantuariensis. (C,D) Cladogramas circulares exibindo a diversidade filogenética de (C) ITS fúngico e (D) sequências bacterianas 16S recuperadas do meio alvo após vários experimentos fúngicos em rodovias usando esterco de cavalo. As seções são coloridas e rotuladas por filo, com nós finais representando gêneros únicos. Clique aqui para ver uma versão maior desta figura.
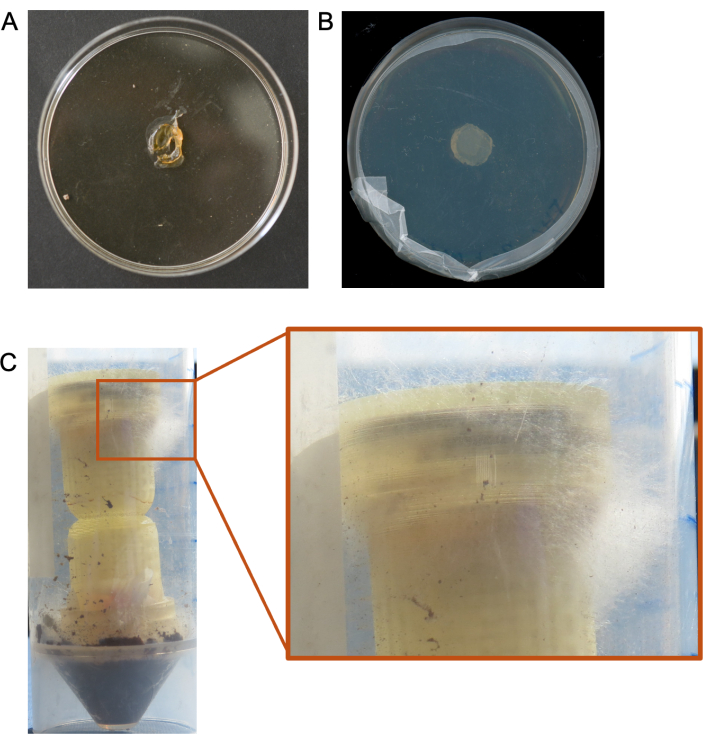
Figura 5. Resultados abaixo do ideal de experimentos de coluna. (A) Um exemplo de um tampão de mídia desidratado resultante de condições ambientais de baixa umidade. (B) Exemplo de nenhum crescimento microbiano de um tampão de mídia de coluna. (C) Exemplo de crescimento excessivo do fungo através do topo (meio alvo) da coluna. Clique aqui para ver uma versão maior desta figura.
Discussão
Ao gerar os componentes da coluna, a seleção de uma impressora 3D e do material de impressão pode ser modificada com base na disponibilidade e nas propriedades desejadasdo material 17,18. A biocompatibilidade, textura da superfície, autoclavabilidade, capacidade de imprimir detalhes em escala fina e transparência relativa foram considerados na seleção de materiais do nosso grupo. Outras características, como porosidade, hidrofobicidade, parâmetros de impressão, etc., também devem ser consideradas. Várias resinas foram testadas (ver Tabela de Materiais) antes da seleção final, e muitos materiais biocompatíveis funcionarão para a impressão dessas colunas. O material escolhido para a construção dos componentes da coluna determinará quais abordagens de limpeza, pós-cura e esterilização devem ser usadas. Nem todos os materiais serão autoclaváveis e luz ultravioleta, alvejante ou outras técnicas de esterilização podem ser necessárias, dependendo das instruções do fabricante do material. Algumas técnicas de esterilização ou limpeza também podem danificar ou não ser compatíveis com o material escolhido, pelo que deve ser dada especial atenção a esta informação por parte do fabricante do material. Para impressoras 3D, algumas considerações incluem tempo de impressão, compatibilidade de materiais, tamanho da plataforma de construção, tecnologia de impressão e custo19. Os componentes impressos em 3D das colunas podem ser frágeis e podem quebrar se manuseados com muita força. A rosca do anel e das tampas nem sempre se alinham exatamente, portanto, recomendamos que componentes extras sejam impressos e esterilizados antes da etapa de montagem, ou a impressão preliminar seja feita para testar como os parâmetros e o material escolhido afetam a rosca. As especificações de design para o rosqueamento dentro das tampas e do anel podem precisar ser ajustadas dependendo do material de impressão 3D escolhido. As dimensões, complexidade da rede e outras características físicas podem ser modificadas no software de design CAD antes da impressão. Conforme projetado, a coluna em si tem 4 cm de altura e a estrutura da treliça dentro do centro da coluna tem uma célula unitária de 2 mm, um diâmetro de escora de 0,5 mm e toda a altura da treliça é de 22 mm9. Esses parâmetros podem ser ajustados se um pesquisador quiser, por exemplo, uma estrutura de rede maior ou mais complexa. No geral, a fabricação impressa em 3D desses dispositivos permite flexibilidade de design, ao mesmo tempo em que garante que um único design possa ser usado de maneira padronizada em organizações e grupos, e até mesmo usado como ferramentas de ensino em sala de aula9.
Várias etapas do protocolo podem exigir solução de problemas, dependendo do ambiente ou da configuração experimental. As colunas de rodovias fúngicas não são muito eficazes em condições de baixa umidade, pois os tampões de mídia secam rapidamente antes de facilitar o crescimento de fungos, o que pode limitar a duração dos experimentos nesses ambientes (Figura 5A). As técnicas que melhoraram a eficácia das colunas em ambientes de baixa umidade incluem o aumento artificial da umidade por meio da adição de umidade ao substrato e/ou a vedação da coluna e do substrato em um recipiente secundário com uma fonte de água (por exemplo, um pequeno recipiente de água pura). A forma de ampulheta e a estrutura da treliça foram incorporadas para evitar o movimento bacteriano sozinho (sem o estabelecimento de uma rodovia fúngica) se a condensação se formasse em ambientes de alta umidade. Os fungos de crescimento rápido podem crescer demais na área de superfície do alvo e do meio de isca e se estender para fora da parte superior ou inferior da coluna (Figura 5C). Diminuir o tempo de incubação do fungo da isca ou a duração do experimento pode minimizar ou eliminar esse crescimento excessivo. Além disso, uma limitação desses dispositivos é que fungos de crescimento rápido no substrato de interesse podem limitar a colonização da isca e do meio-alvo por fungos de crescimento lento, potencialmente influenciando quais interações de rodovias são observadas. Alguns fungos, especialmente fungos de crescimento mais lento, podem não colonizar o meio de isca de uma maneira que lhes permita crescer através do tampão de ágar e na estrutura da rede. Se houver umidade suficiente no ambiente, plugues de ágar mais finos podem ser usados para estimular o crescimento na rede após a colonização do tampão de ágar isca. O meio pode ser escolhido com base no fato de um pesquisador querer selecionar o crescimento de fungos ou bactérias, mas isso também pode limitar a subcultura a organismos que preferem esse tipode meio 20. Se nenhum crescimento for observado no meio alvo, pode ser necessário inocular o meio ou substrato da isca com um fungo conhecido por criar rodovias fúngicas.
O sequenciamento metagenômico ou amplicon pode ser realizado como parte desses experimentos, e ambas as estratégias transmitem suas próprias limitações e pontos fortes21. O sequenciamento metagenômico é ideal para obter informações genômicas adicionais sobre os micróbios. No entanto, a quantidade recuperável de ácidos nucléicos diretamente do meio alvo pode ser muito baixa, o que pode exigir a utilização de sequenciamento de amplicon ou outros métodos de amplificação antes do sequenciamento. As bibliotecas de sequenciamento de amplicon devem ser preparadas separadamente (16S e ITS), e este método carece de resolução taxonômica e limita quaisquer avaliações sobre características do genoma ou potencial funcional que possam ser alcançados usando sequenciamento metagenômico. Métodos de sequenciamento direto dos plugues podem ser preferidos nos casos em que os micróbios não podem ser subcultivados. Recomenda-se que os plugues sejam divididos em várias seções para permitir abordagens de cultura e sequenciamento.
Um benefício desses dispositivos é que eles podem ser usados tanto em laboratório quanto em campo. Cuidados especiais devem ser tomados para garantir que as colunas no campo possam permanecer na vertical e sejam protegidas de animais e perturbações ambientais que possam perturbar sua colocação. As colunas ainda não foram testadas na posição horizontal, em uma posição em que estejam totalmente cobertas por um substrato, e não foram testadas em ambientes expostos a chuvas ou neve substanciais. Como dito acima, a estrutura da rede foi projetada para minimizar a probabilidade de as bactérias serem capazes de se mover para o meio alvo em ambientes de alta umidade. No entanto, é possível que, se a coluna fosse exposta a volumes maiores de água e essa água saturasse totalmente a coluna, o movimento bacteriano seria facilitado em toda a coluna, independentemente de quaisquer rodovias fúngicas presentes. Para experimentos baseados em laboratório, as colunas podem ser usadas dentro de tubos cônicos de 50 mL, pequenos microcosmos de substratos, no solo ao redor de vasos de plantas, em caixas ou em outros sistemas experimentais controlados. As colunas têm sido utilizadas com sucesso no solo, rizosferas e esterco, e sua utilidade pode ser expandida para outros substratos, incluindo serapilheira, lodo, areia, neve, composto, etc.
As colunas de rodovias fúngicas permitem uma série de comparações para entender esse fenótipo BFI em diversos tipos de amostras. A comparação da composição da comunidade entre a isca e o meio alvo pode indicar quais bactérias podem utilizar rodovias fúngicas e quais fungos podem servir como rodovias potenciais9. Se o sequenciamento do metagenoma for usado, as características genômicas que distinguem os organismos da isca versus o meio alvo também podem ser examinadas. Também é possível comparar os meios alvo de colunas colocadas em diferentes substratos (por exemplo, solo versus esterco) ou colocadas no mesmo substrato sob diferentes condições (por exemplo, temperatura ou umidade). No geral, as colunas de rodovias fúngicas expandem as capacidades dos métodos anteriores para interrogar essa forma de BFI e permitem exames extensivos dessas interações que moldam a dinâmica espacial de microbiomas ambientais complexos.
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Esta pesquisa foi apoiada por uma Bolsa de Área de Foco Científico do Departamento de Energia dos EUA (DOE), Pesquisa Biológica e Ambiental (BER), Divisão de Ciência do Sistema Biológico (BSSD) sob o número de concessão LANLF59T.
Materiais
| Name | Company | Catalog Number | Comments |
| 50 mL tubes | Greiner BIO-ONE | 5622-7261 | 50 mL tubes for performing column experiments in the lab |
| 90 mm Petri dishes | Thermo Scientific Nunc | 08-757-099 | Petri dishes for preparation of agar and for microbial growth |
| Asiga Freeform Pico Plus 39 digital light processing (DLP) 3D printer | Asiga Germany | Freeform Pico Plus 39 | 3D printer used to generate batches of the columns; other 3D printers can be used |
| Autoclave | Fisher Scientific | LS40F20 | Benchtop autoclave to sterilize the column components |
| Beaker | Fisher Scientific | FB100600 | 600 mL beaker for various uses throughout the protocol |
| Dental LT Clear Resin V2 | Formlabs | RS-F2-DLCL-02 | Alternative resin for 3D printing that was tested |
| Dental Surgical Guide Resin | Formlabs | RS-F2-SGAM-01 | Was used to generate the columns discussed in manuscript; Other photosensitive resins can be used in place of this material |
| DNA Low Bind 1.5 mL tubes | Eppendorf | 13-698-791 | Tubes used for various preparations including nucleic acid extractions |
| DNA/RNA shield preservative | Zymo Research | R1100-50 | Preservative used prior to nucleic acid extractions |
| EDGE Bioinformatics | Open source; Developed by the Los Alamos National Laboratory (LANL) | n/a | Bioinformatics platform for processing amplicon data |
| FastDNA spin kit for soil | MP Biomedicals LLC | 116560200-CF | DNA extraction kit option for soil |
| Forceps | Fisher Scientific | 10-300 | Forceps that can be sterilized |
| Formlabs BioMed Clear Resin | Formlabs | RS-F2-BMCL-01 | Alternative resin for 3D printing that was tested |
| Formlabs Form 3B+ stereolithography (SLA) 3D printer | Formlabs | Form 3B+ | Alternative 3D printer |
| Formlabs IBT Resin | Formlabs | RS-F2-IBCL-01 | Alternative resin for 3D printing that was tested |
| Inoculating Loops | Fisher Scientific | 22-363-598 | Used to isolate/transfer microbes |
| Malt Extract Agar (MEA) | Criterion | 89405-654 | A media type used in columns |
| MiSeq sequencer + MiSeq sequencing kit | Illumina | SY-410-1003 | Can use other sequencers |
| Mortar & Pestle | Fisher Scientific | FB961K; FB961A | Can use any common mortar & pestle that can be sterilized between uses |
| NEBNext Ultra II DNA Library Prep Kit for Illumina | New England Biolabs | E7805S | Library prep kit for metagenomic sequencing |
| Nextera XT DNA Library Preparation Kit (24 samples) | Illumina | FC-131-1024 | Library prep kit for amplicon sequencing |
| NMDC EDGE | Open source: Developed by the National Microbiome Data Collaborative (NMDC) | n/a | Bioinformatics platform for processing metagenomic data |
| Nylon mesh | Sefar | 03-25/19 | The mesh used as part of the column construction |
| Pipette tips | Rainin | 30807966 | Can use many different sterilized pipette tips for the protocol steps |
| Potato Dextrose Agar | Cole Parmer | EW-14200-28 | A media type used in columns |
| QIIME2 | Open source | n/a | Software for processing amplicon data |
| Qubit dsDNA HS assay kit | Thermo Fisher Scientific | Q32851 | Used to quantify DNA after extractions |
| Qubit Fluorometer | Thermo Fisher Scientific | Q33238 | Used to quantify DNA after extractions |
| Quick-DNA Fungal/Bacterial Miniprep Kit | Zymo Research | D6005 | DNA extraction kit option that works with both bacteria and fungi |
| R2A agar | BD Difco | 218263 | A media type used in columns (bacterial media) |
| Rack for 50 mL tubes | Fisher Scientific | 03-448-11 | Rack to hold 50 mL tubes upright |
| Scissors | Fisher Scientific | 12-000-155 | Fine precision scissors that can be sterilized |
| Sodium carboxymethyl cellulose medium | Aldrich | 419273-100G | A media type used in columns |
| SolidWorks CAD software | SolidWorks | n/a | Software used to design the columns |
| Trowel scoop | Fisher Scientific | S41701 | To make a depression in the substrate prior to adding the column |
| UltraPure DNase/RNase-Free Distilled Water | Invitrogen: ThermoFisher Scientific | 10977015 | Water for the ultrasonicator water bath |
| Ultrasonicator | Fisher Scientific | FB-11201 | Ultrasonicator for cleaning the columns |
Referências
- Or, D., Smets, B. F., Wraith, J. M., Dechesne, A., Friedman, S. P. Physical constraints affecting bacterial habitats and activity in unsaturated porous media - A review. Adv Water Resour. 30 (6), 1505-1527 (2007).
- Kohlmeier, S., et al. Taking the fungal highway: Mobilization of pollutant-degrading bacteria by fungi. Environ Sci Technol. 39 (12), 4640-4646 (2005).
- Simon, A., Hervé, V., Al-Dourobi, A., Verrecchia, E., Junier, P. An in situ inventory of fungi and their associated migrating bacteria in forest soils using fungal highway columns. FEMS Microbiol Ecol. 93 (1), 217 (2017).
- Wick, L. Y., et al. Effect of fungal hyphae on the access of bacteria to phenanthrene in soil. Environ Sci Technol. 41 (2), 500-505 (2007).
- Bravo, D., et al. Isolation of oxalotrophic bacteria able to disperse on fungal mycelium. FEMS Microbiol Lett. 348 (2), 157-166 (2013).
- Furuno, S., Remer, R., Chatzinotas, A., Harms, H., Wick, L. Y. Use of mycelia as paths for the isolation of contaminant-degrading bacteria from soil. Microb Biotechnol. 5 (1), 142-148 (2012).
- Buffi, M., et al. Fungal drops: A novel approach for macro- and microscopic analyses of fungal mycelial growth. Microlife. 4, 042 (2023).
- Kuhn, T., et al. Design and construction of 3D printed devices to investigate active and passive bacterial dispersal on hydrated surfaces. BMC Biol. 20 (1), 203 (2022).
- Junier, P., et al. Democratization of fungal highway columns as a tool to investigate bacteria associated with soil fungi. FEMS Microbiol Ecol. 97 (2), 003 (2021).
- Reasoner, D. J., Geldreich, E. E. A new medium for the enumeration and subculture of bacteria from potable water. Appl Environ Microbiol. 49 (1), 1-7 (1985).
- Bolyen, E., et al. Reproducible, interactive, scalable, and extensible microbiome data science using QIIME 2. Nat Biotechnol. 37 (8), 852-857 (2019).
- Callahan, B. J., et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nat Methods. 13 (7), 581-583 (2016).
- Vázquez-Baeza, Y., Pirrung, M., Gonzalez, A., Knight, R. EMPeror: A tool for visualizing high-throughput microbial community data. Gigascience. 2 (1), 16 (2013).
- Li, P. -. E., et al. Enabling the democratization of the genomics revolution with a fully integrated web-based bioinformatics platform. Nucleic Acids Res. 45 (1), 67-80 (2017).
- Eloe-Fadrosh, E. A., et al. The National Microbiome Data Collaborative Data Portal: An integrated multi-omics microbiome data resource. Nucleic Acids Res. 50 (1), D828-D836 (2022).
- Entrez Direct: E-utilities on the Unix Command Line. Entrez Programming Utilities Help Available from: https://www.ncbi.nlm.nih.gov/books/NBK179288/ (2024)
- Palmara, G., Frascella, F., Roppolo, I., Chiappone, A., Chiadò, A. Functional 3D printing: Approaches and bioapplications. Biosens Bioelectron. 175, 112849 (2021).
- Guttridge, C., Shannon, A., O'Sullivan, A., O'Sullivan, K. J., O'Sullivan, L. W. Biocompatible 3D printing resins for medical applications: A review of marketed intended use, biocompatibility certification, and post-processing guidance. Ann 3D Print Med. 5, 100044 (2022).
- Yao, L., et al. Comparison of accuracy and precision of various types of photo-curing printing technology. J Phys Conf Ser. 1549 (3), 032151 (2020).
- Basu, S., et al. Evolution of bacterial and fungal growth media. Bioinformation. 11 (4), 182-184 (2015).
- Liu, Y. -. X., et al. A practical guide to amplicon and metagenomic analysis of microbiome data. Protein Cell. 12 (5), 315-330 (2021).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados