Method Article
다양한 환경 및 기질에서 Fungal Highway Column을 사용한 박테리아-진균 상호 작용 조사
요약
이 프로토콜은 다양한 환경 기질에서 곰팡이 고속도로를 통해 상호 작용하는 박테리아-곰팡이 쌍을 농축하기 위해 곰팡이 고속도로 기둥을 구성, 멸균, 조립, 활용 및 재사용하는 방법에 대한 자세한 지침을 제공합니다.
초록
박테리아-진균 상호작용(BFI)은 미생물 군집 구성, 생지화학적 기능, 공간 역학 및 미생물 분산을 형성하는 데 필수적인 역할을 합니다. 사상균 또는 기타 사상균 미생물(예: 난균류)에 의해 생성된 균사체 네트워크는 박테리아가 이질적인 환경을 가로지르는 수송을 위해 활용할 수 있는 '진균 고속도로' 역할을 하여 박테리아의 이동성을 크게 촉진하고 스스로 도달하기 어렵거나 불가능할 수 있는 영역(예: 토양 내의 공기 주머니로 인해)에 접근할 수 있도록 합니다. 곰팡이 고속도로 기둥을 포함하여 이러한 곰팡이 고속도로를 연구하기 위해 여러 장치와 실험 프로토콜이 만들어졌습니다. 우리 그룹이 설계한 곰팡이 고속도로 기둥은 다양한 현장 또는 체외 응용 분야뿐만 아니라 다양한 환경 및 숙주 관련 시료 유형에도 사용할 수 있습니다. 여기에서는 장치의 설계, 인쇄, 멸균 및 준비를 포함하여 이러한 컬럼을 사용하여 실험을 수행하는 방법을 설명합니다. 이러한 장치를 사용하여 얻은 데이터를 분석하기 위한 옵션도 여기에서 논의되며, 곰팡이 고속도로 기둥을 사용한 실험과 관련된 잠재적 위험에 대한 문제 해결 조언이 제공됩니다. 이러한 장치는 곰팡이 고속도로 BFI의 다양성, 메커니즘 및 역학에 대한 보다 포괄적인 이해를 얻는 데 사용할 수 있으며, 복잡한 환경(예: 토양) 및 박테리아와 곰팡이가 공존하는 다양한 서식지 내에서 구조적 및 기능적 역학에 대한 귀중한 통찰력을 제공할 수 있습니다.
서문
박테리아-진균 상호작용(BFI)은 환경 마이크로바이옴의 구조적, 공간적, 기능적 특성을 형성하는 데 매우 중요합니다. 예를 들어, 사상균 또는 기타 균류와 유사한 사상균 미생물의 성장과 확장은 박테리아와 같은 다른 미생물의 이동을 촉진하는 '고속도로'로 기능할 수 있는 생물학적 네트워크를 생성합니다. 환경 기질 내의 이질성과 일관되지 않은 포화는 박테리아의 운동성을 방해할 수 있습니다. 그러나 박테리아는 이러한 고속도로를 사용하여 환경의 추가 영역에 쉽게 접근할 수 있습니다 1,2. 이러한 상호 작용은 미생물 군집의 공간 역학을 이해하는 데 중요합니다. 곰팡이 고속도로를 조사하기 위해 여러 가지 기술과 방법이 사용되었지만, 대부분 실험실 기반 조사에 국한되어 있습니다 3,4.
한 플레이트 기반 방법에서는 페트리 접시 중간에서 한천의 큰 부분을 제거하여 두 한천 섬 사이에 틈을 만듭니다. 곰팡이 균사는 이 틈을 통과할 수 있으며, 호환 가능한 박테리아가 한 한천 섬에서 다른 한천 섬으로 교차할 수 있는 수단을 제공합니다5. 다른 변형된 페트리 접시 방법으로는 곰팡이 균사가 수직으로 성장하고 직접적인 접촉 없이 배지에 군집화할 수 있도록 뚜껑에 흙을 넣는 거꾸로 된 판이 있으며, 이는 박테리아 수송 수단을 제공합니다 5,6. 최근에 개발된 성장 배지 액적 기반 방법은 특정 영양 프로필로 향하는 박테리아의 선택적 균사 이동을 평가하는 데 사용할 수 있습니다7. 박테리아 다리 및 트레일 장치는 또한 박테리아의 이동에 대한 비생물적 요인의 영향을 조사하는 데 사용되었습니다8. 곰팡이 고속도로를 조사하기 위해 여러 가지 방법과 기법이 사용되었지만, 배설물, 토양 및 근권과 같은 복잡한 환경 기질에서 곰팡이 고속도로 건설을 촉진하면서 멸균 미세 환경을 유지하는 표준화된 장치에 대한 필요성은 여전히 남아 있습니다.
우리 그룹은 곰팡이가 한쪽 끝에서 다른 쪽 끝으로 박테리아를 운반할 수 있는 곰팡이 고속도로 기둥의 3D 프린팅 버전을 설계했습니다9. 이러한 장치는 모래시계 모양과 복잡한 내부 격자 구조의 컬럼 자체, 나사산 링, 두 개의 캡(큰 캡 및 작은 캡), 멸균된 나일론 메쉬 조각의 4가지 인쇄 부품으로 조립됩니다(그림 1). 조립된 컬럼은 원하는 환경 기판에 직접 추가됩니다. 그런 다음 컬럼은 미생물이 컬럼 바닥에 있고 메쉬를 통해 환경 기질과 접촉하는 '미끼' 배지 플러그로 알려진 한천 성장 배지 플러그에 서식할 수 있도록 합니다. 이 나일론 메쉬 조각은 박테리아를 운반할 수 있는 다른 토양 거주자를 제외하므로 기둥 내의 박테리아 이동을 곰팡이 고속도로로 제한합니다. 이 미끼 플러그가 군집화되면 사상균은 토양(또는 기타 불포화 매체)과 유사한 불포화 시스템을 생성하고 미끼 매체의 잠재적 오염을 최소화하도록 설계된 기둥 중앙 내의 내부 격자를 통해 확장 및 성장할 수 있습니다. 그런 다음 곰팡이는 기둥 상단에 있는 대상 매체 플러그를 향해 성장하고 군락화합니다. 컬럼에 특정 곰팡이 분리물을 접종하여 박테리아를 운반하는 능력을 테스트하거나, 기질에서 어떤 곰팡이가 박테리아를 운반할 수 있는지 확인하기 위해 접종하지 않은 상태로 둘 수 있습니다. 타겟 배지에 도달한 유기체는 추가로 배양, 분리 및 염기서열분석 분석을 수행할 수 있습니다(순수 배양 또는 앰플리콘 또는 메타유전체학(metagenomic) 염기서열분석 접근법을 사용하는 혼합 군집에서). 전반적으로 이 컬럼은 다양한 기질에서 곰팡이 고속도로를 조사하기 위한 표준화되고 재현 가능하며 재사용 가능하고 직관적인 방법을 제공합니다. 이러한 장치는 연구 및 교실 교육 도구로 사용할 수 있으며, 여기에서는 과거에 수행된 실험을 기반으로 이를 사용하기 위한 교육 단계를 제공합니다. 이 방법은 프로토콜 표준화를 용이하게 하지만, 장치의 설계 및 구성은 다른 애플리케이션 및 추가 기판에 맞게 수정될 수 있습니다.
프로토콜
연구에 사용된 시약 및 장비의 세부 사항은 재료 표에 나열되어 있습니다.
1. 컬럼 설계, 재료 및 매개변수 수정
- 공개적으로 사용 가능한 컬럼 디자인9 을 다운로드하여 그대로 사용하거나 호환 가능한 CAD(Computer-Aided Design) 소프트웨어에서 컬럼 디자인을 수정하십시오.
- 치과 수술 가이드 레진을 구하거나 다른 감광성 투명 레진과 같은 대체 3D 프린팅 재료를 선택합니다.
- 선택한 3D 프린터, 3D 프린팅 기술 또는 3D 프린팅 재료가 이전에 사용된 것과 변경된 경우 필요에 따라 열의 사양을 조정하십시오9.
2. 3D 기둥 인쇄
- 0.05mm 슬라이스 두께와 0.8초의 노출 시간을 사용하도록 인쇄 매개변수를 설정하거나 선택한 프린터, 인쇄 재료 및 인쇄 소프트웨어에 따라 인쇄 매개변수를 조정합니다.
- 호환되는 3D 프린터를 사용하여 곰팡이 고속도로 기둥, 나사산 고리 및 캡을 인쇄합니다(그림 1).

그림 1: 곰팡이 고속도로 기둥의 구성 요소. (A,B) 작은 캡, 나사산 링 및 큰 캡의 상단 및 측면 모습(왼쪽에서 오른쪽으로). (씨,디) 작은 모자의 상단 및 측면 보기. (E,F) 나사산 링의 상단 및 측면 뷰. (G,H) 큰 모자의 상단과 측면도. (I) 컬럼 끝에 나일론 메쉬 필터(25μm) 조각을 배치하고 환경 기판에 삽입하여 소동물이 컬럼에 진입하는 것을 방지합니다. (J) 조립되지 않은 기둥. (K) 조립된 기둥: '미끼' 끝은 기판 속으로 들어가고 '타겟' 끝은 덮개가 없는 상태로 기판 밖으로 나옵니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
3. 3D 프린팅 컬럼 구성 요소 청소
- 인쇄된 기둥, 캡 및 나사산 링을 실온에서 99% 이소프로필 알코올에 15분 동안 담그고 1분마다 10-15초 동안 손으로 수조를 앞뒤로 움직여 교반하여 과도한 수지를 제거합니다.
- 구성 요소를 새로운 99% 이소프로필 알코올 수조로 옮깁니다. 5분 동안 물에 담그고 3.1단계와 같은 방식으로 손으로 저어줍니다.
- 구성 요소를 순수한 물로 채워진 초음파 세척기 장치로 옮기고 구성 요소를 담근 다음 중간 속도 설정에서 2분 동안 교반합니다. 물을 가열하지 마십시오. 구성 요소를 제거합니다.
- 모든 구성 요소를 최소 30분 동안 자연 건조하십시오.
- 수지의 후경화를 수행하려면 모든 3D 프린팅 부품을 60°C에서 30분 동안 405nm 광에 노출시킵니다.
4. 컬럼 살균
- 선택한 재료가 고압증기멸균이 가능한 경우 컬럼, 나사산 링, 캡 및 25μm 나일론 메쉬 필터 시트를 개별적으로 또는 더 큰 비커 내에서 121°C, 1기압에서 20-30분 동안 고압증기멸균합니다.
참고: 컬럼의 구성 요소는 고압증기멸균 후 모양과 색상이 변경될 수 있지만 원하는 재료 특성은 유지됩니다. 오토클레이빙 후 컬럼 성분의 최종 크기, 모양 및 색상은 그림 1에 나와 있습니다.
5. 컬럼에 대한 매체 준비
- 나트륨 카르복시메틸 셀룰로오스 배지(CMC), 맥아 추출물 한천(MEA), 감자 포도당 한천(PDA) 또는 Reasoner의 2A 한천(R2A)9,10과 같은 멸균된 한천 기반 배지의 90mm 페트리 접시를 준비합니다. 제조업체의 지침에 따라 미디어를 준비합니다.
- 21°C, 121기압에서 제조업체가 권장하는 시간 동안 고압증기멸균하여 매체를 살균하고 한천이 페트리 접시의 측면 상단에 가까워질 때까지 90mm 페트리 접시에 붓습니다(그림 2). 생물 안전 작업대에서 이 단계를 수행하여 멸균 상태를 높이십시오.
- 한천이 제조업체의 지침에 따라 응고되고 건조되도록 합니다.

그림 2: 곰팡이 고속도로 기둥의 조립 공정. 컬럼 자체의 열린 끝을 사용하여 플러그를 잘라내어 삽입하고, 연구원은 미디어에서 컬럼을 제거할 때 컬럼을 비틀어 플러그가 컬럼 끝 내에 유지되도록 합니다. 그 끝은 작은 캡 조각으로 덮여 있습니다. 그런 다음 미디어 플러그가 동일한 방식으로 열의 다른 쪽 끝에 추가됩니다. 그런 다음 메쉬 조각을 이 끝 부분에 놓고 나사산 링으로 고정합니다. 그런 다음 큰 캡이 나사산 링 위의 이 '미끼' 끝에 사용됩니다. 메쉬가 있는 면은 환경 기판에 배치됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
6. 곰팡이 고속도로 기둥 준비
참고: 이 단계는 컬럼 구성 요소와 매체의 멸균 상태를 유지하기 위해 생물 안전 작업대에서 수행해야 합니다. 그림 2 는 곰팡이 고속도로 기둥의 조립 과정을 보여줍니다.
- 컬럼 끝 자체(캡이 없음)를 사용하여 컬럼의 한쪽 끝에 단단히 맞는 미디어 플러그를 추출합니다. 한천이 열 끝 내에 유지되도록 기둥이 미디어에서 들어 올려질 때 비틀기 동작을 사용합니다. 또는 컬럼 끝을 템플릿으로 사용하고, 미디어를 분리하고, 피펫 팁을 사용하여 컬럼으로 옮깁니다.
- 첫 번째 플러그를 컬럼 끝에 추가한 후 이 끝에 작은 캡을 추가하여 대상 매체에 대한 멸균 미세 환경을 유지합니다.
- 열을 뒤집고 열의 다른 쪽 끝에 대해 6.1단계를 반복합니다.
- 멸균된 가위를 사용하여 오토클레이브 처리된 나일론 메쉬(25μm 기공 크기)의 ~2cm 직경의 원형 조각을 자릅니다(그림 1). 기둥의 노출된 끝 부분에 메쉬를 놓습니다. 나사산 링을 컬럼의 이 미끼 미디어 끝에 비틀면서 나사산 내부의 메쉬를 고정합니다.
- 메쉬 위의 컬럼 바닥에 큰 캡과 나사산 링의 다른 쪽 끝을 놓고 컬럼을 보관하거나 운반할 때 이 캡을 계속 씌우십시오.
7. 기질 또는 미끼 배지에 관심 곰팡이를 사전 접종
참고: 이 단계는 선택 사항입니다.
- 페트리 접시 내의 고체 진균 성장 배지(예: MEA, PDA, 5단계에서 설명한 대로 준비)에서 곰팡이를 먼저 성장시키고 눈에 띄는 진균 성장이 있는 한천의 작은(~1cm) 부분을 기질로 이동시켜 원하는 기질에 관심 진균(예: 진균 고속도로를 생성하는 것으로 알려진 진균)을 접종합니다.
참고: 이전 실험9 은 사전 집락화된 기질에 컬럼을 추가하기 전에 10일 동안 Coprinopsis cinerea 와 함께 사전 집락화된 배설물 기질을 사용했습니다. - 기질을 미리 집락화하는 대신 미끼 곰팡이를 페트리 접시에 추가하거나 소량의 곰팡이 균사체를 사용하여 컬럼 내의 미끼 배지 바닥에 직접 추가합니다(예: 멸균 루프의 스와이프).
- 페트리 접시의 경우 페트리 접시의 많은 부분(덮인 접시의 약 50%-75%)을 덮는 눈에 띄는 성장이 있을 때까지 기다린 다음 곰팡이 성장의 바깥쪽 가장자리에 가장 가까운 집락화된 페트리 접시에서 직접 미끼 배지 부분을 찍어냅니다(6.1단계 설명 참조).
- 미끼 배지를 직접 사전 집락화하는 경우 기질에 컬럼을 추가하기 전에 미끼 배지 전체에 명확한 성장이 있을 때까지 기다립니다(며칠이 걸릴 수 있음).
참고: 이전 실험9 은 C. cinerea 를 미끼 배지로 사전 식민지화한 14일 된 배지를 사용했습니다. 군락화 전 시간은 곰팡이의 성장 속도, 배지 유형 및 배양 조건에 따라 달라집니다.
8. 대조군 처리 및 복제 준비
- negative control column(negative control column)을 준비하고(단계 1-6에 설명된 대로), 이를 접종하거나 이러한 column을 기판에 배치하지 마십시오. 이를 사용하여 후속 분석을 위한 기준선을 제공하고 준비 공정의 오염을 평가할 수 있습니다.
- 모든 실험에 대해 최소 3개의 열 반복실험을 포함합니다.
9. 기판에 컬럼 추가
- 실험실 소우주(예: 상자, 냄비 또는 튜브 안의 흙; 그림 3A) 또는 열을 관심 있는 필드 사이트로 가져옵니다.
- 컬럼 바닥에서 큰 캡을 제거하여 나사산 링과 메쉬를 노출시킵니다.
- 관심 있는 기질(그림 3)에 컬럼을 추가하고 아래에 설명된 각 기질 유형에 대한 특정 고려 사항에 따라 추가합니다. 필요한 경우, 메쉬의 손상을 최소화하기 위해 컬럼을 기판에 삽입하기 전에 장갑을 낀 손이나 작은 도구(예: 작은 흙손)를 사용하여 기판을 움푹 패입니다.
- 배설물: 이 기둥은 이전에 신선한 말똥에 성공적으로 사용되었습니다. 사용하기 전에 배설물을 4°C에서 보관하고 컬럼을 추가하기 전에 마젠타색 상자와 같은 챔버에 추가합니다. 전체 컬럼 높이의 1-2cm를 덮도록 컬럼을 기판에 삽입합니다.
- 토양: 전체 바닥 부분이 토양(토양 깊이의 ~1-2cm) 내에 있도록 기둥을 삽입합니다(그림 3B). 기둥은 토양 위에 놓이거나 대상 쪽의 작은 캡 바로 아래에 묻힐 수 있습니다.
- 근권(Rhizosphere): 9.3.2단계의 지시에 따라 식물 뿌리를 둘러싼 토양에 기둥을 삽입하면서 기둥을 각도를 조절하고 특정 뿌리 가까이에 배치하여 뿌리권 미생물을 포획할 가능성을 높입니다(그림 3C).
- 튜브 내 기질: 50mL 원뿔형 튜브 또는 이와 유사한 튜브에 ~10mL의 관심 기질을 추가하고 원하는 경우(예: 습도가 매우 낮은 환경에서) 순수한 물을 사용하여 기질을 적십니다. 바닥이 묻히고 나사산 링이나 메쉬의 일부가 보이지 않도록 튜브에 컬럼을 삽입합니다(그림 3A). 50mL 튜브의 뚜껑을 나사로 닫고 50mL 튜브 랙에 넣어 똑바로 세웁니다.
10. 기질에 있는 란 남겨두기
- 컬럼을 기판에 3-21일 동안 그대로 두십시오.
참고: 컬럼을 기질에 남겨두어야 하는 시간은 사용된 미끼 곰팡이의 성장률, 미끼 배지에 서식하는 곰팡이의 성장 속도 및 환경 조건에 따라 다릅니다(예: 건조한 조건은 배지 건조가 더 빠르므로 오래 사용할 수 없음). - 실험을 위해 원하는 명암 주기를 설정합니다. 이전 실험에서는 실험실 내의 기둥을 어둠 속에 두었지만, 현장의 기둥은 현지의 낮과 밤 조건9을 적용받았다.
11. 기질에서 컬럼 제거
- 컬럼이 원하는 시간 동안 기질과 접촉한 후 컬럼을 제거하고 여분의 기질을 조심스럽게 털어낸 다음 메쉬 아래의 컬럼 바닥에 큰 캡을 다시 추가하여 컬럼을 운반합니다.
- 이송을 위해 컬럼을 멸균 비커 또는 50mL 튜브에 넣습니다. 운반하는 동안 기둥을 똑바로 세우십시오.
12. 컬럼의 타겟 매체에서 분리물을 배양하기
- 생물 안전 캐비닛과 같은 멸균 환경에서는 미끼 매체, 대형 캡, 나사산 링 및 메쉬를 제거합니다. 메시를 버립니다.
참고: 연구자가 어떤 유기체가 미끼 배지에 군집화했는지에 관심이 있는 경우 대상 배지 플러그 외에 미끼 배지 플러그를 사용하여 12.3-12.4단계를 따르십시오. - 대상 매체를 포함하는 컬럼 끝에서 작은 캡을 제거합니다.
- 타겟 플러그가 컬럼에서 떨어지도록 컬럼을 뒤집거나 멸균된 집게 또는 피펫 팁을 사용하여 플러그를 빼내십시오. 선택한 한천 매체로 채워진 90mm 페트리 접시의 중앙에 대상 매체 플러그를 직접 놓습니다(5단계에서 준비한 대로).
참고: 곰팡이 또는 박테리아 특이적 배지를 여기에서 사용하여 두 왕국 구성원의 성장을 향상시킬 수 있습니다. 또한 멸균된 가위 또는 피펫 팁을 사용하여 플러그를 두 개 이상의 조각으로 나눌 수 있으므로 여러 배지 유형에 도금하거나 DNA 추출을 위해 표적 배지를 보존할 수 있습니다(14단계 참조). - 표적 매체 플러그가 접종된 페트리 접시를 어둠 속에서 25°C에서 최소 72시간 동안 배양합니다.
13. 미생물 분리를 위한 계대배양
참고: 이 단계는 선택 사항입니다.
- 박테리아의 경우: 멸균 접종 루프를 사용하여 페트리 접시의 다른 영역과의 접촉을 최소화하면서 관심 있는 단일 군체를 가로질러 스와이프합니다. 단일 콜로니가 형성될 수 있는 방법(예: 4개 구역으로 줄무늬)을 사용하여 R2A(5단계에서 준비)와 같은 선호되는 박테리아 배지가 포함된 신선한 90mm 페트리 접시에 콜로니를 줄무늬로 만듭니다. 페트리 접시를 25-37 ° C에서 24-72 시간 동안 배양하여 식민지 성장을 확인합니다.
- 곰팡이의 경우: 멸균 접종 루프, 멸균 면도기 또는 멸균 피펫 팁을 사용하여 균사 성장을 포함하는 한천의 최소한(1mm2) 부분을 조각합니다. 90단계에서 준비한 대로 MEA 또는 PDA와 같은 선호하는 곰팡이 배지가 들어 있는 신선한 5mm 페트리 접시에 작은 한천 조각을 놓습니다. 페트리 접시를 어둠 속에서 25 ° C에서 최대 1 주일 동안 부화시키고 과잉 성장을 방지하기 위해 매일 확인하십시오.
- 유기체 형태가 균일해질 때까지 필요에 따라 1단계 또는 2단계를 반복합니다.
14. 플레이트에서 또는 타겟 매체에서 직접 DNA를 추출합니다.
- 전체 한천 플러그(표적 및/또는 미끼) 또는 1.5mL 원심분리 튜브의 컬럼에서 선택한 조각을 -20°C에서 동결하거나 추출이 즉시 이루어지지 않는 경우 추출하기 전에 1.5mL 원심분리 튜브의 방부제에 플러그 조각을 담그십시오.
- 표적 및/또는 미끼 배지에서 배양이 수행된 경우, 후속 하위 배양 단계를 수행한 경우, 곰팡이 또는 박테리아 성장이 있는 ~1cm 한천 조각을 스탬프로 찍거나 조각합니다(또는 후속 분리 단계를 수행하지 않은 경우 둘 다의 혼합).
- 분리된 박테리아 콜로니를 추출하려면 멸균 접종 루프를 사용하여 플레이트에서 콜로니를 스와이프하고 상용 DNA 추출 키트와 관련된 추출 버퍼에서 직접 소용돌이칩니다(단계 14.4).
- 모르타르와 절구를 사용하여 한천 조각을 액체 질소로 개별적으로 분쇄하고 분쇄 조직을 추출 튜브로 옮깁니다.
- 토양 또는 박테리아 및 곰팡이에 최적화된 상용 DNA 추출 키트를 사용하고 제조업체의 지침에 따라 DNA를 추출합니다( 재료 표 참조).
- 결과 DNA를 형광측정기 또는 유사한 시스템을 사용하여 정량화합니다.
15. 앰플리콘 또는 메타유전체 염기서열분석 접근법을 사용하여 표적 및/또는 미끼 배지의 미생물 분류학적 다양성 평가
- 아래 단계에 따라 앰플리콘 염기서열분석(16S 및/또는 내부 전사 스페이서[ITS]) 또는 메타유전체 염기서열분석을 수행합니다.
- 앰플리콘 염기서열분석: 프라이머 Bakt 341F(5'-CCT ACG GGN GGC WGC AG-3') 및 Bakt 805R(5'-GAC TAC HVG GGT, ATC TAA TCC-3')을 사용하여 V3-V4 16S rRNA 유전자 영역을 증폭합니다. 프라이머 ITS3, KYO2(5'-GAT, GAA, GAA, CGY, AGY, RAA-3') 및 ITS4(5'-TCC, TCC, GCT, TAT, TGA, TAT, GC-3')9를 사용하여 진균 ITS2 영역을 증폭합니다.
- 선택한 염기서열분석기와 호환되는 상용 라이브러리 준비 키트를 사용하여 앰플리콘 라이브러리를 준비합니다. 짧은 판독 염기서열분석기를 사용하여 앰플리콘을 염기서열분석하고, 염기서열분석 플랫폼 제조업체의 로딩 농도에 대한 지침에 따라 충분한 커버리지를 가진 150 또는 250 염기쌍 쌍 판독을 생성합니다.
- 메타유전체(metagenomic) 염기서열분석(sequogenic sequencing): 선택한 염기서열분석기와 호환되는 상업적으로 이용 가능한 메타유전체학(metagenomic) 라이브러리 준비 키트를 사용하여 추출된 DNA로부터 메타유전체학(metagenomic) 라이브러리를 생성합니다. 로딩 농도에 대한 염기서열분석 플랫폼 제조업체의 지침에 따라 충분한 적용 범위(메타게놈당 ~10-20Gb)로 150 또는 250 염기쌍 쌍 말단 판독을 생성하도록 설정된 짧은 판독 시퀀서를 사용하여 메타게놈 라이브러리를 시퀀싱합니다.
16. 염기서열분석 데이터 분석
- 앰플리콘 데이터 분석: 결과를 시각화하기 전에 QIIME2 플랫폼11 을 DADA212 와 함께 활용하여 앰플리콘 데이터를 분석한다13. EDGE(Empowering the Development of Genomics Expertise)14 웹 기반 생물정보학 플랫폼에서 QIIME2를 실행하려면 아래 단계를 따르십시오. QIIME2는 온라인으로 제공되는 "동영상" 가이드(https://docs.qiime2.org/2024.2/tutorials/moving-pictures/)와 같이 공개적으로 사용 가능한 자습서를 사용하여 실행할 수도 있습니다.
- https://edgebioinformatics.org/ 로 이동하여 로그인하거나 계정을 만듭니다.
- 홈페이지에서 RUN QIIME2 를 선택합니다. 왼쪽 메뉴 모음에서 Upload Files(파일 업로드 )를 선택하여 amplicon 염기서열분석 실행에서 파일을 업로드합니다.
- 'Metadata Mapping File' 옆의 'i'를 마우스로 가리킬 때 제공되는 지침에 따라 메타데이터 매핑 파일을 만듭니다.
- 필요한 모든 정보(프로젝트/실행 이름, 읽기 유형, 매개 변수 등)를 추가하고 업로드된 올바른 입력 데이터를 선택합니다. DADA2가 품질 관리 방법12로 선택되었는지 확인하십시오. 다른 매개변수는 다른 수정이 필요하지 않는 한 기본값으로 둘 수 있습니다.
- 박테리아 앰플리콘의 경우 16S Greengenes (http://greengenes.lbl.gov) 또는 진균 앰플리콘의 경우 Fungal ITS로 앰플리콘 유형을 선택합니다. 박테리아(16S) 및 곰팡이(ITS) 데이터를 독립적으로 분석합니다.
- 제출을 누르고 실행이 완료될 때까지 기다렸다가 결과를 확인합니다.
- 앰플리콘 데이터 시각화 생성(선택 사항): GitHub에 제공된 지침 및 R Studio에서 사용할 수 있는 R 패키지 도움말을 통해 패키지 메뉴로 이동하여 다운로드한 패키지를 클릭하고 설명서를 확인하여 phyloseq(https://github.com/joey711/phyloseq), VEGAN(https://github.com/vegandevs/vegan) 및 APE(https://emmanuelparadis.github.io/)와 같은 일반 패키지를 사용하여 R에서 추가 커뮤니티 데이터 분석을 수행합니다.
- 메타유전체학(metagenomic) 데이터 분석
- NMDC EDGE 사이트15(https://nmdc-edge.org/home)로 이동하여 ORCiD 계정(https://orcid.org/)을 사용하여 로그인합니다.
- 화면 왼쪽의 메뉴 표시줄에서 파일 업로드 를 선택하고 올바른 입력 FASTQ 파일을 끌어다 놓거나 찾습니다.
- Metagenomics(균유전체학)를 선택한 다음, 화면 왼쪽의 메뉴 표시줄에서 Run Multiple Workflows(여러 워크플로우 실행) 옵션을 선택하고 모든 워크플로우를 On(켜기)으로 설정합니다. 프로젝트 이름과 선택적 설명을 추가합니다.
- 업로드된 원시 읽기(fastq) 파일을 선택하고 적절한 파일 형식(인터리브 또는 페어링)을 선택합니다.
- 제출을 클릭하고 실행이 완료되면 화면 맨 위에 있는 내 프로젝트 탭에서 프로젝트를 선택하여 요약 테이블 및 시각화를 확인하여 실행을 시작합니다.
17. 앰플리콘 및/또는 메타유전체학(metagenomic) 결과에서 분류 데이터의 추가 시각화 생성
- GitHub에 제공된 지침에 따라 GraPhalan(https://github.com/biobakery/graphlan) 패키지를 사용하여 원형 클라도그램을 생성합니다.
참고: NCBI(National Center for Biotechnology Information) 분류학(https://www.ncbi.nlm.nih.gov/taxonomy) 식별자는 대표 염기서열 할당에서 얻을 수 있으며 Entrez Direct E-utilities의 'eftech' 프로그램으로 전달하여 GraPhlAn16에서 요구하는 분류 롤업 정보를 수집할 수 있습니다.
18. 열 재사용
- 기둥이 여전히 조립되어 있으면 나사를 풀고 캡, 나사산 링 및 메쉬를 제거합니다. 메시를 버립니다. 남아 있는 한천 플러그를 제거하고 99% 이소프로필 알코올과 정제수로 컬럼을 세척한 다음 3.1-3.4단계에 설명된 대로 구성 요소를 세척 및 건조합니다.
- 고압증기멸균할 새 나일론 메쉬 시트를 준비합니다.
- 4.1단계에서 제공된 지침에 따라 구성 요소를 오토클레이브합니다.
결과
완전히 조립된 곰팡이 고속도로 기둥의 길이는 약 5cm입니다(그림 1). 컬럼은 어떤 영역에서도 파손되어서는 안 되며 캡과 나사산 링이 쉽고 단단히 맞아 컬럼 내부에 미세 환경을 생성해야 합니다. 필터 메쉬는 나사산 링을 넘어 확장되거나( 그림 1 및 그림 2 참조) 멸균된 가위로 트리밍할 수 있습니다. 한천 플러그는 컬럼의 양쪽 끝에 꼭 맞아야 합니다. 기판에 배치할 때 필터 메쉬가 기판과 접촉해야 하며 컬럼이 완전히 묻히지 않아야 합니다.
기둥은 이전에 말똥9에서 테스트되었습니다. 컬럼은 또한 연구 현장 현장의 벌크 및 뿌리권 토양과 실험실의 50mL 튜브에 있는 소량의 토양에 배치되었습니다(그림 3). 곰팡이 고속도로 기둥을 기질에서 제거하고 분해한 후 미끼와 표적 매체 플러그 모두에서 미생물 성장이 관찰되었습니다(그림 4A에 표시된 예). 박테리아와 곰팡이는 subculturing 기술을 통해 표적 및 미끼 배지에서 분리되었으며(그림 4B), 배지 플러그에 존재하는 미생물은 앰플리콘 염기서열분석을 사용하여 분류학적으로 식별되었습니다(그림 4C, D). 그림 4C,D는 여러 실험에 걸친 앰플리콘 염기서열분석의 결합된 결과를 보여주며, 어떤 미생물이 말똥9에 첨가된 컬럼에서 표적 배지 플러그에 도달할 수 있었는지를 보여줍니다. 이 박테리아 및 진균 데이터의 시각화는 17단계에서 설명한 대로 생성되었습니다. 결과는 분류군의 상대적 풍부도로 표시될 수도 있습니다.
습도가 극도로 낮은 환경에 컬럼을 추가하고 미디어 플러그가 며칠 내에 완전히 건조되어 집락화된 미생물이 회수되지 않는 경우 최적이 아닌 결과를 얻었습니다(그림 5A). 또한 미생물이 타겟 미디어 플러그에서 성장하지 않는 경우(그림 5B)와 의미 있는 분석을 위해 타겟 미디어 플러그에서 충분한 염기서열분석 데이터를 복구하지 못하는 경우도 보았습니다. 곰팡이가 기둥에서 과도하게 자라는 다른 경우에도 실험을 다시 수행해야 했습니다(그림 5C).

그림 3: 실험실 및 현장 환경에서 환경 샘플에 배치된 컬럼의 예. (A) 실험실 환경에서 축축한 토양이 있는 50mL 튜브 내부에 배치된 컬럼. 또한 크기에 대한 눈금자와 함께 표시됩니다. (B) 밭의 토양에 놓인 기둥. (C) 밭에 있는 식물의 뿌리 네트워크에 배치된 기둥. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
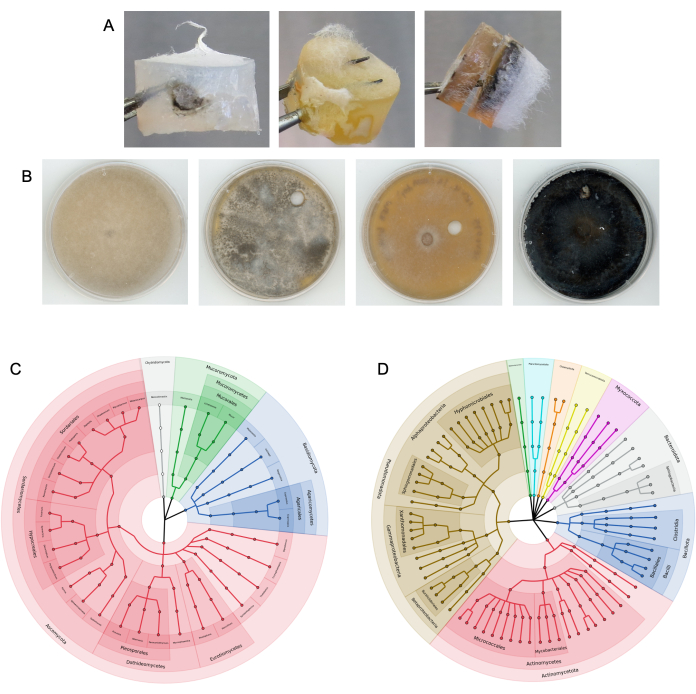
그림 4: 성공적인 컬럼 실험의 대표적인 결과. (A) 컬럼에서 추출한 집락화된 배지 플러그의 예. (B) 표적 배지에서 하대배양된 진균 분리물의 예. 기질은 흙이었습니다. 맨 위 ITS 염기서열 NCBI BLAST 정체성 왼쪽에서 방향으로: Rhizopus azygosporous, Aspergillus novofumigatus, Curvularia subpapendorfii, Phaeomycocentrospora cantuariensis. (C,D) 말똥을 사용한 여러 진균 고속도로 실험에 따라 표적 배지에서 회수된 (C) 진균 ITS 및 (D) 박테리아 16S 염기서열의 계통발생학적 다양성을 보여주는 원형 클라도그램. 섹션은 문에 의해 색상이 지정되고 레이블이 지정되며 끝 노드는 고유한 속을 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
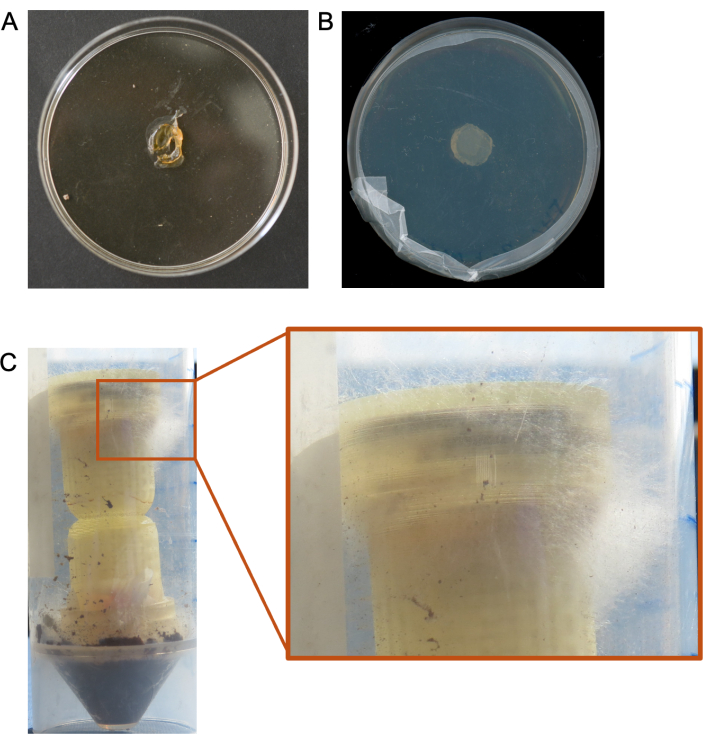
그림 5. 컬럼 실험에서 최적이 아닌 결과. (A) 낮은 습도의 환경 조건으로 인한 건조된 매체 플러그의 예. (B) 컬럼 배지 플러그에서 미생물이 성장하지 않는 예. (C) 기둥의 상단(대상 매체)을 통해 곰팡이가 과도하게 증식하는 예. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
컬럼 구성 요소를 생성할 때 3D 프린터 및 인쇄 재료의 선택은 가용성 및 원하는 재료 특성에 따라 수정할 수 있습니다17,18. 생체 적합성, 표면 질감, 오토클레이브성, 미세한 세부 사항을 인쇄할 수 있는 능력 및 상대적 투명도는 모두 우리 그룹의 재료 선택에서 고려되었습니다. 다공성(porosity), 소수성(hydrophobicity), 인쇄 매개변수 등과 같은 다른 기능도 고려해야 합니다. 최종 선택 전에 다양한 수지를 테스트했으며(재료 표 참조) 많은 생체 적합성 재료가 이러한 컬럼의 인쇄에 사용할 수 있습니다. 컬럼 구성 요소의 구성을 위해 선택한 재료에 따라 어떤 세척, 경화 후 및 멸균 방법을 사용해야 하는지 결정됩니다. 모든 재료가 고압멸균이 가능한 것은 아니며 재료 제조업체의 지침에 따라 자외선, 표백제 또는 기타 살균 기술이 필요할 수 있습니다. 일부 멸균 또는 세척 기술은 선택한 재료를 손상시키거나 호환되지 않을 수도 있으므로 재료 제조업체에서 이 정보에 특별한 주의를 기울여야 합니다. 3D 프린터의 경우 몇 가지 고려 사항에는 인쇄 시간, 재료 호환성, 빌드 플랫폼 크기, 인쇄 기술 및 비용19가 포함됩니다. 3D 프린팅으로 제작된 기둥 구성 요소는 깨지기 쉬우며 너무 세게 다루면 파손될 수 있습니다. 링과 캡의 나사산이 항상 정확하게 정렬되는 것은 아니므로 조립 단계 전에 추가 부품을 인쇄하고 멸균하거나 선택한 매개변수와 재료가 나사산에 어떤 영향을 미치는지 테스트하기 위해 예비 인쇄를 수행하는 것이 좋습니다. 캡과 링 내부의 나사산에 대한 설계 사양은 선택한 3D 프린팅 재료에 따라 조정해야 할 수 있습니다. 치수, 격자 복잡성 및 기타 물리적 특징은 모두 인쇄 전에 CAD 설계 소프트웨어에서 수정할 수 있습니다. 설계한 바와 같이 기둥 자체의 높이는 4cm이고, 기둥 중앙 내의 격자 구조는 2mm 크기의 단위 셀, 0.5mm의 스트럿 직경, 전체 격자 높이는 22mm입니다9. 이러한 매개변수는 연구자가 예를 들어 더 크거나 더 복잡한 격자 구조를 원하는 경우 조정할 수 있습니다. 전반적으로 이러한 장치의 3D 프린팅 제조는 설계 유연성을 가능하게 하는 동시에 단일 설계를 조직 및 그룹 전반에 걸쳐 표준화된 방식으로 사용할 수 있도록 하고 교실 교육 도구로도 사용할 수 있도록 합니다9.
프로토콜의 여러 단계에서 환경 또는 실험 설정에 따라 문제 해결이 필요할 수 있습니다. 곰팡이 고속도로 기둥은 곰팡이 성장을 촉진하기 전에 매체 플러그가 빠르게 건조되어 이러한 환경에서의 실험 기간을 제한할 수 있기 때문에 습도가 낮은 조건에서는 그다지 효과적이지 않습니다(그림 5A). 습도가 낮은 환경에서 컬럼의 효과를 개선한 기술에는 기질에 수분을 추가하여 인위적으로 습도를 높이거나 수원(예: 작은 순수 용기)으로 2차 용기에서 컬럼과 기질을 밀봉하는 것이 포함됩니다. 모래시계 모양과 격자 구조는 습도가 높은 환경에서 응결이 형성되는 경우 박테리아 이동만으로(곰팡이 고속도로를 설정하지 않고) 박테리아 이동을 방지하기 위해 통합되었습니다. 빠르게 자라는 곰팡이는 표적 및 미끼 매체 표면적을 과도하게 성장시켜 컬럼의 상단 또는 하단으로 확장될 수 있습니다(그림 5C). 미끼 곰팡이의 배양 시간이나 실험 기간을 줄이면 이러한 과잉 성장을 최소화하거나 제거할 수 있습니다. 또한 이러한 장치의 한계는 관심 기질에서 빠르게 성장하는 곰팡이가 느리게 성장하는 곰팡이에 의한 미끼 및 표적 매체의 집락화를 제한할 수 있으며, 잠재적으로 관찰되는 고속도로 상호 작용에 편향을 일으킬 수 있다는 것입니다. 일부 곰팡이, 특히 느리게 자라는 곰팡이는 한천 플러그를 통해 격자 구조로 자랄 수 있는 방식으로 미끼 배지를 식민지화하지 않을 수 있습니다. 환경에 충분한 습도가 있는 경우 더 얇은 한천 플러그를 사용하여 미끼 한천 플러그 집락화 후 격자로의 성장을 촉진할 수 있습니다. 연구자가 곰팡이 또는 박테리아 성장을 위해 선택하기를 원하는지 여부에 따라 배지를 선택할 수 있지만, 이는 또한 해당 배지 유형20을 선호하는 유기체에 대한 하위 배양을 제한할 수 있습니다. 표적 배지에서 성장이 보이지 않으면 미끼 배지 또는 기질에 곰팡이 고속도로를 생성하는 것으로 알려진 곰팡이를 접종해야 할 수 있습니다.
이러한 실험의 일환으로 메타게놈(metagenomic) 또는 앰플리콘(amplicon) 염기서열분석을 수행할 수 있으며, 이 두 가지 전략 모두 고유한 한계와 강점을 가지고 있습니다21. 균유전체 염기서열분석(metagenomic sequencing)은 미생물에 대한 추가적인 게놈 정보를 얻는 데 이상적입니다. 그러나, 타겟 배지에서 직접 회수할 수 있는 핵산의 양은 매우 낮을 수 있으며, 이는 염기서열분석 전에 앰플리콘 염기서열분석 또는 다른 증폭 방법의 활용이 필요할 수 있습니다. Amplicon 염기서열분석 라이브러리는 별도로 준비해야 하며(16S 및 ITS), 이 방법은 분류학적 해상도가 부족하고 메타유전체 염기서열분석을 사용하여 달성할 수 있는 게놈 특징 또는 기능 잠재력에 대한 평가를 제한합니다. 플러그로부터의 직접 시퀀싱 방법은 미생물을 하대배양할 수 없는 경우에 선호될 수 있습니다. 배양 및 염기서열분석 접근 방식을 모두 사용할 수 있도록 플러그를 여러 섹션으로 분할하는 것이 좋습니다.
이러한 장치의 장점은 실험실과 현장 모두에서 사용할 수 있다는 것입니다. 필드의 기둥이 똑바로 유지될 수 있고 배치를 방해할 수 있는 동물 및 환경 교란으로부터 보호되도록 특별한 주의를 기울여야 합니다. 컬럼은 기판으로 완전히 덮인 수평 위치에서 아직 테스트되지 않았으며 상당한 강우량이나 눈에 노출되는 환경에서 테스트되지 않았습니다. 위에서 언급한 바와 같이, 격자 구조는 습도가 높은 환경에서 박테리아가 대상 매질로 이동할 수 있는 가능성을 최소화하도록 설계되었습니다. 그러나 컬럼이 더 많은 양의 물에 노출되고 이 물이 컬럼을 완전히 포화시킨 경우 존재하는 곰팡이 고속도로와 무관하게 컬럼 전체에서 박테리아 이동이 촉진될 수 있습니다. 실험실 기반 실험의 경우, 컬럼은 50mL 원뿔형 튜브, 기질의 작은 소우주 내부, 화분에 심은 식물 주변의 토양, 상자 또는 기타 통제된 실험 시스템 내에서 사용할 수 있습니다. 기둥은 토양, 뿌리 줄기 및 배설물에서 성공적으로 활용되었으며 그 유용성은 잎 쓰레기, 슬러지, 모래, 눈, 퇴비 등을 포함한 다른 기질로 확장될 수 있습니다.
fungal highway column을 사용하면 다양한 샘플 유형 내에서 이 BFI 표현형을 이해하기 위한 여러 비교를 수행할 수 있습니다. 미끼와 표적 배지 사이의 군집 구성을 비교하면 어떤 박테리아가 곰팡이 고속도로를 이용할 수 있고 어떤 곰팡이가 잠재적인 고속도로 역할을 할 수 있는지 나타낼 수 있습니다9. 메타게놈 염기서열분석(metagenome sequencing)을 사용하는 경우, 미생물을 미끼와 표적 배지와 구별하는 게놈 특징도 조사할 수 있습니다. 또한 다른 기질(예: 토양 대 배설물)에 배치되거나 다른 조건(예: 온도 또는 습도)에서 동일한 기질에 배치된 컬럼의 타겟 배지를 비교할 수도 있습니다. 전반적으로 곰팡이 고속도로 칼럼은 이러한 형태의 BFI를 조사하는 이전 방법의 기능을 확장하고 복잡한 환경 미생물 군집의 공간 역학을 형성하는 이러한 상호 작용에 대한 광범위한 검사를 가능하게 합니다.
공개
저자는 공개할 이해 상충이 없습니다.
감사의 말
이 연구는 미국 에너지부(DOE), 생물 및 환경 연구(BER), 생물 시스템 과학과(BSSD)의 과학 초점 영역 보조금(Science Focus Area Grant)의 지원을 받았습니다(보조금 번호 LANLF59T).
자료
| Name | Company | Catalog Number | Comments |
| 50 mL tubes | Greiner BIO-ONE | 5622-7261 | 50 mL tubes for performing column experiments in the lab |
| 90 mm Petri dishes | Thermo Scientific Nunc | 08-757-099 | Petri dishes for preparation of agar and for microbial growth |
| Asiga Freeform Pico Plus 39 digital light processing (DLP) 3D printer | Asiga Germany | Freeform Pico Plus 39 | 3D printer used to generate batches of the columns; other 3D printers can be used |
| Autoclave | Fisher Scientific | LS40F20 | Benchtop autoclave to sterilize the column components |
| Beaker | Fisher Scientific | FB100600 | 600 mL beaker for various uses throughout the protocol |
| Dental LT Clear Resin V2 | Formlabs | RS-F2-DLCL-02 | Alternative resin for 3D printing that was tested |
| Dental Surgical Guide Resin | Formlabs | RS-F2-SGAM-01 | Was used to generate the columns discussed in manuscript; Other photosensitive resins can be used in place of this material |
| DNA Low Bind 1.5 mL tubes | Eppendorf | 13-698-791 | Tubes used for various preparations including nucleic acid extractions |
| DNA/RNA shield preservative | Zymo Research | R1100-50 | Preservative used prior to nucleic acid extractions |
| EDGE Bioinformatics | Open source; Developed by the Los Alamos National Laboratory (LANL) | n/a | Bioinformatics platform for processing amplicon data |
| FastDNA spin kit for soil | MP Biomedicals LLC | 116560200-CF | DNA extraction kit option for soil |
| Forceps | Fisher Scientific | 10-300 | Forceps that can be sterilized |
| Formlabs BioMed Clear Resin | Formlabs | RS-F2-BMCL-01 | Alternative resin for 3D printing that was tested |
| Formlabs Form 3B+ stereolithography (SLA) 3D printer | Formlabs | Form 3B+ | Alternative 3D printer |
| Formlabs IBT Resin | Formlabs | RS-F2-IBCL-01 | Alternative resin for 3D printing that was tested |
| Inoculating Loops | Fisher Scientific | 22-363-598 | Used to isolate/transfer microbes |
| Malt Extract Agar (MEA) | Criterion | 89405-654 | A media type used in columns |
| MiSeq sequencer + MiSeq sequencing kit | Illumina | SY-410-1003 | Can use other sequencers |
| Mortar & Pestle | Fisher Scientific | FB961K; FB961A | Can use any common mortar & pestle that can be sterilized between uses |
| NEBNext Ultra II DNA Library Prep Kit for Illumina | New England Biolabs | E7805S | Library prep kit for metagenomic sequencing |
| Nextera XT DNA Library Preparation Kit (24 samples) | Illumina | FC-131-1024 | Library prep kit for amplicon sequencing |
| NMDC EDGE | Open source: Developed by the National Microbiome Data Collaborative (NMDC) | n/a | Bioinformatics platform for processing metagenomic data |
| Nylon mesh | Sefar | 03-25/19 | The mesh used as part of the column construction |
| Pipette tips | Rainin | 30807966 | Can use many different sterilized pipette tips for the protocol steps |
| Potato Dextrose Agar | Cole Parmer | EW-14200-28 | A media type used in columns |
| QIIME2 | Open source | n/a | Software for processing amplicon data |
| Qubit dsDNA HS assay kit | Thermo Fisher Scientific | Q32851 | Used to quantify DNA after extractions |
| Qubit Fluorometer | Thermo Fisher Scientific | Q33238 | Used to quantify DNA after extractions |
| Quick-DNA Fungal/Bacterial Miniprep Kit | Zymo Research | D6005 | DNA extraction kit option that works with both bacteria and fungi |
| R2A agar | BD Difco | 218263 | A media type used in columns (bacterial media) |
| Rack for 50 mL tubes | Fisher Scientific | 03-448-11 | Rack to hold 50 mL tubes upright |
| Scissors | Fisher Scientific | 12-000-155 | Fine precision scissors that can be sterilized |
| Sodium carboxymethyl cellulose medium | Aldrich | 419273-100G | A media type used in columns |
| SolidWorks CAD software | SolidWorks | n/a | Software used to design the columns |
| Trowel scoop | Fisher Scientific | S41701 | To make a depression in the substrate prior to adding the column |
| UltraPure DNase/RNase-Free Distilled Water | Invitrogen: ThermoFisher Scientific | 10977015 | Water for the ultrasonicator water bath |
| Ultrasonicator | Fisher Scientific | FB-11201 | Ultrasonicator for cleaning the columns |
참고문헌
- Or, D., Smets, B. F., Wraith, J. M., Dechesne, A., Friedman, S. P. Physical constraints affecting bacterial habitats and activity in unsaturated porous media - A review. Adv Water Resour. 30 (6), 1505-1527 (2007).
- Kohlmeier, S., et al. Taking the fungal highway: Mobilization of pollutant-degrading bacteria by fungi. Environ Sci Technol. 39 (12), 4640-4646 (2005).
- Simon, A., Hervé, V., Al-Dourobi, A., Verrecchia, E., Junier, P. An in situ inventory of fungi and their associated migrating bacteria in forest soils using fungal highway columns. FEMS Microbiol Ecol. 93 (1), 217 (2017).
- Wick, L. Y., et al. Effect of fungal hyphae on the access of bacteria to phenanthrene in soil. Environ Sci Technol. 41 (2), 500-505 (2007).
- Bravo, D., et al. Isolation of oxalotrophic bacteria able to disperse on fungal mycelium. FEMS Microbiol Lett. 348 (2), 157-166 (2013).
- Furuno, S., Remer, R., Chatzinotas, A., Harms, H., Wick, L. Y. Use of mycelia as paths for the isolation of contaminant-degrading bacteria from soil. Microb Biotechnol. 5 (1), 142-148 (2012).
- Buffi, M., et al. Fungal drops: A novel approach for macro- and microscopic analyses of fungal mycelial growth. Microlife. 4, 042 (2023).
- Kuhn, T., et al. Design and construction of 3D printed devices to investigate active and passive bacterial dispersal on hydrated surfaces. BMC Biol. 20 (1), 203 (2022).
- Junier, P., et al. Democratization of fungal highway columns as a tool to investigate bacteria associated with soil fungi. FEMS Microbiol Ecol. 97 (2), 003 (2021).
- Reasoner, D. J., Geldreich, E. E. A new medium for the enumeration and subculture of bacteria from potable water. Appl Environ Microbiol. 49 (1), 1-7 (1985).
- Bolyen, E., et al. Reproducible, interactive, scalable, and extensible microbiome data science using QIIME 2. Nat Biotechnol. 37 (8), 852-857 (2019).
- Callahan, B. J., et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nat Methods. 13 (7), 581-583 (2016).
- Vázquez-Baeza, Y., Pirrung, M., Gonzalez, A., Knight, R. EMPeror: A tool for visualizing high-throughput microbial community data. Gigascience. 2 (1), 16 (2013).
- Li, P. -. E., et al. Enabling the democratization of the genomics revolution with a fully integrated web-based bioinformatics platform. Nucleic Acids Res. 45 (1), 67-80 (2017).
- Eloe-Fadrosh, E. A., et al. The National Microbiome Data Collaborative Data Portal: An integrated multi-omics microbiome data resource. Nucleic Acids Res. 50 (1), D828-D836 (2022).
- Entrez Direct: E-utilities on the Unix Command Line. Entrez Programming Utilities Help Available from: https://www.ncbi.nlm.nih.gov/books/NBK179288/ (2024)
- Palmara, G., Frascella, F., Roppolo, I., Chiappone, A., Chiadò, A. Functional 3D printing: Approaches and bioapplications. Biosens Bioelectron. 175, 112849 (2021).
- Guttridge, C., Shannon, A., O'Sullivan, A., O'Sullivan, K. J., O'Sullivan, L. W. Biocompatible 3D printing resins for medical applications: A review of marketed intended use, biocompatibility certification, and post-processing guidance. Ann 3D Print Med. 5, 100044 (2022).
- Yao, L., et al. Comparison of accuracy and precision of various types of photo-curing printing technology. J Phys Conf Ser. 1549 (3), 032151 (2020).
- Basu, S., et al. Evolution of bacterial and fungal growth media. Bioinformation. 11 (4), 182-184 (2015).
- Liu, Y. -. X., et al. A practical guide to amplicon and metagenomic analysis of microbiome data. Protein Cell. 12 (5), 315-330 (2021).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유