Method Article
Farklı Ortamlarda ve Yüzeylerde Mantar Otoyol Kolonları Kullanılarak Bakteri-Mantar Etkileşimlerinin İncelenmesi
Bu Makalede
Özet
Bu protokol, çeşitli çevresel substratlardan mantar otoyolları yoluyla etkileşime giren bakteriyel-mantar çiftlerini zenginleştirmek için mantar otoyol kolonlarının nasıl oluşturulacağı, sterilize edileceği, birleştirileceği, kullanılacağı ve yeniden kullanılacağı hakkında ayrıntılı talimatlar sağlar.
Özet
Bakteriyel-fungal etkileşimler (BFI'ler), mikrobiyal topluluk bileşimini, biyojeokimyasal fonksiyonları, mekansal dinamikleri ve mikrobiyal dağılımı şekillendirmede ayrılmaz bir rol oynar. Filamentli mantarlar veya diğer filamentli mikroorganizmalar (örneğin, Oomycetes) tarafından oluşturulan misel ağları, bakteriler tarafından heterojen ortamlarda taşınmak için kullanılabilen, hareketliliklerini büyük ölçüde kolaylaştıran ve kendi başlarına ulaşılması zor veya imkansız olabilecek bölgelere erişim sağlayan 'mantar otoyolları' görevi görür (örneğin, topraktaki hava cepleri nedeniyle). Mantar otoyol sütunları da dahil olmak üzere bu mantar otoyollarını incelemek için çeşitli cihazlar ve deneysel protokoller oluşturulmuştur. Grubumuz tarafından tasarlanan mantar otoyol kolonu, çeşitli yerinde veya in vitro uygulamaların yanı sıra çeşitli çevresel ve konakçı ile ilişkili numune türleri için kullanılabilir. Burada, cihazların tasarlanması, yazdırılması, sterilize edilmesi ve hazırlanması dahil olmak üzere bu sütunlarla deney yapma yöntemlerini açıklıyoruz. Bu cihazların kullanımından elde edilen verileri analiz etme seçenekleri de burada tartışılmakta ve mantar otoyol sütunları kullanılarak yapılan deneylerle ilişkili olası tuzaklarla ilgili sorun giderme önerileri sunulmaktadır. Bu cihazlar, karmaşık ortamlardaki (örneğin topraklar) ve bakteri ve mantarların bir arada bulunduğu çeşitli habitatlardaki yapısal ve işlevsel dinamikler hakkında değerli bilgiler sağlamak için mantar otoyolu BFI'lerinin çeşitliliği, mekanizmaları ve dinamikleri hakkında daha kapsamlı bir anlayış kazanmak için kullanılabilir.
Giriş
Bakteriyel-fungal etkileşimler (BFI'ler), çevresel mikrobiyomların yapısal, mekansal ve fonksiyonel özelliklerini şekillendirmede son derece önemlidir. Örneğin, filamentli mantarların veya diğer mantar benzeri filamentli mikroorganizmaların büyümesi ve genişlemesi, bakteriler gibi diğer mikroorganizmaların hareketini kolaylaştırmak için bir 'otoyol' işlevi görebilen biyolojik bir ağ oluşturur. Çevresel substratlar içindeki heterojenlik ve tutarsız doygunluk, bakteri hareketliliğini engelleyebilir; Bununla birlikte, bakteriler bu otoyolları çevrenin ek alanlarına erişimi kolaylaştırmak için kullanabilir 1,2. Bu etkileşimler, mikrobiyal toplulukların mekansal dinamiklerini anlamak için kritik öneme sahiptir. Mantar otoyollarını incelemek için çeşitli teknikler ve yöntemler kullanılmıştır, ancak bunlar büyük ölçüde laboratuvar temelli araştırmalarla sınırlıdır 3,4.
Plaka bazlı bir yöntemde, Petri kabının ortasından büyük bir agar bölümü çıkarılır ve iki agar adası arasında bir boşluk oluşturulur. Mantar hifleri bu boşluğu geçebilir ve uyumlu bakterilerin bir agar adasından diğer5'e geçmesi için araçlar sağlar. Diğer modifiye edilmiş Petri-dish yöntemleri arasında, mantar hiflerinin dikey olarak büyüyebilmesi ve ortamı doğrudan temas olmadan kolonize edebilmesi ve bakteri taşınması için araçlar sağlaması için toprağın kapağa yerleştirildiği ters çevrilmiş plakalar bulunur 5,6. Yakın zamanda geliştirilen büyüme ortamı damlacık bazlı bir yöntem, bakterilerin belirli besin profillerine doğru seçici hifal taşınmasını değerlendirmek için kullanılabilir7. Abiyotik faktörlerin bakteri hareketi üzerindeki etkisini araştırmak için bakteri köprüsü ve iz cihazları da kullanılmıştır8. Mantar otoyollarını araştırmak için çeşitli yöntem ve teknikler kullanılmış olsa da, gübre, toprak ve rizosferler gibi karmaşık çevresel substratlardan mantar otoyollarının kurulmasını teşvik ederken steril bir mikro ortamı koruyan standartlaştırılmış cihazlara ihtiyaç vardır.
Grubumuz, mantarların bakterileri bir uçtan diğeruca taşıyabildiği mantar otoyol kolonlarının 3D baskılı bir versiyonunu tasarladı 9. Bu cihazlar dört basılı bileşenden monte edilir: bir kum saati şeklinde ve karmaşık bir iç kafes yapısına sahip sütunun kendisi, dişli bir halka ve iki kapak (büyük bir kapak ve küçük bir kapak) ve ayrıca bir parça sterilize edilmiş naylon ağ (Şekil 1). Monte edilen kolon doğrudan istenen çevresel alt tabakaya eklenir. Kolon daha sonra mikropların, kolonun altında bulunan ve ağ boyunca çevresel substrat ile temas halinde olan 'yem' medya tapası olarak bilinen bir agar büyüme ortamı tıkacını kolonize etmesine izin verir. Bu naylon ağ boyutu, bakterileri taşıyabilen diğer toprak sakinlerini dışlar, böylece sütunlar içindeki bakteri hareketini mantar otoyollarına sınırlar. Bu yem tıkacı kolonize edildikten sonra, filamentli mantarlar, toprağa (veya diğer doymamış ortamlara) benzeyen doymamış bir sistem oluşturmak ve yem ortamından kaynaklanan potansiyel kontaminasyonu en aza indirmek için tasarlanmış kolonun ortasındaki iç kafes boyunca uzayabilir ve büyüyebilir. Mantarlar daha sonra kolonun üst kısmındaki hedef ortam tıkacına doğru büyür ve kolonize olur. Sütunlar, bakteri taşıma yeteneklerini test etmek için spesifik mantar izolatları ile aşılanabilir veya substrattan hangi mantarların bakteri taşıyabildiğini belirlemek için aşılanmadan bırakılabilir. Hedef ortama ulaşan organizmalar daha fazla kültürlenebilir, izole edilebilir ve dizileme analizlerine tabi tutulabilir (saf kültürlerden veya amplikon veya metagenomik dizileme yaklaşımları kullanılarak karışık topluluklardan). Genel olarak, sütunlar, çeşitli yüzeylerdeki mantar otoyollarını sorgulamak için standartlaştırılmış, tekrarlanabilir, yeniden kullanılabilir ve sezgisel bir yöntem sağlar. Bu cihazlar araştırma için ve bir sınıf öğretim aracı olarak kullanılabilir ve burada, geçmişte gerçekleştirilen deneylere dayalı olarak bunları kullanmak için öğretim adımları sunuyoruz. Bu yöntem protokol standardizasyonunu kolaylaştırsa da, cihazların tasarımı ve yapısı diğer uygulamalar ve ek alt tabakalar için değiştirilebilir.
Protokol
Çalışmada kullanılan reaktiflerin ve ekipmanların detayları Malzeme Tablosunda listelenmiştir.
1. Sütun tasarımını, malzemelerini ve parametrelerini değiştirme
- Genel kullanıma açık sütun tasarımlarını9 indirin ve olduğu gibi kullanın veya sütun tasarımlarını uyumlu bilgisayar destekli tasarım (CAD) yazılımında değiştirin.
- Diş cerrahisi kılavuz reçinesi edinin veya diğer ışığa duyarlı şeffaf reçineler gibi alternatif bir 3D baskı malzemesi seçin.
- Seçilen 3D yazıcı, 3D baskı teknolojisi veya 3D baskı malzemesi daha önce kullanılandan değiştirilirse, sütunun özelliklerini gerektiği gibi ayarlayın9.
2. 3D Sütunların baskısı
- Yazdırma parametrelerini 0,05 mm dilim kalınlığı ve 0,8 sn pozlama süresi kullanacak şekilde ayarlayın veya seçilen yazıcıya, yazdırma malzemesine ve yazdırma yazılımına bağlı olarak yazdırma parametrelerini ayarlayın.
- Mantar otoyol sütunlarını, dişli halkaları ve kapakları uyumlu bir 3D yazıcı kullanarak yazdırın (Şekil 1).

Şekil 1: Mantar otoyol kolonunun bileşenleri. (A,B) Küçük kapağın, dişli halkanın ve büyük kapağın üstten ve yandan görünümleri (soldan sağa). (C,D) Küçük kapağın üstten ve yandan görünümü. (E,F) Dişli halkanın üstten ve yandan görünümü. (G,H) Büyük kapağın üstten ve yandan görünümü. (I) Kolonun ucuna yerleştirilmiş ve mikrofaunanın kolona girmesini önlemek için çevresel alt tabakaya yerleştirilmiş bir naylon ağ filtre (25 μm) parçası. (J) Demonte edilmemiş kolon. (K) Monte edilmiş sütun: 'Yem' ucu alt tabakaya girer ve 'Hedef' ucu açıkta ve alt tabakanın dışında kalır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
3. 3D yazdırılmış sütun bileşenlerinin temizlenmesi
- Basılı sütunları, kapakları ve dişli halkaları oda sıcaklığında 15 dakika boyunca %99 izopropil alkole batırın ve fazla reçineyi çıkarmak için banyoyu dakikada 10-15 saniye boyunca elle ileri geri hareket ettirerek çalkalayın.
- Bileşenleri taze bir %99 izopropil alkol banyosuna aktarın. 5 dakika daldırın ve adım 3.1'deki gibi elle çalkalayın.
- Bileşenleri saf suyla doldurulmuş bir ultrasonik temizleme cihazına aktarın, bileşenleri daldırın ve orta hız ayarında 2 dakika çalkalayın. Suyu ısıtmayın. Bileşenleri çıkarın.
- Tüm bileşenleri en az 30 dakika havayla kurutun.
- Reçine için son kürleme yapmak için, tüm 3D baskılı bileşenleri 60 °C'de 30 dakika boyunca 405 nm ışığa maruz bırakın.
4. Sütunların sterilize edilmesi
- Seçilen malzeme otoklavlanabilir ise, kolonları, dişli halkaları, kapakları ve 25 μm naylon ağ filtre tabakalarını ayrı ayrı veya daha büyük bir beher içinde 121 °C, 1 atm'de 20-30 dakika otoklavlayın.
NOT: Kolonun bileşenleri otoklavlamadan sonra şekil ve renk değiştirebilir, ancak istenen malzeme özelliklerini koruyacaklardır. Otoklavlamadan sonra kolon bileşenlerinin nihai boyutu, şekli ve rengi Şekil 1'de gösterilmiştir.
5. Kolonlar için ortam hazırlama
- Sterilize edilmiş agar bazlı ortamlardan 90 mm'lik Petri kapları hazırlayın: sodyum karboksimetil selüloz ortamı (CMC), Malt Özü Agar (MEA), Patates Dekstroz Agar (PDA) veya Reasoner'ın 2A agar (R2A)9,10. Üreticinin talimatlarına göre ortamı hazırlayın.
- Ortamı 21 dakika veya üretici tarafından önerilen süre boyunca 121 °C, 1 atm'de otoklavlayarak sterilize edin ve agar Petri kaplarının kenarlarının üst kısmına yakın olana kadar 90 mm Petri kaplarına dökün (Şekil 2). Steriliteyi artırmak için bu adımı bir biyolojik güvenlik kabininde gerçekleştirin.
- Agarın üreticinin talimatlarına göre katılaşmasına ve kurumasına izin verin.

Şekil 2: Mantar otoyol kolonları için montaj işlemi. Kolonun kendisinin açık bir ucu kullanılarak, bir tapa kesilir ve yerleştirilir ve araştırmacı, tapanın kolonun ucunda kalmasını sağlamak için kolonu ortamdan çıkarılırken büker. Bu uç küçük kapak parçası ile kapatılmıştır. Daha sonra sütunun diğer ucuna da aynı şekilde bir medya fişi eklenir. Ağ parçası daha sonra bu ucun üzerine yerleştirilir ve dişli halka ile sabitlenir. Büyük kapak daha sonra bu 'Yem' ucunda dişli halka üzerinde kullanılır. Ağın bulunduğu taraf, çevresel alt tabakaya yerleştirilecektir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
6. Mantar otoyol kolonlarının hazırlanması
NOT: Bu adım, kolon bileşenlerinin ve ortamın sterilitesini korumak için bir biyolojik güvenlik kabininde gerçekleştirilmelidir. Şekil 2 , mantar otoyol kolonunun montaj sürecini göstermektedir.
- Sütunun bir ucuna sıkıca oturan bir ortam tapasını çıkarmak için sütun ucunun kendisini (üzerinde herhangi bir kapak olmadan) kullanın. Kolon ortamdan kaldırılırken, agar kolon ucunun içinde kalacak şekilde bir büküm hareketi kullanın. Alternatif olarak, sütunun ucunu şablon olarak kullanın, ortamı kesin ve bir pipet ucu kullanarak sütuna aktarın.
- Kolonun ucuna ilk fiş eklendikten sonra, hedef ortam için steril bir mikro ortam sağlamak için küçük kapağı bu uca ekleyin.
- Sütunu ters çevirin ve sütunun diğer ucu için adım 6.1'i yineleyin.
- Sterilize makas kullanarak otoklavlanmış naylon ağın ~2 cm çapında dairesel bir parçasını (25 μm gözenek boyutu) kesin (Şekil 1). Ağı, sütunun açıkta kalan ucunun üzerine yerleştirin. Ağı ipliklerin içine sabitlerken dişli halkayı sütunun bu yem ortamı ucuna bükün.
- Kolonun altındaki büyük kapağı ağın üzerine ve dişli halkanın diğer ucuna yerleştirin ve kolonu saklarken veya taşırken bu kapağı açık tutun.
7. Substratın veya yem ortamının ilgilenilen bir mantarla önceden aşılanması
NOT: Bu adım isteğe bağlıdır.
- İlgilenilen bir mantarı (örneğin, mantar otoyolları oluşturduğu bilinen bir mantar) istenen substrat üzerine, mantarı önce katı bir mantar büyüme ortamı üzerinde büyüterek aşılayın (örneğin, MEA, PDA; adım 5'te açıklandığı gibi hazırlanır) bir Petri kabı içinde ve görünür mantar büyümesi olan küçük (~ 1 cm) bir agar bölümünü substrata aktarın.
NOT: Önceki deneyler9 , sütunları önceden kolonize edilmiş substrata eklemeden önce 10 gün boyunca Coprinopsis cinerea ile önceden kolonize edilmiş gübre substratı kullanmıştır. - Alt tabakayı önceden kolonize etmek yerine, az miktarda mantar miselini kullanarak (örneğin, steril bir döngünün kaydırılmasından) bir Petri kabına veya doğrudan sütun içindeki yem ortamının dibine bir yem mantarı ekleyin.
- Petri kabı için, Petri kabının büyük bir bölümünü kaplayan gözle görülür bir büyüme olana kadar bekleyin (kaplanan plakanın yaklaşık %50-75'i), ardından yem ortamı bölümünü doğrudan mantar büyümesinin dış kenarına en yakın kolonize Petri kabından damgalayın (adım 6.1'de açıklandığı gibi).
- Yem ortamını doğrudan ön kolonize ederken, sütunu alt tabakaya eklemeden önce yem ortamı boyunca net bir büyüme olmasını bekleyin (bu muhtemelen birkaç gün sürecektir).
NOT: Önceki deneyler9 , yem ortamı olarak C. cinerea ile önceden kolonize edilmiş 14 günlük ortamı kullanmıştır; Kolonizasyon öncesi süreler, mantarın büyüme hızına, ortam türüne ve kuluçka koşullarına bağlı olarak değişecektir.
8. Kontrol tedavilerinin ve replikalarının hazırlanması
- Negatif kontrol kolonları hazırlayın (adım 1-6'da açıklandığı gibi) ve bunları aşılamayın veya bu kolonları alt tabakaya yerleştirmeyin. Bunları, sonraki analizler için bir temel sağlamak ve hazırlama sürecindeki herhangi bir kontaminasyonu değerlendirmek için kullanın.
- Herhangi bir deneme için en az üç sütun çoğaltması ekleyin.
9. Sütunun alt tabakaya eklenmesi
- Bir laboratuvar mikrokozmosu kurun (örneğin, bir kutu, saksı veya tüp içindeki toprak; Şekil 3A) Veya sütunları ilgilendiğiniz bir alan sitesine getirin.
- Dişli halkayı ve ağı ortaya çıkarmak için sütunun altından büyük kapağı çıkarın.
- Aşağıda açıklanan her bir alt tabaka türü için özel hususları izleyerek sütunu ilgilenilen alt tabakaya ekleyin (Şekil 3). Gerekirse ve ağa verilen hasarı en aza indirmek için, kolonu alt tabakaya yerleştirmeden önce eldivenli bir el veya küçük bir alet (örneğin küçük bir mala) kullanarak alt tabakada bir çöküntü yapın.
- Gübre: Sütunlar daha önce taze at gübresinde başarıyla kullanılıyordu. Kullanmadan önce gübreyi 4 °C'de saklayın ve sütunları eklemeden önce Eflatun kutular gibi bölmelere ekleyin. Toplam sütun yüksekliğinin 1-2 cm'sini kaplayacak şekilde sütunu alt tabakaya yerleştirin.
- Toprak: Sütunları, tüm alt kısım toprağın içinde olacak şekilde (~1-2 cm toprak derinliği) yerleştirin (Şekil 3B). Sütunlar toprağın üstüne dayanabilir veya hedef taraftaki küçük kapağın hemen altına kadar gömülebilir.
- Rizosfer: Rizosfer mikroorganizmalarını yakalama şansını artırmak için sütunu belirli köklere yakın açı verirken ve yerleştirirken sütunları adım 9.3.2'de belirtildiği gibi bitki köklerini çevreleyen toprağa yerleştirin (Şekil 3C).
- Tüplerin içindeki alt tabakalar: 50 mL'lik bir konik tüpe veya benzerine ~ 10 mL ilgilenilen bir alt tabaka ekleyin ve istenirse alt tabakayı (örn. çok düşük nemli ortamlarda) saf su kullanarak ıslatın. Sütunu, alt kısım gömülü olacak ve dişli halkanın veya ağın hiçbir parçası görünmeyecek şekilde boruya yerleştirin (Şekil 3A). 50 mL'lik tüpün kapağını kapatın ve dik durması için 50 mL'lik bir tüp rafına yerleştirin.
10. Alt tabakada sütun bırakma
- Sütunu 3-21 gün boyunca alt tabakada bırakın.
NOT: Sütunların alt tabakalarında bırakılması gereken süre, kullanılan herhangi bir yem mantarının büyüme hızına, yem ortamını kolonize eden mantarların büyüme hızına ve çevresel koşullara bağlıdır (örneğin, kuru koşullar ortamın daha hızlı kurumasına neden olur, bu nedenle uzun süre kullanılamaz). - Deney için istenen bir aydınlık/karanlık döngüsü oluşturun; Önceki deneylerde laboratuvardaki sütunlar karanlıkta tutulurken, sahadaki sütunlar yerel gündüz/gece koşullarına maruz kalmıştır9.
11. Sütunun alt tabakadan çıkarılması
- Sütun, istenen süre boyunca alt tabaka ile temas ettikten sonra, sütunu çıkarın, fazla alt tabakayı dikkatlice silkeleyin ve sütunu taşımak için büyük kapağı ağın altındaki sütunun altına geri ekleyin.
- Kolonu taşımak için steril bir behere veya 50 mL'lik tüpe yerleştirin. Taşıma sırasında sütunu dik tutun.
12. Kolonun hedef ortamından izolatların kültürlenmesi
- Biyolojik güvenlik kabini gibi steril bir ortamda, yem ortamının büyük kapağını, dişli halkayı ve ağı çıkarın. Ağı atın.
NOT: Bir araştırmacı yem ortamını hangi organizmaların kolonize ettiğiyle ilgileniyorsa, hedef ortam tapasına ek olarak yem ortamı tapası ile 12.3-12.4 adımlarını izleyin. - Hedef ortamı içeren sütun ucundan küçük kapağı çıkarın.
- Hedef tapanın sütundan düşmesini sağlamak için sütunu çevirerek veya tapayı çıkarmak için sterilize edilmiş forseps veya pipet uçları kullanarak hedef orta tapasını çıkarın. Hedef orta tapasını doğrudan tercih edilen bir agar ortamıyla doldurulmuş 90 mm'lik bir Petri kabının ortasına yerleştirin (5. adımda hazırlandığı gibi).
NOT: Mantar veya bakteriye özgü ortamlar burada her iki krallıktan üyelerin büyümesini artırmak için kullanılabilir; tıkaç ayrıca sterilize edilmiş makas veya bir pipet ucu kullanılarak iki veya daha fazla parçaya bölünebilir, bu da birden fazla ortam türü üzerinde kaplamaya ve/veya DNA ekstraksiyonları için hedef ortamın korunmasına olanak tanır (bkz. adım 14). - Hedef ortam tıkaç ile aşılanmış Petri kabını karanlıkta 25 ° C'de en az 72 saat inkübe edin.
13. Mikroorganizmaları izole etmek için alt kültürleme
NOT: Bu adım isteğe bağlıdır.
- Bakteriler için: Steril bir aşılama döngüsü kullanarak, Petri kabının diğer alanlarıyla teması en aza indirirken tek bir ilgili koloni boyunca kaydırın. Koloniyi, tek kolonilerin oluşmasına izin veren herhangi bir yöntem kullanarak (örneğin, dört bölgeye ayırma) R2A (5. adımda hazırlandığı gibi) gibi tercih edilen bakteri ortamını içeren 90 mm'lik yeni bir Petri kabına geçirin. Petri kabını 25-37 °C'de 24-72 saat inkübe edin ve koloni büyümesini kontrol edin.
- Mantarlar için: Steril bir aşılama halkası, steril tıraş bıçağı veya steril pipet ucu kullanarak, hif büyümesi içeren minimum (1 mm2) bir agar bölümü açın. Küçük agar parçasını, 5. adımda hazırlandığı gibi MEA veya PDA gibi tercih edilen mantar ortamını içeren 90 mm'lik taze bir Petri kabına yerleştirin. Petri kabını karanlıkta 25 °C'de bir haftaya kadar inkübe edin ve aşırı büyümeyi önlemek için günlük olarak kontrol edin.
- Organizma morfolojileri tekdüze olana kadar adım 1 veya adım 2'yi gerektiği kadar tekrarlayın.
14. DNA'nın plakadan veya doğrudan hedef ortamdan çıkarılması
- Ekstraksiyon hemen gerçekleşmeyecekse, tüm agar tıkaçlarını (hedef ve/veya yem) veya kolonlardan seçilen parçaları 1,5 mL'lik santrifüj tüplerinde -20 °C'de dondurun veya ekstraksiyondan önce tapaların parçalarını 1,5 mL'lik bir santrifüj tüpüne bir koruyucuya batırın.
- Hedef ve/veya yem ortamından kültürleme yapıldıysa ve müteakip alt kültürleme adımları atıldıysa, mantar veya bakteri üremesi olan ~ 1 cm'lik bir agar parçasını (veya sonraki izolasyon adımları atılmadıysa her ikisinin bir karışımını) damgalayın veya oyun.
- İzole edilmiş bakteri kolonilerinin ekstraksiyonları için, steril bir aşılama halkası kullanarak bir koloniyi plakadan kaydırın ve doğrudan ticari DNA ekstraksiyon kiti ile ilişkili ekstraksiyon tamponunda döndürün (adım 14.4).
- Agar parçalarını bir havan ve havan tokmağı kullanarak sıvı nitrojen içinde ayrı ayrı öğütün ve öğütülmüş dokuyu ekstraksiyon tüplerine aktarın.
- Toprak veya bakteri ve mantarlar için optimize edilmiş ticari bir DNA ekstraksiyon kiti kullanın ve DNA'yı çıkarmak için üreticinin talimatlarını izleyin (Malzeme Tablosuna bakın).
- Elde edilen DNA'yı bir florometre veya karşılaştırılabilir bir sistem kullanarak ölçün.
15. Hedef ve/veya yem ortamındaki mikrobiyal taksonomik çeşitliliğin amplikon veya metagenomik dizileme yaklaşımları kullanılarak değerlendirilmesi
- Aşağıdaki adımları izleyerek amplikon dizilemesi (16S ve/veya dahili transkripsiyonlu aralayıcı [ITS]) veya metagenomik dizileme gerçekleştirin:
- Amplikon dizilimi: Bakt 341F (5′-CCT, ACG, GGN, GGC, WGC, AG-3′) ve Bakt 805R (5′-GAC TAC, HVG, GGT, GGT, ATC, TAA, TCC-3′) primerlerini kullanarak V3-V4 16S rRNA gen bölgesini amplifiye edin. ITS3 KYO2 (5′-GAT GAA, GAA CGY AGY RAA-3′) ve ITS4 (5′-TCC, TCC, GCT, TAT, TGA, TAT, GC-3")9 primerlerini kullanarak mantar ITS2 bölgesini güçlendirin.
- Seçilen sıralayıcı ile uyumlu ticari kitaplık hazırlama kitlerini kullanarak amplikon kitaplıkları hazırlayın. Sıralama platformu üreticisinin yükleme konsantrasyonu talimatlarını izleyerek yeterli kapsama alanına sahip 150 veya 250 baz çifti eşleştirilmiş uç okumaları oluşturmak için bir kısa okuma sıralayıcı kullanarak amplikonları sıralayın.
- Metagenomik dizileme: Seçilen dizileyici ile uyumlu, ticari olarak temin edilebilen bir metagenomik kitaplık hazırlama kitini kullanarak ekstrakte edilen DNA'dan bir metagenomik kitaplık oluşturun. Sıralama platformu üreticisinin yükleme konsantrasyonu talimatlarını izleyerek yeterli kapsama alanına (metagenom başına ~10-20 Gb) sahip 150 veya 250 baz çifti eşleştirilmiş uç okumaları oluşturmak için bir kısa okuma dizileyici seti kullanarak metagenom kitaplığını sıralayın.
16. Sıralama verilerinin analiz edilmesi
- Amplikon verilerini analiz edin: Sonuçları görselleştirmeden önce amplikon verilerini analiz etmek için DADA211 ile QIIME2 platformunu 12 13 kullanın. Empowering the Development of Genomics Expertise (EDGE)14 web tabanlı biyoinformatik platformunda QIIME2'yi çalıştırmak için aşağıdaki adımları izleyin. QIIME2, çevrimiçi olarak sağlanan "Hareketli Resimler" kılavuzu (https://docs.qiime2.org/2024.2/tutorials/moving-pictures/) gibi halka açık eğitimler kullanılarak da çalıştırılabilir.
- https://edgebioinformatics.org/'ye gidin ve giriş yapın veya bir hesap oluşturun.
- Ana sayfadan QIIME2'yi ÇALIŞTIR'ı seçin. Amplikon sıralama çalıştırmalarından dosya yüklemek için sol menü çubuğunda Dosyaları Yükle'yi seçin.
- 'Meta Veri Eşleme Dosyası'nın yanındaki 'i' işaretinin üzerine gelindiğinde sağlanan talimatları kullanarak bir Meta Veri Eşleme dosyası oluşturun.
- Gerekli tüm bilgileri (Proje/Çalıştırma Adı, Okuma Türü, Parametreler vb.) ekleyin ve yüklenen doğru giriş verilerini seçin. Kalite kontrol yöntemi12 olarak DADA2'nin seçildiğinden emin olun; Diğer parametreler, başka değişiklikler istenmedikçe varsayılan değerleri olarak bırakılabilir.
- Amplikon tipini bakteri amplikonları için 16S Greengenes (http://greengenes.lbl.gov) veya fungal amplikonlar için Fungal ITS olarak seçin. Bakteri (16S) ve mantar (ITS) verilerini bağımsız olarak analiz edin.
- Gönder tuşuna basın ve sonuçları görüntülemek için çalıştırmanın bitmesini bekleyin.
- Amplikon veri görselleştirmeleri oluşturma (İsteğe bağlı): GitHub'da sağlanan yönergeleri izleyerek ve Paketler menüsüne giderek, indirilen pakete tıklayarak ve belgeleri görüntüleyerek R Studio'da bulunan R paketi Yardımı aracılığıyla phyloseq (https://github.com/joey711/phyloseq), VEGAN (https://github.com/vegandevs/vegan) ve APE (https://emmanuelparadis.github.io/) gibi yaygın paketleri kullanarak R'de ek topluluk veri analizleri gerçekleştirin.
- Metagenomik verileri analiz edin
- NMDC EDGE sitesi15'e (https://nmdc-edge.org/home) gidin ve bir ORCiD hesabı (https://orcid.org/) kullanarak oturum açın.
- Ekranın sol tarafındaki menü çubuğunda Dosyaları Yükle'yi seçin ve doğru giriş FASTQ dosyalarını sürükleyip bırakın veya göz atın.
- Metagenomiks'i seçin, ardından ekranın sol tarafındaki menü çubuğunda Birden Çok İş Akışı Çalıştır seçeneğini belirleyin ve tüm iş akışlarını Açık olarak ayarlayın. Bir proje adı ve isteğe bağlı bir açıklama ekleyin.
- Yüklenen ham okuma (fastq) dosyalarını seçin ve uygun dosya biçimini (araya eklenmiş veya eşleştirilmiş) seçin.
- Gönder'e tıklayarak çalıştırmayı başlatın ve ekranın üst kısmındaki Projelerim sekmesinde projeyi seçerek çalıştırma tamamlandığında özet tabloları ve görselleştirmeleri görüntüleyin.
17. Amplikon ve/veya metagenomik sonuçlardan taksonomi verilerinin ek görselleştirmelerinin oluşturulması
- GitHub'da sağlanan yönergeleri izleyerek GraPhlAn (https://github.com/biobakery/graphlan) paketini kullanarak dairesel kladogramlar oluşturun.
NOT: Ulusal Biyoteknoloji Bilgi Merkezi (NCBI) taksonomisi (https://www.ncbi.nlm.nih.gov/taxonomy) tanımlayıcıları, temsili dizi atamalarından elde edilebilir ve GraPhlAn16'nın gerektirdiği taksonomi toplama bilgilerini toplamak için Entrez Direct E-yardımcı programlarının 'eftech' programına iletilebilir.
18. Sütunları yeniden kullanma
- Sütunlar hala monte edilmişse, kapakları, dişli halkayı ve ağı sökün ve çıkarın. Ağı atın. Kalan agar tapalarını çıkarın, kolonları %99 izopropil alkol ve arıtılmış su ile yıkayın ve bileşenleri adım 3.1-3.4'te açıklandığı gibi temizleyin ve kurulayın.
- Otoklavlanacak yeni naylon ağ tabakaları hazırlayın.
- Adım 4.1'de sağlanan yönergeleri izleyerek bileşenleri otoklavlayın.
Sonuçlar
Tamamen monte edilmiş mantar otoyol sütunu yaklaşık 5 cm uzunluğundadır (Şekil 1). Kolon hiçbir alanda kırılmamalı ve kolon içinde mikro ortamlar oluşturmak için kapaklar ve dişli halka kolayca ve sıkıca birbirine oturmalıdır. Filtre ağı, dişli halkanın ötesine uzanabilir ( Şekil 1 ve Şekil 2'de gösterildiği gibi) veya sterilize edilmiş makasla kesilebilir. Agar tapaları kolonun her iki ucuna sıkıca oturmalıdır. Alt tabakaya yerleştirildiğinde, filtre ağı alt tabaka ile temas etmeli ve kolon tamamen gömülmemelidir.
Sütunlar daha önce at gübresi9'da test edilmiştir. Sütunlar ayrıca bir araştırma sahasında toplu ve rizosfer toprağına ve ayrıca laboratuvarda 50 mL'lik tüplerde küçük hacimli toprağa yerleştirildi (Şekil 3). Mantar otoyol kolonları alt tabakadan çıkarıldıktan ve demonte edildikten sonra, hem yem hem de hedef ortam tapalarında mikrobiyal büyüme görüldü (örnekler Şekil 4A'da gösterilmiştir). Bakteri ve mantarlar, alt kültürleme teknikleri ile hedef ve yem ortamlarından izole edildi (Şekil 4B) ve ortam tıkaçlarında bulunan mikroplar, amplikon dizilemesi kullanılarak taksonomik olarak tanımlandı (Şekil 4C,D). Şekil 4C,D, at gübresi9'a eklenen sütunlardan hangi mikropların hedef ortam fişine ulaşabildiğini gösteren, birden fazla deneyde amplikon dizilemesinin birleşik sonuçlarını göstermektedir. Bu bakteri ve mantar verilerinin görselleştirmeleri, adım 17'de belirtildiği gibi oluşturulmuştur. Sonuçlar ayrıca taksonların nispi bollukları olarak da görüntülenebilir.
Kolonların son derece düşük nemli ortamlara eklendiği ve medya tapalarının birkaç gün içinde tamamen kurutulduğu ve kolonize mikropların geri kazanılmasına yol açmadığı durumlarda optimal olmayan sonuçlar elde edilmiştir (Şekil 5A). Ayrıca, mikropların hedef ortam fişinden basitçe büyümediği durumları da gördük (Şekil 5B) ve anlamlı analizler için hedef ortam fişinden yeterli sıralama verisini kurtarmadığımız durumlar. Mantarların sütunlardan aşırı büyüdüğü diğer durumlar da deneylerin yeniden yapılması ihtiyacını doğurmuştur (Şekil 5C).

Şekil 3: Laboratuvar ve saha ortamlarında çevresel numunelere yerleştirilen sütun örnekleri. (A) Laboratuvar ortamında nemlendirilmiş toprakla 50 mL'lik bir tüpün içine yerleştirilen sütun. Ayrıca ölçek için bir cetvel ile gösterilir. (B) Tarlada toprağa yerleştirilen sütun. (C) Tarladaki bir bitkinin kök ağına yerleştirilen kolon. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
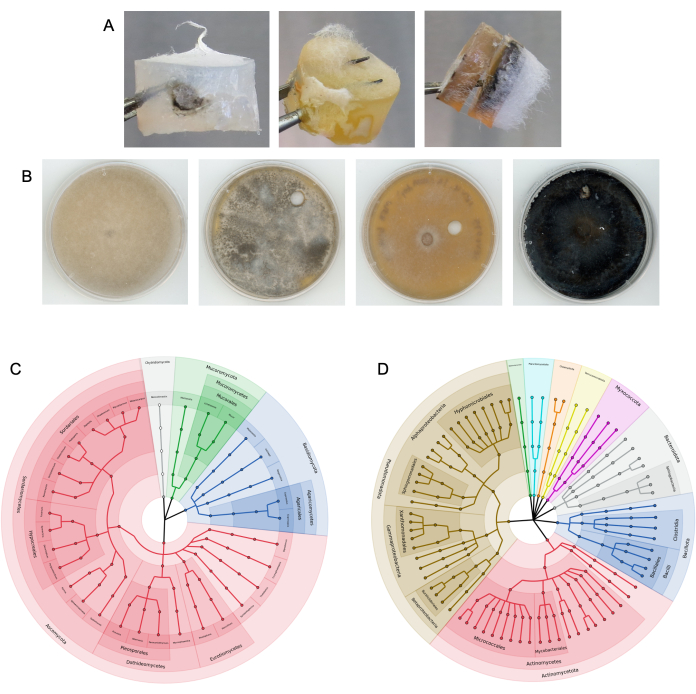
Şekil 4: Başarılı kolon deneylerinden elde edilen temsili sonuçlar. (A) Sütunlardan çıkarılan kolonize medya fişlerine örnekler. (B) Hedef ortamdan alt kültürlenmiş mantar izolatlarının örnekleri. Substrat topraktı. Soldan sağa üst ITS dizisi NCBI BLAST kimliği: Rhizopus azygosporous, Aspergillus novofumigatus, Curvularia subpapendorfii ve Phaeomycocentrospora cantuariensis. (C,D) At gübresi kullanılarak yapılan çoklu mantar otoyolu deneylerinin ardından hedef ortamdan elde edilen (C) mantar ITS ve (D) bakteriyel 16S dizilerinin filogenetik çeşitliliğini gösteren dairesel kladogramlar. Bölümler filum ile renklendirilir ve etiketlenir, uç düğümler benzersiz cinsleri temsil eder. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
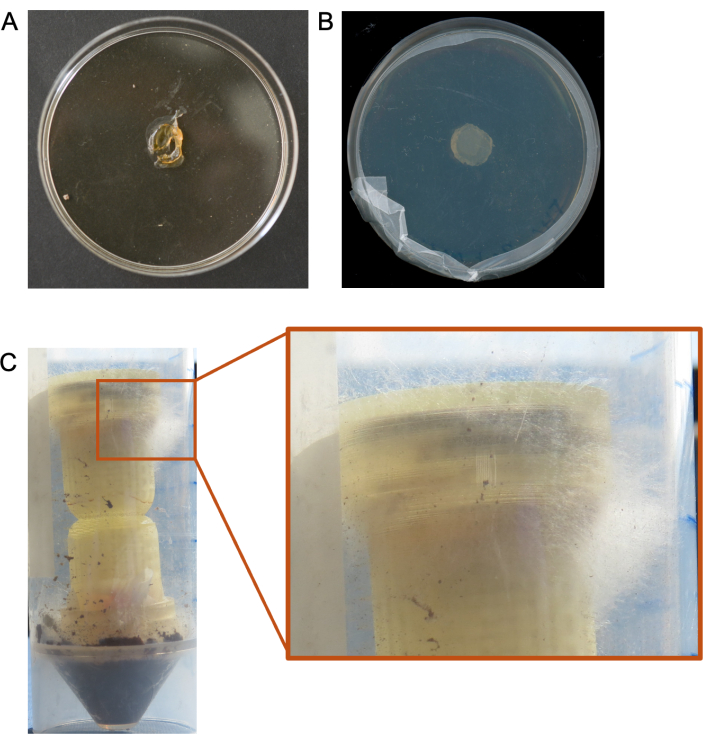
Şekil 5. Kolon deneylerinden elde edilen optimal olmayan sonuçlar. (A) Düşük nemli ortam koşullarından kaynaklanan kurumuş bir ortam fişi örneği. (B) Bir kolon medya fişinden mikrobiyal büyüme olmaması örneği. (C) Mantarın kolonun üst kısmından (hedef ortam) aşırı büyümesi örneği. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Tartışmalar
Kolon bileşenleri oluşturulurken, bir 3D yazıcı ve baskı malzemesinin seçimi, kullanılabilirliğe ve istenen malzeme özelliklerine göre değiştirilebilir17,18. Grubumuzun malzeme seçiminde biyouyumluluk, yüzey dokusu, otoklavlanabilirlik, ince ölçekli detaylar basabilme yeteneği ve göreceli şeffaflık göz önünde bulunduruldu. Gözeneklilik, hidrofobiklik, baskı parametreleri vb. gibi diğer özellikler de dikkate alınmalıdır. Son seçimden önce çeşitli reçineler test edilmiştir (Malzeme Tablosuna bakınız) ve bu sütunların baskısı için birçok biyouyumlu malzeme çalışacaktır. Kolon bileşenlerinin inşası için seçilen malzeme, hangi temizleme, son kürleme ve sterilizasyon yaklaşımlarının kullanılması gerektiğini belirleyecektir. Tüm malzemeler otoklavlanabilir olmayacaktır ve malzeme üreticisinin talimatlarına bağlı olarak ultraviyole ışık, ağartıcı veya diğer sterilizasyon teknikleri gerekebilir. Bazı sterilizasyon veya temizleme teknikleri de seçilen malzemeye zarar verebilir veya onunla uyumlu olmayabilir, bu nedenle malzeme üreticisinin bu bilgilere özellikle dikkat etmesi gerekir. 3D yazıcılar için bazı hususlar arasında baskı süresi, malzeme uyumluluğu, yapı platformu boyutu, baskı teknolojisi vemaliyet 19 yer alır. Sütunların 3D baskılı bileşenleri kırılgan olabilir ve çok güçlü bir şekilde kullanılırsa kırılabilir. Halka ve kapaklar için diş açma her zaman tam olarak hizalanmayabilir, bu nedenle montaj adımından önce ekstra bileşenlerin basılmasını ve sterilize edilmesini veya seçilen parametrelerin ve malzemenin diş açmayı nasıl etkilediğini test etmek için ön baskı yapılmasını öneririz. Kapaklar ve halka içindeki diş açma için tasarım özelliklerinin, seçilen 3D baskı malzemesine bağlı olarak ayarlanması gerekebilir. Boyutlar, kafes karmaşıklığı ve diğer fiziksel özelliklerin tümü, baskıdan önce CAD tasarım yazılımında değiştirilebilir. Tasarlandığı gibi, kolonun kendisi 4 cm yüksekliğindedir ve kolonun ortasındaki kafes yapısı 2 mm boyutunda bir birim hücreye, 0,5 mm'lik bir dikme çapına ve tüm kafes yüksekliği 22 mm'dir9. Bu parametreler, bir araştırmacı, örneğin daha büyük veya daha karmaşık bir kafes yapısı isterse ayarlanabilir. Genel olarak, bu cihazların 3D baskılı üretimi, tasarım esnekliği sağlarken, aynı zamanda tek bir tasarımın organizasyonlar ve gruplar arasında standart bir şekilde kullanılabilmesini ve hatta sınıf öğretim araçları olarak kullanılabilmesini sağlar9.
Protokoldeki birkaç adım, ortama veya deney düzeneğine bağlı olarak sorun giderme gerektirebilir. Mantar otoyol kolonları, düşük nem koşullarında çok etkili değildir, çünkü ortam tıkaçları mantar büyümesini kolaylaştırmadan önce hızla kurur ve bu da bu ortamlardaki deneylerin süresini sınırlayabilir (Şekil 5A). Düşük nemli ortamlarda kolonların etkinliğini artıran teknikler arasında, alt tabakaya nem eklenmesi yoluyla nemin yapay olarak artırılması ve/veya kolonun ve alt tabakanın bir su kaynağı (örneğin, küçük bir saf su kabı) ile ikincil bir kapta kapatılması yer alır. Kum saati şekli ve kafes yapısı, yüksek nemli ortamlarda yoğuşma oluşması durumunda tek başına bakteri hareketini önlemek için (bir mantar otoyolu kurulmadan) dahil edildi. Hızlı büyüyen mantarlar, hedef ve yem ortamı yüzey alanını aşırı büyütebilir ve kolonun üstünden veya altından dışarı doğru uzanabilir (Şekil 5C). Yem mantarının kuluçka süresinin veya deney süresinin azaltılması bu aşırı büyümeyi en aza indirebilir veya ortadan kaldırabilir. Ek olarak, bu cihazların bir sınırlaması, ilgilenilen substrattaki hızlı büyüyen mantarların, yavaş büyüyen mantarlar tarafından yem ve hedef ortamın kolonizasyonunu sınırlayabilmesi ve potansiyel olarak hangi otoyol etkileşimlerinin gözlemlendiğini önyargılı hale getirebilmesidir. Bazı mantarlar, özellikle daha yavaş büyüyen mantarlar, yem ortamını agar tıkacından ve kafes yapısına büyümelerine izin verecek şekilde kolonize etmeyebilir. Ortamda yeterli nem varsa, yem agar tıkacı kolonizasyonundan sonra kafes içine büyümeyi teşvik etmek için daha ince agar tıkaçları kullanılabilir. Ortam, bir araştırmacının mantar veya bakteri üremesini seçmek isteyip istemediğine bağlı olarak seçilebilir, ancak bu aynı zamanda alt kültürlemeyi, bu ortam tipi20'yi tercih eden organizmalarla sınırlayabilir. Hedef ortamda herhangi bir büyüme görülmezse, yem ortamını veya substratı mantar otoyolları oluşturduğu bilinen bir mantarla aşılamak gerekebilir.
Metagenomik veya amplikon dizileme bu deneylerin bir parçası olarak gerçekleştirilebilir ve bu stratejilerin her ikisi de kendi sınırlamalarını ve güçlü yönlerini verir21. Metagenomik dizileme, mikroplar hakkında ek genomik bilgi elde etmek için idealdir. Bununla birlikte, doğrudan hedef ortamdan geri kazanılabilir nükleik asit miktarı çok düşük olabilir, bu da dizilemeden önce amplikon dizileme veya diğer amplifikasyon yöntemlerinin kullanılmasını gerektirebilir. Amplikon dizileme kütüphaneleri ayrı ayrı hazırlanmalıdır (16S ve ITS) ve bu yöntem taksonomik çözünürlükten yoksundur ve metagenomik dizileme kullanılarak elde edilebilecek genom özellikleri veya fonksiyonel potansiyel hakkındaki değerlendirmeleri sınırlar. Mikropların alt kültüre alınamadığı durumlarda tıkaçlardan doğrudan dizileme yöntemleri tercih edilebilir. Hem kültürleme hem de sıralama yaklaşımlarını etkinleştirmek için tapaların birden çok bölüme ayrılması önerilir.
Bu cihazların bir avantajı da hem laboratuvarda hem de sahada kullanılabilmeleridir. Sahadaki sütunların dik kalabilmesi ve yerleşimlerini bozabilecek hayvanlardan ve çevresel rahatsızlıklardan korunabilmesi için özel dikkat gösterilmelidir. Sütunlar henüz yatay bir konumda, bir alt tabaka ile tamamen kaplandıkları bir konumda test edilmemiştir ve önemli miktarda yağış veya kara maruz kalan ortamlarda test edilmemiştir. Yukarıda belirtildiği gibi, kafes yapısı, yüksek nemli ortamlarda bakterilerin hedef ortama hareket etme olasılığını en aza indirecek şekilde tasarlanmıştır. Bununla birlikte, kolon daha büyük hacimlerde suya maruz kalırsa ve bu su kolonu tamamen doyurursa, mevcut herhangi bir mantar otoyolundan bağımsız olarak kolon boyunca bakteri hareketinin kolaylaştırılması mümkündür. Laboratuar tabanlı deneyler için, sütunlar 50 mL'lik konik tüplerde, küçük substrat mikrokozmoslarında, saksı bitkilerini çevreleyen toprakta, kutularda veya diğer kontrollü deney sistemlerinde kullanılabilir. Sütunlar toprakta, rizosferlerde ve gübrede başarıyla kullanılmıştır ve kullanımları yaprak çöpü, çamur, kum, kar, kompost vb. dahil olmak üzere diğer alt tabakalara genişletilebilir.
Mantar otoyol sütunları, çeşitli numune türleri içinde bu BFI fenotipini anlamak için bir dizi karşılaştırmaya olanak tanır. Yem ve hedef ortam arasındaki topluluk bileşiminin karşılaştırılması, hangi bakterilerin mantar otoyollarını kullanabileceğini ve hangi mantarların potansiyel otoyollar olarak hizmet edebileceğini gösterebilir9. Metagenom dizilimi kullanılırsa, organizmaları yemden hedef ortama karşı ayıran genomik özellikler de incelenebilir. Farklı alt tabakalara yerleştirilen (örneğin, toprağa karşı gübre) veya farklı koşullar altında (örneğin, sıcaklık veya nem) aynı alt tabakaya yerleştirilen sütunlardan hedef ortamı karşılaştırmak da mümkündür. Genel olarak, mantar otoyol sütunları, bu BFI formunu sorgulamak için önceki yöntemlerin yeteneklerini genişletir ve karmaşık çevresel mikrobiyomların mekansal dinamiklerini şekillendiren bu etkileşimlerin kapsamlı bir şekilde incelenmesini sağlar.
Açıklamalar
Yazarların açıklanacak herhangi bir çıkar çatışması yoktur.
Teşekkürler
Bu araştırma, ABD Enerji Bakanlığı (DOE), Biyolojik ve Çevresel Araştırma (BER), Biyolojik Sistem Bilimi Bölümü'nden (BSSD) LANLF59T numaralı hibe numarası altında bir Bilim Odak Alanı Hibesi ile desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 50 mL tubes | Greiner BIO-ONE | 5622-7261 | 50 mL tubes for performing column experiments in the lab |
| 90 mm Petri dishes | Thermo Scientific Nunc | 08-757-099 | Petri dishes for preparation of agar and for microbial growth |
| Asiga Freeform Pico Plus 39 digital light processing (DLP) 3D printer | Asiga Germany | Freeform Pico Plus 39 | 3D printer used to generate batches of the columns; other 3D printers can be used |
| Autoclave | Fisher Scientific | LS40F20 | Benchtop autoclave to sterilize the column components |
| Beaker | Fisher Scientific | FB100600 | 600 mL beaker for various uses throughout the protocol |
| Dental LT Clear Resin V2 | Formlabs | RS-F2-DLCL-02 | Alternative resin for 3D printing that was tested |
| Dental Surgical Guide Resin | Formlabs | RS-F2-SGAM-01 | Was used to generate the columns discussed in manuscript; Other photosensitive resins can be used in place of this material |
| DNA Low Bind 1.5 mL tubes | Eppendorf | 13-698-791 | Tubes used for various preparations including nucleic acid extractions |
| DNA/RNA shield preservative | Zymo Research | R1100-50 | Preservative used prior to nucleic acid extractions |
| EDGE Bioinformatics | Open source; Developed by the Los Alamos National Laboratory (LANL) | n/a | Bioinformatics platform for processing amplicon data |
| FastDNA spin kit for soil | MP Biomedicals LLC | 116560200-CF | DNA extraction kit option for soil |
| Forceps | Fisher Scientific | 10-300 | Forceps that can be sterilized |
| Formlabs BioMed Clear Resin | Formlabs | RS-F2-BMCL-01 | Alternative resin for 3D printing that was tested |
| Formlabs Form 3B+ stereolithography (SLA) 3D printer | Formlabs | Form 3B+ | Alternative 3D printer |
| Formlabs IBT Resin | Formlabs | RS-F2-IBCL-01 | Alternative resin for 3D printing that was tested |
| Inoculating Loops | Fisher Scientific | 22-363-598 | Used to isolate/transfer microbes |
| Malt Extract Agar (MEA) | Criterion | 89405-654 | A media type used in columns |
| MiSeq sequencer + MiSeq sequencing kit | Illumina | SY-410-1003 | Can use other sequencers |
| Mortar & Pestle | Fisher Scientific | FB961K; FB961A | Can use any common mortar & pestle that can be sterilized between uses |
| NEBNext Ultra II DNA Library Prep Kit for Illumina | New England Biolabs | E7805S | Library prep kit for metagenomic sequencing |
| Nextera XT DNA Library Preparation Kit (24 samples) | Illumina | FC-131-1024 | Library prep kit for amplicon sequencing |
| NMDC EDGE | Open source: Developed by the National Microbiome Data Collaborative (NMDC) | n/a | Bioinformatics platform for processing metagenomic data |
| Nylon mesh | Sefar | 03-25/19 | The mesh used as part of the column construction |
| Pipette tips | Rainin | 30807966 | Can use many different sterilized pipette tips for the protocol steps |
| Potato Dextrose Agar | Cole Parmer | EW-14200-28 | A media type used in columns |
| QIIME2 | Open source | n/a | Software for processing amplicon data |
| Qubit dsDNA HS assay kit | Thermo Fisher Scientific | Q32851 | Used to quantify DNA after extractions |
| Qubit Fluorometer | Thermo Fisher Scientific | Q33238 | Used to quantify DNA after extractions |
| Quick-DNA Fungal/Bacterial Miniprep Kit | Zymo Research | D6005 | DNA extraction kit option that works with both bacteria and fungi |
| R2A agar | BD Difco | 218263 | A media type used in columns (bacterial media) |
| Rack for 50 mL tubes | Fisher Scientific | 03-448-11 | Rack to hold 50 mL tubes upright |
| Scissors | Fisher Scientific | 12-000-155 | Fine precision scissors that can be sterilized |
| Sodium carboxymethyl cellulose medium | Aldrich | 419273-100G | A media type used in columns |
| SolidWorks CAD software | SolidWorks | n/a | Software used to design the columns |
| Trowel scoop | Fisher Scientific | S41701 | To make a depression in the substrate prior to adding the column |
| UltraPure DNase/RNase-Free Distilled Water | Invitrogen: ThermoFisher Scientific | 10977015 | Water for the ultrasonicator water bath |
| Ultrasonicator | Fisher Scientific | FB-11201 | Ultrasonicator for cleaning the columns |
Referanslar
- Or, D., Smets, B. F., Wraith, J. M., Dechesne, A., Friedman, S. P. Physical constraints affecting bacterial habitats and activity in unsaturated porous media - A review. Adv Water Resour. 30 (6), 1505-1527 (2007).
- Kohlmeier, S., et al. Taking the fungal highway: Mobilization of pollutant-degrading bacteria by fungi. Environ Sci Technol. 39 (12), 4640-4646 (2005).
- Simon, A., Hervé, V., Al-Dourobi, A., Verrecchia, E., Junier, P. An in situ inventory of fungi and their associated migrating bacteria in forest soils using fungal highway columns. FEMS Microbiol Ecol. 93 (1), 217 (2017).
- Wick, L. Y., et al. Effect of fungal hyphae on the access of bacteria to phenanthrene in soil. Environ Sci Technol. 41 (2), 500-505 (2007).
- Bravo, D., et al. Isolation of oxalotrophic bacteria able to disperse on fungal mycelium. FEMS Microbiol Lett. 348 (2), 157-166 (2013).
- Furuno, S., Remer, R., Chatzinotas, A., Harms, H., Wick, L. Y. Use of mycelia as paths for the isolation of contaminant-degrading bacteria from soil. Microb Biotechnol. 5 (1), 142-148 (2012).
- Buffi, M., et al. Fungal drops: A novel approach for macro- and microscopic analyses of fungal mycelial growth. Microlife. 4, 042 (2023).
- Kuhn, T., et al. Design and construction of 3D printed devices to investigate active and passive bacterial dispersal on hydrated surfaces. BMC Biol. 20 (1), 203 (2022).
- Junier, P., et al. Democratization of fungal highway columns as a tool to investigate bacteria associated with soil fungi. FEMS Microbiol Ecol. 97 (2), 003 (2021).
- Reasoner, D. J., Geldreich, E. E. A new medium for the enumeration and subculture of bacteria from potable water. Appl Environ Microbiol. 49 (1), 1-7 (1985).
- Bolyen, E., et al. Reproducible, interactive, scalable, and extensible microbiome data science using QIIME 2. Nat Biotechnol. 37 (8), 852-857 (2019).
- Callahan, B. J., et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nat Methods. 13 (7), 581-583 (2016).
- Vázquez-Baeza, Y., Pirrung, M., Gonzalez, A., Knight, R. EMPeror: A tool for visualizing high-throughput microbial community data. Gigascience. 2 (1), 16 (2013).
- Li, P. -. E., et al. Enabling the democratization of the genomics revolution with a fully integrated web-based bioinformatics platform. Nucleic Acids Res. 45 (1), 67-80 (2017).
- Eloe-Fadrosh, E. A., et al. The National Microbiome Data Collaborative Data Portal: An integrated multi-omics microbiome data resource. Nucleic Acids Res. 50 (1), D828-D836 (2022).
- Entrez Direct: E-utilities on the Unix Command Line. Entrez Programming Utilities Help Available from: https://www.ncbi.nlm.nih.gov/books/NBK179288/ (2024)
- Palmara, G., Frascella, F., Roppolo, I., Chiappone, A., Chiadò, A. Functional 3D printing: Approaches and bioapplications. Biosens Bioelectron. 175, 112849 (2021).
- Guttridge, C., Shannon, A., O'Sullivan, A., O'Sullivan, K. J., O'Sullivan, L. W. Biocompatible 3D printing resins for medical applications: A review of marketed intended use, biocompatibility certification, and post-processing guidance. Ann 3D Print Med. 5, 100044 (2022).
- Yao, L., et al. Comparison of accuracy and precision of various types of photo-curing printing technology. J Phys Conf Ser. 1549 (3), 032151 (2020).
- Basu, S., et al. Evolution of bacterial and fungal growth media. Bioinformation. 11 (4), 182-184 (2015).
- Liu, Y. -. X., et al. A practical guide to amplicon and metagenomic analysis of microbiome data. Protein Cell. 12 (5), 315-330 (2021).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır