Method Article
Studio delle interazioni batterio-fungine utilizzando colonne autostradali fungine in diversi ambienti e substrati
In questo articolo
Riepilogo
Questo protocollo fornisce istruzioni dettagliate su come costruire, sterilizzare, assemblare, utilizzare e riutilizzare le colonne autostradali fungine per arricchire le coppie batterio-fungine che interagiscono attraverso le autostrade fungine da diversi substrati ambientali.
Abstract
Le interazioni batterio-fungine (BFI) svolgono un ruolo fondamentale nel plasmare la composizione della comunità microbica, le funzioni biogeochimiche, le dinamiche spaziali e la dispersione microbica. Le reti miceliali create da funghi filamentosi o altri microrganismi filamentosi (ad esempio, oomiceti) agiscono come "autostrade fungine" che possono essere utilizzate dai batteri per il trasporto in ambienti eterogenei, facilitando notevolmente la loro mobilità e garantendo loro l'accesso a regioni che possono essere difficili o impossibili da raggiungere da sole (ad esempio, a causa di sacche d'aria all'interno del suolo). Diversi dispositivi e protocolli sperimentali sono stati creati per studiare queste autostrade fungine, comprese le colonne autostradali fungine. La colonna autostradale fungina progettata dal nostro gruppo può essere utilizzata per una varietà di applicazioni in situ o in vitro , nonché con diversi tipi di campioni associati all'ambiente e all'ospite. In questo articolo, descriviamo i metodi per eseguire esperimenti con queste colonne, tra cui la progettazione, la stampa, la sterilizzazione e la preparazione dei dispositivi. Qui vengono discusse anche le opzioni per l'analisi dei dati ottenuti dall'uso di questi dispositivi e vengono offerti consigli per la risoluzione dei problemi relativi alle potenziali insidie associate agli esperimenti che utilizzano colonne autostradali fungine. Questi dispositivi possono essere utilizzati per ottenere una comprensione più completa della diversità, dei meccanismi e delle dinamiche dei BFI autostradali fungini per fornire preziose informazioni sulle dinamiche strutturali e funzionali all'interno di ambienti complessi (ad esempio, suoli) e in diversi habitat in cui coesistono batteri e funghi.
Introduzione
Le interazioni batterio-fungine (BFI) sono estremamente importanti nel plasmare le proprietà strutturali, spaziali e funzionali dei microbiomi ambientali. Ad esempio, la crescita e l'espansione di funghi filamentosi o di altri microrganismi filamentosi simili a funghi genera una rete biologica che può funzionare come una "autostrada" per facilitare il movimento di altri microrganismi, come i batteri. L'eterogeneità e la saturazione incoerente all'interno dei substrati ambientali possono ostacolare la motilità batterica; Tuttavia, i batteri possono utilizzare queste autostrade per facilitare l'accesso ad altre aree dell'ambiente 1,2. Queste interazioni sono fondamentali per comprendere le dinamiche spaziali delle comunità microbiche. Diverse tecniche e metodi sono stati utilizzati per esaminare le autostrade fungine, tuttavia, sono in gran parte limitati a indagini di laboratorio 3,4.
In un metodo basato su piastra, un'ampia sezione di agar viene rimossa dal centro della piastra di Petri, creando uno spazio tra due isole di agar. Le ife fungine possono attraversare questo divario, fornendo i mezzi per i batteri compatibili per passare da un'isola di agar all'altra5. Altri metodi modificati della capsula di Petri includono piastre invertite in cui il terreno viene posto nel coperchio in modo che le ife fungine possano crescere verticalmente e colonizzare il terreno senza contatto diretto, fornendo i mezzi per il trasporto batterico 5,6. Un metodo basato su goccioline di terreno di crescita che è stato recentemente sviluppato può essere utilizzato per valutare il trasporto ifale selettivo dei batteri verso determinati profili nutrizionali7. Sono stati utilizzati anche dispositivi a ponte e a sentiero batterico per studiare l'effetto di fattori abiotici sul movimento batterico8. Sebbene siano stati utilizzati diversi metodi e tecniche per studiare le autostrade fungine, rimane la necessità di dispositivi standardizzati che mantengano un microambiente sterile, promuovendo al contempo la creazione di autostrade fungine da substrati ambientali complessi come sterco, suolo e rizosfere.
Il nostro gruppo ha progettato una versione stampata in 3D di colonne autostradali fungine in cui i funghi possono trasportare i batteri da un'estremità all'altra9. Questi dispositivi sono assemblati da quattro componenti stampati: la colonna stessa con una forma a clessidra e una complessa struttura reticolare interna, un anello filettato e due tappi (un tappo grande e un tappo piccolo), nonché un pezzo di rete di nylon sterilizzata (Figura 1). La colonna assemblata viene aggiunta direttamente al substrato ambientale desiderato. La colonna consente quindi ai microbi di colonizzare un tappo del terreno di crescita dell'agar noto come tappo del terreno di coltura "esca" che si trova nella parte inferiore della colonna e a contatto con il substrato ambientale attraverso la rete. Questo pezzo di rete di nylon esclude altri abitanti del suolo che possono trasportare batteri, limitando così il movimento batterico all'interno delle colonne verso le autostrade fungine. Una volta che questo tappo dell'esca è stato colonizzato, i funghi filamentosi possono estendersi e crescere attraverso il reticolo interno all'interno del centro della colonna che è progettato per creare un sistema insaturo che assomiglia al suolo (o altri mezzi insaturi) e ridurre al minimo la potenziale contaminazione dal mezzo dell'esca. I funghi quindi crescono e colonizzano il tappo del mezzo bersaglio nella parte superiore della colonna. Le colonne possono essere inoculate con isolati fungini specifici per testare la loro capacità di trasportare batteri, oppure possono essere lasciate non inoculate per identificare quali funghi del substrato sono in grado di trasportare batteri. Gli organismi che raggiungono il terreno bersaglio possono essere ulteriormente coltivati, isolati e sottoposti ad analisi di sequenziamento (sia da colture pure che da comunità miste utilizzando approcci di sequenziamento ampliconico o metagenomico). Nel complesso, le colonne forniscono un metodo standardizzato, riproducibile, riutilizzabile e intuitivo per interrogare le autostrade fungine in diversi substrati. Questi dispositivi possono essere utilizzati per la ricerca e come strumento didattico in classe e, in questo articolo, forniamo passaggi didattici per il loro utilizzo basati sugli esperimenti eseguiti in passato. Sebbene questo metodo faciliti la standardizzazione del protocollo, la progettazione e la costruzione dei dispositivi possono essere modificate per altre applicazioni e substrati aggiuntivi.
Protocollo
I dettagli dei reagenti e delle attrezzature utilizzate nello studio sono elencati nella tabella dei materiali.
1. Modifica del design, dei materiali e dei parametri della colonna
- Scaricate i progetti di colonnadisponibili al pubblico 9 e utilizzateli così come sono o modificate i progetti di colonna in un software CAD (Computer-Aided Design) compatibile.
- Ottieni la resina per guide chirurgiche dentali o seleziona un materiale di stampa 3D alternativo, come altre resine trasparenti fotosensibili.
- Regolare le specifiche della colonna se necessario se la stampante 3D, la tecnologia di stampa 3D o il materiale di stampa 3D scelto vengono modificati rispetto a quelli utilizzati in precedenza9.
2. 3D stampa delle colonne
- Impostare i parametri di stampa in modo da utilizzare uno spessore della fetta di 0,05 mm e un tempo di esposizione di 0,8 s, oppure regolare i parametri di stampa in base alla stampante, al materiale di stampa e al software di stampa selezionati.
- Stampa le colonne autostradali fungine, gli anelli filettati e i cappucci utilizzando una stampante 3D compatibile (Figura 1).

Figura 1: Componenti della colonna fungina dell'autostrada. (A,B) Viste dall'alto e laterali del tappo piccolo, dell'anello filettato e del tappo grande (da sinistra a destra). (C, D) Vista dall'alto e laterale del tappo piccolo. (E,F) Vista dall'alto e laterale dell'anello filettato. (G,H) Vista dall'alto e laterale del tappo grande. (I) Un pezzo di filtro a rete di nylon (25 μm) posizionato all'estremità della colonna e inserito nel substrato ambientale per impedire alla microfauna di entrare nella colonna. (J) Colonna non montata. (K) Colonna assemblata: l'estremità 'Esca' entra nel substrato e l'estremità 'Target' rimane scoperta e fuori dal substrato. Clicca qui per visualizzare una versione più grande di questa figura.
3. Pulizia dei componenti della colonna stampati in 3D
- Immergere le colonne stampate, i cappucci e gli anelli filettati in alcol isopropilico al 99% a temperatura ambiente per 15 minuti e agitare muovendo il bagno avanti e indietro a mano per 10-15 secondi ogni minuto per rimuovere la resina in eccesso.
- Trasferire i componenti in un bagno fresco di alcol isopropilico al 99%. Immergere per 5 minuti e agitare a mano come al punto 3.1.
- Trasferire i componenti in un dispositivo di pulizia a ultrasuoni riempito con acqua pura, immergere i componenti e agitare per 2 minuti a velocità media. Non riscaldare l'acqua. Rimuovere i componenti.
- Asciugare all'aria tutti i componenti per almeno 30 minuti.
- Per eseguire la polimerizzazione post-stampa della resina, esporre tutti i componenti stampati in 3D a una luce a 405 nm per 30 minuti a 60 °C.
4. Sterilizzazione delle colonne
- Se il materiale scelto è autoclavabile, sterilizzare in autoclave le colonne, gli anelli filettati, i tappi e i fogli di filtro a rete di nylon da 25 μm singolarmente o all'interno di un becher più grande per 20-30 minuti a 121 °C, 1 atm.
NOTA: I componenti della colonna possono cambiare forma e colore dopo la sterilizzazione in autoclave, ma manterranno le proprietà del materiale desiderate. Le dimensioni, la forma e il colore finali dei componenti della colonna dopo la sterilizzazione in autoclave sono mostrati nella Figura 1.
5. Preparazione dei supporti per le colonne
- Preparare piastre di Petri da 90 mm di terreni sterilizzati a base di agar: carbossimetilcellulosa di sodio (CMC), agar estratto di malto (MEA), agar destrosio di patate (PDA) o agar 2A di Reasoner (R2A)9,10. Preparare il supporto seguendo le istruzioni del produttore.
- Sterilizzare il terreno in autoclave per 21 minuti o il tempo consigliato dal produttore a 121 °C, 1 atm, e versare in piastre di Petri da 90 mm fino a quando l'agar non si trova vicino alla parte superiore dei lati delle piastre di Petri (Figura 2). Eseguire questo passaggio in una cabina di sicurezza biologica per aumentare la sterilità.
- Lasciare solidificare e asciugare l'agar secondo le istruzioni del produttore.

Figura 2: Processo di assemblaggio delle colonne autostradali fungine. Utilizzando un'estremità aperta della colonna stessa, viene ritagliato e inserito un tappo e il ricercatore torce la colonna mentre viene rimossa dal supporto per garantire che il tappo rimanga all'interno dell'estremità della colonna. Quell'estremità è chiusa con il pezzo piccolo del tappo. Allo stesso modo, viene quindi aggiunto un connettore multimediale all'altra estremità della colonna. Il pezzo di rete viene quindi posizionato su questa estremità e fissato con l'anello filettato. Il tappo grande viene quindi utilizzato su questa estremità "esca" sopra l'anello filettato. Il lato con la rete verrà inserito nel substrato ambientale. Clicca qui per visualizzare una versione più grande di questa figura.
6. Preparazione delle colonne autostradali fungine
NOTA: Questa fase deve essere eseguita in una cabina di sicurezza biologica per mantenere la sterilità dei componenti della colonna e dei terreni. La Figura 2 illustra il processo di assemblaggio della colonna fungina dell'autostrada.
- Utilizzare l'estremità della colonna stessa (senza cappucci) per estrarre un tappo del supporto che si inserisca saldamente in un'estremità della colonna. Utilizzare un movimento rotatorio mentre la colonna viene sollevata dal terreno in modo che l'agar rimanga all'interno dell'estremità della colonna. In alternativa, utilizzare l'estremità della colonna come modello, ritagliare il terreno e trasferirlo nella colonna utilizzando la punta di una pipetta.
- Dopo aver aggiunto il primo tappo all'estremità della colonna, aggiungere il tappo piccolo a questa estremità per mantenere un microambiente sterile per il terreno di destinazione.
- Capovolgere la colonna e ripetere il passaggio 6.1 per l'altra estremità della colonna.
- Tagliare un pezzo circolare di ~2 cm di diametro della rete di nylon autoclavata (dimensione dei pori di 25 μm) utilizzando forbici sterilizzate (Figura 1). Posare la rete sull'estremità esposta della colonna. Attorcigliare l'anello filettato su questa estremità del supporto dell'esca della colonna fissando la rete all'interno dei fili.
- Posizionare il cappuccio grande sul fondo della colonna sopra la rete e l'altra estremità dell'anello filettato e tenerlo acceso durante lo stoccaggio o il trasporto della colonna.
7. Pre-inoculare il substrato o il terreno di coltura con un fungo di interesse
NOTA: Questo passaggio è facoltativo.
- Inoculare un fungo di interesse (ad esempio, un fungo noto per creare autostrade fungine) sul substrato desiderato coltivando prima il fungo su un terreno di crescita fungino solido (ad esempio, MEA, PDA; preparato come descritto nel passaggio 5) all'interno di una capsula di Petri e trasferendo una piccola sezione (~ 1 cm) di agar con crescita fungina visibile sul substrato.
NOTA: Esperimenti precedenti9 hanno utilizzato un substrato di sterco pre-colonizzato con Coprinopsis cinerea per 10 giorni prima di aggiungere le colonne al substrato pre-colonizzato. - Invece di pre-colonizzare il substrato, aggiungere un fungo esca a una capsula di Petri o direttamente sul fondo del terreno di coltura all'interno della colonna utilizzando una piccola quantità di miceli fungini (ad esempio, dal passaggio di un anello sterile).
- Per la capsula di Petri, attendere che vi sia una crescita visibile che copre gran parte della piastra di Petri (circa il 50%-75% della piastra coperta), quindi eliminare la sezione del terreno di coltura dell'esca direttamente dalla piastra di Petri colonizzata più vicina al bordo esterno della crescita fungina (come descritto nel passaggio 6.1).
- Quando si precolonizza direttamente il mezzo dell'esca, attendere che ci sia una crescita chiara in tutto il mezzo dell'esca prima di aggiungere la colonna al substrato (questo probabilmente richiederà diversi giorni).
NOTA: Esperimenti precedenti9 hanno utilizzato terreni di 14 giorni pre-colonizzati con C. cinerea come terreno esca; I tempi di pre-colonizzazione variano a seconda del tasso di crescita del fungo, del tipo di terreno e delle condizioni di incubazione.
8. Preparazione dei trattamenti di controllo e delle repliche
- Preparare le colonne di controllo negativo (come descritto nei passaggi 1-6) e non inocularle né posizionare queste colonne nel substrato. Utilizzali per fornire una base di riferimento per le analisi successive e per valutare eventuali contaminazioni nel processo di preparazione.
- Includere almeno tre repliche di colonne per qualsiasi esperimento.
9. Aggiunta della colonna al substrato
- Allestire un microcosmo di laboratorio (ad esempio, terriccio all'interno di una scatola, un vaso o un tubo; Figura 3A) o portare le colonne in un sito di campo di interesse.
- Rimuovere il tappo grande dalla parte inferiore della colonna per esporre l'anello filettato e la rete.
- Aggiungere la colonna al substrato di interesse (Figura 3) seguendo le considerazioni specifiche per ciascun tipo di substrato descritte di seguito. Se necessario, e per ridurre al minimo i danni alla rete, praticare una depressione nel substrato utilizzando una mano guantata o un piccolo strumento (ad esempio, una piccola spatola) prima di inserire la colonna nel substrato.
- Letame: Le colonne sono state precedentemente utilizzate con successo nello sterco di cavallo fresco. Conservare lo sterco a 4 °C prima dell'uso e aggiungerlo alle camere, come le scatole magenta, prima di aggiungere le colonne. Inserire la colonna nel substrato per coprire 1-2 cm dell'altezza totale della colonna.
- Terreno: Inserire le colonne in modo che l'intera sezione inferiore si trovi all'interno del terreno (~1-2 cm di profondità del suolo) (Figura 3B). Le colonne possono poggiare sulla parte superiore del terreno o essere sepolte fino a sotto il piccolo tappo sul lato bersaglio.
- Rizosfera: inserire le colonne nel terreno che circonda le radici delle piante come indicato nel passaggio 9.3.2 mentre si angola e si posiziona la colonna vicino a determinate radici per aumentare le possibilità di catturare i microrganismi della rizosfera (Figura 3C).
- Substrati all'interno delle provette: aggiungere ~10 ml di un substrato di interesse a una provetta conica da 50 ml o simile e, se lo si desidera, bagnare il substrato (ad esempio, in ambienti con umidità molto bassa) utilizzando acqua pura. Inserire la colonna nel tubo in modo che il fondo sia interrato e non siano visibili parti dell'anello filettato o della rete (Figura 3A). Avvitare il coperchio della provetta da 50 ml e posizionarla in un rack per provette da 50 ml per mantenerla in posizione verticale.
10. Lasciare la colonna nel substrato
- Lasciare la colonna nel substrato per 3-21 giorni.
NOTA: La quantità di tempo che le colonne devono lasciare nei loro substrati dipende dal tasso di crescita di qualsiasi fungo esca utilizzato, dal tasso di crescita dei funghi che colonizzano il mezzo esca e dalle condizioni ambientali (ad esempio, le condizioni asciutte portano a una più rapida essiccazione del terreno, quindi non possono essere utilizzate per così tanto tempo). - Stabilire un ciclo luce/buio desiderato per l'esperimento; Esperimenti precedenti hanno mantenuto le colonne all'interno del laboratorio al buio, mentre le colonne sul campo sono state sottoposte alle condizioni locali giorno/notte9.
11. Rimozione della colonna dal substrato
- Dopo che la colonna è rimasta a contatto con il substrato per il periodo di tempo desiderato, rimuovere la colonna, scrollarsi di dosso con cura il substrato in eccesso e riaggiungere il tappo grande sul fondo della colonna sotto la rete per trasportare la colonna.
- Posizionare la colonna in un becher sterile o in una provetta da 50 mL per il trasporto. Mantenere la colonna in posizione verticale durante il trasporto.
12. Coltura di isolati dal mezzo target della colonna
- In un ambiente sterile come una cabina di sicurezza biologica, rimuovere il tappo grande del mezzo esca, l'anello filettato e la rete. Scartare la mesh.
NOTA: Se un ricercatore è interessato a quali organismi hanno colonizzato il mezzo esca, seguire i passaggi 12.3-12.4 con il tappo del mezzo esca oltre al tappo del mezzo bersaglio. - Rimuovere il tappo piccolo dall'estremità della colonna contenente il mezzo di destinazione.
- Estrarre il tappo del fluido target capovolgendo la colonna per consentire al tappo target di cadere dalla colonna o utilizzando pinze sterilizzate o punte di pipette per estrarre il tappo. Posizionare il tappo del terreno target direttamente al centro di una piastra di Petri da 90 mm riempita con un terreno di agar a scelta (come preparato al punto 5).
NOTA: I mezzi specifici per funghi o batteri possono essere utilizzati qui per migliorare la crescita dei membri di entrambi i regni; il tappo può anche essere diviso in due o più pezzi utilizzando forbici sterilizzate o una punta per pipetta, consentendo la placcatura su più tipi di terreno e/o preservando il terreno target per l'estrazione del DNA (vedere il passaggio 14). - Incubare la capsula di Petri inoculata con il tappo del mezzo bersaglio per almeno 72 ore a 25 °C al buio.
13. Subcoltura per isolare microrganismi
NOTA: Questo passaggio è facoltativo.
- Per i batteri: utilizzando un circuito di inoculazione sterile, scorrere su una singola colonia di interesse riducendo al minimo il contatto con altre aree della capsula di Petri. Strisciare la colonia su una piastra di Petri fresca da 90 mm contenente il mezzo batterico preferito, come R2A (come preparato nel passaggio 5), utilizzando qualsiasi metodo che consenta la formazione di singole colonie (ad esempio, strisciando in quattro zone). Incubare la capsula di Petri per 24-72 ore a 25-37 °C, controllando la crescita delle colonie.
- Per i funghi: utilizzando un'ansa di inoculazione sterile, un rasoio sterile o una punta di pipetta sterile, ritagliare una sezione minima (1 mm2) di agar contenente la crescita ifale. Posizionare il pezzetto di agar su una piastra di Petri fresca da 90 mm contenente il terreno fungino preferito, come MEA o PDA, come preparato al punto 5. Incubare la capsula di Petri per un massimo di una settimana a 25 °C al buio, controllando quotidianamente per prevenire la crescita eccessiva.
- Ripetere il passaggio 1 o il passaggio 2 secondo necessità fino a quando le morfologie dell'organismo non sono uniformi.
14. Estrazione del DNA dalla lastra o direttamente dal mezzo target
- Congelare tutti i tappi di agar (bersaglio e/o esca) o i pezzi selezionati dalle colonne in provette da centrifuga da 1,5 ml a -20 °C o immergere i pezzi dei tappi in un conservante in una provetta da centrifuga da 1,5 ml prima dell'estrazione, se le estrazioni non avvengono immediatamente.
- Se è stata eseguita la coltura dal bersaglio e/o dal terreno di coltura e se sono state eseguite le successive fasi di subcoltura, stampigliare o ritagliare un pezzo di agar di ~1 cm con crescita fungina o batterica (o un mix di entrambi se non sono state eseguite le successive fasi di isolamento).
- Per le estrazioni di colonie batteriche isolate, estrarre una colonia dalla piastra utilizzando un circuito di inoculazione sterile e agitare direttamente nel tampone di estrazione associato al kit di estrazione del DNA commerciale (passaggio 14.4).
- Macinare i pezzi di agar separatamente in azoto liquido utilizzando un mortaio e un pestello e trasferire il tessuto macinato nelle provette di estrazione.
- Utilizzare un kit di estrazione del DNA commerciale ottimizzato per il suolo o per batteri e funghi e seguire le istruzioni del produttore per estrarre il DNA (vedere la Tabella dei materiali).
- Quantificare il DNA risultante utilizzando un fluorimetro o un sistema comparabile.
15. Valutazione della diversità tassonomica microbica nel terreno bersaglio e/o esca utilizzando approcci di sequenziamento ampliconico o metagenomico
- Eseguire il sequenziamento degli ampliconi (16S e/o spaziatore trascritto interno [ITS]) o il sequenziamento metagenomico seguendo i passaggi seguenti:
- Sequenziamento dell'amplicona: amplificare la regione del gene RRNA V3-V4 16S utilizzando i primer Bakt 341F (5'-CCT ACG GGN GGC WGC AG-3') e Bakt 805R (5'-GAC TAC HVG GGT ATC TAA TCC-3'). Amplificare la regione fungina ITS2 utilizzando i primer ITS3 KYO2 (5′-GAT GAA GAA CGY AGY RAA-3′) e ITS4 (5′-TCC TCC GCT TAT TGA TAT GC-3′)9.
- Prepara le librerie di amplificatori utilizzando kit di preparazione di librerie commerciali compatibili con il sequencer scelto. Sequenziare gli ampliconi utilizzando un sequenziatore a lettura breve per generare 150 o 250 letture di coppie di basi con una copertura sufficiente, seguendo le istruzioni del produttore della piattaforma di sequenziamento per la concentrazione di carico.
- Sequenziamento metagenomico: crea una libreria metagenomica dal DNA estratto utilizzando un kit di preparazione della libreria metagenomica disponibile in commercio compatibile con il sequenziatore scelto. Sequenziare la libreria del metagenoma utilizzando un sequenziatore a lettura breve impostato per generare 150 o 250 letture paired-end di coppie di basi con copertura sufficiente (~10-20 Gb per metagenoma) seguendo le istruzioni del produttore della piattaforma di sequenziamento per la concentrazione di carico.
16. Analisi dei dati di sequenziamento
- Analizza i dati degli ampliconi: utilizza la piattaforma QIIME2 11 con DADA212 per analizzare i dati degli ampliconi prima di visualizzare i risultati13. Segui i passaggi seguenti per eseguire QIIME2 nella piattaforma bioinformatica basata sul Web Empowering the Development of Genomics Expertise (EDGE)14. QIIME2 può essere eseguito anche utilizzando tutorial disponibili pubblicamente come la guida "Moving Pictures" fornita online (https://docs.qiime2.org/2024.2/tutorials/moving-pictures/).
- Vai su https://edgebioinformatics.org/ e accedi o crea un account.
- Seleziona RUN QIIME2 dalla homepage. Selezionare Carica file nella barra dei menu a sinistra per caricare i file dalle esecuzioni di sequenziamento di amplicon.
- Crea un file di mappatura dei metadati utilizzando le istruzioni fornite quando si passa il mouse sulla "i" accanto a "File di mappatura dei metadati".
- Aggiungi tutte le informazioni richieste (nome del progetto/esecuzione, tipo di lettura, parametri, ecc.) e seleziona i dati di input caricati corretti. Assicurarsi che DADA2 sia selezionato come metodo di controllo qualità12; Gli altri parametri possono essere lasciati come valori predefiniti, a meno che non si desiderino altre modifiche.
- Seleziona il tipo di amplicone come 16S Greengenes (http://greengenes.lbl.gov) per gli ampliconi batterici o Fungal ITS per gli ampliconi fungini. Analizza i dati batterici (16S) e fungini (ITS) in modo indipendente.
- Premere Invia e attendere il completamento dell'esecuzione per visualizzare i risultati.
- Generare visualizzazioni di dati amplicon (facoltativo): eseguire ulteriori analisi dei dati della community in R usando pacchetti comuni come phyloseq (https://github.com/joey711/phyloseq), VEGAN (https://github.com/vegandevs/vegan) e APE (https://emmanuelparadis.github.io/) seguendo le indicazioni fornite in GitHub e tramite la Guida del pacchetto R disponibile in R Studio passando al menu Pacchetti , facendo clic sul pacchetto scaricato e visualizzando la documentazione.
- Analizza i dati metagenomici
- Passare al sito NMDC EDGE 15 (https://nmdc-edge.org/home) ed effettuare l'accesso utilizzando un account ORCiD (https://orcid.org/).
- Seleziona Carica file nella barra dei menu sul lato sinistro dello schermo e trascina e rilascia o cerca i file FASTQ di input corretti.
- Seleziona Metagenomics, quindi l'opzione Esegui più flussi di lavoro nella barra dei menu sul lato sinistro dello schermo e imposta tutti i flussi di lavoro su Attivato. Aggiungi un nome al progetto e una descrizione facoltativa.
- Selezionare i file di lettura raw (fastq) caricati e selezionare il formato di file appropriato (interlacciato o accoppiato).
- Avvia l'esecuzione facendo clic su Invia e visualizzando le tabelle di riepilogo e le visualizzazioni quando l'esecuzione è stata completata selezionando il progetto nella scheda I miei progetti nella parte superiore dello schermo.
17. Creazione di visualizzazioni aggiuntive dei dati della tassonomia da risultati ampliconi e/o metagenomici
- Genera cladogrammi circolari utilizzando il pacchetto GraPhlAn (https://github.com/biobakery/graphlan) seguendo le istruzioni fornite in GitHub.
NOTA: Gli identificatori di tassonomia (https://www.ncbi.nlm.nih.gov/taxonomy) del National Center for Biotechnology Information (NCBI) possono essere ottenuti da assegnazioni di sequenze rappresentative e passati al programma "eftech" di Entrez Direct E-utilities per raccogliere le informazioni di rollup della tassonomia richieste da GraPhlAn16.
18. Riutilizzo delle colonne
- Se le colonne sono ancora assemblate, svitare e rimuovere i tappi, l'anello filettato e la rete. Scartare la mesh. Rimuovere eventuali tappi di agar rimanenti, lavare le colonne con alcol isopropilico al 99% e acqua purificata, quindi pulire e asciugare i componenti come descritto nei passaggi 3.1-3.4.
- Preparare nuovi fogli di rete di nylon da sterilizzare in autoclave.
- Autoclavare i componenti seguendo le indicazioni fornite al punto 4.1.
Risultati
La colonna dell'autostrada fungina completamente assemblata è lunga circa 5 cm (Figura 1). La colonna non deve essere rotta in nessuna area e i cappucci e l'anello filettato devono combaciare facilmente e saldamente per creare microambienti all'interno della colonna. La rete del filtro può estendersi oltre l'anello filettato (come mostrato nella Figura 1 e nella Figura 2), oppure può essere tagliata con forbici sterilizzate. I tappi di agar devono adattarsi perfettamente a ciascuna estremità della colonna. Quando viene posizionata nel substrato, la rete del filtro deve entrare in contatto con il substrato e la colonna non deve essere completamente interrata .
Le colonne sono state precedentemente testate in sterco di cavallo9. Le colonne sono state anche collocate in terreno sfuso e rizosferico in un sito di ricerca, nonché in piccoli volumi di terreno in provette da 50 mL in laboratorio (Figura 3). Dopo che le colonne dell'autostrada fungina sono state rimosse dal substrato e smontate, la crescita microbica era visibile sia sull'esca che sui tappi del terreno bersaglio (esempi mostrati nella Figura 4A). Batteri e funghi sono stati isolati da terreni bersaglio ed esca tramite tecniche di subcoltura (Figura 4B) e i microbi presenti sui tappi dei terreni sono stati identificati tassonomicamente utilizzando il sequenziamento degli ampliconi (Figura 4C, D). Le Figure 4C,D illustrano i risultati combinati del sequenziamento degli ampliconi in più esperimenti, mostrando quali microbi sono stati in grado di raggiungere il tappo del terreno bersaglio dalle colonne aggiunte allo sterco di cavallo9. Le visualizzazioni di questi dati batterici e fungini sono state generate come descritto nel passaggio 17. I risultati possono anche essere visualizzati come abbondanze relative di taxa.
Risultati non ottimali sono stati ottenuti nei casi in cui le colonne sono state aggiunte ad ambienti con umidità estremamente bassa e i tappi dei mezzi sono stati completamente essiccati nel giro di pochi giorni, senza portare al recupero dei microbi colonizzati (Figura 5A). Abbiamo anche visto casi in cui i microbi semplicemente non crescono dal tappo del terreno target (Figura 5B) e casi in cui non recuperiamo dati di sequenziamento sufficienti dal plug del terreno target per analisi significative. Anche altri casi in cui i funghi crescono eccessivamente fuori dalle colonne hanno comportato la necessità di rifare gli esperimenti (Figura 5C).

Figura 3: Esempi di colonne collocate in campioni ambientali in laboratorio e sul campo. (A) Colonna collocata all'interno di una provetta da 50 mL con terreno inumidito in un ambiente di laboratorio. Mostrato anche con un righello per la scala. (B) Colonna collocata nel terreno nel campo. (C) Colonna inserita nella rete radicale di una pianta in campo. Clicca qui per visualizzare una versione più grande di questa figura.
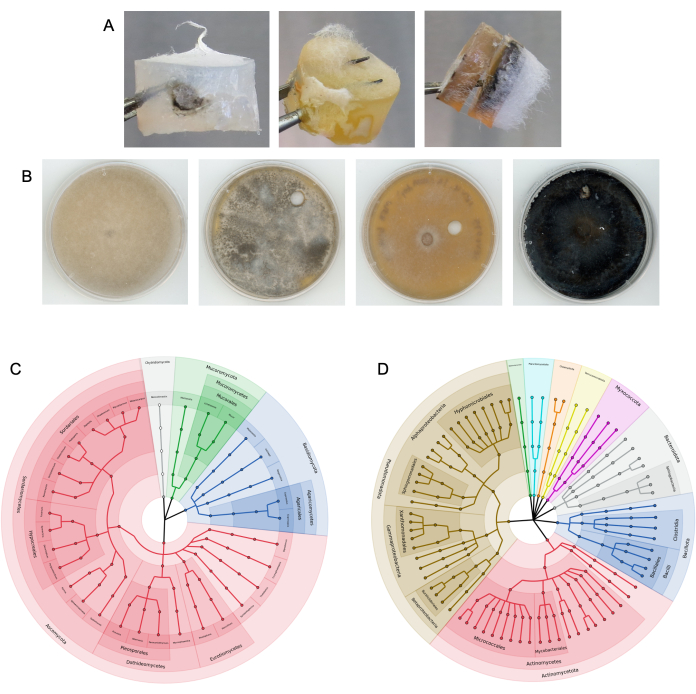
Figura 4: Risultati rappresentativi di esperimenti di successo su colonne. (A) Esempi di tappi di terreni colonizzati estratti da colonne. (B) Esempi di isolati fungini subcoltivati da terreni bersaglio. Il substrato era terra. Da sinistra a destra: Rhizopus azygosporous, Aspergillus novofumigatus, Curvularia subpapendorfii, e Phaeomycocentrospora cantuariensis. (C,D) cladogrammi circolari che mostrano la diversità filogenetica di (C) sequenze ITS fungine e (D) 16S batteriche recuperate dai terreni bersaglio a seguito di molteplici esperimenti sulle autostrade fungine utilizzando sterco di cavallo. Le sezioni sono colorate ed etichettate per phylum, con i nodi terminali che rappresentano generi unici. Clicca qui per visualizzare una versione più grande di questa figura.
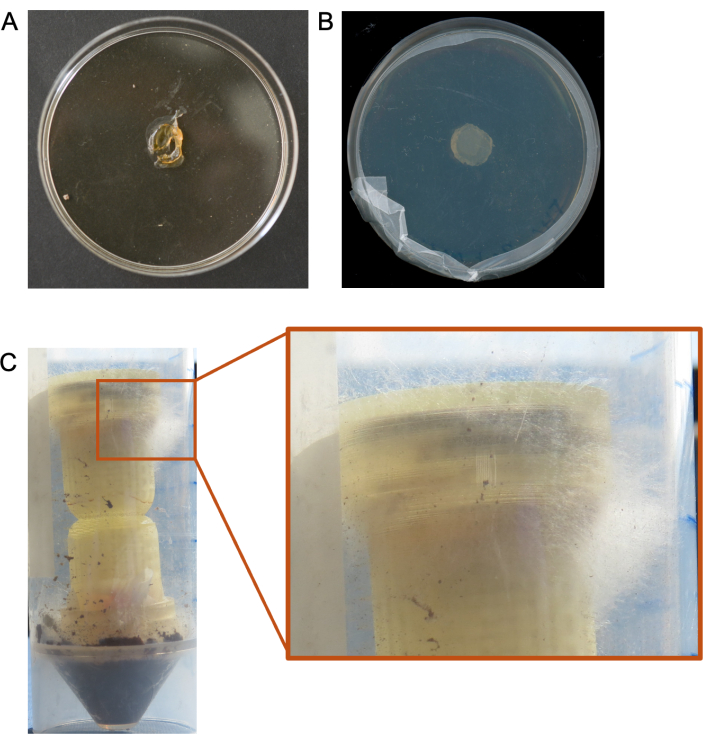
Figura 5. Risultati non ottimali dagli esperimenti su colonna. (A) Un esempio di tappo di media essiccato derivante da condizioni ambientali di bassa umidità. (B) Esempio di assenza di crescita microbica da un tappo di terreno di colonna. (C) Esempio di crescita eccessiva del fungo attraverso la parte superiore (mezzo bersaglio) della colonna. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Durante la generazione dei componenti della colonna, la selezione di una stampante 3D e del materiale di stampa può essere modificata in base alla disponibilità e alle proprietà del materiale desiderate17,18. La biocompatibilità, la struttura superficiale, l'autoclavabilità, la capacità di stampare dettagli su scala fine e la relativa trasparenza sono stati tutti considerati nella selezione dei materiali del nostro gruppo. Dovrebbero essere considerate anche altre caratteristiche, come porosità, idrofobicità, parametri di stampa, ecc. Varie resine sono state testate (vedi Tabella dei materiali) prima della selezione finale e molti materiali biocompatibili funzioneranno per la stampa di queste colonne. Il materiale scelto per la costruzione dei componenti della colonna determinerà quali approcci di pulizia, post-polimerizzazione e sterilizzazione devono essere utilizzati. Non tutti i materiali saranno autoclavabili e potrebbero essere necessari luce ultravioletta, candeggina o altre tecniche di sterilizzazione, a seconda delle istruzioni del produttore del materiale. Alcune tecniche di sterilizzazione o pulizia possono anche danneggiare o non essere compatibili con il materiale scelto, quindi è necessario prestare particolare attenzione a queste informazioni da parte del produttore del materiale. Per le stampanti 3D, alcune considerazioni includono il tempo di stampa, la compatibilità dei materiali, le dimensioni della piattaforma di stampa, la tecnologia di stampa e il costo19. I componenti stampati in 3D delle colonne possono essere fragili e rompersi se maneggiati con troppa forza. La filettatura dell'anello e dei cappucci potrebbe non essere sempre allineata esattamente, pertanto si consiglia di stampare e sterilizzare i componenti aggiuntivi prima della fase di assemblaggio, oppure di eseguire una stampa preliminare per verificare in che modo i parametri e il materiale scelto influiscono sulla filettatura. Potrebbe essere necessario regolare le specifiche di progettazione per la filettatura all'interno dei cappucci e dell'anello a seconda del materiale di stampa 3D scelto. Le dimensioni, la complessità del reticolo e altre caratteristiche fisiche possono essere modificate nel software di progettazione CAD prima della stampa. Come progettato, la colonna stessa è alta 4 cm e la struttura reticolare all'interno del centro della colonna ha una cella unitaria di 2 mm, un diametro del montante di 0,5 mm e l'intera altezza del reticolo è di 22 mm9. Questi parametri possono essere regolati se un ricercatore desidera, ad esempio, una struttura reticolare più grande o più complessa. Nel complesso, la produzione stampata in 3D di questi dispositivi consente flessibilità di progettazione, garantendo al contempo che un singolo progetto possa essere utilizzato in modo standardizzato tra organizzazioni e gruppi e persino utilizzato come strumenti didattici in classe9.
Diversi passaggi del protocollo possono richiedere la risoluzione dei problemi a seconda dell'ambiente o della configurazione sperimentale. Le colonne autostradali fungine non sono molto efficaci in condizioni di bassa umidità, poiché i tappi dei terreni si seccano rapidamente prima di facilitare la crescita dei funghi, il che può limitare la durata degli esperimenti in questi ambienti (Figura 5A). Le tecniche che hanno migliorato l'efficacia delle colonne in ambienti a bassa umidità includono l'aumento artificiale dell'umidità attraverso l'aggiunta di umidità al substrato e/o la sigillatura della colonna e del substrato in un contenitore secondario con una fonte d'acqua (ad esempio, un piccolo contenitore di acqua pura). La forma a clessidra e la struttura reticolare sono state incorporate per prevenire il movimento batterico da solo (senza la creazione di un'autostrada fungina) se la condensa dovesse formarsi in ambienti ad alta umidità. I funghi a crescita rapida possono crescere eccessivamente sulla superficie del bersaglio e del terreno di coltura dell'esca ed estendersi dalla parte superiore o inferiore della colonna (Figura 5C). Ridurre il tempo di incubazione del fungo esca o la durata dell'esperimento può ridurre al minimo o eliminare questa crescita eccessiva. Inoltre, una limitazione di questi dispositivi è che i funghi a crescita rapida nel substrato di interesse possono limitare la colonizzazione dell'esca e del terreno bersaglio da parte di funghi a crescita lenta, influenzando potenzialmente le interazioni autostradali osservate. Alcuni funghi, in particolare i funghi a crescita più lenta, potrebbero non colonizzare il terreno di coltura dell'esca in modo da consentire loro di crescere attraverso il tappo dell'agar e nella struttura reticolare. Se c'è sufficiente umidità nell'ambiente, è possibile utilizzare tappi di agar più sottili per incoraggiare la crescita nel reticolo dopo la colonizzazione del tappo di agar esca. I terreni possono essere scelti in base al fatto che un ricercatore voglia selezionare per la crescita fungina o batterica, ma questo può anche limitare la subcoltura agli organismi che preferiscono quel tipodi terreno 20. Se non si osserva alcuna crescita nel terreno di destinazione, potrebbe essere necessario inoculare il terreno di coltura o il substrato con un fungo noto per creare autostrade fungine.
Il sequenziamento metagenomico o ampliconico può essere eseguito come parte di questi esperimenti ed entrambe queste strategie impartiscono i propri limiti e punti di forza21. Il sequenziamento metagenomico è ideale per ottenere ulteriori informazioni genomiche sui microbi. Tuttavia, la quantità recuperabile di acidi nucleici direttamente dal terreno di destinazione può essere molto bassa, il che può richiedere l'utilizzo del sequenziamento degli ampliconi o di altri metodi di amplificazione prima del sequenziamento. Le librerie di sequenziamento degli ampliconi devono essere preparate separatamente (16S e ITS) e questo metodo manca di risoluzione tassonomica e limita qualsiasi valutazione sulle caratteristiche del genoma o sul potenziale funzionale che può essere ottenuta utilizzando il sequenziamento metagenomico. I metodi di sequenziamento diretto dai tappi possono essere preferiti nei casi in cui i microbi non possono essere subcoltivati. Si consiglia di dividere i tappi in più sezioni per consentire approcci sia di coltura che di sequenziamento.
Un vantaggio di questi dispositivi è che possono essere utilizzati sia in laboratorio che sul campo. Particolare attenzione deve essere posta per garantire che le colonne in campo possano rimanere in posizione verticale e siano protette da animali e perturbazioni ambientali che potrebbero disturbarne il posizionamento. Le colonne non sono ancora state testate in posizione orizzontale, in una posizione in cui sono completamente coperte da un substrato, e non sono state testate in ambienti esposti a piogge o neve consistenti. Come affermato in precedenza, la struttura reticolare è stata progettata per ridurre al minimo la probabilità che i batteri siano in grado di spostarsi verso il mezzo bersaglio in ambienti ad alta umidità. Tuttavia, è possibile che se la colonna fosse esposta a volumi d'acqua maggiori e quest'acqua saturasse completamente la colonna, il movimento batterico sarebbe facilitato in tutta la colonna indipendentemente da eventuali autostrade fungine presenti. Per esperimenti di laboratorio, le colonne possono essere utilizzate all'interno di provette coniche da 50 mL, piccoli microcosmi di substrati, nel terreno che circonda le piante in vaso, in scatole o all'interno di altri sistemi sperimentali controllati. Le colonne sono state utilizzate con successo nel suolo, nelle rizosfere e nello sterco e la loro utilità può essere estesa ad altri substrati, tra cui lettiera di foglie, fanghi, sabbia, neve, compost, ecc.
Le colonne dell'autostrada fungina consentono una serie di confronti per comprendere questo fenotipo BFI all'interno di diversi tipi di campioni. Il confronto della composizione della comunità tra l'esca e il terreno bersaglio può indicare quali batteri possono utilizzare le autostrade fungine e quali funghi possono fungere da potenziali autostrade9. Se si utilizza il sequenziamento del metagenoma, è possibile esaminare anche le caratteristiche genomiche che distinguono gli organismi dall'esca rispetto ai mezzi bersaglio. È anche possibile confrontare i mezzi target da colonne posizionate in substrati diversi (ad esempio, terreno o sterco) o posizionate nello stesso substrato in condizioni diverse (ad esempio, temperatura o umidità). Nel complesso, le colonne dell'autostrada fungina espandono le capacità dei metodi precedenti per interrogare questa forma di BFI e consentono esami approfonditi su queste interazioni che modellano le dinamiche spaziali di microbiomi ambientali complessi.
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Questa ricerca è stata supportata da una sovvenzione Science Focus Area dal Dipartimento dell'Energia degli Stati Uniti (DOE), Biological and Environmental Research (BER), Biological System Science Division (BSSD) con il numero di sovvenzione LANLF59T.
Materiali
| Name | Company | Catalog Number | Comments |
| 50 mL tubes | Greiner BIO-ONE | 5622-7261 | 50 mL tubes for performing column experiments in the lab |
| 90 mm Petri dishes | Thermo Scientific Nunc | 08-757-099 | Petri dishes for preparation of agar and for microbial growth |
| Asiga Freeform Pico Plus 39 digital light processing (DLP) 3D printer | Asiga Germany | Freeform Pico Plus 39 | 3D printer used to generate batches of the columns; other 3D printers can be used |
| Autoclave | Fisher Scientific | LS40F20 | Benchtop autoclave to sterilize the column components |
| Beaker | Fisher Scientific | FB100600 | 600 mL beaker for various uses throughout the protocol |
| Dental LT Clear Resin V2 | Formlabs | RS-F2-DLCL-02 | Alternative resin for 3D printing that was tested |
| Dental Surgical Guide Resin | Formlabs | RS-F2-SGAM-01 | Was used to generate the columns discussed in manuscript; Other photosensitive resins can be used in place of this material |
| DNA Low Bind 1.5 mL tubes | Eppendorf | 13-698-791 | Tubes used for various preparations including nucleic acid extractions |
| DNA/RNA shield preservative | Zymo Research | R1100-50 | Preservative used prior to nucleic acid extractions |
| EDGE Bioinformatics | Open source; Developed by the Los Alamos National Laboratory (LANL) | n/a | Bioinformatics platform for processing amplicon data |
| FastDNA spin kit for soil | MP Biomedicals LLC | 116560200-CF | DNA extraction kit option for soil |
| Forceps | Fisher Scientific | 10-300 | Forceps that can be sterilized |
| Formlabs BioMed Clear Resin | Formlabs | RS-F2-BMCL-01 | Alternative resin for 3D printing that was tested |
| Formlabs Form 3B+ stereolithography (SLA) 3D printer | Formlabs | Form 3B+ | Alternative 3D printer |
| Formlabs IBT Resin | Formlabs | RS-F2-IBCL-01 | Alternative resin for 3D printing that was tested |
| Inoculating Loops | Fisher Scientific | 22-363-598 | Used to isolate/transfer microbes |
| Malt Extract Agar (MEA) | Criterion | 89405-654 | A media type used in columns |
| MiSeq sequencer + MiSeq sequencing kit | Illumina | SY-410-1003 | Can use other sequencers |
| Mortar & Pestle | Fisher Scientific | FB961K; FB961A | Can use any common mortar & pestle that can be sterilized between uses |
| NEBNext Ultra II DNA Library Prep Kit for Illumina | New England Biolabs | E7805S | Library prep kit for metagenomic sequencing |
| Nextera XT DNA Library Preparation Kit (24 samples) | Illumina | FC-131-1024 | Library prep kit for amplicon sequencing |
| NMDC EDGE | Open source: Developed by the National Microbiome Data Collaborative (NMDC) | n/a | Bioinformatics platform for processing metagenomic data |
| Nylon mesh | Sefar | 03-25/19 | The mesh used as part of the column construction |
| Pipette tips | Rainin | 30807966 | Can use many different sterilized pipette tips for the protocol steps |
| Potato Dextrose Agar | Cole Parmer | EW-14200-28 | A media type used in columns |
| QIIME2 | Open source | n/a | Software for processing amplicon data |
| Qubit dsDNA HS assay kit | Thermo Fisher Scientific | Q32851 | Used to quantify DNA after extractions |
| Qubit Fluorometer | Thermo Fisher Scientific | Q33238 | Used to quantify DNA after extractions |
| Quick-DNA Fungal/Bacterial Miniprep Kit | Zymo Research | D6005 | DNA extraction kit option that works with both bacteria and fungi |
| R2A agar | BD Difco | 218263 | A media type used in columns (bacterial media) |
| Rack for 50 mL tubes | Fisher Scientific | 03-448-11 | Rack to hold 50 mL tubes upright |
| Scissors | Fisher Scientific | 12-000-155 | Fine precision scissors that can be sterilized |
| Sodium carboxymethyl cellulose medium | Aldrich | 419273-100G | A media type used in columns |
| SolidWorks CAD software | SolidWorks | n/a | Software used to design the columns |
| Trowel scoop | Fisher Scientific | S41701 | To make a depression in the substrate prior to adding the column |
| UltraPure DNase/RNase-Free Distilled Water | Invitrogen: ThermoFisher Scientific | 10977015 | Water for the ultrasonicator water bath |
| Ultrasonicator | Fisher Scientific | FB-11201 | Ultrasonicator for cleaning the columns |
Riferimenti
- Or, D., Smets, B. F., Wraith, J. M., Dechesne, A., Friedman, S. P. Physical constraints affecting bacterial habitats and activity in unsaturated porous media - A review. Adv Water Resour. 30 (6), 1505-1527 (2007).
- Kohlmeier, S., et al. Taking the fungal highway: Mobilization of pollutant-degrading bacteria by fungi. Environ Sci Technol. 39 (12), 4640-4646 (2005).
- Simon, A., Hervé, V., Al-Dourobi, A., Verrecchia, E., Junier, P. An in situ inventory of fungi and their associated migrating bacteria in forest soils using fungal highway columns. FEMS Microbiol Ecol. 93 (1), 217 (2017).
- Wick, L. Y., et al. Effect of fungal hyphae on the access of bacteria to phenanthrene in soil. Environ Sci Technol. 41 (2), 500-505 (2007).
- Bravo, D., et al. Isolation of oxalotrophic bacteria able to disperse on fungal mycelium. FEMS Microbiol Lett. 348 (2), 157-166 (2013).
- Furuno, S., Remer, R., Chatzinotas, A., Harms, H., Wick, L. Y. Use of mycelia as paths for the isolation of contaminant-degrading bacteria from soil. Microb Biotechnol. 5 (1), 142-148 (2012).
- Buffi, M., et al. Fungal drops: A novel approach for macro- and microscopic analyses of fungal mycelial growth. Microlife. 4, 042 (2023).
- Kuhn, T., et al. Design and construction of 3D printed devices to investigate active and passive bacterial dispersal on hydrated surfaces. BMC Biol. 20 (1), 203 (2022).
- Junier, P., et al. Democratization of fungal highway columns as a tool to investigate bacteria associated with soil fungi. FEMS Microbiol Ecol. 97 (2), 003 (2021).
- Reasoner, D. J., Geldreich, E. E. A new medium for the enumeration and subculture of bacteria from potable water. Appl Environ Microbiol. 49 (1), 1-7 (1985).
- Bolyen, E., et al. Reproducible, interactive, scalable, and extensible microbiome data science using QIIME 2. Nat Biotechnol. 37 (8), 852-857 (2019).
- Callahan, B. J., et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nat Methods. 13 (7), 581-583 (2016).
- Vázquez-Baeza, Y., Pirrung, M., Gonzalez, A., Knight, R. EMPeror: A tool for visualizing high-throughput microbial community data. Gigascience. 2 (1), 16 (2013).
- Li, P. -. E., et al. Enabling the democratization of the genomics revolution with a fully integrated web-based bioinformatics platform. Nucleic Acids Res. 45 (1), 67-80 (2017).
- Eloe-Fadrosh, E. A., et al. The National Microbiome Data Collaborative Data Portal: An integrated multi-omics microbiome data resource. Nucleic Acids Res. 50 (1), D828-D836 (2022).
- Entrez Direct: E-utilities on the Unix Command Line. Entrez Programming Utilities Help Available from: https://www.ncbi.nlm.nih.gov/books/NBK179288/ (2024)
- Palmara, G., Frascella, F., Roppolo, I., Chiappone, A., Chiadò, A. Functional 3D printing: Approaches and bioapplications. Biosens Bioelectron. 175, 112849 (2021).
- Guttridge, C., Shannon, A., O'Sullivan, A., O'Sullivan, K. J., O'Sullivan, L. W. Biocompatible 3D printing resins for medical applications: A review of marketed intended use, biocompatibility certification, and post-processing guidance. Ann 3D Print Med. 5, 100044 (2022).
- Yao, L., et al. Comparison of accuracy and precision of various types of photo-curing printing technology. J Phys Conf Ser. 1549 (3), 032151 (2020).
- Basu, S., et al. Evolution of bacterial and fungal growth media. Bioinformation. 11 (4), 182-184 (2015).
- Liu, Y. -. X., et al. A practical guide to amplicon and metagenomic analysis of microbiome data. Protein Cell. 12 (5), 315-330 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon