Method Article
Disección de ovarios, intestino medio y glándulas salivales de mosquitos para análisis del microbioma a nivel de órganos
En este artículo
Resumen
Las comunidades microbianas de los mosquitos son muy prometedoras para las estrategias de biocontrol de vectores. La mayoría de los simbiontes son incultivables, lo que requiere análisis metagenómicos. Describimos un método para diseccionar mosquitos hembra y separar los ovarios, el intestino medio y las glándulas salivales evitando la contaminación cruzada, facilitando los estudios del microbioma a nivel de órgano y mejorando la comprensión de las funciones de los microorganismos en la biología de los mosquitos.
Resumen
La carga mundial de enfermedades transmitidas por mosquitos, como el paludismo, el dengue, el virus del Nilo Occidental, el zika, el usutu y la fiebre amarilla, sigue aumentando, lo que supone una importante amenaza para la salud pública. Con el aumento de la resistencia a los insecticidas y la ausencia de vacunas eficaces, están surgiendo nuevas estrategias centradas en la microbiota del mosquito. Sin embargo, la mayoría de los simbiontes siguen siendo resistentes al cultivo. Por lo tanto, la caracterización de la diversidad y la función de los genomas bacterianos en especímenes de mosquitos se basa en la metenómica y en las estrategias posteriores de ensamblaje y agrupación. La obtención y el análisis de genomas ensamblados por metagenomas (MAG) a partir de órganos separados puede proporcionar información clave sobre el papel específico de los microbios asociados a los mosquitos en los ovarios (los órganos reproductivos), el intestino medio (clave para la digestión de los alimentos y la inmunidad) o las glándulas salivales (esenciales para la transmisión de enfermedades transmitidas por vectores, ya que los patógenos deben colonizarlas para entrar en la saliva y llegar al torrente sanguíneo durante una ingesta de sangre). Estos genomas recién reconstruidos pueden allanar el camino para el desarrollo de nuevas estrategias de biocontrol de vectores. Para ello, es necesario aislar los órganos de los mosquitos evitando la contaminación cruzada entre ellos o con microorganismos presentes en otros órganos de los mosquitos. Aquí, describimos un protocolo de disección optimizado y libre de contaminación para estudiar el microbioma de mosquitos a nivel de órganos.
Introducción
Los mosquitos propagan una amplia gama de patógenos que causan enfermedades y son una grave amenaza para la salud pública. Debido a una mayor prevalencia de resistencia a los insecticidas entre las poblaciones de mosquitos y en ausencia de vacunas efectivas contra estos patógenos, están surgiendo nuevos métodos de control biológico que se centran en el microbioma del mosquito. En particular, se destaca la bacteria intracelular Wolbachia, que puede interferir con la transmisión de patógenos y manipular la reproducción del huésped 1,2,3. Además, otros mosquitos simbiontes son fundamentales para la supervivencia, el desarrollo o el sistema inmunitario de su huésped, así como para la infección y la transmisión de patógenos, y son muy prometedores para su explotación en la lucha contra las enfermedades transmitidas por vectores 4,5,6,7,8.
Los microorganismos asociados a los mosquitos abarcan todos los dominios de la vida microbiana (incluyendo bacterias, eucariotas y hongos) que interactúan íntimamente con su huésped pero también entre sí en los diferentes compartimentos del cuerpo 9,10. Por lo tanto, una mejor comprensión de la microbiota, de su potencial variación estacional11 y de los mecanismos por los que sus miembros interactúan directamente en los distintos tejidos de los mosquitos puede ayudar a desarrollar nuevos métodos de biocontrol específicos o mejorar los existentes. Sin embargo, la mayoría de los simbiontes siguen siendo resistentes al cultivo, lo que hace imposible su caracterización.
El advenimiento de los métodos de secuenciación de segunda y tercera generación, junto con los enfoques de ensamblaje y agrupamiento de última generación, han permitido la reconstrucción de los genomas microbianos y el acceso a la diversidad y el potencial funcional de los microbios no cultivables. Aquí, presentamos un método para la disección de los ovarios, el intestino medio y las glándulas salivales de mosquitos hembra mientras se previene la contaminación cruzada. Este protocolo puede ser seguido por la extracción genómica de ADN y la posterior metabarcoding o secuenciación metagenómica de escopeta para explorar la diversidad y la función de la microbiota de mosquitos a nivel de órganos. Proporcionamos un ejemplo de disección de mosquitos y datos de microbioma para especímenes de Culex spp., aunque este protocolo puede extenderse a vectores de otros géneros como Anopheles o Aedes.
Protocolo
NOTA: La Figura 1 muestra un esquema del método que resume los diferentes pasos del protocolo.
1. Preparación del material
- Antes de comenzar el experimento, prepare una solución de lejía con un 0,5% de cloro activo (recomendado por la Organización Mundial de la Salud) para desinfectar el equipo y el banco de trabajo.
- En 900 ml de agua, disuelva tres comprimidos solubles, cada uno con 1,5 g de cloro activo. Espere 15 minutos para que las tabletas se disuelvan por completo, lo que da como resultado una solución de lejía al 0,5%.
- Coloca la solución de lejía al 0,5% en una botella con atomizador. Luego, rocíe la solución en todo el equipo de disección, incluidos los portaobjetos microscópicos y el banco de trabajo.
NOTA: El equipo y el banco de trabajo deben dejarse secar durante ~ 10 minutos antes de su uso.
- Para enjuagar los mosquitos antes de cada disección y evitar la contaminación de las muestras con bacterias externas, prepare cuatro tubos recién esterilizados de 1,5 mL: dos que contengan etanol al 96% y otros dos que contengan 1x tampón PBS (solución salina tamponada con fosfato).
- Prepare los tubos para almacenar las muestras después de la disección de cada mosquito: cuatro tubos estériles de 1,5 mL, cada uno con 100 μL de tampón de conservación.
NOTA: Prepare un tubo para las glándulas salivales, uno para los ovarios, uno para el intestino medio y otro para el cadáver del mosquito.
2. Recolección de mosquitos
- Recoja y mantenga vivos a los individuos femeninos adultos (en el campo o en el laboratorio, de 4 a 7 días) hasta la esterilización en la superficie.
3. Esterilización superficial de mosquitos
NOTA: Limpie las pinzas y agujas y use tubos nuevos y esterilizados entre cada órgano y cada individuo. Si no se dispone de lejía, asegúrese de esterilizar a fondo con llama las pinzas y agujas de disección entre la disección de cada individuo y órgano para evitar la contaminación cruzada del material biológico. Los instrumentos de disección deben estar secos, sin ningún residuo de lejía/etanol, antes del próximo uso.
- Para evitar que las hembras vacíen su contenido intestinal cuando se exponen al alcohol, anestesiarlas en frío con hielo o en un congelador a -20 °C (durante 4 minutos para que los tejidos y órganos no se congelen).
- Transfiera los mosquitos individuales a un nuevo tubo esterilizado de 1,5 mL con 1 mL de etanol al 96%. Agite suavemente el mosquito (manualmente) durante 30 s (agite bien el mosquito en el tubo para separar las escamas y los posibles contaminantes bacterianos en la superficie de los insectos). Enjuague dos veces con tubos que contengan etanol.
- Transfiera el mosquito a un nuevo tubo esterilizado de 1,5 mL con 1 mL de solución estéril 1x PBS y haga un vórtice suavemente (manualmente) durante 30 s (para evitar la precipitación de ADN con etanol). Enjuague dos veces con tubos que contengan PBS estéril 1x.
4. Disección de mosquitos
- Coloque varias gotas pequeñas de 1x PBS estéril en el portaobjetos para facilitar la colocación del mosquito y el enjuague de los órganos disecados.
NOTA: Las gotas de PBS estéril deben ser lo suficientemente grandes como para acomodar los órganos del mosquito. - Tome el mosquito limpio y colóquelo en una gota de PBS estéril 1x en el portaobjetos del microscopio.
- Diseccione las glándulas salivales con pinzas y aguja esterilizadas como se describe a continuación.
- Sostén el tórax del mosquito con pinzas para evitar que se mueva. Luego, coloque una aguja en el cuello, debajo de la cabeza, y tire suavemente de la cabeza del mosquito para separarlo del tórax.
NOTA: Las glándulas salivales se encuentran en la parte superior del tórax y están conectadas a la cabeza por el conducto salival. Durante la disección, es crucial no romper este conducto, ya que las glándulas salivales no vendrán con la cabeza cuando la saque del tórax. Por eso es importante tener un mosquito fresco, no uno congelado. - Cuando la cabeza se separa del tórax, observa cómo emergen las dos glándulas salivales, cada una compuesta por tres lóbulos. Corte las glándulas salivales a nivel del conducto salival, recójalas con una aguja y colóquelas en una nueva gota de PBS estéril 1x para enjuagarla y evitar contaminaciones interorgánicas.
- Recupere las glándulas salivales con una aguja y colóquelas en un nuevo tubo esterilizado que contenga 100 μL de tampón de conservación.
- Sostén el tórax del mosquito con pinzas para evitar que se mueva. Luego, coloque una aguja en el cuello, debajo de la cabeza, y tire suavemente de la cabeza del mosquito para separarlo del tórax.
- Diseccione los ovarios con pinzas esterilizadas y aguja de la siguiente manera.
- Coloca el mosquito boca arriba. Usando fórceps para sujetar el mosquito por el tórax para evitar el movimiento durante la disección, perfore el tercer segmento abdominal (comenzando desde la parte inferior) con una aguja.
NOTA: Opcionalmente, retire las patas y alas del mosquito para facilitar la disección. - Mientras perforas el tercer segmento abdominal, tira hacia abajo del abdomen para abrirlo y revelar los órganos internos. Retire los restos del exoesqueleto del mosquito para facilitar la liberación de los órganos.
NOTA: Los órganos del abdomen, como el intestino, los túbulos de Malpighi y los ovarios, son visibles. - Recupere ambos ovarios con pinzas esterilizadas y colóquelos en una nueva gota de 1x PBS estéril en el portaobjetos del microscopio para enjuagarlos. Agarre los ovarios y colóquelos en el tubo de 1,5 ml con 100 μl de tampón de conservación.
- Coloca el mosquito boca arriba. Usando fórceps para sujetar el mosquito por el tórax para evitar el movimiento durante la disección, perfore el tercer segmento abdominal (comenzando desde la parte inferior) con una aguja.
- Ahora que el abdomen está abierto y los órganos están expuestos, recupere el intestino medio con pinzas y/o aguja nuevas esterilizadas.
- Separe el intestino medio del intestino anterior y el intestino posterior; luego, retire los túbulos de Malpighi del intestino medio. Agarre el intestino medio con una aguja estéril y colóquelo en una nueva gota estéril de PBS 1x para enjuagarlo y evitar contaminaciones interorgánicas.
- Transfiera a una nueva gota estéril 1x PBS. Abra el intestino medio con pinzas para liberar y enriquecer las bacterias como se describe en el Archivo Suplementario 1. Agarre el intestino medio con pinzas y pipetee todas las bacterias que contengan PBS para almacenar todo el material (líquido y tejido) en un nuevo tubo esterilizado de 1,5 ml con 100 μL de tampón de conservación.
- Añada de nuevo 5 μL de PBS 1x en el lugar utilizado anteriormente en el portaobjetos del microscopio y vuelva a pipetear directamente el PBS en el mismo tubo de 1,5 mL para recuperar las bacterias que puedan haberse adherido al portaobjetos del microscopio.
- Utilice pinzas estériles para agarrar el cadáver (cuerpo restante) y guárdelo en un nuevo tubo esterilizado de 1,5 ml con 100 μl de tampón de conservación.
- Almacenamiento del material hasta su posterior procesamiento
- Etiquete los tubos con 'Especie, Órgano, Fecha, Nombre del investigador'.
- Almacene los ovarios, el intestino medio, las glándulas salivales y la canal disecados a -20 °C hasta su procesamiento.
- Realice un seguimiento en una hoja de cálculo de todos los metadatos asociados a las muestras, incluida la especie (solo a nivel de género o género), la fecha de recolección, el país, la provincia, la latitud, la longitud, el nombre de la persona que realizó la disección, la hembra grávida o no, la harina de sangre o no.
Resultados
x'Disecciones de mosquitos
Siguiendo el protocolo, recolectamos y aislamos los ovarios, el intestino medio, las glándulas salivales y el cadáver de dos especímenes de Culex pipiens molestus (incluida una hembra grávida) de una colonia de laboratorio. Confirmamos disecciones limpias después de la observación de órganos completos (es decir, intactos) y bien aislados, sin restos restantes bajo los binoculares. En la Figura 2 se muestra todo el cuerpo, la cabeza y el tórax, las glándulas salivales disecadas, los ovarios y el intestino medio de un espécimen de Culex pipiens molestus. Como era de esperar, el intestino medio y las glándulas salivales eran un orden de magnitud más pequeños que los ovarios de los mosquitos. Los huevos de un espécimen grávido de Culex, junto con su intestino medio y túbulos de Malpighi, se presentan en la Figura 3. Cabe destacar que las disecciones son más propensas a fallar cuando el material no está fresco y es probable que el tejido se rompa (Figura 4B, C). Por lo tanto, sugerimos diseccionar el material inmediatamente después de la recolección (dentro de menos de 12 h) cuando sea posible, ya que los tejidos aún son elásticos (Figura 4A). Del mismo modo, se puede realizar la disección del material congelado, pero existe un riesgo mucho mayor de fracaso y contaminación cruzada entre órganos debido a la fragilidad de los tejidos (Figura 4).
Datos del microbioma
Además, recolectamos y separamos el intestino medio y los ovarios de tres especímenes individuales de Culex quinquefasciatus de Noumea, Nueva Caledonia, siguiendo el mismo procedimiento. Extrajeron ADN de cada órgano, preparamos muestras para la secuenciación del genoma completo y realizamos análisis de datos del microbioma como se detalla en el Archivo Suplementario 1. Un análisis de diversidad taxonómica bacteriana en lecturas cortas filtradas de calidad sin ensamblar utilizando PhyloFlash12 mostró distintos taxones dominantes en el intestino medio en comparación con los ovarios (Figura 5A). En particular, las comunidades bacterianas ováricas estaban dominadas por Wolbachia, con la presencia adicional de un filotipo de Spirochaetaceae en los ovarios del individuo 3, mientras que las comunidades del intestino medio exhibían una diversidad más amplia, incluidas Gammaproteobacterias, Spirochaetaceae y Firmicutes. A partir de los mismos datos de secuenciación, reconstruimos cuatro Genomas Ensamblados con Metagenoma (MAG) con un > de finalización del 80% y una redundancia < 5%, pertenecientes a las clases Spirochaetia, Alpha- y Gammaproteobacteria (Tabla 1).
Como se esperaba, los MAG reconstruidos en este documento no cubrieron la diversidad taxonómica completa predicha por los resultados de PhyloFlash debido a las deficiencias específicas de la reconstrucción del genoma mediante lecturas cortas metagenómicas. El MAG asignado a Wolbachia (Ref. MAG 3 en la Tabla 1) se detectó en todos los ovarios y dos del intestino medio y tuvo mayor cobertura en los ovarios (Figura 5B). También reconstruimos dos MAG pertenecientes a la familia Enterobacteriaceae , incluyendo el género Pantoea (Ref. MAG 1 y 2 en la Tabla 1), en los intestinos medios obtenidos de los individuos 2 y 3 de Culex , que no fueron detectados en los ovarios correspondientes (Figura 5B). Finalmente, también reconstruimos un genoma bacteriano de Spirochaetaceae , Ref. MAG 4 (Tabla 1), asignado al género BR149 que fue aislado con éxito del intestino medio de Culex pipiens por Graña-Miraglia y colaboradores13. Curiosamente, este MAG se detectó en el intestino medio de los individuos 1 y 3, así como en los ovarios del individuo 3 (Figura 5B).
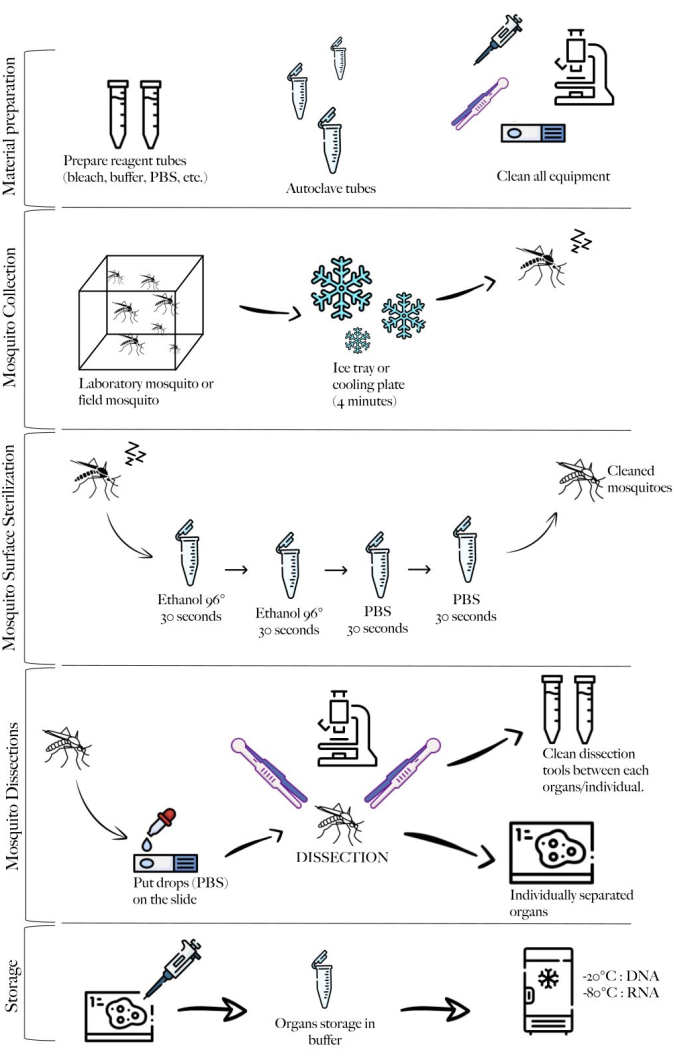
Figura 1: Esquema del método que resume los diferentes pasos. Preparación de materiales, recolección de mosquitos, limpieza de mosquitos, disecciones de mosquitos y almacenamiento. Haga clic aquí para ver una versión más grande de esta figura.
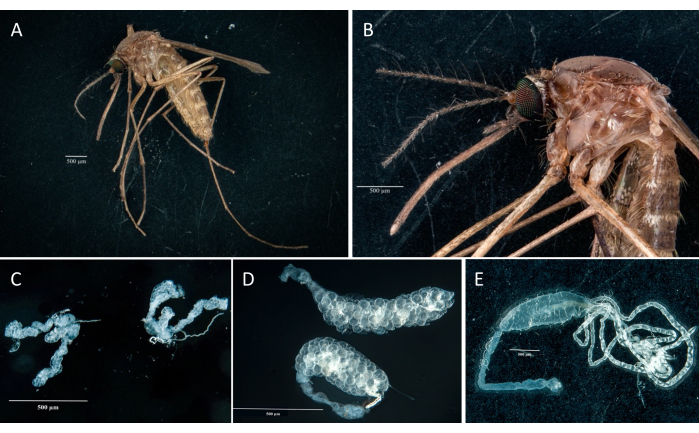
Figura 2: Culex pipiens molestus hembra. (A) Cuerpo entero. (B) Cabeza y tórax. (C) Glándulas salivales disecadas. (D) Ovarios disecados. (E) Intestino disecado con túbulos de Malpighi. Barras de escala = 500 μm. Haga clic aquí para ver una versión más grande de esta figura.
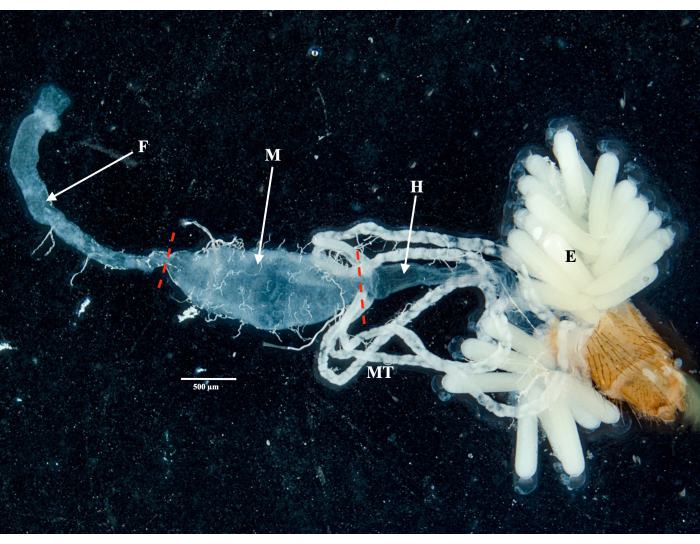
Figura 3: Abdomen disecado de una hembra grávida de Culex pipiens molestus . Se muestran el intestino anterior, el intestino medio, el intestino posterior, los túbulos de Malpighi y los huevos del espécimen. Los guiones rojos indican dónde cortar para separar el intestino medio del intestino anterior y el intestino posterior. Barra de escala = 500 μm. Abreviaturas: F = intestino anterior; M = intestino medio; H = intestino posterior; E = huevos; MT = Túbulos de Malpighi. Haga clic aquí para ver una versión más grande de esta figura.
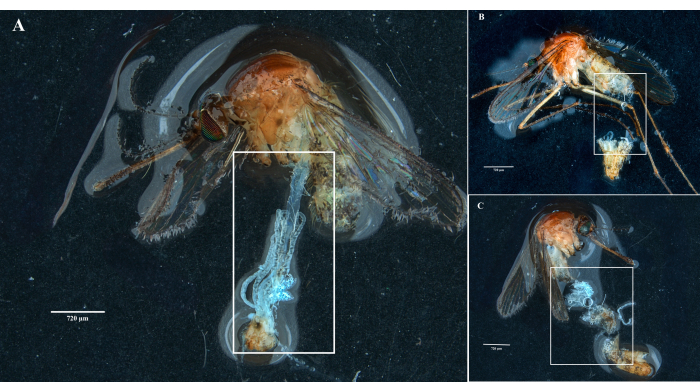
Figura 4: Disección de especímenes de mosquitos Culex frescos y congelados. Los rectángulos blancos ilustran (A) tejidos de mosquitos con órganos intactos de un espécimen recién diseccionado y (B,C) material biológico roto de especímenes que se han congelado antes de la disección. Barras de escala = 720 μm. Haga clic aquí para ver una versión más grande de esta figura.
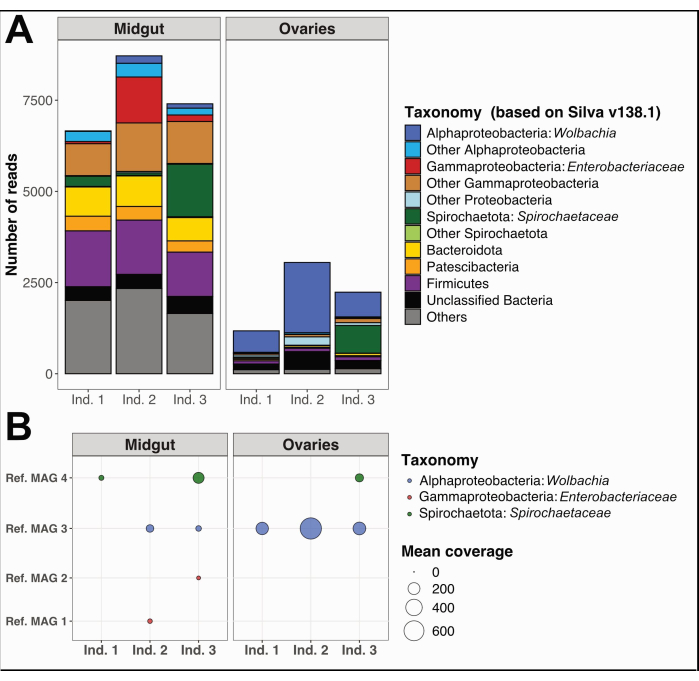
Figura 5: Ejemplo de análisis del microbioma en tres individuos de Culex quinquefasciatus . (A) Visualización de la diversidad bacteriana estimada a través de la extracción de lecturas de ARNr de SSU con PhyloFlash12 en el intestino medio y los ovarios de los tres especímenes. (B) Cobertura media de los MAG reconstruidos sobre las muestras. Abreviaturas: MAG = Genoma Ensamblado en Metagenoma; Ind = Individual. Haga clic aquí para ver una versión más grande de esta figura.
| MAG | Ref. MAG 4 | Ref. MAG 2 | Ref. MAG 1 | Ref. MAG 3 |
| Longitud (bp) | 1,287,790 | 4,910,866 | 4,751,276 | 1,298,266 |
| Número de contigs | 9 | 162 | 149 | 123 |
| GC % | 34.05 | 55.45 | 54.05 | 34.15 |
| Finalización (%) | 84.5 | 97.18 | 98.59 | 91.55 |
| Redundancia (%) | 0 | 2.82 | 4.22 | 0 |
| Dominio | Gérmenes | Gérmenes | Gérmenes | Gérmenes |
| Filo | Spirochaetota | Proteobacterias | Proteobacterias | Proteobacterias |
| Clase | Espiroquetía | Gammaproteobacterias | Gammaproteobacterias | Alfaproteobacterias |
| Orden | WRBN01 | Enterobacterales | Enterobacterales | Rickettsiales |
| Familia | WRBN01 | Enterobacteriaceae | Enterobacteriaceae | Anaplasmatáceas |
| Género | BR149 | Pantoea | - | Wolbachia |
Tabla 1: MAGs reconstruidos de los tres individuos de Culex quinquefasciatus . Tamaño del genoma, número de contigs, proporción de GC, estimaciones de finalización y redundancia basadas en la colección de genes centrales de una sola copia disponible en Anvi'o17 y la taxonomía obtenida utilizando GTDB18.
Archivo complementario 1: Ejemplo detallado de procedimiento para el análisis de datos del microbioma a partir de la recolección de muestras, la extracción de ADN y la secuenciación del genoma completo, seguido del flujo de trabajo bioinformático para la reconstrucción del genoma y la estimación de la proporción de lectura procariota y eucariota . Haga clic aquí para descargar este archivo.
Discusión
Recomendamos prestar especial atención a la secuencia de disección de los órganos, empezando por las glándulas salivales. De hecho, observamos que se extraían más fácilmente del tórax de los especímenes de Culex si se preservaba la integridad del mosquito. El daño en el abdomen o el tórax podría reducir la presión en el cuerpo del mosquito, lo que dificultaría el procedimiento. Sin embargo, también es posible cortar entre el tórax y el abdomen y luego extraer las glándulas salivales de la cabeza y el tórax (A. B. Failloux, comunicación personal). Además, adquirir habilidades de disección competentes puede ser un desafío, por lo que sugerimos practicar con un número adecuado de especímenes antes del experimento.
Aislar los órganos de los mosquitos y evitar la contaminación cruzada de manera sistemática es crucial para una amplia gama de análisis posteriores de la microbiota de los mosquitos. Un estudio metagenómico resuelto genómicamente tras la disección de ovarios individuales de especímenes de Culex pipiens en el sur de Francia permitió descubrir el primer plásmido de Wolbachia en Culex pipiens (pWCP14). Utilizando un enfoque similar, investigamos la distribución y frecuencia de pWCP en especímenes de Culex pipiens y Culex quinquefasciatus de áreas continentales e insulares de todo el mundo, en diversas condiciones ambientales y de laboratorio. En general, los datos revelaron un elemento plásmido Wolbachia notablemente conservado en los mosquitos Culex , lo que sugiere un papel crucial de este elemento móvil en la biología endosimbionte15, lo que justifica un análisis más detallado.
A continuación, proporcionamos ejemplos adicionales de análisis de microbioma de mosquitos en muestras de intestino medio y ovario obtenidas mediante este procedimiento sistemático. Observamos una clara diferencia en la microbiota entre los tejidos (Figura 5), con taxones bacterianos compartidos y específicos de órganos. Como era de esperar, se detectó la presencia de Wolbachia en ambos órganos, con mayor abundancia relativa (basada en lecturas cortas no ensambladas) y cobertura media de MAG en los ovarios de los mosquitos en comparación con los intestinos medios, lo que concuerda con la observación de que este endosimbionte se transmite a través de los ovarios y posteriormente se propaga a los tejidos somáticos. Aunque este estudio se limitó a muestras de Nueva Caledonia, este protocolo puede facilitar la investigación de la variabilidad genómica de Wolbachia a escala mundial, así como su papel en varios fenotipos, incluida la regulación de la densidad de sí misma y la protección viral. Además, este trabajo ejemplifica cómo el procedimiento de disección presentado aquí permite examinar la diversidad taxonómica y las capacidades funcionales potenciales de los simbiontes mosquitos dentro de muestras del intestino medio.
Obtuvimos dos genomas preliminares de enterobacterias que solo estaban presentes en dos muestras del intestino medio, lo que confirma la falta de contaminación entre órganos para estos dos especímenes. En cuanto a las espiroquetas, detectadas tanto en el intestino medio como en los ovarios del individuo 3, Juma y colaboradores, en 2020 observaron la presencia de estas bacterias en la superficie de las balsas de huevos. Los autores sugirieron que las comunidades bacterianas encontradas en las balsas de huevos podrían ser principalmente heredadas por la madre de los ovarios, dado que las balsas de huevos se mantuvieron en agua desionizada y libre de bacterias. Sin embargo, no pudieron descartar la posibilidad de que la colonización bacteriana ocurriera justo después de la oviposición y recomendaron más estudios sobre el microbioma ovárico16.
Aunque inicialmente se diseñó para especímenes del complejo de especies Culex pipiens , prevemos la aplicabilidad de este protocolo a una comunidad más amplia de entomólogos médicos que estudian otros vectores como Anopheles o Aedes. Al operar a nivel de órganos individuales, este método puede permitir comparaciones genómicas tanto intra como interindividuales, ofreciendo información sobre la variabilidad genómica de los simbiontes a una escala fina, con el potencial de avanzar en las estrategias de control de vectores. Este método de disección y aislamiento de las glándulas salivales, los ovarios y el intestino medio, evitando la contaminación cruzada microbiana, también podría ser un protocolo útil para los estudios de dinámica de infecciones virales dentro de estos tres órganos.
Divulgaciones
Los autores declaran que no tienen conflictos de intereses.
Agradecimientos
Agradecemos a Gilbert Legoff por enseñarle a Jordan Tutagata cómo diseccionar las glándulas salivales de los mosquitos y a Giuliano Mucci por ayudar con las fotos de los órganos de los mosquitos. Agradecemos a Anna-Bella Failloux y Nonito Pages por su útil discusión sobre el protocolo. Este trabajo contó con el apoyo de la subvención inicial RosaLind del ERC "948135" a JR. Agradecemos a la plataforma Vectopole (IRD, Montpellier) por brindar apoyo técnico y por la cría y mantenimiento de las poblaciones de mosquitos.
Materiales
| Name | Company | Catalog Number | Comments |
| Alcohol 96° | Fisher scientific | 10332562 | |
| Binocular magnifier | |||
| Bleach | RAJA | 145517 | 150 tablets of 1.5 g |
| DNA prep kit | Illumina | Provided by MGX sequencing platform | Previously known as Nextera DNA Flex |
| DNA-RNA Shield (50 mL) | Zymo research | ZR1100-50 | Preservation buffer |
| DNeasy Blood and Tissue Kit | Qiagen | 69504 | DNA extraction kit |
| Filter tips 20 µL | Starlab | S1120-3810 | |
| Filter tips 200 µL | Starlab | S1120-8710 | |
| Filter tips 1000 µL | Starlab | S1122-1730-C | |
| Forceps | FST (Fine Science Tools) | 11252-20 | Dumont Forceps #5 |
| Library quantification kit | Roche | Provided by MGX sequencing platform | KAPA Library Quanitification Kits |
| Micropipettes 2-20 µL | Eppendorf | 6.291704 | |
| Micropipettes 20-200 µL | Eppendorf | 6.291703 | |
| Micropipettes 100-1000 µL | Eppendorf | 7.648488 | |
| Microscope slides | Epredia | J1800BMNZ | dimension : 75 mm x 50 mm |
| Needles | Terumo | AN*2719R1 | |
| NGS kit | Agilent | Provided by MGX sequencing platform | Fragment Analyzer Systems HS Genomic DNA 50kb Kit |
| NovaSeq 6000 | Illumina | Provided by MGX sequencing platform | Sequencer |
| PBS Phosphate Buffered Saline (Sterile) | Fisher scientific | 10212990 | |
| Permanent black marker | |||
| Sterile Eppendorf | Dutscher | 33871 | 1.5 mL |
| Support for needles | FST (Fine Science Tools) | 26016-12 | Moria MC1 Pin Holder 12 cm |
Referencias
- Achee, N. L., et al. Alternative strategies for mosquito-borne arbovirus control. PLOS Negl Trop Dis. 13 (1), e0006822 (2019).
- Hoffmann, A. A., et al. Successful establishment of Wolbachia in Aedes populations to suppress dengue transmission. Nature. 476 (7361), 454-457 (2011).
- Porter, J., Sullivan, W. The cellular lives of Wolbachia. Nat Rev Microbiol. 21 (11), 750-766 (2023).
- Coon, K. L., Brown, M. R., Strand, M. R. Mosquitoes host communities of bacteria that are essential for development but vary greatly between local habitats. Mol Ecol. 25 (22), 5806-5826 (2016).
- Didion, E. M., et al. Microbiome reduction prevents lipid accumulation during early diapause in the northern house mosquito, Culex pipiens pipiens. J Insect Physiol. 134, 104295 (2021).
- Gabrieli, P., et al. Mosquito trilogy: Microbiota, immunity and pathogens, and their implications for the control of disease transmission. Front Microbiol. 12, 630438 (2021).
- Garrigós, M., Garrido, M., Panisse, G., Veiga, J., Martínez-de La Puente, J. Interactions between West Nile Virus and the microbiota of Culex pipiens vectors: A literature review. Pathogens. 12 (11), 1287 (2023).
- Zheng, R., et al. Holobiont perspectives on tripartite interactions among microbiota, mosquitoes, and pathogens. ISME J. 17 (8), 1143-1152 (2023).
- Flores, G. A. M., et al. Wolbachia dominance influences the Culex quinquefasciatus microbiota. Sci Rep. 13 (1), 18980 (2023).
- Hegde, S., et al. Interkingdom interactions shape the fungal microbiome of mosquitoes. Anim Microbiome. 6 (1), 11 (2024).
- Suo, P., et al. Seasonal variation of midgut bacterial diversity in Culex quinquefasciatus populations in Haikou city, Hainan province, China. Biology. 11 (8), 1166 (2022).
- Gruber-Vodicka, H. R., Seah, B. K. B., Pruesse, E. phyloFlash: Rapid small-subunit rRNA profiling and targeted assembly from metagenomes. mSystems. 5 (5), e00920-e00920 (2020).
- Graña-Miraglia, L., et al. Spirochetes isolated from arthropods constitute a novel genus Entomospira genus novum within the order Spirochaetales. Sci Rep. 10 (1), 17053 (2020).
- Reveillaud, J., et al. The Wolbachia mobilome in Culex pipiens includes a putative plasmid. Nat Commun. 10 (1), 1051 (2019).
- Ghousein, A., et al. pWCP is a widely distributed and highly conserved Wolbachia plasmid in Culex pipiens and Culex quinquefasciatus mosquitoes worldwide. ISME Commun. 3 (1), 40 (2023).
- Juma, E. O., Kim, C. -. H., Dunlap, C., Allan, B. F., Stone, C. M. Culex pipiens and Culex restuans egg rafts harbor diverse bacterial communities compared to their midgut tissues. Parasit Vectors. 13 (1), 532 (2020).
- Lee, M. D. GToTree: a user-friendly workflow for phylogenomics. Bioinformatics. 35 (20), 4162-4164 (2019).
- Parks, D. H., et al. A complete domain-to-species taxonomy for bacteria and Archaea. Nat Biotechnology. 38 (9), 1079-1086 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados