Method Article
Präparierung von Mücken-Eierstöcken, Mitteldarm- und Speicheldrüsen für Mikrobiomanalysen auf Organebene
In diesem Artikel
Zusammenfassung
Mikrobielle Gemeinschaften in Mücken sind vielversprechend für biologische Vektorkontrollstrategien. Die meisten Symbionten sind nicht kultivierbar und erfordern metagenomische Analysen. Wir beschreiben eine Methode zur Sezierung weiblicher Mücken und zur Trennung von Eierstöcken, Mitteldarm- und Speicheldrüsen, um Kreuzkontaminationen zu verhindern, Mikrobiomstudien auf Organebene zu erleichtern und das Verständnis der Rolle von Mikroorganismen in der Mückenbiologie zu verbessern.
Zusammenfassung
Die weltweite Belastung durch durch Stechmücken übertragene Krankheiten wie Malaria, Dengue-Fieber, West-Nil-Fieber, Zika, Usutu und Gelbfieber nimmt weiter zu und stellt eine erhebliche Bedrohung für die öffentliche Gesundheit dar. Mit der Zunahme von Insektizidresistenzen und dem Fehlen wirksamer Impfstoffe entstehen neue Strategien, die sich auf die Mikrobiota der Mücke konzentrieren. Dennoch bleibt der Großteil der Symbionten resistent gegen die Kultivierung. Die Charakterisierung der Diversität und Funktion bakterieller Genome in Mückenproben beruht daher auf Metagenomik und anschließenden Assemblierungs- und Binning-Strategien. Die Erfassung und Analyse von Metagenom-assemblierten Genomen (MAGs) aus getrennten Organen kann insbesondere wichtige Informationen über die spezifische Rolle von Mücken-assoziierten Mikroben in den Eierstöcken (den Fortpflanzungsorganen), dem Mitteldarm (Schlüssel für die Nahrungsverdauung und Immunität) oder den Speicheldrüsen (essentiell für die Übertragung von vektorübertragenen Krankheiten, da Krankheitserreger sie besiedeln müssen, um in den Speichel zu gelangen und den Blutkreislauf während einer Blutmahlzeit zu erreichen) liefern. Diese neu rekonstruierten Genome können dann den Weg für die Entwicklung neuartiger Vektor-Biokontrollstrategien ebnen. Zu diesem Zweck ist es erforderlich, Mückenorgane zu isolieren und gleichzeitig eine Kreuzkontamination zwischen ihnen oder mit Mikroorganismen in anderen Mückenorganen zu vermeiden. Hier beschreiben wir ein optimiertes und kontaminationsfreies Dissektionsprotokoll zur Untersuchung des Mückenmikrobioms auf Organebene.
Einleitung
Stechmücken verbreiten eine Vielzahl von Krankheitserregern und stellen eine ernsthafte Bedrohung für die öffentliche Gesundheit dar. Aufgrund einer erhöhten Prävalenz von Insektizidresistenzen in Mückenpopulationen und in Ermangelung wirksamer Impfstoffe gegen diese Krankheitserreger entstehen neue biologische Bekämpfungsmethoden, die sich auf das Mückenmikrobiom konzentrieren. Insbesondere sticht das intrazelluläre Bakterium Wolbachia hervor, das die Übertragung von Krankheitserregern stören und die Wirtsvermehrung manipulieren kann 1,2,3. Darüber hinaus sind andere Mückensymbionten von zentraler Bedeutung für das Überleben, die Entwicklung oder das Immunsystem ihres Wirts sowie für die Infektion und Übertragung von Krankheitserregern und vielversprechend für ihre Nutzung zur Bekämpfung vektorübertragener Krankheiten 4,5,6,7,8.
Mikroorganismen, die mit Mücken assoziiert sind, erstrecken sich über alle Bereiche des mikrobiellen Lebens (einschließlich Bakterien, Eukaryoten und Pilze), die eng mit ihrem Wirt, aber auch miteinander in den verschiedenen Körperkompartimenten interagieren 9,10. Daher kann ein besseres Verständnis der Mikrobiota, ihrer potenziellen saisonalen Variation11 und der Mechanismen, durch die ihre Mitglieder direkt in verschiedenen Mückengeweben interagieren, dazu beitragen, neue gezielte biologische Bekämpfungsmethoden zu entwickeln oder bestehende zu verbessern. Die Mehrzahl der Symbionten bleibt jedoch resistent gegen die Kultivierung, so dass ihre Charakterisierung unmöglich ist.
Das Aufkommen von Sequenzierungsmethoden der zweiten und dritten Generation in Verbindung mit modernsten Assemblierungs- und Binning-Ansätzen hat die Rekonstruktion mikrobieller Genome und den Zugang zur Vielfalt und zum funktionellen Potenzial nicht kultivierbarer Mikroben ermöglicht. Hier stellen wir eine Methode vor, mit der die Eierstöcke, der Mitteldarm und die Speicheldrüsen weiblicher Mücken dissektioniert und gleichzeitig eine Kreuzkontamination verhindert werden kann. Diesem Protokoll kann eine genomische DNA-Extraktion und eine anschließende Metabarcodierung oder metagenomische Sequenzierung folgen, um die Vielfalt und Funktion der Mückenmikrobiota auf Organebene zu untersuchen. Wir stellen ein Beispiel für die Stechmückendissektion und Mikrobiomdaten für Culex spp.-Proben zur Verfügung, obwohl dieses Protokoll auf Vektoren anderer Gattungen wie Anopheles oder Aedes ausgeweitet werden kann.
Protokoll
HINWEIS: Abbildung 1 zeigt ein Schema der Methode, das die verschiedenen Schritte des Protokolls zusammenfasst.
1. Materialvorbereitung
- Bereiten Sie vor Beginn des Experiments eine Bleichlösung mit 0,5 % aktivem Chlor (empfohlen von der Weltgesundheitsorganisation) vor, um das Gerät und die Werkbank zu desinfizieren.
- Lösen Sie in 900 ml Wasser drei lösliche Tabletten auf, die jeweils 1,5 g aktives Chlor enthalten. Warten Sie 15 Minuten, bis sich die Tabletten vollständig aufgelöst haben, was zu einer 0,5%igen Bleichlösung führt.
- Geben Sie die 0,5%ige Bleichlösung in eine Sprühflasche. Sprühen Sie dann die Lösung auf alle Seziergeräte, einschließlich mikroskopisch kleiner Objektträger und Werkbank.
HINWEIS: Das Gerät und die Werkbank sollten vor dem Gebrauch ~10 Minuten trocknen gelassen werden.
- Um die Mücken vor jeder Dissektion zu spülen und eine Kontamination der Proben mit externen Bakterien zu verhindern, bereiten Sie vier neu sterilisierte 1,5-ml-Röhrchen vor: zwei mit 96 % Ethanol und zwei weitere mit 1x PBS-Puffer (phosphatgepufferte Kochsalzlösung).
- Bereiten Sie die Röhrchen für die Lagerung der Proben nach der Dissektion jeder Mücke vor: vier sterile 1,5-ml-Röhrchen mit jeweils 100 μl Konservierungspuffer.
HINWEIS: Bereiten Sie einen Schlauch für die Speicheldrüsen, einen für die Eierstöcke, einen für den Mitteldarm und einen für den Mückenkadaver vor.
2. Sammlung von Mücken
- Sammeln und halten Sie erwachsene weibliche Individuen am Leben (aus dem Feld oder Labor, 4-7 Tage) bis zur Oberflächensterilisation.
3. Oberflächensterilisation von Mücken
HINWEIS: Reinigen Sie Pinzetten und Nadeln und verwenden Sie neue, sterilisierte Schläuche zwischen jedem Organ und jeder Person. Wenn kein Bleichmittel verfügbar ist, stellen Sie sicher, dass die Präparierzange und die Nadeln zwischen der Dissektion jedes Individuums und jedes Organs gründlich flammsterilisiert werden, um eine Kreuzkontamination von biologischem Material zu verhindern. Präparierinstrumente sollten vor dem nächsten Gebrauch trocken und ohne Bleichmittel-/Ethanolrückstände sein.
- Um zu vermeiden, dass Weibchen ihren Darminhalt entleeren, wenn sie Alkohol ausgesetzt sind, betäuben Sie sie auf Eis oder in einem Gefrierschrank bei -20 °C (4 Minuten lang, damit das Gewebe und die Organe nicht einfrieren).
- Übertragen Sie einzelne Mücken in ein neues sterilisiertes 1,5-ml-Röhrchen mit 1 ml 96%igem Ethanol. 30 s lang vorsichtig (manuell) vortexen (die Mücke im Röhrchen gut schütteln, um die Schuppen und mögliche bakterielle Verunreinigungen auf der Oberfläche der Insekten zu entfernen). Spülen Sie zweimal mit Tuben, die Ethanol enthalten.
- Übertragen Sie die Mücke in ein neues sterilisiertes 1,5-ml-Röhrchen mit 1 mL steriler 1x PBS-Lösung und wirbeln Sie sie vorsichtig (manuell) für 30 s vor (um DNA-Ausfällung mit Ethanol zu vermeiden). Spülen Sie zweimal mit Röhrchen, die steriles 1x PBS enthalten.
4. Sezieren von Mücken
- Geben Sie einige kleine Tropfen steriles 1x PBS auf den Objektträger, um das Platzieren der Mücke und das Spülen der präparierten Organe zu erleichtern.
HINWEIS: Die Tropfen sterilen PBS sollten groß genug sein, um die Mückenorgane aufzunehmen. - Nehmen Sie die gereinigte Mücke und geben Sie sie auf einen Tropfen steriles 1x PBS auf den Objektträger.
- Präparieren Sie die Speicheldrüsen mit einer sterilisierten Pinzette und Nadel wie unten beschrieben.
- Halten Sie den Brustkorb der Mücke mit einer Pinzette fest, damit sie sich nicht bewegt. Platzieren Sie dann eine Nadel am Hals unterhalb des Kopfes und ziehen Sie vorsichtig am Kopf der Mücke, um ihn vom Brustkorb zu trennen.
HINWEIS: Die Speicheldrüsen befinden sich im oberen Brustkorb und sind über den Speichelgang mit dem Kopf verbunden. Während der Dissektion ist es wichtig, diesen Kanal nicht zu brechen, da die Speicheldrüsen nicht mit dem Kopf kommen, wenn Sie ihn aus dem Thorax ziehen. Aus diesem Grund ist es wichtig, eine frische Mücke zu haben, keine gefrorene. - Wenn sich der Kopf vom Thorax trennt, beobachte, wie die beiden Speicheldrüsen herauskommen, die jeweils aus drei Lappen bestehen. Schneiden Sie die Speicheldrüsen auf Höhe des Speichelgangs, entnehmen Sie sie mit einer Nadel und geben Sie sie in einen neuen Tropfen steriles 1x PBS, um ihn zu spülen und Kontaminationen zwischen den Organen zu verhindern.
- Entnehmen Sie die Speicheldrüsen mit einer Nadel und geben Sie sie in ein neues sterilisiertes Röhrchen mit 100 μl Konservierungspuffer.
- Halten Sie den Brustkorb der Mücke mit einer Pinzette fest, damit sie sich nicht bewegt. Platzieren Sie dann eine Nadel am Hals unterhalb des Kopfes und ziehen Sie vorsichtig am Kopf der Mücke, um ihn vom Brustkorb zu trennen.
- Präparieren Sie die Eierstöcke mit einer sterilisierten Pinzette und Nadel wie folgt.
- Lege die Mücke auf den Rücken. Halten Sie die Mücke mit einer Zange am Brustkorb fest, um eine Bewegung während der Dissektion zu verhindern, und stechen Sie mit einer Nadel in das dritte Bauchsegment (von unten beginnend).
HINWEIS: Entfernen Sie optional die Beine und Flügel der Mücke, um das Sezieren zu erleichtern. - Während Sie das dritte Bauchsegment durchstechen, ziehen Sie den Bauch nach unten, um ihn zu öffnen und die inneren Organe freizulegen. Entfernen Sie alle verbleibenden Teile des Mücken-Exoskeletts, um die Freisetzung der Organe zu erleichtern.
HINWEIS: Organe im Bauchraum wie der Darm, die Malpighischen Tubuli und die Eierstöcke sind sichtbar. - Entnehmen Sie beide Eierstöcke mit einer sterilisierten Pinzette und legen Sie sie zum Spülen in einen neuen Tropfen steriles 1x PBS auf den Objektträger. Nehmen Sie die Eierstöcke und legen Sie sie in das 1,5-ml-Röhrchen mit dem 100 μl Konservierungspuffer.
- Lege die Mücke auf den Rücken. Halten Sie die Mücke mit einer Zange am Brustkorb fest, um eine Bewegung während der Dissektion zu verhindern, und stechen Sie mit einer Nadel in das dritte Bauchsegment (von unten beginnend).
- Jetzt, da der Bauch offen ist und die Organe freiliegen, entnehmen Sie den Mitteldarm mit einer neuen sterilisierten Pinzette und/oder Nadel.
- Trennen Sie den Mitteldarm vom Vorderdarm und Hinterdarm; Entfernen Sie dann die Malpighischen Tubuli aus dem Mitteldarm. Fassen Sie den Mitteldarm mit einer sterilen Nadel und legen Sie ihn auf einen neuen sterilen 1x PBS-Tropfen, um ihn zu spülen und Kontaminationen zwischen den Organen zu verhindern.
- Umfüllen mit einem neuen sterilen 1x PBS Tropfen. Öffnen Sie den Mitteldarm mit einer Pinzette, um Bakterien zu befreien und anzureichern, wie in der Zusatzdatei 1 beschrieben. Fassen Sie den Mitteldarm mit einer Pinzette und pipettieren Sie alle PBS-haltigen Bakterien, um das gesamte Material (Flüssigkeit und Gewebe) in einem neuen sterilisierten 1,5-ml-Röhrchen mit 100 μl Konservierungspuffer aufzubewahren.
- Geben Sie erneut 5 μl 1x PBS auf die zuvor verwendete Stelle im Objektträger und pipettieren Sie PBS direkt in dasselbe 1,5-ml-Röhrchen zurück, um Bakterien zu entfernen, die möglicherweise am Objektträger haften geblieben sind.
- Verwenden Sie eine sterile Pinzette, um den Schlachtkörper (verbleibenden Körper) zu greifen, und lagern Sie ihn in einem neuen sterilisierten 1,5-ml-Röhrchen mit 100 μl Konservierungspuffer.
- Lagerung des Materials bis zur Weiterverarbeitung
- Beschriften Sie die Röhrchen mit "Spezies, Organ, Datum, Name des Forschers".
- Präparierte Eierstöcke, Mitteldarm, Speicheldrüsen und Schlachtkörper bis zur Verarbeitung bei -20 °C lagern.
- Behalten Sie den Überblick in einer Tabelle mit allen zugehörigen Metadaten für die Proben, einschließlich Spezies (oder nur Gattungsebene), Entnahmedatum, Land, Provinz, Breitengrad, Längengrad, Name der Person, die die Sektion durchgeführt hat, trächtiges Weibchen oder nicht, Blutmehl oder nicht.
Ergebnisse
x'Mosquito Dissektionen
Gemäß dem Protokoll sammelten und isolierten wir die Eierstöcke, den Mitteldarm, die Speicheldrüsen und den Schlachtkörper von zwei Culex pipiens molestus-Exemplaren (einschließlich eines trächtigen Weibchens) aus einer Laborkolonie. Wir bestätigten saubere Dissektionen nach der Beobachtung ganzer (d.h. ungebrochener) und gut isolierter Organe ohne verbleibende Trümmer unter dem Fernglas. Der gesamte Körper, der Kopf und der Thorax, die präparierten Speicheldrüsen, Eierstöcke und der Mitteldarm eines Exemplars von Culex pipiens molestus sind in Abbildung 2 dargestellt. Erwartungsgemäß waren die Mitteldarm- und Speicheldrüsen um eine Größenordnung kleiner als die Eierstöcke der Mücke. Die Eier eines trächtigen Culex-Exemplars sind zusammen mit seinen Mitteldarm- und Malpighischen Tubuli in Abbildung 3 dargestellt. Bemerkenswert ist, dass Dissektionen anfälliger für Fehler sind, wenn das Material nicht frisch ist und das Gewebe wahrscheinlich bricht (Abbildung 4B, C). Wir empfehlen daher, das Material nach Möglichkeit direkt nach der Entnahme (innerhalb von weniger als 12 Stunden) zu präparieren, da das Gewebe noch elastisch ist (Abbildung 4A). In ähnlicher Weise kann die Dissektion von gefrorenem Material durchgeführt werden, aber es besteht ein viel höheres Risiko für Versagen und Kreuzkontamination zwischen Organen aufgrund von zerbrechlichem Gewebe (Abbildung 4).
Mikrobiom-Daten
Darüber hinaus sammelten und trennten wir die Mitteldärme und Eierstöcke von drei einzelnen Culex quinquefasciatus-Exemplaren aus Noumea, Neukaledonien, nach dem gleichen Verfahren. Wir extrahierten DNA aus jedem Organ, bereiteten Proben für die Sequenzierung des gesamten Genoms vor und führten Mikrobiomdatenanalysen durch, wie in der ergänzenden Datei 1 beschrieben. Eine bakterielle taxonomische Diversitätsanalyse an nicht assemblierten, qualitätsgefilterten Short Reads mit PhyloFlash12 zeigte deutlich dominante Taxa im Mitteldarm im Vergleich zu den Eierstöcken (Abbildung 5A). Bemerkenswert ist, dass die Bakteriengemeinschaften der Eierstöcke von Wolbachia dominiert wurden, mit dem zusätzlichen Vorhandensein eines Spirochaetaceae-Phylotyps in den Eierstöcken von Individuum 3, während die Gemeinschaften des Mitteldarms eine größere Diversität aufwiesen, einschließlich Gammaproteobakterien, Spirochaetaceae und Firmicutes. Aus den gleichen Sequenzierungsdaten rekonstruierten wir vier Metagenom-Assemblierte Genome (MAGs) mit einer Vervollständigung > 80% und einer Redundanz < 5%, die den Klassen Spirochätien, Alpha- und Gammaproteobakterien angehören (Tabelle 1).
Wie erwartet, deckten die hier rekonstruierten MAGs aufgrund der spezifischen Mängel der Genomrekonstruktion mit metagenomischen Short-Reads nicht die gesamte taxonomische Vielfalt ab, die von den PhyloFlash-Ergebnissen vorhergesagt wurde. Der Wolbachia zugeordnete MAG (Ref. MAG 3 in Tabelle 1) wurde in allen Eierstöcken und zwei der Mitteldärme nachgewiesen und hatte eine höhere Abdeckung in den Eierstöcken (Abbildung 5B). Wir rekonstruierten auch zwei MAGs, die zur Familie der Enterobacteriaceae gehören, einschließlich der Gattung Pantoea (Ref. MAG 1 und 2 in Tabelle 1), in den Mitteldärmen, die von Culex-Individuen 2 und 3 gewonnen wurden und in den entsprechenden Eierstöcken nicht nachgewiesen wurden (Abbildung 5B). Schließlich rekonstruierten wir auch ein bakterielles Genom von Spirochaetaceae, Ref. MAG 4 (Tabelle 1), das der Gattung BR149 zugeordnet wurde und von Graña-Miraglia und Kollegen13 erfolgreich aus dem Mitteldarm von Culex pipiens isoliert wurde. Interessanterweise wurde dieser MAG sowohl im Mitteldarm von Individuum 1 und 3 als auch in den Eierstöcken von Individuum 3 nachgewiesen (Abbildung 5B).
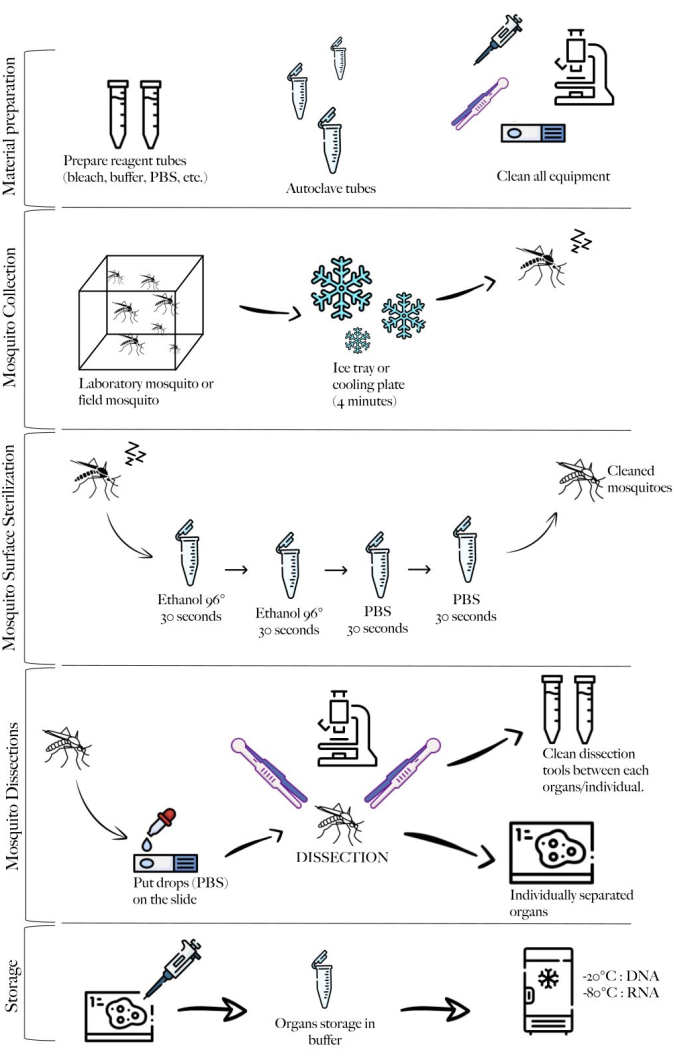
Abbildung 1: Schematische Darstellung der Methode, die die verschiedenen Schritte zusammenfasst. Materialvorbereitung, Mückensammlung, Mückenreinigung, Mückensektion und Lagerung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
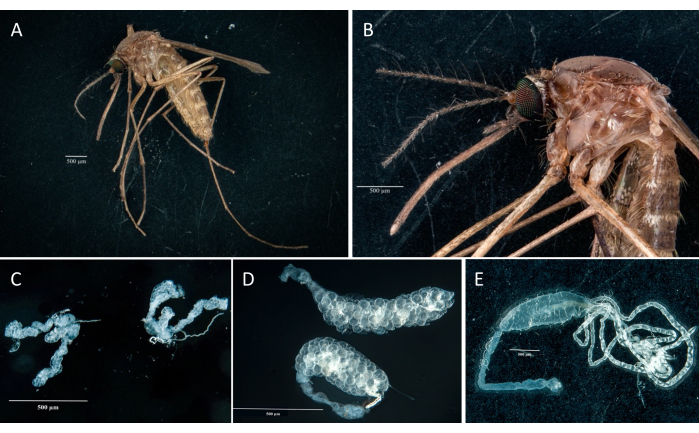
Abbildung 2: Culex pipiens molestus Weibchen. (A) Ganzer Körper. (B) Kopf und Thorax. (C) Präparierte Speicheldrüsen. (D) Präparierte Eierstöcke. (E) Präparierter Darm mit Malpighi-Tubuli. Maßstabsbalken = 500 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
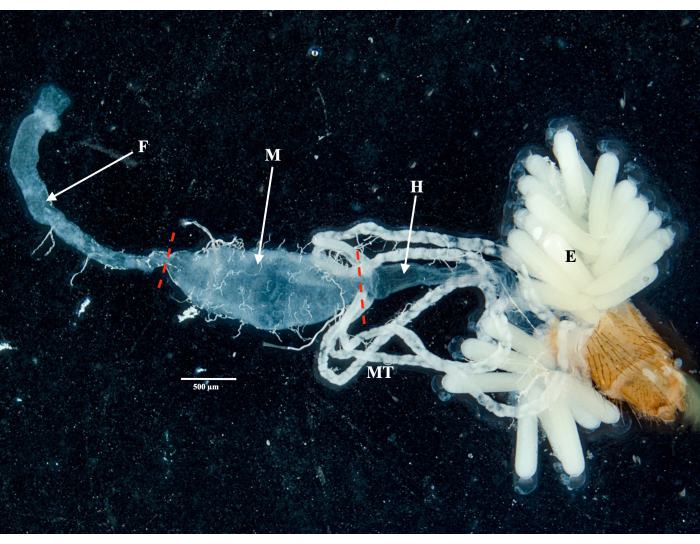
Abbildung 3: Präpariertes Abdomen eines trächtigen Culex pipiens molestus-Weibchens . Gezeigt sind der Vorderdarm, der Mitteldarm, der Hinterdarm, die Malpighischen Tubuli und die Eier des Exemplars. Rote Striche zeigen an, wo geschnitten werden muss, um den Mitteldarm vom Vorderdarm und Hinterdarm zu trennen. Maßstabsbalken = 500 μm. Abkürzungen: F = Vorderdarm; M = Mitteldarm; H = Hinterdarm; E = Eier; MT = Malpighische Tubuli. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
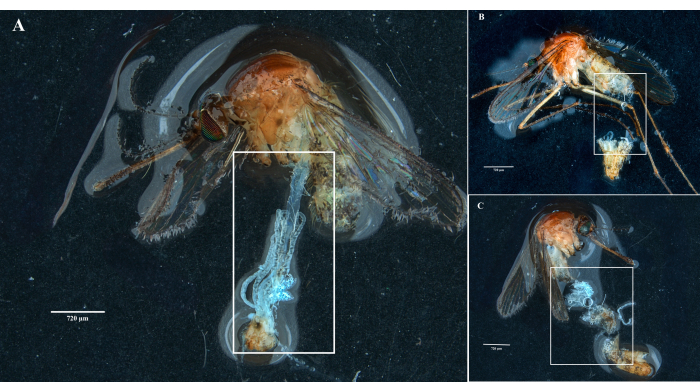
Abbildung 4: Sektion von frischen und gefrorenen Culex-Mückenproben. Weiße Rechtecke zeigen (A) Mückengewebe mit intakten Organen aus einer frisch präparierten Probe und (B,C) gebrochenes biologisches Material aus Proben, die vor der Sektion eingefroren wurden. Maßstabsbalken = 720 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
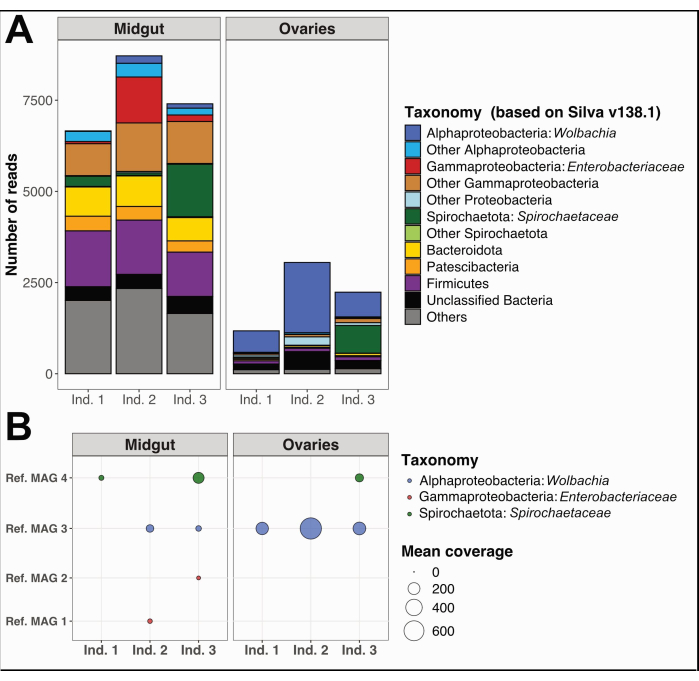
Abbildung 5: Beispiel für eine Mikrobiomanalyse an drei Culex quinquefasciatus-Individuen . (A) Visualisierung der bakteriellen Diversität, die durch die Extraktion von SSU-rRNA-Reads mit PhyloFlash12 im Mitteldarm und in den Eierstöcken der drei Proben geschätzt wurde. (B) Mittlere Abdeckung der rekonstruierten MAGs über die Proben. Abkürzungen: MAG = Metagenome-Assembled Genome; Ind = Individuell. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| ILLUSTRIERTE | Referenz MAG 4 | Referenz MAG 2 | Referenz MAG 1 | Referenz MAG 3 |
| Länge (bp) | 1,287,790 | 4,910,866 | 4,751,276 | 1,298,266 |
| Anzahl der Contigs | 9 | 162 | 149 | 123 |
| GC % | 34.05 | 55.45 | 54.05 | 34.15 |
| Fertigstellung (%) | 84.5 | 97.18 | 98.59 | 91.55 |
| Redundanz (%) | 0 | 2.82 | 4.22 | 0 |
| Domäne | Bakterien | Bakterien | Bakterien | Bakterien |
| Stamm | Spirochäte | Proteobakterien | Proteobakterien | Proteobakterien |
| Klasse | Spirochätien | Gammaproteobakterien | Gammaproteobakterien | Alphaproteobakterien |
| Bestellung | WRBN01 | Enterobacterales | Enterobacterales | Rickettsiales |
| Familie | WRBN01 | Enterobacteriaceae | Enterobacteriaceae | Anaplasmataceae |
| Gattung | BR149 | Pantoea | - | Wolbachia |
Tabelle 1: Rekonstruierte MAGs von den drei Culex quinquefasciatus-Individuen . Genomgröße, Anzahl der Contigs, Anteil der GC, Schätzungen der Fertigstellung und Redundanz basierend auf der in Anvi'o17 verfügbaren Einzelkopie-Kerngensammlung und der mit GTDB18 erhaltenen Taxonomie.
Ergänzende Datei 1: Detailliertes Beispielverfahren für die Analyse von Mikrobiomdaten, beginnend mit der Probenentnahme, der DNA-Extraktion und der Sequenzierung des gesamten Genoms, gefolgt von dem bioinformatischen Workflow für die Genomrekonstruktion und die Schätzung des prokaryotischen und eukaryotischen Leseanteils Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Wir empfehlen, besonders auf die Abfolge der Organdissektion zu achten, beginnend mit den Speicheldrüsen. Tatsächlich beobachteten wir, dass sie leichter aus dem Thorax von Culex-Exemplaren extrahiert werden konnten, wenn die Integrität der Mücke erhalten blieb. Eine Schädigung des Bauches oder des Brustkorbs kann den Druck im Körper der Mücke verringern und den Eingriff erschweren. Es ist aber auch möglich, zwischen Thorax und Bauch zu schneiden und dann die Speicheldrüsen aus Kopf und Thorax herauszuziehen (A. B. Failloux, persönliche Mitteilung). Darüber hinaus kann es eine Herausforderung sein, kompetente Präparierfähigkeiten zu erwerben, daher empfehlen wir, vor dem Experiment an einer ausreichenden Anzahl von Proben zu üben.
Die Isolierung von Mückenorganen bei gleichzeitiger systematischer Vermeidung von Kreuzkontaminationen ist entscheidend für eine Vielzahl von nachgelagerten Mikrobiota-Analysen von Mücken. Eine genomaufgelöste metagenomische Studie nach der Dissektion einzelner Eierstöcke aus Culex pipiens-Proben in Südfrankreich ermöglichte die Entdeckung des ersten Plasmids von Wolbachia in Culex pipiens (pWCP14). Mit einem ähnlichen Ansatz untersuchten wir die Verteilung und Häufigkeit von pWCP in Culex pipiens- und Culex quinquefasciatus-Proben sowohl aus kontinentalen als auch aus Inselgebieten weltweit, unter verschiedenen Umwelt- und Laborbedingungen. Insgesamt zeigten die Daten ein bemerkenswert konserviertes Wolbachia-Plasmidelement in Culex-Mücken, was auf eine entscheidende Rolle dieses mobilen Elements in der Endosymbiontenbiologie hindeutet15, was eine weitere Analyse rechtfertigt.
Hier stellen wir weitere Beispiele für Mücken-Mikrobiom-Analysen an Mitteldarm- und Eierstockproben vor, die mit diesem systematischen Verfahren gewonnen wurden. Wir beobachteten einen deutlichen Unterschied in der Mikrobiota zwischen den Geweben (Abbildung 5), wobei sowohl gemeinsame als auch organspezifische bakterielle Taxa vorhanden waren. Wie erwartet wurde das Vorhandensein von Wolbachia in beiden Organen nachgewiesen, mit einer höheren relativen Abundanz (basierend auf unassemblierten Short Reads) und einer mittleren Abdeckung von MAGs in den Mücken-Ovarien im Vergleich zu den Mitteldärmen, was mit der Beobachtung übereinstimmt, dass dieser Endosymbiont über die Eierstöcke übertragen wird und sich anschließend auf das somatische Gewebe ausbreitet. Obwohl diese Studie auf Proben aus Neukaledonien beschränkt war, kann dieses Protokoll die Untersuchung der genomischen Variabilität von Wolbachia auf globaler Ebene sowie seiner Rolle bei verschiedenen Phänotypen, einschließlich der Dichteregulation von sich selbst und des Virusschutzes, erleichtern. Darüber hinaus zeigt diese Arbeit beispielhaft, wie das hier vorgestellte Präparierungsverfahren die Untersuchung der taxonomischen Diversität und der potentiellen funktionellen Fähigkeiten von Mückensymbionten in Mitteldarmproben ermöglicht.
Wir erhielten zwei enterobakterielle Entwurfsgenome, die nur in zwei Mitteldarmproben vorhanden waren, was die fehlende Kontamination zwischen den Organen für diese beiden Proben bestätigt. In Bezug auf Spirochäten, die sowohl im Mitteldarm als auch in den Eierstöcken von Individuum nachgewiesen wurden 3, beobachteten Juma und Kollegen im Jahr 2020 das Vorhandensein dieser Bakterien auf der Oberfläche von Eierflößen. Die Autoren schlugen vor, dass die Bakteriengemeinschaften, die in den Eierflößen gefunden wurden, in erster Linie mütterlich von den Eierstöcken vererbt werden könnten, da die Eierflöße in entionisiertem und bakterienfreiem Wasser gehalten wurden. Sie konnten jedoch nicht ausschließen, dass es direkt nach der Eiablage zu einer bakteriellen Besiedlung kommt und empfahlen weitere Untersuchungen des Mikrobioms der Eierstöcke16.
Obwohl dieses Protokoll ursprünglich für Exemplare des Culex pipiens-Artenkomplexes entwickelt wurde, sehen wir die Anwendbarkeit dieses Protokolls auf eine breitere Gemeinschaft von medizinischen Entomologen, die andere Vektoren wie Anopheles oder Aedes untersuchen. Durch die Arbeit auf der Ebene einzelner Organe kann diese Methode sowohl intra- als auch interindividuelle genomische Vergleiche ermöglichen und Einblicke in die genomische Variabilität von Symbionten auf einer feinen Skala bieten, mit dem Potenzial, Vektorkontrollstrategien voranzutreiben. Diese Methode der Dissektion und Isolierung der Speicheldrüsen, Eierstöcke und des Mitteldarms, die eine mikrobielle Kreuzkontamination verhindert, könnte auch ein nützliches Protokoll für Studien zur Dynamik von Virusinfektionen in diesen drei Organen sein.
Offenlegungen
Die Autoren erklären, dass sie keine Interessenkonflikte haben.
Danksagungen
Wir danken Gilbert Legoff dafür, dass er Jordan Tutagata beigebracht hat, wie man Speicheldrüsen von Mücken präpariert, und Giuliano Mucci für seine Hilfe mit Fotos der Mückenorgane. Wir danken Anna-Bella Failloux und Nonito Pages für die hilfreiche Diskussion über das Protokoll. Diese Arbeit wurde durch den ERC RosaLind Starting Grant "948135" für JR unterstützt. Wir danken der Vectopole-Plattform (IRD, Montpellier) für die technische Unterstützung sowie für die Aufzucht und Pflege der Mückenpopulationen.
Materialien
| Name | Company | Catalog Number | Comments |
| Alcohol 96° | Fisher scientific | 10332562 | |
| Binocular magnifier | |||
| Bleach | RAJA | 145517 | 150 tablets of 1.5 g |
| DNA prep kit | Illumina | Provided by MGX sequencing platform | Previously known as Nextera DNA Flex |
| DNA-RNA Shield (50 mL) | Zymo research | ZR1100-50 | Preservation buffer |
| DNeasy Blood and Tissue Kit | Qiagen | 69504 | DNA extraction kit |
| Filter tips 20 µL | Starlab | S1120-3810 | |
| Filter tips 200 µL | Starlab | S1120-8710 | |
| Filter tips 1000 µL | Starlab | S1122-1730-C | |
| Forceps | FST (Fine Science Tools) | 11252-20 | Dumont Forceps #5 |
| Library quantification kit | Roche | Provided by MGX sequencing platform | KAPA Library Quanitification Kits |
| Micropipettes 2-20 µL | Eppendorf | 6.291704 | |
| Micropipettes 20-200 µL | Eppendorf | 6.291703 | |
| Micropipettes 100-1000 µL | Eppendorf | 7.648488 | |
| Microscope slides | Epredia | J1800BMNZ | dimension : 75 mm x 50 mm |
| Needles | Terumo | AN*2719R1 | |
| NGS kit | Agilent | Provided by MGX sequencing platform | Fragment Analyzer Systems HS Genomic DNA 50kb Kit |
| NovaSeq 6000 | Illumina | Provided by MGX sequencing platform | Sequencer |
| PBS Phosphate Buffered Saline (Sterile) | Fisher scientific | 10212990 | |
| Permanent black marker | |||
| Sterile Eppendorf | Dutscher | 33871 | 1.5 mL |
| Support for needles | FST (Fine Science Tools) | 26016-12 | Moria MC1 Pin Holder 12 cm |
Referenzen
- Achee, N. L., et al. Alternative strategies for mosquito-borne arbovirus control. PLOS Negl Trop Dis. 13 (1), e0006822 (2019).
- Hoffmann, A. A., et al. Successful establishment of Wolbachia in Aedes populations to suppress dengue transmission. Nature. 476 (7361), 454-457 (2011).
- Porter, J., Sullivan, W. The cellular lives of Wolbachia. Nat Rev Microbiol. 21 (11), 750-766 (2023).
- Coon, K. L., Brown, M. R., Strand, M. R. Mosquitoes host communities of bacteria that are essential for development but vary greatly between local habitats. Mol Ecol. 25 (22), 5806-5826 (2016).
- Didion, E. M., et al. Microbiome reduction prevents lipid accumulation during early diapause in the northern house mosquito, Culex pipiens pipiens. J Insect Physiol. 134, 104295 (2021).
- Gabrieli, P., et al. Mosquito trilogy: Microbiota, immunity and pathogens, and their implications for the control of disease transmission. Front Microbiol. 12, 630438 (2021).
- Garrigós, M., Garrido, M., Panisse, G., Veiga, J., Martínez-de La Puente, J. Interactions between West Nile Virus and the microbiota of Culex pipiens vectors: A literature review. Pathogens. 12 (11), 1287 (2023).
- Zheng, R., et al. Holobiont perspectives on tripartite interactions among microbiota, mosquitoes, and pathogens. ISME J. 17 (8), 1143-1152 (2023).
- Flores, G. A. M., et al. Wolbachia dominance influences the Culex quinquefasciatus microbiota. Sci Rep. 13 (1), 18980 (2023).
- Hegde, S., et al. Interkingdom interactions shape the fungal microbiome of mosquitoes. Anim Microbiome. 6 (1), 11 (2024).
- Suo, P., et al. Seasonal variation of midgut bacterial diversity in Culex quinquefasciatus populations in Haikou city, Hainan province, China. Biology. 11 (8), 1166 (2022).
- Gruber-Vodicka, H. R., Seah, B. K. B., Pruesse, E. phyloFlash: Rapid small-subunit rRNA profiling and targeted assembly from metagenomes. mSystems. 5 (5), e00920-e00920 (2020).
- Graña-Miraglia, L., et al. Spirochetes isolated from arthropods constitute a novel genus Entomospira genus novum within the order Spirochaetales. Sci Rep. 10 (1), 17053 (2020).
- Reveillaud, J., et al. The Wolbachia mobilome in Culex pipiens includes a putative plasmid. Nat Commun. 10 (1), 1051 (2019).
- Ghousein, A., et al. pWCP is a widely distributed and highly conserved Wolbachia plasmid in Culex pipiens and Culex quinquefasciatus mosquitoes worldwide. ISME Commun. 3 (1), 40 (2023).
- Juma, E. O., Kim, C. -. H., Dunlap, C., Allan, B. F., Stone, C. M. Culex pipiens and Culex restuans egg rafts harbor diverse bacterial communities compared to their midgut tissues. Parasit Vectors. 13 (1), 532 (2020).
- Lee, M. D. GToTree: a user-friendly workflow for phylogenomics. Bioinformatics. 35 (20), 4162-4164 (2019).
- Parks, D. H., et al. A complete domain-to-species taxonomy for bacteria and Archaea. Nat Biotechnology. 38 (9), 1079-1086 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten