Method Article
Dissection des ovaires, de l’intestin moyen et des glandes salivaires de moustiques pour l’analyse du microbiome au niveau des organes
Dans cet article
Résumé
Les communautés microbiennes chez les moustiques sont très prometteuses pour les stratégies de biocontrôle vectoriel. La plupart des symbiotes sont incultivables et nécessitent des analyses métagénomiques. Nous décrivons une méthode pour disséquer les moustiques femelles et séparer les ovaires, l’intestin moyen et les glandes salivaires, en évitant la contamination croisée, en facilitant les études du microbiome au niveau des organes et en améliorant la compréhension des rôles des micro-organismes dans la biologie des moustiques.
Résumé
Le fardeau mondial des maladies transmises par les moustiques, notamment le paludisme, la dengue, le Nil occidental, le Zika, l’Usutu et la fièvre jaune, continue d’augmenter, ce qui constitue une menace importante pour la santé publique. Avec l’augmentation de la résistance aux insecticides et l’absence de vaccins efficaces, de nouvelles stratégies émergent qui se concentrent sur le microbiote du moustique. Néanmoins, la majorité des symbiotes restent résistants à la culture. La caractérisation de la diversité et de la fonction des génomes bactériens dans les spécimens de moustiques repose donc sur la métagénomique et les stratégies d’assemblage et de regroupement subséquentes. L’obtention et l’analyse de génomes assemblés par métagénome (MAG) à partir d’organes séparés peuvent notamment fournir des informations clés sur le rôle spécifique des microbes associés aux moustiques dans les ovaires (les organes reproducteurs), l’intestin moyen (clé pour la digestion des aliments et l’immunité) ou les glandes salivaires (essentielles pour la transmission des maladies à transmission vectorielle car les agents pathogènes doivent les coloniser pour pénétrer dans la salive et atteindre la circulation sanguine lors d’un repas sanguin). Ces génomes nouvellement reconstruits peuvent ensuite ouvrir la voie au développement de nouvelles stratégies de biocontrôle vectoriel. Pour ce faire, il est nécessaire d’isoler les organes des moustiques tout en évitant la contamination croisée entre eux ou avec des micro-organismes présents dans d’autres organes de moustiques. Nous décrivons ici un protocole de dissection optimisé et sans contamination pour étudier le microbiome des moustiques au niveau des organes.
Introduction
Les moustiques propagent un large éventail d’agents pathogènes causant des maladies et constituent une menace sérieuse pour la santé publique. En raison d’une prévalence accrue de la résistance aux insecticides parmi les populations de moustiques et en l’absence de vaccins efficaces contre ces agents pathogènes, de nouvelles méthodes de biocontrôle axées sur le microbiome des moustiques émergent. En particulier, la bactérie intracellulaire Wolbachia, qui peut interférer avec la transmission des agents pathogènes et manipuler la reproduction de l’hôte, se distingue 1,2,3. De plus, d’autres symbiotes de moustiques sont essentiels à la survie, au développement ou au système immunitaire de leur hôte, ainsi qu’à l’infection et à la transmission d’agents pathogènes, et sont très prometteurs pour leur exploitation dans la lutte contre les maladies à transmission vectorielle 4,5,6,7,8.
Les micro-organismes associés aux moustiques couvrent tous les domaines de la vie microbienne (y compris les bactéries, les eucaryotes et les champignons) qui interagissent intimement avec leur hôte mais aussi les uns avec les autres dans les différents compartiments du corps 9,10. Par conséquent, une meilleure compréhension du microbiote, de ses variations saisonnières potentielles11 et des mécanismes par lesquels ses membres interagissent directement dans des tissus distincts de moustiques peut aider à développer de nouvelles méthodes de biocontrôle ciblées ou à améliorer celles existantes. Cependant, la majorité des symbiotes restent résistants à la culture, rendant leur caractérisation impossible.
L’avènement des méthodes de séquençage de deuxième et troisième générations, ainsi que des approches d’assemblage et de binning de pointe, ont permis de reconstruire les génomes microbiens et d’accéder à la diversité et au potentiel fonctionnel des microbes non cultivables. Ici, nous présentons une méthode de dissection des ovaires, de l’intestin moyen et des glandes salivaires des moustiques femelles tout en prévenant la contamination croisée. Ce protocole peut être suivi d’une extraction d’ADN génomique et d’un métacodage à barres ou d’un séquençage métagénomique ultérieur pour explorer la diversité et la fonction du microbiote des moustiques au niveau des organes. Nous fournissons un exemple de dissection de moustiques et de données sur le microbiome pour les spécimens de Culex spp., bien que ce protocole puisse être étendu à des vecteurs d’autres genres tels que Anopheles ou Aedes.
Protocole
REMARQUE : La figure 1 montre un schéma de la méthode résumant les différentes étapes du protocole.
1. Préparation du matériel
- Avant de commencer l’expérience, préparez une solution d’eau de Javel avec 0,5% de chlore actif (recommandée par l’Organisation mondiale de la santé) pour désinfecter l’équipement et l’établi.
- Dans 900 mL d’eau, dissoudre trois comprimés solubles contenant chacun 1,5 g de chlore actif. Attendez 15 minutes pour que les pastilles se dissolvent complètement, ce qui donne une solution d’eau de Javel à 0,5 %.
- Placez la solution d’eau de Javel à 0,5 % dans un flacon pulvérisateur. Ensuite, vaporisez la solution sur tout l’équipement de dissection, y compris les lames microscopiques et l’établi.
REMARQUE : L’équipement et l’établi doivent être laissés à sécher pendant ~10 min avant utilisation.
- Pour rincer les moustiques avant chaque dissection afin d’éviter la contamination des échantillons par des bactéries externes, préparez quatre tubes de 1,5 mL nouvellement stérilisés : deux contenant de l’éthanol à 96 % et deux autres contenant 1 tampon PBS (solution saline tamponnée au phosphate).
- Préparez les tubes pour stocker les échantillons après la dissection de chaque moustique : quatre tubes stériles de 1,5 mL, contenant chacun 100 μL de tampon de conservation.
REMARQUE : Préparez un tube pour les glandes salivaires, un pour les ovaires, un pour l’intestin moyen et un pour la carcasse du moustique.
2. Collecte des moustiques
- Prélever et garder en vie les femelles adultes (sur le terrain ou en laboratoire, 4 à 7 jours) jusqu’à la stérilisation en surface.
3. Stérilisation de surface des moustiques
REMARQUE : Nettoyez les pinces et les aiguilles et utilisez des tubes neufs et stérilisés entre chaque organe et individu. Si vous n’avez pas accès à de l’eau de Javel, assurez-vous de bien stériliser à la flamme la pince et les aiguilles de dissection entre chaque individu et chaque organe pour éviter la contamination croisée du matériel biologique. Les instruments de dissection doivent être secs, sans aucun résidu d’eau de Javel ou d’éthanol, avant la prochaine utilisation.
- Pour éviter que les femelles ne vident leur contenu intestinal lorsqu’elles sont exposées à l’alcool, anesthésiez-les froidement sur de la glace ou dans un congélateur à -20 °C (pendant 4 min pour que les tissus et les organes ne gèlent pas).
- Transférez les moustiques individuels dans un nouveau tube stérilisé de 1,5 ml contenant 1 ml d’éthanol à 96 %. Agitez doucement (manuellement) pendant 30 s (secouez bien le moustique dans le tube pour détacher les écailles et les contaminants bactériens potentiels à la surface des insectes). Rincer deux fois à l’aide de tubes contenant de l’éthanol.
- Transférez le moustique dans un nouveau tube stérilisé de 1,5 mL avec 1 mL de solution stérile 1x PBS et agitez doucement (manuellement) pendant 30 s (pour éviter la précipitation de l’ADN avec de l’éthanol). Rincer deux fois à l’aide de tubes contenant 1x PBS stérile.
4. Dissection des moustiques
- Placez plusieurs petites gouttes de 1x PBS stérile sur la lame pour faciliter le placement du moustique et le rinçage des organes disséqués.
REMARQUE : Les gouttes de PBS stérile doivent être suffisamment grandes pour accueillir les organes du moustique. - Prenez le moustique nettoyé et placez-le sur une goutte de PBS stérile 1x sur la lame du microscope.
- Disséquez les glandes salivaires à l’aide d’une pince et d’une aiguille stérilisées comme décrit ci-dessous.
- Tenez le thorax du moustique avec une pince à épiler pour l’empêcher de bouger. Ensuite, placez une aiguille au niveau du cou, sous la tête, et tirez doucement sur la tête du moustique pour la séparer du thorax.
REMARQUE : Les glandes salivaires sont situées dans la partie supérieure du thorax et sont reliées à la tête par le canal salivaire. Lors de la dissection, il est crucial de ne pas casser ce conduit car les glandes salivaires ne viendront pas avec la tête lorsque vous la retirerez du thorax. C’est pourquoi il est important d’avoir un moustique frais, pas un moustique congelé. - Lorsque la tête se sépare du thorax, observez l’émergence des deux glandes salivaires, chacune composée de trois lobes. Coupez les glandes salivaires au niveau du canal salivaire, récupérez-les à l’aide d’une aiguille et placez-les dans une nouvelle goutte de PBS stérile 1x pour la rincer et prévenir les contaminations inter-organes.
- Récupérez les glandes salivaires à l’aide d’une aiguille et placez-les dans un nouveau tube stérilisé contenant 100 μL de tampon de conservation.
- Tenez le thorax du moustique avec une pince à épiler pour l’empêcher de bouger. Ensuite, placez une aiguille au niveau du cou, sous la tête, et tirez doucement sur la tête du moustique pour la séparer du thorax.
- Disséquez les ovaires à l’aide d’une pince et d’une aiguille stérilisées comme suit.
- Placez le moustique sur son dos. À l’aide d’une pince pour tenir le moustique par le thorax afin d’empêcher tout mouvement pendant la dissection, percez le troisième segment abdominal (en commençant par le bas) avec une aiguille.
REMARQUE : En option, retirez les pattes et les ailes du moustique pour faciliter la dissection. - Tout en perçant le troisième segment abdominal, tirez vers le bas pour l’ouvrir et révéler les organes internes. Retirez tous les morceaux restants de l’exosquelette du moustique pour faciliter la libération des organes.
REMARQUE : Les organes de l’abdomen tels que l’intestin, les tubules de Malpighi et les ovaires sont visibles. - Prélevez les deux ovaires à l’aide d’une pince stérilisée et placez-les dans une nouvelle goutte de PBS stérile 1x sur la lame de microscope pour les rincer. Saisissez les ovaires et placez-les dans le tube de 1,5 ml avec les 100 μL de tampon de conservation.
- Placez le moustique sur son dos. À l’aide d’une pince pour tenir le moustique par le thorax afin d’empêcher tout mouvement pendant la dissection, percez le troisième segment abdominal (en commençant par le bas) avec une aiguille.
- Maintenant que l’abdomen est ouvert et que les organes sont exposés, récupérez l’intestin moyen à l’aide d’une nouvelle pince et/ou d’une aiguille stérilisée.
- Séparez l’intestin moyen de l’intestin antérieur et de l’intestin postérieur ; ensuite, retirez les tubules de Malpighi de l’intestin moyen. Saisissez l’intestin moyen à l’aide d’une aiguille stérile et placez-le sur une nouvelle goutte stérile 1x PBS pour le rincer et prévenir les contaminations inter-organes.
- Transférer dans une nouvelle goutte stérile 1x PBS. Ouvrez l’intestin moyen avec une pince pour libérer et enrichir les bactéries comme décrit dans le fichier supplémentaire 1. Saisissez l’intestin moyen à l’aide d’une pince et pipetez tous les PBS contenant des bactéries pour stocker l’ensemble du matériel (liquide et tissu) dans un nouveau tube stérilisé de 1,5 ml avec 100 μL de tampon de conservation.
- Ajoutez à nouveau 5 μL de PBS 1x à l’endroit précédemment utilisé dans la lame de microscope et pipetez directement le PBS dans le même tube de 1,5 mL pour récupérer les bactéries qui auraient pu coller à la lame de microscope.
- À l’aide d’une pince stérile, saisissez la carcasse (corps restant) et rangez-la dans un nouveau tube stérilisé de 1,5 mL avec 100 μL de tampon de conservation.
- Stockage du matériel jusqu’à la suite du traitement
- Étiquetez les tubes avec « Espèce, Orgue, Date, Nom du chercheur ».
- Conservez les ovaires, l’intestin moyen, les glandes salivaires et la carcasse disséqués à -20 °C jusqu’à la transformation.
- Gardez une trace dans une feuille de calcul de toutes les métadonnées associées aux échantillons, y compris l’espèce (ou le niveau du genre seulement), la date de collecte, le pays, la province, la latitude, la longitude, le nom de la personne qui a fait la dissection, la femelle gravide ou non, le repas de sang ou non.
Résultats
x’Dissections de moustiques
Conformément au protocole, nous avons collecté et isolé les ovaires, l’intestin moyen, les glandes salivaires et la carcasse de deux spécimens de Culex pipiens molestus (dont une femelle gravide) provenant d’une colonie de laboratoire. Nous avons confirmé des dissections propres après l’observation d’organes entiers (c’est-à-dire non brisés) et bien isolés, sans débris restants sous les jumelles. L’ensemble du corps, la tête et le thorax, les glandes salivaires disséquées, les ovaires et l’intestin moyen d’un spécimen de Culex pipiens molestus sont illustrés à la figure 2. Comme prévu, l’intestin moyen et les glandes salivaires étaient d’un ordre de grandeur plus petit que les ovaires des moustiques. Les œufs d’un spécimen gravide de Culex, ainsi que ses tubules de l’intestin moyen et de Malpighian, sont présentés sur la figure 3. Il convient de noter que les dissections sont plus susceptibles d’échouer lorsque le matériau n’est pas frais et que le tissu est susceptible de se rompre (Figure 4B,C). Nous suggérons donc de disséquer le matériel juste après le prélèvement (dans les moins de 12 h) lorsque cela est possible, car les tissus sont encore élastiques (Figure 4A). De même, la dissection de matériaux congelés peut être effectuée, mais le risque d’échec et de contamination croisée entre les organes est beaucoup plus élevé en raison de la fragilité des tissus (Figure 4).
Données sur le microbiome
De plus, nous avons collecté et séparé les intestins moyens et les ovaires de trois spécimens individuels de Culex quinquefasciatus de Nouméa, en Nouvelle-Calédonie, en suivant la même procédure. Nous avons extrait l’ADN de chaque organe, préparé des échantillons pour le séquençage du génome entier et effectué des analyses de données sur le microbiome, comme détaillé dans le fichier supplémentaire 1. Une analyse de la diversité taxonomique bactérienne sur des lectures courtes non assemblées filtrées de qualité à l’aide de PhyloFlash12 a montré des taxons dominants distincts dans l’intestin moyen par rapport aux ovaires (Figure 5A). Notamment, les communautés bactériennes ovariennes étaient dominées par Wolbachia, avec la présence supplémentaire d’un phylotype Spirochaetaceae dans les ovaires de l’individu 3, tandis que les communautés de l’intestin moyen présentaient une plus grande diversité, y compris les Gammaprotéobactéries, les Spirochaetaceae et les Firmicutes. À partir des mêmes données de séquençage, nous avons reconstruit quatre génomes assemblés par métagénome (MAG) avec un achèvement > 80 % et une redondance < 5 %, appartenant aux classes Spirochaetia, Alpha- et Gammaproteobacteria (Tableau 1).
Comme prévu, les MAG reconstruits ici n’ont pas couvert toute la diversité taxonomique prédite par les résultats de PhyloFlash en raison des lacunes spécifiques de la reconstruction du génome à l’aide de lectures courtes métagénomiques. La MAG attribuée à Wolbachia (réf. MAG 3 dans le tableau 1) a été détectée dans tous les ovaires et deux des intestins moyens et avait une couverture plus élevée dans les ovaires (Figure 5B). Nous avons également reconstruit deux MAG appartenant à la famille des Enterobacteriaceae , y compris le genre Pantoea (Réf. MAG 1 et 2 dans le tableau 1), dans les intestins moyens obtenus sur les individus Culex 2 et 3, qui n’ont pas été détectés dans les ovaires correspondants (Figure 5B). Enfin, nous avons également reconstruit un génome bactérien de Spirochaetaceae , réf. MAG 4 (tableau 1), attribué au genre BR149 qui a été isolé avec succès de l’intestin moyen de Culex pipiens par Graña-Miraglia et ses collègues13. Il est intéressant de noter que ce MAG a été détecté dans l’intestin moyen des individus 1 et 3, ainsi que dans les ovaires de l’individu 3 (Figure 5B).
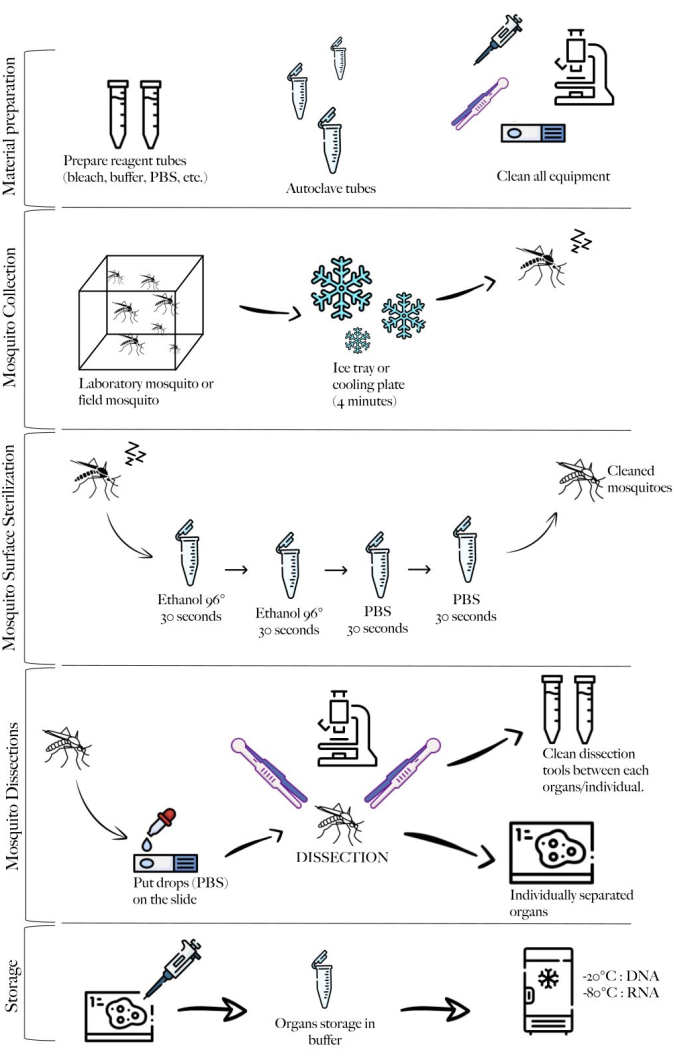
Figure 1 : Schéma de la méthode résumant les différentes étapes. Préparation du matériel, collecte des moustiques, nettoyage des moustiques, dissections de moustiques et stockage. Veuillez cliquer ici pour voir une version agrandie de cette figure.
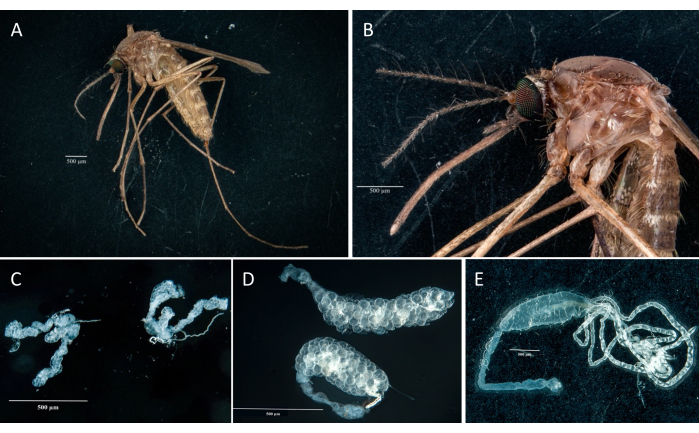
Figure 2 : Culex pipiens molestus femelle. (A) Corps entier. (B) Tête et thorax. (C) Glandes salivaires disséquées. (D) Ovaires disséqués. (E) Intestin disséqué avec des tubules de Malpighi. Barres d’échelle = 500 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
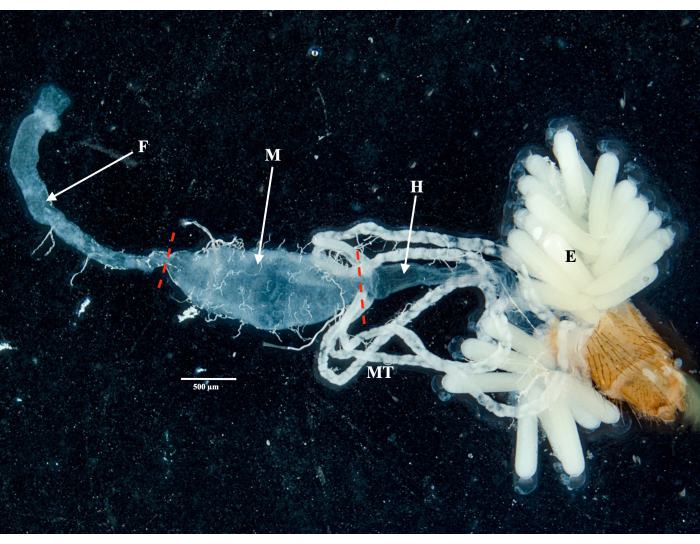
Figure 3 : Abdomen disséqué d’une femelle gravide Culex pipiens molestus. L’intestin antérieur, l’intestin moyen, l’intestin postérieur, les tubules de Malpighi et les œufs du spécimen sont montrés. Des tirets rouges indiquent où couper pour séparer l’intestin moyen de l’intestin antérieur et postérieur. Barre d’échelle = 500 μm. Abréviations : F = intestin antérieur ; M = intestin moyen ; H = intestin postérieur ; E = œufs ; MT = Tubules de Malpighian. Veuillez cliquer ici pour voir une version agrandie de cette figure.
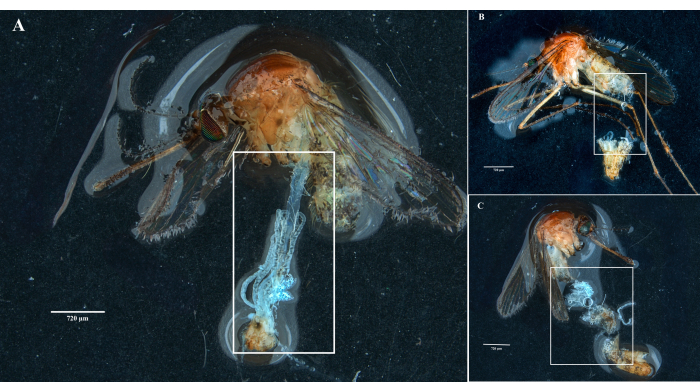
Figure 4 : Dissection d’échantillons de moustiques Culex frais et congelés. Les rectangles blancs illustrent (A) des tissus de moustiques avec des organes intacts d’un échantillon fraîchement disséqué et (B,C) du matériel biologique brisé provenant d’échantillons qui ont été congelés avant la dissection. Barres d’échelle = 720 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
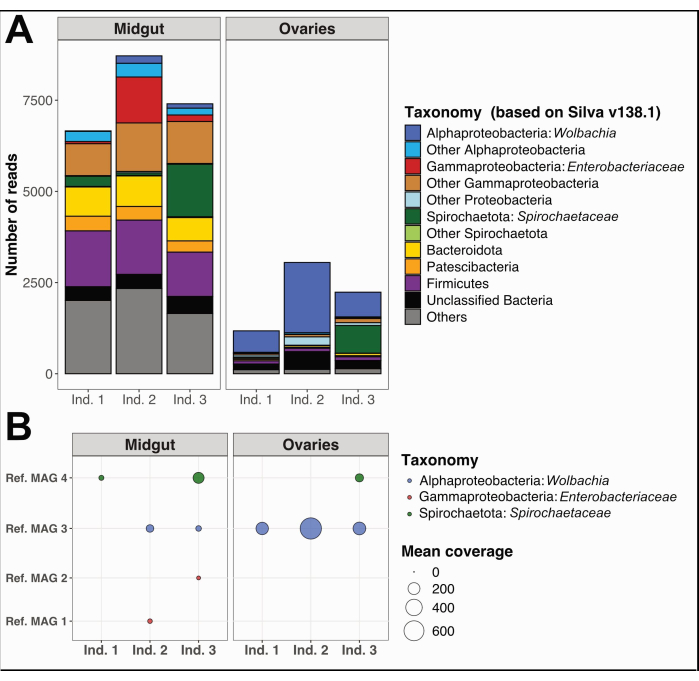
Figure 5 : Exemple d’analyse du microbiome sur trois individus de Culex quinquefasciatus. (A) Visualisation de la diversité bactérienne estimée par l’extraction des lectures d’ARNr SSU avec PhyloFlash12 dans les intestins moyens et les ovaires des trois échantillons. (B) Couverture moyenne des MAG reconstitués sur les échantillons. Abréviations : MAG = Metagenome-Assembled Genome ; Ind = Individuel. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| REVUE | Réf. MAG 4 | Réf. MAG 2 | Réf. MAG 1 | Réf. MAG 3 |
| Longueur (bp) | 1,287,790 | 4,910,866 | 4,751,276 | 1,298,266 |
| Nombre de contigs | 9 | 162 | 149 | 123 |
| % GC | 34.05 | 55.45 | 54.05 | 34.15 |
| Achèvement (%) | 84.5 | 97.18 | 98.59 | 91.55 |
| Redondance (%) | 0 | 2.82 | 4.22 | 0 |
| Domaine | Bactéries | Bactéries | Bactéries | Bactéries |
| Embranchement | Spirochaetota | Protéobactéries | Protéobactéries | Protéobactéries |
| Classe | Spirochaetia | Gammaprotéobactéries | Gammaprotéobactéries | Alphaprotéobactéries |
| Commande | WRBN01 | Entérobactéries | Entérobactéries | Rickettsiales |
| Famille | WRBN01 | Entérobactéries | Entérobactéries | Anaplasmatacées |
| Genre | BR149 | Pantoea | - | Wolbachia |
Tableau 1 : MAG reconstitués des trois individus de Culex quinquefasciatus . La taille du génome, le nombre de contigs, la proportion de GC, les estimations de l’achèvement et la redondance basées sur la collection de gènes de base à copie unique disponible dans Anvi’o17 et la taxonomie obtenue à l’aide de GTDB18.
Fichier supplémentaire 1 : Exemple détaillé de procédure pour l’analyse des données du microbiome à partir de la collecte d’échantillons, de l’extraction de l’ADN et du séquençage du génome entier, suivie du flux de travail bioinformatique pour la reconstruction du génome et l’estimation de la proportion de lecture procaryote et eucaryote Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Nous vous recommandons de porter une attention particulière à la séquence de dissection de l’organe, en commençant par les glandes salivaires. En effet, nous avons observé qu’ils étaient plus facilement extraits du thorax des spécimens de Culex si l’intégrité du moustique était préservée. Des dommages à l’abdomen ou au thorax pourraient réduire la pression dans le corps du moustique, entravant la procédure. Cependant, il est également possible de couper entre le thorax et l’abdomen puis d’extraire les glandes salivaires de la tête et du thorax (A. B. Failloux, communication personnelle). De plus, il peut être difficile d’acquérir des compétences de dissection, c’est pourquoi nous vous suggérons de vous entraîner sur un nombre adéquat d’échantillons avant l’expérience.
Isoler les organes des moustiques tout en évitant la contamination croisée de manière systématique est crucial pour un large éventail d’analyses du microbiote des moustiques en aval. Une étude métagénomique résolue par le génome suite à la dissection d’ovaires uniques de spécimens de Culex pipiens dans le sud de la France a permis de découvrir le premier plasmide de Wolbachia chez Culex pipiens (pWCP14). En utilisant une approche similaire, nous avons étudié la distribution et la fréquence de la pWCP chez les spécimens de Culex pipiens et de Culex quinquefasciatus provenant de zones continentales et insulaires du monde entier, dans diverses conditions environnementales et de laboratoire. Dans l’ensemble, les données ont révélé un élément plasmidique Wolbachia remarquablement conservé chez les moustiques Culex , suggérant un rôle crucial pour cet élément mobile dans la biologie des endosymbiotes15, justifiant une analyse plus approfondie.
Nous fournissons ici d’autres exemples d’analyses du microbiome des moustiques sur des échantillons d’intestin moyen et d’ovaire obtenus à l’aide de cette procédure systématique. Nous avons observé une nette différence de microbiote entre les tissus (Figure 5), avec des taxons bactériens communs et spécifiques à un organe. Comme prévu, la présence de Wolbachia a été détectée dans les deux organes, avec une abondance relative plus élevée (basée sur de courtes lectures non assemblées) et une couverture moyenne des MAG dans les ovaires des moustiques par rapport aux intestins moyens, ce qui correspond à l’observation selon laquelle cet endosymbiote est transmis par les ovaires et se propage ensuite aux tissus somatiques. Bien que cette étude ait été limitée à des échantillons de Nouvelle-Calédonie, ce protocole pourrait faciliter l’étude de la variabilité génomique de Wolbachia à l’échelle mondiale, ainsi que de son rôle dans divers phénotypes, y compris la régulation de la densité et la protection virale. De plus, ce travail illustre comment la procédure de dissection présentée ici permet d’examiner la diversité taxonomique et les capacités fonctionnelles potentielles des symbiotes de moustiques dans des échantillons d’intestin moyen.
Nous avons obtenu deux ébauches de génomes entérobactériens qui n’étaient présentes que dans deux échantillons d’intestin moyen, confirmant l’absence de contamination entre les organes pour ces deux échantillons. En ce qui concerne les spirochètes, détectés à la fois dans l’intestin moyen et les ovaires de l’individu 3, Juma et ses collègues ont observé en 2020 la présence de ces bactéries à la surface des radeaux d’œufs. Les auteurs ont suggéré que les communautés bactériennes trouvées dans les radeaux d’œufs pourraient être principalement héritées de la mère des ovaires, étant donné que les radeaux d’œufs étaient conservés dans de l’eau désionisée et exempte de bactéries. Cependant, ils n’ont pas pu exclure la possibilité d’une colonisation bactérienne juste après la ponte et ont recommandé une étude plus approfondie sur le microbiome ovaire16.
Bien qu’initialement conçu pour des spécimens du complexe d’espèces Culex pipiens , nous prévoyons l’applicabilité de ce protocole à une communauté plus large d’entomologistes médicaux étudiant d’autres vecteurs tels que les anophèles ou les aedes. En opérant au niveau des organes individuels, cette méthode peut permettre des comparaisons génomiques intra et interindividuelles, offrant des informations sur la variabilité génomique des symbiotes à une petite échelle, avec le potentiel de faire progresser les stratégies de lutte antivectorielle. Cette méthode de dissection et d’isolement des glandes salivaires, des ovaires et de l’intestin moyen, prévenant la contamination croisée microbienne, pourrait également être un protocole utile pour les études de dynamique des infections virales au sein de ces trois organes.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont aucun conflit d’intérêts.
Remerciements
Nous remercions Gilbert Legoff d’avoir enseigné à Jordan Tutagata comment disséquer les glandes salivaires des moustiques et Giuliano Mucci de nous avoir aidés avec des photos des organes des moustiques. Nous remercions Anna-Bella Failloux et Nonito Pages pour la discussion utile sur le protocole. Ce travail a été soutenu par la bourse de démarrage ERC RosaLind « 948135 » à JR. Nous remercions la plateforme Vectopole (IRD, Montpellier) pour l’appui technique et pour l’élevage et le maintien des populations de moustiques.
matériels
| Name | Company | Catalog Number | Comments |
| Alcohol 96° | Fisher scientific | 10332562 | |
| Binocular magnifier | |||
| Bleach | RAJA | 145517 | 150 tablets of 1.5 g |
| DNA prep kit | Illumina | Provided by MGX sequencing platform | Previously known as Nextera DNA Flex |
| DNA-RNA Shield (50 mL) | Zymo research | ZR1100-50 | Preservation buffer |
| DNeasy Blood and Tissue Kit | Qiagen | 69504 | DNA extraction kit |
| Filter tips 20 µL | Starlab | S1120-3810 | |
| Filter tips 200 µL | Starlab | S1120-8710 | |
| Filter tips 1000 µL | Starlab | S1122-1730-C | |
| Forceps | FST (Fine Science Tools) | 11252-20 | Dumont Forceps #5 |
| Library quantification kit | Roche | Provided by MGX sequencing platform | KAPA Library Quanitification Kits |
| Micropipettes 2-20 µL | Eppendorf | 6.291704 | |
| Micropipettes 20-200 µL | Eppendorf | 6.291703 | |
| Micropipettes 100-1000 µL | Eppendorf | 7.648488 | |
| Microscope slides | Epredia | J1800BMNZ | dimension : 75 mm x 50 mm |
| Needles | Terumo | AN*2719R1 | |
| NGS kit | Agilent | Provided by MGX sequencing platform | Fragment Analyzer Systems HS Genomic DNA 50kb Kit |
| NovaSeq 6000 | Illumina | Provided by MGX sequencing platform | Sequencer |
| PBS Phosphate Buffered Saline (Sterile) | Fisher scientific | 10212990 | |
| Permanent black marker | |||
| Sterile Eppendorf | Dutscher | 33871 | 1.5 mL |
| Support for needles | FST (Fine Science Tools) | 26016-12 | Moria MC1 Pin Holder 12 cm |
Références
- Achee, N. L., et al. Alternative strategies for mosquito-borne arbovirus control. PLOS Negl Trop Dis. 13 (1), e0006822 (2019).
- Hoffmann, A. A., et al. Successful establishment of Wolbachia in Aedes populations to suppress dengue transmission. Nature. 476 (7361), 454-457 (2011).
- Porter, J., Sullivan, W. The cellular lives of Wolbachia. Nat Rev Microbiol. 21 (11), 750-766 (2023).
- Coon, K. L., Brown, M. R., Strand, M. R. Mosquitoes host communities of bacteria that are essential for development but vary greatly between local habitats. Mol Ecol. 25 (22), 5806-5826 (2016).
- Didion, E. M., et al. Microbiome reduction prevents lipid accumulation during early diapause in the northern house mosquito, Culex pipiens pipiens. J Insect Physiol. 134, 104295 (2021).
- Gabrieli, P., et al. Mosquito trilogy: Microbiota, immunity and pathogens, and their implications for the control of disease transmission. Front Microbiol. 12, 630438 (2021).
- Garrigós, M., Garrido, M., Panisse, G., Veiga, J., Martínez-de La Puente, J. Interactions between West Nile Virus and the microbiota of Culex pipiens vectors: A literature review. Pathogens. 12 (11), 1287 (2023).
- Zheng, R., et al. Holobiont perspectives on tripartite interactions among microbiota, mosquitoes, and pathogens. ISME J. 17 (8), 1143-1152 (2023).
- Flores, G. A. M., et al. Wolbachia dominance influences the Culex quinquefasciatus microbiota. Sci Rep. 13 (1), 18980 (2023).
- Hegde, S., et al. Interkingdom interactions shape the fungal microbiome of mosquitoes. Anim Microbiome. 6 (1), 11 (2024).
- Suo, P., et al. Seasonal variation of midgut bacterial diversity in Culex quinquefasciatus populations in Haikou city, Hainan province, China. Biology. 11 (8), 1166 (2022).
- Gruber-Vodicka, H. R., Seah, B. K. B., Pruesse, E. phyloFlash: Rapid small-subunit rRNA profiling and targeted assembly from metagenomes. mSystems. 5 (5), e00920-e00920 (2020).
- Graña-Miraglia, L., et al. Spirochetes isolated from arthropods constitute a novel genus Entomospira genus novum within the order Spirochaetales. Sci Rep. 10 (1), 17053 (2020).
- Reveillaud, J., et al. The Wolbachia mobilome in Culex pipiens includes a putative plasmid. Nat Commun. 10 (1), 1051 (2019).
- Ghousein, A., et al. pWCP is a widely distributed and highly conserved Wolbachia plasmid in Culex pipiens and Culex quinquefasciatus mosquitoes worldwide. ISME Commun. 3 (1), 40 (2023).
- Juma, E. O., Kim, C. -. H., Dunlap, C., Allan, B. F., Stone, C. M. Culex pipiens and Culex restuans egg rafts harbor diverse bacterial communities compared to their midgut tissues. Parasit Vectors. 13 (1), 532 (2020).
- Lee, M. D. GToTree: a user-friendly workflow for phylogenomics. Bioinformatics. 35 (20), 4162-4164 (2019).
- Parks, D. H., et al. A complete domain-to-species taxonomy for bacteria and Archaea. Nat Biotechnology. 38 (9), 1079-1086 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon