Method Article
Dissezione delle ovaie, dell'intestino medio e delle ghiandole salivari delle zanzare per l'analisi del microbioma a livello d'organo
In questo articolo
Riepilogo
Le comunità microbiche nelle zanzare sono molto promettenti per le strategie di biocontrollo dei vettori. La maggior parte dei simbionti non sono coltivabili e richiedono analisi metagenomiche. Descriviamo un metodo per sezionare le zanzare femmine e separare le ovaie, l'intestino medio e le ghiandole salivari prevenendo la contaminazione incrociata, facilitando gli studi sul microbioma a livello di organo e migliorando la comprensione dei ruoli dei microrganismi nella biologia delle zanzare.
Abstract
L'onere globale delle malattie trasmesse dalle zanzare, tra cui la malaria, la dengue, il Nilo occidentale, la Zika, l'Usutu e la febbre gialla, continua ad aumentare, rappresentando una significativa minaccia per la salute pubblica. Con l'aumento della resistenza agli insetticidi e l'assenza di vaccini efficaci, stanno emergendo nuove strategie che si concentrano sul microbiota della zanzara. Tuttavia, la maggior parte dei simbionti rimane resistente alla coltivazione. La caratterizzazione della diversità e della funzione dei genomi batterici negli esemplari di zanzara, quindi, si basa sulla metagenomica e sulle successive strategie di assemblaggio e binning. L'ottenimento e l'analisi dei genomi assemblati con metagenoma (MAG) da organi separati possono fornire informazioni chiave sul ruolo specifico dei microbi associati alle zanzare nelle ovaie (gli organi riproduttivi), nell'intestino medio (chiave per la digestione e l'immunità del cibo) o nelle ghiandole salivari (essenziali per la trasmissione di malattie trasmesse da vettori poiché gli agenti patogeni devono colonizzarle per entrare nella saliva e raggiungere il flusso sanguigno durante un pasto di sangue). Questi genomi appena ricostruiti possono quindi aprire la strada allo sviluppo di nuove strategie di biocontrollo dei vettori. A tal fine, è necessario isolare gli organi delle zanzare evitando la contaminazione incrociata tra di loro o con microrganismi presenti in altri organi delle zanzare. Qui, descriviamo un protocollo di dissezione ottimizzato e privo di contaminazioni per lo studio del microbioma delle zanzare a livello di organo.
Introduzione
Le zanzare diffondono una vasta gamma di agenti patogeni che causano malattie e rappresentano una seria minaccia per la salute pubblica. A causa di una maggiore prevalenza di resistenza agli insetticidi tra le popolazioni di zanzare e in assenza di vaccini efficaci contro questi agenti patogeni, stanno emergendo nuovi metodi di biocontrollo che si concentrano sul microbioma delle zanzare. In particolare, spicca il batterio intracellulare Wolbachia, che può interferire con la trasmissione di agenti patogeni e manipolare la riproduzione dell'ospite 1,2,3. Inoltre, altri simbionti di zanzara sono fondamentali per la sopravvivenza, lo sviluppo o il sistema immunitario del loro ospite, nonché nell'infezione e nella trasmissione di agenti patogeni, e mostrano grandi promesse per il loro sfruttamento per combattere le malattie trasmesse da vettori 4,5,6,7,8.
I microrganismi associati alle zanzare abbracciano tutti i domini della vita microbica (inclusi batteri, eucarioti e funghi) che interagiscono intimamente con il loro ospite ma anche tra loro nei diversi compartimenti corporei 9,10. Pertanto, una migliore comprensione del microbiota, della sua potenziale variazione stagionale11 e dei meccanismi attraverso i quali i suoi membri interagiscono direttamente nei distinti tessuti delle zanzare può aiutare a sviluppare nuovi metodi di biocontrollo mirati o migliorare quelli esistenti. Tuttavia, la maggior parte dei simbionti rimane resistente alla coltivazione, rendendo impossibile la loro caratterizzazione.
L'avvento di metodi di sequenziamento di seconda e terza generazione, insieme ad approcci di assemblaggio e binning all'avanguardia, hanno consentito la ricostruzione dei genomi microbici e l'accesso alla diversità e al potenziale funzionale dei microbi non coltivabili. Qui, presentiamo un metodo per la dissezione delle ovaie, dell'intestino medio e delle ghiandole salivari delle zanzare femmine, prevenendo la contaminazione incrociata. Questo protocollo può essere seguito dall'estrazione del DNA genomico e dal successivo metabarcoding o sequenziamento metagenomico shotgun per esplorare la diversità e la funzione del microbiota delle zanzare a livello d'organo. Forniamo un esempio di dati sulla dissezione delle zanzare e sul microbioma per gli esemplari di Culex spp., sebbene questo protocollo possa essere esteso a vettori di altri generi come Anopheles o Aedes.
Protocollo
NOTA: La Figura 1 mostra uno schema del metodo che riassume le diverse fasi del protocollo.
1. Preparazione del materiale
- Prima di iniziare l'esperimento, preparare una soluzione di candeggina con cloro attivo allo 0,5% (raccomandata dall'Organizzazione Mondiale della Sanità) per disinfettare l'attrezzatura e il banco di lavoro.
- In 900 ml di acqua, sciogliere tre compresse solubili, ciascuna contenente 1,5 g di cloro attivo. Attendere 15 minuti affinché le compresse si dissolvano completamente, ottenendo una soluzione di candeggina allo 0,5%.
- Metti la soluzione di candeggina allo 0,5% in un flacone spray. Quindi, spruzzare la soluzione su tutte le apparecchiature di dissezione, compresi i vetrini microscopici e il banco di lavoro.
NOTA: L'attrezzatura e il banco da lavoro devono essere lasciati asciugare per ~10 minuti prima dell'uso.
- Per sciacquare le zanzare prima di ogni dissezione per evitare la contaminazione dei campioni con batteri esterni, preparare quattro provette da 1,5 ml appena sterilizzate: due contenenti etanolo al 96% e altre due contenenti 1x tampone PBS (soluzione salina tamponata con fosfato).
- Preparare le provette per conservare i campioni dopo la dissezione di ogni zanzara: quattro provette sterili da 1,5 mL, ciascuna contenente 100 μL di tampone di conservazione.
NOTA: Preparare un tubo per le ghiandole salivari, uno per le ovaie, uno per l'intestino medio e uno per la carcassa della zanzara.
2. Raccolta delle zanzare
- Raccogliere e mantenere in vita individui di sesso femminile adulto (dal campo o dal laboratorio, 4-7 giorni) fino alla sterilizzazione superficiale.
3. Sterilizzazione superficiale delle zanzare
NOTA: Pulire pinze e aghi e utilizzare tubi nuovi e sterilizzati tra ogni organo e individuo. Se la candeggina non è disponibile, assicurarsi di sterilizzare accuratamente a fiamma le pinze e gli aghi da dissezione tra la dissezione di ciascun individuo e organo per prevenire la contaminazione incrociata del materiale biologico. Gli strumenti di dissezione devono essere asciutti, senza residui di candeggina/etanolo, prima dell'uso successivo.
- Per evitare che le femmine svuotino il loro contenuto intestinale quando esposte all'alcol, anestetizzarle con ghiaccio o in congelatore a -20 °C (per 4 minuti in modo che i tessuti e gli organi non si congelino).
- Trasferire le singole zanzare in una nuova provetta sterilizzata da 1,5 ml con 1 ml di etanolo al 96%. Agitare delicatamente (manualmente) per 30 s (agitare bene la zanzara nel tubo per staccare le squame e i potenziali contaminanti batterici sulla superficie degli insetti). Sciacquare due volte utilizzando tubi contenenti etanolo.
- Trasferire la zanzara in una nuova provetta sterilizzata da 1,5 ml con 1 ml di soluzione sterile 1x PBS e agitare delicatamente (manualmente) per 30 s (per evitare la precipitazione del DNA con l'etanolo). Risciacquare due volte utilizzando tubi contenenti 1x PBS sterile.
4. Dissezione delle zanzare
- Posizionare alcune piccole gocce di 1x PBS sterile sul vetrino per facilitare il posizionamento della zanzara e il risciacquo degli organi sezionati.
NOTA: Le gocce di PBS sterile devono essere abbastanza grandi da accogliere gli organi della zanzara. - Prendi la zanzara pulita e posizionala su una goccia di PBS sterile 1x sul vetrino del microscopio.
- Sezionare le ghiandole salivari con una pinza sterilizzata e un ago come descritto di seguito.
- Tieni il torace della zanzara con una pinzetta per evitare che si muova. Quindi, posiziona un ago sul collo, sotto la testa, e tira delicatamente la testa della zanzara per separarla dal torace.
NOTA: Le ghiandole salivari si trovano nella parte superiore del torace e sono collegate alla testa dal dotto salivare. Durante la dissezione, è fondamentale non rompere questo dotto poiché le ghiandole salivari non verranno con la testa quando la si estrae dal torace. Questo è il motivo per cui è importante avere una zanzara fresca, non congelata. - Quando la testa si separa dal torace, si osservino emergere le due ghiandole salivari, ciascuna composta da tre lobi. Tagliare le ghiandole salivari a livello del dotto salivare, recuperarle con un ago e metterle in una nuova goccia di PBS sterile 1x per risciacquarle e prevenire contaminazioni interorgani.
- Recuperare le ghiandole salivari utilizzando un ago e inserirle in una nuova provetta sterilizzata contenente 100 μL di tampone di conservazione.
- Tieni il torace della zanzara con una pinzetta per evitare che si muova. Quindi, posiziona un ago sul collo, sotto la testa, e tira delicatamente la testa della zanzara per separarla dal torace.
- Sezionare le ovaie con una pinza sterilizzata e l'ago come segue.
- Posiziona la zanzara sul dorso. Usando una pinza per tenere la zanzara per il torace per impedirne il movimento durante la dissezione, perforare il terzo segmento addominale (partendo dal basso) con un ago.
NOTA: Facoltativamente, rimuovere le zampe e le ali della zanzara per facilitare la dissezione. - Durante il piercing al terzo segmento addominale, tirare verso il basso l'addome per aprirlo e rivelare gli organi interni. Rimuovere eventuali pezzi rimanenti di esoscheletro di zanzara per facilitare il rilascio degli organi.
NOTA: Sono visibili organi nell'addome come l'intestino, i tubuli di Malpighian e le ovaie. - Recuperare entrambe le ovaie con una pinza sterilizzata e posizionarle in una nuova goccia di PBS sterile 1x sul vetrino del microscopio per sciacquarle. Afferrare le ovaie e posizionarle nella provetta da 1,5 ml con 100 μl di tampone di conservazione.
- Posiziona la zanzara sul dorso. Usando una pinza per tenere la zanzara per il torace per impedirne il movimento durante la dissezione, perforare il terzo segmento addominale (partendo dal basso) con un ago.
- Ora che l'addome è aperto e gli organi sono esposti, recupera l'intestino medio con una nuova pinza sterilizzata e/o un ago.
- Separare l'intestino medio dall'intestino anteriore e posteriore; quindi, rimuovere i tubuli malpighiani dall'intestino medio. Afferra l'intestino medio usando un ago sterile e posizionalo su una nuova goccia sterile 1x PBS per sciacquarlo e prevenire contaminazioni interorganiche.
- Trasferire in una nuova goccia sterile 1x PBS. Aprire l'intestino medio con una pinza per liberare e arricchire i batteri come descritto nel File supplementare 1. Afferrare l'intestino medio con una pinza e pipettare tutto il PBS contenente batteri per conservare l'intero materiale (liquido e tessuto) in una nuova provetta sterilizzata da 1,5 ml con 100 μl di tampone di conservazione.
- Aggiungere nuovamente 5 μl di PBS 1x nel punto precedentemente utilizzato nel vetrino del microscopio e pipettare direttamente il PBS nella stessa provetta da 1,5 mL per recuperare i batteri che potrebbero essersi attaccati al vetrino del microscopio.
- Utilizzare una pinza sterile per afferrare la carcassa (il corpo rimanente) e conservarla in una nuova provetta sterilizzata da 1,5 ml con 100 μl di tampone di conservazione.
- Stoccaggio del materiale fino a nuova lavorazione
- Etichetta i tubi con "Specie, Organo, Data, Nome del ricercatore".
- Conservare le ovaie sezionate, l'intestino medio, le ghiandole salivari e la carcassa a -20 °C fino alla lavorazione.
- Tieni traccia in un foglio di calcolo di tutti i metadati associati ai campioni, tra cui specie (o solo a livello di genere), data di raccolta, paese, provincia, latitudine, longitudine, nome della persona che ha eseguito la dissezione, femmina gravida o meno, pasto di sangue o meno.
Risultati
x'Dissezioni delle zanzare
Seguendo il protocollo, abbiamo raccolto e isolato le ovaie, l'intestino medio, le ghiandole salivari e la carcassa di due esemplari di Culex pipiens molestus (tra cui una femmina gravida) da una colonia di laboratorio. Abbiamo confermato dissezioni pulite a seguito dell'osservazione di organi interi (cioè integri) e ben isolati senza detriti residui sotto il binocolo. L'intero corpo, la testa e il torace, le ghiandole salivari sezionate, le ovaie e l'intestino medio di un esemplare di Culex pipiens molestus sono mostrati nella Figura 2. Come previsto, l'intestino medio e le ghiandole salivari erano di un ordine di grandezza più piccole delle ovaie delle zanzare. Le uova di un esemplare gravido di Culex , insieme al suo intestino medio e ai tubuli malpighiani, sono presentate nella Figura 3. Da notare che le dissezioni sono più inclini a fallire quando il materiale non è fresco ed è probabile che il tessuto si rompa (Figura 4B, C). Si consiglia quindi di dissezionare il materiale subito dopo la raccolta (entro meno di 12 ore) quando possibile, poiché i tessuti sono ancora elastici (Figura 4A). Allo stesso modo, è possibile eseguire la dissezione di materiale congelato, ma esiste un rischio molto più elevato di fallimento e contaminazione incrociata tra gli organi a causa di tessuti fragili (Figura 4).
Dati del microbioma
Inoltre, abbiamo raccolto e separato l'intestino medio e le ovaie da tre singoli esemplari di Culex quinquefasciatus di Noumea, Nuova Caledonia, seguendo la stessa procedura. Abbiamo estratto il DNA da ciascun organo, preparato campioni per il sequenziamento dell'intero genoma ed eseguito analisi dei dati del microbioma come descritto nel File supplementare 1. Un'analisi della diversità tassonomica batterica su letture brevi non assemblate e filtrate di qualità utilizzando PhyloFlash12 ha mostrato taxa dominanti distinti nell'intestino medio rispetto alle ovaie (Figura 5A). In particolare, le comunità batteriche ovariche erano dominate da Wolbachia, con l'ulteriore presenza di un filotipo di Spirochaetaceae nelle ovaie degli individui 3, mentre le comunità dell'intestino medio mostravano una più ampia diversità, tra cui Gammaproteobacteria, Spirochaetaceae e Firmicutes. Dagli stessi dati di sequenziamento, abbiamo ricostruito quattro genomi assemblati in metagenoma (MAG) con completamento > 80% e ridondanza < 5%, appartenenti alle classi Spirochezia, Alfa- e Gammaproteobatteri (Tabella 1).
Come previsto, i MAG qui ricostruiti non coprivano l'intera diversità tassonomica prevista dai risultati di PhyloFlash a causa delle carenze specifiche della ricostruzione del genoma utilizzando letture brevi metagenomiche. Il MAG assegnato a Wolbachia (Rif. MAG 3 nella Tabella 1) è stato rilevato in tutte le ovaie e in due dell'intestino medio e ha avuto una copertura più elevata nelle ovaie (Figura 5B). Abbiamo anche ricostruito due MAG appartenenti alla famiglia delle Enterobacteriaceae , tra cui il genere Pantoea (Rif. MAG 1 e 2 nella Tabella 1), nell'intestino medio ottenuto dagli individui Culex 2 e 3, che non sono stati rilevati nelle ovaie corrispondenti (Figura 5B). Infine, abbiamo anche ricostruito un genoma batterico delle Spirochaetaceae , Ref. MAG 4 (Tabella 1), assegnato al genere BR149 che è stato isolato con successo da Culex pipiens midguts da Graña-Miraglia e colleghi13. È interessante notare che questo MAG è stato rilevato nell'intestino medio degli individui 1 e 3, così come nelle ovaie dell'individuo 3 (Figura 5B).
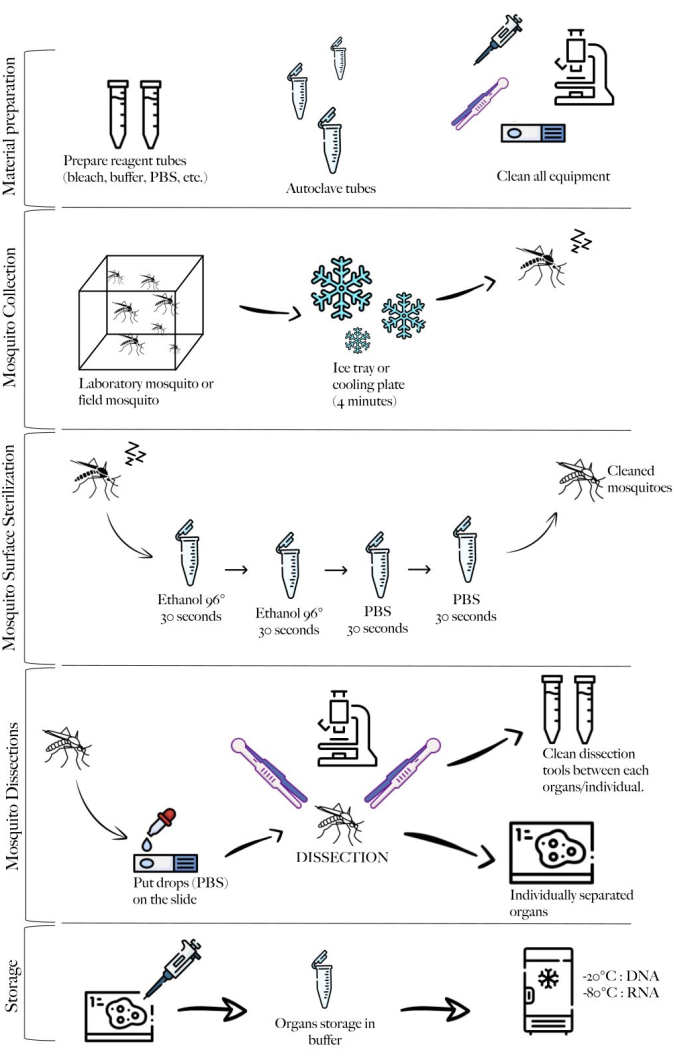
Figura 1: Schema del metodo che riassume i diversi passaggi. Preparazione del materiale, raccolta delle zanzare, pulizia delle zanzare, dissezione delle zanzare e stoccaggio. Clicca qui per visualizzare una versione più grande di questa figura.
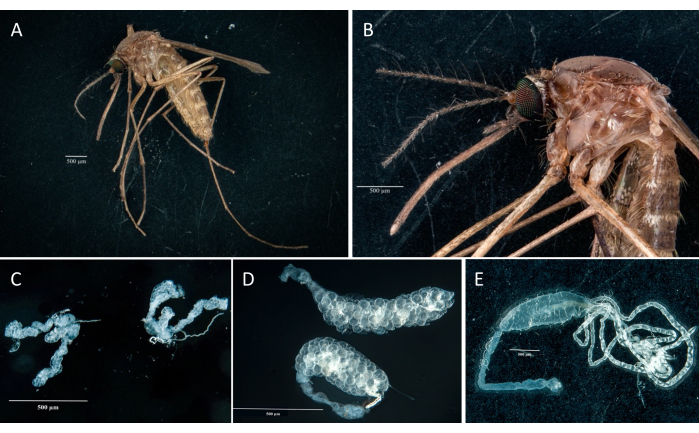
Figura 2: Culex pipiens molestus femmina. (A) Tutto il corpo. (B) Testa e torace. (C) Ghiandole salivari sezionate. (D) Ovaie sezionate. (E) Budello sezionato con tubuli di Malpighi. Barre di scala = 500 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
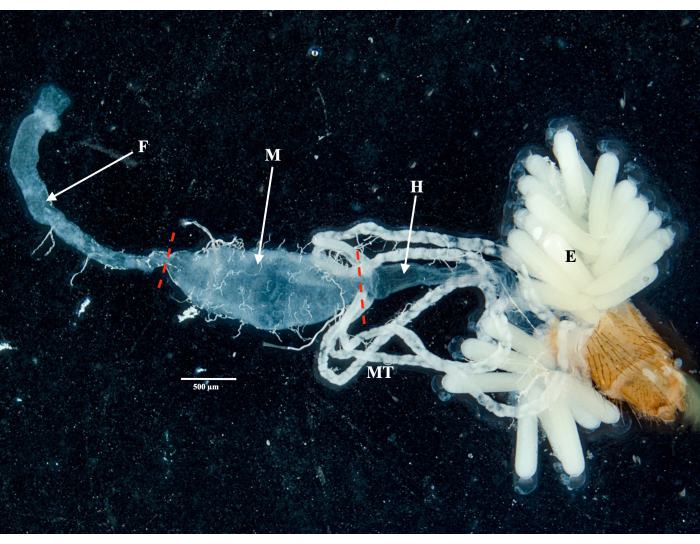
Figura 3: Addome sezionato di una femmina gravida di Culex pipiens molestus . Sono mostrati l'intestino anteriore, l'intestino medio, l'intestino posteriore, i tubuli malpighiani e le uova dell'esemplare. I trattini rossi indicano dove tagliare per separare l'intestino medio dall'intestino anteriore e dall'intestino posteriore. Barra della scala = 500 μm. Abbreviazioni: F = intestino anteriore; M = intestino medio; H = intestino posteriore; E = uova; MT = tubuli malpighiani. Clicca qui per visualizzare una versione più grande di questa figura.
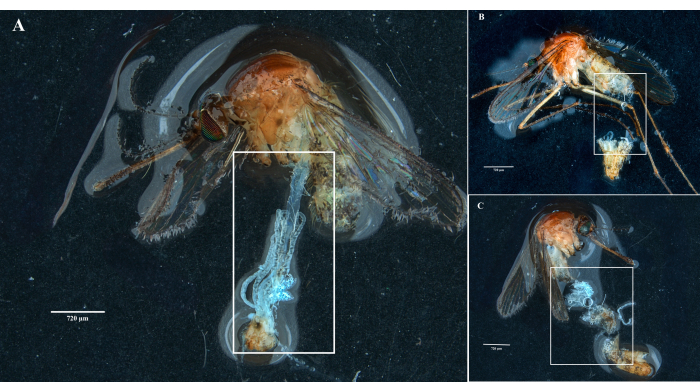
Figura 4: Dissezione di esemplari di zanzara Culex freschi e congelati. Rettangoli bianchi illustrano (A) tessuti di zanzara con organi intatti da un esemplare appena sezionato e (B,C) materiale biologico rotto da campioni che sono stati congelati prima della dissezione. Barre di scala = 720 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
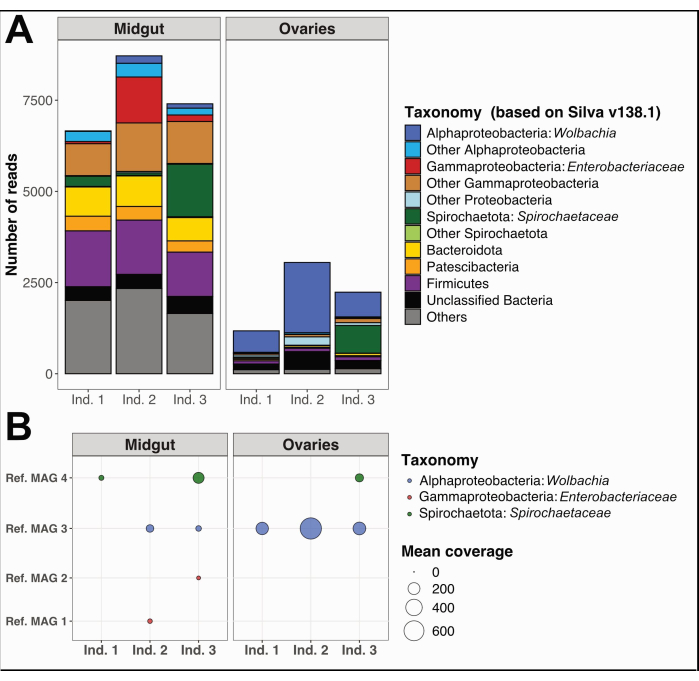
Figura 5: Esempio di analisi del microbioma su tre individui di Culex quinquefasciatus . (A) Visualizzazione della diversità batterica stimata attraverso l'estrazione di reads di rRNA SSU con PhyloFlash12 nell'intestino medio e nelle ovaie dei tre campioni. (B) Copertura media dei MAG ricostruiti sui campioni. Abbreviazioni: MAG = Metagenome-Assembled Genome; Ind = Individuale. Clicca qui per visualizzare una versione più grande di questa figura.
| MAG | Rif. MAG 4 | Rif. MAG 2 | Rif. MAG 1 | Rif. MAG 3 |
| Lunghezza (bp) | 1,287,790 | 4,910,866 | 4,751,276 | 1,298,266 |
| Numero di contig | 9 | 162 | 149 | 123 |
| % GC | 34.05 | 55.45 | 54.05 | 34.15 |
| Completamento (%) | 84.5 | 97.18 | 98.59 | 91.55 |
| Ridondanza (%) | 0 | 2.82 | 4.22 | 0 |
| Dominio | Batteri | Batteri | Batteri | Batteri |
| Phylum | Spirochaetota | Proteobatteri | Proteobatteri | Proteobatteri |
| Classe | Spirochezia | Gammaproteobatteri | Gammaproteobatteri | Alfaproteobatteri |
| Ordine | WRBN01 | Enterobacterales | Enterobacterales | Rickettsiales |
| Famiglia | WRBN01 | Enterobacteriaceae | Enterobacteriaceae | Anaplasmataceae |
| Genere | BR149 | Pantoea | - | Wolbachia |
Tabella 1: MAG ricostruiti dai tre individui di Culex quinquefasciatus . Dimensione del genoma, numero di contig, proporzione di GC, stime di completamento e ridondanza basate sulla collezione di geni core a copia singola disponibile in Anvi'o17 e sulla tassonomia ottenuta utilizzando GTDB18.
File supplementare 1: Esempio dettagliato di procedura per l'analisi dei dati del microbioma a partire dalla raccolta del campione, dall'estrazione del DNA e dal sequenziamento dell'intero genoma, seguita dal flusso di lavoro bioinformatico per la ricostruzione del genoma e la stima della proporzione di lettura procariotica ed eucariotica Fare clic qui per scaricare questo file.
Discussione
Si consiglia di prestare particolare attenzione alla sequenza di dissezione degli organi, a partire dalle ghiandole salivari. Infatti, abbiamo osservato che venivano estratti più facilmente dal torace degli esemplari di Culex se veniva preservata l'integrità della zanzara. I danni all'addome o al torace potrebbero ridurre la pressione nel corpo della zanzara, ostacolando la procedura. Tuttavia, è anche possibile tagliare tra il torace e l'addome e quindi estrarre le ghiandole salivari dalla testa e dal torace (A. B. Failloux, comunicazione personale). Inoltre, l'acquisizione di competenze di dissezione può essere impegnativa, quindi suggeriamo di esercitarsi su un numero adeguato di campioni prima dell'esperimento.
Isolare gli organi delle zanzare evitando la contaminazione incrociata in modo sistematico è fondamentale per un'ampia gamma di analisi del microbiota delle zanzare a valle. Uno studio metagenomico risolto nel genoma a seguito della dissezione di singole ovaie da campioni di Culex pipiens nel sud della Francia ha permesso la scoperta del primo plasmide di Wolbachia in Culex pipiens (pWCP14). Utilizzando un approccio simile, abbiamo studiato la distribuzione e la frequenza di pWCP in esemplari di Culex pipiens e Culex quinquefasciatus provenienti da aree continentali e insulari di tutto il mondo, in varie condizioni ambientali e di laboratorio. Nel complesso, i dati hanno rivelato un elemento plasmidico di Wolbachia notevolmente conservato nelle zanzare Culex , suggerendo un ruolo cruciale per questo elemento mobile nella biologia dell'endosimbionte15, giustificando ulteriori analisi.
Qui, forniamo ulteriori esempi di analisi del microbioma delle zanzare su campioni di intestino medio e ovaie ottenuti utilizzando questa procedura sistematica. Abbiamo osservato una chiara differenza nel microbiota tra i tessuti (Figura 5), sia con taxa batterici condivisi che organo-specifici. Come previsto, la presenza di Wolbachia è stata rilevata in entrambi gli organi, con una maggiore abbondanza relativa (basata su letture brevi non assemblate) e una copertura media di MAG nelle ovaie delle zanzare rispetto all'intestino medio, coerentemente con l'osservazione che questo endosimbionte viene trasmesso attraverso le ovaie e successivamente si diffonde ai tessuti somatici. Sebbene questo studio sia stato limitato a campioni provenienti dalla Nuova Caledonia, questo protocollo può facilitare lo studio della variabilità genomica di Wolbachia su scala globale, nonché del suo ruolo in vari fenotipi, tra cui la regolazione della densità di se stesso e la protezione virale. Inoltre, questo lavoro esemplifica come la procedura di dissezione qui presentata consenta l'esame della diversità tassonomica e delle potenziali capacità funzionali dei simbionti di zanzara all'interno di campioni di intestino medio.
Abbiamo ottenuto due genomi di bozza enterobatterica che erano presenti solo in due campioni di intestino medio, confermando l'assenza di contaminazione tra gli organi per questi due campioni. Per quanto riguarda le spirochete, rilevate sia nell'intestino medio che nelle ovaie dell'individuo 3, Juma e colleghi, 2020 hanno osservato la presenza di questi batteri sulla superficie delle zattere di uova. Gli autori hanno suggerito che le comunità batteriche trovate nelle zattere delle uova potrebbero essere ereditate principalmente dalla madre dalle ovaie, dato che le zattere sono state conservate in acqua deionizzata e priva di batteri. Tuttavia, non hanno potuto escludere la possibilità che la colonizzazione batterica si verifichi subito dopo l'ovideposizione e hanno raccomandato ulteriori studi sul microbioma ovarico16.
Sebbene inizialmente progettato per esemplari del complesso di specie Culex pipiens , prevediamo l'applicabilità di questo protocollo a una più ampia comunità di entomologi medici che studiano altri vettori come Anopheles o Aedes. Operando a livello di singoli organi, questo metodo può consentire confronti genomici sia intra che interindividuali, offrendo approfondimenti sulla variabilità genomica simbionte su scala fine, con il potenziale di far progredire le strategie di controllo dei vettori. Questo metodo di dissezione e isolamento delle ghiandole salivari, delle ovaie e dell'intestino medio, prevenendo la contaminazione crociata microbica, potrebbe anche essere un protocollo utile per gli studi sulla dinamica dell'infezione virale all'interno di questi tre organi.
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Ringraziamo Gilbert Legoff per aver insegnato a Jordan Tutagata come sezionare le ghiandole salivari dalle zanzare e Giuliano Mucci per l'aiuto con le foto degli organi delle zanzare. Ringraziamo Anna-Bella Failloux e Nonito Pages per l'utile discussione sul protocollo. Questo lavoro è stato sostenuto dal RosaLind Starting Grant "948135" dell'ERC a JR. Ringraziamo la piattaforma Vectopole (IRD, Montpellier) per il supporto tecnico e per l'allevamento e il mantenimento delle popolazioni di zanzare.
Materiali
| Name | Company | Catalog Number | Comments |
| Alcohol 96° | Fisher scientific | 10332562 | |
| Binocular magnifier | |||
| Bleach | RAJA | 145517 | 150 tablets of 1.5 g |
| DNA prep kit | Illumina | Provided by MGX sequencing platform | Previously known as Nextera DNA Flex |
| DNA-RNA Shield (50 mL) | Zymo research | ZR1100-50 | Preservation buffer |
| DNeasy Blood and Tissue Kit | Qiagen | 69504 | DNA extraction kit |
| Filter tips 20 µL | Starlab | S1120-3810 | |
| Filter tips 200 µL | Starlab | S1120-8710 | |
| Filter tips 1000 µL | Starlab | S1122-1730-C | |
| Forceps | FST (Fine Science Tools) | 11252-20 | Dumont Forceps #5 |
| Library quantification kit | Roche | Provided by MGX sequencing platform | KAPA Library Quanitification Kits |
| Micropipettes 2-20 µL | Eppendorf | 6.291704 | |
| Micropipettes 20-200 µL | Eppendorf | 6.291703 | |
| Micropipettes 100-1000 µL | Eppendorf | 7.648488 | |
| Microscope slides | Epredia | J1800BMNZ | dimension : 75 mm x 50 mm |
| Needles | Terumo | AN*2719R1 | |
| NGS kit | Agilent | Provided by MGX sequencing platform | Fragment Analyzer Systems HS Genomic DNA 50kb Kit |
| NovaSeq 6000 | Illumina | Provided by MGX sequencing platform | Sequencer |
| PBS Phosphate Buffered Saline (Sterile) | Fisher scientific | 10212990 | |
| Permanent black marker | |||
| Sterile Eppendorf | Dutscher | 33871 | 1.5 mL |
| Support for needles | FST (Fine Science Tools) | 26016-12 | Moria MC1 Pin Holder 12 cm |
Riferimenti
- Achee, N. L., et al. Alternative strategies for mosquito-borne arbovirus control. PLOS Negl Trop Dis. 13 (1), e0006822 (2019).
- Hoffmann, A. A., et al. Successful establishment of Wolbachia in Aedes populations to suppress dengue transmission. Nature. 476 (7361), 454-457 (2011).
- Porter, J., Sullivan, W. The cellular lives of Wolbachia. Nat Rev Microbiol. 21 (11), 750-766 (2023).
- Coon, K. L., Brown, M. R., Strand, M. R. Mosquitoes host communities of bacteria that are essential for development but vary greatly between local habitats. Mol Ecol. 25 (22), 5806-5826 (2016).
- Didion, E. M., et al. Microbiome reduction prevents lipid accumulation during early diapause in the northern house mosquito, Culex pipiens pipiens. J Insect Physiol. 134, 104295 (2021).
- Gabrieli, P., et al. Mosquito trilogy: Microbiota, immunity and pathogens, and their implications for the control of disease transmission. Front Microbiol. 12, 630438 (2021).
- Garrigós, M., Garrido, M., Panisse, G., Veiga, J., Martínez-de La Puente, J. Interactions between West Nile Virus and the microbiota of Culex pipiens vectors: A literature review. Pathogens. 12 (11), 1287 (2023).
- Zheng, R., et al. Holobiont perspectives on tripartite interactions among microbiota, mosquitoes, and pathogens. ISME J. 17 (8), 1143-1152 (2023).
- Flores, G. A. M., et al. Wolbachia dominance influences the Culex quinquefasciatus microbiota. Sci Rep. 13 (1), 18980 (2023).
- Hegde, S., et al. Interkingdom interactions shape the fungal microbiome of mosquitoes. Anim Microbiome. 6 (1), 11 (2024).
- Suo, P., et al. Seasonal variation of midgut bacterial diversity in Culex quinquefasciatus populations in Haikou city, Hainan province, China. Biology. 11 (8), 1166 (2022).
- Gruber-Vodicka, H. R., Seah, B. K. B., Pruesse, E. phyloFlash: Rapid small-subunit rRNA profiling and targeted assembly from metagenomes. mSystems. 5 (5), e00920-e00920 (2020).
- Graña-Miraglia, L., et al. Spirochetes isolated from arthropods constitute a novel genus Entomospira genus novum within the order Spirochaetales. Sci Rep. 10 (1), 17053 (2020).
- Reveillaud, J., et al. The Wolbachia mobilome in Culex pipiens includes a putative plasmid. Nat Commun. 10 (1), 1051 (2019).
- Ghousein, A., et al. pWCP is a widely distributed and highly conserved Wolbachia plasmid in Culex pipiens and Culex quinquefasciatus mosquitoes worldwide. ISME Commun. 3 (1), 40 (2023).
- Juma, E. O., Kim, C. -. H., Dunlap, C., Allan, B. F., Stone, C. M. Culex pipiens and Culex restuans egg rafts harbor diverse bacterial communities compared to their midgut tissues. Parasit Vectors. 13 (1), 532 (2020).
- Lee, M. D. GToTree: a user-friendly workflow for phylogenomics. Bioinformatics. 35 (20), 4162-4164 (2019).
- Parks, D. H., et al. A complete domain-to-species taxonomy for bacteria and Archaea. Nat Biotechnology. 38 (9), 1079-1086 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon