Method Article
Aprovechamiento de la realidad virtual para la segmentación y el análisis inmersivos de datos de tomografía crioelectrónica
* Estos autores han contribuido por igual
En este artículo
Resumen
La criotomografía electrónica (crio-ET) permite la visualización en 3D de la ultraestructura celular con una resolución nanométrica, pero la segmentación manual sigue siendo lenta y compleja. Presentamos un novedoso flujo de trabajo que integra un software avanzado de realidad virtual para la segmentación de tomografías crio-ET, mostrando su efectividad a través de la segmentación de mitocondrias en células de mamíferos.
Resumen
La criotomografía electrónica (crio-ET) es una poderosa técnica para visualizar la ultraestructura de las células en tres dimensiones (3D) con una resolución nanométrica. Sin embargo, la segmentación manual de los componentes celulares en los datos de crio-ET sigue siendo un cuello de botella importante debido a su complejidad y a su naturaleza lenta. En este trabajo, presentamos un novedoso flujo de trabajo de segmentación que integra software avanzado de realidad virtual (VR) para mejorar tanto la eficiencia como la precisión de la segmentación de conjuntos de datos de crio-ET. Este flujo de trabajo aprovecha una herramienta de realidad virtual inmersiva con interacción 3D intuitiva, lo que permite a los usuarios navegar y anotar estructuras celulares complejas en un entorno más natural e interactivo. Para evaluar la efectividad del flujo de trabajo, lo aplicamos a la segmentación de mitocondrias en células del epitelio pigmentario de la retina (RPE1). Las mitocondrias, esenciales para la producción de energía celular y la señalización, exhiben cambios morfológicos dinámicos, lo que las convierte en una muestra de prueba ideal. El software VR facilitó la delineación precisa de las membranas mitocondriales y las estructuras internas, lo que permitió el análisis posterior de las estructuras de membrana segmentadas. Demostramos que este flujo de trabajo de segmentación basado en realidad virtual mejora significativamente la experiencia del usuario al tiempo que mantiene una segmentación precisa de estructuras celulares complejas en datos crio-ET. Este enfoque es prometedor para amplias aplicaciones en biología celular estructural y educación científica, ya que ofrece una herramienta transformadora para los investigadores que se dedican al análisis celular detallado.
Introducción
La criotomografía electrónica (crio-ET) ha revolucionado nuestra capacidad para visualizar componentes celulares en su estado casi nativo a alta resolución 1,2. Esta poderosa técnica permite a los investigadores caracterizar la ultraestructura celular, proporcionando información sin precedentes sobre la arquitectura y la función celular. Sin embargo, la crio-ET no está exenta de limitaciones, la principal de ellas es el requisito de que las muestras sean lo suficientemente delgadas como para ser transparentes a los electrones (normalmente <0,5 μm) para la obtención de imágenes en microscopios electrónicos de transmisión con capacidad criogénica estándar. Los avances recientes en el molienda de haces de iones criofocalizados han permitido el adelgazamiento de muestras gruesas para el análisis de crio-ET 3,4.
Los flujos de trabajo típicos de crio-ET comienzan con la recolección de una serie de inclinación, donde la muestra se visualiza en varios ángulos, que van desde +60° a -60°. A continuación, estas imágenes se alinean computacionalmente y se retroproyectan para crear un volumen tridimensional (3D) o un tomograma 5,6,7. Este tomograma sirve como un mapa detallado en 3D del paisaje celular, ofreciendo información espacial y temporal sobre las estructuras celulares. Un mayor refinamiento a través del promedio de subtomograma, donde se alinean y promedian múltiples copias de la misma estructura, puede empujar los límites de resolución aún más, a veces logrando una resolución subnanométricade 7,8,9,10.
Un paso crucial en la extracción de información biológica significativa de estos tomogramas es la segmentación. Este proceso implica anotar estructuras celulares específicas, como membranas, dentro del volumen 3D. La segmentación permite realizar análisis avanzados, incluido el cálculo de las distancias entre membranas y la curvatura de las membranas, lo que proporciona información valiosa sobre los procesos celulares11,12. Si bien hay varios paquetes de software disponibles para esta tarea, incluidos Dragonfly, Amira, MemBrain, EMAN2 y tomomemsegtv 13,14,15,16,17, el proceso de segmentación sigue siendo un cuello de botella importante en el análisis de datos crio-ET. A menudo es un proceso manual que requiere mucho trabajo y tiempo, y puede tardar semanas o meses en completarse. Muchos de estos paquetes ofrecen funciones de segmentación automática, pero con frecuencia requieren una corrección manual extensa para eliminar falsos positivos, un proceso que puede ser laborioso y poco intuitivo cuando se realiza corte por corte o en 3D.
Proponemos un enfoque alternativo aprovechando la tecnología de realidad virtual (RV) para abordar estos desafíos. La realidad virtual ofrece un método inmersivo e interactivo para la visualización de datos, lo que permite a los usuarios navegar a través del volumen tomográfico como si estuviera dentro del propio entorno celular. Este enfoque también proporciona una plataforma valiosa para la educación científica y la exploración científica y el descubrimiento de datos crio-ET in situ al proporcionar una experiencia inmersiva y atractiva única. En este trabajo, presentamos un protocolo para la segmentación de datos crio-ET utilizando syGlass18, software de realidad virtual diseñado para la visualización científica. Este software proporciona un conjunto de herramientas completo para el análisis de datos de crio-ET, incluida la segmentación manual, el refinamiento de las segmentaciones generadas automáticamente e incluso la selección de partículas dentro de los tomogramas. Nuestro estudio demuestra la viabilidad de la RV como una poderosa herramienta para la limpieza de segmentaciones, la selección de partículas y la segmentación manual en el análisis de datos crio-ET.
Para ilustrar la utilidad del software de realidad virtual para la segmentación crio-ET, nos centramos en el análisis de la morfología mitocondrial en células del epitelio pigmentario de la retina (RPE1). Las mitocondrias sirven como un excelente caso de prueba para la segmentación debido a su compleja estructura y la presencia de características fácilmente cuantificables, como la distancia entre las membranas mitocondriales externas e internas. Estas características se pueden medir con precisión utilizando las herramientas de análisis de morfometría de superficie12, lo que proporciona métricas sólidas para evaluar la calidad de la segmentación. Este protocolo proporciona instrucciones paso a paso para segmentar datos de crio-ET utilizando syGlass, lo que demuestra su utilidad dentro de la canalización de segmentación de crio-ET. Al incorporar la segmentación basada en realidad virtual en el flujo de trabajo de crio-ET, nuestro objetivo es mejorar tanto la eficiencia como la precisión del análisis estructural manual en biología celular.
Protocolo
1. Preparación de datos de crio-ET para la segmentación
- Convierta tomografías Cryo-ET sin procesar a un formato de datos compatible con syGlass, como pilas TIFF. Configure la señal para que las partículas sean blancas sobre negro y realice la ecualización del histograma con ImageJ19.
NOTA: Para mejorar la relación señal-ruido, se recomienda que los tomogramas se eliminen con software como Warp20, Topaz Denoise21, cryoCARE22,23 o IsoNet24 antes de importarlos al software. - Inicie el software de realidad virtual en la computadora. Vaya al menú Archivo y seleccione Crear proyecto.
- Haga clic en Crear nuevo proyecto | Agregar archivos. Navegue hasta la ubicación donde se guardan los archivos TIFF e impórtelos en el software.
- Cuando se le pida que confirme si los archivos son una serie temporal, haga clic en No.
- Asigne un nombre al proyecto y, a continuación, haga clic en Guardar para crear un nuevo proyecto en la lista de proyectos.
- Haga doble clic en el proyecto para abrir el tomograma y cargarlo en el entorno de realidad virtual interactiva del software.
2. Configuración de la realidad virtual (RV)
- Conecte el casco de realidad virtual y los controladores de mano a la computadora.
- Siga las instrucciones en pantalla para calibrar el entorno de realidad virtual.
NOTA: La calibración se refiere al espacio tridimensional donde se realizará la segmentación. Los parámetros (longitud, anchura y altura) del espacio de trabajo deben definirse antes de que se muestre la proyección de realidad virtual. La proyección de realidad virtual solo será visible en estos parámetros. - La segmentación se realizará solo dentro del espacio tridimensional de trabajo definido. Asegúrese de que el campo de visión contenga el área en la que se desea trabajar en el entorno de realidad virtual.
3. Optimización de la visualización 3D
- Haga clic en el botón Visualización en la interfaz del software. Ajuste varias opciones de visualización, como el contraste, las ventanas, el brillo y los controles deslizantes de umbral para mejorar la señal y minimizar el ruido.
- Use los controladores manuales para acercar el tomograma o alejarlo según sea necesario.
- Active la herramienta Cortar usando el controlador de la izquierda para inspeccionar visualmente diferentes cortes dentro del tomograma.
4. Proceso de segmentación
- Navegue a través del tomograma hasta el corte deseado donde comenzará la segmentación.
- Active la opción Región de interés (ROI) en el menú de anotaciones utilizando los controladores manuales. Aparecerá un recuadro verde dentro del tomograma.
- Ajuste el tamaño y la posición del cuadro verde, moviéndolo al área a segmentar.
- Bloquee el retorno de la inversión con el controlador de la izquierda e inicie la segmentación. Después de bloquear el ROI, la herramienta cambia automáticamente a un modo de pintura que permite la segmentación de los datos volumétricos.
- Acérquese o aléjese del tomograma para una segmentación precisa.
- Ajuste el tamaño del pincel girándolo en el sentido de las agujas del reloj o en el sentido contrario a las agujas del reloj para un control óptimo.
- Segmente cuidadosamente el ROI (por ejemplo, membranas mitocondriales) dentro del área 3D seleccionada. Si se produce un error durante la segmentación, mantenga presionado el activador del controlador secundario para activar el modo de borrado y, a continuación, borre el error con el mismo movimiento que la segmentación.
- Repita este proceso región por región hasta que todo el tomograma esté segmentado.
5. Exportación de datos y análisis segmentados
- Después de completar la segmentación, seleccione el proyecto haciendo clic en él para asegurarse de que esté resaltado.
- Haz clic en la pestaña Proyectos en la esquina superior izquierda y, a continuación, selecciona ROI.
- Elija exportar todo el volumen o un ROI específico. Especifique la ubicación de exportación de los datos segmentados.
- Cargue y analice los datos segmentados utilizando el software de su elección y genere cifras para su publicación.
6. Importar máscara binaria en el software para la limpieza
- Realice los pasos 1.1 a 1.6.
- Haga clic con el botón derecho en el proyecto y haga clic en Agregar datos de máscara. A continuación, navegue hasta donde se guarda la segmentación inicial e impórtela en el mismo proyecto.
- Activar la anotación de ROI para realizar ediciones en la segmentación inicial.
- Agregar/borrar segmentación para limpiar la segmentación inicial.
- Ajuste el ROI de la máscara en la opción ROI de la pestaña máscaras . Para cambiar el ROI de la máscara de una a otra, active la única opción de sobrescribir máscara y vaya al modo de pintura con el controlador manual. Este modo solo sobrescribirá los datos de máscara que estén visibles.
- Exporte el ROI de la máscara ajustado siguiendo los pasos descritos en los pasos 5.1-5.3.
7. Selección de coordenadas de partículas usando el software VR
- Cree un proyecto utilizando un tomograma (pila TIFF) siguiendo los pasos descritos en la sección 1.
- Haga clic en Anotación | Recuento, que abre una ventana con opciones manuales o automatizadas .
- Para la opción manual , elija un color para los recuentos y comience a marcar las coordenadas.
- Para la opción automatizada, elija un tamaño de celda e ingrese la resolución. Haga clic en Ejecutar conteo automático, edite los recuentos (mantenga las buenas selecciones y descarte las malas) y guarde el progreso.
- Exporte las coordenadas de recuento haciendo clic en la pestaña proyecto | anotaciones.
- Para el origen de la anotación, elija Esquina inferior izquierda y para Unidades de anotación, elija Vóxeles.
- Haga clic en la celda de puntos de conteo para resaltar la celda.
- Haga clic en Exportar seleccionado para guardar las coordenadas de la selección en un archivo .csv.
NOTA: Este archivo se puede utilizar para extraer subtomogramas del tomograma para el promedio de subtomogramas
Resultados
En este estudio, segmentamos tomografías que contenían mitocondrias y orgánulos membranosos adicionales (p. ej., vesículas, retículo endoplásmico) utilizando syGlass. Los tomogramas se reconstruyeron inicialmente en Warp utilizando una proyección ponderada a 16,00 Å/píxel y se sometieron a la corrección de cuña faltante y a la eliminación de ruido utilizando el software IsoNet. Los siguientes tomogramas se sometieron a un procesamiento adicional para la importación, como se muestra en la Figura 1A (consulte también Video Suplementario S1, Video Suplementario S2, Video Suplementario S3 y Video Suplementario S4).
Después del preprocesamiento, los tomogramas en formato MRC se convirtieron en pilas TIFF utilizando ImageJ, con inversión de contraste aplicada para hacer que las membranas aparecieran blancas sobre un fondo negro. A continuación, se realizó la ecualización del histograma para mejorar aún más el contraste; Esto también permite un umbral más eficaz. Las pilas TIFF se importaron y visualizaron en 3D dentro de un entorno de realidad virtual inmersivo, proporcionando una inspección detallada de las estructuras membranosas utilizando la herramienta de corte en el software que se muestra en la Figura 1B.
La segmentación manual se realizó en el software, con ventanas configuradas en automático y ajustes de brillo/umbral para optimizar la visibilidad de las funciones celulares. Los ROI en torno a las mitocondrias y otras estructuras se definieron utilizando los controladores de realidad virtual. La herramienta de segmentación permitió una delineación precisa de los límites de la membrana, con errores corregidos mediante la función de borrado. La navegación corte por corte o en 3D utilizando la herramienta ROI para cuadrar regiones en todo el tomograma, combinada con la herramienta de pincel ajustable, garantizó una segmentación precisa de las membranas mitocondriales y otros orgánulos, como se muestra en la Figura 1C.
Los datos segmentados se visualizaron generando una malla utilizando la opción de superficies de la herramienta ROI, con iteraciones de suavizado establecidas en 12 y nivel de resolución establecido en 3. Las representaciones 3D finales muestran claramente las estructuras mitocondriales, incluidas las membranas externas e internas, las crestas y los depósitos de fosfato de calcio como se muestra en la Figura 1D (consulte también el Video Suplementario S5, el Video Suplementario S6, el Video Suplementario S7 y el Video Suplementario S8).
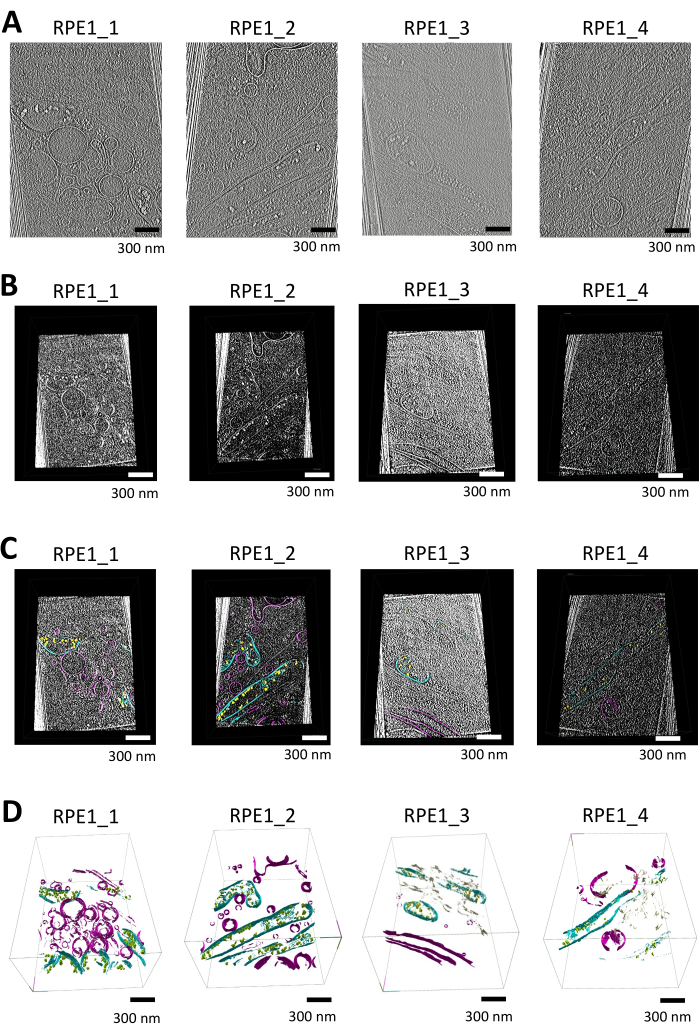
Figura 1: Flujo de trabajo de visualización, segmentación y renderizado 3D de tomografías con sirglass. (A) Cortes tomográficos de células RPE1 de borde delgado. Tomogramas corregidos por IsoNet reconstruidos en Warp y utilizados para la segmentación con syGlass, visualizados con IMOD25. Las tomografías se recogieron en un microscopio Titan-Krios de 300 keV equipado con un detector K3 y se centraron en los bordes finos de las células del epitelio de la retina. Cada tomografía contiene al menos una mitocondria junto con varios otros orgánulos unidos a la membrana. Estas imágenes son imágenes fijas de los vídeos que se muestran en el vídeo complementario S1, el vídeo complementario S2, el vídeo complementario S3 y el vídeo complementario S4. (B) Tomogramas correspondientes visualizados en el software. Corte tomográfico creado con la herramienta de corte en el software VR después de optimizar el umbral, el brillo y la ventana, revelando estructuras celulares distintas. (C) Tomogramas visualizados en el software con membranas segmentadas. Corte tomográfico creado con la herramienta de corte en el software de realidad virtual, superpuesto con la segmentación correspondiente. Los depósitos de fosfato de calcio se muestran en amarillo, las membranas mitocondriales en cian y otras membranas como vesículas y la membrana plasmática en púrpura. (D) Renderizado 3D de las membranas segmentadas en syGlass. Las membranas mitocondriales, incluidas las crestas, las membranas mitocondriales externas e internas, se muestran en cian. Los depósitos de fosfato de calcio son de color amarillo o verde, las vesículas y la membrana plasmática de color púrpura y el retículo endoplásmico de color canela. Estas imágenes son imágenes fijas de los vídeos que se muestran en Vídeos complementarios S5, Vídeo complementario S6, Vídeo complementario S7 y Vídeo complementario S8. Haga clic aquí para ver una versión más grande de esta figura.
Video Suplementario S1: Tomograma de la muestra RPE-1_1 utilizada para la segmentación en este estudio. Este tomograma se reconstruye a 16 Å/píxel con Warp, luego se elimina el ruido y se corrige la cuña faltante con IsoNet. Haga clic aquí para descargar este archivo.
Video suplementario S2: Tomograma de la muestra RPE-1_2 utilizada para la segmentación en este estudio. Este tomograma se reconstruye a 16 Å/píxel con Warp, luego se elimina el ruido y se corrige la cuña faltante con IsoNet. Haga clic aquí para descargar este archivo.
Video Suplementario S3: Tomograma de la muestra RPE-1_3 utilizada para la segmentación en este estudio. Este tomograma se reconstruye a 16 Å/píxel con Warp, luego se elimina el ruido y se corrige la cuña faltante con IsoNet. Haga clic aquí para descargar este archivo.
Video Suplementario S4: Tomograma de la muestra RPE-1_4 utilizada para la segmentación en este estudio. Este tomograma se reconstruye a 16 Å/píxel con Warp, luego se elimina el ruido y se corrige la cuña faltante con IsoNet. Haga clic aquí para descargar este archivo.
Vídeo complementario S5: La segmentación resultante de la muestra RPE-1_1 después de generar superficies. Las membranas mitocondriales se muestran en cian; Los fosfatos de calcio dentro de las mitocondrias se representan en amarillo; el retículo endoplásmico se muestra en color canela; y otras membranas se muestran en color púrpura. Haga clic aquí para descargar este archivo.
Vídeo complementario S6: La segmentación resultante de la muestra RPE-1_2 después de generar superficies. Las membranas mitocondriales se muestran en cian; Los fosfatos de calcio dentro de las mitocondrias se representan en amarillo; el retículo endoplásmico se muestra en color canela; y otras membranas se muestran en color púrpura. Haga clic aquí para descargar este archivo.
Vídeo complementario S7: La segmentación resultante de la muestra RPE-1_3 después de generar superficies. Las membranas mitocondriales se muestran en cian; Los fosfatos de calcio dentro de las mitocondrias se representan en amarillo; el retículo endoplásmico se muestra en color canela; y otras membranas se muestran en color púrpura. Haga clic aquí para descargar este archivo.
Vídeo complementario S8: La segmentación resultante de la muestra RPE-1_4 después de generar superficies. Las membranas mitocondriales se muestran en cian; Los fosfatos de calcio dentro de las mitocondrias se representan en amarillo; el retículo endoplásmico se muestra en color canela; y otras membranas se muestran en color púrpura. Haga clic aquí para descargar este archivo.
Discusión
En este trabajo, demostramos cómo la realidad virtual, específicamente usando syGlass, se puede integrar en la tubería crio-ET para segmentar de manera efectiva las estructuras celulares. Aunque nos centramos principalmente en las membranas, no existe ninguna limitación inherente que impida la segmentación de otras estructuras celulares, como los filamentos o los ribosomas, ya que se puede ajustar la forma y el tamaño del pincel, y el umbral para garantizar que sólo se marquen los vóxeles correspondientes a los objetos celulares deseados. Una de las principales ventajas de la realidad virtual en la segmentación es la interacción intuitiva y práctica con los datos, lo que permite a los usuarios visualizar y manipular volúmenes como si estuvieran físicamente dentro de ellos. Los métodos tradicionales de segmentación manual o limpieza de máscaras de segmentación inicial suelen implicar trabajar directamente en un equipo, que puede ser menos envolvente y más lento de anotar.
Al incorporar la realidad virtual en el flujo de trabajo de crio-ET, los usuarios no solo pueden interactuar rápidamente con las máscaras de segmentación generadas por otro software, sino también usar la realidad virtual para guiar la segmentación de estructuras parcialmente segmentadas y limpiar de manera eficiente los falsos positivos. En la actualidad, la segmentación manual sigue siendo necesaria para la mayoría de los casos de uso de anotaciones, y el flujo de trabajo presentado permite a los usuarios generar segmentaciones adecuadas para el análisis posterior con mayor facilidad y velocidad. Para este estudio utilizamos los auriculares HTC Vive VR, pero el software es compatible con dispositivos que tienen soporte para SteamVR u OculusVR.
Para una aplicación óptima de este protocolo, los datos de crio-ET deben cumplir criterios específicos, como tomografías de alta calidad con una alta relación señal-ruido (SNR). La SNR es un parámetro esencial, ya que la claridad de las características estructurales influye directamente en la eficacia de la segmentación manual en la RV. Los pasos de preprocesamiento, como la corrección de cuña faltante y la eliminación de ruido, son partes clave del flujo de trabajo; en este protocolo, utilizamos IsoNet para estos fines. Los tomogramas deben reconstruirse a una resolución adecuada, utilizando un tamaño de vóxel que proporcione suficientes detalles estructurales y suficiente separación entre las estructuras celulares para permitir una segmentación efectiva entre las estructuras, al tiempo que se mantienen tamaños de datos manejables para la visualización de realidad virtual. Además, se debe aplicar un contraste invertido con estructuras celulares que aparecen blancas sobre un fondo negro y una ecualización del histograma para mejorar la visibilidad de las membranas y otras estructuras dentro del entorno de syGlass.
Hay que tener en cuenta varios puntos críticos para garantizar el éxito de este protocolo. En primer lugar, durante el preprocesamiento de datos, es vital la aplicación precisa de la inversión de contraste y la ecualización del histograma; los ajustes incorrectos pueden dar lugar a una visualización subóptima en la realidad virtual, lo que dificulta la segmentación. En segundo lugar, dentro del software de realidad virtual, los ajustes adecuados de las ventanas, el brillo y la configuración del umbral son esenciales para una visualización óptima de las estructuras. Los usuarios deben experimentar con esta configuración para lograr los mejores resultados para sus conjuntos de datos específicos y configuraciones de realidad virtual.
Cuando se trata de solucionar problemas, los usuarios pueden encontrar problemas como limitaciones de rendimiento del sistema de realidad virtual, especialmente cuando se manejan grandes conjuntos de datos de tomografías. Si el entorno de realidad virtual se vuelve lento o no responde, considere reducir la resolución de los datos o segmentar el tomograma en secciones más pequeñas ajustando el cuadro delimitador en las herramientas de ROI en syGlass y moviéndolo a lo largo del tomograma a medida que se realiza la segmentación. El control deslizante de rendimiento en el menú de visualización también se puede ajustar para reducir el retraso durante el proceso de segmentación. Además, para mitigar el mareo por movimiento, los usuarios pueden ajustar la configuración de realidad virtual para reducir los efectos del movimiento o tomar descansos regulares durante las sesiones de segmentación.
Nuestro protocolo de segmentación basado en VR ofrece beneficios significativos para los flujos de trabajo actuales de segmentación crio-ET. Los métodos convencionales de segmentación manual a menudo implican anotaciones 2D en segmentos individuales, lo que puede llevar mucho tiempo y puede no capturar completamente la continuidad tridimensional13. Además, la segmentación basada en la realidad virtual introduce una vista mucho más inmersiva de las estructuras celulares, lo que ayuda a la visualización. Las segmentaciones automatizadas de aprendizaje automático están emergiendo como un método poderoso para obtener segmentaciones a partir de tomografías celulares 14,16,17, aunque los altos niveles de ruido y las estructuras complejas presentes en los datos de crio-ET conducen a brechas y falsos positivos que requieren intervención manual. Este protocolo ofrece un enfoque alternativo para segmentar manualmente los tomogramas crioelectrónicos, para generar segmentaciones iniciales que pueden usarse potencialmente como datos de entrenamiento para otro software de redes neuronales, o para limpiar las segmentaciones iniciales generadas a partir de otros enfoques automatizados.
En conclusión, este estudio destaca la segmentación basada en la realidad virtual como una herramienta prometedora para el análisis y la educación de datos crio-ET, que ofrece una mayor eficiencia y una experiencia de usuario más inmersiva. Con un mayor desarrollo, la tecnología de realidad virtual tiene el potencial de revolucionar la forma en que interpretamos y difundimos los descubrimientos científicos de estructuras celulares complejas en conjuntos de datos crio-ET, proporcionando una valiosa alternativa a los métodos tradicionales de segmentación y educación.
Divulgaciones
Los autores declaran que no tienen conflictos de intereses.
Agradecimientos
Este trabajo se realizó en el Centro Nacional de Acceso y Capacitación en CrioEM (NCCAT) y el Centro de Microscopía Electrónica Simons ubicado en el Centro de Biología Estructural de Nueva York, con el apoyo de los NIH (Fondo Común U24GM129539, U24GM139171 y NIGMS R24GM154192), la Fundación Simons (SF349247) y la Asamblea del Estado de Nueva York.
Materiales
| Name | Company | Catalog Number | Comments |
| CryoET Data | Format:TIFF-stack, TIFF | ||
| HTC VIVE Cosmos | HTC | 99HARL000-00 | https://www.vive.com/sea/product/vive-cosmos/features/ |
| Intel(R) Core(TM) i7-10870H CPU @ 2.20 GHz 2.21 GHz | Intel | https://ark.intel.com/content/www/us/en/ark/products/208018/intel-core-i7-10870h-processor-16m-cache-up-to-5-00-ghz.html | |
| NVIDIA GeForce RTX 3070 Laptop GPU | NVIDIA | https://www.nvidia.com/en-us/geforce/laptops/compare/30-series/ | |
| syGlass Software | syGlass | syGlass Software installed on a compatible Windows PC | |
| VIVE Cosmos Hand Controllers | HTC | 99HAFR001-00 | https://www.vive.com/us/accessory/cosmos-controller-right/ |
| Windows 11 Home | Microsoft | Microsoft Windows 11 Home |
Referencias
- Plitzko, J. M., Schuler, B., Selenko, P. Structural biology outside the box-inside the cell. Curr Opin Struct Biol. 46, 110-121 (2017).
- Beck, M., Baumeister, W. Cryo-electron tomography: can it reveal the molecular sociology of cells in atomic detail. Trends Cell Biol. 26 (11), 825-837 (2016).
- Lam, V., Villa, E. Practical approaches for cryo-FIB milling and applications for cellular cryo-electron tomography. Methods Mol Biol. 2215, 49-82 (2021).
- Berger, C., et al. Cryo-electron tomography on focused ion beam lamellae transforms structural cell biology. Nat Methods. 20 (4), 499-511 (2023).
- Watson, A. J. I., Bartesaghi, A. Advances in cryo-ET data processing: meeting the demands of visual proteomics. Curr Opin Struct Biol. 87, 102861 (2024).
- Pyle, E., Zanetti, G. Current data processing strategies for cryo-electron tomography and subtomogram averaging. Biochem J. 478 (10), 1827-1845 (2021).
- Wan, W., Briggs, J. A. G. Cryo-electron tomography and subtomogram averaging. Methods Enzymol. 579, 329-367 (2016).
- Ni, T., Liu, K., Zhang, J., Atanasov, I., Mladenov, M. G., Jiang, W. High-resolution in situ structure determination by cryo-electron tomography and subtomogram averaging using emClarity. Nat Protoc. 17 (2), 421-444 (2022).
- Obr, M., et al. Exploring high-resolution cryo-ET and subtomogram averaging capabilities of contemporary DEDs. J Struct Biol. 214 (2), 107852 (2021).
- Tegunov, D., Xue, L., Dienemann, C., Cramer, P., Mahamid, J. Multi-particle cryo-EM refinement with M visualizes ribosome-antibiotic complex at 3.5 Å in cells. Nat Methods. 18 (2), 186-193 (2021).
- Salfer, M., Collado, J. F., Baumeister, W., Fernández-Busnadiego, R., Martínez-Sánchez, A. Reliable estimation of membrane curvature for cryo-electron tomography. PLoS Comput Biol. 16 (8), e1007962 (2020).
- Barad, B. A., et al. Quantifying organellar ultrastructure in cryo-electron tomography using a surface morphometrics pipeline. J Cell Biol. 222 (2), e202204093 (2023).
- Martinez-Sanchez, A., Garcia, I., Asano, S., Lucic, V., Fernandez, J. J. Robust membrane detection based on tensor voting for electron tomography. J Struct Biol. 186 (1), 49-61 (2014).
- Heebner, J. E., et al. Deep learning-based segmentation of cryo-electron tomograms. J Vis Exp. (189), e64435 (2022).
- Garza-López, E., Robles-Flores, M. Protocols for generating surfaces and measuring 3D organelle morphology using Amira. Cells. 11 (1), 65 (2021).
- Chen, M., et al. Convolutional neural networks for automated annotation of cellular cryo-electron tomograms. Nat Methods. 14 (10), 983-985 (2017).
- Lamm, L., et al. MemBrain v2: an end-to-end tool for the analysis of membranes in cryo-electron tomography. bioRxiv. , (2024).
- Pidhorskyi, S., Morehead, M., Jones, Q., Spirou, G., Doretto, G. syGlass: interactive exploration of multidimensional images using virtual reality head-mounted displays. arXiv. , (2018).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Tegunov, D., Cramer, P. Real-time cryo-electron microscopy data preprocessing with Warp. Nat Methods. 16 (11), 1146-1152 (2019).
- Bepler, T., Kelley, K., Noble, A. J., Berger, B. Topaz-Denoise: general deep denoising models for cryoEM and cryoET. Nat Commun. 11, 5208 (2020).
- Buchholz, T. -. O., Berninger, L., Maurer, M., Jug, F. Cryo-CARE: content-aware image restoration for cryo-transmission electron microscopy data. Proc IEEE Int Symp Biomed Imaging. 16, 502-506 (2019).
- Buchholz, T. -. O., Jordan, M., Pigino, G., Jug, F. Content-aware image restoration for electron microscopy. Methods Cell Biol. 152, 277-289 (2019).
- Liu, Y. -. T., et al. Isotropic reconstruction for electron tomography with deep learning. Nat Commun. 13, 6482 (2022).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. J Struct Biol. 116 (1), 71-76 (1996).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados