Method Article
バーチャルリアリティを活用したクライオ電子線トモグラフィーデータの没入型セグメンテーションと解析
要約
クライオ電子線トモグラフィー(クライオET)は、ナノメートルの分解能で細胞の超微細構造を3D可視化することを可能にしますが、手作業によるセグメンテーションは依然として時間がかかり、複雑です。クライオET断層撮影のための高度なバーチャルリアリティソフトウェアを統合した新しいワークフローを提示し、哺乳類細胞のミトコンドリアのセグメンテーションを通じてその有効性を示します。
要約
クライオ電子線トモグラフィー(クライオET)は、細胞の超微細構造をナノメートルの分解能で3次元(3D)に視覚化するための強力な技術です。しかし、クライオETデータにおける細胞成分の手動セグメンテーションは、その複雑さと時間のかかる性質のために、依然として大きなボトルネックとなっています。この研究では、高度な仮想現実(VR)ソフトウェアを統合して、クライオETデータセットのセグメンテーションの効率と精度の両方を向上させる新しいセグメンテーションワークフローを紹介します。このワークフローは、直感的な3Dインタラクションを備えた没入型VRツールを活用しており、ユーザーはより自然でインタラクティブな環境で複雑な細胞構造をナビゲートし、注釈を付けることができます。ワークフローの有効性を評価するために、網膜色素上皮(RPE1)細胞のミトコンドリアのセグメンテーションに適用しました。細胞のエネルギー産生とシグナル伝達に不可欠なミトコンドリアは、動的な形態学的変化を示すため、理想的なテストサンプルとなっています。このVRソフトウェアは、ミトコンドリア膜と内部構造の正確な描写を容易にし、セグメント化された膜構造の下流分析を可能にしました。このVRベースのセグメンテーションワークフローにより、クライオETデータの複雑な細胞構造の正確なセグメンテーションを維持しながら、ユーザーエクスペリエンスが大幅に向上することを示しています。このアプローチは、構造細胞生物学や科学教育への幅広い応用が期待されており、詳細な細胞解析に従事する研究者に変革的なツールを提供します。
概要
クライオ電子線トモグラフィー(クライオET)は、細胞成分をほぼ天然の状態の高解像度1,2で視覚化する能力に革命をもたらしました。この強力な技術により、研究者は細胞の超微細構造を特徴付け、細胞の構造と機能に関する前例のない洞察を得ることができます。しかし、クライオETには限界がないわけではなく、その中でも最も重要なのは、標準的なクライオ対応透過型電子顕微鏡でのイメージングには、サンプルが電子透明(通常は<0.5μm)であるほど薄いという要件です。クライオ集束イオンビームミリングの最近の進歩により、クライオET分析のための厚いサンプルの薄化が可能になりました3,4。
一般的なクライオETワークフローは、サンプルが+60°から-60°の範囲のさまざまな角度でイメージングされるチルトシリーズの収集から始まります。次に、これらの画像は計算によって位置合わせされ、後方投影されて、3次元(3D)ボリュームまたは断層像5,6,7が作成されます。この断層撮影は、細胞の風景の詳細な3Dマップとして機能し、細胞構造に関する空間的および時間的情報を提供します。サブトモグラム平均化によるさらなる改良は、同じ構造の複数のコピーを整列させ、平均化することで、分解能の限界をさらに押し上げ、時にはサブナノメートルの分解能7,8,9,10を達成することもあります。
これらの断層撮影から意味のある生物学的情報を抽出するための重要なステップは、セグメンテーションです。このプロセスでは、3Dボリューム内の特定の細胞構造(膜など)にアノテーションを付与します。セグメンテーションは、膜間距離や膜曲率の計算などの高度な分析を可能にし、細胞プロセスに関する貴重な洞察を提供します11,12。このタスクには、Dragonfly、Amira、MemBrain、EMAN2、tomomemsegtv13、14、15、16、17など、いくつかのソフトウェアパッケージが利用可能ですが、クライオETデータ解析では、セグメンテーションプロセスが依然として大きなボトルネックとなっています。多くの場合、これは労働集約的で時間のかかる手動プロセスであり、完了するまでに数週間から数か月かかる可能性があります。これらのパッケージの多くは自動セグメンテーション機能を提供しますが、誤検出を排除するために大規模な手動修正が必要になることが多く、このプロセスはスライスごとまたは3Dで実行すると面倒で直感的ではありません。
これらの課題に対処するために、仮想現実(VR)技術を活用する別のアプローチを提案します。VRは、データ視覚化のための没入型でインタラクティブな方法を提供し、ユーザーは細胞環境自体の中にいるかのように断層撮影ボリュームをナビゲートできます。このアプローチは、没入型でユニークで魅力的な体験を提供することにより、科学教育や in situ クライオETデータの科学的探索と発見のための貴重なプラットフォームも提供します。この研究では、科学的な視覚化用に設計されたVRソフトウェアであるsyGlass18を使用したクライオETデータセグメンテーションのプロトコルを紹介します。このソフトウェアは、手動セグメンテーション、自動生成されたセグメンテーションの改良、さらには断層撮影内の粒子ピッキングなど、クライオETデータ分析のための包括的なツールキットを提供します。私たちの研究は、クライオETデータ分析におけるセグメンテーションのクリーニング、粒子ピッキング、および手動セグメンテーションの強力なツールとしてのVRの実行可能性を示しています。
クライオETセグメンテーションにおけるVRソフトウェアの有用性を説明するために、網膜色素上皮(RPE1)細胞のミトコンドリア形態の解析に焦点を当てています。ミトコンドリアは、その複雑な構造と、ミトコンドリアの外膜と内膜の間の距離など、容易に定量化できる特徴が存在するため、セグメンテーションの優れたテストケースとして機能します。これらの特徴は、表面形態測定分析ツール12を使用して正確に測定でき、セグメンテーションの品質を評価するための堅牢な指標を提供します。このプロトコルは、syGlassを使用してクライオETデータをセグメンテーションするための段階的な手順を提供し、クライオETセグメンテーションパイプライン内での有用性を実証しています。VRベースのセグメンテーションをクライオETワークフローに組み込むことで、細胞生物学における手動構造解析の効率と精度の両方を向上させることを目指しています。
プロトコル
1. セグメンテーションのためのクライオETデータの準備
- 生のCryo-ET断層撮影を、TIFFスタックなどのsyGlassと互換性のあるデータ形式に変換します。粒子が黒地に白になるように信号を設定し、ImageJ19でヒストグラムの均等化を行います。
注:信号対雑音比を向上させるために、ソフトウェアにインポートする前に、Warp20、Topaz Denoise21、cryoCARE22,23、IsoNet24などのソフトウェアで断層撮影のノイズを除去することをお勧めします。 - コンピューターでVRソフトウェアを起動します。 [ファイル ] メニューに移動し、[ プロジェクトの作成] を選択します。
- [ Create New Project (新しいプロジェクトを作成)] |ファイルを追加します。TIFFファイルが保存されている場所に移動し、ソフトウェアにインポートします。
- ファイルが時系列であるかどうかを確認するメッセージが表示されたら、[ いいえ] をクリックします。
- プロジェクトに名前を付け、「 保存 」をクリックして、プロジェクト・リストの下に新しいプロジェクトを作成します。
- プロジェクトをダブルクリックして断層撮影を開き、ソフトウェアのインタラクティブバーチャルリアリティ環境にロードします。
2. バーチャルリアリティ(VR)の設定
- VRヘッドセットとハンドコントローラーをコンピューターに接続します。
- 画面の指示に従って、VR環境を調整します。
注:キャリブレーションとは、セグメンテーションが行われる3次元空間を指します。作業スペースのパラメータ(長さ、幅、高さ)は、VRプロジェクションを表示する前に定義する必要があります。VR プロジェクションは、これらのパラメーターでのみ表示されます。 - セグメンテーションは、定義された作業 3 次元空間内でのみ行われます。視野に、VR環境で作業したい領域が含まれていることを確認します。
3. 3Dビジュアライゼーションの最適化
- ソフトウェアインターフェースの [視覚化 ]ボタンをクリックします。コントラスト、ウィンドウ処理、明るさ、しきい値スライダーなどのさまざまな視覚化オプションを調整して、信号を強化し、ノイズを最小限に抑えます。
- ハンドコントローラーを使用して、必要に応じて断層撮影を近づけたり押したりします。
- 左側のコントローラーを使用して カット ツールをアクティブにし、断層撮影内のさまざまなスライスを視覚的に検査します。
4. セグメンテーションプロセス
- 断層撮影をナビゲートして、セグメンテーションが始まる目的のスライスに移動します。
- ハンドコントローラーを使用して、注釈メニューの下にある関心領域(ROI)オプションをアクティブにします。断層撮影内に緑色のボックスが表示されます。
- 緑色のボックスのサイズと位置を調整し、セグメント化する領域に移動します。
- 左側のコントローラーを使用して ROI をロックし、セグメンテーションを開始します。ROIをロックすると、ツールは自動的に ペイント モードに切り替わり、ボリュームデータのセグメンテーションが可能になります。
- 断層撮影をズームインまたはズームアウトして、正確なセグメンテーションを実現します。
- ペイントブラシのサイズを時計回りまたは反時計回りに回転させて調整すると、最適な制御が可能になります。
- 選択した3D領域内のROI(ミトコンドリア膜など)を慎重にセグメント化します。セグメンテーション中にエラーが発生した場合は、セカンダリコントローラーのトリガーを押したまま 消去 モードにしてから、セグメンテーションと同じモーションでエラーを消去します。
- このプロセスを領域ごとに繰り返して、トモグラム全体がセグメント化されるまで繰り返します。
5. セグメント化されたデータのエクスポートと分析
- セグメンテーションが完了したら、プロジェクトをクリックして選択し、ハイライトされていることを確認します。
- 左上隅の 「プロジェクト 」タブをクリックし、「 ROI」を選択します。
- ボリューム全体をエクスポートするか、特定のROIをエクスポートするかを選択します。セグメント化されたデータのエクスポート場所を指定します。
- 選択したソフトウェアを使用してセグメント化されたデータをロードして分析し、公開用の図を生成します。
6.クリーンアップのためにバイナリマスクをソフトウェアにインポートする
- 手順1.1〜1.6を実行します。
- プロジェクトを右クリックし、[ マスク データの追加] をクリックします。次に、最初のセグメンテーションが保存されている場所に移動し、同じプロジェクトの下にインポートします。
- ROI アノテーションを使用して、初期セグメンテーションを編集します。
- セグメンテーションの追加/消去は、初期セグメンテーションをクリーンアップします。
- [マスク] タブの [ROI] オプションで [ROI] を調整します。マスクROIを一方から他方に変更するには、マスクの上書きのみオプションをオンにして、ハンドコントローラーでペイントモードに移動します。このモードは、表示されているマスクデータのみを上書きします。
- 調整されたマスク ROI を、手順 5.1 から 5.3 で説明されている手順を使用してエクスポートします。
7. VRソフトウェアを使用してパーティクル座標をピッキングする
- セクション1で説明されている手順を使用して、断層撮影(TIFFスタック)を使用してプロジェクトを作成します。
- [注釈] |[カウント] は、手動または自動のオプションを含むウィンドウを開きます。
- 手動オプションの場合は、カウントの色を選択し、座標のマーキングを開始します。
- 自動オプションの場合は、セル サイズを選択し、 解像度を入力します。[ 自動カウントの実行]をクリックし、カウントを編集し(良いピックを保持し、悪いピックを破棄します)、進行状況を保存します。
- カウント座標をエクスポートするには、 プロジェクト タブ の「注釈」をクリックします。
- アノテーションの原点として [左下コーナー ] を選択し、[ アノテーション単位] で [ボクセル] を選択します。
- カウントポイントセルをクリックして、セルを強調表示します。
- [選択項目のエクスポート] をクリックして、選択した項目の座標を .csv ファイルに保存します。
注:このファイルを使用して、サブトモグラムの平均化のためにトモグラムからサブトモグラムを抽出できます
結果
この研究では、syGlassを使用して、ミトコンドリアと追加の膜性オルガネラ(小胞、小胞体など)を含む断層撮影をセグメント化しました。断層撮影は、最初に 16.00 Å/ピクセルのウェイトバック投影を使用して Warp で再構築され、ソフトウェアである IsoNet を使用して欠落ウェッジ補正とノイズ除去が行われました。以下の断層撮影は、 図1A に示すように、インポートのための追加処理を行った( 補足ビデオS1、 補足ビデオS2、 補足ビデオS3、および 補足ビデオS4も参照)。
前処理の後、MRC形式の断層撮影をImageJを使用してTIFFスタックに変換し、コントラスト反転を適用して、黒い背景に膜が白く見えるようにしました。次に、コントラストをさらに強化するためにヒストグラムの均等化を実行しました。これにより、より効果的なしきい値設定も可能になります。TIFFスタックは、没入型VR環境内で3Dでインポートおよび視覚化され、 図1Bに示すソフトウェアのカットツールを使用して膜構造の詳細な検査を提供しました。
ソフトウェアでは手動セグメンテーションが行われ、ウィンドウ処理は自動に設定され、輝度/しきい値の調整はセルラー機能の可視性を最適化するために行われました。ミトコンドリアやその他の構造の周りのROIは、VRコントローラーを使用して定義しました。セグメンテーションツールにより、膜境界の正確な描写が可能になり、消去機能を使用してエラーが修正されました。スライスごとのナビゲーションまたは ROI ツールを使用して3Dで断層撮影全体の領域をボックス化し、調整可能な ペイントブラシ ツールと組み合わせることで、 図1Cに示すように、ミトコンドリア膜と他の細胞小器官の正確なセグメンテーションが保証されました。
セグメント化されたデータは、ROI ツールの surfaces オプションを使用してメッシュを生成し、スムージング反復回数を 12 に、解像度レベルを 3 に設定して視覚化しました。最終的な3Dレンダリングでは、 図1D に示すように、外膜と内膜、クリステ、リン酸カルシウム沈着物などのミトコンドリア構造が明確に示されています( 補足ビデオS5、 補足ビデオS6、 補足ビデオS7、および 補足ビデオS8も参照してください)。
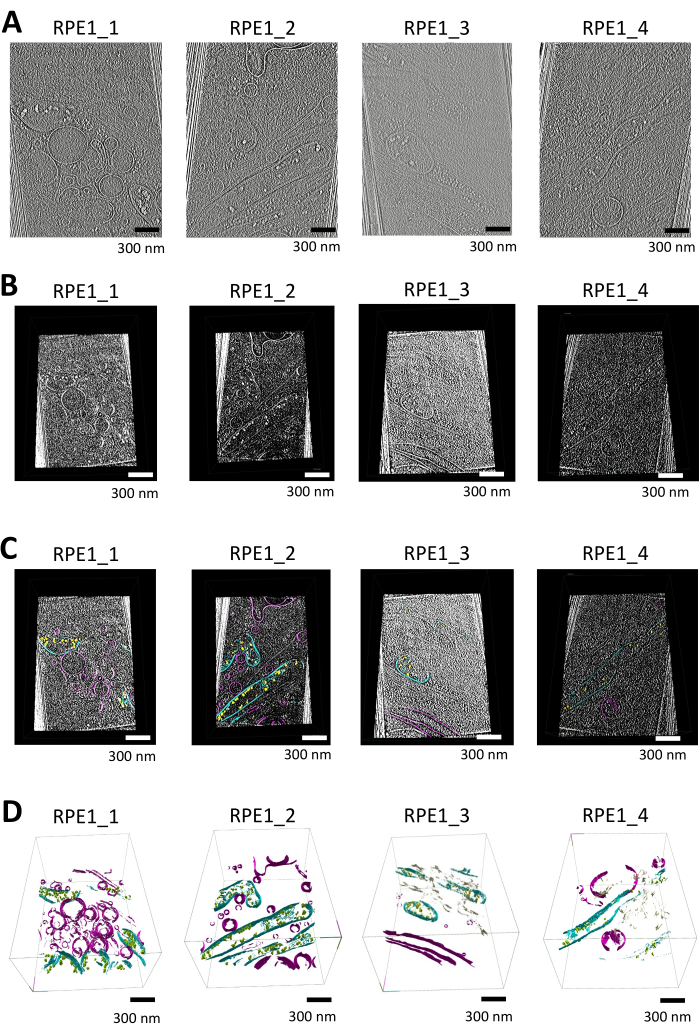
図1:シグラスを使用した断層撮影の視覚化、セグメンテーション、および3Dレンダリングのワークフロー(A)薄いエッジRPE1細胞の断層撮影スライス。IsoNet補正断層撮影をWarpで再構築し、syGlassによるセグメンテーションに使用し、IMOD25で可視化しました。断層撮影は、K3検出器を備えた300 keVのTitan-Krios顕微鏡で収集され、網膜上皮細胞の薄いエッジに焦点を合わせました。各断層図には、少なくとも1つのミトコンドリアと、他のさまざまな膜結合オルガネラが含まれています。これらの画像は、サプリメンタルビデオS1、サプリメンタルビデオS2、サプリメンタルビデオS3、およびサプリメンタルビデオS4に表示されているビデオの静止画です。(B)ソフトウェアで視覚化された対応する断層撮影。VRソフトウェアのカットツールを使用して、しきい値、輝度、ウィンドウ化を最適化した後、断層スライスを作成し、明確な細胞構造を明らかにします。(C)セグメント化された膜を使用してソフトウェアで視覚化された断層撮影。VRソフトウェアのカットツールで作成された断層撮影スライスに、対応するセグメンテーションが重ねられています。リン酸カルシウムの沈着物は黄色、ミトコンドリア膜はシアン、小胞や原形質膜などの他の膜は紫色で示されています。(D)syGlassのセグメント化された膜の3Dレンダリング。ミトコンドリア膜(クリステ、外側、内側のミトコンドリア膜を含む)はシアンで示されています。リン酸カルシウム沈着物は黄色または緑色、小胞体と原形質膜は紫色、小胞体は黄褐色です。これらの画像は、補足動画S5、補足動画S6、補足動画S7、補足動画S8に表示されている動画の静止画です。この図の拡大版を表示するには、ここをクリックしてください。
補足ビデオ S1: この研究でセグメンテーションに使用したサンプル RPE-1_1 の断層撮影。 この断層撮影は、ワープを使用して16 Å/ピクセルで再構築され、ノイズ除去され、欠落しているウェッジがIsoNetで補正されます。 このファイルをダウンロードするには、ここをクリックしてください。
補足ビデオ S2: この研究でセグメンテーションに使用したサンプル RPE-1_2 の断層撮影。 この断層撮影は、ワープを使用して16 Å/ピクセルで再構築され、ノイズ除去され、欠落しているウェッジがIsoNetで補正されます。 このファイルをダウンロードするには、ここをクリックしてください。
補足ビデオ S3: この研究でセグメンテーションに使用したサンプル RPE-1_3 の断層撮影。 この断層撮影は、ワープを使用して16 Å/ピクセルで再構築され、ノイズ除去され、欠落しているウェッジがIsoNetで補正されます。 このファイルをダウンロードするには、ここをクリックしてください。
補足ビデオ S4: この研究でセグメンテーションに使用したサンプル RPE-1_4 の断層撮影。 この断層撮影は、ワープを使用して16 Å/ピクセルで再構築され、ノイズ除去され、欠落しているウェッジがIsoNetで補正されます。 このファイルをダウンロードするには、ここをクリックしてください。
補足ビデオ S5: サーフェス生成後のサンプル RPE-1_1 のセグメンテーション結果。 ミトコンドリア膜はシアンで示されています。ミトコンドリア内のリン酸カルシウムは黄色で示されています。小胞体は黄褐色で示されています。その他の膜は紫色で表示されます。 このファイルをダウンロードするには、ここをクリックしてください。
補足ビデオ S6: サーフェス生成後のサンプル RPE-1_2 のセグメンテーション結果。 ミトコンドリア膜はシアンで示されています。ミトコンドリア内のリン酸カルシウムは黄色で示されています。小胞体は黄褐色で示されています。その他の膜は紫色で表示されます。 このファイルをダウンロードするには、ここをクリックしてください。
補足ビデオ S7: サーフェス生成後のサンプル RPE-1_3 のセグメンテーション結果。 ミトコンドリア膜はシアンで示されています。ミトコンドリア内のリン酸カルシウムは黄色で示されています。小胞体は黄褐色で示されています。その他の膜は紫色で表示されます。 このファイルをダウンロードするには、ここをクリックしてください。
補足ビデオ S8: サーフェス生成後のサンプル RPE-1_4 のセグメンテーション結果。 ミトコンドリア膜はシアンで示されています。ミトコンドリア内のリン酸カルシウムは黄色で示されています。小胞体は黄褐色で示されています。その他の膜は紫色で表示されます。 このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
この研究では、特にsyGlassを使用したVRをクライオETパイプラインに統合して、細胞構造を効果的にセグメント化する方法を示しました。私たちは主にメンブレンに焦点を当てていましたが、ブラシの形状とサイズを調整できるため、フィラメントやリボソームなどの他の細胞構造のセグメンテーションを妨げる固有の制限はなく、目的の細胞オブジェクトに対応するボクセルのみがマークされるように閾値化できます。セグメンテーションにおける VR の主な利点の 1 つは、直感的で実践的なデータ操作により、ユーザーは物理的にボリュームの中にいるかのように視覚化および操作できることです。手動セグメンテーションや初期セグメンテーションマスクのクリーニングを行う従来の方法では、通常、コンピュータで直接作業するため、没入感が低く、注釈を付けるのに時間がかかる可能性があります。
クライオETのワークフローにVRを組み込むことで、ユーザーは他のソフトウェアで生成されたセグメンテーションマスクと迅速に対話できるだけでなく、VRを使用して部分的にセグメント化された構造のセグメンテーションをガイドし、誤検知を効率的にクリーンアップすることができます。現在、ほとんどのアノテーションのユースケースでは手動セグメンテーションが依然として必要であり、提示されたワークフローにより、ユーザーはダウンストリーム分析に適したセグメンテーションを簡単かつ迅速に生成できます。この調査ではHTC Vive VRヘッドセットを使用しましたが、このソフトウェアはSteamVRまたはOculusVRをサポートするデバイスと互換性があります。
このプロトコルを最適に適用するには、クライオETデータは、高い信号対雑音比(SNR)を備えた高品質の断層撮影など、特定の基準を満たす必要があります。SNRは重要なパラメータであり、構造的特徴の明瞭さはVRの手動セグメンテーションの有効性に直接影響します。欠落しているウェッジの修正やノイズ除去などの前処理ステップは、ワークフローの重要な部分です。このプロトコルでは、これらの目的でIsoNetを利用しました。断層撮影は、適切な解像度で再構築する必要があります -- 十分な構造の詳細と細胞構造間の十分な分離を提供するボクセルサイズを使用して、構造間の効果的なセグメンテーションを可能にしながら、VR視覚化のための管理可能なデータサイズをさらに維持します。さらに、syGlass環境内の膜やその他の構造の視認性を高めるために、黒い背景に白く見える細胞構造との反転コントラストとヒストグラムの均等化を適用する必要があります。
このプロトコルを確実に成功させるためには、いくつかの重要なポイントを考慮する必要があります。まず、データの前処理では、コントラスト反転とヒストグラムの均等化を正確に適用することが重要です。不適切な調整は、VRでの視覚化が最適でなく、セグメンテーションを困難にする可能性があります。次に、VRソフトウェア内では、構造を最適に視覚化するために、ウィンドウ処理、明るさ、およびしきい値設定の適切な調整が不可欠です。ユーザーは、特定のデータセットと VR セットアップで最良の結果を得るために、これらの設定を試す必要があります。
トラブルシューティングに関しては、特に大規模な断層撮影データセットを処理する際に、VRシステムのパフォーマンス制限などの問題に遭遇する可能性があります。VR 環境が遅延したり、応答しなくなったりした場合は、syGlass の ROI ツールでバウンディングボックスを調整し、セグメンテーションの実行中にトモグラムに沿って移動することで、データをダウンサンプリングするか、トモグラムを小さなセクションにセグメント化することを検討してください。ビジュアライゼーション メニューのパフォーマンス スライダーを調整して、セグメンテーション プロセス中の遅延を減らすこともできます。さらに、乗り物酔いを軽減するために、ユーザーはVR設定を調整してモーション効果を減らしたり、セグメンテーションセッション中に定期的に休憩を取ったりすることができます。
当社のVRベースのセグメンテーションプロトコルは、現在のクライオETセグメンテーションワークフローに大きなメリットをもたらします。従来の手動セグメンテーション法は、個々のスライスに2Dアノテーションを施すことが多く、これには時間がかかり、3次元の連続性を完全に捉えることができない場合がある13。さらに、VRベースのセグメンテーションにより、細胞構造の没入感が大幅に向上し、視覚化に役立ちます。自動機械学習セグメンテーションは、細胞断層画像14、16、17からセグメンテーションを取得するための強力な方法として浮上していますが、クライオETデータに存在する高いノイズレベルと複雑な構造は、手動介入を必要とするギャップと偽陽性につながります。このプロトコルは、クライオ電子断層撮影を手動でセグメンテーションしたり、他のニューラルネットワークソフトウェアのトレーニングデータとして使用できる可能性のある初期セグメンテーションを生成したり、他の自動化されたアプローチから生成された初期セグメンテーションをクリーンアップしたりするための代替アプローチを提供します。
結論として、この研究では、VRベースのセグメンテーションがクライオETデータ分析と教育の有望なツールとして強調されており、効率の向上とより没入感のあるユーザーエクスペリエンスを提供します。さらなる開発により、VRテクノロジーは、クライオETデータセットの複雑な細胞構造の科学的発見を解釈し、広める方法に革命をもたらす可能性を秘めており、従来のセグメンテーションや教育方法に代わる貴重な方法を提供します。
開示事項
著者は、利益相反がないことを宣言します。
謝辞
この研究は、NIH(Common Fund U24GM129539、U24GM139171、NIGMS R24GM154192)、Simons Foundation(SF349247)、およびNY州議会の支援を受けて、National Center for CryoEM Access and Training(NCCAT)およびNew York Structural Biology CenterにあるSimons Electron Microscopy Centerで行われました。
資料
| Name | Company | Catalog Number | Comments |
| CryoET Data | Format:TIFF-stack, TIFF | ||
| HTC VIVE Cosmos | HTC | 99HARL000-00 | https://www.vive.com/sea/product/vive-cosmos/features/ |
| Intel(R) Core(TM) i7-10870H CPU @ 2.20 GHz 2.21 GHz | Intel | https://ark.intel.com/content/www/us/en/ark/products/208018/intel-core-i7-10870h-processor-16m-cache-up-to-5-00-ghz.html | |
| NVIDIA GeForce RTX 3070 Laptop GPU | NVIDIA | https://www.nvidia.com/en-us/geforce/laptops/compare/30-series/ | |
| syGlass Software | syGlass | syGlass Software installed on a compatible Windows PC | |
| VIVE Cosmos Hand Controllers | HTC | 99HAFR001-00 | https://www.vive.com/us/accessory/cosmos-controller-right/ |
| Windows 11 Home | Microsoft | Microsoft Windows 11 Home |
参考文献
- Plitzko, J. M., Schuler, B., Selenko, P. Structural biology outside the box-inside the cell. Curr Opin Struct Biol. 46, 110-121 (2017).
- Beck, M., Baumeister, W. Cryo-electron tomography: can it reveal the molecular sociology of cells in atomic detail. Trends Cell Biol. 26 (11), 825-837 (2016).
- Lam, V., Villa, E. Practical approaches for cryo-FIB milling and applications for cellular cryo-electron tomography. Methods Mol Biol. 2215, 49-82 (2021).
- Berger, C., et al. Cryo-electron tomography on focused ion beam lamellae transforms structural cell biology. Nat Methods. 20 (4), 499-511 (2023).
- Watson, A. J. I., Bartesaghi, A. Advances in cryo-ET data processing: meeting the demands of visual proteomics. Curr Opin Struct Biol. 87, 102861 (2024).
- Pyle, E., Zanetti, G. Current data processing strategies for cryo-electron tomography and subtomogram averaging. Biochem J. 478 (10), 1827-1845 (2021).
- Wan, W., Briggs, J. A. G. Cryo-electron tomography and subtomogram averaging. Methods Enzymol. 579, 329-367 (2016).
- Ni, T., Liu, K., Zhang, J., Atanasov, I., Mladenov, M. G., Jiang, W. High-resolution in situ structure determination by cryo-electron tomography and subtomogram averaging using emClarity. Nat Protoc. 17 (2), 421-444 (2022).
- Obr, M., et al. Exploring high-resolution cryo-ET and subtomogram averaging capabilities of contemporary DEDs. J Struct Biol. 214 (2), 107852 (2021).
- Tegunov, D., Xue, L., Dienemann, C., Cramer, P., Mahamid, J. Multi-particle cryo-EM refinement with M visualizes ribosome-antibiotic complex at 3.5 Å in cells. Nat Methods. 18 (2), 186-193 (2021).
- Salfer, M., Collado, J. F., Baumeister, W., Fernández-Busnadiego, R., Martínez-Sánchez, A. Reliable estimation of membrane curvature for cryo-electron tomography. PLoS Comput Biol. 16 (8), e1007962 (2020).
- Barad, B. A., et al. Quantifying organellar ultrastructure in cryo-electron tomography using a surface morphometrics pipeline. J Cell Biol. 222 (2), e202204093 (2023).
- Martinez-Sanchez, A., Garcia, I., Asano, S., Lucic, V., Fernandez, J. J. Robust membrane detection based on tensor voting for electron tomography. J Struct Biol. 186 (1), 49-61 (2014).
- Heebner, J. E., et al. Deep learning-based segmentation of cryo-electron tomograms. J Vis Exp. (189), e64435 (2022).
- Garza-López, E., Robles-Flores, M. Protocols for generating surfaces and measuring 3D organelle morphology using Amira. Cells. 11 (1), 65 (2021).
- Chen, M., et al. Convolutional neural networks for automated annotation of cellular cryo-electron tomograms. Nat Methods. 14 (10), 983-985 (2017).
- Lamm, L., et al. MemBrain v2: an end-to-end tool for the analysis of membranes in cryo-electron tomography. bioRxiv. , (2024).
- Pidhorskyi, S., Morehead, M., Jones, Q., Spirou, G., Doretto, G. syGlass: interactive exploration of multidimensional images using virtual reality head-mounted displays. arXiv. , (2018).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Tegunov, D., Cramer, P. Real-time cryo-electron microscopy data preprocessing with Warp. Nat Methods. 16 (11), 1146-1152 (2019).
- Bepler, T., Kelley, K., Noble, A. J., Berger, B. Topaz-Denoise: general deep denoising models for cryoEM and cryoET. Nat Commun. 11, 5208 (2020).
- Buchholz, T. -. O., Berninger, L., Maurer, M., Jug, F. Cryo-CARE: content-aware image restoration for cryo-transmission electron microscopy data. Proc IEEE Int Symp Biomed Imaging. 16, 502-506 (2019).
- Buchholz, T. -. O., Jordan, M., Pigino, G., Jug, F. Content-aware image restoration for electron microscopy. Methods Cell Biol. 152, 277-289 (2019).
- Liu, Y. -. T., et al. Isotropic reconstruction for electron tomography with deep learning. Nat Commun. 13, 6482 (2022).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. J Struct Biol. 116 (1), 71-76 (1996).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved