Method Article
Aproveitando a realidade virtual para segmentação imersiva e análise de dados de tomografia crioeletrônica
Neste Artigo
Resumo
A tomografia crioeletrônica (cryo-ET) permite a visualização 3D da ultraestrutura celular com resolução nanométrica, mas a segmentação manual permanece demorada e complexa. Apresentamos um novo fluxo de trabalho que integra software avançado de realidade virtual para segmentar tomogramas crio-ET, mostrando sua eficácia por meio da segmentação de mitocôndrias em células de mamíferos.
Resumo
A tomografia crioeletrônica (crio-ET) é uma técnica poderosa para visualizar a ultraestrutura das células em três dimensões (3D) com resolução nanométrica. No entanto, a segmentação manual de componentes celulares em dados crio-ET continua sendo um gargalo significativo devido à sua complexidade e natureza demorada. Neste trabalho, apresentamos um novo fluxo de trabalho de segmentação que integra software avançado de realidade virtual (VR) para aumentar a eficiência e a precisão da segmentação de conjuntos de dados crio-ET. Esse fluxo de trabalho aproveita uma ferramenta de RV imersiva com interação 3D intuitiva, permitindo que os usuários naveguem e anotem estruturas celulares complexas em um ambiente mais natural e interativo. Para avaliar a eficácia do fluxo de trabalho, aplicamo-lo à segmentação de mitocôndrias em células do epitélio pigmentar da retina (RPE1). As mitocôndrias, essenciais para a produção e sinalização de energia celular, exibem mudanças morfológicas dinâmicas, tornando-as uma amostra de teste ideal. O software VR facilitou o delineamento preciso das membranas mitocondriais e estruturas internas, permitindo a análise a jusante das estruturas de membrana segmentadas. Demonstramos que esse fluxo de trabalho de segmentação baseado em VR melhora significativamente a experiência do usuário, mantendo a segmentação precisa de estruturas celulares intrincadas em dados crio-ET. Essa abordagem é promissora para amplas aplicações em biologia celular estrutural e educação científica, oferecendo uma ferramenta transformadora para pesquisadores envolvidos em análises celulares detalhadas.
Introdução
A tomografia crioeletrônica (crio-ET) revolucionou nossa capacidade de visualizar componentes celulares em seu estado quase nativo em alta resolução 1,2. Essa técnica poderosa permite que os pesquisadores caracterizem a ultraestrutura celular, fornecendo insights sem precedentes sobre a arquitetura e a função celular. No entanto, o cryo-ET tem suas limitações, a principal delas é a exigência de que as amostras sejam finas o suficiente para serem transparentes em elétrons (normalmente <0,5 μm) para imagens em microscópios eletrônicos de transmissão com capacidade criogênica padrão. Avanços recentes na moagem de feixe de íons criofocado permitiram o afinamento de amostras espessas para análise crio-ET 3,4.
Os fluxos de trabalho típicos de crio-ET começam com a coleta de uma série de inclinação, onde a amostra é fotografada em vários ângulos, variando de +60° a -60°. Essas imagens são então alinhadas computacionalmente e retroprojetadas para criar um volume tridimensional (3D) ou tomograma 5,6,7. Este tomograma serve como um mapa 3D detalhado da paisagem celular, oferecendo informações espaciais e temporais sobre as estruturas celulares. Um refinamento adicional por meio da média do subtomograma, onde várias cópias da mesma estrutura são alinhadas e calculadas em média, pode aumentar ainda mais os limites de resolução, às vezes alcançando uma resolução subnanométricade 7,8,9,10.
Um passo crucial na extração de informações biológicas significativas desses tomogramas é a segmentação. Esse processo envolve a anotação de estruturas celulares específicas, como membranas, dentro do volume 3D. A segmentação permite análises avançadas, incluindo o cálculo de distâncias intermembranares e curvatura da membrana, fornecendo informações valiosas sobre os processos celulares11,12. Embora vários pacotes de software estejam disponíveis para esta tarefa, incluindo Dragonfly, Amira, MemBrain, EMAN2 e tomomemsegtv 13,14,15,16,17, o processo de segmentação continua sendo um gargalo significativo na análise de dados crio-ET. Muitas vezes, é um processo manual trabalhoso e demorado, podendo levar semanas a meses para ser concluído. Muitos desses pacotes oferecem recursos de segmentação automática, mas frequentemente exigem uma extensa correção manual para remover falsos positivos, um processo que pode ser trabalhoso e pouco intuitivo quando executado fatia por fatia ou em 3D.
Propomos uma abordagem alternativa, aproveitando a tecnologia de realidade virtual (VR) para enfrentar esses desafios. A RV oferece um método imersivo e interativo para visualização de dados, permitindo que os usuários naveguem pelo volume tomográfico como se estivessem dentro do próprio ambiente celular. Essa abordagem também fornece uma plataforma valiosa para educação científica e exploração científica e descoberta de dados crio-ET in situ , fornecendo uma experiência imersiva e envolvente única. Neste trabalho, apresentamos um protocolo para segmentação de dados crio-ET utilizando o syGlass18, software de RV projetado para visualização científica. Este software fornece um kit de ferramentas abrangente para análise de dados crio-ET, incluindo segmentação manual, refinamento de segmentações geradas automaticamente e até mesmo coleta de partículas em tomogramas. Nosso estudo demonstra a viabilidade da RV como uma ferramenta poderosa para segmentações de limpeza, coleta de partículas e segmentação manual na análise de dados crio-ET.
Para ilustrar a utilidade do software VR para segmentação crio-ET, nos concentramos na análise da morfologia mitocondrial em células do epitélio pigmentar da retina (RPE1). As mitocôndrias servem como um excelente caso de teste para segmentação devido à sua estrutura complexa e à presença de características facilmente quantificáveis, como a distância entre as membranas mitocondriais externa e interna. Esses recursos podem ser medidos com precisão usando ferramentas de análise morfométrica de superfície12, fornecendo métricas robustas para avaliar a qualidade da segmentação. Este protocolo fornece instruções passo a passo para segmentar dados crio-ET usando syGlass, demonstrando sua utilidade dentro do pipeline de segmentação crio-ET. Ao incorporar a segmentação baseada em VR no fluxo de trabalho crio-ET, pretendemos melhorar a eficiência e a precisão da análise estrutural manual em biologia celular.
Protocolo
1. Preparação de dados crio-ET para segmentação
- Converta tomogramas Cryo-ET brutos em um formato de dados compatível com syGlass, como pilhas TIFF. Defina o sinal para que as partículas fiquem brancas sobre pretas e execute a equalização do histograma com ImageJ19.
NOTA: Para melhorar a relação sinal-ruído, recomenda-se que os tomogramas sejam removidos com software como Warp20, Topaz Denoise21, cryoCARE22,23 ou IsoNet24 antes de serem importados para o software. - Inicie o software VR no computador. Navegue até o menu Arquivo e selecione Criar projeto.
- Clique em Criar Novo Projeto | Adicionar arquivos. Navegue até o local onde os arquivos TIFF são salvos e importe-os para o software.
- Quando solicitado a confirmar se os arquivos são uma série temporal, clique em Não.
- Nomeie o projeto e clique em Salvar para criar um novo projeto na lista de projetos.
- Clique duas vezes no projeto para abrir o tomograma e carregá-lo no ambiente de realidade virtual interativo do software.
2. Configurando a realidade virtual (VR)
- Conecte o fone de ouvido VR e os controladores manuais ao computador.
- Siga as instruções na tela para calibrar o ambiente de RV.
NOTA: A calibração refere-se ao espaço tridimensional onde a segmentação será feita. Os parâmetros (comprimento, largura e altura) do espaço de trabalho precisam ser definidos antes que a projeção VR seja exibida. A projeção de RV só será visível nesses parâmetros. - A segmentação será feita apenas dentro do espaço tridimensional de trabalho definido. Certifique-se de que o campo de visão contenha a área desejada para ser trabalhada no ambiente de RV.
3. Otimizando a visualização 3D
- Clique no botão Visualização na interface do software. Ajuste várias opções de visualização, como contraste, janelas, brilho e controles deslizantes de limite para aprimorar o sinal e minimizar o ruído.
- Use os controladores manuais para aproximar o tomograma ou afastá-lo conforme necessário.
- Ative a ferramenta Cortar usando o controlador esquerdo para inspecionar visualmente diferentes fatias dentro do tomograma.
4. Processo de segmentação
- Navegue pelo tomograma até a fatia desejada onde a segmentação começará.
- Ative a opção Região de interesse (ROI) no menu de anotação usando os controladores manuais. Uma caixa verde aparecerá dentro do tomograma.
- Ajuste o tamanho e a posição da caixa verde, movendo-a para a área a ser segmentada.
- Bloqueie o ROI usando o controlador esquerdo e inicie a segmentação. Depois de bloquear o ROI, a ferramenta muda automaticamente para um modo de pintura que permite a segmentação dos dados volumétricos.
- Aumente ou diminua o zoom do tomograma para uma segmentação precisa.
- Ajuste o tamanho do pincel girando no sentido horário ou anti-horário para obter o controle ideal.
- Segmente cuidadosamente o ROI (por exemplo, membranas mitocondriais) dentro da área 3D selecionada. Se ocorrer um erro durante a segmentação, pressione e segure o gatilho do controlador secundário para ativar o modo de apagamento e, em seguida, apague o erro usando o mesmo movimento da segmentação.
- Repita esse processo região por região até que todo o tomograma seja segmentado.
5. Exportação de dados e análises segmentados
- Depois de concluir a segmentação, selecione o projeto clicando nele para garantir que ele seja destacado.
- Clique na guia Projetos no canto superior esquerdo e selecione ROIs.
- Escolha exportar o volume inteiro ou um ROI específico. Especifique o local de exportação para os dados segmentados.
- Carregue e analise os dados segmentados usando o software de sua escolha e gere figuras para publicação.
6. Importando a máscara binária para o software para limpeza
- Execute as etapas 1.1 a 1.6.
- Clique com o botão direito do mouse no projeto e clique em Adicionar dados de máscara. Em seguida, navegue até onde a segmentação inicial é salva e importe-a no mesmo projeto.
- Ative a anotação de ROI para fazer edições na segmentação inicial.
- Adicione/apague a segmentação para limpar a segmentação inicial.
- Ajuste os ROIs da máscara na opção ROI na guia de máscaras . Para alterar o ROI da máscara de um para o outro, ative a única opção de substituição de máscara e vá para o modo de pintura com o controlador manual. Este modo substituirá apenas os dados da máscara visíveis.
- Exporte os ROIs da máscara ajustados utilizando as etapas descritas nas etapas 5.1 a 5.3.
7. Escolhendo coordenadas de partículas usando o software VR
- Crie um projeto usando tomograma (pilha TIFF) utilizando as etapas descritas na seção 1.
- Clique em Anotação | Contagem, que abre uma janela com opções manuais ou automatizadas .
- Para a opção manual , escolha uma cor para as contagens e comece a marcar as coordenadas.
- Para a opção automatizada, escolha um tamanho de célula e insira a resolução. Clique em Executar contagem automática, edite as contagens (mantenha as boas escolhas e descarte as ruins) e salve o progresso.
- Exporte as coordenadas de contagem clicando na guia do projeto | anotações.
- Para a origem da anotação, escolha Canto inferior esquerdo e, para Unidades de anotação, escolha Voxels.
- Clique na célula de pontos de contagem para realçá-la.
- Clique em Exportar selecionado para salvar as coordenadas da seleção em um arquivo .csv.
NOTA: Este arquivo pode ser usado para extrair subtomogramas do tomograma para média de subtomograma
Resultados
Neste estudo, segmentamos tomogramas contendo mitocôndrias e organelas membranosas adicionais (por exemplo, vesículas, retículo endoplasmático) usando syGlass. Os tomogramas foram inicialmente reconstruídos em Warp usando projeção ponderada a 16,00 Å / pixel e foram submetidos à correção de cunha ausente e redução de ruído utilizando o software IsoNet. Os tomogramas a seguir foram submetidos a processamento adicional para importação, conforme mostrado na Figura 1A (consulte também Vídeo Suplementar S1, Vídeo Suplementar S2, Vídeo Suplementar S3 e Vídeo Suplementar S4).
Após o pré-processamento, os tomogramas no formato MRC foram convertidos em pilhas TIFF usando ImageJ, com inversão de contraste aplicada para fazer as membranas parecerem brancas em um fundo preto. A equalização do histograma foi então realizada para aumentar ainda mais o contraste; Isso também permite um limite mais eficaz. As pilhas TIFF foram importadas e visualizadas em 3D em um ambiente imersivo de RV, fornecendo inspeção detalhada de estruturas membranosas utilizando a ferramenta de corte no software mostrado na Figura 1B.
A segmentação manual foi realizada no software, com janelas definidas para automático e ajustes de brilho/limite feitos para otimizar a visibilidade do recurso celular. As ROIs em torno das mitocôndrias e outras estruturas foram definidas usando os controladores de RV. A ferramenta de segmentação permitiu o delineamento preciso dos limites da membrana, com erros corrigidos usando a função de apagamento. A navegação fatia a fatia ou em 3D usando a ferramenta ROI para encaixotar regiões em todo o tomograma, combinada com a ferramenta pincel ajustável, garantiu a segmentação precisa das membranas mitocondriais e outras organelas, conforme mostrado na Figura 1C.
Os dados segmentados foram visualizados gerando uma malha usando a opção de superfícies da ferramenta ROI, com iterações de suavização definidas como 12 e nível de resolução definido como 3. As renderizações 3D finais demonstram claramente as estruturas mitocondriais, incluindo as membranas externa e interna, cristas e depósitos de fosfato de cálcio, conforme mostrado na Figura 1D (consulte também o Vídeo Suplementar S5, o Vídeo Suplementar S6, o Vídeo Suplementar S7 e o Vídeo Suplementar S8).
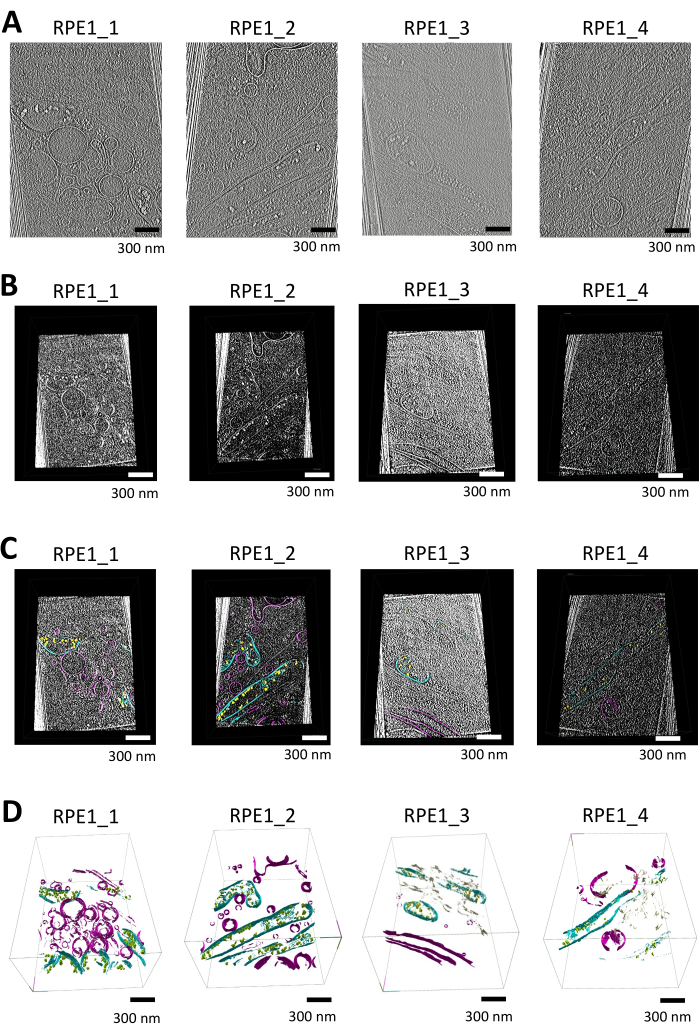
Figura 1: Fluxo de trabalho de visualização de tomograma, segmentação e renderização 3D usando syglass. (A) Fatias tomográficas de células RPE1 de borda fina. Tomografias corrigidas por IsoNet reconstruídas em Warp e usadas para segmentação com syGlass, visualizadas com IMOD25. Os tomogramas foram coletados em um microscópio Titan-Krios de 300 keV equipado com um detector K3 e focados nas bordas finas das células do epitélio retiniano. Cada tomograma contém pelo menos uma mitocôndria junto com várias outras organelas ligadas à membrana. Essas imagens são imagens estáticas de vídeos mostrados no Vídeo Suplementar S1, Vídeo Suplementar S2, Vídeo Suplementar S3 e Vídeo Suplementar S4. (B) Tomografias correspondentes visualizadas no software. Corte tomográfico criado usando a ferramenta de corte no software VR após otimizar o limite, o brilho e as janelas, revelando estruturas celulares distintas. (C) Tomografias visualizadas no software com membranas segmentadas. Corte tomográfico criado com a ferramenta de corte no software VR, sobreposto com a segmentação correspondente. Os depósitos de fosfato de cálcio são mostrados em amarelo, as membranas mitocondriais em ciano e outras membranas, como vesículas e a membrana plasmática em roxo. (D) Renderização 3D das membranas segmentadas em syGlass. As membranas mitocondriais, incluindo as cristas, as membranas mitocondriais externas e internas, são mostradas em ciano. Os depósitos de fosfato de cálcio estão em amarelo ou verde, as vesículas e a membrana plasmática estão em roxo e o retículo endoplasmático está em bronzeado. Essas imagens são imagens estáticas de vídeos mostrados em Vídeos Suplementares S5, Vídeo Suplementar S6, Vídeo Suplementar S7 e Vídeo Suplementar S8. Clique aqui para ver uma versão maior desta figura.
Vídeo Suplementar S1: Tomografia da amostra RPE-1_1 usada para segmentação neste estudo. Este tomograma é reconstruído a 16 Å / pixel usando Warp, depois removido e corrigido para a cunha ausente com IsoNet. Clique aqui para baixar este arquivo.
Vídeo Suplementar S2: Tomografia da amostra RPE-1_2 usada para segmentação neste estudo. Este tomograma é reconstruído a 16 Å / pixel usando Warp, depois removido e corrigido para a cunha ausente com IsoNet. Clique aqui para baixar este arquivo.
Vídeo Suplementar S3: Tomografia da amostra RPE-1_3 usada para segmentação neste estudo. Este tomograma é reconstruído a 16 Å / pixel usando Warp, depois removido e corrigido para a cunha ausente com IsoNet. Clique aqui para baixar este arquivo.
Vídeo Suplementar S4: Tomografia da amostra RPE-1_4 usada para segmentação neste estudo. Este tomograma é reconstruído a 16 Å / pixel usando Warp, depois removido e corrigido para a cunha ausente com IsoNet. Clique aqui para baixar este arquivo.
Vídeo Suplementar S5: A segmentação resultante da amostra RPE-1_1 após a geração de superfícies. As membranas mitocondriais são mostradas em ciano; os fosfatos de cálcio dentro das mitocôndrias são representados em amarelo; o retículo endoplasmático é mostrado em bronzeado; e outras membranas são exibidas em roxo. Clique aqui para baixar este arquivo.
Vídeo Suplementar S6: A segmentação resultante da amostra RPE-1_2 após a geração de superfícies. As membranas mitocondriais são mostradas em ciano; os fosfatos de cálcio dentro das mitocôndrias são representados em amarelo; o retículo endoplasmático é mostrado em bronzeado; e outras membranas são exibidas em roxo. Clique aqui para baixar este arquivo.
Vídeo Suplementar S7: A segmentação resultante da amostra RPE-1_3 após a geração de superfícies. As membranas mitocondriais são mostradas em ciano; os fosfatos de cálcio dentro das mitocôndrias são representados em amarelo; o retículo endoplasmático é mostrado em bronzeado; e outras membranas são exibidas em roxo. Clique aqui para baixar este arquivo.
Vídeo suplementar S8: A segmentação resultante da amostra RPE-1_4 após a geração de superfícies. As membranas mitocondriais são mostradas em ciano; os fosfatos de cálcio dentro das mitocôndrias são representados em amarelo; o retículo endoplasmático é mostrado em bronzeado; e outras membranas são exibidas em roxo. Clique aqui para baixar este arquivo.
Discussão
Neste trabalho, demonstramos como a RV, especificamente usando o syGlass, pode ser integrada ao pipeline crio-ET para segmentar efetivamente as estruturas celulares. Embora nosso foco tenha sido principalmente nas membranas, não há limitação inerente que impeça a segmentação de outras estruturas celulares, como filamentos ou ribossomos, pois é possível ajustar a forma e o tamanho do pincel e o limiar para garantir que apenas os voxels correspondentes aos objetos celulares desejados sejam marcados. Uma das principais vantagens da RV na segmentação é a interação intuitiva e prática com os dados, permitindo que os usuários visualizem e manipulem volumes como se estivessem fisicamente dentro deles. Os métodos tradicionais de segmentação manual ou limpeza de máscaras de segmentação inicial geralmente envolvem trabalhar diretamente em um computador, que pode ser menos imersivo e mais lento para anotar.
Ao incorporar a RV ao fluxo de trabalho crio-ET, os usuários podem não apenas interagir rapidamente com máscaras de segmentação geradas por outro software, mas também usar a RV para orientar a segmentação de estruturas parcialmente segmentadas e limpar com eficiência os falsos positivos. Atualmente, a segmentação manual ainda é necessária para a maioria dos casos de uso de anotação, e o fluxo de trabalho apresentado permite que os usuários gerem segmentações adequadas para análise downstream com maior facilidade e velocidade. Para este estudo, usamos o fone de ouvido HTC Vive VR, mas o software é compatível com dispositivos que possuem suporte a SteamVR ou OculusVR.
Para uma aplicação ideal deste protocolo, os dados crio-ET devem atender a critérios específicos, como tomogramas de alta qualidade com uma alta relação sinal-ruído (SNR). A SNR é um parâmetro essencial, pois a clareza das características estruturais influencia diretamente na eficácia da segmentação manual em RV. As etapas de pré-processamento, como correção de cunha ausente e redução de ruído, são partes fundamentais do fluxo de trabalho; neste protocolo, utilizamos o IsoNet para esses fins. Os tomogramas devem ser reconstruídos em uma resolução adequada - usando um tamanho de voxel que forneça detalhes estruturais suficientes e separação suficiente entre as estruturas celulares para permitir a segmentação eficaz entre as estruturas, mantendo ainda mais os tamanhos de dados gerenciáveis para visualização de RV. Além disso, o contraste invertido com estruturas celulares que aparecem em branco em um fundo preto e a equalização do histograma devem ser aplicados para aumentar a visibilidade das membranas e outras estruturas dentro do ambiente syGlass.
Vários pontos críticos devem ser considerados para garantir o sucesso deste protocolo. Primeiro, durante o pré-processamento de dados, a aplicação precisa da inversão de contraste e equalização do histograma é vital; ajustes inadequados podem resultar em visualização abaixo do ideal em RV, tornando a segmentação desafiadora. Em segundo lugar, dentro do software VR, ajustes apropriados de janelas, brilho e configurações de limite são essenciais para a visualização ideal das estruturas. Os usuários devem experimentar essas configurações para obter os melhores resultados para seus conjuntos de dados específicos e configurações de RV.
Quando se trata de solução de problemas, os usuários podem encontrar problemas como limitações de desempenho do sistema VR, especialmente ao lidar com grandes conjuntos de dados de tomograma. Se o ambiente de RV ficar lento ou sem resposta, considere reduzir a amostragem dos dados ou segmentar o tomograma em seções menores, ajustando a caixa delimitadora nas ferramentas ROI no syGlass e movendo-a ao longo do tomograma à medida que a segmentação está sendo executada. O controle deslizante de desempenho no menu de visualização também pode ser ajustado para reduzir o atraso durante o processo de segmentação. Além disso, para mitigar o enjôo, os usuários podem ajustar as configurações de RV para reduzir os efeitos de movimento ou fazer pausas regulares durante as sessões de segmentação.
Nosso protocolo de segmentação baseado em VR oferece benefícios significativos para os fluxos de trabalho de segmentação crio-ET atuais. Os métodos convencionais de segmentação manual geralmente envolvem anotação 2D em fatias individuais, o que pode ser demorado e pode não capturar totalmente a continuidade tridimensional13. Além disso, a segmentação baseada em VR introduz uma visão muito mais imersiva das estruturas celulares, auxiliando na visualização. As segmentações automatizadas de aprendizado de máquina estão emergindo como um método poderoso para obter segmentações de tomogramas celulares 14,16,17, embora os altos níveis de ruído e as estruturas complexas presentes nos dados crio-ET levem a lacunas e falsos positivos que requerem intervenção manual. Este protocolo oferece uma abordagem alternativa para segmentar manualmente tomogramas crioeletrônicos, para gerar segmentações iniciais que podem ser usadas como dados de treinamento para outro software de rede neural ou para limpar segmentações iniciais geradas a partir de outras abordagens automatizadas.
Em conclusão, este estudo destaca a segmentação baseada em RV como uma ferramenta promissora para análise e educação de dados crio-ET, oferecendo maior eficiência e uma experiência de usuário mais imersiva. Com mais desenvolvimento, a tecnologia VR tem o potencial de revolucionar a maneira como interpretamos e disseminamos descobertas científicas de estruturas celulares complexas em conjuntos de dados crio-ET, fornecendo uma alternativa valiosa aos métodos tradicionais de segmentação e educação.
Divulgações
Os autores declaram não ter conflitos de interesse.
Agradecimentos
Este trabalho foi realizado no Centro Nacional de Acesso e Treinamento em CryoEM (NCCAT) e no Centro de Microscopia Eletrônica Simons localizado no Centro de Biologia Estrutural de Nova York, apoiado pelo NIH (Common Fund U24GM129539, U24GM139171 e NIGMS R24GM154192), a Simons Foundation (SF349247) e a Assembleia Estadual de NY.
Materiais
| Name | Company | Catalog Number | Comments |
| CryoET Data | Format:TIFF-stack, TIFF | ||
| HTC VIVE Cosmos | HTC | 99HARL000-00 | https://www.vive.com/sea/product/vive-cosmos/features/ |
| Intel(R) Core(TM) i7-10870H CPU @ 2.20 GHz 2.21 GHz | Intel | https://ark.intel.com/content/www/us/en/ark/products/208018/intel-core-i7-10870h-processor-16m-cache-up-to-5-00-ghz.html | |
| NVIDIA GeForce RTX 3070 Laptop GPU | NVIDIA | https://www.nvidia.com/en-us/geforce/laptops/compare/30-series/ | |
| syGlass Software | syGlass | syGlass Software installed on a compatible Windows PC | |
| VIVE Cosmos Hand Controllers | HTC | 99HAFR001-00 | https://www.vive.com/us/accessory/cosmos-controller-right/ |
| Windows 11 Home | Microsoft | Microsoft Windows 11 Home |
Referências
- Plitzko, J. M., Schuler, B., Selenko, P. Structural biology outside the box-inside the cell. Curr Opin Struct Biol. 46, 110-121 (2017).
- Beck, M., Baumeister, W. Cryo-electron tomography: can it reveal the molecular sociology of cells in atomic detail. Trends Cell Biol. 26 (11), 825-837 (2016).
- Lam, V., Villa, E. Practical approaches for cryo-FIB milling and applications for cellular cryo-electron tomography. Methods Mol Biol. 2215, 49-82 (2021).
- Berger, C., et al. Cryo-electron tomography on focused ion beam lamellae transforms structural cell biology. Nat Methods. 20 (4), 499-511 (2023).
- Watson, A. J. I., Bartesaghi, A. Advances in cryo-ET data processing: meeting the demands of visual proteomics. Curr Opin Struct Biol. 87, 102861 (2024).
- Pyle, E., Zanetti, G. Current data processing strategies for cryo-electron tomography and subtomogram averaging. Biochem J. 478 (10), 1827-1845 (2021).
- Wan, W., Briggs, J. A. G. Cryo-electron tomography and subtomogram averaging. Methods Enzymol. 579, 329-367 (2016).
- Ni, T., Liu, K., Zhang, J., Atanasov, I., Mladenov, M. G., Jiang, W. High-resolution in situ structure determination by cryo-electron tomography and subtomogram averaging using emClarity. Nat Protoc. 17 (2), 421-444 (2022).
- Obr, M., et al. Exploring high-resolution cryo-ET and subtomogram averaging capabilities of contemporary DEDs. J Struct Biol. 214 (2), 107852 (2021).
- Tegunov, D., Xue, L., Dienemann, C., Cramer, P., Mahamid, J. Multi-particle cryo-EM refinement with M visualizes ribosome-antibiotic complex at 3.5 Å in cells. Nat Methods. 18 (2), 186-193 (2021).
- Salfer, M., Collado, J. F., Baumeister, W., Fernández-Busnadiego, R., Martínez-Sánchez, A. Reliable estimation of membrane curvature for cryo-electron tomography. PLoS Comput Biol. 16 (8), e1007962 (2020).
- Barad, B. A., et al. Quantifying organellar ultrastructure in cryo-electron tomography using a surface morphometrics pipeline. J Cell Biol. 222 (2), e202204093 (2023).
- Martinez-Sanchez, A., Garcia, I., Asano, S., Lucic, V., Fernandez, J. J. Robust membrane detection based on tensor voting for electron tomography. J Struct Biol. 186 (1), 49-61 (2014).
- Heebner, J. E., et al. Deep learning-based segmentation of cryo-electron tomograms. J Vis Exp. (189), e64435 (2022).
- Garza-López, E., Robles-Flores, M. Protocols for generating surfaces and measuring 3D organelle morphology using Amira. Cells. 11 (1), 65 (2021).
- Chen, M., et al. Convolutional neural networks for automated annotation of cellular cryo-electron tomograms. Nat Methods. 14 (10), 983-985 (2017).
- Lamm, L., et al. MemBrain v2: an end-to-end tool for the analysis of membranes in cryo-electron tomography. bioRxiv. , (2024).
- Pidhorskyi, S., Morehead, M., Jones, Q., Spirou, G., Doretto, G. syGlass: interactive exploration of multidimensional images using virtual reality head-mounted displays. arXiv. , (2018).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Tegunov, D., Cramer, P. Real-time cryo-electron microscopy data preprocessing with Warp. Nat Methods. 16 (11), 1146-1152 (2019).
- Bepler, T., Kelley, K., Noble, A. J., Berger, B. Topaz-Denoise: general deep denoising models for cryoEM and cryoET. Nat Commun. 11, 5208 (2020).
- Buchholz, T. -. O., Berninger, L., Maurer, M., Jug, F. Cryo-CARE: content-aware image restoration for cryo-transmission electron microscopy data. Proc IEEE Int Symp Biomed Imaging. 16, 502-506 (2019).
- Buchholz, T. -. O., Jordan, M., Pigino, G., Jug, F. Content-aware image restoration for electron microscopy. Methods Cell Biol. 152, 277-289 (2019).
- Liu, Y. -. T., et al. Isotropic reconstruction for electron tomography with deep learning. Nat Commun. 13, 6482 (2022).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. J Struct Biol. 116 (1), 71-76 (1996).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados