Method Article
利用虚拟现实对冷冻电子断层扫描数据进行沉浸式分割和分析
摘要
冷冻电子断层扫描 (cryo-ET) 能够以纳米分辨率对细胞超微结构进行 3D 可视化,但手动分割仍然耗时且复杂。我们提出了一种新的工作流程,它集成了先进的虚拟现实软件,用于分割冷冻 ET 断层扫描,通过分割哺乳动物细胞中的线粒体来展示其有效性。
摘要
冷冻电子断层扫描 (cryo-ET) 是一种强大的技术,可以纳米分辨率在三维 (3D) 中可视化细胞的超微结构。然而,由于其复杂性和耗时性,冷冻电子断层扫描数据中细胞成分的手动分割仍然是一个重大瓶颈。在这项工作中,我们提出了一种新颖的分割工作流程,该工作流程集成了先进的虚拟现实 (VR) 软件,以提高分割冷冻电子断层扫描数据集的效率和准确性。该工作流程利用具有直观 3D 交互的沉浸式 VR 工具,使用户能够在更自然和交互式的环境中导航和注释复杂的细胞结构。为了评估工作流程的有效性,我们将其应用于视网膜色素上皮 (RPE1) 细胞中线粒体的分割。线粒体对细胞能量产生和信号传导至关重要,表现出动态形态变化,使其成为理想的测试样品。VR 软件有助于精确描绘线粒体膜和内部结构,从而能够对分段膜结构进行下游分析。我们证明,这种基于 VR 的分割工作流程显著改善了用户体验,同时保持了对冷冻电子断层扫描数据中复杂细胞结构的准确分割。这种方法有望在结构细胞生物学和科学教育中得到广泛应用,为从事详细细胞分析的研究人员提供变革性工具。
引言
冷冻电子断层扫描 (cryo-ET) 彻底改变了我们以高分辨率可视化处于近天然状态的细胞成分的能力 1,2。这种强大的技术使研究人员能够表征细胞超微结构,为细胞结构和功能提供前所未有的见解。然而,冷冻电子断层扫描并非没有局限性,其中最主要的是要求样品足够薄,以便具有电子透明性(通常为 <0.5 μm),以便在标准冷冻透射电子显微镜中成像。冷冻聚焦离子束铣削的最新进展使用于冷冻电子断层扫描分析的厚样品变得薄化 3,4。
典型的冷冻电子断层扫描工作流程从收集倾斜序列开始,其中样品以各种角度成像,范围从 +60° 到 -60°。然后,这些图像经过计算对齐和反向投影,以创建三维 (3D) 体积或断层照片 5,6,7。该断层照片用作细胞景观的详细 3D 图,提供有关细胞结构的空间和时间信息。通过亚断层照片平均进行进一步细化,其中同一结构的多个副本对齐并平均,可以进一步突破分辨率极限,有时达到亚纳米分辨率 7,8,9,10。
从这些断层照片中提取有意义的生物信息的关键步骤是分割。这个过程涉及注释 3D 体积内的特定细胞结构,例如膜。分割可实现高级分析,包括膜间距离和膜曲率的计算,为细胞过程提供有价值的见解11,12。虽然有几种软件包可用于此任务,包括 Dragonfly、Amira、MemBrain、EMAN2 和 tomomemsegtv 13,14,15,16,17,但分割过程仍然是冷冻电子断层扫描数据分析中的一个重要瓶颈。这通常是一个劳动密集型且耗时的手动过程,可能需要数周到数月才能完成。其中许多软件包都提供自动分割功能,但通常需要大量的手动校正以消除误报,当逐切片或 3D 执行时,这个过程可能很费力且不直观。
我们提出了一种利用虚拟现实 (VR) 技术来应对这些挑战的替代方法。VR 为数据可视化提供了一种沉浸式和交互式方法,允许用户在断层扫描体积中导航,就像在蜂窝环境本身内部一样。这种方法通过提供身临其境且独特而引人入胜的体验,为科学教育以及 原位 冷冻电子断层扫描数据的科学探索和发现提供了一个有价值的平台。在这项工作中,我们提出了一种使用 syGlass 18 进行冷冻电子断层扫描数据分割的协议,syGlass18 是专为科学可视化而设计的 VR 软件。该软件为冷冻电子断层扫描数据分析提供了全面的工具包,包括手动分割、自动生成分割的优化,甚至断层扫描中的颗粒拾取。我们的研究证明了 VR 作为冷冻电子断层扫描数据分析中清洁分割、颗粒拾取和手动分割的强大工具的可行性。
为了说明 VR 软件在冷冻 ET 分割中的效用,我们专注于分析视网膜色素上皮 (RPE1) 细胞中的线粒体形态。线粒体由于其复杂的结构和易于量化的特征(例如线粒体外膜和内线粒体膜之间的距离)的存在,因此可以作为一个很好的分割测试用例。这些特征可以使用表面形态计量分析工具12 准确测量,为评估分割质量提供可靠的指标。该实验步骤提供了使用 syGlass 分割冷冻电子断层扫描数据的分步说明,展示了其在冷冻电子断层扫描分割流程中的实用性。通过将基于 VR 的分割整合到冷冻电子断层扫描工作流程中,我们的目标是提高细胞生物学中手动结构分析的效率和准确性。
研究方案
1. 准备用于分割的冷冻电子断层扫描数据
- 将原始冷冻电子断层扫描图转换为与 syGlass 兼容的数据格式,例如 TIFF 堆栈。设置信号,使颗粒为黑底白字,并使用 ImageJ19 执行直方图均衡。
注:为了提高信噪比,建议在将断层照片导入软件之前,使用 Warp20、Topaz Denoise21、cryoCARE22,23 或 IsoNet24 等软件对断层照片进行去噪。 - 在计算机上启动 VR 软件。导航到 文件 菜单,然后选择 创建项目。
- 单击 Create New Project |添加文件。导航到保存 TIFF 文件的位置,然后将它们导入软件。
- 当系统提示确认文件是否为时间序列时,单击 No (否)。
- 为项目命名,然后单击 Save 在项目列表下创建一个新项目。
- 双击项目以打开断层图并将其加载到软件交互式虚拟现实环境中。
2. 设置虚拟现实 (VR)
- 将 VR 头戴式设备和手持控制器连接到计算机。
- 按照屏幕上的说明校准 VR 环境。
注意:校准是指将进行分割的三维空间。在显示 VR 投影之前,需要定义工作空间的参数(长度、宽度和高度)。VR 投影仅在这些参数中可见。 - 分割将仅在定义的工作 3D 空间内进行。确保视野包含要在 VR 环境中处理的区域。
3. 优化 3D 可视化
- 单击软件界面中的 Visualization 按钮。调整各种可视化选项,如对比度、窗口化、亮度和阈值滑块,以增强信号并最大限度地减少杂色。
- 根据需要,使用手动控制器将断层扫描拉近或推开。
- 使用左侧控制器激活 Cut 工具,以直观地检查断层照片中的不同切片。
4. 细分流程
- 在断层图中导航到将开始分割的所需切片。
- 使用手动控制器激活注释菜单下的 Region of Interest (ROI) 选项。断层照片中将出现一个绿色框。
- 调整绿色框的大小和位置,将其移动到要分割的区域。
- 使用左侧控制器锁定 ROI 并启动分段。锁定 ROI 后,该工具会自动切换到允许分割体积数据的 绘制 模式。
- 放大或缩小断层照片以进行精确分割。
- 通过顺时针或逆时针旋转来调整 画笔 大小,以实现最佳控制。
- 仔细分割所选 3D 区域内的 ROI(例如,线粒体膜)。如果在分段过程中发生错误,请按住辅助控制器触发器以启用 擦除 模式,然后使用与分段相同的动作擦除错误。
- 逐个区域重复此过程,直到整个断层照片被分割。
5. 导出细分数据和分析
- 完成分段后,通过单击项目来选择项目,以确保它突出显示。
- 单击左上角的 Projects 选项卡,然后选择 ROI。
- 选择导出整个卷或特定 ROI。指定分段数据的导出位置。
- 使用所选软件加载和分析分割数据,并生成用于发布的图表。
6. 将二进制掩码导入软件进行清理
- 执行步骤 1.1-1.6。
- 右键单击项目,然后单击 Add mask data(添加掩码数据)。然后,导航到初始分段的保存位置,并将其导入到同一项目下。
- 使用 ROI 注释对初始分段进行编辑。
- 添加/擦除分段以清理初始分段。
- 调整 遮罩 ROI 在 遮罩 选项卡的 ROI 选项下。要将蒙版 ROI 从一个更改为另一个,请打开唯一的覆盖蒙版选项,然后使用手动控制器进入绘画模式。此模式将仅覆盖可见的掩码数据。
- 使用步骤 5.1-5.3 中描述的步骤导出调整后的蒙版 ROI。
7. 使用 VR 软件拾取粒子坐标
- 使用断层照片(TIFF 堆栈)使用第 1 节中描述的步骤创建一个项目。
- 单击 Annotation |Count,这将打开一个包含 手动 或 自动 选项的窗口。
- 对于 手动 选项,选择计数 的颜色 ,然后开始标记坐标。
- 对于 automated 选项,请选择像元大小并输入 分辨率。点击 Run auto counting,编辑计数(保留好的选择并丢弃坏的选择),并保存进度。
- 通过单击 Project 选项卡 |annotations 导出计数坐标。
- 对于注释原点,选择 Bottom Left Corner ,对于 Annotation Units (注释单位),选择 Voxels。
- 单击 计数点单元格 以突出显示该单元格。
- 单击 Export selected 将所选内容的坐标保存在.csv文件中。
注意:此文件可用于从断层图中提取子断层图以进行亚断层图平均
结果
在这项研究中,我们使用 syGlass 分割了包含线粒体和其他膜细胞器(例如,囊泡、内质网)的断层照片。断层图最初使用 16.00 Å/像素的加权回投影在 Warp 中重建,并使用 IsoNet 软件进行缺失楔形校正和去噪。如图 1A 所示,以下断层照片经过额外处理以进行导入(另请参见 补充视频 S1、 补充视频 S2、 补充视频 S3 和 补充视频 S4)。
预处理后,使用 ImageJ 将 MRC 格式的断层照片转换为 TIFF 堆栈,并应用对比反转以使膜在黑色背景上显示为白色。然后进行直方图均衡以进一步增强对比度;这也允许更有效的阈值设置。TIFF 堆栈被导入并在沉浸式 VR 环境中以 3D 形式可视化,使用 图 1B 所示软件中的切割工具对膜结构进行详细检查。
在软件中执行手动分割,将窗口设置为自动,并进行亮度/阈值调整以优化蜂窝特征的可见性。使用 VR 控制器定义线粒体和其他结构的 ROI。分割工具允许精确描绘膜边界,并使用擦除功能纠正错误。逐片导航或使用 ROI 工具在整个断层照片中框住区域,结合可调节的 画笔 工具,确保线粒体膜和其他细胞器的准确分割,如图 1C 所示。
通过使用 ROI 工具的 surfaces 选项生成网格来可视化分割数据,平滑迭代设置为 12,分辨率级别设置为 3。最终的 3D 渲染清楚地展示了线粒体结构,包括外膜和内膜、嵴和磷酸钙沉积物,如图 1D 所示(另见 补充视频 S5、 补充视频 S6、 补充视频 S7 和 补充视频 S8)。
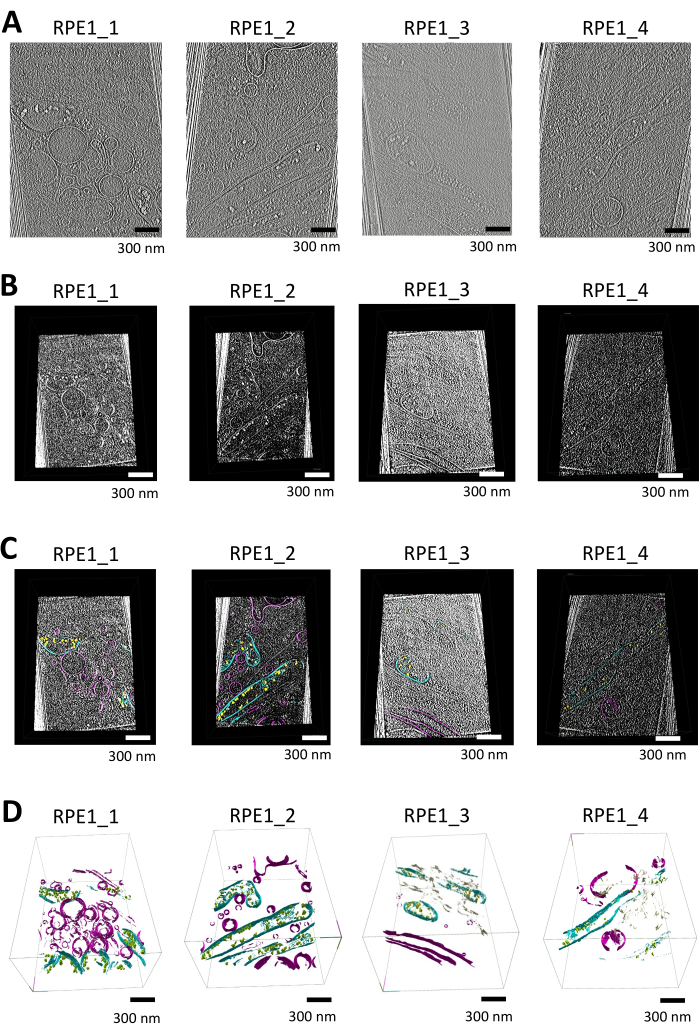
图 1:使用 syglass 进行断层照片可视化、分割和 3D 渲染的工作流程。 (A) 薄边缘 RPE1 细胞的断层扫描切片。在 Warp 中重建 IsoNet 校正的断层照片,并使用 syGlass 进行分割,使用 IMOD25 进行可视化。在配备 K3 检测器的 300 keV Titan-Krios 显微镜上收集断层照片,并聚焦于视网膜上皮细胞的薄边缘。每个断层照片包含至少一个线粒体以及各种其他膜结合细胞器。这些图像是 补充视频 S1、 补充视频 S2、 补充视频 S3 和 补充视频 S4 中显示的视频中的静止图像。(B) 在软件中可视化的相应断层照片。在优化阈值、亮度和窗口化后,使用 VR 软件中的 剪切 工具创建的断层切片,揭示了不同的细胞结构。(C) 在软件中可视化的断层图与分段膜。使用 VR 软件中的 剪切 工具创建的断层扫描切片,与相应的分割重叠。磷酸钙沉积物以黄色显示,线粒体膜以青色显示,其他膜(如囊泡和质膜)以紫色显示。(D) syGlass 中分段膜的 3D 渲染。线粒体膜,包括嵴膜、外膜和线粒体内膜,以青色显示。磷酸钙沉积物呈黄色或绿色,囊泡和质膜呈紫色,内质网呈棕褐色。这些图像是 补充视频 S5、 补充视频 S6、 补充视频 S7 和 补充视频 S8 中显示的视频中的静止图像。 请单击此处查看此图的较大版本。
补充视频 S1:本研究中用于分割的样本 RPE-1_1 的断层图。 该断层图使用 Warp 以 16 Å/像素重建,然后使用 IsoNet 对缺失的楔形进行去噪和校正。 请点击此处下载此文件。
补充视频 S2:本研究中用于分割的样本 RPE-1_2 的断层图。 该断层图使用 Warp 以 16 Å/像素重建,然后使用 IsoNet 对缺失的楔形进行去噪和校正。 请点击此处下载此文件。
补充视频 S3:本研究中用于分割的样本 RPE-1_3 的断层图。 该断层图使用 Warp 以 16 Å/像素重建,然后使用 IsoNet 对缺失的楔形进行去噪和校正。 请点击此处下载此文件。
补充视频 S4:本研究中用于分割的样本 RPE-1_4 的断层图。 该断层图使用 Warp 以 16 Å/像素重建,然后使用 IsoNet 对缺失的楔形进行去噪和校正。 请点击此处下载此文件。
补充视频 S5:生成表面后样品 RPE-1_1 的结果分割。 线粒体膜以青色显示;线粒体内的磷酸钙用黄色表示;内质网以 tan 表示;和其他膜显示为紫色。 请点击此处下载此文件。
补充视频 S6:生成表面后样本 RPE-1_2 的结果分割。 线粒体膜以青色显示;线粒体内的磷酸钙用黄色表示;内质网以 tan 表示;和其他膜显示为紫色。 请点击此处下载此文件。
补充视频 S7:生成表面后样本 RPE-1_3 的结果分割。 线粒体膜以青色显示;线粒体内的磷酸钙用黄色表示;内质网以 tan 表示;和其他膜显示为紫色。 请点击此处下载此文件。
补充视频 S8:生成表面后样本 RPE-1_4 的结果分割。 线粒体膜以青色显示;线粒体内的磷酸钙用黄色表示;内质网以 tan 表示;和其他膜显示为紫色。 请点击此处下载此文件。
讨论
在这项工作中,我们展示了如何将 VR,特别是使用 syGlass,集成到冷冻电子断层扫描管道中,以有效地分割细胞结构。尽管我们的重点主要放在膜上,但没有固有的限制阻止其他细胞结构的分割,例如细丝或核糖体,因为可以调整画笔的形状和大小,以及阈值以确保仅标记与所需细胞对象相对应的体素。VR 在分割方面的主要优势之一是与数据进行直观、动手交互,从而允许用户可视化和作体积,就像他们身处其中一样。手动分割或清理初始分割掩码的传统方法通常涉及直接在计算机上工作,这可能不那么沉浸且注释速度较慢。
通过将 VR 整合到冷冻电子断层扫描工作流程中,用户不仅可以与其他软件生成的分割掩码快速交互,还可以使用 VR 指导部分分割结构的分割并有效清理假阳性。目前,大多数注释使用案例仍然需要手动分割,并且提供的工作流程使用户能够以更高的易用性和速度生成适合下游分析的分割。在这项研究中,我们使用了 HTC Vive VR 头戴式设备,但该软件与支持 SteamVR 或 OculusVR 的设备兼容。
为了实现该方案的最佳应用,冷冻电子断层扫描数据应满足特定标准,例如具有高信噪比 (SNR) 的高质量断层照片。SNR 是一个基本参数,因为结构特征的清晰度直接影响 VR 中手动分割的有效性。预处理步骤(例如缺失楔区校正和去噪)是工作流的关键部分;在该协议中,我们将 IsoNet 用于这些目的。断层照片应以适当的分辨率重建 - 使用体素大小,该体素大小可提供足够的结构细节和细胞结构之间的足够分离,以实现结构之间的有效分割,同时进一步保持可管理的数据大小以进行 VR 可视化。此外,应应用与黑底白色细胞结构的倒置对比和直方图均衡,以提高 syGlass 环境中膜和其他结构的可见性。
为了确保该协议的成功,必须考虑几个关键点。首先,在数据预处理过程中,准确应用对比度反转和直方图均衡至关重要;不当的调整会导致 VR 中的可视化效果不佳,从而使分割变得具有挑战性。其次,在 VR 软件中,适当调整窗口、亮度和阈值设置对于结构的最佳可视化至关重要。用户应尝试使用这些设置,以便为其特定数据集和 VR 设置获得最佳结果。
在故障排除方面,用户可能会遇到 VR 系统性能限制等问题,尤其是在处理大型断层照片数据集时。如果 VR 环境变得滞后或无响应,请考虑通过调整 syGlass 中 ROI 工具中的边界框并在执行分割时沿断层图移动,对数据进行缩减采样或将断层图分割成更小的部分。还可以调整可视化菜单中的性能滑块,以减少分段过程中的滞后。此外,为了缓解晕动症,用户可以调整 VR 设置以减少运动效果或在分段会话期间定期休息。
我们基于 VR 的分割方案为当前的冷冻电子断层扫描分割工作流程提供了显著优势。传统的手动分割方法通常涉及对单个切片进行 2D 注释,这可能非常耗时,并且可能无法完全捕获三维连续性13。此外,基于 VR 的分割引入了更加身临其境的细胞结构视图,有助于可视化。自动机器学习分割正在成为一种从细胞断层照片14、16、17 中获取分割的强大方法,尽管冷冻电子断层扫描数据中存在的高噪声水平和复杂结构会导致需要人工干预的空白和假阳性。该协议提供了一种手动分割冷冻电子断层照片的替代方法,以生成可能用作其他神经网络软件的训练数据的初始分割,或清理由其他自动化方法生成的初始分割。
总之,本研究强调基于 VR 的分割是冷冻电子断层扫描数据分析和教育的一种有前途的工具,可提供更高的效率和更身临其境的用户体验。随着技术的进一步发展,VR 技术有可能彻底改变我们在冷冻电子断层扫描数据集中解释和传播复杂细胞结构的科学发现的方式,为传统的分割和教育方法提供有价值的替代方案。
披露声明
作者声明他们没有利益冲突。
致谢
这项工作是在国家冷冻电镜访问和培训中心 (NCCAT) 和位于纽约结构生物学中心的西蒙斯电子显微镜中心进行的,得到了 NIH(共同基金 U24GM129539、U24GM139171 和 NIGMS R24GM154192)、西蒙斯基金会 (SF349247) 和纽约州议会的支持。
材料
| Name | Company | Catalog Number | Comments |
| CryoET Data | Format:TIFF-stack, TIFF | ||
| HTC VIVE Cosmos | HTC | 99HARL000-00 | https://www.vive.com/sea/product/vive-cosmos/features/ |
| Intel(R) Core(TM) i7-10870H CPU @ 2.20 GHz 2.21 GHz | Intel | https://ark.intel.com/content/www/us/en/ark/products/208018/intel-core-i7-10870h-processor-16m-cache-up-to-5-00-ghz.html | |
| NVIDIA GeForce RTX 3070 Laptop GPU | NVIDIA | https://www.nvidia.com/en-us/geforce/laptops/compare/30-series/ | |
| syGlass Software | syGlass | syGlass Software installed on a compatible Windows PC | |
| VIVE Cosmos Hand Controllers | HTC | 99HAFR001-00 | https://www.vive.com/us/accessory/cosmos-controller-right/ |
| Windows 11 Home | Microsoft | Microsoft Windows 11 Home |
参考文献
- Plitzko, J. M., Schuler, B., Selenko, P. Structural biology outside the box-inside the cell. Curr Opin Struct Biol. 46, 110-121 (2017).
- Beck, M., Baumeister, W. Cryo-electron tomography: can it reveal the molecular sociology of cells in atomic detail. Trends Cell Biol. 26 (11), 825-837 (2016).
- Lam, V., Villa, E. Practical approaches for cryo-FIB milling and applications for cellular cryo-electron tomography. Methods Mol Biol. 2215, 49-82 (2021).
- Berger, C., et al. Cryo-electron tomography on focused ion beam lamellae transforms structural cell biology. Nat Methods. 20 (4), 499-511 (2023).
- Watson, A. J. I., Bartesaghi, A. Advances in cryo-ET data processing: meeting the demands of visual proteomics. Curr Opin Struct Biol. 87, 102861 (2024).
- Pyle, E., Zanetti, G. Current data processing strategies for cryo-electron tomography and subtomogram averaging. Biochem J. 478 (10), 1827-1845 (2021).
- Wan, W., Briggs, J. A. G. Cryo-electron tomography and subtomogram averaging. Methods Enzymol. 579, 329-367 (2016).
- Ni, T., Liu, K., Zhang, J., Atanasov, I., Mladenov, M. G., Jiang, W. High-resolution in situ structure determination by cryo-electron tomography and subtomogram averaging using emClarity. Nat Protoc. 17 (2), 421-444 (2022).
- Obr, M., et al. Exploring high-resolution cryo-ET and subtomogram averaging capabilities of contemporary DEDs. J Struct Biol. 214 (2), 107852 (2021).
- Tegunov, D., Xue, L., Dienemann, C., Cramer, P., Mahamid, J. Multi-particle cryo-EM refinement with M visualizes ribosome-antibiotic complex at 3.5 Å in cells. Nat Methods. 18 (2), 186-193 (2021).
- Salfer, M., Collado, J. F., Baumeister, W., Fernández-Busnadiego, R., Martínez-Sánchez, A. Reliable estimation of membrane curvature for cryo-electron tomography. PLoS Comput Biol. 16 (8), e1007962 (2020).
- Barad, B. A., et al. Quantifying organellar ultrastructure in cryo-electron tomography using a surface morphometrics pipeline. J Cell Biol. 222 (2), e202204093 (2023).
- Martinez-Sanchez, A., Garcia, I., Asano, S., Lucic, V., Fernandez, J. J. Robust membrane detection based on tensor voting for electron tomography. J Struct Biol. 186 (1), 49-61 (2014).
- Heebner, J. E., et al. Deep learning-based segmentation of cryo-electron tomograms. J Vis Exp. (189), e64435 (2022).
- Garza-López, E., Robles-Flores, M. Protocols for generating surfaces and measuring 3D organelle morphology using Amira. Cells. 11 (1), 65 (2021).
- Chen, M., et al. Convolutional neural networks for automated annotation of cellular cryo-electron tomograms. Nat Methods. 14 (10), 983-985 (2017).
- Lamm, L., et al. MemBrain v2: an end-to-end tool for the analysis of membranes in cryo-electron tomography. bioRxiv. , (2024).
- Pidhorskyi, S., Morehead, M., Jones, Q., Spirou, G., Doretto, G. syGlass: interactive exploration of multidimensional images using virtual reality head-mounted displays. arXiv. , (2018).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Tegunov, D., Cramer, P. Real-time cryo-electron microscopy data preprocessing with Warp. Nat Methods. 16 (11), 1146-1152 (2019).
- Bepler, T., Kelley, K., Noble, A. J., Berger, B. Topaz-Denoise: general deep denoising models for cryoEM and cryoET. Nat Commun. 11, 5208 (2020).
- Buchholz, T. -. O., Berninger, L., Maurer, M., Jug, F. Cryo-CARE: content-aware image restoration for cryo-transmission electron microscopy data. Proc IEEE Int Symp Biomed Imaging. 16, 502-506 (2019).
- Buchholz, T. -. O., Jordan, M., Pigino, G., Jug, F. Content-aware image restoration for electron microscopy. Methods Cell Biol. 152, 277-289 (2019).
- Liu, Y. -. T., et al. Isotropic reconstruction for electron tomography with deep learning. Nat Commun. 13, 6482 (2022).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. J Struct Biol. 116 (1), 71-76 (1996).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。