Method Article
Sfruttare la realtà virtuale per la segmentazione e l'analisi immersive dei dati di tomografia crioelettronica
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
La tomografia crioelettronica (cryo-ET) consente la visualizzazione 3D dell'ultrastruttura cellulare con risoluzione nanometrica, ma la segmentazione manuale rimane lunga e complessa. Presentiamo un nuovo flusso di lavoro che integra un software avanzato di realtà virtuale per la segmentazione dei tomogrammi crio-ET, dimostrandone l'efficacia attraverso la segmentazione dei mitocondri nelle cellule di mammifero.
Abstract
La crio-tomografia elettronica (cryo-ET) è una potente tecnica per visualizzare l'ultrastruttura delle cellule in tre dimensioni (3D) con risoluzione nanometrica. Tuttavia, la segmentazione manuale dei componenti cellulari nei dati crio-ET rimane un collo di bottiglia significativo a causa della sua complessità e della sua natura dispendiosa in termini di tempo. In questo lavoro, presentiamo un nuovo flusso di lavoro di segmentazione che integra un software avanzato di realtà virtuale (VR) per migliorare sia l'efficienza che l'accuratezza della segmentazione dei set di dati crio-ET. Questo flusso di lavoro sfrutta uno strumento VR immersivo con un'interazione 3D intuitiva, che consente agli utenti di navigare e annotare strutture cellulari complesse in un ambiente più naturale e interattivo. Per valutare l'efficacia del flusso di lavoro, lo abbiamo applicato alla segmentazione dei mitocondri nelle cellule dell'epitelio pigmentato retinico (RPE1). I mitocondri, essenziali per la produzione di energia cellulare e la segnalazione, mostrano cambiamenti morfologici dinamici, rendendoli un campione di prova ideale. Il software VR ha facilitato la delineazione precisa delle membrane mitocondriali e delle strutture interne, consentendo l'analisi a valle delle strutture segmentate della membrana. Dimostriamo che questo flusso di lavoro di segmentazione basato sulla realtà virtuale migliora significativamente l'esperienza dell'utente, mantenendo al contempo una segmentazione accurata di complesse strutture cellulari nei dati crio-ET. Questo approccio è promettente per ampie applicazioni nella biologia cellulare strutturale e nell'educazione scientifica, offrendo uno strumento trasformativo per i ricercatori impegnati nell'analisi cellulare dettagliata.
Introduzione
La tomografia crioelettronica (cryo-ET) ha rivoluzionato la nostra capacità di visualizzare i componenti cellulari nel loro stato quasi nativo ad alta risoluzione 1,2. Questa potente tecnica consente ai ricercatori di caratterizzare l'ultrastruttura cellulare, fornendo informazioni senza precedenti sull'architettura e la funzione cellulare. Tuttavia, la crio-ET non è priva di limiti, il principale tra questi è il requisito che i campioni siano abbastanza sottili da essere trasparenti agli elettroni (in genere <0,5 μm) per l'imaging nei microscopi elettronici a trasmissione standard criogenici. I recenti progressi nella fresatura a fascio ionico criofocalizzato hanno consentito l'assottigliamento di campioni spessi per l'analisi crio-ET 3,4.
I tipici flussi di lavoro crio-ET iniziano con la raccolta di una serie di inclinazione, in cui il campione viene ripreso a varie angolazioni, da +60° a -60°. Queste immagini vengono quindi allineate computazionalmente e retroproiettate per creare un volume tridimensionale (3D) o tomogramma 5,6,7. Questo tomogramma funge da mappa 3D dettagliata del paesaggio cellulare, offrendo informazioni sia spaziali che temporali sulle strutture cellulari. Un ulteriore perfezionamento attraverso la media del sottotomogramma, in cui più copie della stessa struttura sono allineate e mediate, può spingere ulteriormente i limiti di risoluzione, a volte raggiungendo una risoluzione sub-nanometricadi 7,8,9,10.
Un passaggio cruciale nell'estrazione di informazioni biologiche significative da questi tomogrammi è la segmentazione. Questo processo comporta l'annotazione di strutture cellulari specifiche, come le membrane, all'interno del volume 3D. La segmentazione consente analisi avanzate, tra cui il calcolo delle distanze intermembrana e della curvatura della membrana, fornendo preziose informazioni sui processi cellulari11,12. Sebbene siano disponibili diversi pacchetti software per questo compito, tra cui Dragonfly, Amira, MemBrain, EMAN2 e tomomemsegtv 13,14,15,16,17, il processo di segmentazione rimane un collo di bottiglia significativo nell'analisi dei dati crio-ET. Spesso si tratta di un processo manuale laborioso e dispendioso in termini di tempo, che potrebbe richiedere settimane o mesi per essere completato. Molti di questi pacchetti offrono funzionalità di segmentazione automatica, ma spesso richiedono un'ampia correzione manuale per rimuovere i falsi positivi, un processo che può essere laborioso e poco intuitivo se eseguito sezione per fetta o in 3D.
Proponiamo un approccio alternativo sfruttando la tecnologia della realtà virtuale (VR) per affrontare queste sfide. La realtà virtuale offre un metodo immersivo e interattivo per la visualizzazione dei dati, consentendo agli utenti di navigare attraverso il volume tomografico come se si trovassero all'interno dell'ambiente cellulare stesso. Questo approccio fornisce anche una preziosa piattaforma per l'educazione scientifica e l'esplorazione scientifica e la scoperta di dati crio-ET in situ , fornendo un'esperienza coinvolgente e unica. In questo lavoro, presentiamo un protocollo per la segmentazione dei dati cryo-ET utilizzando syGlass18, software VR progettato per la visualizzazione scientifica. Questo software fornisce un toolkit completo per l'analisi dei dati crio-ET, inclusa la segmentazione manuale, il perfezionamento delle segmentazioni generate automaticamente e persino il prelievo di particelle all'interno dei tomogrammi. Il nostro studio dimostra la fattibilità della realtà virtuale come potente strumento per la pulizia delle segmentazioni, il prelievo di particelle e la segmentazione manuale nell'analisi dei dati crio-ET.
Per illustrare l'utilità del software VR per la segmentazione crio-ET, ci concentriamo sull'analisi della morfologia mitocondriale nelle cellule dell'epitelio pigmentato retinico (RPE1). I mitocondri fungono da eccellente banco di prova per la segmentazione grazie alla loro struttura complessa e alla presenza di caratteristiche facilmente quantificabili, come la distanza tra le membrane mitocondriali esterne e interne. Queste caratteristiche possono essere misurate con precisione utilizzando strumenti di analisi morfometrica di superficie12, fornendo metriche solide per valutare la qualità della segmentazione. Questo protocollo fornisce istruzioni dettagliate per la segmentazione dei dati crio-ET utilizzando syGlass, dimostrando la sua utilità all'interno della pipeline di segmentazione crio-ET. Incorporando la segmentazione basata sulla realtà virtuale nel flusso di lavoro crio-ET, miriamo a migliorare sia l'efficienza che l'accuratezza dell'analisi strutturale manuale in biologia cellulare.
Protocollo
1. Preparazione dei dati crio-ET per la segmentazione
- Converti i tomogrammi Cryo-ET grezzi in un formato di dati compatibile con syGlass, come le pile TIFF. Impostare il segnale in modo che le particelle siano bianche su nero ed eseguire l'equalizzazione dell'istogramma con ImageJ19.
NOTA: Per migliorare il rapporto segnale/rumore, si consiglia di denoificare i tomogrammi con software come Warp20, Topaz Denoise21, cryoCARE22,23 o IsoNet24 prima di essere importati nel software. - Avvia il software VR sul computer. Passare al menu File e selezionare Crea progetto.
- Clicca su Crea nuovo progetto | Aggiungi file. Passare alla posizione in cui sono salvati i file TIFF e importarli nel software.
- Quando viene richiesto di confermare se i file sono una serie temporale, fare clic su No.
- Assegna un nome al progetto, quindi fai clic su Salva per creare un nuovo progetto nell'elenco dei progetti.
- Fare doppio clic sul progetto per aprire il tomogramma e caricarlo nell'ambiente di realtà virtuale interattivo del software.
2. Configurazione della realtà virtuale (VR)
- Collega il visore VR e i controller manuali al computer.
- Segui le istruzioni sullo schermo per calibrare l'ambiente VR.
NOTA: La calibrazione si riferisce allo spazio tridimensionale in cui verrà eseguita la segmentazione. I parametri (lunghezza, larghezza e altezza) dello spazio di lavoro devono essere definiti prima che la proiezione VR venga visualizzata. La proiezione VR sarà visibile solo in questi parametri. - La segmentazione verrà eseguita solo all'interno dello spazio tridimensionale di lavoro definito. Assicurarsi che il campo visivo contenga l'area su cui si desidera lavorare nell'ambiente VR.
3. Ottimizzazione della visualizzazione 3D
- Fare clic sul pulsante Visualizzazione nell'interfaccia del software. Regola varie opzioni di visualizzazione come contrasto, finestra, luminosità e cursori di soglia per migliorare il segnale e ridurre al minimo il rumore.
- Usa i controller manuali per avvicinare il tomogramma o spingerlo via secondo necessità.
- Attiva lo strumento Taglia utilizzando il controller sinistro per ispezionare visivamente diverse fette all'interno del tomogramma.
4. Processo di segmentazione
- Navigare attraverso il tomogramma fino alla sezione desiderata in cui inizierà la segmentazione.
- Attiva l'opzione Regione di interesse (ROI) nel menu delle annotazioni utilizzando i controller manuali. All'interno del tomogramma apparirà una casella verde.
- Regola le dimensioni e la posizione della casella verde, spostandola nell'area da segmentare.
- Blocca il ROI utilizzando il controller sinistro e avvia la segmentazione. Dopo aver bloccato il ROI, lo strumento passa automaticamente a una modalità di verniciatura che consente la segmentazione dei dati volumetrici.
- Ingrandisci o rimpicciolisci il tomogramma per una segmentazione precisa.
- Regola le dimensioni del pennello ruotandolo in senso orario o antiorario per un controllo ottimale.
- Segmentare attentamente la ROI (ad esempio, le membrane mitocondriali) all'interno dell'area 3D selezionata. Se si verifica un errore durante la segmentazione, tenere premuto il grilletto del controller secondario per attivare la modalità di cancellazione , quindi cancellare l'errore utilizzando lo stesso movimento della segmentazione.
- Ripeti questo processo regione per regione fino a quando l'intero tomogramma non è segmentato.
5. Esportazione di dati segmentati e analisi
- Dopo aver completato la segmentazione, seleziona il progetto facendo clic su di esso per assicurarti che sia evidenziato.
- Fai clic sulla scheda Progetti nell'angolo in alto a sinistra, quindi seleziona ROI.
- Scegli di esportare l'intero volume o un ROI specifico. Specificare la posizione di esportazione per i dati segmentati.
- Carica e analizza i dati segmentati utilizzando il software preferito e genera cifre per la pubblicazione.
6. Importazione della maschera binaria nel software per la pulizia
- Eseguire i passaggi 1.1-1.6.
- Fare clic con il pulsante destro del mouse sul progetto e fare clic su Aggiungi dati maschera. Quindi, vai alla posizione in cui è salvata la segmentazione iniziale e importala nello stesso progetto.
- Attiva l'annotazione ROI per apportare modifiche alla segmentazione iniziale.
- Aggiungere/cancellare la segmentazione per ripulire la segmentazione iniziale.
- Regola le ROI delle maschere sotto l'opzione ROI nella scheda maschere . Per modificare il ROI della maschera da una all'altra, attiva l'unica opzione di sovrascrittura della maschera e passa alla modalità di pittura con il controller manuale. Questa modalità sovrascriverà solo i dati della maschera visibili.
- Esporta le ROI della maschera regolate utilizzando i passaggi descritti nei passaggi da 5.1 a 5.3.
7. Selezione delle coordinate delle particelle utilizzando il software VR
- Crea un progetto utilizzando il tomogramma (stack TIFF) utilizzando i passaggi descritti nella sezione 1.
- Fare clic su Annotazione | Conteggio, che apre una finestra con opzioni manuali o automatizzate .
- Per l'opzione manuale , scegli un colore per i conteggi e inizia a segnare le coordinate.
- Per l'opzione automatica, scegli una dimensione della cella e inserisci la risoluzione. Fai clic su Esegui conteggio automatico, modifica i conteggi (conserva le scelte buone e scarta quelle cattive) e salva i progressi.
- Esporta le coordinate di conteggio facendo clic sulla scheda progetto | annotazioni.
- Per l'origine dell'annotazione, scegliere Angolo in basso a sinistra e per Unità di annotazione, scegliere Voxels.
- Fare clic sulla cella dei punti di conteggio per evidenziare la cella.
- Fare clic su Esporta selezionato per salvare le coordinate della selezione in un file .csv.
NOTA: Questo file può essere utilizzato per estrarre i sottotomogrammi dal tomogramma per la media del sottotomogramma
Risultati
In questo studio, abbiamo segmentato tomogrammi contenenti mitocondri e organelli membranosi aggiuntivi (ad esempio, vescicole, reticolo endoplasmatico) utilizzando syGlass. I tomogrammi sono stati inizialmente ricostruiti in Warp utilizzando una proiezione pesata a 16.00 Å/pixel e sono stati sottoposti a correzione del cuneo mancante e denoising utilizzando il software IsoNet. I seguenti tomogrammi sono stati sottoposti a un'ulteriore elaborazione per l'importazione, come illustrato nella Figura 1A (vedere anche Video supplementare S1, Video supplementare S2, Video supplementare S3 e Video supplementare S4).
Dopo la pre-elaborazione, i tomogrammi in formato MRC sono stati convertiti in pile TIFF utilizzando ImageJ, con l'inversione del contrasto applicata per far apparire le membrane bianche su sfondo nero. L'equalizzazione dell'istogramma è stata quindi eseguita per migliorare ulteriormente il contrasto; Ciò consente anche una soglia più efficace. Le pile TIFF sono state importate e visualizzate in 3D all'interno di un ambiente VR immersivo, fornendo un'ispezione dettagliata delle strutture membranose utilizzando l'utensile di taglio nel software mostrato nella Figura 1B.
La segmentazione manuale è stata eseguita nel software, con la finestra impostata su auto e le regolazioni di luminosità/soglia effettuate per ottimizzare la visibilità delle funzioni cellulari. Le ROI intorno ai mitocondri e ad altre strutture sono state definite utilizzando i controller VR. Lo strumento di segmentazione ha permesso di delineare con precisione i confini della membrana, correggendo gli errori utilizzando la funzione di cancellazione. La navigazione sezione per fetta o in 3D utilizzando lo strumento ROI per inscatolare le regioni in tutto il tomogramma, combinata con lo strumento pennello regolabile, ha garantito una segmentazione accurata delle membrane mitocondriali e di altri organelli, come mostrato nella Figura 1C.
I dati segmentati sono stati visualizzati generando una mesh utilizzando l'opzione delle superfici dello strumento ROI, con le iterazioni di levigatura impostate su 12 e il livello di risoluzione impostato su 3. I rendering 3D finali mostrano chiaramente le strutture mitocondriali, comprese le membrane esterne e interne, le cristae e i depositi di fosfato di calcio, come mostrato nella Figura 1D (vedi anche Video supplementare S5, Video supplementare S6, Video supplementare S7 e Video supplementare S8).
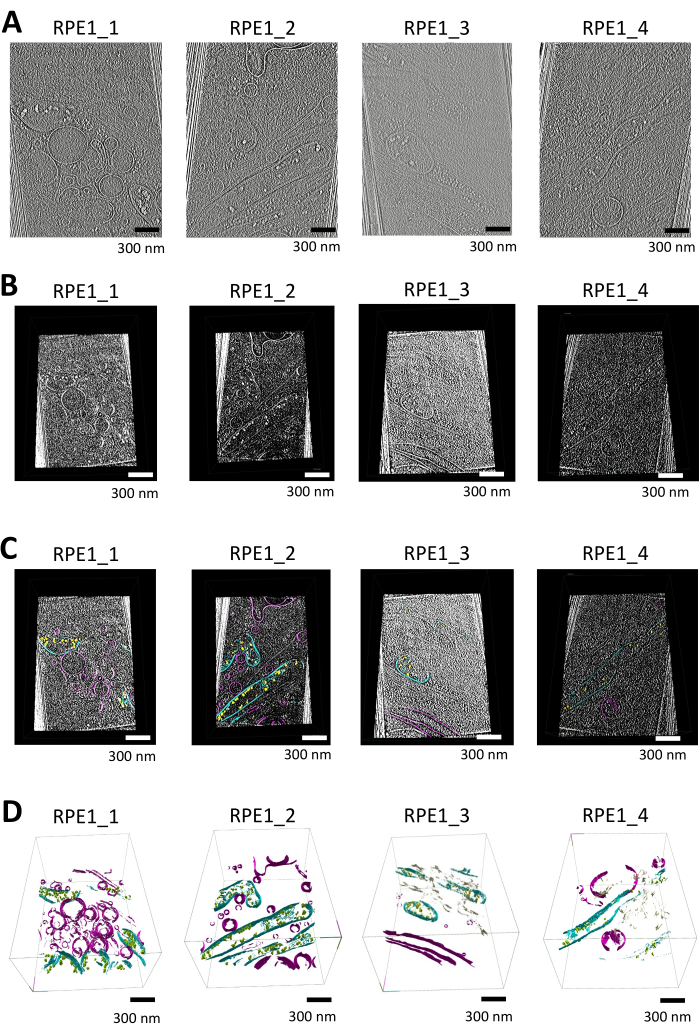
Figura 1: Flusso di lavoro della visualizzazione, segmentazione e rendering 3D del tomogramma utilizzando la clessidra. (A) Sezioni tomografiche di cellule RPE1 a bordo sottile. Tomogrammi corretti IsoNet ricostruiti in Warp e utilizzati per la segmentazione con syGlass, visualizzati con IMOD25. I tomogrammi sono stati raccolti su un microscopio Titan-Krios da 300 keV dotato di un rivelatore K3 e focalizzato sui bordi sottili delle cellule dell'epitelio retinico. Ogni tomogramma contiene almeno un mitocondrio insieme a vari altri organelli legati alla membrana. Queste immagini sono immagini fisse di video mostrati in Video supplementare S1, Video supplementare S2, Video supplementare S3 e Video supplementare S4. (B) Tomogrammi corrispondenti visualizzati nel software. Fetta tomografica creata utilizzando lo strumento di taglio nel software VR dopo aver ottimizzato la soglia, la luminosità e la finestratura, rivelando strutture cellulari distinte. (C) Tomogrammi visualizzati nel software con membrane segmentate. Sezione tomografica creata con lo strumento di taglio nel software VR, sovrapposta alla segmentazione corrispondente. I depositi di fosfato di calcio sono mostrati in giallo, le membrane mitocondriali in ciano e altre membrane come le vescicole e la membrana plasmatica in viola. (D) Rendering 3D delle membrane segmentate in syGlass. Le membrane mitocondriali, comprese le cristae, le membrane mitocondriali esterne e interne, sono mostrate in ciano. I depositi di fosfato di calcio sono in giallo o verde, le vescicole e la membrana plasmatica sono in viola e il reticolo endoplasmatico è in marrone chiaro. Queste immagini sono immagini fisse di video mostrati in Video supplementari S5, Video supplementari S6, Video supplementari S7 e Video supplementari S8. Clicca qui per visualizzare una versione più grande di questa figura.
Video supplementare S1: Tomogramma del campione RPE-1_1 utilizzato per la segmentazione in questo studio. Questo tomogramma viene ricostruito a 16 Å/pixel utilizzando Warp, quindi denoised e corretto per il cuneo mancante con IsoNet. Clicca qui per scaricare questo file.
Video supplementare S2: Tomogramma del campione RPE-1_2 utilizzato per la segmentazione in questo studio. Questo tomogramma viene ricostruito a 16 Å/pixel utilizzando Warp, quindi denoised e corretto per il cuneo mancante con IsoNet. Clicca qui per scaricare questo file.
Video supplementare S3: Tomogramma del campione RPE-1_3 utilizzato per la segmentazione in questo studio. Questo tomogramma viene ricostruito a 16 Å/pixel utilizzando Warp, quindi denoised e corretto per il cuneo mancante con IsoNet. Clicca qui per scaricare questo file.
Video supplementare S4: Tomogramma del campione RPE-1_4 utilizzato per la segmentazione in questo studio. Questo tomogramma viene ricostruito a 16 Å/pixel utilizzando Warp, quindi denoised e corretto per il cuneo mancante con IsoNet. Clicca qui per scaricare questo file.
Video supplementare S5: La segmentazione risultante del campione RPE-1_1 dopo la generazione delle superfici. Le membrane mitocondriali sono mostrate in ciano; i fosfati di calcio all'interno dei mitocondri sono raffigurati in giallo; il reticolo endoplasmatico è mostrato in marrone chiaro; e altre membrane sono visualizzate in viola. Clicca qui per scaricare questo file.
Video supplementare S6: La segmentazione risultante del campione RPE-1_2 dopo la generazione delle superfici. Le membrane mitocondriali sono mostrate in ciano; i fosfati di calcio all'interno dei mitocondri sono raffigurati in giallo; il reticolo endoplasmatico è mostrato in marrone chiaro; e altre membrane sono visualizzate in viola. Clicca qui per scaricare questo file.
Video supplementare S7: la segmentazione risultante del campione RPE-1_3 dopo la generazione delle superfici. Le membrane mitocondriali sono mostrate in ciano; i fosfati di calcio all'interno dei mitocondri sono raffigurati in giallo; il reticolo endoplasmatico è mostrato in marrone chiaro; e altre membrane sono visualizzate in viola. Clicca qui per scaricare questo file.
Video supplementare S8: La segmentazione risultante del campione RPE-1_4 dopo la generazione delle superfici. Le membrane mitocondriali sono mostrate in ciano; i fosfati di calcio all'interno dei mitocondri sono raffigurati in giallo; il reticolo endoplasmatico è mostrato in marrone chiaro; e altre membrane sono visualizzate in viola. Clicca qui per scaricare questo file.
Discussione
In questo lavoro, abbiamo dimostrato come la realtà virtuale, in particolare utilizzando syGlass, possa essere integrata nella pipeline cryo-ET per segmentare efficacemente le strutture cellulari. Sebbene la nostra attenzione si sia concentrata principalmente sulle membrane, non vi è alcuna limitazione intrinseca che impedisca la segmentazione di altre strutture cellulari, come filamenti o ribosomi, in quanto è possibile regolare la forma e le dimensioni del pennello e la soglia per garantire che vengano contrassegnati solo i voxel corrispondenti agli oggetti cellulari desiderati. Uno dei principali vantaggi della realtà virtuale nella segmentazione è l'interazione intuitiva e pratica con i dati, che consente agli utenti di visualizzare e manipolare i volumi come se fossero fisicamente al loro interno. I metodi tradizionali di segmentazione manuale o di pulizia delle maschere di segmentazione iniziale in genere comportano il lavoro diretto su un computer, che può essere meno coinvolgente e più lento da annotare.
Incorporando la realtà virtuale nel flusso di lavoro crio-ET, gli utenti possono non solo interagire rapidamente con le maschere di segmentazione generate da altri software, ma anche utilizzare la realtà virtuale per guidare la segmentazione di strutture parzialmente segmentate e ripulire in modo efficiente i falsi positivi. Attualmente, la segmentazione manuale è ancora necessaria per la maggior parte dei casi d'uso di annotazione e il flusso di lavoro presentato consente agli utenti di generare segmentazioni adatte all'analisi a valle con maggiore facilità e velocità. Per questo studio abbiamo utilizzato il visore HTC Vive VR, ma il software è compatibile con i dispositivi che supportano SteamVR o OculusVR.
Per un'applicazione ottimale di questo protocollo, i dati crio-ET devono soddisfare criteri specifici, come tomogrammi di alta qualità con un elevato rapporto segnale/rumore (SNR). L'SNR è un parametro essenziale, poiché la chiarezza delle caratteristiche strutturali influenza direttamente l'efficacia della segmentazione manuale in VR. Le fasi di pre-elaborazione, come la correzione del cuneo mancante e il denoising, sono parti fondamentali del flusso di lavoro; in questo protocollo, abbiamo utilizzato IsoNet per questi scopi. I tomogrammi devono essere ricostruiti con una risoluzione adeguata, utilizzando una dimensione del voxel che fornisca dettagli strutturali sufficienti e una separazione sufficiente tra le strutture cellulari per consentire una segmentazione efficace tra le strutture, mantenendo ulteriormente le dimensioni dei dati gestibili per la visualizzazione VR. Inoltre, è necessario applicare il contrasto invertito con le strutture cellulari che appaiono bianche su sfondo nero e l'equalizzazione dell'istogramma per migliorare la visibilità delle membrane e di altre strutture all'interno dell'ambiente syGlass.
Diversi punti critici devono essere considerati per garantire il successo di questo protocollo. In primo luogo, durante la pre-elaborazione dei dati, è fondamentale applicare accuratamente l'inversione del contrasto e l'equalizzazione dell'istogramma; Regolazioni improprie possono portare a una visualizzazione non ottimale in VR, rendendo difficile la segmentazione. In secondo luogo, all'interno del software VR, le regolazioni appropriate delle impostazioni di finestra, luminosità e soglia sono essenziali per una visualizzazione ottimale delle strutture. Gli utenti dovrebbero sperimentare queste impostazioni per ottenere i migliori risultati per i loro set di dati specifici e le configurazioni VR.
Quando si tratta di risoluzione dei problemi, gli utenti possono riscontrare problemi come limitazioni delle prestazioni del sistema VR, soprattutto quando si gestiscono set di dati di tomogrammi di grandi dimensioni. Se l'ambiente VR diventa lento o non risponde, prendi in considerazione la possibilità di sottocampionare i dati o segmentare il tomogramma in sezioni più piccole regolando il riquadro di delimitazione negli strumenti ROI in syGlass e spostandolo lungo il tomogramma durante l'esecuzione della segmentazione. Il cursore delle prestazioni nel menu di visualizzazione può anche essere regolato per ridurre il ritardo durante il processo di segmentazione. Inoltre, per mitigare la cinetosi, gli utenti possono regolare le impostazioni VR per ridurre gli effetti di movimento o fare pause regolari durante le sessioni di segmentazione.
Il nostro protocollo di segmentazione basato su realtà virtuale offre vantaggi significativi agli attuali flussi di lavoro di segmentazione crio-ET. I metodi convenzionali di segmentazione manuale spesso comportano l'annotazione 2D su singole sezioni, che può richiedere molto tempo e potrebbe non catturare completamente la continuità tridimensionale13. Inoltre, la segmentazione basata sulla realtà virtuale introduce una visione molto più immersiva delle strutture cellulari, facilitando la visualizzazione. Le segmentazioni automatizzate dell'apprendimento automatico stanno emergendo come un metodo potente per ottenere segmentazioni dai tomogrammi cellulari 14,16,17, sebbene gli elevati livelli di rumore e le strutture complesse presenti nei dati crio-ET portino a lacune e falsi positivi che richiedono un intervento manuale. Questo protocollo offre un approccio alternativo per segmentare manualmente i tomogrammi crioelettronici, per generare segmentazioni iniziali che possono potenzialmente essere utilizzate come dati di addestramento per altri software di reti neurali o per ripulire le segmentazioni iniziali generate da altri approcci automatizzati.
In conclusione, questo studio evidenzia la segmentazione basata sulla realtà virtuale come uno strumento promettente per l'analisi e l'educazione dei dati crio-ET, offrendo una maggiore efficienza e un'esperienza utente più coinvolgente. Con l'ulteriore sviluppo, la tecnologia VR ha il potenziale per rivoluzionare il modo in cui interpretiamo e diffondiamo le scoperte scientifiche di strutture cellulari complesse nei set di dati crio-ET, fornendo una valida alternativa ai tradizionali metodi di segmentazione e istruzione.
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Questo lavoro è stato svolto presso il National Center for CryoEM Access and Training (NCCAT) e il Simons Electron Microscopy Center situato presso il New York Structural Biology Center, supportati da NIH (Common Fund U24GM129539, U24GM139171 e NIGMS R24GM154192), dalla Simons Foundation (SF349247) e dall'Assemblea dello Stato di New York.
Materiali
| Name | Company | Catalog Number | Comments |
| CryoET Data | Format:TIFF-stack, TIFF | ||
| HTC VIVE Cosmos | HTC | 99HARL000-00 | https://www.vive.com/sea/product/vive-cosmos/features/ |
| Intel(R) Core(TM) i7-10870H CPU @ 2.20 GHz 2.21 GHz | Intel | https://ark.intel.com/content/www/us/en/ark/products/208018/intel-core-i7-10870h-processor-16m-cache-up-to-5-00-ghz.html | |
| NVIDIA GeForce RTX 3070 Laptop GPU | NVIDIA | https://www.nvidia.com/en-us/geforce/laptops/compare/30-series/ | |
| syGlass Software | syGlass | syGlass Software installed on a compatible Windows PC | |
| VIVE Cosmos Hand Controllers | HTC | 99HAFR001-00 | https://www.vive.com/us/accessory/cosmos-controller-right/ |
| Windows 11 Home | Microsoft | Microsoft Windows 11 Home |
Riferimenti
- Plitzko, J. M., Schuler, B., Selenko, P. Structural biology outside the box-inside the cell. Curr Opin Struct Biol. 46, 110-121 (2017).
- Beck, M., Baumeister, W. Cryo-electron tomography: can it reveal the molecular sociology of cells in atomic detail. Trends Cell Biol. 26 (11), 825-837 (2016).
- Lam, V., Villa, E. Practical approaches for cryo-FIB milling and applications for cellular cryo-electron tomography. Methods Mol Biol. 2215, 49-82 (2021).
- Berger, C., et al. Cryo-electron tomography on focused ion beam lamellae transforms structural cell biology. Nat Methods. 20 (4), 499-511 (2023).
- Watson, A. J. I., Bartesaghi, A. Advances in cryo-ET data processing: meeting the demands of visual proteomics. Curr Opin Struct Biol. 87, 102861 (2024).
- Pyle, E., Zanetti, G. Current data processing strategies for cryo-electron tomography and subtomogram averaging. Biochem J. 478 (10), 1827-1845 (2021).
- Wan, W., Briggs, J. A. G. Cryo-electron tomography and subtomogram averaging. Methods Enzymol. 579, 329-367 (2016).
- Ni, T., Liu, K., Zhang, J., Atanasov, I., Mladenov, M. G., Jiang, W. High-resolution in situ structure determination by cryo-electron tomography and subtomogram averaging using emClarity. Nat Protoc. 17 (2), 421-444 (2022).
- Obr, M., et al. Exploring high-resolution cryo-ET and subtomogram averaging capabilities of contemporary DEDs. J Struct Biol. 214 (2), 107852 (2021).
- Tegunov, D., Xue, L., Dienemann, C., Cramer, P., Mahamid, J. Multi-particle cryo-EM refinement with M visualizes ribosome-antibiotic complex at 3.5 Å in cells. Nat Methods. 18 (2), 186-193 (2021).
- Salfer, M., Collado, J. F., Baumeister, W., Fernández-Busnadiego, R., Martínez-Sánchez, A. Reliable estimation of membrane curvature for cryo-electron tomography. PLoS Comput Biol. 16 (8), e1007962 (2020).
- Barad, B. A., et al. Quantifying organellar ultrastructure in cryo-electron tomography using a surface morphometrics pipeline. J Cell Biol. 222 (2), e202204093 (2023).
- Martinez-Sanchez, A., Garcia, I., Asano, S., Lucic, V., Fernandez, J. J. Robust membrane detection based on tensor voting for electron tomography. J Struct Biol. 186 (1), 49-61 (2014).
- Heebner, J. E., et al. Deep learning-based segmentation of cryo-electron tomograms. J Vis Exp. (189), e64435 (2022).
- Garza-López, E., Robles-Flores, M. Protocols for generating surfaces and measuring 3D organelle morphology using Amira. Cells. 11 (1), 65 (2021).
- Chen, M., et al. Convolutional neural networks for automated annotation of cellular cryo-electron tomograms. Nat Methods. 14 (10), 983-985 (2017).
- Lamm, L., et al. MemBrain v2: an end-to-end tool for the analysis of membranes in cryo-electron tomography. bioRxiv. , (2024).
- Pidhorskyi, S., Morehead, M., Jones, Q., Spirou, G., Doretto, G. syGlass: interactive exploration of multidimensional images using virtual reality head-mounted displays. arXiv. , (2018).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Tegunov, D., Cramer, P. Real-time cryo-electron microscopy data preprocessing with Warp. Nat Methods. 16 (11), 1146-1152 (2019).
- Bepler, T., Kelley, K., Noble, A. J., Berger, B. Topaz-Denoise: general deep denoising models for cryoEM and cryoET. Nat Commun. 11, 5208 (2020).
- Buchholz, T. -. O., Berninger, L., Maurer, M., Jug, F. Cryo-CARE: content-aware image restoration for cryo-transmission electron microscopy data. Proc IEEE Int Symp Biomed Imaging. 16, 502-506 (2019).
- Buchholz, T. -. O., Jordan, M., Pigino, G., Jug, F. Content-aware image restoration for electron microscopy. Methods Cell Biol. 152, 277-289 (2019).
- Liu, Y. -. T., et al. Isotropic reconstruction for electron tomography with deep learning. Nat Commun. 13, 6482 (2022).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. J Struct Biol. 116 (1), 71-76 (1996).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon