Method Article
Nutzung von Virtual Reality für die immersive Segmentierung und Analyse von Kryo-Elektronentomographie-Daten
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Die Kryo-Elektronentomographie (Kryo-ET) ermöglicht die 3D-Visualisierung der zellulären Ultrastruktur mit Nanometerauflösung, aber die manuelle Segmentierung bleibt zeitaufwändig und komplex. Wir stellen einen neuartigen Workflow vor, der fortschrittliche Virtual-Reality-Software zur Segmentierung von Kryo-ET-Tomogrammen integriert und seine Wirksamkeit durch die Segmentierung von Mitochondrien in Säugetierzellen demonstriert.
Zusammenfassung
Die Kryo-Elektronentomographie (Kryo-ET) ist eine leistungsstarke Technik, um die Ultrastruktur von Zellen in drei Dimensionen (3D) mit Nanometerauflösung sichtbar zu machen. Die manuelle Segmentierung zellulärer Komponenten in Kryo-ET-Daten bleibt jedoch aufgrund ihrer Komplexität und ihres Zeitaufwands ein erheblicher Engpass. In dieser Arbeit stellen wir einen neuartigen Segmentierungs-Workflow vor, der fortschrittliche Virtual-Reality-Software (VR) integriert, um sowohl die Effizienz als auch die Genauigkeit der Segmentierung von Kryo-ET-Datensätzen zu verbessern. Dieser Workflow nutzt ein immersives VR-Tool mit intuitiver 3D-Interaktion, das es den Nutzern ermöglicht, komplexe zelluläre Strukturen in einer natürlicheren und interaktiveren Umgebung zu navigieren und zu kommentieren. Um die Effektivität des Workflows zu bewerten, haben wir ihn auf die Segmentierung von Mitochondrien in retinalen Pigmentepithelzellen (RPE1) angewendet. Mitochondrien, die für die zelluläre Energieproduktion und Signalübertragung unerlässlich sind, weisen dynamische morphologische Veränderungen auf, was sie zu einer idealen Testprobe macht. Die VR-Software ermöglichte eine präzise Abgrenzung der Mitochondrienmembranen und internen Strukturen und ermöglichte so eine nachgelagerte Analyse der segmentierten Membranstrukturen. Wir zeigen, dass dieser VR-basierte Segmentierungs-Workflow die Benutzererfahrung erheblich verbessert und gleichzeitig eine genaue Segmentierung komplizierter zellulärer Strukturen in Kryo-ET-Daten beibehält. Dieser Ansatz ist vielversprechend für breite Anwendungen in der strukturellen Zellbiologie und in der wissenschaftlichen Bildung und bietet ein transformatives Werkzeug für Forscher, die sich mit detaillierten zellulären Analysen beschäftigen.
Einleitung
Die Kryo-Elektronentomographie (Kryo-ET) hat unsere Fähigkeit revolutioniert, zelluläre Komponenten in ihrem nahezu nativen Zustand mit hoher Auflösung sichtbar zu machen 1,2. Diese leistungsstarke Technik ermöglicht es Forschern, die zelluläre Ultrastruktur zu charakterisieren und bietet beispiellose Einblicke in die zelluläre Architektur und Funktion. Die Kryo-ET ist jedoch nicht ohne Einschränkungen, vor allem durch die Anforderung, dass die Proben dünn genug sein müssen, um elektronentransparent zu sein (typischerweise <0,5 μm) für die Bildgebung in standardmäßigen kryofähigen Transmissionselektronenmikroskopen. Jüngste Fortschritte beim kryofokussierten Ionenstrahlfräsen haben die Ausdünnung dicker Proben für die Kryo-ET-Analyse ermöglicht 3,4.
Typische Kryo-ET-Arbeitsabläufe beginnen mit der Entnahme einer Tilt-Serie, bei der die Probe in verschiedenen Winkeln von +60° bis -60° abgebildet wird. Diese Bilder werden dann rechnerisch ausgerichtet und zurückprojiziert, um ein dreidimensionales (3D) Volumen oder Tomogramm 5,6,7 zu erzeugen. Dieses Tomogramm dient als detaillierte 3D-Karte der zellulären Landschaft und bietet sowohl räumliche als auch zeitliche Informationen über zelluläre Strukturen. Eine weitere Verfeinerung durch Subtomogramm-Mittelwertbildung, bei der mehrere Kopien derselben Struktur ausgerichtet und gemittelt werden, kann die Auflösungsgrenzen noch weiter verschieben und manchmal eine Sub-Nanometer-Auflösungvon 7,8,9,10 erreichen.
Ein entscheidender Schritt bei der Extraktion aussagekräftiger biologischer Informationen aus diesen Tomogrammen ist die Segmentierung. Bei diesem Prozess werden bestimmte zelluläre Strukturen, wie z. B. Membranen, innerhalb des 3D-Volumens annotiert. Die Segmentierung ermöglicht fortschrittliche Analysen, einschließlich der Berechnung von Intermembranabständen und Membrankrümmung, und liefert wertvolle Einblicke in zelluläre Prozesse11,12. Obwohl mehrere Softwarepakete für diese Aufgabe verfügbar sind, darunter Dragonfly, Amira, MemBrain, EMAN2 und tomomemsegtv 13,14,15,16,17, bleibt der Segmentierungsprozess ein erheblicher Engpass bei der Datenanalyse von Kryo-ETs. Es handelt sich oft um einen arbeits- und zeitaufwändigen manuellen Prozess, der möglicherweise Wochen bis Monate dauern kann. Viele dieser Pakete bieten automatische Segmentierungsfunktionen, erfordern jedoch häufig umfangreiche manuelle Korrekturen, um Fehlalarme zu entfernen, ein Prozess, der mühsam und unintuitiv sein kann, wenn er Slice für Slice oder in 3D durchgeführt wird.
Wir schlagen einen alternativen Ansatz vor, indem wir die Virtual-Reality-Technologie (VR) nutzen, um diese Herausforderungen zu bewältigen. VR bietet eine immersive und interaktive Methode zur Datenvisualisierung, die es dem Benutzer ermöglicht, durch das tomographische Volumen zu navigieren, als ob er sich in der zellulären Umgebung selbst befände. Dieser Ansatz bietet auch eine wertvolle Plattform für die wissenschaftliche Bildung und die wissenschaftliche Erforschung und Entdeckung von In-situ-Kryo-ET-Daten, indem er ein immersives und einzigartig ansprechendes Erlebnis bietet. In dieser Arbeit stellen wir ein Protokoll zur Kryo-ET-Datensegmentierung mit syGlass18 vor, einer VR-Software für die wissenschaftliche Visualisierung. Diese Software bietet ein umfassendes Toolkit für die Datenanalyse von Kryo-ETs, einschließlich manueller Segmentierung, Verfeinerung automatisch generierter Segmentierungen und sogar Partikelauswahl innerhalb von Tomogrammen. Unsere Studie zeigt, dass VR als leistungsstarkes Werkzeug für die Reinigung von Segmentierungen, die Partikelauswahl und die manuelle Segmentierung in der Kryo-ET-Datenanalyse geeignet ist.
Um den Nutzen der VR-Software für die Kryo-ET-Segmentierung zu veranschaulichen, konzentrieren wir uns auf die Analyse der mitochondrialen Morphologie in retinalen Pigmentepithelzellen (RPE1). Mitochondrien eignen sich aufgrund ihrer komplexen Struktur und des Vorhandenseins leicht quantifizierbarer Merkmale, wie z. B. des Abstands zwischen äußerer und innerer Mitochondrienmembran, als hervorragender Testfall für die Segmentierung. Diese Merkmale können mit Hilfe von Oberflächenmorphometrie-Analysewerkzeugen12 genau gemessen werden, die robuste Metriken zur Bewertung der Segmentierungsqualität liefern. Dieses Protokoll bietet eine Schritt-für-Schritt-Anleitung für die Segmentierung von Kryo-ET-Daten mit syGlass und demonstriert damit seinen Nutzen innerhalb der Kryo-ET-Segmentierungspipeline. Durch die Integration von VR-basierter Segmentierung in den Kryo-ET-Workflow wollen wir sowohl die Effizienz als auch die Genauigkeit der manuellen Strukturanalyse in der Zellbiologie verbessern.
Protokoll
1. Aufbereitung von Kryo-ET-Daten für die Segmentierung
- Konvertieren Sie rohe Cryo-ET-Tomogramme in ein Datenformat, das mit syGlass kompatibel ist, z. B. TIFF-Stacks. Stellen Sie das Signal so ein, dass die Partikel weiß auf schwarz sind, und führen Sie die Histogrammentzerrung mit ImageJ19 durch.
HINWEIS: Um das Signal-Rausch-Verhältnis zu verbessern, wird empfohlen, die Tomogramme mit Software wie Warp20, Topaz Denoise21, cryoCARE22,23 oder IsoNet24 zu entrauschen, bevor sie in die Software importiert werden. - Starten Sie die VR-Software auf dem Computer. Navigieren Sie zum Menü Datei , und wählen Sie Projekt erstellen aus.
- Klicken Sie auf Neues Projekt erstellen | Dateien hinzufügen. Navigieren Sie zu dem Speicherort, an dem die TIFF-Dateien gespeichert sind, und importieren Sie sie in die Software.
- Wenn Sie aufgefordert werden, zu bestätigen, ob es sich bei den Dateien um Zeitreihen handelt, klicken Sie auf Nein.
- Benenne das Projekt und klicke dann auf "Sichern ", um ein neues Projekt in der Projektliste zu erstellen.
- Doppelklicken Sie auf das Projekt, um das Tomogramm zu öffnen und es in die interaktive Virtual-Reality-Umgebung der Software zu laden.
2. Einrichten von Virtual Reality (VR)
- Verbinden Sie das VR-Headset und die Handcontroller mit dem Computer.
- Befolgen Sie die Anweisungen auf dem Bildschirm, um die VR-Umgebung zu kalibrieren.
HINWEIS: Die Kalibrierung bezieht sich auf den dreidimensionalen Raum, in dem die Segmentierung durchgeführt wird. Die Parameter (Länge, Breite und Höhe) des Arbeitsbereichs müssen definiert werden, bevor die VR-Projektion angezeigt wird. Die VR-Projektion ist nur in diesen Parametern sichtbar. - Die Segmentierung erfolgt nur innerhalb des definierten dreidimensionalen Arbeitsraums. Stellen Sie sicher, dass das Sichtfeld den Bereich enthält, der in der VR-Umgebung bearbeitet werden soll.
3. Optimierung der 3D-Visualisierung
- Klicken Sie auf die Schaltfläche Visualisierung in der Softwareoberfläche. Passen Sie verschiedene Visualisierungsoptionen an, z. B. Schieberegler für Kontrast, Fenster, Helligkeit und Schwellenwert, um das Signal zu verbessern und das Rauschen zu minimieren.
- Verwenden Sie die Handcontroller, um das Tomogramm je nach Bedarf näher zu ziehen oder wegzuschieben.
- Aktivieren Sie das Schnittwerkzeug mit dem linken Controller, um verschiedene Slices innerhalb des Tomogramms visuell zu untersuchen.
4. Prozess der Segmentierung
- Navigieren Sie durch das Tomogramm zum gewünschten Segment, in dem die Segmentierung beginnt.
- Aktivieren Sie die Option Region of Interest (ROI) im Anmerkungsmenü mit den Handcontrollern. Im Tomogramm erscheint ein grüner Kasten.
- Passen Sie die Größe und Position des grünen Quaders an und verschieben Sie es in den Bereich, der segmentiert werden soll.
- Sperren Sie den ROI mit dem linken Controller und initiieren Sie die Segmentierung. Nach dem Sperren des ROI wechselt das Werkzeug automatisch in einen Malmodus , der eine Segmentierung der volumetrischen Daten ermöglicht.
- Vergrößern oder verkleinern Sie das Tomogramm für eine präzise Segmentierung.
- Passen Sie die Größe des Pinsels an, indem Sie ihn für eine optimale Steuerung im oder gegen den Uhrzeigersinn drehen.
- Segmentieren Sie den ROI (z. B. mitochondriale Membranen) innerhalb des ausgewählten 3D-Bereichs sorgfältig. Wenn während der Segmentierung ein Fehler auftritt, halten Sie den Auslöser des sekundären Controllers gedrückt, um den Löschmodus zu aktivieren, und löschen Sie den Fehler dann mit der gleichen Bewegung wie bei der Segmentierung.
- Wiederholen Sie diesen Vorgang Region für Region, bis das gesamte Tomogramm segmentiert ist.
5. Exportieren von segmentierten Daten und Analysen
- Nachdem Sie die Segmentierung abgeschlossen haben, wählen Sie das Projekt aus, indem Sie darauf klicken, um sicherzustellen, dass es hervorgehoben wird.
- Klicken Sie auf die Registerkarte Projekte in der oberen linken Ecke und wählen Sie dann ROIs aus.
- Wählen Sie, ob Sie entweder das gesamte Volumen oder einen bestimmten ROI exportieren möchten. Geben Sie den Exportspeicherort für die segmentierten Daten an.
- Laden und analysieren Sie die segmentierten Daten mit der Software Ihrer Wahl und generieren Sie Zahlen für die Veröffentlichung.
6. Importieren der Binärmaske in die Software zur Bereinigung
- Führen Sie die Schritte 1.1-1.6 aus.
- Klicken Sie mit der rechten Maustaste auf das Projekt und klicken Sie auf Maskendaten hinzufügen. Navigieren Sie dann zu dem Speicherort, an dem die ursprüngliche Segmentierung gespeichert ist, und importieren Sie sie unter demselben Projekt.
- Nutzen Sie die ROI-Annotation, um Änderungen an der ursprünglichen Segmentierung vorzunehmen.
- Segmentierung hinzufügen/löschen, um die ursprüngliche Segmentierung zu bereinigen.
- Passen Sie die ROIs der Masken unter der Option ROI auf der Registerkarte "Masken" an. Um den ROI der Maske von einer zur anderen zu ändern, aktivieren Sie die Option "Nur Maske überschreiben " und wechseln Sie mit dem Handcontroller in den Malmodus . In diesem Modus werden nur sichtbare Maskendaten überschrieben.
- Exportieren Sie die angepassten ROIs der Maske mithilfe der in den Schritten 5.1 bis 5.3 beschriebenen Schritte.
7. Auswählen von Partikelkoordinaten mit der VR-Software
- Erstellen Sie ein Projekt mit Tomogramm (TIFF-Stack), indem Sie die in Abschnitt 1 beschriebenen Schritte ausführen.
- Klicken Sie auf Anmerkung | Count, wodurch ein Fenster mit manuellen oder automatisierten Optionen geöffnet wird.
- Wählen Sie für die manuelle Option eine Farbe für die Zählungen und beginnen Sie mit dem Markieren der Koordinaten.
- Wählen Sie für die automatisierte Option eine Zellengröße aus und geben Sie die Auflösung ein. Klicken Sie auf Automatische Zählung ausführen, bearbeiten Sie die Zählungen (gute Picks behalten und schlechte verwerfen) und speichern Sie den Fortschritt.
- Exportieren Sie die Zählkoordinaten, indem Sie auf die Registerkarte Projekt | Anmerkungen klicken.
- Wählen Sie für den Annotationsursprung die Option Bottom Left Corner (Untere linke Ecke ) und für Annotationseinheiten (Annotation Units) die Option Voxels (Voxels) aus.
- Klicken Sie auf die Zelle mit den Zählpunkten , um die Zelle zu markieren.
- Klicken Sie auf Ausgewählte exportieren , um die Koordinaten der Auswahl in einer .csv Datei zu speichern.
HINWEIS: Diese Datei kann verwendet werden, um Subtomogramme aus dem Tomogramm für die Subtomogramm-Mittelung zu extrahieren
Ergebnisse
In dieser Studie segmentierten wir Tomogramme, die Mitochondrien und zusätzliche membranöse Organellen (z.B. Vesikel, endoplasmatisches Retikulum) enthielten, mit syGlass. Die Tomogramme wurden zunächst in Warp mittels Weighted-Back-Projektion bei 16.00 Å/Pixel rekonstruiert und mit der Software IsoNet einer fehlenden Keilkorrektur und Rauschunterdrückung unterzogen. Die folgenden Tomogramme wurden für den Import einer zusätzlichen Verarbeitung unterzogen, wie in Abbildung 1A gezeigt (siehe auch Ergänzendes Video S1, Ergänzendes Video S2, Ergänzendes Video S3 und Ergänzendes Video S4).
Nach der Vorverarbeitung wurden Tomogramme im MRC-Format mit ImageJ in TIFF-Stacks konvertiert, wobei die Kontrastinversion angewendet wurde, um die Membranen auf schwarzem Hintergrund weiß erscheinen zu lassen. Anschließend wurde eine Histogramm-Entzerrung durchgeführt, um den Kontrast weiter zu verbessern. Dies ermöglicht auch eine effektivere Festlegung von Schwellenwerten. Die TIFF-Stacks wurden importiert und in 3D in einer immersiven VR-Umgebung visualisiert, um eine detaillierte Inspektion membranöser Strukturen mit dem Cut-Tool in der in Abbildung 1B gezeigten Software zu ermöglichen.
In der Software wurde eine manuelle Segmentierung durchgeführt, wobei die Fensterfunktion auf "Auto" eingestellt wurde und Helligkeits-/Schwellenwertanpassungen vorgenommen wurden, um die Sichtbarkeit der Mobilfunkfunktionen zu optimieren. ROIs um Mitochondrien und andere Strukturen wurden mit Hilfe der VR-Controller definiert. Das Segmentierungswerkzeug ermöglichte eine präzise Abgrenzung der Membrangrenzen, wobei Fehler mit der Löschfunktion korrigiert wurden. Die Schicht-für-Schicht-Navigation oder in 3D mit dem ROI-Tool zum Einrahmen von Regionen im gesamten Tomogramm in Kombination mit dem einstellbaren Pinselwerkzeug gewährleistete eine genaue Segmentierung der Mitochondrienmembranen und anderer Organellen, wie in Abbildung 1C gezeigt.
Die segmentierten Daten wurden visualisiert, indem ein Netz mit der Oberflächenoption des ROI-Werkzeugs generiert wurde, wobei die Glättungsiterationen auf 12 und die Auflösungsstufe auf 3 festgelegt waren. Die endgültigen 3D-Renderings zeigen deutlich die mitochondrialen Strukturen, einschließlich der äußeren und inneren Membranen, Crista und Calciumphosphatablagerungen, wie in Abbildung 1D gezeigt (siehe auch Ergänzendes Video S5, Ergänzendes Video S6, Ergänzendes Video S7 und Ergänzendes Video S8).
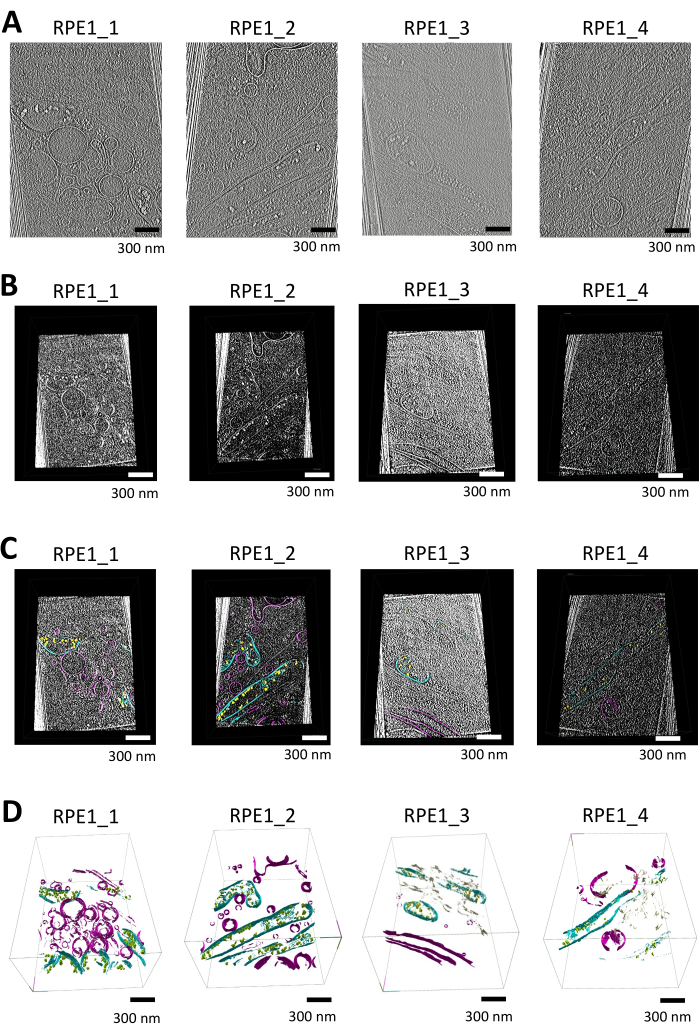
Abbildung 1: Arbeitsablauf der Tomogramm-Visualisierung, Segmentierung und 3D-Rendering mit Syglass. (A) Tomographische Schnitte von RPE1-Zellen mit dünnen Kanten. IsoNet-korrigierte Tomogramme, die in Warp rekonstruiert und für die Segmentierung mit syGlass verwendet wurden, visualisiert mit IMOD25. Die Tomogramme wurden an einem 300 keV Titan-Krios-Mikroskop gesammelt, das mit einem K3-Detektor ausgestattet war, und auf die dünnen Ränder von retinalen Epithelzellen fokussiert. Jedes Tomogramm enthält mindestens ein Mitochondrium zusammen mit verschiedenen anderen membrangebundenen Organellen. Bei diesen Bildern handelt es sich um Standbilder von Videos, die in Ergänzendes Video S1, Ergänzendes Video S2, Ergänzendes Video S3 und Ergänzendes Video S4 gezeigt werden. (B) Entsprechende Tomogramme, die in der Software visualisiert werden. Tomographische Schnitte, die mit dem Schnittwerkzeug in der VR-Software erstellt wurden, nachdem Schwellenwerte, Helligkeit und Fensterbildung optimiert wurden, wodurch unterschiedliche zelluläre Strukturen sichtbar wurden. (C) Tomogramme, die in der Software mit segmentierten Membranen visualisiert werden. Tomographische Schnitte, die mit dem Schnittwerkzeug in der VR-Software erstellt und mit der entsprechenden Segmentierung überlagert wurden. Calciumphosphatablagerungen sind gelb, Mitochondrienmembranen in Cyan und andere Membranen wie Vesikel und die Plasmamembran violett dargestellt. (D) 3D-Rendering der segmentierten Membranen in syGlass. Die Mitochondrienmembranen, einschließlich der Cristae, der äußeren und inneren Mitochondrienmembranen, sind in Cyan dargestellt. Calciumphosphatablagerungen sind gelb oder grün, Vesikel und die Plasmamembran sind violett und das endoplasmatische Retikulum ist hellbraun. Bei diesen Bildern handelt es sich um Standbilder von Videos, die in "Ergänzende Videos S5", "Ergänzende Videos S6", "Ergänzende Videos S7" und "Ergänzende Videos S8" gezeigt werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzendes Video S1: Tomogramm der Probe RPE-1_1, die für die Segmentierung in dieser Studie verwendet wurde. Dieses Tomogramm wird mit 16 Å/Pixel mittels Warp rekonstruiert, dann entrauscht und mit IsoNet um das fehlende Wedge korrigiert. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzendes Video S2: Tomogramm der Probe RPE-1_2, die für die Segmentierung in dieser Studie verwendet wurde. Dieses Tomogramm wird mit 16 Å/Pixel mittels Warp rekonstruiert, dann entrauscht und mit IsoNet um das fehlende Wedge korrigiert. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzendes Video S3: Tomogramm der Probe RPE-1_3, die für die Segmentierung in dieser Studie verwendet wurde. Dieses Tomogramm wird mit 16 Å/Pixel mittels Warp rekonstruiert, dann entrauscht und mit IsoNet um das fehlende Wedge korrigiert. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzendes Video S4: Tomogramm der Probe RPE-1_4, die für die Segmentierung in dieser Studie verwendet wurde. Dieses Tomogramm wird mit 16 Å/Pixel mittels Warp rekonstruiert, dann entrauscht und mit IsoNet um das fehlende Wedge korrigiert. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzendes Video S5: Die resultierende Segmentierung des Beispiels RPE-1_1 nach der Generierung von Oberflächen. Die mitochondrialen Membranen sind in Cyan dargestellt; Kalziumphosphate in den Mitochondrien sind gelb dargestellt; Das endoplasmatische Retikulum ist in Tan dargestellt; und andere Membranen werden violett dargestellt. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzendes Video S6: Die resultierende Segmentierung des Beispiels RPE-1_2 nach der Generierung von Oberflächen. Die mitochondrialen Membranen sind in Cyan dargestellt; Kalziumphosphate in den Mitochondrien sind gelb dargestellt; Das endoplasmatische Retikulum ist in Tan dargestellt; und andere Membranen werden violett dargestellt. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzendes Video S7: Die resultierende Segmentierung der Probe RPE-1_3 nach der Generierung von Oberflächen. Die mitochondrialen Membranen sind in Cyan dargestellt; Kalziumphosphate in den Mitochondrien sind gelb dargestellt; Das endoplasmatische Retikulum ist in Tan dargestellt; und andere Membranen werden violett dargestellt. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzendes Video S8: Die resultierende Segmentierung von Sample RPE-1_4 nach der Generierung von Oberflächen. Die mitochondrialen Membranen sind in Cyan dargestellt; Kalziumphosphate in den Mitochondrien sind gelb dargestellt; Das endoplasmatische Retikulum ist in Tan dargestellt; und andere Membranen werden violett dargestellt. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
In dieser Arbeit haben wir gezeigt, wie VR, insbesondere unter Verwendung von syGlass, in die Kryo-ET-Pipeline integriert werden kann, um zelluläre Strukturen effektiv zu segmentieren. Obwohl unser Fokus in erster Linie auf Membranen lag, gibt es keine inhärente Einschränkung, die die Segmentierung anderer zellulärer Strukturen, wie Filamente oder Ribosomen, verhindert, da man die Form und Größe des Pinsels sowie den Schwellenwert anpassen kann, um sicherzustellen, dass nur Voxel markiert werden, die den gewünschten zellulären Objekten entsprechen. Einer der Hauptvorteile von VR bei der Segmentierung ist die intuitive, praktische Interaktion mit den Daten, die es den Benutzern ermöglicht, Volumen so zu visualisieren und zu manipulieren, als ob sie sich physisch in ihnen befänden. Herkömmliche Methoden der manuellen Segmentierung oder der Bereinigung der anfänglichen Segmentierungsmasken beinhalten in der Regel die direkte Arbeit auf einem Computer, was weniger immersiv und langsamer zu kommentieren sein kann.
Durch die Integration von VR in den Kryo-ET-Workflow können Benutzer nicht nur schnell mit Segmentierungsmasken interagieren, die von anderer Software generiert werden, sondern VR auch verwenden, um die Segmentierung von teilweise segmentierten Strukturen zu steuern und Fehlalarme effizient zu bereinigen. Derzeit ist für die meisten Anwendungsfälle von Annotationen noch eine manuelle Segmentierung erforderlich, und der vorgestellte Workflow ermöglicht es den Benutzern, Segmentierungen, die für die nachgelagerte Analyse geeignet sind, einfacher und schneller zu erstellen. Für diese Studie haben wir das HTC Vive VR-Headset verwendet, aber die Software ist mit Geräten kompatibel, die SteamVR- oder OculusVR-Unterstützung haben.
Für eine optimale Anwendung dieses Protokolls sollten die Kryo-ET-Daten bestimmte Kriterien erfüllen, wie z. B. qualitativ hochwertige Tomogramme mit einem hohen Signal-Rausch-Verhältnis (SNR). Das SNR ist ein wesentlicher Parameter, da die Klarheit der strukturellen Merkmale die Wirksamkeit der manuellen Segmentierung in VR direkt beeinflusst. Vorverarbeitungsschritte wie die Korrektur fehlender Keile und die Rauschunterdrückung sind wichtige Bestandteile des Arbeitsablaufs. In diesem Protokoll haben wir IsoNet für diese Zwecke verwendet. Die Tomogramme sollten mit einer geeigneten Auflösung rekonstruiert werden – unter Verwendung einer Voxelgröße, die ausreichend strukturelle Details und einen ausreichenden Abstand zwischen den zellulären Strukturen bietet, um eine effektive Segmentierung zwischen den Strukturen zu ermöglichen und gleichzeitig überschaubare Datengrößen für die VR-Visualisierung beizubehalten. Zusätzlich sollten ein invertierter Kontrast mit zellulären Strukturen, die weiß auf schwarzem Hintergrund erscheinen, und eine Histogrammentzerrung angewendet werden, um die Sichtbarkeit von Membranen und anderen Strukturen in der syGlass-Umgebung zu verbessern.
Um den Erfolg dieses Protokolls zu gewährleisten, müssen mehrere kritische Punkte berücksichtigt werden. Erstens ist bei der Datenvorverarbeitung die genaue Anwendung der Kontrastinversion und der Histogrammentzerrung von entscheidender Bedeutung. Unsachgemäße Anpassungen können zu einer suboptimalen Visualisierung in VR führen, was die Segmentierung zu einer Herausforderung macht. Zweitens sind innerhalb der VR-Software entsprechende Anpassungen der Fenster-, Helligkeits- und Schwellenwerteinstellungen für eine optimale Visualisierung von Strukturen unerlässlich. Benutzer sollten mit diesen Einstellungen experimentieren, um die besten Ergebnisse für ihre spezifischen Datensätze und VR-Setups zu erzielen.
Bei der Fehlerbehebung können Benutzer auf Probleme stoßen, wie z. B. Leistungseinschränkungen des VR-Systems, insbesondere beim Umgang mit großen Tomogramm-Datensätzen. Wenn die VR-Umgebung verzögert oder nicht mehr reagiert, sollten Sie in Erwägung ziehen, die Daten zu verkleinern oder das Tomogramm in kleinere Abschnitte zu segmentieren, indem Sie den Begrenzungsrahmen in den ROI-Tools in syGlass anpassen und ihn während der Segmentierung entlang des Tomogramms verschieben. Der Leistungsschieberegler im Visualisierungsmenü kann ebenfalls angepasst werden, um Verzögerungen während des Segmentierungsprozesses zu reduzieren. Um die Reisekrankheit zu mildern, können Benutzer außerdem die VR-Einstellungen anpassen, um Bewegungseffekte zu reduzieren, oder während der Segmentierungssitzungen regelmäßige Pausen einlegen.
Unser VR-basiertes Segmentierungsprotokoll bietet erhebliche Vorteile für die aktuellen Arbeitsabläufe der Kryo-ET-Segmentierung. Herkömmliche manuelle Segmentierungsmethoden beinhalten häufig 2D-Annotationen auf einzelnen Segmenten, was zeitaufwändig sein kann und die dreidimensionale Kontinuität möglicherweise nicht vollständig erfasst13. Darüber hinaus ermöglicht die VR-basierte Segmentierung eine viel intensivere Sicht auf die zellulären Strukturen, was die Visualisierung erleichtert. Automatisierte Segmentierungen durch maschinelles Lernen entwickeln sich zu einer leistungsfähigen Methode, um Segmentierungen aus zellulären Tomogrammen zu erhalten 14,16,17, obwohl die hohen Rauschpegel und komplexen Strukturen in Kryo-ET-Daten zu Lücken und falsch positiven Ergebnissen führen, die manuelle Eingriffe erfordern. Dieses Protokoll bietet einen alternativen Ansatz zur manuellen Segmentierung von Kryo-Elektronen-Tomogrammen, zur Generierung anfänglicher Segmentierungen, die möglicherweise als Trainingsdaten für andere neuronale Netzwerksoftware verwendet werden können, oder zur Bereinigung von anfänglichen Segmentierungen, die von anderen automatisierten Ansätzen generiert wurden.
Zusammenfassend hebt diese Studie hervor, dass die VR-basierte Segmentierung ein vielversprechendes Werkzeug für die Datenanalyse und -aufklärung von Kryo-ET ist, das eine verbesserte Effizienz und ein immersiveres Benutzererlebnis bietet. Mit der Weiterentwicklung hat die VR-Technologie das Potenzial, die Art und Weise, wie wir wissenschaftliche Entdeckungen komplexer zellulärer Strukturen in Kryo-ET-Datensätzen interpretieren und verbreiten, zu revolutionieren und eine wertvolle Alternative zu herkömmlichen Segmentierungs- und Bildungsmethoden zu bieten.
Offenlegungen
Die Autoren erklären, dass sie keine Interessenkonflikte haben.
Danksagungen
Diese Arbeit wurde am National Center for CryoEM Access and Training (NCCAT) und am Simons Electron Microscopy Center am New York Structural Biology Center durchgeführt, unterstützt von NIH (Common Fund U24GM129539, U24GM139171 und NIGMS R24GM154192), der Simons Foundation (SF349247) und der NY State Assembly.
Materialien
| Name | Company | Catalog Number | Comments |
| CryoET Data | Format:TIFF-stack, TIFF | ||
| HTC VIVE Cosmos | HTC | 99HARL000-00 | https://www.vive.com/sea/product/vive-cosmos/features/ |
| Intel(R) Core(TM) i7-10870H CPU @ 2.20 GHz 2.21 GHz | Intel | https://ark.intel.com/content/www/us/en/ark/products/208018/intel-core-i7-10870h-processor-16m-cache-up-to-5-00-ghz.html | |
| NVIDIA GeForce RTX 3070 Laptop GPU | NVIDIA | https://www.nvidia.com/en-us/geforce/laptops/compare/30-series/ | |
| syGlass Software | syGlass | syGlass Software installed on a compatible Windows PC | |
| VIVE Cosmos Hand Controllers | HTC | 99HAFR001-00 | https://www.vive.com/us/accessory/cosmos-controller-right/ |
| Windows 11 Home | Microsoft | Microsoft Windows 11 Home |
Referenzen
- Plitzko, J. M., Schuler, B., Selenko, P. Structural biology outside the box-inside the cell. Curr Opin Struct Biol. 46, 110-121 (2017).
- Beck, M., Baumeister, W. Cryo-electron tomography: can it reveal the molecular sociology of cells in atomic detail. Trends Cell Biol. 26 (11), 825-837 (2016).
- Lam, V., Villa, E. Practical approaches for cryo-FIB milling and applications for cellular cryo-electron tomography. Methods Mol Biol. 2215, 49-82 (2021).
- Berger, C., et al. Cryo-electron tomography on focused ion beam lamellae transforms structural cell biology. Nat Methods. 20 (4), 499-511 (2023).
- Watson, A. J. I., Bartesaghi, A. Advances in cryo-ET data processing: meeting the demands of visual proteomics. Curr Opin Struct Biol. 87, 102861 (2024).
- Pyle, E., Zanetti, G. Current data processing strategies for cryo-electron tomography and subtomogram averaging. Biochem J. 478 (10), 1827-1845 (2021).
- Wan, W., Briggs, J. A. G. Cryo-electron tomography and subtomogram averaging. Methods Enzymol. 579, 329-367 (2016).
- Ni, T., Liu, K., Zhang, J., Atanasov, I., Mladenov, M. G., Jiang, W. High-resolution in situ structure determination by cryo-electron tomography and subtomogram averaging using emClarity. Nat Protoc. 17 (2), 421-444 (2022).
- Obr, M., et al. Exploring high-resolution cryo-ET and subtomogram averaging capabilities of contemporary DEDs. J Struct Biol. 214 (2), 107852 (2021).
- Tegunov, D., Xue, L., Dienemann, C., Cramer, P., Mahamid, J. Multi-particle cryo-EM refinement with M visualizes ribosome-antibiotic complex at 3.5 Å in cells. Nat Methods. 18 (2), 186-193 (2021).
- Salfer, M., Collado, J. F., Baumeister, W., Fernández-Busnadiego, R., Martínez-Sánchez, A. Reliable estimation of membrane curvature for cryo-electron tomography. PLoS Comput Biol. 16 (8), e1007962 (2020).
- Barad, B. A., et al. Quantifying organellar ultrastructure in cryo-electron tomography using a surface morphometrics pipeline. J Cell Biol. 222 (2), e202204093 (2023).
- Martinez-Sanchez, A., Garcia, I., Asano, S., Lucic, V., Fernandez, J. J. Robust membrane detection based on tensor voting for electron tomography. J Struct Biol. 186 (1), 49-61 (2014).
- Heebner, J. E., et al. Deep learning-based segmentation of cryo-electron tomograms. J Vis Exp. (189), e64435 (2022).
- Garza-López, E., Robles-Flores, M. Protocols for generating surfaces and measuring 3D organelle morphology using Amira. Cells. 11 (1), 65 (2021).
- Chen, M., et al. Convolutional neural networks for automated annotation of cellular cryo-electron tomograms. Nat Methods. 14 (10), 983-985 (2017).
- Lamm, L., et al. MemBrain v2: an end-to-end tool for the analysis of membranes in cryo-electron tomography. bioRxiv. , (2024).
- Pidhorskyi, S., Morehead, M., Jones, Q., Spirou, G., Doretto, G. syGlass: interactive exploration of multidimensional images using virtual reality head-mounted displays. arXiv. , (2018).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Tegunov, D., Cramer, P. Real-time cryo-electron microscopy data preprocessing with Warp. Nat Methods. 16 (11), 1146-1152 (2019).
- Bepler, T., Kelley, K., Noble, A. J., Berger, B. Topaz-Denoise: general deep denoising models for cryoEM and cryoET. Nat Commun. 11, 5208 (2020).
- Buchholz, T. -. O., Berninger, L., Maurer, M., Jug, F. Cryo-CARE: content-aware image restoration for cryo-transmission electron microscopy data. Proc IEEE Int Symp Biomed Imaging. 16, 502-506 (2019).
- Buchholz, T. -. O., Jordan, M., Pigino, G., Jug, F. Content-aware image restoration for electron microscopy. Methods Cell Biol. 152, 277-289 (2019).
- Liu, Y. -. T., et al. Isotropic reconstruction for electron tomography with deep learning. Nat Commun. 13, 6482 (2022).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. J Struct Biol. 116 (1), 71-76 (1996).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten