Method Article
Exploiter la réalité virtuelle pour la segmentation et l’analyse immersives des données de cryo-tomographie électronique
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
La cryo-tomographie électronique (cryo-ET) permet de visualiser en 3D l’ultrastructure cellulaire à une résolution nanométrique, mais la segmentation manuelle reste longue et complexe. Nous présentons un nouveau flux de travail qui intègre un logiciel de réalité virtuelle avancé pour segmenter les tomogrammes cryo-ET, démontrant ainsi son efficacité grâce à la segmentation des mitochondries dans les cellules de mammifères.
Résumé
La cryotomographie électronique (cryo-ET) est une technique puissante pour visualiser l’ultrastructure des cellules en trois dimensions (3D) à une résolution nanométrique. Cependant, la segmentation manuelle des composants cellulaires dans les données cryo-ET reste un goulot d’étranglement important en raison de sa complexité et de sa nature chronophage. Dans ce travail, nous présentons un nouveau flux de travail de segmentation qui intègre un logiciel de réalité virtuelle (VR) avancé pour améliorer à la fois l’efficacité et la précision de la segmentation des ensembles de données cryo-ET. Ce flux de travail s’appuie sur un outil de réalité virtuelle immersif avec une interaction 3D intuitive, permettant aux utilisateurs de naviguer et d’annoter des structures cellulaires complexes dans un environnement plus naturel et interactif. Pour évaluer l’efficacité du flux de travail, nous l’avons appliqué à la segmentation des mitochondries dans les cellules de l’épithélium pigmentaire rétinien (RPE1). Les mitochondries, essentielles à la production d’énergie cellulaire et à la signalisation, présentent des changements morphologiques dynamiques, ce qui en fait un échantillon de test idéal. Le logiciel VR a facilité la délimitation précise des membranes mitochondriales et des structures internes, permettant une analyse en aval des structures membranaires segmentées. Nous démontrons que ce flux de travail de segmentation basé sur la RV améliore considérablement l’expérience utilisateur tout en maintenant une segmentation précise des structures cellulaires complexes dans les données cryo-ET. Cette approche est prometteuse pour de vastes applications en biologie cellulaire structurale et dans l’enseignement des sciences, offrant un outil transformateur pour les chercheurs engagés dans l’analyse cellulaire détaillée.
Introduction
La cryotomographie électronique (cryo-ET) a révolutionné notre capacité à visualiser les composants cellulaires dans leur état quasi natif à haute résolution 1,2. Cette technique puissante permet aux chercheurs de caractériser l’ultrastructure cellulaire, fournissant des informations sans précédent sur l’architecture et la fonction cellulaires. Cependant, la cryo-ET n’est pas sans limites, la principale d’entre elles étant l’exigence que les échantillons soient suffisamment minces pour être transparents aux électrons (généralement <0,5 μm) pour l’imagerie dans les microscopes électroniques à transmission cryo-capables standard. Les progrès récents dans le broyage par faisceau d’ions cryofocalisés ont permis l’amincissement d’échantillons épais pour l’analyse cryo-ET 3,4.
Les flux de travail cryo-ET typiques commencent par la collecte d’une série d’inclinaisons, où l’échantillon est imagé sous différents angles, allant de +60° à -60°. Ces images sont ensuite alignées par calcul et rétroprojetées pour créer un volume tridimensionnel (3D) ou tomogramme 5,6,7. Ce tomogramme sert de carte 3D détaillée du paysage cellulaire, offrant des informations à la fois spatiales et temporelles sur les structures cellulaires. Un raffinement supplémentaire grâce à la moyenne du sous-tomogramme, où plusieurs copies de la même structure sont alignées et moyennées, peut repousser encore plus loin les limites de résolution, atteignant parfois une résolution subnanométriquede 7,8,9,10.
La segmentation est une étape cruciale dans l’extraction d’informations biologiques significatives à partir de ces tomogrammes. Ce processus consiste à annoter des structures cellulaires spécifiques, telles que des membranes, dans le volume 3D. La segmentation permet des analyses avancées, y compris le calcul des distances intermembranaires et de la courbure de la membrane, fournissant des informations précieuses sur les processus cellulaires11,12. Bien que plusieurs progiciels soient disponibles pour cette tâche, notamment Dragonfly, Amira, MemBrain, EMAN2 et tomomemsegtv 13,14,15,16,17, le processus de segmentation reste un goulot d’étranglement important dans l’analyse des données cryo-ET. Il s’agit souvent d’un processus manuel chronophage et laborieux, qui peut prendre des semaines, voire des mois. Beaucoup de ces packages offrent des fonctionnalités de segmentation automatique, mais nécessitent souvent une correction manuelle importante pour supprimer les faux positifs, un processus qui peut être laborieux et peu intuitif lorsqu’il est effectué tranche par tranche ou en 3D.
Nous proposons une approche alternative en tirant parti de la technologie de réalité virtuelle (VR) pour relever ces défis. La RV offre une méthode immersive et interactive de visualisation des données, permettant aux utilisateurs de naviguer dans le volume tomographique comme s’ils étaient à l’intérieur de l’environnement cellulaire lui-même. Cette approche fournit également une plate-forme précieuse pour l’enseignement des sciences, l’exploration scientifique et la découverte de données cryo-ET in situ en offrant une expérience immersive et unique en son genre. Dans ce travail, nous présentons un protocole de segmentation de données cryo-ET à l’aide de syGlass18, un logiciel VR conçu pour la visualisation scientifique. Ce logiciel fournit une boîte à outils complète pour l’analyse des données cryo-ET, y compris la segmentation manuelle, l’affinement des segmentations générées automatiquement et même le prélèvement de particules dans les tomogrammes. Notre étude démontre la viabilité de la RV en tant qu’outil puissant pour les segmentations de nettoyage, la sélection de particules et la segmentation manuelle dans l’analyse de données cryo-ET.
Pour illustrer l’utilité du logiciel VR pour la segmentation cryo-ET, nous nous concentrons sur l’analyse de la morphologie mitochondriale dans les cellules de l’épithélium pigmentaire rétinien (RPE1). Les mitochondries constituent un excellent test pour la segmentation en raison de leur structure complexe et de la présence de caractéristiques facilement quantifiables, telles que la distance entre les membranes mitochondriales externes et internes. Ces caractéristiques peuvent être mesurées avec précision à l’aide d’outils d’analyse morphométrique de surface12, fournissant des mesures robustes pour évaluer la qualité de la segmentation. Ce protocole fournit des instructions étape par étape pour segmenter les données cryo-ET à l’aide de syGlass, démontrant ainsi son utilité dans le pipeline de segmentation cryo-ET. En intégrant la segmentation basée sur la RV dans le flux de travail cryo-ET, nous visons à améliorer à la fois l’efficacité et la précision de l’analyse structurelle manuelle en biologie cellulaire.
Protocole
1. Préparation des données cryo-ET pour la segmentation
- Convertissez les tomogrammes Cryo-ET bruts dans un format de données compatible avec syGlass, tel que les piles TIFF. Réglez le signal de manière à ce que les particules soient blanches sur noires et effectuez l’égalisation de l’histogramme avec ImageJ19.
REMARQUE : Pour améliorer le rapport signal/bruit, il est recommandé que les tomogrammes soient débruités avec des logiciels tels que Warp20, Topaz Denoise21, cryoCARE22,23 ou IsoNet24 avant d’être importés dans le logiciel. - Lancez le logiciel VR sur l’ordinateur. Accédez au menu Fichier et sélectionnez Créer un projet.
- Cliquez sur Créer un nouveau projet | Ajouter des fichiers. Naviguez jusqu’à l’emplacement où les fichiers TIFF sont enregistrés et importez-les dans le logiciel.
- Lorsque vous êtes invité à confirmer s’il s’agit d’une série chronologique, cliquez sur Non.
- Nommez le projet, puis cliquez sur Enregistrer pour créer un nouveau projet dans la liste des projets.
- Double-cliquez sur le projet pour ouvrir le tomogramme et le charger dans l’environnement de réalité virtuelle interactive du logiciel.
2. Mise en place de la réalité virtuelle (VR)
- Connectez le casque VR et les manettes à l’ordinateur.
- Suivez les instructions à l’écran pour calibrer l’environnement VR.
REMARQUE : L’étalonnage fait référence à l’espace tridimensionnel où la segmentation sera effectuée. Les paramètres (longueur, largeur et hauteur) de l’espace de travail doivent être définis avant l’affichage de la projection VR. La projection VR ne sera visible que dans ces paramètres. - La segmentation ne sera effectuée que dans l’espace tridimensionnel de travail défini. Assurez-vous que le champ de vision contient la zone sur laquelle vous souhaitez travailler dans l’environnement VR.
3. Optimiser la visualisation 3D
- Cliquez sur le bouton Visualisation dans l’interface du logiciel. Ajustez diverses options de visualisation telles que le contraste, le fenêtrage, la luminosité et les curseurs de seuil pour améliorer le signal et minimiser le bruit.
- Utilisez les manettes pour rapprocher le tomogramme ou le repousser au besoin.
- Activez l’outil Couper à l’aide de la manette de gauche pour inspecter visuellement différentes tranches du tomogramme.
4. Processus de segmentation
- Naviguez dans le tomogramme jusqu’à la tranche souhaitée où la segmentation commencera.
- Activez l’option Région d’intérêt (ROI) dans le menu d’annotation à l’aide des manettes. Une boîte verte apparaîtra dans le tomogramme.
- Ajustez la taille et la position de la boîte verte, en la déplaçant vers la zone à segmenter.
- Verrouillez le retour sur investissement à l’aide du contrôleur de gauche et lancez la segmentation. Après avoir verrouillé le ROI, l’outil passe automatiquement en mode peinture qui permet la segmentation des données volumétriques.
- Zoomez ou dézoomez sur le tomogramme pour une segmentation précise.
- Ajustez la taille du pinceau en le tournant dans le sens des aiguilles d’une montre ou dans le sens inverse des aiguilles d’une montre pour un contrôle optimal.
- Segmentez soigneusement le retour sur investissement (par exemple, les membranes mitochondriales) dans la zone 3D sélectionnée. Si une erreur se produit pendant la segmentation, maintenez enfoncée la gâchette de la manette secondaire pour activer le mode d’effacement , puis effacez l’erreur en utilisant le même mouvement que la segmentation.
- Répétez ce processus région par région jusqu’à ce que l’ensemble du tomogramme soit segmenté.
5. Exportation de données segmentées et d’analyses
- Une fois la segmentation terminée, sélectionnez le projet en cliquant dessus pour vous assurer qu’il est mis en surbrillance.
- Cliquez sur l’onglet Projets dans le coin supérieur gauche, puis sélectionnez ROI.
- Choisissez d’exporter soit l’intégralité du volume, soit un retour sur investissement spécifique. Spécifiez l’emplacement d’exportation des données segmentées.
- Charger et analyser les données segmentées à l’aide du logiciel de votre choix et générer des chiffres pour publication.
6. Importation du masque binaire dans le logiciel pour le nettoyage
- Effectuez les étapes 1.1 à 1.6.
- Faites un clic droit sur le projet, puis cliquez sur Ajouter des données de masque. Ensuite, naviguez jusqu’à l’endroit où la segmentation initiale est enregistrée et importez-la sous le même projet.
- Engagez l’annotation ROI pour apporter des modifications à la segmentation initiale.
- Ajoutez/effacez la segmentation pour nettoyer la segmentation initiale.
- Ajustez les retours sur investissement des masques sous l’option ROI dans l’onglet masques . Pour changer la ROI du masque de l’un à l’autre, activez la seule option de remplacement du masque et passez en mode peinture avec la manette. Ce mode n’écrasera que les données de masque visibles.
- Exportez les retours d’intérêt du masque ajustés en suivant les étapes décrites aux étapes 5.1 à 5.3.
7. Prélèvement des coordonnées des particules à l’aide du logiciel VR
- Créez un projet à l’aide d’un tomogramme (pile TIFF) en suivant les étapes décrites à la section 1.
- Cliquez sur Annotation | Compter, qui ouvre une fenêtre avec des options manuelles ou automatisées .
- Pour l’option manuelle , choisissez une couleur pour les comptages et commencez à marquer les coordonnées.
- Pour l’option automatisée, choisissez une taille de cellule et entrez la résolution. Cliquez sur Exécuter le comptage automatique, modifiez les comptes (conservez les bons choix et éliminez les mauvais) et enregistrez la progression.
- Exportez les coordonnées de comptage en cliquant sur l’onglet projet | annotations.
- Pour l’origine de l’annotation, choisissez Coin inférieur gauche et pour Unités d’annotation, choisissez Voxels.
- Cliquez sur la cellule de comptage des points pour mettre la cellule en surbrillance.
- Cliquez sur Exporter la sélection pour enregistrer les coordonnées de la sélection dans un fichier .csv.
REMARQUE : Ce fichier peut être utilisé pour extraire des sous-tomogrammes du tomogramme afin de calculer la moyenne des sous-tomogrammes
Résultats
Dans cette étude, nous avons segmenté des tomogrammes contenant des mitochondries et des organites membraneux supplémentaires (par exemple, des vésicules, un réticulum endoplasmique) à l’aide de syGlass. Les tomogrammes ont d’abord été reconstruits en Warp à l’aide d’une projection pondérée à 16,00 Å/pixel et ont été soumis à une correction de coin manquante et à un débruitage à l’aide du logiciel IsoNet. Les tomogrammes suivants ont fait l’objet d’un traitement supplémentaire pour l’importation, comme le montre la figure 1A (voir également Vidéo supplémentaire S1, Vidéo supplémentaire S2, Vidéo supplémentaire S3 et Vidéo supplémentaire S4).
Après le prétraitement, les tomogrammes au format MRC ont été convertis en piles TIFF à l’aide d’ImageJ, avec une inversion de contraste appliquée pour faire apparaître les membranes blanches sur un fond noir. L’égalisation de l’histogramme a ensuite été effectuée pour améliorer encore le contraste ; Cela permet également un seuillage plus efficace. Les piles TIFF ont été importées et visualisées en 3D dans un environnement VR immersif, permettant une inspection détaillée des structures membraneuses à l’aide de l’outil de coupe dans le logiciel illustré à la figure 1B.
La segmentation manuelle a été effectuée dans le logiciel, avec le fenêtrage réglé sur auto et des ajustements de luminosité/seuil effectués pour optimiser la visibilité des fonctions cellulaires. Les zones d’intérêt autour des mitochondries et d’autres structures ont été définies à l’aide des contrôleurs VR. L’outil de segmentation a permis de délimiter avec précision les limites de la membrane, les erreurs étant corrigées à l’aide de la fonction d’effacement. La navigation tranche par tranche ou en 3D à l’aide de l’outil ROI pour boxer les régions du tomogramme, combinée à l’outil pinceau réglable, a assuré une segmentation précise des membranes mitochondriales et d’autres organites, comme le montre la figure 1C.
Les données segmentées ont été visualisées en générant un maillage à l’aide de l’option de surfaces de l’outil ROI, avec des itérations de lissage définies sur 12 et un niveau de résolution défini sur 3. Les rendus 3D finaux montrent clairement les structures mitochondriales, y compris les membranes externe et interne, les crêtes et les dépôts de phosphate de calcium, comme le montre la figure 1D (voir également la vidéo supplémentaire S5, la vidéo supplémentaire S6, la vidéo supplémentaire S7 et la vidéo supplémentaire S8).
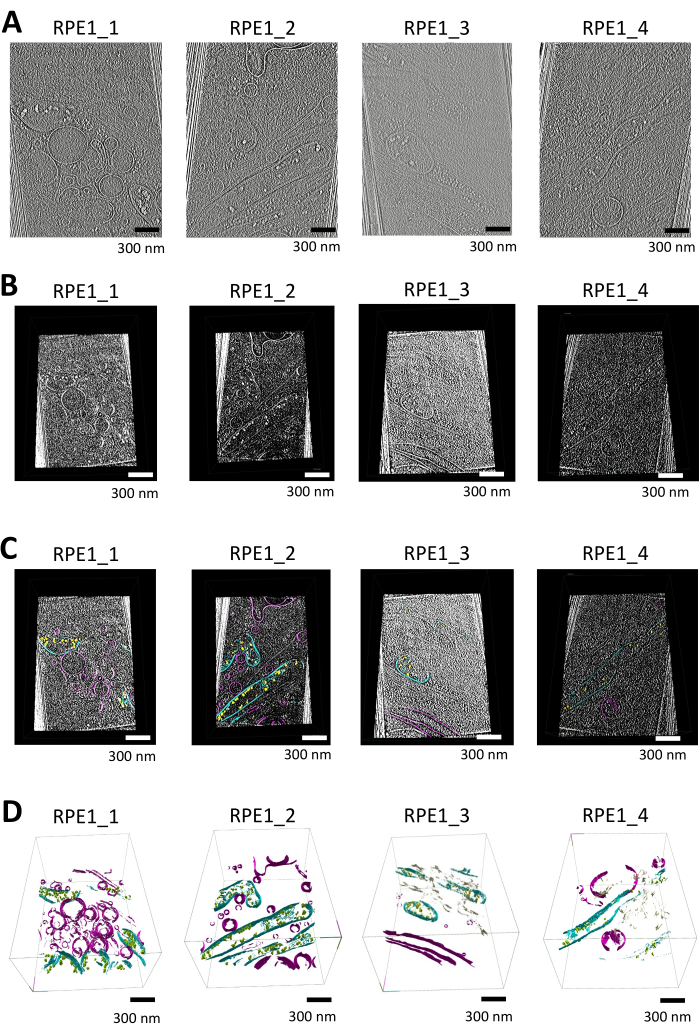
Figure 1 : Flux de travail de visualisation de tomogrammes, de segmentation et de rendu 3D à l’aide de syglass. (A) Tranches tomographiques de cellules RPE1 à bords fins. Tomogrammes corrigés IsoNet reconstruits dans Warp et utilisés pour la segmentation avec syGlass, visualisés avec IMOD25. Les tomogrammes ont été collectés sur un microscope Titan-Krios de 300 keV équipé d’un détecteur K3 et se sont concentrés sur les bords minces des cellules de l’épithélium rétinien. Chaque tomogramme contient au moins une mitochondrie ainsi que divers autres organites liés à une membrane. Ces images sont des images fixes de vidéos présentées dans les vidéos supplémentaires S1, S2, S3 et S4. (B) Les tomogrammes correspondants visualisés dans le logiciel. Tranche tomographique créée à l’aide de l’outil de coupe dans le logiciel VR après optimisation du seuillage, de la luminosité et du fenêtrage, révélant des structures cellulaires distinctes. (C) Tomogrammes visualisés dans le logiciel avec des membranes segmentées. Tranche tomographique créée avec l’outil de coupe dans le logiciel VR, superposée à la segmentation correspondante. Les dépôts de phosphate de calcium sont représentés en jaune, les membranes mitochondriales en cyan et d’autres membranes telles que les vésicules et la membrane plasmique en violet. (D) Rendu 3D des membranes segmentées dans syGlass. Les membranes mitochondriales, y compris les crêtes, les membranes mitochondriales externes et internes, sont illustrées en cyan. Les dépôts de phosphate de calcium sont en jaune ou en vert, les vésicules et la membrane plasmique sont en violet, et le réticulum endoplasmique sont en beige. Ces images sont des images fixes de vidéos présentées dans les vidéos supplémentaires S5, Vidéos supplémentaires S6, Vidéo supplémentaire S7 et Vidéo supplémentaire S8. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Vidéo supplémentaire S1 : Tomogramme de l’échantillon RPE-1_1 utilisé pour la segmentation dans cette étude. Ce tomogramme est reconstruit à 16 Å/pixel à l’aide de Warp, puis débruité et corrigé pour le coin manquant à l’aide d’IsoNet. Veuillez cliquer ici pour télécharger ce fichier.
Vidéo supplémentaire S2 : Tomogramme de l’échantillon RPE-1_2 utilisé pour la segmentation dans cette étude. Ce tomogramme est reconstruit à 16 Å/pixel à l’aide de Warp, puis débruité et corrigé pour le coin manquant à l’aide d’IsoNet. Veuillez cliquer ici pour télécharger ce fichier.
Vidéo supplémentaire S3 : Tomogramme de l’échantillon RPE-1_3 utilisé pour la segmentation dans cette étude. Ce tomogramme est reconstruit à 16 Å/pixel à l’aide de Warp, puis débruité et corrigé pour le coin manquant à l’aide d’IsoNet. Veuillez cliquer ici pour télécharger ce fichier.
Vidéo supplémentaire S4 : Tomogramme de l’échantillon RPE-1_4 utilisé pour la segmentation dans cette étude. Ce tomogramme est reconstruit à 16 Å/pixel à l’aide de Warp, puis débruité et corrigé pour le coin manquant à l’aide d’IsoNet. Veuillez cliquer ici pour télécharger ce fichier.
Vidéo supplémentaire S5 : La segmentation résultante de l’échantillon RPE-1_1 après génération de surfaces. Les membranes mitochondriales sont représentées en cyan ; les phosphates de calcium dans les mitochondries sont représentés en jaune ; le réticulum endoplasmique est montré en beige ; et les autres membranes sont affichées en violet. Veuillez cliquer ici pour télécharger ce fichier.
Vidéo supplémentaire S6 : La segmentation résultante de l’échantillon RPE-1_2 après génération de surfaces. Les membranes mitochondriales sont représentées en cyan ; les phosphates de calcium dans les mitochondries sont représentés en jaune ; le réticulum endoplasmique est montré en beige ; et les autres membranes sont affichées en violet. Veuillez cliquer ici pour télécharger ce fichier.
Vidéo supplémentaire S7 : La segmentation résultante de l’échantillon RPE-1_3 après génération de surfaces. Les membranes mitochondriales sont représentées en cyan ; les phosphates de calcium dans les mitochondries sont représentés en jaune ; le réticulum endoplasmique est montré en beige ; et les autres membranes sont affichées en violet. Veuillez cliquer ici pour télécharger ce fichier.
Vidéo supplémentaire S8 : La segmentation résultante de l’échantillon RPE-1_4 après génération de surfaces. Les membranes mitochondriales sont représentées en cyan ; les phosphates de calcium dans les mitochondries sont représentés en jaune ; le réticulum endoplasmique est montré en beige ; et les autres membranes sont affichées en violet. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Dans ce travail, nous avons démontré comment la RV, en particulier à l’aide de syGlass, peut être intégrée dans le pipeline cryo-ET pour segmenter efficacement les structures cellulaires. Bien que nous nous soyons principalement concentrés sur les membranes, il n’y a aucune limitation inhérente empêchant la segmentation d’autres structures cellulaires, telles que les filaments ou les ribosomes, car on peut ajuster la forme et la taille du pinceau, et le seuillage pour s’assurer que seuls les voxels correspondant aux objets cellulaires souhaités sont marqués. L’un des principaux avantages de la RV dans la segmentation est l’interaction intuitive et pratique avec les données, permettant ainsi aux utilisateurs de visualiser et de manipuler les volumes comme s’ils étaient physiquement à l’intérieur. Les méthodes traditionnelles de segmentation manuelle ou de nettoyage des masques de segmentation initiale impliquent généralement de travailler directement sur un ordinateur, qui peut être moins immersif et plus lent à annoter.
En intégrant la réalité virtuelle dans le flux de travail cryo-ET, les utilisateurs peuvent non seulement interagir rapidement avec les masques de segmentation générés par d’autres logiciels, mais aussi utiliser la réalité virtuelle pour guider la segmentation des structures partiellement segmentées et nettoyer efficacement les faux positifs. Actuellement, la segmentation manuelle est toujours nécessaire pour la plupart des cas d’utilisation d’annotation, et le flux de travail présenté permet aux utilisateurs de générer des segmentations adaptées à l’analyse en aval avec une facilité et une rapidité améliorées. Pour cette étude, nous avons utilisé le casque VR HTC Vive, mais le logiciel est compatible avec les appareils prenant en charge SteamVR ou OculusVR.
Pour une application optimale de ce protocole, les données cryo-ET doivent répondre à des critères spécifiques, tels que des tomogrammes de haute qualité avec un rapport signal/bruit (SNR) élevé. Le SNR est un paramètre essentiel, car la clarté des caractéristiques structurelles influence directement l’efficacité de la segmentation manuelle en VR. Les étapes de prétraitement telles que la correction du coin manquant et le débruitage sont des éléments clés du flux de travail ; dans ce protocole, nous avons utilisé IsoNet à ces fins. Les tomogrammes doivent être reconstruits à une résolution appropriée - en utilisant une taille de voxel qui fournit suffisamment de détails structurels et une séparation suffisante entre les structures cellulaires pour permettre une segmentation efficace entre les structures tout en maintenant des tailles de données gérables pour la visualisation VR. De plus, un contraste inversé avec des structures cellulaires apparaissant blanches sur un fond noir et une égalisation de l’histogramme doivent être appliqués pour améliorer la visibilité des membranes et autres structures dans l’environnement syGlass.
Plusieurs points critiques doivent être pris en compte pour assurer le succès de ce protocole. Tout d’abord, lors du prétraitement des données, l’application précise de l’inversion de contraste et de l’égalisation de l’histogramme est essentielle ; des ajustements inappropriés peuvent entraîner une visualisation sous-optimale en VR, ce qui rend la segmentation difficile. Deuxièmement, dans le logiciel de réalité virtuelle, des ajustements appropriés du fenêtrage, de la luminosité et des paramètres de seuil sont essentiels pour une visualisation optimale des structures. Les utilisateurs doivent expérimenter ces paramètres pour obtenir les meilleurs résultats pour leurs ensembles de données spécifiques et leurs configurations VR.
Lorsqu’il s’agit de dépanner, les utilisateurs peuvent rencontrer des problèmes tels que des limitations de performances du système VR, en particulier lors de la manipulation de grands ensembles de données de tomogrammes. Si l’environnement VR devient lent ou ne répond plus, envisagez de sous-échantillonner les données ou de segmenter le tomogramme en sections plus petites en ajustant le cadre englobant dans les outils de retour sur investissement de syGlass et en le déplaçant le long du tomogramme au fur et à mesure que la segmentation est effectuée. Le curseur de performance dans le menu de visualisation peut également être ajusté pour réduire le décalage pendant le processus de segmentation. De plus, pour atténuer le mal des transports, les utilisateurs peuvent ajuster les paramètres VR pour réduire les effets de mouvement ou faire des pauses régulières pendant les sessions de segmentation.
Notre protocole de segmentation basé sur la réalité virtuelle offre des avantages significatifs aux flux de travail actuels de segmentation cryo-ET. Les méthodes de segmentation manuelle conventionnelles impliquent souvent une annotation 2D sur des tranches individuelles, ce qui peut prendre beaucoup de temps et ne pas capturer pleinement la continuité tridimensionnelle13. De plus, la segmentation basée sur la réalité virtuelle introduit une vue beaucoup plus immersive des structures cellulaires, ce qui facilite la visualisation. Les segmentations automatisées d’apprentissage automatique apparaissent comme une méthode puissante pour obtenir des segmentations à partir de tomogrammes cellulaires 14,16,17, bien que les niveaux de bruit élevés et les structures complexes présentes dans les données cryo-ET entraînent des lacunes et des faux positifs qui nécessitent une intervention manuelle. Ce protocole offre une approche alternative pour segmenter manuellement les tomogrammes cryo-électroniques, pour générer des segmentations initiales qui peuvent potentiellement être utilisées comme données d’entraînement pour d’autres logiciels de réseau neuronal, ou pour nettoyer les segmentations initiales générées par d’autres approches automatisées.
En conclusion, cette étude met en évidence la segmentation basée sur la RV comme un outil prometteur pour l’analyse et l’éducation des données cryo-ET, offrant une efficacité accrue et une expérience utilisateur plus immersive. Avec de nouveaux développements, la technologie VR a le potentiel de révolutionner la façon dont nous interprétons et diffusons les découvertes scientifiques de structures cellulaires complexes dans les ensembles de données cryo-ET, offrant une alternative précieuse aux méthodes traditionnelles de segmentation et d’éducation.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont aucun conflit d’intérêts.
Remerciements
Ce travail a été effectué au National Center for CryoEM Access and Training (NCCAT) et au Simons Electron Microscopy Center situé au New York Structural Biology Center, avec le soutien du NIH (Common Fund U24GM129539, U24GM139171 et NIGMS R24GM154192), de la Fondation Simons (SF349247) et de l’Assemblée de l’État de New York.
matériels
| Name | Company | Catalog Number | Comments |
| CryoET Data | Format:TIFF-stack, TIFF | ||
| HTC VIVE Cosmos | HTC | 99HARL000-00 | https://www.vive.com/sea/product/vive-cosmos/features/ |
| Intel(R) Core(TM) i7-10870H CPU @ 2.20 GHz 2.21 GHz | Intel | https://ark.intel.com/content/www/us/en/ark/products/208018/intel-core-i7-10870h-processor-16m-cache-up-to-5-00-ghz.html | |
| NVIDIA GeForce RTX 3070 Laptop GPU | NVIDIA | https://www.nvidia.com/en-us/geforce/laptops/compare/30-series/ | |
| syGlass Software | syGlass | syGlass Software installed on a compatible Windows PC | |
| VIVE Cosmos Hand Controllers | HTC | 99HAFR001-00 | https://www.vive.com/us/accessory/cosmos-controller-right/ |
| Windows 11 Home | Microsoft | Microsoft Windows 11 Home |
Références
- Plitzko, J. M., Schuler, B., Selenko, P. Structural biology outside the box-inside the cell. Curr Opin Struct Biol. 46, 110-121 (2017).
- Beck, M., Baumeister, W. Cryo-electron tomography: can it reveal the molecular sociology of cells in atomic detail. Trends Cell Biol. 26 (11), 825-837 (2016).
- Lam, V., Villa, E. Practical approaches for cryo-FIB milling and applications for cellular cryo-electron tomography. Methods Mol Biol. 2215, 49-82 (2021).
- Berger, C., et al. Cryo-electron tomography on focused ion beam lamellae transforms structural cell biology. Nat Methods. 20 (4), 499-511 (2023).
- Watson, A. J. I., Bartesaghi, A. Advances in cryo-ET data processing: meeting the demands of visual proteomics. Curr Opin Struct Biol. 87, 102861 (2024).
- Pyle, E., Zanetti, G. Current data processing strategies for cryo-electron tomography and subtomogram averaging. Biochem J. 478 (10), 1827-1845 (2021).
- Wan, W., Briggs, J. A. G. Cryo-electron tomography and subtomogram averaging. Methods Enzymol. 579, 329-367 (2016).
- Ni, T., Liu, K., Zhang, J., Atanasov, I., Mladenov, M. G., Jiang, W. High-resolution in situ structure determination by cryo-electron tomography and subtomogram averaging using emClarity. Nat Protoc. 17 (2), 421-444 (2022).
- Obr, M., et al. Exploring high-resolution cryo-ET and subtomogram averaging capabilities of contemporary DEDs. J Struct Biol. 214 (2), 107852 (2021).
- Tegunov, D., Xue, L., Dienemann, C., Cramer, P., Mahamid, J. Multi-particle cryo-EM refinement with M visualizes ribosome-antibiotic complex at 3.5 Å in cells. Nat Methods. 18 (2), 186-193 (2021).
- Salfer, M., Collado, J. F., Baumeister, W., Fernández-Busnadiego, R., Martínez-Sánchez, A. Reliable estimation of membrane curvature for cryo-electron tomography. PLoS Comput Biol. 16 (8), e1007962 (2020).
- Barad, B. A., et al. Quantifying organellar ultrastructure in cryo-electron tomography using a surface morphometrics pipeline. J Cell Biol. 222 (2), e202204093 (2023).
- Martinez-Sanchez, A., Garcia, I., Asano, S., Lucic, V., Fernandez, J. J. Robust membrane detection based on tensor voting for electron tomography. J Struct Biol. 186 (1), 49-61 (2014).
- Heebner, J. E., et al. Deep learning-based segmentation of cryo-electron tomograms. J Vis Exp. (189), e64435 (2022).
- Garza-López, E., Robles-Flores, M. Protocols for generating surfaces and measuring 3D organelle morphology using Amira. Cells. 11 (1), 65 (2021).
- Chen, M., et al. Convolutional neural networks for automated annotation of cellular cryo-electron tomograms. Nat Methods. 14 (10), 983-985 (2017).
- Lamm, L., et al. MemBrain v2: an end-to-end tool for the analysis of membranes in cryo-electron tomography. bioRxiv. , (2024).
- Pidhorskyi, S., Morehead, M., Jones, Q., Spirou, G., Doretto, G. syGlass: interactive exploration of multidimensional images using virtual reality head-mounted displays. arXiv. , (2018).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Tegunov, D., Cramer, P. Real-time cryo-electron microscopy data preprocessing with Warp. Nat Methods. 16 (11), 1146-1152 (2019).
- Bepler, T., Kelley, K., Noble, A. J., Berger, B. Topaz-Denoise: general deep denoising models for cryoEM and cryoET. Nat Commun. 11, 5208 (2020).
- Buchholz, T. -. O., Berninger, L., Maurer, M., Jug, F. Cryo-CARE: content-aware image restoration for cryo-transmission electron microscopy data. Proc IEEE Int Symp Biomed Imaging. 16, 502-506 (2019).
- Buchholz, T. -. O., Jordan, M., Pigino, G., Jug, F. Content-aware image restoration for electron microscopy. Methods Cell Biol. 152, 277-289 (2019).
- Liu, Y. -. T., et al. Isotropic reconstruction for electron tomography with deep learning. Nat Commun. 13, 6482 (2022).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. J Struct Biol. 116 (1), 71-76 (1996).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon