Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Quantification des γH2AX Foci en réponse aux radiations ionisantes
Dans cet article
Résumé
La quantification de l'ADN double-brin de stries en utilisant la formation γH2AX comme marqueur moléculaire est devenu un outil précieux dans la radiobiologie. Ici nous démontrons l'utilisation d'un test d'immunofluorescence pour la quantification des γH2AX foyers après exposition des cellules aux radiations.
Résumé
L'ADN des cassures double brin (CDB), qui sont induits soit par des processus métaboliques endogènes ou par des sources exogènes, sont l'une des lésions de l'ADN les plus critiques à l'égard de la survie et la préservation de l'intégrité du génome. Une réponse précoce à l'induction de CDB est la phosphorylation de la H2A variant d'histone, H2AX, à la sérine-139 résidus, dans le très conservé C-terminal motif SQEY, formant γH2AX
Protocole
Préparation des cellules
- Les kératinocytes humains (FEP-1811) ont été cultivées dans des kératinocytes milieu sans sérum (K-GDF; Invitrogen) supplémenté avec du facteur de croissance épidermique, extrait pituitaire bovin et 20 ug / ml de gentamicine, à 37 ° C et CO 2 5%.
- Une suspension cellulaire unique a été préparé en détachant à la trypsine-EDTA (0,05% v / v)
- Les cellules ont été ensemencées dans 8 puits Lab Tek diapositives microchambre II (10.000 cellules / puits) et les diapositives ont été incubées pendant 3 jours à 37 ° C et 5% de CO 2.
L'irradiation
- Les cellules ont été irradiées sur la glace avec 2 Gy en utilisant une source de 137 Cs (Gammacell 1000 irradiateur Elite; Nordion International, ON, Canada; 20,6 secondes / Gy)
- Le contrôle non irradié et 2 Gy cellules irradiées ont été incubées pendant 1 heure à 37 ° C et 5% de CO 2.
Immunofluorescence
- Media a été averti et les cellules ont été lavées avec 300μl de PBS (w / o Ca 2 + ou Mg 2 +) par puits et ont été tourné sur une table de mixage orbitale pendant 5 minutes.
- Le tampon a été averti et 100 pi d'fraîchement préparés à 4% (v / v) paraformaldéhyde a été ajouté à chaque puits et lames ont été incubées à température ambiante pendant 10 minutes.
Toutes les incubations ont été effectuées dans une auge coloration humidifié - Les cellules ont ensuite été lavées avec du PBS (w / o Ca 2 + ou Mg 2 +). Les lames ont été placées dans une jarre de Coplin et tourné sur un mélangeur orbitale pendant 5 minutes. Cette étape de lavage a été répété encore deux fois.
- Le tampon a été averti et l'excès de PBS a été doucement effacé.
- Les cellules ont été perméabilisées avec 100ml Triton X-100 (0,1% v / v) par puits et une incubation de 10 minutes à température ambiante.
- Les cellules ont été lavées avec du PBS (w / o Ca 2 + ou Mg 2 +) tel que décrit dans les étapes 3 et 4 ci-dessus.
- La liaison aux protéines non spécifique a été bloqué avec 100 ml de BSA (1% v / v) par puits et 20 minutes d'incubation à température ambiante.
- Excès de BSA a été averti et 100 pi d'anticorps monoclonal de souris primaires anti-phospho-histone H2AX anticorps (dilution 1:500, dans 1% de BSA; Millipore), a été ajouté à chaque puits pour une incubation de 1 heure à température ambiante.
- Les cellules ont été lavées avec du PBS (w / o Ca 2 + ou Mg 2 +) tel que décrit dans les étapes 3 et 4 ci-dessus et incubées avec 100 pi d'anticorps secondaire (Alexa Fluor 488 de chèvre anti-IgG de souris dilué 1:500, dans 1% de BSA ; Invitrogen) par puits pendant 45 minutes à température ambiante dans l'obscurité. (Anticorps dilué a été maintenu dans l'obscurité pendant toute la procédure)
- Les cellules ont été lavées avec du PBS (w / o Ca 2 + ou Mg 2 +) tel que décrit dans les étapes 3 et 4 ci-dessus. Toutefois, l'exposition à la lumière a été minimisé en utilisant une feuille.
- Contre-coloration nucléaire a été réalisée avec 100ml Topro3 (dilué 1:500; Invitrogen) par puits et une incubation de 10 minutes à température ambiante
- Les cellules ont été lavées avec du PBS (w / o Ca 2 + ou Mg 2 +) tel que décrit dans l'étape 10 ci-dessus.
- Les chambres ont été soigneusement retiré de la glisse, l'excès d'humidité a été effacé et les diapositives ont été autorisés à l'air sec.
- Une goutte de prolonger OR anti-fade solution (Invitrogen) a été ajouté par puits et lames ont été montées (22x50 mm lamelle) et tout excès de liquide autour des bords de lame a été effacé.
- Les lames ont été maintenus dans l'obscurité pendant 30 minutes à température ambiante avant de sceller avec du vernis à ongles.
- Les lames ont été enregistrées durant la nuit à 4 ° C dans l'obscurité avant l'analyse.
Microscopie / Analyse
- Zeiss LSM510 Meta confocale Microscpe utilisé pour acquérir des images à l'aide de la GFP standard (pour γH2AX - Alexa Fluor 488 de chèvre anti-IgG de souris) et les lasers rouge lointain (pour TOPRO-3). Typiquement, une huile de 63 x objectif d'immersion est utilisé.
Les images sont acquises dans un modèle de série Z avec un pas de 0,5 um. Une taille de pas de 0,5 um a été choisi pour minimiser la perte de présenter des foyers dans des plans différents dans les noyaux. Pendant l'analyse, des avions individuels sont déconvolué et empilés pour produire une image maximale projetée pour minimiser le chevauchement des foyers (Haut-chapeau filtre appliqué). - Metamorph (Molecular Devices, USA) a été utilisé pour analyser nombre de foyers.
Le programme quantifie le nombre de foyers dans chaque cellule après que le seuil a été appliqué pour exclure de fond. Les informations sont enregistrées dans un tableur Microsoft Excel pour une analyse ultérieure.

Figure 1. Visualisation par immunofluorescence de γH2AX foyers (en vert) dans les kératinocytes humains non traités et dans les cellules irradiées à 2 Gy et incubé pendant 1 heure à 37 ° C, 5% de CO 2. L'ADN a été coloré avec TOPRO-3 (bleu). Les images ont été acquises en utilisant un microscope Zeiss LSM 510 Meta confocale. Barre = 10 um.
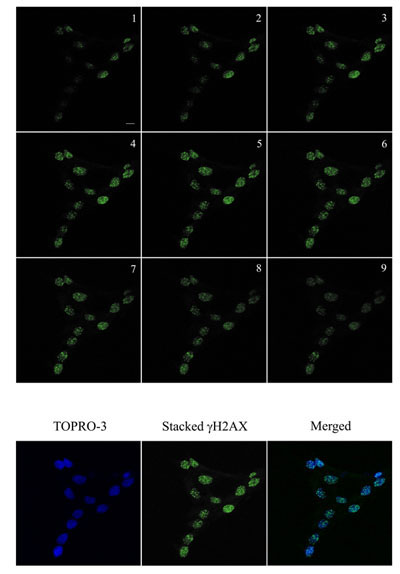
Figure 2. Visualisation par immunofluorescence de γH2AX foyers (en vert) dans les kératinocytes humains et dans les cellules irradiées à 2 Gy et incubé pendant 1 heure à 37 ° C, 5% de CO 2. Les images ont été acquises en utilisant un microscope Zeiss LSM 510 Meta confocale utilisant 0,5 um-Z sectionnement (1-9) pour s'assurer que tous les foyers ont été acquises. Les images a ensuite été empilées pour la quantification en utilisant Metamorph. L'ADN a été coloré avec TOPRO-3 (bleu). Le γH2AX empilés et images bleues ont été empilées pour la visualisation. Barre = 10 um.
Discussion
Après une exposition à des rayonnements ionisants (γ-rayons), γH2AX foyers se forment rapidement et les numéros de foyers atteindre un maximum entre 30-60 minutes 2. Par conséquent, nos 1 point de temps heures après l'irradiation initiale reflète la formation de l'ORD. Nous avons utilisé la dose de rayonnement cliniquement pertinente de 2 Gy pour notre expérience. Cependant, la méthode peut être utilisée pour des doses de rayonnement jusqu'à 4 Gy pour la détection de l'ORD forma...
Déclarations de divulgation
Remerciements
Le soutien de l'Institut australien des sciences et techniques nucléaires est reconnu. TCK a été récipiendaire du prix AINSE. Laboratoire de médecine épigénomique est soutenu par la National Health and Medical Research Council de l'Australie (566 559). LM est soutenue par la recherche de Melbourne (Université de Melbourne) et biomédicale bourses d'imagerie complémentaires CRC. Le soutien de Monash Micro Imaging (Drs Stephen Cody et Iska Carmichael) a été inestimable pour ce travail.
matériels
| Name | Company | Catalog Number | Comments | |
| Keratinocyte-Serum Free Medium (K-SFM) | Media | Invitrogen | 17005042 | Keratinocyte media supplemented with human recombinant Epidermal Growth Factor 1-53 (EGF 1-53), and Bovine Pituitary Extract (BPE). |
| Lab Tek lI Chamber Slides (8-well) | Chamber Slides | Nalge Nunc international | NUN154534 | |
| Coverslips (22x50mm) | Coverslips | Menzel-Glaser | CS2250100 | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A7906 | BSA (1%) is used to block any non-specific antibody binding. Primary and secondary antibodies are diluted in BSA. | |
| PBS (without Ca2+ and Mg2+) | Invitrogen | 17-517Q | ||
| 0.5% Trypsin-EDTA x10 | Invitrogen | 15400-054 | Trypsin-EDTA (0.05%) used to detach cells from culture flasks. | |
| Triton X-100 | Reagent | Sigma-Aldrich | T8787 | Triton X-100 (0.1%) used to permeabilise cells. |
| Paraformaldehyde | Reagent | Sigma-Aldrich | 158127 | Paraformaldehyde (4%) used to fix cells. |
| Mouse monoclonal anti-phospho histone-H2AX antibody | Primary Antibody | EMD Millipore | 16193 | Dilution of primary antibody (1:500), in 1% BSA. |
| Alexa Fluor 488 goat anti-mouse IgG (H+L) | Secondary Antibody | Invitrogen | 11029 | Dilution of secondary antibody (1:500), in 1% BSA. |
| TOPRO3 | DNA Stain | Invitrogen | T3605 | DNA stain commonly used: 4,6-diamidino-2-phenylindole dihydrochloride (DAPI). Can only be used with microscopes with the appropriate excitation laser. |
| ProLong Gold | Anti-fade solution | Invitrogen | P36930 | Refractive index of 1.42 at 20°C. |
| Tissue Culture Flask, Vented Cap | Culture Flask | BD Biosciences | 353112 | |
| Tissue Culture Dish (150x25mm) | Petridish | BD Biosciences | 353025 | |
| Coplin Jar, glass | Grale Scientific P/L | 1771-OG | ||
| Staining Trough | Grale Scientific P/L | V1991.99 | ||
| Gammacell 1000 Elite Irradiator | Gamma Irradiator | Nordion International Inc. | ||
| Zeiss LSM 510 Meta Confocal | Confocal Microscope | |||
| Metamorph | Software for Imaging analysis | Molecular Devices |
Références
- Rogakou, E. P., Boon, C., Redon, C., Bonner, W. M. Megabase chromatin domains involved in DNA double-strand breaks in vivo. J. Cell Biol. 146, 905-916 (1999).
- Bonner, W. M. Gamma H2AX and cancer. Nature Reviews Cancer. 8 (12), 957-967 (2008).
- Savic, V. Formation of Dynamic [gamma]-H2AX Domains along Broken DNA Strands Is Distinctly Regulated by ATM and MDC1 and Dependent upon H2AX Densities in Chromatin. Molecular Cell. 34 (3), 298-310 (2009).
- Downs, J. Binding of chromatin-modifying activities to phosphorylated histone H2A at DNA damage sites. Mol Cell. 16 (6), 979-990 (2004).
- Chowdhury, D. [gamma]-H2AX Dephosphorylation by Protein Phosphatase 2A Facilitates DNA Double-Strand Break Repair. Molecular Cell. 20 (5), 801-809 (2005).
- Nakada, S., Chen, G., Gingras, A., Durocher, D. PP4 is a gamma H2AX phosphatase required for recovery from the DNA damage checkpoint. EMBO Rep. 9 (10), 1019-1026 (2008).
- Altaf, M., Auger, A., Covic, M., Côté, J. Connection between histone H2A variants and chromatin remodeling complexes. Biochem Cell Biol. 87 (1), 35-50 (2009).
- Kusch, T. Acetylation by Tip60 Is Required for Selective Histone Variant Exchange at DNA Lesions. Science. 306, 2084-2087 (2004).
- Hurlin, P. Progression of human papillomavirus type 18-immortalized human keratinocytes to a malignant phenotype. Proc Natl Acad Sci U S A. 88 (2), 570-574 (1991).
- Dickey, J. H2AX: functional roles and potential applications. Chromosoma. , (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon