Method Article
Un test visuel pour surveiller T6SS à médiation bactérienne concurrence
Dans cet article
Résumé
Nous décrivons un test qualitatif de surveiller la concurrence bactérienne médiée par le Pseudomonas aeruginosa sécrétion système VI (T6SS). Le test repose sur la survie / destruction des cellules d'Escherichia coli cibles portant un LacZ-Reporter. Cette technique est réglable afin d'évaluer l'activité bactéricide / bactériostatique de T6SS-compétents micro-organismes.
Résumé
Systèmes de sécrétion de type VI (T6SSs) sont des nanomachines moléculaires permettant bactéries Gram-négatives à transporter et injecter des protéines dans une grande variété de cellules cibles 1,2. Le T6SS est composé de 13 éléments de base et présente des similitudes structurelles avec la queue de bactériophages tube 3. Le phage utilise un tube et un dispositif de perforation pour percer l'enveloppe cellulaire des bactéries cibles et injecter l'ADN. Il est proposé que le T6SS est un dispositif bactériophage inversé la création d'un chemin spécifique dans l'enveloppe cellulaire bactérienne à conduire des effecteurs et de toxines à la surface. Le processus pourrait être plus poussée et l'appareil pourrait perforer T6SS autres cellules de la bactérie qui est en contact, donc l'injection des effecteurs dans ces objectifs. Le tube de queue et des parties de dispositif de perforation de la T6SS sont réalisés avec les protéines et Hcp VgrG, respectivement 4,5.
La polyvalence de la T6SS a été démontrée par ses études utilisant diverses bactéries pathogènes. Les T6SS Vibrio cholerae peuvent remodeler le cytosquelette des cellules hôtes eucaryotes par injection d'un «évolué» VgrG portant un actine C-terminale de réticulation de domaine 6,7. Un autre exemple frappant a été récemment documentés à l'aide de Pseudomonas aeruginosa qui est capable de cibler et de tuer les bactéries d'une manière T6SS-dépendante, favorisant ainsi la création de bactéries dans des niches microbiennes spécifiques et l'environnement concurrentiel 8,9,10.
Dans ce dernier cas, trois T6SS-protéines sécrétées, à savoir Tse1, TSE2 et tse3 ont été identifiés comme les toxines injectées dans les bactéries cibles (figure 1). La cellule donneuse est protégé contre les effets délétères de ces effecteurs par l'intermédiaire d'un mécanisme anti-toxine, médiée par les protéines de l'immunité TSI1, Tsi2 et Tsi3 8,9,10. Cette activité antimicrobienne peut être surveillée en cas T6SS-compétents bactéries sont co-cultivés sur sOlid surfaces en concurrence avec d'autres espèces bactériennes ou avec T6SS-inactifs les bactéries de la même espèce 8,11,12,13.
Les données disponibles accent sur une approche numérique pour le test de compétition bactérienne, y compris le comptage CFU temps que dépend en grande partie responsables des antibiotiques. Dans le cas de souches résistantes aux antibiotiques comme P. aeruginosa, ces méthodes peuvent être inappropriés. En outre, avec l'identification d'environ 200 différents loci T6SS dans plus de 100 génomes bactériens 14, un outil de dépistage pratique est hautement souhaitable. Nous avons développé un test qui est facile à utiliser et nécessite du matériel de laboratoire standard et de réactifs. La méthode propose une technique rapide et qualitative de surveiller l'activité bactéricide dépendant T6SS / bactériostase en utilisant une souche journaliste comme une proie (dans ce cas, Escherichia coli DH5a) permettant une complémentation 'du gène lacZ. Dans l'ensemble, cette méthode est graphic et permet l'identification rapide des phénotypes liés T6SS sur des plaques d'agar. Ce protocole expérimental peut être adapté à d'autres souches ou espèces bactériennes en tenant compte des conditions spécifiques, tels que les milieux de croissance, de la température ou du temps de contact.
Protocole
1. Souches bactériennes et les cultures
- Ingénieur une cellule d'Escherichia coli destinataire (Prey, P) par la transformation (en utilisant la norme de CaCl2 traitement ou électroporation) 15 E. coli DH5a cellules avec un plasmide permettant la complémentation d'une-du gène lacZ (tableau 1). Plaque les cellules transformées sur des plaques d'agar Luria-Bertani (LBA, l'agar 1,5%) contenant du 5-bromo-4-chloro-indolyl-β-D-galactopyranoside (X-gal) à 40 mg / ml de concentration finale et un antibiotique approprié. Incuber à 37 ° C pendant la nuit et le lendemain matin sélectionner les transformants bleu (voir § 1.3).
- Cultiver les cellules de Pseudomonas aeruginosa donateurs qui sont T6SS-actif (D +) ou T6SS-inactive (D-) (tableau 1) toute la nuit LBA à 37 ° C. Dans l'exemple présenté ici, nous avons utilisé un P. TER aeruginosa souche possédantun H1-T6SS constitutivement active 16 et un mutant isogénique avec une délétion du gène H1-grappe T6SS (tableau 1).
- Le lendemain, préparer une culture de nuit de liquide dans un flacon aéré par inoculation d'un clone bleu vif parmi les E. transformants coli (P) de la plaque décrite au § 1.1, dans 5 ml de bouillon de soja tryptique (TSB) additionné de l'antibiotique approprié. Croître sous agitation à 37 ° C. Procéder également avec une seule colonie de (D +) et de (D -) de la plaque décrite au § 1.2.
2. Test de compétition
- Préparer les plaques LBA pour l'expérience lendemain. Assurez-vous que ces plaques sont correctement séché (soit près du brûleur Bunsen ou hotte à flux laminaire). Préparez un "test d'entrée" (A-Entrée) plaque pour les souches (D +), (D -) et (P ). Préparez un "test-output" (A-sortie) pour les souches plaque (D - + P) et (D + + P). Diviser pour marquer les plaques en conséquence.
- Après une croissance de nuit (voir § 1.3), mesurer la densité optique (DO 600 nm) de la culture bactérienne d'entrée (D - D + et P) et calculer le volume nécessaire pour obtenir une densité cellulaire équivalent à 1 unité de DO 600 nm de chaque souche . Chaque culture "input" cellule (D - D + et P) est d'abord recueillie dans un 1.5 stériles tubes Eppendorf ml.
- Centrifuger les échantillons bactériens à 13.000 rpm pendant 1 min à température ambiante et jeter le surnageant.
- Remettre en suspension les culots de la D -, D + cultures, et P, dans 100 ul de frais TSB par pipetage doux et inoculer 10 ml de chaque cordant souche comme un seul point sur la "A-entrée" plaque (préparé au § 2.1).
- Inoculer le "A-sortie" plaque (préparé au § 2.1). Plus précisément, mélanger doucement 30 ul de (D +) avec 30 pi (P) et 30 ul de (D -) avec 30 ml (P) dans deux tubes Eppendorf distinctes (notez que les cultures utilisées sont celles décrites au § 2.4) . Inoculer 20 ul du mélange (D + / P) et (D - / P) sous forme de points individuels sur la "A-sortie" plaque. Laisser sécher les gouttes à proximité d'un bec Bunsen et placer la plaque dans un incubateur à 37 ° C pendant une période de temps appropriée au cours de laquelle l'effet bactéricide est en cours. Dans le cas de P. aeruginosa, une mise à mort efficaces bactérienne n'est observée après 5 heures d'incubation lorsque l'on compare un T6SS actifs avec une souche T6SS défectueux.
3. Observation qualitative de til bactéricide
- Préparer les plaques LBA contenant 40 ug / ml de X-gal pour la lecture de l'essai. Les plaques seront appelés "entrée Lecture" ou "entrée R" pour repérer les bactéries isolées ou les "Lecture" ou "R-sortie" pour repérer cultures mixtes de bactéries et donc lu la performance tuer. Ces plaques doivent également être séché. Diviser la plaque en quatre parties égales sur son dos et annoter ces pièces 0, 10 -1, 10 -2 et 10 -3, désignant les dilutions de culture bactérienne à repérer sur les plaques d'agar (voir § 3.3). La dilution permet une évaluation semi-quantitative du rapport bleu / blanc bactéries dans le même endroit.
- Ramasser à l'aide d'une anse stérile les taches individuelles bactériennes de l'"Une entrée" plaque (voir § 2.4) et "A-sortie" plaque (voir § 2.5) et remettre chaque point dans différents tubes Eppendorf de 1,5 ml contenant 1 ml de TSB. Placez-les dans un bloc agitateur Eppendorf pendant 30 min pour remettre efficacement l'bactéries.
- Préparer une série de 5 fois des tubes Eppendorf 3 contenant 900 ul de TSB et procéder pour chaque tache remis en suspension à dix dilutions allant jusqu'à 10 -3. Assurez-vous de changer les embouts de pipette et de vortex 5 secondes entre chaque étape de dilution.
- Passez à l'repérage de vos dilutions bactériennes préparées au § 3.3 sur les plaques LBA bien séchées contenant 40 ug / ml de X-gal, en commençant par la plus diluée à la suspension non diluée, ce qui rend ces les "R-" plaques d'entrée pour le "A d'intrants et les "points" R-sortie "pour les plaques" A-"points de sortie (figure 2). Vortex brièvement chaque tube avant de procéder à la repérer à maintenir une suspension homogène bactérienne. Pour la reproductibilité des résultats, spot de 20 pi en trois exemplaires au sein d'un quart de cercle (figure 2). La sécheresse relative de votre assiette est important à ce stade car les points doivent rester séparés individuellement sur la plaque et ne glissera pas vers l'autre.
Une quantification de l'essai de compétition est également possible à ce stade de l'expérimentation. Après l'étape 3.3, épandage 100 ul de dilution des 10 -3 sur des plaques LBA contenant 40 ug / ml de X-gal. Pour la reproductibilité des résultats, procéder à un placage en trois exemplaires pour chaque point de la "A-sortie" plaque. Placez les plaques de dilution dans un incubateur à 37 ° C pendant la nuit (ce qui correspond à 16 heures d'incubation). Procéder au comptage des colonies bleues (les cellules qui sont restés en vie P). Des résultats typiques obtenus après 5 heures de compétition sont présentés dans la figure 3. - Laisser la plaque (sans couvercle) ouverte dans une zone stérile pour permettre l'absorption du liquide en excès à l'intérieur de chaque tache.
- Placer la plaque dans un incubateur à 37 ° C pendant la nuit. Pendant ce temps d'incubation, la mort est toujours en cours dans le mélange (D + / P) puisque les deux souches sont toujours en contact.
- Prendre une photoou numériser vos assiettes pour l'analyse de sortie (Figure 2). Où points restent en grande partie bleue indique que E. coli n'a pas été tué par P. aeruginosa. C'est le cas lorsque E. coli est mélangé avec un p-T6SS défectueuse aeruginosa souche (D-) (figure 2, la plaque en bas à droite).
Résultats
Des résultats typiques sont présentés dans la figure 1, les souches et les réactifs décrits dans le tableau 1. Les plaques représentées dans cette figure ont été scannés après une nuit d'incubation. Les «Lecture-Input" plaques montrent un modèle de dilution en série pour les souches utilisées dans cet essai. Comme prévu, le E. coli proie points (P) surexprimant le gène lacZ apparaissent en bleu sur des milieux supplémentés avec X-gal, tandis que le P. donateurs aeruginosa (D +, T6SS actif) et (D -, inactif T6SS) restent blancs. Les «Lecture-sortie" plaques sur lesquelles le mélange entre la proie et une souche T6SS actif (D + / P) a été repéré montrent la disparition du bleu proie indiquant ainsi qu'il a été tué. Ceci démontre la capacité du donneur à supplanter la proie. La persistance de la couleur bleue sur le (D - / P) Plaque démontre l'incapacité d'un donneur T6SS inactif pour tuer la proie bleu.
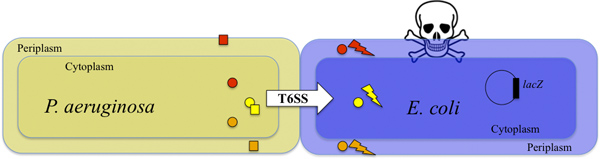
Figure 1. Meurtre de E. coli par T6SS compétents dans P. aeruginosa. P. aeruginosa injecte des toxines dans l'E. cellule cible coli d'une manière T6SS-dépendant (indiqué par la flèche blanche). Deux toxines et Tse1 tse3 (orange et rouge cercles) sont injectés dans le E. périplasme coli et de dégrader le peptidoglycane 9. La toxine TSE2 (cercle jaune) est injecté dans le E. coli cytoplasme et possède une action bactériostatique 8,10. L'action combinée des toxines tue les cellules cibles (l'éclair et le crâne). La survie des cellules cibles peuvent être détectées en surveillant l'activité de la β-galactosidase produite (voir aussi la figure 2). P. uneruginosa est protégé contre l'activité des toxines par les protéines de l'immunité TSI1, Tsi2 et Tsi3 (orange, carrés jaunes et rouges, respectivement) 8,9,10.
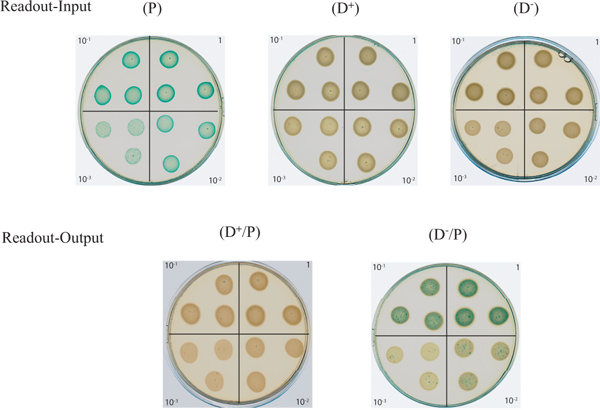
Figure 2. Essai sur plaque de gélose pour surveiller T6SS dépendant de la bactéricidie Dans cette figure sont représentés sur la partie supérieure des «Lecture-Input" plaques comprenant des dilutions en série du D -., D + et les cellules d'entrée P. Les cellules d'entrée P sont bleues en raison de l'α-complémentation du gène lacZ et donc à des β-galactosidase qui clive X-gal. Dans la partie inférieure est montré les «Lecture-sorties" plaques constituées de dilutions successives du mélange bactérienne entre un actif (D + / P) , ou un inactif (D - / P), T6SS donneur P. souche E. aeruginosa avec l' coli proie. Cliquez ici pour agrandir la figure .
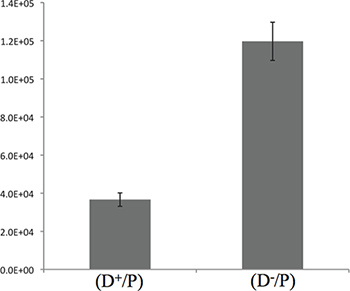
Figure 3. Quantification de l'assassinat de E. coli par P. aeruginosa après 5 heures d'incubation. Le graphique présente le comptage CFU de E. coli décrite dans l'étape de 3,4. Les résultats présentés ici montrent une différence de 3 fois entre le T6SS + et les T6SS-souches, ce qui suggère que la plupart des meurtres se déroule pendant les 5 premières heures de contact.
Discussion
La méthode présentée dans cet article permet une observation visuelle de T6SS activité médiée / bactériostatique bactéricide. Le dosage est réalisé sur la surface d'une plaque de gélose. Il a été précédemment montré que le dosage en fonction de meurtre T6SS réalisée avec liquide de culture bactérienne mixte n'est pas efficace, probablement en raison de l'absence d'un contact permanent entre les deux bactéries 8. Le T6SS est censé fonctionner avec un mécanisme semblable à celui utilisé par les bactériophages pour injecter l'ADN dans des 17 cellules cibles. En culture liquide, la structure en forme de tube de l'T6SS peut se casser plus facilement, entre-bactérien de contact peuvent être perdus, et les toxines ne sont pas fournis efficacement.
En termes de temps d'incubation, les 5 premières heures de contact que nous décrivons entre la souche donneuse et la proie sont suffisantes pour observer la bactéricidie entre P. aeruginosa et E. coli, comme illustré dans la figure 3 . Néanmoins, il est conseillé de régler le temps d'incubation en effectuant une cinétique afin d'optimiser les conditions expérimentales.
Étant donné que cette méthode est une technique basée sur la couleur, les résultats de sortie peut être compromise par la pigmentation de la souche donneuse. Par exemple, dans le cas de P. aeruginosa, certaines souches produisent des niveaux élevés de pigments colorés tels que pyocyanine et pyoverdine, qui peuvent interférer avec la lecture d'essai, ce qui rend la distinction de la proie relativement difficile. D'autres chromogènes β-galactosidase substrats, tels que le magenta ou le gal gal rouge, peut être utilisé à la place du X-gal (tableau 1).
Le test de compétition peut faire usage de gènes rapporteurs autres pour la lecture. Par exemple, le dosage similaire a également été réalisée en utilisant la protéine fluorescente verte marquée proies 12.
Notre analyse, tout en n'étant pas quantitative, donne une bonne indicationde l'activité T6SS car elle est basée sur la survie ou la mort d'un journaliste proie. Cette technique présente l'avantage d'être simple et pratique pour évaluer l'activité bactéricide / bactériostatique de T6SSs de toutes les espèces bactériennes. Jusqu'à présent, l'activité de la T6SS a été démontré contre les bactéries Gram-négatives et aucun exemple clair de T6SS sensibles à bactéries Gram-positives a été signalé à ce jour 12. Il est également évident que l'incompatibilité de la culture des différentes espèces bactériennes à tester (par exemple, la croissance de la température, l'oxygénation, les médias spécifiques) doit être pris en considération.
Notre test peut également être utilisé pour évaluer laquelle des composants T6SS sont absolument indispensables puisque même les traces d'une toxine sécrétée pourrait être suffisante pour tuer la proie. Même une faible activité des T6SS pourrait alors être clairement détectée par notre analyse par rapport à la procédure standard de test T6SS-dépendante la sécrétion de l'aide blot du surnageant de culture et de l'Ouestanalyse. Cependant, une bonne unité formant colonie (UFC) de comptage est toujours nécessaire pour la quantification précise de cette activité T6SS.
Déclarations de divulgation
Nous n'avons rien à déclarer.
Remerciements
Ce travail a été financé par le Wellcome Trust WT091939MA subvention. Alain Filloux est soutenue par la Royal Society.
matériels
| Name | Company | Catalog Number | Comments |
| Nom de réactif / Matériel | Entreprise | Numéro de catalogue | Commentaires |
| P. aeruginosa PAKΔ TER (D +) (T6SS actifs) | Souche de laboratoire | Décrit dans Référence 16 | |
| P. aeruginosa PAKΔ TER Δ S1-T6SS (D -) (T6SS inactifs) | Cette étude | Le groupe H1-T6SS (englobant les gènes PA0070 PA0095 à) a été supprimé par échange allélique suivant la procédure décrite dans la référence 18. Le fragment mutateur a été généré avec l'ensemble d'amorces suivant: Les amorces fragment Up: 5'-ATGGTCAACGACATGGAGCTGGAG-3 ', et 5'CGAGGCCGATCAGGCCTTCAGAACTGA-3 '. Les amorces de fragments Down: 5'-TCAGGCCTTCAGAACTGAAGCGGCGCA-3 ', 5'-GGTGGCGTTCAACAGTTCCATGTC-3 ' | |
| E. coli DH5a | Invitrogen | 18258-012 | F-φ80 lac ZΔM15 Δ (lac ZYA-arg F) U169 rec A1 A1 fin hsd R17 (r k -, m k +) pho Un soutien E44 λ-thi -1 gyr A96 rel A1 |
| pBluescript II SK (+) | Agilent | 212205 | Ce vecteur exprime le peptide β α-galactosidase d'utiliser pour α-complémentation. |
| X-gal | Invitrogen | 15520-018 | Utilisez à 40 pg / ml |
| Luria Bertani agar | Merck-chimiques | 1.10283.0500 | |
| TSB (caséine de soja bouillon) | Oxoid | CM109 | |
| Vortex Agitateur Genius 3 | IKA | 3340000 | |
| Scanner | Epson | V700 | |
| Spectrophotomètre | WPA Biowave | CO8000 Densimètre portable | |
| Magenta-gal | Bioworld | 30350001-1 (715241) | |
| Red-gal | Organiques recherche | 1364c | |
Tableau 1. Souches, plasmide, la matière et le réactif utilisé. |
Références
- Filloux, A., et al. The bacterial type VI secretion machine: yet another player for protein transport across membranes. Microbiology. 154 (6), 1570(2008).
- Cascales, E., Cambillau, C. Structural biology of type VI secretion systems. Philos. Trans. R. Soc. Lond. B. Biol. Sci. 367 (1592), 1102(2012).
- Leiman, P. G., et al. Morphogenesis of the T4 tail and tail fibers. Virol. J. 7, 355(2010).
- Ballister, E. R., et al. In vitro self-assembly of tailorable nanotubes from a simple protein building block. Proc. Natl. Acad. Sci. U.S.A. 105 (10), 3733(2008).
- Leiman, P. G., et al. Type VI secretion apparatus and phage tail-associated protein complexes share a common evolutionary origin. Proc. Natl. Acad. Sci. U.S.A. 106 (11), 4154(2009).
- Pukatzki, S., et al. Type VI secretion system translocates a phage tail spike-like protein into target cells where it cross-links actin. Proc. Natl. Acad. Sci. U.S.A. 104 (39), 15508(2007).
- Ma, A. T., et al. Translocation of a Vibrio cholerae type VI secretion effector requires bacterial endocytosis by host cells. Cell Host Microbe. 5 (3), 234(2009).
- Hood, R. D., et al. A type VI secretion system of Pseudomonas aeruginosa targets a toxin to bacteria. Cell Host Microbe. 7 (1), 25(2010).
- Russell, A. B., et al. Type VI secretion delivers bacteriolytic effectors to target cells. Nature. 475 (7356), 343(2011).
- Li, M., et al. Structural basis for type VI secretion effector recognition by a cognate immunity protein. PLoS Pathog. 8 (4), e1002613(2012).
- Zheng, J., et al. Genetic analysis of anti-amoebae and anti-bacterial activities of the type VI secretion system in Vibrio cholerae. PLoS One. 6 (8), (2011).
- Schwarz, S., et al. Burkholderia type VI secretion systems have distinct roles in eukaryotic and bacterial cell interactions. PLoS Pathog. 6 (8), e23876(2010).
- Murdoch, S. L., et al. The opportunistic pathogen Serratia marcescens utilizes type VI secretion to target bacterial competitors. J. Bacteriol. 193 (21), 6057(2011).
- Boyer, F., et al. Dissecting the bacterial type VI secretion system by a genome wide in silico analysis: what can be learned from available microbial genomic resources? BMC Genomics. 10, 104(2009).
- Maniatis, T., et al. Molecular cloning: A Laboratory Manual. , Cold Spring Harbor Laboratory. Cold Spring, NY. (1982).
- Mougous, J. D., et al. A virulence locus of Pseudomonas aeruginosa encodes a protein secretion apparatus. Science. 312 (5779), 1526(2006).
- Records, A. R., et al. The type VI secretion system: a multipurpose delivery system with a phage-like machinery. Mol. Plant Microbe Interact. 24 (7), 751(2011).
- Hachani, A., et al. Type VI secretion system in Pseudomonas aeruginosa: secretion and multimerization of VgrG proteins. J. Biol. Chem. 286 (14), 12317(2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon