Method Article
Um Ensaio visual para o Monitor T6SS mediada Competição bacteriana
Neste Artigo
Resumo
Descreve-se um ensaio qualitativo para monitorar competição bacteriana mediada pela Pseudomonas aeruginosa Tipo de sistema de secreção VI (T6SS). O ensaio baseia-se na sobrevivência / morte de células de Escherichia coli que transportam um alvo LacZ Repórter. Esta técnica é ajustável para avaliar a actividade bactericida / bacteriostase de T6SS-proficientes microorganismos.
Resumo
Sistemas de secreção de tipo VI (T6SSs) são nanomáquinas moleculares que permitem bactérias Gram-negativas e para o transporte de proteínas para injectar uma ampla variedade de células de 1,2-alvo. O T6SS é composta por 13 componentes do núcleo e exibe semelhanças estruturais com a cauda de bacteriófagos tubo 3. O fago usa um tubo e um dispositivo de puncionar para penetrar o envelope celular das bactérias alvo e injectar o DNA. Propõe-se que o T6SS é um dispositivo de bacteriófago invertido criação de um caminho específico no envelope da célula bacteriana para dirigir efectores e toxinas para a superfície. O processo pode ser levado mais longe e o dispositivo pode perfurar T6SS outras células com as quais a bactéria estiver em contacto, assim injectar os efectores para estes alvos. O tubo de cauda e partes do dispositivo de punção dos T6SS são feitos com Hcp e proteínas VgrG, respectivamente 4,5.
A versatilidade do T6SS tem sido demonstrada através datudies usando vários patógenos bacterianos. Os T6SS Vibrio cholerae pode remodelar o citoesqueleto das células hospedeiras eucarióticas injectando uma "evoluiu" VgrG carregando uma actina C-terminal de ligação cruzada de domínio 6,7. Outro exemplo notável foi recentemente documentado usando Pseudomonas aeruginosa, que é capaz de atacar e eliminar as bactérias de uma maneira dependente da T6SS, promovendo assim o estabelecimento de bactérias em nichos microbianos específicos e ambiente competitivo 8,9,10.
Neste último caso, três T6SS-secretadas proteínas, ou seja, Tse1, Tse2 Tse3 e têm sido identificados como as toxinas injectados na bactéria alvo (figura 1). A célula do dador é protegido contra o efeito prejudicial destes efectores por meio de um mecanismo de anti-toxina, mediados pelas proteínas Tsi1, Tsi2 e Tsi3 imunidade 8,9,10. Esta actividade anti-microbiana pode ser monitorizado quando T6SS-proficientes bactérias são co-cultivados em sOlid superfícies em competição com outras espécies bacterianas ou com T6SS-inactivos bactérias da mesma espécie 8,11,12,13.
Os dados disponíveis enfatizou uma abordagem numérica para o ensaio de competição de bactérias, incluindo demorada contagem CFU que depende em grande parte responsáveis antibióticas. No caso de estirpes resistentes aos antibióticos, como P. aeruginosa, estes métodos podem ser inadequadas. Além disso, com a identificação de cerca de 200 diferentes loci T6SS em mais de 100 genomas bacterianos 14, uma ferramenta de rastreio conveniente é altamente desejável. Foi desenvolvido um ensaio que é fácil de utilizar e requer materiais e reagentes de laboratório padrão. O método oferece uma técnica rápida e qualitativa para monitorizar a actividade bactericida dependente do T6SS / bacteriostase usando uma estirpe repórter como presa (neste caso, a Escherichia coli DH5a), permitindo uma complementação do gene lacZ. No geral, este método é graphic e permite a rápida identificação de fenótipos relacionados T6SS em placas de agar. Este protocolo experimental pode ser adaptado para outras estirpes ou espécies bacterianas, tomando em consideração as condições específicas, tais como meios de crescimento, a temperatura ou o tempo de contacto.
Protocolo
1. As cepas bacterianas e Culturas
- Um engenheiro Escherichia coli célula receptora (Prey, P), transformando (usando padrão de CaCl 2 tratamento ou eletroporação) 15 E. coli DH5a células com um plasmídeo que permita a complementação do gene lacZ (Tabela 1). Placa das células transformadas em placas de ágar Luria-Bertani (LBA, agar 1,5%) contendo 5-bromo-4-cloro-indolil-β-D-galactopiranosido (X-gal) a 40 mg / ml de concentração final e antibiótico apropriado. Incubar a 37 ° C durante a noite e na manhã seguinte para selecionar transformantes azul (ver § 1.3).
- Crescer as células de Pseudomonas aeruginosa de dadores que são T6SS-activo (D +) ou inactivos T6SS-(D-) (Tabela 1) em LBA durante a noite a 37 ° C. No exemplo aqui apresentado foi utilizado um P. Rets aeruginosa estirpe possuindoum H1-T6SS constitutivamente activo 16 e um mutante isogênica com uma deleção do aglomerado de genes de H1 T6SS-(Tabela 1).
- No dia seguinte, preparar uma cultura durante a noite de líquido no frasco gaseificado através da inoculação de um clone azul brilhante entre a E. Os transformantes de E. coli (P) a partir da placa, conforme descrito em § 1.1, em 5 ml de caldo de soja tríptica (TSB) suplementada com o antibiótico apropriado. Crescer sob agitação a 37 ° C. Proceder igualmente com uma única colónia de (D +) e de (D -) a partir da placa, conforme descrito em § 1.2.
2. Ensaio de competição
- Prepare placas LBA para o experimento dia seguinte. Certifique-se estas placas são bem seco (ou perto do bico de Bunsen ou em câmara de fluxo laminar). Prepara-se uma placa de "Ensaio de entrada" (A-Input) para as amostras (D +), (D -), e (P ). Prepara-se uma "Ensaio de saída" (A saída) da placa para as estirpes (D - + P) e (D + + P). Dividir e rotular as placas em conformidade.
- Após crescimento durante a noite (ver § 1.3), medir a densidade óptica (DO 600 nm) da cultura bacteriana de entrada (D -, D + e P) e calcular o volume necessário para obter uma densidade celular equivalente a 1 unidade de OD 600nm de cada estirpe . Cada "entrada" cultura de células (D -, D + e P) é inicialmente coletado em um recipiente estéril tubos de 1,5 ml Eppendorf.
- Centrifugar as amostras bacterianas a 13.000 rpm durante 1 min à temperatura ambiente e desprezar o sobrenadante.
- Voltar a suspender as pílulas da D -, as culturas de D +, P e, em 100 ul de TSB fresco por pipetagem suave e inocular 10 ml de cada correspondente estirpe como uma única mancha no "A entrada de" prato (preparado em § 2.1).
- Inocular o "A saída de" placa (preparado no § 2.1). Mais precisamente, misturar suavemente ul de 30 (D +) com 30 ul de (P) e 30 ul de (D -) com 30 ml (P) em dois tubos Eppendorf separados (note que as culturas utilizadas são as descritas em § 2.4) . Inocular 20 ul da mistura (D + / P) e (D - / P) como pontos individuais na "A-saída" placa. Permitir que os pontos de secar de um queimador de Bunsen próxima e colocar a placa numa incubadora a 37 ° C, durante um período adequado de tempo durante o qual a morte bacteriana está a ter lugar. No caso de P. aeruginosa, uma matança eficaz bacteriana é observada após 5 horas de incubação quando se compara um T6SS activas com uma estirpe T6SS defeituoso.
3. A observação qualitativa da tele Killing bacteriana
- Preparar as placas de LBA contendo 40 ug / ml de X-gal para a leitura do ensaio. As placas serão chamados de "entrada de Leitura" ou "R-entrada" para detectar bactérias isoladas ou "saída de Leitura" ou "R-saída" para detectar culturas mistas de bactérias e assim ler o desempenho de matar. Estas placas também devem ser bem seco. Divida o prato em quatro partes iguais em suas costas e anotar essas peças 0, 10 -1, 10 -2 e 10 -3, designando as diluições de cultura bacteriana de detectar as placas de ágar (ver § 3.3). A diluição permitirá uma avaliação semi-quantitativa da taxa de bactérias azul / branco dentro do mesmo local.
- Recolher com uma agulha esterilizada os pontos individuais de bactérias do "A entrada de" placa (ver § 2.4) e "A-saída" placa (ver § 2.5) e ressuspender cada local em diferentes tubos de 1,5 ml Eppendorf contendo 1 ml de TSB. Coloque em um agitador Eppendorf bloco durante 30 minutos para voltar a suspender de forma eficiente obactérias.
- Prepara-se uma série de 5 vezes 3 tubos de Eppendorf contendo 900 uL de TSB e prossiga para cada mancha ressuspenso a 10 diluições em série até 10 -3. Certifique-se de trocar a ponteira e vortex 5 segundos entre cada etapa de diluição.
- Avance para a focagem de suas diluições bacterianas preparadas em § 3.3 em placas bem secas LBA contendo 40 ug / ml de X-gal, começando com o mais diluída à suspensão não diluída, tornando estes os "R-" placas de entrada para o "A -entrada "pontos e os" R-saída "placas para o" A-saída "pontos (Figura 2). Vortex brevemente cada tubo, antes de prosseguir para a mancha para manter uma suspensão homogénea bacteriana. Para a reprodutibilidade dos resultados, o ponto 20 ul, em triplicado, num quadrante (Figura 2). A secura relativa da sua placa é importante, nesta fase, uma vez que as manchas têm de permanecer separados individualmente na placa e não deslizam um contra o outro.
A quantificação do ensaio de competição é também possível nesta fase do experimento. A seguir ao passo 3.3, espalhar 100 ul de uma diluição 10 -3 em placas LBA contendo 40 ug / ml de X-gal. Para a reprodutibilidade dos resultados, a uma cultura em placas em triplicado para cada ponto do "A-saída" placa. Colocar as placas de diluição de 37 ° C incubadora durante a noite (o que corresponde a 16 horas de incubação). Proceder à contagem das colônias azuis (as células P que permaneceram vivos). Os resultados típicos obtidos após 5 horas de competição são apresentados na Figura 3. - Deixar a placa aberta (sem tampa) dentro de uma zona estéril, para permitir a absorção do líquido em excesso, dentro de cada local.
- Colocar a placa numa incubadora a 37 ° C durante a noite. Durante este período de incubação, a morte ocorre ainda na mistura (D + / P) a partir de ambas as estirpes estão ainda em contacto.
- Tire uma fotoou digitalizar suas chapas para a análise de saída (Figura 2). Onde manchas permanecem em grande parte azul indica que E. coli não foi morto por P. aeruginosa. É esse o caso quando E. coli é misturado com um p-T6SS defeituoso estirpe aeruginosa (D-) (Figura 2, a placa na parte inferior direita).
Resultados
Os resultados típicos são mostrados na Figura 1 com as estirpes e os reagentes descritos na Tabela 1. As placas mostradas nesta figura foram verificados após uma incubação de um dia para o outro. Os "Leitura-Input" placas mostram um padrão de diluição em série para as estirpes utilizadas neste ensaio. Como esperado, a E. coli rapina pontos (P) com superexpressão do gene lacZ azul aparecem no meio suplementado com X-gal, enquanto que a P. doador aeruginosa (D +, T6SS ativa) e (D -, inativos T6SS) permanecem branco. Os "Leitura-saída" placas em que a mistura entre a presa e uma estirpe T6SS ativa (D + / P) foi flagrada mostram o desaparecimento do azul presa, assim, indicando que foi morto. Isto demonstra a capacidade do doador para outcompete a presa. A persistência da cor azul na (D - / P placa) demonstra a incapacidade de um doador T6SS inativo para matar a presa azul.
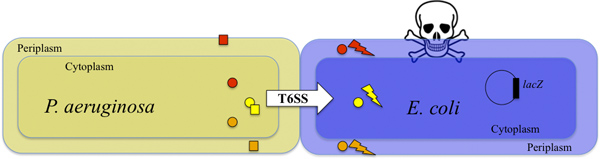
Figura 1. Matança de E. coli por T6SS-proficiente P. aeruginosa. P. aeruginosa injeta toxinas no E. coli célula alvo de uma forma dependente da T6SS (indicado pela seta branca). Duas toxinas Tse1 e Tse3 (laranja e círculos vermelhos) são injetados na E. periplasma coli e degradar o peptidoglicano 9. A toxina Tse2 (círculo amarelo) é injetado no E. coli citoplasma e tem uma atividade bacteriostática 8,10. A ação combinada das toxinas mata as células-alvo (relâmpago e crânio). A sobrevivência de células alvo pode ser detectada por monitorização da actividade de a-galactosidase produzida β (ver também a Figura 2). P. umeruginosa é protegida contra a actividade das toxinas pelas proteínas de imunidade Tsi1, Tsi2 e Tsi3 (laranja, quadrados amarelos e vermelhos, respectivamente) 8,9,10.
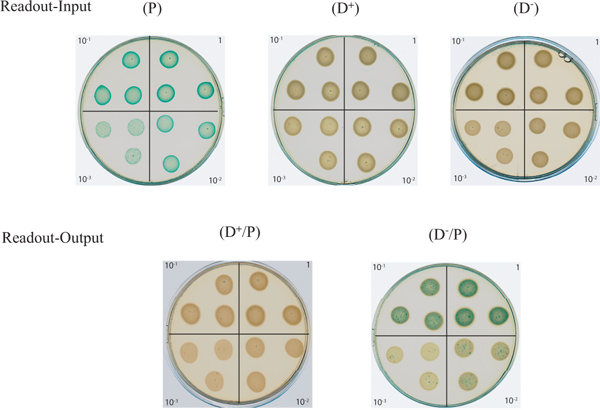
Figura 2. Ensaio de placa de agar para monitorar T6SS dependente de morte bacteriana Nesta figura são mostradas na parte superior do "input" Leitura placas constituídas por diluições em série do D -., D + e células de entrada P. As células de entrada P são azuis devido ao α-complementação do gene lacZ e assim produzido β-galactosidase que cliva X-gal. Na parte mais inferior é mostrada a "Leitura-Output" placas constituídas por uma série de diluições da mistura de bactérias entre um activo (D + / P) , ou um inactivo (D - / P), T6SS doador P. aeruginosa com a estirpe de E. coli presa. Clique aqui para ver maior figura .
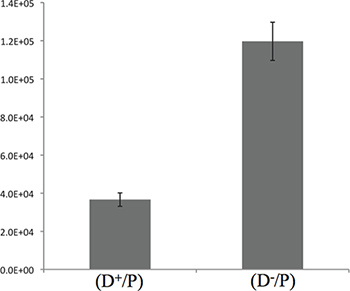
Figura 3. Quantificação da morte de E. coli por P. aeruginosa, após 5 horas de incubação. O gráfico apresenta a contagem de CFU de E. coli descrito no passo 3.4. Os resultados aqui apresentados mostram uma diferença de 3 vezes entre o T6SS + e as T6SS-estirpes, sugerindo que a maior parte da morte tem lugar durante as 5 primeiras horas de contacto.
Discussão
O método apresentado neste artigo permite uma observação visual de T6SS mediada atividade bactericida / bacteriostase. O ensaio é efectuado sobre a superfície de uma placa de agar. Foi previamente demonstrado que T6SS dependente de ensaio realizado com matança cultura bacteriana mista líquido não é eficaz, provavelmente devido à falta de contacto constante entre as duas bactérias 8. O T6SS é acreditado para operar com um mecanismo semelhante ao utilizado por bacteriófagos para injectar o DNA em células-alvo 17. Na cultura líquido, a estrutura tubular do T6SS podem quebrar com mais facilidade, inter-bacteriana contato pode ser perdida, e as toxinas não são eficientemente entregue.
Em termos de tempos de incubação, as 5 primeiras horas de contacto que descrevem entre a estirpe doadora e a presa são suficientes para observar a morte bacteriana entre P. aeruginosa e E. coli, tal como ilustrado na Figura 3 . No entanto, é aconselhável ajustar o tempo de incubação através da realização de uma cinética de modo a optimizar as condições experimentais.
Uma vez que este método é uma técnica baseada em cor, os resultados de saída pode ser comprometida pela pigmentação da estirpe doadora. Por exemplo, no caso do P. aeruginosa, algumas linhagens produzem níveis elevados de pigmentos de cor, tais como piocianina e pyoverdine, o que pode interferir com a leitura de ensaio, fazendo com que a distinção da presa relativamente difícil. Outros cromogénicos β-galactosidase substratos, tais como o magenta gal-gal ou o vermelho-, podem ser usados em vez de X-gal (Tabela 1).
O ensaio de competição pode fazer uso de outros genes repórter para a leitura. Por exemplo, o ensaio semelhante foi também realizada usando a proteína verde fluorescente marcado com presas 12.
O nosso ensaio, ao passo que não quantitativas, dá uma boa indicaçãoda actividade T6SS uma vez que é baseada na sobrevivência ou a morte de uma presa repórter. Esta técnica apresenta a vantagem de ser fácil e conveniente para avaliar a actividade bactericida / bacteriostase de T6SSs de quaisquer espécies de bactérias. Até agora, a actividade do T6SS foi demonstrado contra bactérias Gram-negativas e nenhum exemplo claro de T6SS sensíveis bactérias Gram-positivas foi relatado ainda 12. É também óbvio que a incompatibilidade na cultura das diferentes espécies bacterianas para testar (por exemplo, temperatura de crescimento, oxigenação, meios específicos), deve ser considerado.
O nosso ensaio pode também ser utilizado para avaliar quais dos componentes T6SS são absolutamente essenciais uma vez que mesmo vestígios de uma toxina secretada pode ser suficiente para matar a presa. Mesmo fraca actividade dos T6SS poderia então ser claramente detectado pelo nosso ensaio, em comparação com o padrão de secreção procedimento de teste dependente da T6SS blot utilizando sobrenadante de cultura e ocidentalanálise. No entanto, uma adequada colónia formadora de unidade de contagem (UFC) é ainda necessário para a quantificação exacta desta actividade T6SS.
Divulgações
Não temos nada a revelar.
Agradecimentos
Este trabalho foi financiado pelo Wellcome Trust concessão WT091939MA. Alain Filloux é apoiado pela Royal Society.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente / Material | Companhia | Número de Catálogo | Comentários |
| P. aeruginosa PAKΔ RETS (D +) (T6SS ativos) | Tensão laboratório | Descrito na referência 16 | |
| P. aeruginosa PAKΔ Rets Δ H1-T6SS (D -) (T6SS inativos) | Este estudo | O cluster H1 T6SS-(englobando os genes para PA0070 PA0095) foi eliminado por troca alélica seguindo o procedimento descrito na referência 18. O fragmento mutante foi gerado com o seguinte conjunto de iniciadores: Os primers dos fragmentos acima: 5'-ATGGTCAACGACATGGAGCTGGAG-3 ', e 5'CGAGGCCGATCAGGCCTTCAGAACTGA-3 '. Os primers de Down fragmento: 5'-TCAGGCCTTCAGAACTGAAGCGGCGCA-3 ', 5'-GGTGGCGTTCAACAGTTCCATGTC-3 ' | |
| E. coli DH5a | Invitrogen | 18258-012 | F-φ80 lac ZΔM15 Δ (lac ZYA-arg F) U169 rec final A1 A1 HSD R17 (r k -, m k +) pho um sup E44 λ-thi -1 Gir A96 rel A1 |
| pBluescript II SK (+) | Agilent | 212205 | Este vector expressa o peptídeo α de β-galactosidase usado para α-complementação. |
| X-gal | Invitrogen | 15520-018 | Utilize a 40 ug / ml |
| Luria Bertani agar | Merck-químicos | 1.10283.0500 | |
| TSB (caldo de caseína de soja) | Oxoid | CM109 | |
| Vortex Genius agitador 3 | IKA | 3340000 | |
| Scanner | Epson | V700 | |
| Espectrofotômetro | WPA Biowave | CO8000 medidor de densidade celular | |
| Magenta-gal | Bioworld | 30350001-1 (715241) | |
| Red-gal | Research Organics | 1364c | |
Tabela 1. Strain, material, plasmídeo e do reagente utilizado. |
Referências
- Filloux, A., et al. The bacterial type VI secretion machine: yet another player for protein transport across membranes. Microbiology. 154 (6), 1570 (2008).
- Cascales, E., Cambillau, C. Structural biology of type VI secretion systems. Philos. Trans. R. Soc. Lond. B. Biol. Sci. 367 (1592), 1102 (2012).
- Leiman, P. G., et al. Morphogenesis of the T4 tail and tail fibers. Virol. J. 7, 355 (2010).
- Ballister, E. R., et al. In vitro self-assembly of tailorable nanotubes from a simple protein building block. Proc. Natl. Acad. Sci. U.S.A. 105 (10), 3733 (2008).
- Leiman, P. G., et al. Type VI secretion apparatus and phage tail-associated protein complexes share a common evolutionary origin. Proc. Natl. Acad. Sci. U.S.A. 106 (11), 4154 (2009).
- Pukatzki, S., et al. Type VI secretion system translocates a phage tail spike-like protein into target cells where it cross-links actin. Proc. Natl. Acad. Sci. U.S.A. 104 (39), 15508 (2007).
- Ma, A. T., et al. Translocation of a Vibrio cholerae type VI secretion effector requires bacterial endocytosis by host cells. Cell Host Microbe. 5 (3), 234 (2009).
- Hood, R. D., et al. A type VI secretion system of Pseudomonas aeruginosa targets a toxin to bacteria. Cell Host Microbe. 7 (1), 25 (2010).
- Russell, A. B., et al. Type VI secretion delivers bacteriolytic effectors to target cells. Nature. 475 (7356), 343 (2011).
- Li, M., et al. Structural basis for type VI secretion effector recognition by a cognate immunity protein. PLoS Pathog. 8 (4), e1002613 (2012).
- Zheng, J., et al. Genetic analysis of anti-amoebae and anti-bacterial activities of the type VI secretion system in Vibrio cholerae. PLoS One. 6 (8), (2011).
- Schwarz, S., et al. Burkholderia type VI secretion systems have distinct roles in eukaryotic and bacterial cell interactions. PLoS Pathog. 6 (8), e23876 (2010).
- Murdoch, S. L., et al. The opportunistic pathogen Serratia marcescens utilizes type VI secretion to target bacterial competitors. J. Bacteriol. 193 (21), 6057 (2011).
- Boyer, F., et al. Dissecting the bacterial type VI secretion system by a genome wide in silico analysis: what can be learned from available microbial genomic resources?. BMC Genomics. 10, 104 (2009).
- Maniatis, T., et al. . Molecular cloning: A Laboratory Manual. , (1982).
- Mougous, J. D., et al. A virulence locus of Pseudomonas aeruginosa encodes a protein secretion apparatus. Science. 312 (5779), 1526 (2006).
- Records, A. R., et al. The type VI secretion system: a multipurpose delivery system with a phage-like machinery. Mol. Plant Microbe Interact. 24 (7), 751 (2011).
- Hachani, A., et al. Type VI secretion system in Pseudomonas aeruginosa: secretion and multimerization of VgrG proteins. J. Biol. Chem. 286 (14), 12317 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados