Method Article
Peptides antimicrobiens produites par l’Incorporation de la pression sélective d’amino-acides Non canoniques
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Le protocole présente l' Escherichia coli-basée d’incorporation de la pression sélective des acides aminés non canoniques (ANAC) dans la nisine de peptide antimicrobien lactocoques. Ses propriétés peuvent être modifiées au cours de l’expression recombinants par substitution avec désiré ANAC dans les milieux de culture définis. Les changements qui en résultent dans la bioactivité sont mappés par tests d’inhibition de la croissance et la microscopie de fluorescence.
Résumé
La nature a une variété de possibilités pour créer de nouvelles fonctions de la protéine en modifiant la séquence des éléments constitutifs des acides aminés individuels. Cependant, toutes les variantes sont basées sur les 20 acides aminés canoniques (cAAs). Comme un moyen d’introduire davantage de propriétés physico-chimiques des polypeptides, l’incorporation des acides aminés non canoniques (ANAC) est plus utilisée dans l’ingénierie des protéines. En raison de leur longueur relativement courte, la modification des peptides synthétisés ribosomally et modifiées post-traductionnellement par ANAC est particulièrement intéressante. Nouvelles fonctionnalités et poignées chimiques peuvent être générées par des modifications spécifiques des résidus individuels. La méthode d’incorporation (SPI) de pression sélective utilise des souches auxotrophes hôte qui sont privés d’un acide aminé essentiel dans les milieux de culture définis chimiquement. Plusieurs analogues d’acides aminés structurellement et chimiquement similaires puis peuvent être activées par le correspondant aminoacyl-ARNt synthétase et fournissent des résidus spécifiques cAA(s) substitutions ncAA(s) → dans la séquence cible de peptides ou de protéines. Bien que, dans le cadre de la méthode SPI, ANAC est également incorporées dans le protéome hôte pendant la phase d’expression du gène recombinant, la majorité des ressources de la cellule est assignée à l’expression du gène cible. Cette mesure efficace axée sur les résidus incorporation d’ANAC souvent accompagnées de fortes quantités de cible modifiée. Le travail présenté décrit l’incorporation in vivo des six analogues de proline dans la peptide antimicrobien nisine, un lantibiotique naturellement produite par Lactococcus lactis. Propriétés antimicrobiennes de la nisine peuvent être changées et étoffées au cours de sa fermentation et d’expression en auxotrophes Escherichia coli souches dans les milieux de culture définis. Ainsi, les effets des résidus spécifiques remplacement du cAAs avec ANAC peuvent livrer des changements dans la spécificité et l’activité antimicrobienne. Tests d’activité antimicrobienne et microscopie de fluorescence sont utilisés pour tester les nouvelles variantes de nisine pour l’inhibition de la croissance d’une souche témoin de Gram positif Lactococcus lactis . Spectroscopie de masse sert à confirmer l’incorporation de la ncAA en variantes de nisine bioactifs.
Introduction
La découverte des antibiotiques dans le XXe siècle et le développement parallèle de nouveaux composés antimicrobiens contre les microorganismes pathogènes activées ciblée traitements des infections bactériennes. Cependant, en raison de l’émergence de pathogènes multirésistants comme résistant à la méthicilline Staphylococcusaureus (SARM), les entérocoques résistant à la vancomycine (ERV), MDR (pharmaco-résistante) Salmonella typhimurium phage type 10 (DT10 ), et Klebsiella pneumoniae, il est urgent générer de nouveaux agents antimicrobiens1. Peptides antimicrobiens (ampères) sont des composés polyvalents, souvent très spécifiques qui sont prometteurs pour le développement de nouveaux médicaments grâce à leurs propriétés physico-chimiques, flexibilité, taille, hydrophobie et mode d’action2. Ampères sont de petits peptides se compose habituellement d’acides aminés 7-100. Souvent, ils ont une structure cationique riche en résidus chargés positivement d’arginine et la lysine, qui interagissent avec la membrane cellulaire microbienne ciblée, qui est chargée de façon opposée3. Un sous-groupe particulier des amplis sont des peptides ribosomally synthétisés et après modification (RiPPs)4. Elles sont produites par nombreux organismes du Royaume des champignons et du domaine des bactéries. L’un des plus connus et largement utilisés RiPPs est la nisine, naturellement produite par la bactérie lactique acide Lactococcus lactis (L. lactis). Active contre un groupe de bactéries à Gram positif, la nisine a été utilisée comme un biopreservative dans l’industrie alimentaire depuis plus de 50 ans en raison de ses propriétés antimicrobiennes et l’absence de résistance évoluée dans les souches microbiennes ciblées5. Études ont montré que la nisine déstabilise et génère des pores dans les membranes cellulaires bactériennes, conduisant à une activité antimicrobienne contre les deux agents pathogènes Gram-positifs et Gram-négatives6. En se liant au lipide II, synthèse de la paroi cellulaire bactérienne est inhibé7. La nisine est codée par CSRN comme un peptide précurseur linéaire, qui est composé d’un chef de file et une région de peptide de base (Figure 1). Après la synthèse ribosomique, prenisin modifie tout d’abord le déshydratase NisB. Ici, les résidus sérine et thréonine dans la région de noyau prepeptide sont déshydratés à dehydroalanine (Dha) et dehydrobutyrine (Dhb)8. Par la suite, les résidus déshydratés sont couplés avec la cystéine à anneaux lanthionine forme (d'où le nom « lantibiotique » pour lanthionine anneau contenant antibiotiques) par une addition de Michael catalysées par les enzymes. Cette modification post-traductionnelle (PTM) est catalysée par l’adénylcyclase NisC. Chez L. lactis, le mis à jour le prenisin est ensuite transporté hors de la cellule par transporteur NisT et peptide leader est clivé par la protéinase NisP pour libérer la nisine mature et active forme9. La peptidase de dirigeant responsable NisP a une spécificité de substrat élevé, puisqu’il uniquement les processus modifiés nisine efficacement10.
En général, RiPPs actives résultent de l’action des enzymes PTM (par exemple NisBC), qui augmentent considérablement l’espace chimique des peptides courts, par exemple, par acétylation, glycosylation, méthylation ou phosphorylation. Ce niveau de complexité peut être étoffé par l’incorporation directe de l’ANAC. Alors que souvent c’est possible, la synthèse chimique des amplis est un défi pour une production à grande échelle en raison de leur complexité structurelle. Par exemple, la synthèse chimique totale de la lactosin lantibiotique S dans 71 étapes réactionnelles a été atteint avec un rendement final de 10 % et celui de la nisine avec un rendement brut de seulement 0,003 %11,12. Par conséquent, la production biologique offre une alternative viable, en raison de la génération de stéréocentres correcte et de la concentration élevée du produit.
Jusqu'à aujourd'hui, plus de 150 ANAC, par exemple, avoir des groupes fonctionnels contenant du fluor ou azotures, ont été incorporés dans les protéines recombinantes, et plusieurs exemples des amplis ncAA modifiés ont été déclarés13,14, 15,16. Avec l’introduction de l’ANAC, nouvelles propriétés physico-chimiques peuvent être générées par rapport à la mutagenèse classique. La diversité des peptides existants peut être augmentée, pouvant conduire à nouveaux antibiotiques.
Une méthode pour l’incorporation de l’ANAC recombinants peptides est incorporation de pression sélective (SPI) fondée sur l’utilisation de souches de bactéries auxotrophes17. Ces souches ne sont pas capables de synthétiser l’analogique cAA correspondant de la ncAA. La méthode utilise la spécificité de substrat détendue fréquemment observées, une caractéristique de nombreux naturel aminoacyl-ARNt synthétases (aaRSs)18. En dehors de leurs substrats naturels de la cAA, ces enzymes sont souvent capables de reconnaître et d’activer la ncAA désiré et de le charger sur leurs tRNA(s) apparentés. Cela conduit à ribosomique incorporation de la ncAA dans le produit du gène cible de façon spécifique de résidus (c.-à-d., cAA → ncAA substitution). C’est bien sûr possible seulement lorsque la ncAA désirée est structurellement et chimiquement similaire à l’acide aminé canonique et toléré par la physiologie cellulaire, de la machinerie de traduction et de la séquence cible de peptides ou de protéines. Dans un montage expérimental particulier, les cellules auxotrophes hôtes sont cultivées en milieu défini fourni avec une concentration limite de l’acide aminé natif soit substitué. La croissance cellulaire ou l’échange par un milieu sans cAA entraîne une déplétion intracellulaire de la cAA. Dans l’étape suivante, la ncAA est ajouté et l’expression du gène cible est induite. Inévitablement, ANAC est maintenant également incorporées dans plusieurs autres protéines dans la cellule hôte au cours de cette phase d’expression du gène cible. Néanmoins, la toxicité de l’installation SPI est maintenue à un niveau faible, puisque la souche d' Escherichia coli (e. coli) est transformée avec un plasmide porteur du gène cible sous contrôle d’un promoteur fort (généralement très compétitif T7 promoteur / Système de RNA polymérase)19. Immédiatement après l’induction (généralement lorsque la cAA est épuisé), l’hôte les cellules cessent de se développer et leurs mécanismes enzymatiques cytoplasmiques sont utilisés principalement pour l’expression du gène cible axée sur le plasmide. Mutagenèse dirigée peut être utilisé pour définir l’ou les lieux de l’installation de ncAA de résidus spécifiques du gène de cible20.
Comme un peptide de modèle pour l’incorporation de l’ANAC, la nisine de AMP pentacyclique A a été choisie. C’est 34 acides aminés et a seulement un résidu proline unique dans la séquence peptidique de base (Figure 1). Comme dans la subtilisine, ericin A et S et utilise aussi bien comme dans la nisine Z et la nisine Q, la proline conservée semble être essentielle pour l’activité9,21. La proline cAA joue un rôle particulièrement important dans la rotation amide peptidyl-prolyl et stabilisation de la structure secondaire. Sa chaîne latérale bague conformations (exo / endo fronce) sont responsables d’une stabilisation thermodynamique de la liaison amide. Modifications chimiques ciblées (par exemple hydroxylations, fluorinations, méthylations) de prolyl fronces influencent souvent critique stabilité, rigidité de l’échafaudage et de fonctions de nombreuses structures biologiques22pliant. Ainsi, il est plausible d’attendre que les substitutions analogiques de proline Pro→ vont doter l’anneau B, le deuxième cercle de nisine, avec roman et propriétés inhabituelles.
Ici, une proline-auxotrophes souche e. coli a été utilisée pour la production de la nisine recombinant. Cela nécessite l’expression de la prepeptide gène CSRN ainsi que la modification de gènes enzyme nisBC. Le produit de peptide codé génétiquement transporte un leader en position N-terminale His-tag pour purification par chromatographie d’affinité sur. Pour la détermination de l’activité, L. lactis exprimant et sécrétant des NisPT est utilisé pour activer les variantes de la nisine recombinante de lysats de cellules d’Escherichia coli ou des échantillons de peptide purifié (Figure 1). L’AMP mature est sorti après clivage du leader NisP. Dans cette méthode de diffusion en gélose, l’échantillon de AMP diffuse dans le milieu de croissance solide et peut inhiber la croissance des microorganismes Gram positif. Après incubation, cela peut être observée visuellement par les halos de l’inhibition de la croissance. En plus de L. lactis comme indicateur, modifié la nisine variantes ont montré une activité antimicrobienne contre Enterococcus faecalis, Bacillus cereus, Staphylococcus aureus et Lactobacillus johnsonii 21,,23.
Une méthode alternative et expérimentalement différente d’incorporer l’ANAC dans RiPPs est stop codon suppression (SCS)24. Pour ce faire, un ARNt orthogonale / paire d’aminoacyl-tRNA synthétase (RAA) est nécessaire pour la ncAA correspondante. Idéalement, tous ces trois composantes sont bioorthogonal, c'est-à-dire qu’ils n’interagissent pas avec le tRNA endogènes et les aaRSs. Un RAA ncAA spécifiques peut être généré par la modification du site actif de l’enzyme et de criblage des bibliothèques génétiques mutantes synthétases25. En outre, l’introduction d’une ncAA exige un codon qui est réassigné et qui n’encode pas le CAA. Le codon ambre est généralement utilisé24,26.
Récemment, SPI a été créé pour l’incorporation de l’ANAC contenant du α-chloroacétamide et clic-chimie-compatible NisA27. Par exemple, Nε- alloc-lysine a été incorporé dans le captistruin de peptide de lasso avec in situ (SCS) et méthodes d’incorporation de résidus spécifiques (SPI) et par la suite modifié in vitro par métathèse de ruthénium catalyse28 . En comparaison avec SPI, la méthode SCS est plus compliquée depuis un ARNt orthogonale / aaRS paire doit être exprimée conjointement. A ce jour, o-paires pour l’incorporation de la proline ont été développés29, mais au mieux de notre connaissance, aucun exemple d’incorporation analogiques de la proline n’a été signalée.
Il est à noter que pas tous ANAC peut être incorporé en utilisant la méthodologie SPI. Tout d’abord, l’absorption de l’ANAC dans le cytoplasme est régie par une multitude de protéines de transport qui sont incorporées dans la membrane cytoplasmique, qui est la membrane interne des bactéries à Gram négatif comme Escherichia coli. Normalement, les e. coli est capable de transporter une large gamme d’analogues d’acides aminés dans la cellule avec des chaînes latérales structurellement et chimiquement similaire à canonique des acides aminés. Deuxième, nombreux ANAC chimiquement réactives ou instable pourrait agir comme un inhibiteur de la croissance cellulaire, car ils sont toxiques pour le métabolisme et la physiologie de l’hôte cellule30. Ainsi, l’absorption et la toxicité de la ncAA pour l’hôte de production doivent être testés au préalable. Pour éviter l’inactivation de la machinerie PTM comme un effet secondaire, une installation d’expression strictement contrôlée des gènes responsables peut être utilisée pour intégrer les enzymes de modification (par exemple, nisBC) de l’acide aminé naturel et la ncAA dans le gène cible ( par exemple, nisA). Ceci peut être accompli à l’aide de deux promoteurs différents et l’induction de l’expression du gène cible, comme le montre spécialement conçu de protocoles SPI31. Comme indiqué ci-dessus, la méthode SPI s’appuie sur la spécificité de substrat détendue de l’aaRS, qui permet pour l’activation de la ncAA et apparentés tRNA de charge. Par la suite, l’ARNt est distribué dans le ribosome, suivie de la formation de la liaison amide et pliage du peptide cible (poly). Dans ce processus, correction d’épreuves et de mécanismes d’édition peuvent devenir pertinente32. Pour ces raisons, il est très important d’avoir une cible en ncAA structurellement et chimiquement semblable à la cAA. Autres points cruciaux sont une stabilité suffisante (tant dans les milieux de culture et exposés au métabolisme cellulaire) et la solubilité de la ncAA. En outre, il devrait être soit disponible commercialement ou facile à être synthétisé chimiquement.
Nous décrivons ici un protocole pour SPI, permettant l’incorporation de résidus spécifiques d’ANAC dans RiPPs recombinant. En particulier, analogues de différents proline sont incorporés dans la nisine peptide antimicrobien A comme organisme hôte à l’aide d’e. coli . Spectrométrie de masse est utilisée pour vérifier le remplacement de l’acide aminé et produits peptidiques sont analysées à l’aide de tests d’inhibition de la croissance et la microscopie de fluorescence à l’aide de souches microbiennes indicatrices de bioactivité.
L’exigence fondamentale d’expression nisine recombinant réussie avec une définition ANAC nécessite une proline adapté auxotrophe souche e. coli . Pour cela auxotrophie, proA doit être dysfonctionnelle, par exemple obtenus par knock-out génomique. Cellules entièrement privé de biosynthèse de Pro intracellulaire (c'est-à-dire la suppression des proABC) sans possibilité de réversion sont stables auxotrophes. Gène couramment knockout méthodes sont monogéniques opercule selon Datsenko & Wanner33ou transduction du phage. En outre, souches de proA knockout peuvent provenir de dépôts publics tels que Addgene, CGSC ou la collection de Keio. Puisque l’expression recombinante nisABC montrée ici repose sur l’utilisation de promoteurs T7, l’expression hôte possède transporter un gène inductible de l’ARN polymérase T7. Ceci peut être accompli par introduction dans le génome de l’hôte, par exemple à l’aide de la trousse commerciale du prophage λDE3. Alternativement, des souches comme BL21 (DE3) peuvent être faits auxotrophes comme décrit ci-dessus.
Protocole
1. le clonage des vecteurs d’expression et de la transformation d’une souche auxotrophe production
Dans les présentes, les gènes pour la biosynthèse de la nisine, nommément nisABC, ont été tirées de L. lactis et transférés dans des vecteurs d’expression de plasmide T7-basé. Des séquences d’ADN pleins de nisABC se trouvent dans GenBank entrée X6830734. Le gène codant pour le peptide précurseur (CSRN) a été placé sur un vecteur animal-3 a qui confère la résistance à l’ampicilline. Gènes de déshydratase (nisB) et cyclase (nisC) ont été placés sur vecteur pRSFDuet-1, comme signalé plus tôt35, qui confère la résistance à la kanamycine.
Remarque : Pour le CSRN, les codons des quatre derniers acides aminés (ASPR) de la séquence de tête ont été mutés pour encoder VSLR36 pour restituer les résidus de proline dans le peptide de base unique tout en assurant la bonne prépeptide traitement par NisP. À l’extrémité N-terminale, une balise hexa-histidine flanquée par des résidus d’éditeur de liens a été ajoutée à des fins de purification (voir Figure 1).

Figure 1 . Représentation schématique de la biosynthèse et les PTM de NisA chez e. coli ainsi que du clivage de la chef de file de la souche témoin de L. lactis dans un dosage de l’activité subséquente. Dans un premier temps, le prenisin linéaire inactif (composé d’un chef de file et une région peptide contenant un unique proline (rose) à la position 9) codée par CSRN est synthétisé ribosomally. Ensuite, prenisin est posttranslationally modifié par déshydratation des résidus sérine et thréonine dehydroalanine (Dha) et dehydrobutyrine (Dhb) catalysée par NisB. L’adénylcyclase NisC forme la thioéther ponts par addition de Michael de groupes sulfhydryles cystéine avec Dha ou Dhb. Le prenisin mis à jour l’inactif est purifiée d’e. coli et testé pour une activité antimicrobienne. Ici, il est transporté dans les cellules de la souche d’indicateur de Gram positif L. lactis . Le leader est clivé par la protéase NisP (comme indiqué par une flèche) pour libérer la nisine pleinement active. Il peut aussi être retiré in vitro par un traitement avec la trypsine (*). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
- Permet de transformer la proline auxotrophe choc thermique standard protocole37 ou électroporation38 e. coli de souche (voir ci-dessus) avec les plasmides pET-3 a nisA(VSLR) et pRSFDuet-1 nisBC.
- Pipetter 25-100 µL de la suspension de cellules transformées sur plaques de gélose contenant l’ampicilline, kanamycine et 1 % (p/v) de glucose. Utilisez un épandeur de plaque ou de perles de verre pour écarter la solution uniformément sur les plaques.
- Incuber les boîtes pendant une nuit à 37 ° C.
- Le lendemain après-midi, utilisez une seule colonie pour inoculer 10 mL LB milieu contenant l’ampicilline, kanamycine et 1 % (p/v) de glucose dans un ballon jaugé de 50 mL.
- Secouez la culture pendant la nuit (12-16 h) à 37 ° C et 200 tr/min.
- Prendre 250 µL stérile 80 % de glycérol et de la culture de 550 µL, mélangez bien dans un tube de 2 mL et stocker sous forme de stock de cellules congelées à 80 ° C.
2. nouvelles préparations d’un milieu minimal (NMM)
Remarque : Ce protocole utilise NMM20 comme un milieu liquide chimiquement défini de la croissance bactérienne. En outre, il est conseillé de suivre l’ordre de préparation strictement. Dans le cas contraire, les précipitations peuvent se produire. Pour les formes d’acides aminés diffèrent de ceux indiqués dans la table des matières (p. ex., les chlorhydrates), vérifier la solubilité. NMM19 contient 19 acides aminés à l’exception de la cAA à remplacer (ici, proline) par l’analogue de la ncAA. Voir le tableau 1 pour la concentration de l’ingrédient final. Selon la souche bactérienne utilisée pour la production, la biotine et la thiamine peuvent être facultatifs.
- Préparation du mélange d’acides aminés
- Dissoudre 0,5 g de Phe, Trp et Tyr dans 100 mL FD2O additionnée de quelques gouttes de HCl concentré jusqu'à dissolution.
- Peser avec 0,5 g de chacune des 16 acides aminés restants. Mélanger avec 22 mL de 1 M KH2PO4 et 48 mL de 1 M K2HPO4. Ajouter ddH2O à ~ 800 mL. Remuer jusqu'à ce que la solution devienne claire.
- Ajouter dissous Phe, Trp et Tyr et régler le volume de la solution à 1 L avec FD2O.
- Stériliser le mélange d’acides aminés par une filtration sous vide avec une unité de filtration haut de bouteille.
- Solutions courantes pour NMM19
- Tout d’abord, préparer 1 M les solutions des éléments suivants : (NH4)2donc4, KH2PO4, K2HPO4, MgSO4 et un 5 M en stock solution de NaCl. Stériliser à l’autoclave.
- Préparer des stocks de 50 mL de D-glucose (1 M), CaCl2 (1 g/L), FeCl2 (1 g/L), thiamine (10 g/L), biotine (10 g/L) et oligo-éléments (CuSO4, ZnCl2, MnCl2, (NH4)2MoO4; 1 mg/L ). Stériliser par filtration avec un filtre de seringue.
- Préparation de NMM19
- Mélanger les solutions-mères pour obtenir une concentration finale de 7,5 mM (NH4)2SO4, 1,7 mM NaCl, 22 mM KH2PO4, 50 mM K2HPO4, 1 MgSO4 ou 20 mM D-glucose, acides aminés 50 mg/L mélanger, 1 µg/L CaCl2, 1 µg/L FeCl2, 10 thiamine µg/L, biotine de 10 mg/L et 0,01 µg/mL d’oligo-éléments.
3. expression de la nisine Recombinant avec Incorporation de Proline analogues par SPI
Dans cette section, l’expression recombinante de le prépeptide (ici : nisA) et gènes PTM (ici : nisBC) est effectuée. Tout d’abord, les cellules sont cultivées en présence de tous les cAAs, depuis que la LB milieu complexe est utilisé. On ajoute du glucose pour réprimer l’expression du gène cible au niveau de fond qui puisse autrement mener à la production du peptide de type sauvage (ici : la nisine) en raison de la perméabilité des promoteurs. Seulement après la cible cAA (ici : proline) est épuisée, la ncAA est ajouté et l’expression des gènes cible est induite dans un milieu chimiquement défini. L’incubation des cultures liquides doit être effectuée dans des flacons adaptés avec aération (p. ex., 500 mL dans une fiole d’Erlenmeyer de 2 L à 200 tr/min).
- À l’aide d’un embout de la pipette stérile, commencer une culture fraîche d’une nuit ou plus de stock de cellules congelées ou fraîche colonie (voir étape 1). Utiliser le milieu de 25 mL LB contenant l’ampicilline, kanamycine et 1 % (p/v) de glucose et incuber à 37 ° C pendant la nuit (12-16 h) et 200 t/min.
- Ensemencer 1 L de milieu frais stérile avec la culture au jour le jour (1 % v/v) de 10 mL et incuber à 37 ° C et 200 tr/min jusqu'à OD600 = 0,5.
- Centrifuger à 4 ° C pendant 15 min à 4 500 x g.
- Décanter le liquide surnageant et Resuspendre le culot avec antibiotiques contenant de 20 mL NMM19 (préparé à l’étape 2.3) et 1 % (p/v) de glucose. Centrifuger à 4 ° C pendant 10 min à 4 500 x g.
- Resuspendre le culot dans 500 mL du même milieu et incuber à 30 ° C et 200 tr/min pendant 1 h.
NOTE : À cette étape, l’appauvrissement de la cAA (ici, proline) se déroule. - Diviser la culture en parties égales (une pour chaque ncAA). Inciter chaque culture avec 1 mM isopropyl β-D-1-thiogalactopyranoside (IPTG) et fournir des analogues de proline 1 mM (4S/R- fluoroproline, 4S/R- hydroxyproline ou 4S/R- methanoproline).
Remarque : En tant que contrôle, une culture peut être fournie avec proline 1 mM, ayant pour résultat la production de peptide de type sauvage. - Incuber pendant la nuit (12-16 h) à 28 ° C et 200 tr/min.
- Centrifuger le cell cultures en tubes de 50 mL à 4 ° C pendant 20 min à 5 000 x g. décanter le liquide surnageant et stockent les granulés à-80 ° C jusqu'à ce que la purification.
4. isolement et Purification des analogues de la nisine His-tag
Peptides sont purifiées dans des conditions guanidine chlorhydrate (GuHCl)39, une forte dénaturant dénaturantes.
ATTENTION : GuHCl est nocif si avalé ou inhalé et provoque la peau et l’irritation oculaire grave. Porter des gants et des lunettes de protection.
- Préparer 250 mL de liant tampon (5 M GuHCl, 300 mM NaCl, Tris de 25 mM, pH 7,4), tampon de lavage (300 mM NaCl, Tris, imidazole de 25 mM, 25 mM pH 7,4) et tampon d’élution (300 mM NaCl, Tris, imidazole de 250 mM, 25 mM pH 7,4). Pour ces derniers, transférer les solides dans un flacon de 250 mL et remplir jusqu'à 200 mL avec FD2O. Mix bien et ajuster le pH à 7,4 avec 1 NaOH M ou HCl. Puis, remplissez jusqu'à 250 mL avec FD2O. filtre toutes les solutions tampons à l’aide d’une unité de filtration haut de bouteille.
- Lyse cellulaire
Ici, un sonicateur (avec 200 W maximum haute fréquence (HF) sortie) est utilisé ; Notez que les réglages de la puissance nécessaires pour la rupture de la cellule peuvent être différent pour d’autres instruments. Toutes les mesures sont effectuées sur la glace. Alternativement, la lyse des cellules chimiques, un homogénéisateur liquide ou une presse Français peut être utilisée.- Ajouter 12 mL de tampon de liaison dans chaque tube à centrifuger (de l’étape 3,8) et remettre en suspension au vortex.
- Plonger l’extrémité de la sonde sonicateur dans la suspension cellulaire. Définissez le sonicateur à amplitude de 40 % avec une impulsion de 1 s sur / 5 s éteint pendant 15 min.
Remarque : Nettoyer la pointe sonicateur entre les échantillons afin d’éviter le report. Essuyer la sonde sonicateur, éthanol à 70 %. - Centrifuger la suspension de cellules lysées à 4 ° C pendant 40 min à 15 000 x g pour granuler les débris cellulaires. Transférer le surnageant dans un nouveau tube de réaction.
- Chromatographie d’affinité
Pour les ions métalliques immobilisés affinité chromatographie (IMAC)40, une pompe péristaltique ou un système FPLC utilisable avec une cartouche de 1 mL (ici remplie de résine Ni-NTA). Pour la préparation des tampons, voir le point 4.1.
NOTE : Purification de l’IMAC est faisable puisque le peptide recombinant produit porte un N-terminale His-tag leader, qui est supprimé à l’étape 6 de peptidase chef NisP, libérant la nisine mature. Accomplissent la purification à température ambiante ou à 4 ° C. Utiliser un débit de 1 mL / minute, le cas échéant pour la cartouche de l’IMAC.- Tout d’abord, laver la cartouche avec 5 volumes de colonne (cv) de ddH2O pour retirer le tampon de stockage.
- Équilibrer avec 10 cv de tampon de liaison.
- Processus cellulaire lysat (étape 4.2) en utilisant une seringue filtrer pour enlever les particules, puis l’appliquer sur la cartouche.
- Laver avec 15 cv de tampon de lavage afin d’éliminer les matières non spécifique et indépendant.
- Éluer avec 10 cv du tampon d’élution et fractions de recueillir 1 mL dans des tubes de 1,5 mL. Stocker les fractions à 4 ° C à court terme (jusqu'à 3 jours) ou à-20 ° C pour des périodes plus longues.
- Pour le stockage, laver la cartouche avec 10 cv de ddH2O suivie de 5 cv de 20 % d’éthanol.
5. LC-ESI-TOF analyse par spectrométrie de masse de la nisine analogues
NOTE : Table de matières voir par exemple l’instrumentation pour la chromatographie liquide couplée à la spectrométrie masse temps de vol (LC-ESI-TOF-MS) de l’ionisation par électronébulisation.
- Effectuer séparation HPLC de 15-20 µL de solution de peptide (préparé à l’étape 4.3) sur une colonne de C5 avec une phase mobile d’eau (A) et l’acétonitrile (B) les deux additionnés de 0,1 % d’acide formique et une pente de 5 à 80 % B pendant 20 min. Pour la spectrométrie de masse (MS), utilisez l’élution après 5 min.
Remarque : Selon le contenu de peptide et l’affinité à la colonne HPLC, séparation et volume des échantillons devrez optimisation. - Utiliser un logiciel approprié pour deconvolute les spectres de masse mesurées et calculer que les frais de peptides différents États41. Comparer la masse d’espèces observées de peptide à la masse calculée de type sauvage altérée par la substitution de ncAA → cAA. Tenir compte du fait que le prépeptide linéaire est après modification par huit déshydratations (-8 H2O) et cinq cyclisations (voir Figure 1).
Remarque : L’utilisation de tampons contenant du sodium, analyse de MS en mode positif peut afficher adduits de sodium. Ceux-ci apparaissent comme des pics supplémentaires avec masse déconvoluée plus élevée (pour chaque sodium adduit, la masse observée déconvoluée est Da 22.99 plus élevé). Pour supprimer ces adduits, HPLC purification42 ou43 de la dialyse vaste peut être réalisées.
6. Test d’activité antimicrobiens
- Préparation des plaques GM17-agar dans des conditions stériles
- Préparer une culture au jour le jour de la glycémie de souche NZ9000 de L. lactis portant le plasmide pNG nisPT44 à 30 ° C dans la bouillon de M1745 avec 1 % (p/v) indicateur (= GM17) et de 5 µg/mL au chloramphénicol.
- Mesurer OD600, ensemencer un milieu frais à OD600 = 0,1 et incuber jusqu'à OD600 = 0,4 à 0,6. Puis placer le ballon sur glace.
Remarque : Chaque mesure de600 OD consommera volume de culture. N’oubliez pas que pour chaque plaque de gélose test, culture bactérienne 1 mL est nécessaires. Si nécessaire, la milieu de culture liquide quantité suffisante en conséquence. - Pour 1,5 % d’agar, peser 4,5 g de gélose dans une bouteille en verre support. Ajouter 300 mL FD2O, mix et autoclave.
- Préparer le bouillon de x M17 2 (Double concentré) dans 300 mL FD2O et autoclave.
- Mélangez 25 mL bouillon de x M17 2 contenant 10 chloramphénicol µg/mL et 2 % de glucose avec 1 mL L. lactis préculture (4 % v/v).
- Ajouter 25 mL d’agar 1,5 % fondu (fraîchement stérilisés à l’autoclave ou chauffée dans un four à micro-ondes).
NOTE : Auparavant, laissez le refroidir de bouteille au toucher (environ 50 ° C). Ceci est nécessaire car L. lactis est un organisme de mésophiles sensible aux températures élevées. - Versez la solution dans une grande boîte de Pétri. Plaques sèches pendant 10-15 min.
- Stériliser les extrémités d’une pipette Pasteur en verre à la flamme. Attendez qu’il refroidisse, puis utilisez l’extrémité la plus large pour créer des trous dans la GM17-gélose solidifiée.
- Préparation des échantillons
- Prendre 1 mL de la culture d’expression e. coli (créé à l’étape 3,7) dans un tube marqué de 1,5 mL et centrifuger pendant 3 min à 7 000 x g. aspirer le milieu restant et Resuspendre le culot dans 500 µL Na-P (tampon de phosphate de sodium 50 mM pH 7,4 de dihydrogénophosphate de sodium 0,5 M et 0,5 M phosphate disodique).
- Laisser agir les échantillons sur glace (Comparez étape 4.2.2). Plonger l’extrémité de la sonde sonicateur dans la suspension cellulaire. Définissez le sonicateur à amplitude de 30 % avec une impulsion de 1 s s 5 sur off pendant 3 min.
- Centrifuger la cellule lysate pendant 10 min à 13 000 x g pour granuler les débris cellulaires. Transférer le surnageant dans un nouveau tube de réaction sur la glace.
- Diluer et normaliser les surnageants extrait de cellule à 1 mL OD600 = 0,6, par rapport à la densité de cellules récoltées, avec Na-p.
- Test d’activité
- Ajouter 40 µL de chaque échantillon normalisée dans un trou de la gélose de l’indicateur (Figure 3). Utiliser le chloramphénicol à 400 µg/mL comme contrôle antibactérien composé. Utiliser le tampon d’élution comme témoin négatif. Attendez que tous les échantillons sont diffusés dans la gélose. Incuber la plaque du jour au lendemain à 30 ° C.
- Prendre des photos de la gélose à l’aide d’un appareil photo numérique ou un scanner à plat. Tailles de croissance inhibition halo peuvent être mesurée à la main ou à l’aide de ImageJ46.
7. la microscopie en fluorescence
Afin d’observer l’effet d’ampères sur les cellules bactériennes, microscopie photonique et à fluorescence peut être utilisée. Notez que le mode de la nisine d’action s’appuie sur la déstabilisation et la formation de pores dans les membranes bactériennes6. Ici, Nil rouge est utilisé pour colorer la membrane bactérienne, qui devient la lyse des cellules dispersées et agrégées sur.
NOTE : Table de matières voir par exemple l’instrumentation. Quantités de solution AMP ajoutée peuvent être ajustées selon la bioactivité et la concentration de peptide.
- Préparation de cellules
- Préparer 10 mM solution rouge du Nil dans le diméthylsulfoxyde (DMSO).
- Cultiver la souche de L. lactis indicatrice à OD600 = 1.0 comme dans l’étape 6.1.1-6.1.2.
- Centrifuger 1 culture mL pendant 3 min à 4 ° C et 5 000 x g.
- Jeter le surnageant, resuspendre dans 1 mL de solution saline tamponnée au phosphate (PBS)47.
- Centrifugeuse et remettre à nouveau.
- Ajouter 1 µL de solution stock de Nil rouge, mélanger doucement.
- Acquisition d’images microscopiques
- Ajouter 30 µL de préparation de cellules sur une lame de couverture tout en excitant à 520 nm.
- La valeur d’acquisition temps 0,2 s, série cinétique 0,1 Hz, images de longueur 200 series.
- Ajouter 0,3 - 1,5 µL de lysat cellulaire ou d’échantillon de l’IMAC (d’après l’étape 6.2.4 ou 4.3.5, respectivement). Pour les échantillons de l’IMAC, le tampon d’élution peut servir comme témoin négatif.
- Surveiller et enregistrer les émission de fluorescence à λ ≥ 560 nm.
- Analyse des données
- Séquences d’images de microscopie sont stockés sous forme de fichiers vidéo (.avi). Images simples sont analysées avec ImageJ46.
Résultats
Ce protocole est conçu pour permettre la production de variantes de nisine modifiés ncAA avec incorporation de résidus spécifiques des analogues de la proline par la méthode SPI. Auparavant, la faisabilité et les rendements de 24 mg/L ont été signalés pour la production recombinante d’entièrement mis à jour le sauvage nisine39. À l’aide de la méthode SPI, rendements de peptides/protéines cibles sont souvent bons et peuvent atteindre quantités près de type sauvage production48. Comme les premières expériences, production RiPP par recombinaison génétique sauvage doit être testée chez l’hôte auxotrophe choisie. Ici, la proline-auxotrophes Escherichia coli MG1655 ΔproBA:: frt ΔproC:: frt (DE3) a servi de souche hôte. Pour l’incorporation de l’ANAC, culture et induction température et composition calendrier comme moyenne peuvent être optimisés vers le rendement maximal de peptide.
Dosage de l’activité antimicrobienne
Purification et production de nisine recombinant de type sauvage ont été réalisées selon le protocole ci-dessus. Dans ce cas, la proline a été utilisé au lieu de ses dérivés en ncAA à l’étape 3.6. Un dosage de l’activité antimicrobienne a été utilisé pour vérifier la production RiPP et de comparer l’activité antimicrobienne avant et après la purification. Pour le dosage de l’activité, élution fractions et accréditives IMAC ont été utilisés directement et testés contre la souche indicatrice de Gram positif L. lactis (Figure 2). Comme cette souche exprime NisP, les variantes de la nisine contenues dans les lysats de cellules d’Escherichia coli ou des échantillons de peptide purifié, deviennent respectivement, activées par clivage protéolytique du peptide leader. Evidemment, le cheminement a montré une activité inhibiteurs de croissance. Cela peut s’expliquer par matériau bioactif ne lie pas à la colonne de l’IMAC. Les fractions d’élution testé tous ont montré une activité accrue par rapport à l’échantillon non-purifié, ce qui indique une concentration du peptide His marqués par IMAC. Notez que la mémoire tampon d’élution (comme contrôle négatif) n’a pas influencé la croissance de L. lactis dans cet essai.
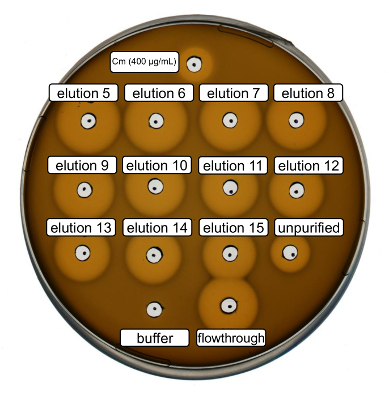
Figure 2 . Activité antimicrobienne d’essai après que purification IMAC d’inoculation produit sauvage nisine. D’élution fractions 5 à 15 et le flux par le biais de la purification de l’IMAC ont été testés en comparaison avec le lysat cellulaire non-purifié (dilué à la normalisation de600 OD) contre la souche témoin de L. lactis . La taille des halos de l’inhibition de la croissance indique une activité antimicrobienne. Chloramphénicol à une concentration de 400 µg/mL a été utilisé comme témoin positif et de la mémoire tampon d’élution IMAC comme contrôle négatif. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Pour prouver l’activité antimicrobienne d’inoculation produit nisine variantes contenant six analogues de différents proline, un dosage de l’activité a été réalisé afin de tester l’inhibition de la souche témoin de L. lactis . La figure 3 montre l’inhibition de la croissance pour cinq des six échantillons produites à l’aide d’analogues de la proline. Les meilleurs résultats (en juger des tailles de halo) ont été observées pour les expériences d’incorporation de l’analogique (4R) - fluoroproline, (4R) - hydroxyproline et (4S)-methanoproline. En comparant la taille de halo de l’inhibition de croissance à la nisine sauvage produit et testé en parallèle, toutes les trois variantes de la nisine a montré force semblable de l’inhibition. Toutefois, la taille de halo seule ne peut pas être utilisé pour évaluer une activité spécifique, étant donné que la concentration de l’AMP n’a pas été déterminée. Par conséquent, les essais servent uniquement à tester qualitativement si l’activité antimicrobienne des variantes nisine qui en résulte est conservée ou perdue. Pour déterminer l’activité spécifique, la concentration des nisine variantes doit être quantifié (voir Discussion).
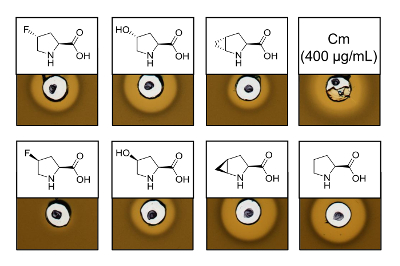
Figure 3 . Analyse de l’activité antimicrobienne des lysats de cellules contenant des variantes de la nisine produite via SPI avec les analogues de la proline. Comparaison des variantes de la nisine avec échantillons recombinants de type sauvage. Tous les échantillons étaient OD600-normalisée après la lyse cellulaire par rapport à la densité de culture de cellules récoltées. Halos indiquent bioactivité sous forme d’inhibition de croissance de la souche témoin. Première rangée, de gauche à droite : (4R)-fluoroproline, (4R) - hydroxyproline, (4R) - methanoproline et le chloramphénicol (400 µg/mL ; antimicrobiens contrôle positif). Deuxième rangée : (4S)-fluoroproline, (4S) - hydroxyproline, (4S) - methanoproline et la proline (contrôle de type sauvage). Remarque la nomenclature chimique ; par exemple, (4R) - fluoroproline est également dénommé trans-4 - fluoroproline. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
LC-ESI-TOF spectrométrie de masse
À la suite de purification de l’IMAC, l’incorporation de l’ANAC nisine a été analysée par spectrométrie de masse LC-ESI-TOF. La figure 4 montre les spectres de masse déconvoluées d’une variante de la nisine contenant (4R)-fluoroproline. Cette variante a été IMAC purifiée comme décrit ci-dessus et par la suite analysé par spectrométrie de masse LC-ESI-TOF, ainsi il a toujours porté le leader. Le pic principal dans la Figure 4A correspond à la nisine mis à jour le contenant (4R)-fluoroproline d’une masse déconvoluée de 6883.18 Da (masse calculée 6882.05 Da, calcule masse du peptide correspondant sauvage avec proline à la position 9 est Da 6864.06). Les deux sommets avec plus faible abondance et de la masse plus élevée correspondent à des adduits de sodium comme indiqué. Figure 4 B regroupe des espèces chargées différemment des principaux composés que l'on trouve par l’algorithme de déconvolution. Par exemple, le pic à m/z 1148.11 correspond à l’espèce chargée sextuplé ([M + 6H]6 +).

Figure 4 . LC-ESI-TOF spectrométrie de masse des variantes de la nisine recombinant purifié IMAC contenant (4R)-fluoroproline. (A) déconvolutée chromatogramme de la spectrométrie de masse (zoomé arrière dans le médaillon) pour la variante de la nisine (portant encore le leader) avec (4R)-fluoroproline (attendue des masses (Da) : [M + H]+ = 6882.05, [M + Na]+ = 6904.03, [M + 2Na]2 + = 6926.02). (B) composé des spectres pour les espèces [M + H]+. Attendus des masses (Da) : [5 M + H]+ 5 = 1377.41, [6 M + H]6 + = 1148.01, [7 M + H]+ 7 = 984.15, [8 M + H]+ 8 = 861.26, [9 M + H]+ 9 = 765.67, [10 M + H]+ 10 = 689.21, [11 M + H]+ 11 = 626.64, [12 M + H] 12 + = 574.50. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Microscopie en fluorescence
Activité antimicrobienne de nisine recombinante et ses variantes contenant du ncAA peut également montrer par l’observation directe de la L. lactis indicateur souche par microscopie à fluorescence. Nil rouge, un colorant fluorescent fortement hydrophobe, a été utilisé pour colorer la membrane de la cellule bactérienne. La figure 5 illustre le changement qualitatif de l’état d’agrégation de la culture cellulaire et la morphologie de la cellule unique. Les cellules ont été colorées au rouge Nil et déposés sur une lame de microscope couverture. La rangée supérieure montre les cellules directement au début quand le buffer, nisine recombinante de type sauvage, ou contenant de la nisine (4R)-fluoroproline, ou 4R-hydroxyproline ont été ajoutés. Le panneau inférieur montre les images correspondantes au bout de 20 minutes d’incubation.
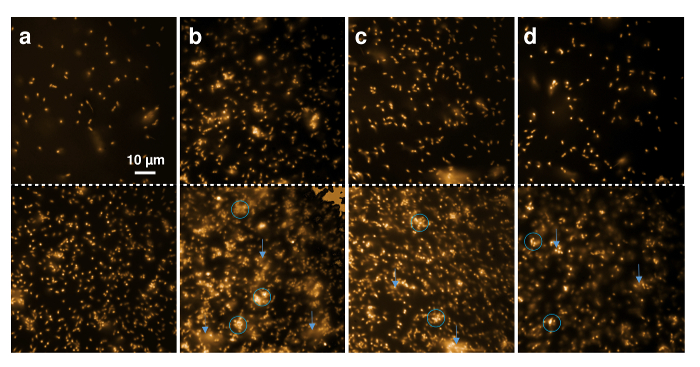
Figure 5 . Microscopie de fluorescence de nisine recombinant effets sur les cellules grampositives. Des images microscopiques de souche d’indicatrice 30 µL L. lactis (OD600 = 1) marqué avec Nil rouge ont été prises avant (du haut) et après (panneau inférieur) 20 min d’incubation avec 1 µL de tampon (A), 0,3 µL recombinant sauvage nisine (B) et Variantes de la nisine µL 0,6 contenant (4R)-fluoroproline (C) et (4R)-hydroxyproline (D). Cercles bleus marquent les régions avec point de flèches de cellules agrégées ou déformées, bleu aux régions où la diffusion des fragments membranaires fluorescents peut être observée. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Figure 5 A montre que l’aspect général des cellules n’a pas changé en 20 minutes d’observation. Que le nombre de cellules qui déposés durant la rose du temps et, par conséquent, une plus grande quantité de cellules sont visibles dans le 80 µm x 80 µm de la région observée. Figure 5 B montre que les cellules Gram positifs apparaissent agrégées et floue (marqués par des cercles bleus) après 20 minutes d’exposition à la nisine sauvage, même en faibles quantités (ici, élution de IMAC 0,3 µL) ont été ajoutées. En outre, matériau léger diffus des cellules dans le tampon, ce qui indique que des fragments de membrane marqués avec Nil rouges ont été mobilisés au cours de la période (indiquée par les flèches bleues). Ces résultats indiquent la lyse cellulaire, comme il a été montré à survenir après traitement avec la nisine6,49. Des effets similaires ont été observés après l’incubation avec la nisine variant contenant (4R) - fluoroproline (Figure 5C) et la nisine contenant(4R)- hydroxyproline (Figure 5D) les deux grandes montrant nombres de cellules déformées et agrégées après 20 minutes, en contraste avec l’échantillon de contrôle (Figure 5A).
| Composant | Concentration de |
| (NH4) 2 OUI4 | 7,5 mM |
| NaCl | 8. 5 mM |
| KH2PO4 | 22 mM |
| K2HPO4 | 50 mM |
| MgSO4 | 1 mM |
| D-Glucose | 20 mM |
| Tous les acides aminés canoniques (à l’exception de celui à remplacer) | 50 mg/L |
| Ca2 + | 1 µg/mL |
| Fe2 + | 1 µg/mL |
| Oligo-éléments (Cu2 +, Zn2 +, Mn2 +, MoOH2 +) | 0,01 µg/mL |
| Thiamine | 10 µg/mL |
| Biotine | 10 µg/mL |
Le tableau 1. Composition du milieu de croissance bactérienne chimiquement défini NMM19 après préparation selon l’étape 2.
Discussion
En utilisant SPI pour l’insertion des analogues de la proline, les structures ciblées nisine peuvent être substantiellement modifiés, créant des variantes de nouveau peptide avec combinaisons de séquence unique et propriétés chimiques. De cette façon, la limite de base de la technologie génétique classique peut être contournée, qui est contraint des chimies de chaîne latérale des 20 cAAs. In vivo la diversification chimique de la nisine comme illustré ci-dessus illustre une approche générale pour compléter la SPTM naturelle et pour rehausser de façon spectaculaire l’espace chimique des RiPPs. Nous croyons qu’élargir le répertoire des acides aminés naturels très prometteur surtout pour amplis. Dans les protéines, un énorme éventail de fonctionnalités peut être réalisé grâce à un arrangement défini des 20 cAAs dans les structures tridimensionnelles. Avec seulement 7-100 aa en longueur3, moyens d’atteindre ces caractéristiques structurales que par le biais de cAAs serait limités pour les amplis. Il n’est donc pas surprenant que naturels ampères sous forme de RiPPs sont généralement largement sera mis à jour le4. De la même façon, ANAC comme blocs de construction alternatives est très prometteurs pour améliorer leurs paramètres pharmacodynamiques et pharmacocinétiques (voir Baumann et coll. 201735 et les références qui y sont).
La méthodologie SPI utilisée dans ce travail bénéficie d’un montage expérimental relativement facile, d’exécution simple et reproductibilité élevée. En raison de la substitution globale, multisite incorporation de ncAA de peptides de la cible est également réalisable. En revanche, la méthode peut ne pas convenir pour le remplacement des acides aminés qui se produisent fréquemment dans le produit du gène cible. En principe, postes non désirés peuvent être modifiés par mutagenèse dirigée, mais ces modifications supplémentaires pourraient également affecter plusieurs propriétés AMP, y compris la structure et la bioactivité. Une fois une souche auxotrophe pour la production disponible, plusieurs ANAC peut être testé sans nécessiter de modifications importantes sur le plan génétique. En outre, la méthode n’est pas limitée à auxotrophes e. coli souches, mais peuvent également être effectuées à l’aide de l’hôte de production naturelle. Par exemple, Zhou et coll. ont montré que SPI pour la production de nouveaux RiPPs travaille également le naturellement Trp-auxotrophe L. lactis: l’utilisation définie des médias, trois analogues de tryptophane ont été incorporées aux quatre positions dans la nisine50.
Étant donné que la méthode SPI conduit à un remplacement global de l’Elu cAA de la ncAA, il est généralement applicable à un large éventail de cibles peptides et protéines. Une gamme d’auxotrophes e. coli souches est disponible (voir étape 1), permettant plusieurs cAAs chaque à tester pour le remplacement de l’ANAC. A rencontré analogues incorporés à l’aide de metA-souches déficients (par exemple, B834(DE3)) sont plus couramment employées. Exemples d’isostructuraux Met analogues sont azidohomoalanine (Aha) et homopropargylglycine (Hpg), ANAC commercialement disponible qui peut être intégré de manière efficace. Les deux introduisent bioorthogonal poignées qui permettent la fixation des molécules transportant un alcyne compatible ou la fonctionnalité de l’azoture, respectivement. Par exemple, les colorants fluorescents ou moitiés de polyéthylène glycol (PEG) peuvent être attachées en cuivre (I)-catalysée de l’azoture alcyne cycloaddition (CuAAC)51.
Bien que les deux méthodes d’incorporation de ncAA recombinante (SPI et SCS) généralement obtenir des quantités suffisantes de cible, les rendements sont souvent réduits par rapport au type sauvage production des protéines et des peptides correspondants. Comme souvent, les puretés sont corrélés à l’efficacité de production, étapes de purification supplémentaire peuvent être nécessaires, surtout pour les espèces peu abondantes. Dans ce cas particulier de la production de la nisine recombinant, la séquence His-tag simplifie grandement la purification RiPP par enrichissement sélectif des lysats cellulaires. La purification montrée dans ce protocole améliore la pureté et la concentration de la nisine, mais ne donne pas souvent de puretés AMP suffisantes pour déterminer le rendement et l’activité spécifique. En plus de l’IMAC, méthodes de purification des AMP couramment utilisées incluent HPLC, chromatographie échangeuse d’ions (IEC) et les précipitations (par exemple. à l’aide d’acétone ou l’acide trichloracétique (TCA)) ou une combinaison - résultant en un système de purification multi-étapes52 . Il est à noter que leur communément polycationiques nature peut faciliter la purification de la CEI. Lyophilisation est fréquemment utilisée pour stocker des amplis purifiées.
Idéalement, ANAC pour incorporation dans ampères devrait être disponible dans le commerce à des prix abordables ou facilement produites par les protocoles de synthèse chimique simple et reproductible. Une condition tout aussi importante pour la méthode SPI est que la ncAA est reconnu, activé et pratiqués sur l’ARNt apparenté par les RAA endogènes ou conjointement exprimées. Cela peut être testé in vitro par acide aminé activation ou ARNt aminoacylation dosage53. Comme une alternative simple, expressions de test SPI de protéines de modèle comme la protéine fluorescente verte (GFP) menée tous les deux en présence et en absence de supplémentation en ncAA peuvent être réalisées. En outre, solubilité dans la perméabilité moyenne et cellules de croissance ainsi que stabilité chimique constituent des facteurs importants.
Dans cet exemple, une activité antimicrobienne a été projetée à l’aide d’une souche de bactéries Gram-positives indicatrice. Comme il exprime la peptidase chef NisP, l’étape finale de la maturation de la nisine est catalysée. Suppression de la séquence de tête (son marqués à des fins de purification) peut également être réalisée in vitro par traitement avec purifiée NisP50 ou la trypsine54. Au-delà de la portée de ce travail, des organismes pathogènes et des souches multirésistantes peuvent ensuite être testés pour l’inhibition bactériostatique ou bactéricide par SAP en utilisant une méthodologie similaire. Espèces cibles cliniquement pertinents comprennent la SARM, MDR, Mycobacterium tuberculosis, ERV, Acinetobacter baumannii, Streptococcus pneumoniae, Pseudomonas aeruginosa et Klebsiella pneumoniae. Outre la diffusion de plaques de gélose test présenté ici, croissance inhibition peut également être effectuée à l’aide des milieux liquides appropriés inoculés avec l’espèce bactérienne et additionné de AMP. À l’aide de méthodes de dilution en bouillon, la concentration minimale inhibitrice (CMI) peut être déterminée à l’aide de peptides pure55. Le dosage de l’activité présenté ici permet également d’estimer la bioactivité et la puissance des solutions contenant de l’AMP par rapport aux composés de référence, la nisine par exemple disponible dans le commerce.
Production par recombinaison génétique de RiPPs est souvent faisable39, qui comprend généralement la co-expression de gènes PTM. Dès que la souche de production est transférée dans un milieu minimal chimiquement défini ou synthétique contenant une ncAA adapté, le remplacement de résidus spécifiques de la cAA correspondant a lieu. Ainsi, autres RiPPs peuvent être produites par la même méthode, pourvu que leur production recombinante est réalisable et conditions se retrouvent lorsque l’incorporation de la ncAA et PTM rendement une quantité suffisante de produit cible. Notez qu’outre le protéome de cellule hôte, la machinerie PTM peptide peut aussi devenir ncAA modifiés au cours de SPI. En conséquence, le moment de l’induction d’expression cible et de la durée de la période d’incubation suivant peuvent exiger optimisation. Étant donné que l’incorporation de ncAA dans les enzymes PTM peut affecter leur stabilité et leur activité, la production de la RiPP transformé peut en principe être affectée. Suffisante efficacité enzymatique PTM est indiquée par la formation de prépeptide transformé, tel que détecté par MS et bioactivité des essais. Tel que présenté ci-dessus, différents promoteurs inductibles pourraient être employées pour produire le PTM gènes tout d’abord (en l’absence de la ncAA) suivie de l’induction du gène peptide précurseur en présence de la ncAA. En général, la production du peptide cible cAA-contenant doit être supprimée avant l’addition de la ncAA, c’est pourquoi la répression serrée du gène précurseur est requise. Dans ce protocole, ceci est réalisé par la répression catabolique par l’addition de glucose dans le milieu de croissance. Surtout depuis que les enzymes PTM requises pour le traitement prepeptide proviennent habituellement d’un hôte génétiquement éloigné, utilisation de température et codon d’expression des gènes correspondants peut nécessiter l’optimisation si production recombinante n’a pas encore été mis en place. En principe, la présence de l’ANAC dans le prépeptide peut interférer avec la reconnaissance et le traitement des enzymes PTM, dans le cas de la nisine, NisBC et NisP. Pour l’incorporation de ncAA dans SAP, il est donc recommandé d’effectuer des expériences à petite échelle expression tout d’abord pour identifier les conditions de l’expression appropriée et la fiabilité du test de l’activité de l’AMP.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont aucun intérêt financier concurrentes.
Remerciements
J.H.N., T.B. et M.B. reconnaissent le financement par le programme UE FW7 (SYNPEPTIDE). F.-J.S. et T.F. reconnaissent le financement par le ministère fédéral de l’éducation et la Science (BMBF programme « HSP 2020 », TU-WIMIplus projet SynTUBio) et la Fondation de recherche allemande (Cluster d’Excellence « Unifying concepts in Catalysis »).
matériels
| Name | Company | Catalog Number | Comments |
| sodium chloride | Carl Roth, Germany | P029 | |
| guanidine hydrochloride | Carl Roth, Germany | 0035.2 | |
| dipotassium hydrogen phosphate | Carl Roth, Germany | P749.3 | |
| potassium dihydrogen phosphate | Carl Roth, Germany | 3904.3 | |
| sodium dihydrogen phosphate monohydrate | Carl Roth, Germany | K300.2 | |
| disodium hydrogen phosphate | Carl Roth, Germany | P030.2 | |
| L-alanine | Carl Roth, Germany | 3076.2 | |
| L-arginine | Carl Roth, Germany | 3144.3 | |
| L-asparagine monohydrate | Carl Roth, Germany | HN23.1 | |
| L-aspartic acid | Carl Roth, Germany | T202.1 | |
| L-cysteine | Carl Roth, Germany | 3467.3 | |
| L-glutamine | Carl Roth, Germany | 3772.1 | |
| L-glutamic acid | Carl Roth, Germany | 3774.1 | |
| L-glycine | Carl Roth, Germany | 3187.3 | |
| L-histidine | Carl Roth, Germany | 3852.3 | |
| L-isoleucine | Carl Roth, Germany | 3922.3 | |
| L-leucine | Carl Roth, Germany | 3984.3 | |
| L-lysine monohydrate | Carl Roth, Germany | 4207.2 | |
| L-methionine | Carl Roth, Germany | 9359.4 | |
| L-phenylalanine | Carl Roth, Germany | 4491.2 | |
| L-proline | Carl Roth, Germany | T205.3 | |
| L-serine | Carl Roth, Germany | 4682.4 | |
| L-threonine | Carl Roth, Germany | T206.2 | |
| L-tryptophan | Carl Roth, Germany | 4858.2 | |
| L-tyrosine | Carl Roth, Germany | T207.2 | |
| L-valine | Carl Roth, Germany | 4879.4 | |
| ammonium sulfate | Carl Roth, Germany | 3746.3 | |
| magnesium sulfate | Carl Roth, Germany | 0261.2 | |
| D-glucose | Carl Roth, Germany | 6780 | prepare a 20% (w/v) solution for addition into molten agar |
| calcium chloride | Carl Roth, Germany | PN93.2 | |
| iron(II) chloride | Sigma-Aldrich, Germany | 372870 | |
| thiamine hydrochloride | Sigma-Aldrich, Germany | T1270 | |
| biotin | Carl Roth, Germany | 3822.2 | |
| copper(II) sulfate | Merck, Germany | 102792 | |
| manganese(II) chloride | Carl Roth, Germany | KK36.2 | |
| zinc chloride | Merck, Germany | 108816 | |
| immonium orthomolybdate | Sigma-Aldrich, Germany | 277908 | |
| glycerol | Carl Roth, Germany | 7533.3 | |
| isopropyl β-D-1-thiogalactopyranoside | Sigma-Aldrich, Germany | I6758 | |
| ampicillin sodium salt | Carl Roth, Germany | K029.5 | working concentration 100 µg/mL for E. coli, prepare 100 mg/mL stock in ddH2O |
| kanamycin sulfate | Carl Roth, Germany | T832.2 | working concentration 50 µg/mL for E. coli, prepare 50 mg/mL stock in ddH2O |
| chloramphenicol | Carl Roth, Germany | 3886.1 | working concentration 5 µg/mL for L. lactis, prepare 37 mg/mL stock in ethanol |
| (4S)-fluoroproline | Bachem, Switzerland | F-3970 | |
| (4R)-fluoroproline | Bachem, Switzerland | F-3975 | |
| (4S)-hydroxyproline | Bachem, Switzerland | F-1395 | |
| (4R)-hydroxyproline | Bachem, Switzerland | F-2980 | |
| (4S)-methanoproline | chemically synthesized | ||
| (4R)-methanoproline | chemically synthesized | ||
| hydrochloric acid (HCl) | Carl Roth, Germany | P074.4 | |
| ethanol | VWR, Germany | 20825.324 | |
| M17-broth | Sigmal-Aldrich, Germany | 56156 | commercial product, see Terzaghi BE & Sandine WE, Appl Microbiol., 1975, 29(6):807-13 for contents and preparation |
| agar-agar | Carl Roth, Germany | 5210.5 | |
| Nisin from Lactococcus lactis | Sigma-Aldrich, Germany | N5764 | commercial product, can be used as reference for bioactivity |
| dimethyl sulfoxide (DMSO) | Carl Roth, Germany | A994.1 | |
| imidazole | Carl Roth, Germany | 3899.3 | |
| 1.5 mL autosampler vial for LC-MS | Sigma-Aldrich, Germany | Supelco 854165 | |
| acetonitrile | VWR, Germany | HiPerSolv CHROMANORM ULTRA for LC-MS, 83642 | LC-MS grade required |
| formic acid | VWR, Germany | HiPerSolv CHROMANORM for LC-MS, 84865 | LC-MS grade required |
| 1 mL Ni-NTA IMAC column, e.g. HisTrap FF Crude | GE Healthcare, UK | 29-0486-31 | different manufacturers and resins available for IMAC |
| 0.45 µm bottle top filter unit | VWR, Germany | 10040-470 | sterile filtration of solutions using a vacuum pump |
| 0.45 µm syringe filter PVDF membrane | Carl Roth, Germany | CCY1.1 | sterile filtration of solutions using a syringe and to remove particles from cell lysates |
| luer-lock syringe, PP, 50 ml | Carl Roth, Germany | T552.2 | sterile filtration of solutions |
| 1.5 mL Eppendorf tubes | Eppendorf, Germany | 30120086 | |
| petri dishes (polystyrene, sterile) | Carl Roth, Germany | TA19 | |
| Nile red | Sigma-Aldrich, Germany | 72485 | |
| E. coli ΔproA strain | CGSC, Keio collection | JW0233-2 | proline-auxotrophic E. coli K-12 strain |
| E. coli B834(DE3) | Novagen (Merck), Germany | 69041 | methionine-auxotrophic E. coli B strain |
| λDE3 Lysogenization Kit | Novagen (Merck), Germany | 69734-3 | |
| Lactococcus lactis NZ9000 pNG nisPT | bacterial indicator strain, see Khusainov R & Kuipers OP, PLoS One, 8 (9), e74890 | ||
| benchtop centrifuge for 1.5 mL Eppendorf tubes | Eppendorf, Germany | 5427 R | |
| peristaltic pump | GE Healthcare, UK | P1 | |
| FPLC system | GE Healthcare, UK | Äkta Purifier 10 or the like | |
| inverted microscope | Nikon | TI Eclipse wide-field fluorescence microscope with 100x (N.A. 1.4) objective and Mercury Lamp | example setup for fluorescence microscopy |
| electron multiplying CCD (EMCCD) camera | Andor Technologies, UK | Andor Luca | example setup for fluorescence microscopy |
| fluorescence excitation filter | Thorlabs, USA | Dichroic cube (TLV-U-MF2-TRITC) | example setup for fluorescence microscopy |
| fluorescence emission filter | AHF Analysentechnik, Germany | T 560 LPXR | example setup for fluorescence microscopy |
| cover slip 24 x 60 mm | Carl Roth, Germany | LH26.1 | example setup for fluorescence microscopy |
| Immersion Oil | Carl Zeiss, Germany | Immersol 518 F | example setup for fluorescence microscopy |
| probe sonicator | Bandelin, Germany | Sonopuls HD3200 with sonotrode MS-72 | 200 W maximum HF output |
| C5 HPLC column (2.1x100 mm, 3 µm particle size) | Sigma-Aldrich, Germany | 567227-U | example setup for mass spectrometry |
| ESI-TOF coupled to HPLC system | Agilent, USA | Agilent 6530 Accurate Mass QTOF with 1260 HPLC | example setup for mass spectrometry |
Références
- Ferri, M., Ranucci, E., Romagnoli, P., Giaccone, V. Antimicrobial resistance: A global emerging threat to public health systems. Crit Rev Food Sci Nutr. 57 (13), 2857-2876 (2017).
- Bahar, A. A., Ren, D. Antimicrobial peptides. Pharmaceuticals. 6 (12), 1543-1575 (2013).
- Ageitos, J. M., Sánchez-Pérez, A., Calo-Mata, P., Villa, T. G. Antimicrobial peptides (AMPs): Ancient compounds that represent novel weapons in the fight against bacteria. Biochem Pharmacol. 133, 117-138 (2017).
- Arnison, P. G., et al. Ribosomally synthesized and post-translationally modified peptide natural products: overview and recommendations for a universal nomenclature. Nat Prod Rep. 30 (1), 108-160 (2013).
- Lubelski, J., Rink, R., Khusainov, R., Moll, G. N., Kuipers, O. P. Biosynthesis, immunity, regulation, mode of action and engineering of the model lantibiotic nisin. Cell Mol Life Sci. 65 (3), 455-476 (2008).
- Shin, J. M., Gwak, J. W., Kamarajan, P., Fenno, J. C., Rickard, A. H., Kapila, Y. L. Biomedical applications of nisin. J Appl Microbiol. 120 (6), 1449-1465 (2016).
- Scherer, K. M., Spille, J. -. H., Sahl, H. -. G., Grein, F., Kubitscheck, U. The lantibiotic nisin induces lipid II aggregation, causing membrane instability and vesicle budding. Biophys J. 108 (5), 1114-1124 (2015).
- Jung, G. Lantibiotica - ribosomal synthetisierte Polypeptidwirkstoffe mit Sulfidbrücken und α,β-Didehydroaminosäuren. Angew Chemie. 103 (9), 1067-1084 (1991).
- Rink, R., et al. Lantibiotic structures as guidelines for the design of peptides that can be modified by lantibiotic enzymes. Biochemistry. 44 (24), 8873-8882 (2005).
- Lagedroste, M., Smits, S. H. J., Schmitt, L. Substrate Specificity of the Secreted Nisin Leader Peptidase NisP. Biochemistry. 56 (30), 4005-4014 (2017).
- Ross, A. C., Liu, H., Pattabiraman, V. R., Vederas, J. C. Synthesis of the lantibiotic lactocin S using peptide cyclizations on solid phase. J Am Chem Soc. 132 (2), 462-463 (2010).
- Fukase, K., et al. Synthetic Study on Peptide Antibiotic Nisin. V. Total Synthesis of Nisin. Bull Chem Soc Jpn. 65 (8), 2227-2240 (1992).
- Dumas, A., Lercher, L., Spicer, C. D., Davis, B. G. Designing logical codon reassignment - Expanding the chemistry in biology. Chem Sci. 6 (1), 50-69 (2015).
- Kuthning, A., Durkin, P., Oehm, S., Hoesl, M. G., Budisa, N., Süssmuth, R. D. Towards Biocontained Cell Factories: An Evolutionarily Adapted Escherichia coli Strain Produces a New-to-nature Bioactive Lantibiotic Containing Thienopyrrole-Alanine. Sci Rep. 6, 33447 (2016).
- Piscotta, F. J., Tharp, J. M., Liu, W. R., Link, A. J. Expanding the chemical diversity of lasso peptide MccJ25 with genetically encoded noncanonical amino acids. Chem Commun (Camb). 51 (2), 409-412 (2015).
- Hartman, M. C. T., Josephson, K., Lin, C. -. W., Szostak, J. W. An expanded set of amino acid analogs for the ribosomal translation of unnatural peptides. PLoS One. 2 (10), 972 (2007).
- Johnson, J. A., Lu, Y. Y., Van Deventer, J. A., Tirrell, D. A. Residue-specific incorporation of non-canonical amino acids into proteins: recent developments and applications. Curr Opin Chem Biol. 14 (6), 774-780 (2010).
- Ibba, M., Söll, D. Aminoacyl-tRNAs: setting the limits of the genetic code. Genes Dev. 18 (7), 731-738 (2004).
- Studier, F. W., Moffatt, B. A. Use of bacteriophage T7 RNA polymerase to direct selective high-level expression of cloned genes. J Mol Biol. 189 (1), 113-130 (1986).
- Budisa, N., Steipe, B., Demange, P., Eckerskorn, C., Kellermann, J., Huber, R. High-level biosynthetic substitution of methionine in proteins by its analogs 2-aminohexanoic acid, selenomethionine, telluromethionine and ethionine in Escherichia coli. Eur J Biochem. 230 (2), 788-796 (1995).
- Rink, R., et al. Dissection and modulation of the four distinct activities of nisin by mutagenesis of rings A and B and by C-terminal truncation. Appl Environ Microbiol. 73 (18), 5809-5816 (2007).
- Kubyshkin, V., Durkin, P., Budisa, N. Energetic contribution to both acidity and conformational stability in peptide models. New J Chem. 40 (6), 5209-5220 (2016).
- Molloy, E. M., Field, D., O'Connor, P. M., Cotter, P. D., Hill, C., Ross, R. P. Saturation mutagenesis of lysine 12 leads to the identification of derivatives of nisin A with enhanced antimicrobial activity. PLoS One. 8 (3), 58530 (2013).
- Wang, L., Brock, A., Herberich, B., Schultz, P. G. Expanding the genetic code of Escherichia coli. Science. 292 (5516), 498-500 (2001).
- Liu, C. C., Schultz, P. G. Adding new chemistries to the genetic code. Annu Rev Biochem. 79, 413-444 (2010).
- Anderson, J. C., Wu, N., Santoro, S. W., Lakshman, V., King, D. S., Schultz, P. G. An expanded genetic code with a functional quadruplet codon. Proc Natl Acad Sci U S A. 101 (20), 7566-7571 (2004).
- Zambaldo, C., Luo, X., Mehta, A. P., Schultz, P. G. Recombinant macrocyclic lanthipeptides incorporating non-canonical amino acids. J Am Chem Soc. 139 (34), 11646-11649 (2017).
- Al Toma, R. S., et al. Site-directed and global incorporation of orthogonal and isostructural noncanonical amino acids into the ribosomal lasso peptide capistruin. Chembiochem. 16 (3), 503-509 (2015).
- Chatterjee, A., Xiao, H., Schultz, P. G. Evolution of multiple, mutually orthogonal prolyl-tRNA synthetase/tRNA pairs for unnatural amino acid mutagenesis in Escherichia coli. Proc Natl Acad Sci U S A. 109 (37), 14841-14846 (2012).
- Budisa, N., Pal, P. P. Designing novel spectral classes of proteins with a tryptophan-expanded genetic code. Biol Chem. 385 (10), 893-904 (2004).
- Voller, J. -. s., Thi To, T. M., Biava, H., Koksch, B., Budisa, N. Global substitution of hemeproteins with noncanonical amino acids in Escherichia coli with intact cofactor maturation machinery. Enzyme Microb Technol. 106, 55-59 (2017).
- Moghal, A., Hwang, L., Faull, K., Ibba, M. Multiple Quality Control Pathways Limit Non-protein Amino Acid Use by Yeast Cytoplasmic Phenylalanyl-tRNA Synthetase. J Biol Chem. 291 (30), 15796-15805 (2016).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc Natl Acad Sci U S A. 97 (12), 6640-6645 (2000).
- Engelke, G., Gutowski-Eckel, Z., Hammelmann, M., Entian, K. D. Biosynthesis of the lantibiotic nisin: genomic organization and membrane localization of the NisB protein. Appl Environ Microbiol. 58 (11), 3730-3743 (1992).
- Baumann, T., Nickling, J. H., Bartholomae, M., Buivydas, A., Kuipers, O. P., Budisa, N. Prospects of In vivo Incorporation of Non-canonical Amino Acids for the Chemical Diversification of Antimicrobial Peptides. Front Microbiol. 8, 124 (2017).
- Plat, A., Kluskens, L. D., Kuipers, A., Rink, R., Moll, G. N. Requirements of the engineered leader peptide of nisin for inducing modification, export, and cleavage. Appl Environ Microbiol. 77 (2), 604-611 (2011).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Bacterial Transformation: The Heat Shock Method. J Vis Exp. , (2017).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Bacterial Transformation: Electroporation. J Vis Exp. , (2017).
- Shi, Y., Yang, X., Garg, N., van der Donk, W. A. Production of lantipeptides in Escherichia coli. J Am Chem Soc. 133 (8), 2338-2341 (2011).
- Hochuli, E., Bannwarth, W., Döbeli, H., Gentz, R., Stüber, D. Genetic Approach to Facilitate Purification of Recombinant Proteins with a Novel Metal Chelate Adsorbent. Nat Biotechnol. 6 (11), 1321-1325 (1988).
- Zhang, Z., Marshall, A. G. A universal algorithm for fast and automated charge state deconvolution of electrospray mass-to-charge ratio spectra. J Am Soc Mass Spectrom. 9 (3), 225-233 (1998).
- JoVE Science Education Database. High-Performance Liquid Chromatography (HPLC). J Vis Exp. , (2017).
- JoVE Science Education Database. Dialysis: Diffusion Based Separation. J Vis Exp. , (2017).
- Khusainov, R., Kuipers, O. P. The presence of modifiable residues in the core peptide part of precursor nisin is not crucial for precursor nisin interactions with NisB- and NisC. PLoS One. 8 (9), 74890 (2013).
- Terzaghi, B. E., Sandine, W. E. Improved medium for lactic streptococci and their bacteriophages. Appl Microbiol. 29 (6), 807-813 (1975).
- Schindelin, J., Rueden, C. T., Hiner, M. C., Eliceiri, K. W. The ImageJ ecosystem: An open platform for biomedical image analysis. Mol Reprod Dev. 82 (7-8), 518-529 (2015).
- . Phosphate-buffered saline (PBS). Cold Spring Harb Protoc. 2006 (1), (2006).
- van Hest, J. C. M., Tirrell, D. A. Efficient introduction of alkene functionality into proteins in vivo. Febs Lett. 428 (1-2), 68-70 (1998).
- Prince, A., et al. Lipid-II Independent Antimicrobial Mechanism of Nisin Depends On Its Crowding And Degree Of Oligomerization. Sci Rep. 6 (1), 37908 (2016).
- Zhou, L., et al. Incorporation of tryptophan analogues into the lantibiotic nisin. Amino Acids. 48 (5), 1309-1318 (2016).
- Presolski, S. I., Hong, V. P., Finn, M. G. Copper-Catalyzed Azide-Alkyne Click Chemistry for Bioconjugation. Curr Protoc Chem Biol. 3 (4), 153-162 (2011).
- Abts, A., et al. Easy and rapid purification of highly active nisin. Int J Pept. 2011, 175145 (2011).
- Francklyn, C. S., First, E. A., Perona, J. J., Hou, Y. -. M. Methods for kinetic and thermodynamic analysis of aminoacyl-tRNA synthetases. Methods. 44 (2), 100-118 (2008).
- van Heel, A. J., et al. Production and Modification of Five Novel Lantibiotics Using the Promiscuous Nisin Modification Machinery. ACS Synth Biol. 5 (10), 1146-1154 (2016).
- Wiegand, I., Hilpert, K., Hancock, R. E. W. Agar and broth dilution methods to determine the minimal inhibitory concentration (MIC) of antimicrobial substances. Nat Protoc. 3 (2), 163-175 (2008).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon