Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Isolant et en incorporant des antennes de lumière de diatomée Cyclotella Meneghiniana dans les Liposomes avec thylakoïdes des lipides
Dans cet article
Résumé
Nous présentons ici un protocole visant à isoler des protéines de liaison d’a/c du chlorophylle fucoxanthine (PCF) de diatomées et de les incorporer dans des liposomes dont la composition lipidique naturel pour étudier le transfert de l’énergie d’excitation sur les changements de composition ionique.
Résumé
Le rendement photosynthétique des plantes, algues et diatomées dépend fortement de la régulation rapide et efficace de la récolte et l’énergie lumineuse des processus de transfert dans la membrane des thylakoïdes des chloroplastes. La lumière récolte antenne de diatomées, qu’on appelle la fucoxanthine chlorophylle a/c les protéines liant la (PCF), sont nécessaires pour l’absorption de la lumière et un transfert efficace de la réaction photosynthétique centres aussi bien en ce qui concerne la photo-protection contre la lumière excessive. Le commutateur entre ces deux fonctions est une question de longue date de la recherche. Bon nombre de ces études ont été réalisées avec PCF dans les micelles de détergent. Pour les études sur les interactions, les détergents, qui a conduit à une agrégation imprécise des complexes du PCF, ont été supprimées. Dans cette approche, il est difficile de distinguer les objets et données physiologiquement pertinentes. On trouvera donc plus de précieuses informations sur FCP et autre liés à la membrane de lumière récolte complexes par l’étude des interactions protéine-protéine, de transfert d’énergie et d’autres caractéristiques spectroscopiques si ils sont intégrés dans leur environnement natif de lipides. Le principal avantage est que les liposomes ont une taille définie et un ratio de lipides/protéines déterminées par laquelle l’étendue de la FCP clustering est contrôlée. En outre, changements dans la composition de pH et des ions qui régulent la lumière récolte in vivo peuvent facilement être simulées. Par rapport à la membrane des thylakoïdes, les liposomes sont plus homogène et moins complexe, qui le rend plus facile à obtenir et à comprendre les données spectroscopiques. Le protocole décrit la procédure d’isolement du PCF et purification, préparation de liposome et incorporation du PCF dans les liposomes avec composition lipidique naturel. Résulte d’une application typique sont donnés et discutés.
Introduction
Organismes photosynthétiques tels que les diatomées doivent faire face aux conditions de lumière changeantes et répondre avec des mécanismes sophistiqués d’acclimatation qui soutiennent haut rendement photosynthétique et protègent contre la photooxydation dommages causés par la lumière excessive. Un important processus de lumière protectrice chez les eucaryotes photosynthétiques est la haute énergie trempe (qE) de la lumière absorbée qui apparaît comme la principale contribution à la trempe non photochimique (QNP) sous stress léger conditions1,2 ,3. Les complexes d’antenne récolte léger (LHC) sont impliqués dans la régulation des voies de transfert énergie excitation. En réponse à la forte luminosité induite par un faible pH dans le lumen du chloroplaste, les commutateurs de système d’antenne de la récolte à l’état de désactivation de la lumière. Cet état d’énergie dissipative protège les photosystèmes (PS) et autres complexes dans la membrane des thylakoïdes de photo-oxydation. Chez les eucaryotes photosynthétiques, le qE est généralement induit par deux facteurs1,2,3. L’un des facteurs sont la lumière spécialisée protéine qui répond au faible pHde récolte. La protéine de l’OSP induit le qE dans les plantes supérieures4. LhcSRs5, modulé par l’activité de l’OSP, induire le qE dans algues vertes6. Les diatomées possèdent des protéines de type Lhcx qui structurellement apparentés aux LHCSRs7,8,9,10.
Le deuxième facteur de qE est le cycle de la xanthophylle où caroténoïdes de l’antenne sont convertis sous une forme de protection photo par de-époxydation et rétablis par époxydation. Chez les plantes et les algues vertes, violaxanthine est converti en zéaxanthine. Dans les diatomées, diadinoxanthine est converti en diatoxanthine, qui ensuite est en corrélation avec la mesure de QNP11. La lumière de diatomées récolte antenne possède quelques particularités, bien qu’il soit évolutif relative aux plantes et algues LHCs. L’interrupteur de la lumière récolte photo-protection est extrêmement rapide et la capacité de QNP est plus élevé comparée aux plantes12. Cela pourrait être une des raisons pourquoi les diatomées sont très réussies dans des niches écologiques différentes dans la manière dont ils sont responsables de jusqu'à 45 % de la production primaire nette océanique13. Diatomées légers, systèmes de récolte sont donc un objet intéressant de recherche de la photosynthèse.
Diatomées, comme les espèces centrée sur Cyclotella meneghiniana, possèdent des thylakoïdes lumière intrinsèque systèmes nommés après les pigments qu’ils se lient - fucoxanthine, chlorophylle (chl) a et c, donc lumière PCF. récolte des protéines, tels que les FCP, sont intégré dans le système de membrane de thylakoid formée de plusieurs couches de membrane. Diatomées forment des bandes de trois thylakoïdes. Ce complexe situation, il est difficile de les étudier au niveau moléculaire dans la membrane des thylakoïdes. En outre, de nombreux composants contribuent à la régulation de la lumière récolte (voir ci-dessus). Donc, dans beaucoup d’approches, les complexes ont été isolés de la membrane à l’aide d’un détergent doux, tels que n-dodécyl-β-D-maltopyranoside (β-DDM), qui solubiliser la membrane, mais préserver les complexes du PCF. Beaucoup d’études spectroscopiques ont été effectuées à l’aide du PCF solubilisé pour enquêter sur le transfert d’énergie intramoléculaire14,15,16,17. Cependant, cette première approche était limitée, étant donné que le règlement du transfert d’énergie doit être excitonique interaction avec d’autres complexes d’antenne ou les photosystèmes. Par conséquent, ces sortes d’études ne peuvent être effectués avec solubilisée complexes parce que l’interaction entre les complexes est perdue.
Une caractéristique importante dans le règlement de l’antenne est « l'encombrement moléculaire » de l’antenne et les photosystèmes dans la membrane de thylakoid18. Auparavant, une approche simple a été réalisée pour simuler cet effet in vitro. Le détergent a été supprimé, ce qui conduit à l’agrégation aléatoire des complexes d’antenne. Bien que certaines données raisonnables a été obtenues par cette approche17,19, la suppression de détergent ne reflète pas la situation in vivo et a quelques limitations, étant donné que les complexes ne sont pas interagir dans leurs tertiaires réguliers structure.
L’utilisation de liposomes surmonte plusieurs des anciennes limites. La structure tertiaire est toujours intacte. La membrane du liposome fournit un environnement quasi natif pour les complexes d’antenne. La membrane sépare l’intérieur du liposome de l’environnement extérieur. Par ces moyens, liposomes offrent deux compartiments de réaction pour les études des gradients ioniques et pH aussi bien en ce qui concerne les processus de transport. En outre, les paramètres du système expérimental peuvent être contrôlés plus facilement pour les études dans la membrane des thylakoïdes. Liposomes se sont déjà avérées être un excellent outil pour l’étude des complexes de la photosynthèse. Une préoccupation majeure dans le passé était sur plante LHC où il a été testé l’effet de la composition lipidique altéré le LHC II20. Dans les autres approches, interaction protéine-protéine entre différents LHC II ont été étudiées21. En outre, certaines études chez les algues vertes ont été réalisées qui décrivent un regroupement spontané entre LHC22. Compte tenu de l’importance des diatomées pour les écosystèmes aquatiques, relativement peu d’études ont été réalisées avec des complexes de l’antenne de diatomées. Deux études ont analysé les complexes d’antenne de la centrée sur Cyclotella meneghiniana, où le regroupement de la FCP antenne23 et la réactivité du PCF aux gradients électrochimiques24 apparaissaient. Ainsi, les liposomes sont un excellent outil pour étudier les antennes de diatomées et de leur interaction et règlement dans des conditions presque natives. Les liposomes sont versatiles depuis plusieurs conditions telles que la composition lipidique, taille de liposome, densité de protéine et la phase aqueuse environnante peut être contrôlée. En outre, la méthode nécessite de faibles quantités d’échantillons. Le système expérimental est robuste et hautement reproductible. Le cloisonnement des liposomes permet pour l’étude de pH et gradients ioniques, qui sont d’importants facteurs dans la régulation des complexes d’antenne.
Nous décrivons ici l’isolement des complexes d’antenne du PCF de c. meneghiniana et leur incorporation dans les liposomes avec composition lipidique naturel thylakoïdes. Aussi, nous fournissent des données exemplaires pour la caractérisation spectroscopique du PCF solubilisée et comparez-les avec le PCF dans les liposomes. La méthode résume les connaissances et les protocoles normalisés obtenus des améliorations de Gundermann et Büchel 201223, Natali et coll. 201622et Ahmad et Dietzel 201724.

Figure 1 : représentation schématique du flux de travail. (1) fait référence au paragraphe 1, qui décrit la croissance cellulaire, perturbation et l’isolement des thylakoïdes avec post-séparation FCP sur des gradients de sucrose de densité ; M. c. -Cellules deCyclotella meneghiniana . (2) préparation du mélange de lipides naturels thylakoïdes (MGDG, corentin et SQDG) décrit au paragraphe 2 et la création des micelles de lipide-détergent avec octylglycoside (OG). Une taille définie lipide-micelle est obtenue par extrusion à l’aide de membranes d’un diamètre de pore définis. FCP et lipides-micelles sont unifiés à un lipide prédéfini : ratio de protéine et les détergents OG et β-DDM sont supprimés via contrôlée dialyse formant des protéoliposomes du PCF. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Protocole
Remarque : Photosynthétiques complexes tels que les FCP sont extrêmement vulnérables à la lumière et de chaleur. Travaillez toujours sur la glace et sous une lumière très faible.
1. isolement du PCF de cellules
-
Isolement de thylakoïdes des cellules meneghiniana c.
- Grandir c. meneghiniana dans cinq flacons de 500 mL que chacun rempli de 300 mL d’ASP-moyen23,25 et 50 millions de cellules. Branchez les flacons avec un bouchon de coton et laisser les cellules à se développer à la phase de croissance exponentielle pendant environ une semaine sur un agitateur à 120 t/mn avec un 16 h de lumière et phase obscure de 8 h à 40 µmol photons/(m²s) blanc lumière et une température comprise entre 15 et 18 ° C. Vérifiez que le nombre de cellules est entre 1,5 millions de cellules/mL avec une chambre de compteur de cellules.
- Centrifuger les cellules à 4 000 x g dans un rotor rafraîchie avec des flacons de 500 mL Centrifugeuse (4 ° C) pendant 15 min dans une centrifugeuse à grande vitesse. En pipettant également, une nouvelle suspension des granulés de cellule dans 12 mL de tampon d’homogénéisation (HB, tableau 1).
- Transférer la suspension dans un tube en plastique unique de 50 mL. Stocker les échantillons à-80 ° C ou passer au point 1.1.3.
- Refroidir préalablement le moulin de perle et l’équipement. Remplissez le bécher de 50 mL de l’usine de perle à 75 % avec un mélange de perles de verre et ajoutez la suspension cellulaire. Pour la rupture de la cellule, utiliser 7 x 45 s impulsions à pleine vitesse avec 30 s de refroidissement entre chaque impulsion. Prendre 20 µL de cellules perturbées pour des contrôles de qualité à l’étape 1.1.6.
- Filtrer les cellules perturbés sur un entonnoir filtrant verre et lavage en versant le HB sur les perles de verre, jusqu'à ce qu’elles apparaissent claires. Piscine la fraction de lavage avec le filtrat. Garder le volume final inférieur à 150 mL.
- Centrifuger l’échantillon pendant 15 min à 140 x g à l’aide de trois tubes en plastique de 50 mL pour granuler les débris cellulaires. Avec précaution, transférer le surnageant pour flacons de 20 mL en polycarbonate ultracentrifugation et jeter le culot.
- Remplir les flacons avec HB, équilibrer le poids et centrifuger dans un rotor adapté pendant 1 h à 300 000 x g et 4 ° C pour granuler les membranes thylakoïdes.
- Le temps de centrifugation permet de vérifier la proportion de cellules perturbées au microscope au grossissement de X 400 avec l’échantillon 20 µL en 1.1.3. Calculer le rapport entre libre des chloroplastes et chloroplaste contenant des frustules (coquilles de silice).
Remarque : Diatomées les parois cellulaires sont faits de silice, qui est visible sous la forme hautement diffractifs substance au microscope. Chloroplastes se produisant des points aussi verts auraient dû être libérés des cellules si la perturbation de la cellule a travaillé. - Resuspendre le culot de membrane avec comme petit tampon de lavage possible (0,5 à 1 mL) à l’aide de la brosse un petit peintre. Remplir les flacons d’ultracentrifugation en polycarbonate avec lavage tampon (tableau 1), d’équilibrer leur poids et centrifuger pendant 20 min à 200 000 x g et 4 ° C.
- Remettre en suspension les membranes lavées avec pinceau d’un peintre. Ajouter le tampon de lavage uniquement si nécessaire pour maintenir la concentration de thylakoïdes aussi haute que possible. Mettre en commun tous les thylakoïdes dans un flacon (15 mL).
- Avant de diluer les échantillons à l’aide de 10 µL de l’échantillon avec 90 ml d’acétone 100 %. Il centrifuger à 12 000 x g pendant 5 min granuler protéines précipitées. Prendre 10 µL de la dilution et mélangez-le avec 990 µL de l’acétone à 90 %.
- Mesurer l’absorbance (ABS) de la chlorophylle a et c à 664 nm et 630 nm dans l’acétone à 90 %. Soustraire l' ABS750nm de ces deux valeurs. Déterminer la teneur en chlorophylle totale à l’aide de la formule suivante :24
1)
2)
- Mesurer l’absorbance (ABS) de la chlorophylle a et c à 664 nm et 630 nm dans l’acétone à 90 %. Soustraire l' ABS750nm de ces deux valeurs. Déterminer la teneur en chlorophylle totale à l’aide de la formule suivante :24
- Les thylakoïdes aliquotes dans des portions de 0,5 mg de chlorophylle totale en un tube de réaction de 1,5 mL, congelez-les dans l’azote liquide et les conserver à-80 ° C jusqu'à l’utilisation ultérieure.
-
Séparation et la concentration de complexes du PCF
- Préparer une solution de gradients de sucrose et remplir les tubes ultracentrifugeuse jusqu'à ce que le dessus moins le volume de chargement (300 – 500 µL). Geler les tubes à-20 ° C jusqu'à ce qu’ils sont complètement gelés. Laisser les tubes décongeler à + 4 ° C, qui prend 3-4 h pour un tube de 17 mL.
- Répétez le cycle gel-dégel, deux fois pour affiner le gradient pour la meilleure résolution.
- Utiliser les échantillons obtenus en 1.1.9 correspondant à 0,5 mg de chlorophylle et ajuster avec le tampon B1 (tableau 1) pour un volume final de 2 mL. De solubilisation, ajoute n-dodécyl-β-D-maltopyranoside (b-DDM) à une concentration finale de 20 mM.
- Renverser le tube 3 fois et placez-le sur la glace pendant 20 min avec agitant doucement pour éviter la mousse. Centrifuger pendant 5 min, à 12 000 x g dans une centrifugeuse de dessus de table refroidis à + 4 ° C.
- Charger le surnageant sur le gradient. Ne chargez pas plus de 125 µg de chlorophylle totale par gradient si 17 mL flacons sont utilisés. Centrifugeuse de 22 h à 100 000 x g et + 4 ° C.
- Récupérez les fractions FCP brunes désirées depuis le dégradé à l’aide d’une seringue (Figure 2 a). Prenez un 5 µL aliquote et diluez-la avec 995 µL de B1a.
- Mesurer le spectre d’absorbance (ABS) entre 370-750 nm dans un spectrophotomètre UV-VIS. Utiliser des cuves semi-micro verre optique.
- Ouvrez le logiciel de gestionnaire de spectres, accédez au mode spectre et enregistrer une planification initiale de 370 à 750 nm suivie de mesures d’échantillons. Utilisez les paramètres suivants : scan vitesse, 200 nm/min ; données de terrain, 0,5 nm ; réponse, moyen.
- Mesurer le volume de l’échantillon récupéré avec une micropipette. Laver les complexes du PCF en ajoutant deux fois le volume récupéré avec tampon de B1 (tableau 1). Se concentrent dans une pompe à membrane avec une coupure à 1 000 x g et + 4 ° C pour un ABS672nm d’au moins 20 de 30 kDa.
Remarque : La concentration de b-DDM pourrait augmenter en raison de l’enrichissement de la micelle dans le compartiment de l’échantillon. Cela pourrait entraîner d’autres solubilisation des complexes du PCF ! Éviter trop de solubilisation en ajoutant le tampon sans détergent B1 si autre lavage étapes sont nécessaires pour supprimer le saccharose résiduel. - Prenez un 20 µL aliquote pour les contrôles. Choc congeler les échantillons dans l’azote liquide et les stocker à-80 ° C.
- Préparer une solution de gradients de sucrose et remplir les tubes ultracentrifugeuse jusqu'à ce que le dessus moins le volume de chargement (300 – 500 µL). Geler les tubes à-20 ° C jusqu'à ce qu’ils sont complètement gelés. Laisser les tubes décongeler à + 4 ° C, qui prend 3-4 h pour un tube de 17 mL.

Figure 2 : Purification des FCP, spectroscopiques contrôles et vérification de la pureté. (A) aspect typique d’un gradient de densité de saccharose après une centrifugation. Toutes les bandes brunes contiennent la piscine FCP consistant en pigment FCPa et FCPb.. - non consolidé les pigments, ch - photosystèmes spectres d’Absorbance (B) du PCF avant (ligne bleue) et après concentration (ligne discontinue orange) à l’aide de dispositifs de filtre centrifuge avec coupure de 30 kDa . En particulier, les caroténoïdes sont sujettes à la perte du PCF, qui se traduirait par absorbance inférieure dans la région entre 500 à 550 nm. Graphiques sont normalisées à chlorophylle Qy maximale à ~ 670 nm. (C) la chlorophylle a des spectres d’émission avec excitation de chl c (465 nm) pour tester le transfert d’énergie excitation fonctionnelle. Si l’énergie de transfert de chl c à chl a est entravée, une bande supplémentaire de fluorescence à ~ 640 nm (LCH c) se produirait. Graphiques sont normalisées à l’émission maximale. (D) enregistré les spectres d’Excitation à 675 nm (chl une fluorescence maximale) pour tester le transfert d’énergie au chl a de tous les pigments absorbants entre 370 nm et 600 nm. Si l’énergie de transfert à chl a est moins efficace, le rendement de fluorescence diminuerait surtout entre 465 et 550 nm. Les graphiques sont normalisées au maximum environ 440 nm. Les spectres (b), (C) et (D) sont presque identiques si la concentration a bien fonctionné. (E) vérifier la pureté du FCP isolé à l’aide d’un gel de Tris-tricine28. FCPa et FCPb ont sous-unités entre 18 et 19 kDa. Toutes les protéines visibles colorés à l’argent plus de 20 kDa sont des contaminants. Thyl. -Les thylakoïdes s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
-
Spectroscopiques et gel à base de contrôles
- Mesurer l’absorbance entre 370-750 nm de 5 µL du PCF en 995 µL de B1a après l’étape 1.2.5. En comparaison avec le spectre obtenu à l’étape 1.2.4. Utilisez la même instrumentation et les paramètres comme indiqué au point 1.2.4.
- Exporter des données sous *.csv et importer les données dans une feuille de calcul. Normaliser les deux spectres au maximum de la chl a dans la bande dey Q d’environ 672 nm comme illustré à la Figure 2 b.
- Calculer le facteur de dilution des échantillons à l’étape 1.2.4 et 1.3.1 en divisant la mesurée de l’ABS672nm par le désiré de l’ABS672nm de 0,03. Diluer les échantillons en conséquence avec B1a et transférez-les vers des cuvettes de fluorescence verre spécial.
- Chl record un spectre d’émission de fluorescence avec un spectrofluorimètre à révéler une énergie lumineuse intacte transfert de chl c chl a (Figure 2).
- Utiliser le logiciel de spectromètre et allez dans le mode de mesure du spectre avec les paramètres suivants : mode, des émissions ; fente de largeur excitation et émission, 3 nm ; sensibilité, moyenne ; Vitesse de balayage, 100 nm/min ; données de terrain, 0,5 nm ; onde d’excitation, 465 nm ; Emission, 600 à 800 nm. Effectuer la mise et mesure.
- Enregistrer les spectres d’excitation avec le même échantillon et équipement de transfert d’énergie intacte de tous les pigments au chl de révéler un (Figure 2D). Modifier les paramètres pour : mode, excitation ; longueur d’onde d’émission 675 nm ; excitation, 370-600 nm. Enregistrer les spectres.
- Corriger avec un spectre de rhodamine dans la même gamme pour des propriétés spectrales de la lampe – FC. instructions dans le manuel de l’utilisateur.
- Échantillons de FCP Mix correspondant à 1 µg de chl a avec 10 µL de tampon de SDS-charge. Incuber pendant 10 min à 25 ° C. Centrifuger 5 min à 12000 x g dans une centrifugeuse de dessus de table.
- Charger le surnageant sur un gel de Tris-tricine28. Séparez-le pendant 2 h à 150 V et argent-tache après séparation40.
Remarque : Les sous-unités du PCF se divisent en deux bandes de protéines importantes entre 18 et 19 kDa, qui sont des constituants de la FCPa et FCPb29 (Figure 2E).
- Charger le surnageant sur un gel de Tris-tricine28. Séparez-le pendant 2 h à 150 V et argent-tache après séparation40.
- Mesurer l’absorbance entre 370-750 nm de 5 µL du PCF en 995 µL de B1a après l’étape 1.2.5. En comparaison avec le spectre obtenu à l’étape 1.2.4. Utilisez la même instrumentation et les paramètres comme indiqué au point 1.2.4.
2. préparation des Liposomes et Incorporation du PCF
-
Préparation du mélange de lipides et micelles détergent lipidique
Remarque : lipides sont sensibles aux chaudes températures combinées avec des conditions oxydatives. Essayez de garder les lipides réfrigérés et sous une atmosphère de2 N.- Calculer les ratios de lipides de thylakoïdes désirée pour c. meneghiniana selon Vieler et al. 200730. Reportez-vous à l’exemple donné en Supplement , tableau 1. Préparer les lipides solutions recommandées par le fabricant dans un solvant contenant étanche.
- Pipeter la quantité désirée de lipides dans un tube à essais 2 mL et évaporer le chloroforme à l’aide d’un courant d’azote doux et essayer de disperser les lipides sur toute la surface de la base du tube. Laisser le flux de2 N jusqu'à ce que tous les solvant s’évapore.
- Solubiliser le mélange lipidique dans 29 µL de solution de β-D-glucopyranoside de n-octyle (OG) à 4 ° C pendant 4 h. Incuber le mélange de lipides pendant 10 min à 30 ° C. Incuber les lipides dans un bain sonicateur pendant 3 x 3 min à 25 ° C, interrompu par 30 s sur la glace.
- Ajouter 221 µL de tampon de tricine et 250 µL de tampon de dialyse 4 x.
- Utilisez une extrudeuse avec des membranes de polycarbonate 0,1 µm pour un diamètre de liposome défini de 50 à 70 nm. Assembler l’extrudeuse avec le soutien de la membrane et le filtre. Éviter les bulles d’air et serrez l’ensemble soigneusement.
- Remplissez une seringue avec un tampon de dialyse x 4 et pré mouillage l’extrudeuse jusqu'à ce qu’aucune bulle ne peut être vu dans la deuxième seringue.
- Appliquer les lipides-détergent-micelles à l’extrudeuse et appuyer sur la solution d’une seringue à l’autre avant et arrière. Répétez cette étape 5 fois jusqu'à ce que la solution apparaît homogène.
Remarque : Cette solution peut être conservée à 4 ° C pendant plusieurs jours. Ne pas congeler !
-
Incorporation des complexes FCP et enlèvement des détergents et des agrégats
Remarque : Dans cet exemple, nous utilisons un lipide/chl un rapport 12:1, qui correspond à un taux de lipides/protéines de près de 100 : 1.- Ajouter FCP égale à 20 µg de chl a dans un volume total de 500 µL de tampon de B1a à 250 μL des lipides-micelles extrudés et 250 µL de 4 x tampon de dialyse. Incuber les échantillons pendant 3 x 3 min à 25 ° C dans un thermomixer à 1 500-3 000 tr/min, interrompu par une pause de 30 s sur la glace.
- Couper le couvercle du tube de réaction de 1,5 mL quatre juste sous le sommet donnant un anneau qui correspond encore sur le couvercle. Préparer des morceaux de 1,5 cm x 1,5 cm de membrane de dialyse et lavez-les dans 20 mL de tampon de dialyse x 1
- Remplir 250 µL de l’échantillon pour chaque couvercle. Attentivement, poser la membrane sur le couvercle afin que le compartiment est complètement rempli avec l’échantillon et sans bulles d’air. Serrez la bague de tube de réaction sur l’Assemblée afin de disposer d’un compartiment fermé.
- Dialyser les échantillons dans 50 mL de 1 x tampon de dialyse pendant la nuit (12-16 h) sur la glace sur un agitateur de culbutage. Remplacer le tampon de dialyse utilisé par fraîche et ajouter 7 mg de perles absorbants pour enlever le résidu de détergent pendant au moins 6 h.
- Replacez le tampon de dialyse et dialyser car un autre 12 h. récupérer les liposomes par perforation de la membrane de dialyse avec une pointe de micropipette 200 µL et aspirer tous les liposomes du couvercle du tube de réaction.
- Étape facultative : si haute pureté (> 95 %) est nécessaire. Préparer un gradient de densité discontinu dans 17 mL flacons ultracentrifugation avec étapes contenant 6 %, 10 %, 15 % et 20 % de sucrose COPOLYMERE d’épichlorhydrine dans le tampon de la dialyse. Charger les liposomes sur le dessus et ultracentrifugation à 100 000 x g pendant 4 h dans un rotor oscillant de seau.
- Récupérer la bande supérieure brune avec une seringue, diluer l’échantillon 1:5 avec DP et passez à l’étape suivante.
- Centrifuger les liposomes FCP dans au moins 2 mL 1 x tampon de dialyse pendant 1,5 h à 100 000 x g et 4 ° C. Récupérer les liposomes en tournant le tube à centrifuger à un angle de 45°. Laissez les liposomes descendre pendant 1 min (Figure 3 a).
- Récupérer les liposomes FCP dans un volume final de 25 à 50 µL. éviter de déranger le précipité.
-
Commandes 1 : absorbance, la spectroscopie de fluorescence
- Ajouter une quantité de 3 µL des FCP-liposomes pour un volume final de 1 mL de 1 x tampon de dialyse et centrifuger pendant 5 min à 12 000 x g.
- Mesurer l’absorbance entre 370 et 750 nm du PCF-liposomes avec le même équipement tel que décrit dans 1.2.4. Normaliser le spectre au maximum dans la région dey Q de la chl a (670-680 nm) et de le comparer au spectre normalisé du PCF solubilisé (Figure 3).
- Préparer du PCF-liposomes dans 1 mL de DP avec une absorbance (ABS) = 0,03 en ce qui concerne la valeur maximale entre 670-680 nm. Ajuster le PCF solubilisé dans un détergent de l’étape 1.2.6 à l’ABS même diluant avec B1a.
- Enregistrer les spectres d’absorbance des deux échantillons comme décrit dans 1.2.4. Enregistrer le chl un spectre d’émission de fluorescence des deux échantillons comme décrit dans 1.3.3.
Remarque : Le rendement de fluorescence est diminué dans l’échantillon du PCF-liposome (Figure 3D et cf. discussion.)
- Ajouter une quantité de 3 µL des FCP-liposomes pour un volume final de 1 mL de 1 x tampon de dialyse et centrifuger pendant 5 min à 12 000 x g.
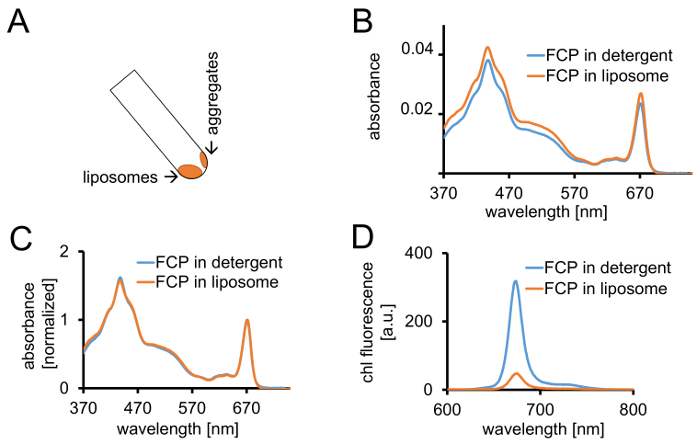
Figure 3 : isolement des FCP protéoliposomes suivie de contrôles spectroscopiques et imagerie confocale. (A) récupération des liposomes FCP après centrifugation. Tourner le tube de centrifugation à 45° et attendre environ 1 min - les liposomes seront déplace vers le bas tandis que le PCF agrégats qui ne sont pas incorporés dans des liposomes bâton à la paroi du tube. (B) la comparaison des spectres d’absorbance de FCP solubilisée dans un détergent (bleu) et du PCF dans les liposomes (orange) (C) les mêmes spectres comme dans (B) normalisé à chl un maximum dans le rouge (~ 670 nm - Qy PIC) ; FCP solubilisée dans un détergent (bleu) et le PCF dans les liposomes (orange). Potentiellement, il y aurait une perte de pigment, principalement des caroténoïdes visible dans la région de 500 à 550 nm. Le regroupement du PCF dans les liposomes peut conduire à un élargissement du PIC et un léger décalage de la LCH un maximum (~ 670 nm) vers le rouge. (D) des spectres d’émission des FCP solubilisée dans un détergent et le PCF dans un liposome. Regroupement du PCF dans le liposome améliore les interactions énergétiques des complexes du PCF qui abaisse le rendement de fluorescence (courbe orange) et décale les maxima d’émission légèrement vers le rouge. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Résultats
Le protocole décrit l’isolement de la fraction totale de FCP de Cyclotella meneghiniana et incorporation dans les liposomes avec composition lipidique native. L’isolement de thylakoïdes est hautement reproductible, mais le rendement de thylakoïdes peut-être changer. Le résultat est acceptable si plus de 50 % de tous les pigments sont récupérés à l’étape 1.1.4. Plus de 80 % est optimale.
La solubilisation...
Discussion
Liposomes FCP avec composition lipidique naturel offrent un outil pratique, simple et reproductible pour étudier les propriétés spectroscopiques in vitro. L’environnement lipidique dans les liposomes FCP ressemble à la situation au sein de la membrane des thylakoïdes, donnant lieu à des résultats expérimentaux qui sont plus proches des conditions naturelles.
Il y a plusieurs avantages de l’utilisation de c. meneghiniana comme système modèle pour antenne FCP. Il ...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions Rana Adeel Ahmad d’assistance dans la purification du PCF. Prof. Claudia Büchel est reconnu pour les discussions utiles et la lecture du manuscrit. Ce travail a été soutenu par la Fondation allemande de recherche à LD (DI1956-1/1) et la Fondation Humboldt pour une bourse de Feodor-Lynen à LD
matériels
| Name | Company | Catalog Number | Comments |
| 500 ml centrifuge vials | |||
| high speed centrifuge | Heraeus | ||
| Bead Mill VI 2 | Edmund-Bühler (edmund-buehler.de) | newer version: Vibrogen-Zellmühle Vl 6 | |
| Silibeads S 400 µm | Sigmund-Lindner.com | 5223-7 | |
| Silibeads S 1,-1,3 mm | Sigmund-Lindner.com | 4504 | |
| VitraPOR filter funnel - por1 | ROBU GmbH | 21121 | |
| polycarbonate ultracentrifuagtion vials (30 mL) for T-865 | Beranek Laborgeräte (Laborgeraete-beranek.de) | 314348 | |
| Ultracentrifuge Discovery 90SE | Sorvall | n.a. | |
| rotor T 865 | ThermoFisher Scientific (thermofisher.com) | 51411 | |
| Neubauer Cell Counter Chamber (improved) | Carl Roth Laborbedarf (Carlroth.com) | T729.1 | |
| Zeiss Mikroskop Primostar (7) | Optik-Pro (optik-pro.de) | 51428 | |
| optical glass cuvettes (6040-OG) | Hellma Analytics (hellma-analytics.com) | "6040-10-10" | |
| V-630 UV-VIS Spectrophotometer (incl. software) | Jasco (jasco.de) | V-630 | |
| n-Dodecyl-β-D-Maltopyranoside | ANATRACE (anatrace.com) | D310LA | |
| Ultra-Clear tubes 17 ml for AH629 | Beranek Laborgeräte (Laborgeraete-beranek.de) | 344061 | |
| rotor AH629-17-mL | ThermoFisher Scientific (thermofisher.com) | 54285 | |
| Membrane concentrator_Centriprep 30 kDa cutoff | Millipore (merckmillipore.com) | 4307 | |
| Biometra Minigel-Twin | Analytik Jena AG (analytik-jena.de) | 846-010-100 | |
| Silver Stain Plus Kit | Bio-Rad (bio-rad.com) | 1610449 | |
| libre office spread sheet | The document foundation | https://de.libreoffice.org/download/libreoffice-still/ | |
| special glass cuvettes for fluorescence (101-0S) | Hellma Analytics (hellma-analytics.com) | 101-10-20 | |
| Spectrofluorometer FP-6500 (incl. Software) | Jasco (jasco.de) | FP-6500 | |
| SDS-loading buffer Roti-Load | ROTH (carlroth.com) | K929.1 | |
| n-octyl β-D-glucopyranoside | ANATRACE (anatrace.com) | O311 | |
| Monogalactosyl Diaclyglycerol (MGDG) | Larodan AB (larodan.com) | 59-1300 | make stock solution in chloroform |
| Digalactosyl Diacylglycerol (DGDG) | Larodan AB (larodan.com) | 59-1310 | make stock solution in chloroform |
| Sulphoquinovosyl Diacylglycerol (SQDG) | Larodan AB (larodan.com) | 59-1230 | make stock solution in chloroform |
| L-alpha-Phosphatidylglycerol (PG) | Larodan AB (larodan.com) | 37-0150 | make stock solution in chloroform |
| L-α-Phosphatidylcholine | Sigma-Aldrich (sigmaaldrich.com) | P3782 SIGMA | make stock solution in chloroform |
| sonicator bath S-50TH | Sonicor (getmedonline.com | SONICOR-S-50TH | |
| mini-Extruder | Avanti Polar Lipids (Avanti.com) | 610000 | |
| Nuleopore polycarbonate membrane | Avanti Polar Lipids (Avanti.com) | 610005 | |
| dialysis membrane Visking 14 kDa cutoff | ROTH (carlroth.com) | 0653.1 | boil in destilled water before use |
| Biobeads SM2 Adsorbent | Biorad (Bio-rad.com) | 152-3920 | |
| sucrose epichlorhydrin copolymer - Ficoll 400 | Sigma-Aldrich (sigmaaldrich.com) | F4375 | |
| Polycarbonate ultracentrifuagtion vials (2.7 mL) for TFT 80.4 | Beranek Laborgeräte (Laborgeraete-beranek.de) | 252150 | |
| rotor TFT 80.4 | Millipore (merckmillipore.com) | 54356 | |
| material listed in order of appearance | For specific safety instructions please refer to material safety sheets and repective manuals. Standard lab material and substances are not listed. |
Références
- Eberhard, S., Finazzi, G., Wollman, F. A. The Dynamics of Photosynthesis. Annual Review of Genetics. 42, 463-515 (2008).
- Li, Z. R., Wakao, S., Fischer, B. B., Niyogi, K. K. Sensing and Responding to Excess Light. Annual Review of Plant Biology. 60, 239-260 (2009).
- Niyogi, K. K., Truong, T. B. Evolution of flexible non-photochemical quenching mechanisms that regulate light harvesting in oxygenic photosynthesis. Current Opinion in Plant Biology. 16 (3), 307-314 (2013).
- Li, X. -. P., et al. A pigment-binding protein essential for regulation of photosynthetic light harvesting. Nature. 403 (6768), 391-395 (2000).
- Peers, G., et al. An ancient light-harvesting protein is critical for the regulation of algal photosynthesis. Nature. 462 (7272), 518-521 (2009).
- Correa-Galvis, V., et al. Photosystem II Subunit PsbS Is Involved in the Induction of LHCSR Protein-dependent Energy Dissipation in Chlamydomonas reinhardtii. The Journal of biological chemistry. 291 (33), 17478-17487 (2016).
- Bailleul, B., et al. An atypical member of the light-harvesting complex stress-related protein family modulates diatom responses to light. Proceedings of the National Academy of Sciences of the United States of America. 107 (42), 18214-18219 (2010).
- Taddei, L., et al. Multisignal control of expression of the LHCX protein family in the marine diatom Phaeodactylum tricornutum. Journal of experimental botany. 67 (13), 3939-3951 (2016).
- Lepetit, B., et al. The diatom Phaeodactylum tricornutum adjusts nonphotochemical fluorescence quenching capacity in response to dynamic light via fine-tuned Lhcx and xanthophyll cycle pigment synthesis. New Phytologist. 214 (1), 205-218 (2017).
- Büchel, C. Evolution and function of light harvesting proteins. Journal of Plant Physiology. 172, 62-75 (2015).
- Lavaud, J., Rousseau, B., van Gorkom, H. J., Etienne, A. -. L. Influence of the Diadinoxanthin Pool Size on Photoprotection in the Marine Planktonic Diatom Phaeodactylum tricornutum. Plant Physiology. 129 (3), 1398-1406 (2002).
- Ruban, A., et al. The super-excess energy dissipation in diatom algae: comparative analysis with higher plants. Photosynthesis Research. 82 (2), 165-175 (2004).
- Mann, D. G. The species concept in diatoms. Phycologia. 38 (6), 437-495 (1999).
- Papagiannakis, E., van Stokkum, I. H. M., Fey, H., Büchel, C., van Grondelle, R. Spectroscopic Characterization of the Excitation Energy Transfer in the Fucoxanthin-Chlorophyll Protein of Diatoms. Photosynthesis Research. 86 (1-2), 241-250 (2005).
- Premvardhan, L., Robert, B., Beer, A., Büchel, C. Pigment organization in fucoxanthin chlorophyll a/c2 proteins (FCP) based on resonance Raman spectroscgopy and sequence analysis. Biochimica et Biophysica Acta-Bioenergetics. 1797 (9), 1647-1656 (2010).
- Gildenhoff, N., Herz, J., Gundermann, K., Büchel, C., Wachtveitl, J. The excitation energy transfer in the trimeric fucoxanthin-chlorophyll protein from Cyclotella meneghiniana analyzed by polarized transient absorption spectroscopy. Chemical Physics. 373 (1), 104-109 (2010).
- Ramanan, C., et al. Exploring the mechanism(s) of energy dissipation in the light harvesting complex of the photosynthetic algae Cyclotella meneghiniana. Biochimica et Biophysica Acta-Bioenergetics. 1837 (9), 1507-1513 (2014).
- Haferkamp, S., Kirchhoff, H. Significance of molecular crowding in grana membranes of higher plants for light harvesting by photosystem II. Photosynthesis Research. 95 (2-3), 129-134 (2008).
- Wahadoszamen, M., et al. Stark fluorescence spectroscopy reveals two emitting sites in the dissipative state of FCP antennas. Biochimica et Biophysica Acta-Bioenergetics. 1837 (1), 193-200 (2014).
- Zhou, F., et al. Effect of monogalactosyldiacylglycerol on the interaction between photosystem II core complex and its antenna complexes in liposomes of thylakoid lipids. Photosynthesis Research. 99 (3), 185-193 (2009).
- Moya, I., Silvestri, M., Vallon, O., Cinque, G., Bassi, R. Time-resolved fluorescence analysis of the photosystem II antenna proteins in detergent micelles and liposomes. Biochemistry. 40 (42), 12552-12561 (2001).
- Natali, A., et al. Light-harvesting Complexes (LHCs) Cluster Spontaneously in Membrane Environment Leading to Shortening of Their Excited State Lifetimes. Journal of Biological Chemistry. 291 (32), 16730-16739 (2016).
- Gundermann, K., Büchel, C. Factors determining the fluorescence yield of fucoxanthin-chlorophyll complexes (FCP) involved in non-photochemical quenching in diatoms. Biochimica et Biophysica Acta-Bioenergetics. 1817 (7), 1044-1052 (2012).
- Ahmad, R. A., Dietzel, L. Relaxation of cellular K+ gradients by valinomycin induces diatoxanthin accumulation in Cyclotella meneghiniana cells and alters FCPa fluorescence yield in vitro. Physiologia Plantarum. , 171-180 (2017).
- Provasoli, L., McLaughlin, J. J. A., Droop, M. R. The development of artificial media for marine algae. Archiv für Mikrobiologie. 25 (4), 392-428 (1957).
- Jeffrey, S., Humphrey, G. New spectrophotometry equations for determining chlorophyll a, chlorophyll b, chlorophyll c-1 and chlorophyll c-2 in higher plants, algae and natural phytoplankton. Biochemie und Physiologie der Pflanzen. 167, 191-194 (1975).
- Beer, A., Gundermann, K., Beckmann, J., Büchel, C. Subunit Composition and Pigmentation of Fucoxanthin−Chlorophyll Proteins in Diatoms: Evidence for a Subunit Involved in Diadinoxanthin and Diatoxanthin Binding. Biochemistry. 45 (43), 13046-13053 (2006).
- Schägger, H., von Jagow, G. Tricine-sodium dodecyl sulfate-polyacrylamide gel electrophoresis for the separation of proteins in the range from 1 to 100 kDa. Analytical Biochemistry. 166 (2), 368-379 (1987).
- Büchel, C. Fucoxanthin-Chlorophyll Proteins in Diatoms: 18 and 19 kDa Subunits Assemble into Different Oligomeric States. Biochemistry. 42 (44), 13027-13034 (2003).
- Vieler, A., Wilhelm, C., Goss, R., Süß, R., Schiller, J. The lipid composition of the unicellular green alga Chlamydomonas reinhardtii and the diatom Cyclotella meneghiniana investigated by MALDI-TOF MS and TLC. Chemistry and Physics of Lipids. 150 (2), 143-155 (2007).
- Gundermann, K., Büchel, C. The fluorescence yield of the trimeric fucoxanthin-chlorophyll-protein FCPa in the diatom Cyclotella meneghiniana is dependent on the amount of bound diatoxanthin. Photosynthesis Research. 95 (2-3), 229-235 (2008).
- Miloslavina, Y., et al. Ultrafast fluorescence study on the location and mechanism of non-photochemical quenching in diatoms. Biochimica et Biophysica Acta-Bioenergetics. 1787 (10), 1189-1197 (2009).
- Grouneva, I., Jakob, T., Wilhelm, C., Goss, R. The regulation of xanthophyll cycle activity and of non-photochemical fluorescence quenching by two alternative electron flows in the diatoms Phaeodactylum tricornutum and Cyclotella meneghiniana. Biochimica et Biophysica Acta-Bioenergetics. 1787 (7), 929-938 (2009).
- Chukhutsina, V. U., Büchel, C., van Amerongen, H. Disentangling two non-photochemical quenching processes in Cyclotella meneghiniana by spectrally-resolved picosecond fluorescence at 77 K. Biochimica et Biophysica Acta-Bioenergetics. 1837 (6), 899-907 (2014).
- Ghazaryan, A., Akhtar, P., Garab, G., Lambrev, P. H., Büchel, C. Involvement of the Lhcx protein Fcp6 of the diatom Cyclotella meneghiniana in the macro-organisation and structural flexibility of thylakoid membranes. Biochimica Et Biophysica Acta-Bioenergetics. 1857 (9), 1373-1379 (2016).
- Darley, W. M. Biochemical composition. The biology of diatoms. 13, 198-223 (1977).
- Milsman, M. H. W., Schwendner, R. A., Weder, H. G. Preparation of large single bilayer liposomes by a fast and controlled dialysis. Biochimica Et Biophysica Acta. 512 (1), 147-155 (1978).
- Zumbuehl, O., Weder, H. G. Liposomes of controllable size in the range of 40 to 180 nm by defined dialysis of lipid-detergent-mixed micelles. Biochimica Et Biophysica Acta. 640 (1), 252-262 (1981).
- Verchere, A., Broutin, I., Picard, M. Photo-induced proton gradients for the in vitro investigation of bacterial efflux pumps. Scientific Reports. 2 (306), (2012).
- Chevallet, M., Luche, S., Rabilloud, T. Silver staining of proteins in polyacrylamide gels. Nature Protocols. 1 (4), 1852-1858 (2006).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon