Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Visualisation et quantification de la signalisation TGFβ/BMP/SMAD dans différentes conditions de contrainte de cisaillement du fluide à l’aide du test de proximité-ligature-assay
Dans cet article
Résumé
Ici, nous établissons un protocole pour visualiser et analyser simultanément plusieurs complexes SMAD en utilisant le test de ligature de proximité (PLA) dans les cellules endothéliales exposées à des conditions pathologiques et physiologiques de stress de cisaillement des fluides.
Résumé
La signalisation du facteur de croissance transformant β (TGFβ) / protéine morphogénétique osseuse (BMP) est étroitement régulée et équilibrée pendant le développement et l’homéostasie du système vasculaire Par conséquent, la dérégulation de cette voie de signalisation entraîne des pathologies vasculaires graves, telles que l’hypertension artérielle pulmonaire, la télangiectasie hémorragique héréditaire et l’athérosclérose. Les cellules endothéliales (CE), en tant que couche la plus interne des vaisseaux sanguins, sont constamment exposées au stress de cisaillement des fluides (SS). Il a été démontré que des schémas anormaux de SS fluide améliorent la signalisation TGFβ / BMP, qui, avec d’autres stimuli, induisent l’athérogenèse. En relation avec cela, l’athéroprorone, faible SS laminaire, a amélioré la signalisation TGFβ / BMP tandis que l’athéroprotecteur, SS laminaire élevé, diminue cette signalisation. Pour analyser efficacement l’activation de ces voies, nous avons conçu un flux de travail pour étudier la formation de complexes de facteurs de transcription dans des conditions SS à faible laminaire et à SS laminaire élevé à l’aide d’un système de pompe pneumatique disponible dans le commerce et d’un test de ligature de proximité (PLA).
La signalisation TGFβ/BMP active nécessite la formation de complexes SMAD trimériques constitués de deux SMAD régulateurs (R-SMAD); SMAD2/3 et SMAD1/5/8 pour la signalisation TGFβ et BMP, respectivement) avec un médiateur commun SMAD (co-SMAD; SMAD4). En utilisant le PLA ciblant différentes sous-unités du complexe SMAD trimérique, c’est-à-dire R-SMAD/co-SMAD ou R-SMAD/R-SMAD, la formation de complexes de facteurs de transcription SMAD actifs peut être mesurée quantitativement et spatialement à l’aide de la microscopie à fluorescence.
L’utilisation de glissières d’écoulement avec 6 petits canaux parallèles, qui peuvent être connectés en série, permet d’étudier la formation complexe du facteur de transcription et d’inclure les contrôles nécessaires.
Le flux de travail expliqué ici peut être facilement adapté pour des études ciblant la proximité des SMAD à d’autres facteurs de transcription ou à des complexes de facteurs de transcription autres que les SMAD, dans différentes conditions de SS fluides. Le flux de travail présenté ici montre un moyen rapide et efficace d’étudier la signalisation TGFβ/BMP induite par les fluides SS dans les CE, à la fois quantitativement et spatialement.
Introduction
Les protéines de la superfamille des facteurs de croissance transformants bêta (TGFβ) sont des cytokines pléiotropes avec une variété de membres, y compris TGFβs, protéines morphogénétiques osseuses (BMP) et Activins1,2. La liaison au ligand induit la formation d’oligomères récepteurs conduisant à la phosphorylation et, par conséquent, à l’activation du SMAD régulateur cytosolique (R-SMAD). Selon la sous-famille de ligands, différents R-SMAD sont activés1,2. Alors que les TGFβs et les Activines induisent principalement la phosphorylation de SMAD2/3, les BMP induisent une phosphorylation SMAD1/5/8. Cependant, il y a de plus en plus de preuves que les BMP et les TGFβs/Activins activent également les R-SMAD de l’autre sous-famille respective, dans un processus appelé « signalisation latérale»3,4,5,6,7,8 et qu’il existe des complexes SMAD mixtes composés à la fois de membres SMAD1/5 et SMAD2/333,9 . Deux R-SMAD activés forment par la suite des complexes trimériques avec le médiateur commun SMAD4. Ces complexes de facteurs de transcription sont alors capables de se transloquer dans le noyau et de réguler la transcription des gènes cibles. Les SMAD peuvent interagir avec une variété de co-activateurs et de co-répresseurs transcriptionnels différents, ce qui conduit à la diversification des possibilités de régulation des gènes cibles10. La déréglementation de la signalisation SMAD a de graves implications dans une variété de maladies. Dans cette optique, une signalisation TGFβ/BMP déséquilibrée peut entraîner des pathologies vasculaires graves, telles que l’hypertension artérielle pulmonaire, la télangiectasie hémorragique héréditaire ou l’athérosclérose3,11,12,13,14.
Les cellules endothéliales (CE) forment la couche la plus interne des vaisseaux sanguins et sont donc exposées au stress de cisaillement (SS), une force de friction exercée par le flux visqueux du sang. Fait intéressant, les CE résidant dans les parties du système vasculaire, qui sont exposées à des niveaux élevés de SS laminaires uniformes, sont maintenus dans un état homéostatique et au repos. En revanche, les CE qui présentent des SS faibles et non uniformes, par exemple aux bifurcations ou à la courbure moindre de l’arc aortique, sont prolifératives et activent les voies inflammatoires15. À leur tour, les sites d’EC dysfonctionnels sont enclins à développer l’athérosclérose. Fait intéressant, les CE dans ces zones d’athéroprone affichent des niveaux aberrants élevés de SMAD2/3 et SMAD1/516,17,18 activés. Dans ce contexte, l’amélioration de la signalisation TGFβ/BMP s’est avérée être un événement précoce dans le développement de lésions athérosclérotiques19 et l’interférence avec la signalisation BMP s’est avérée réduire considérablement l’inflammation vasculaire, la formation d’athérome et la calcification associée20.
Le test de ligature de proximité (PLA) est une technique biochimique permettant d’étudier les interactions protéine-protéine in situ21,22. Il repose sur la spécificité des anticorps de différentes espèces qui peuvent se lier aux protéines cibles d’intérêt, permettant une détection très spécifique des interactions protéiques endogènes au niveau d’une seule cellule. Ici, les anticorps primaires doivent se lier à leur épitope cible à une distance inférieure à 40 nm pour permettre la détection23. Par conséquent, le PLA est très bénéfique par rapport aux approches traditionnelles de co-immunoprécipitation, où plusieurs millions de cellules sont nécessaires pour détecter les interactions protéiques endogènes. Dans le PLA, les anticorps secondaires spécifiques à l’espèce, liés de manière covalente à des fragments d’ADN (appelés sondes Plus et Moins), se lient aux anticorps primaires et si les protéines d’intérêt interagissent, les sondes Plus et Moins sont proches. L’ADN est ligaturé à l’étape suivante et l’amplification en cercle roulant de l’ADN circulaire est rendue possible. Lors de l’amplification, des oligonucléotides complémentaires marqués par fluorescence se lient à l’ADN synthétisé, ce qui permet de visualiser ces interactions protéiques par microscopie à fluorescence conventionnelle.
Le protocole décrit ici permet aux scientifiques de comparer quantitativement le nombre de complexes de transcription SMAD actifs dans des conditions athéroprotectrices et athéroprones SS in vitro à l’aide de PLA. SS est généré via un système de pompe pneumatique programmable capable de générer un débit unidirectionnel laminaire de niveaux définis et permettant d’augmenter progressivement les débits. Cette méthode permet de détecter les interactions entre SMAD1/5 ou SMAD2/3 avec SMAD4, ainsi que les complexes mixtes R-SMAD. Il peut facilement être étendu pour analyser les interactions des SMAD avec les co-régulateurs transcriptionnels ou aux complexes de facteurs de transcription autres que les SMAD. La figure 1 montre les principales étapes du protocole présenté ci-dessous.
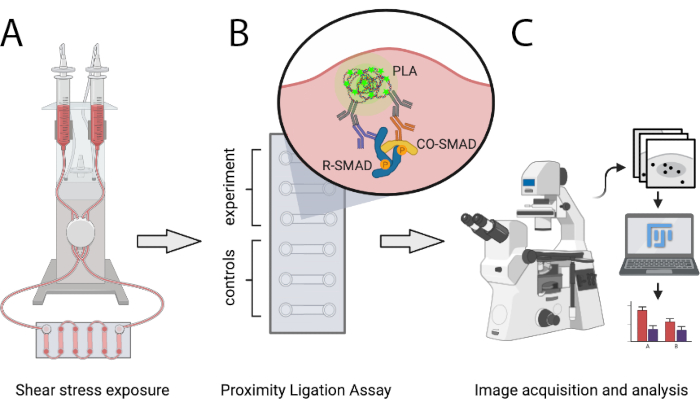
Figure 1 : Représentation schématique du protocole décrit. (A) Les cellules ensemencées dans des lames à 6 canaux sont exposées à une contrainte de cisaillement à l’aide d’un système de pompe pneumatique. (B) Les cellules fixes sont utilisées pour l’expérience du PLA ou pour les conditions de contrôle. (C) Les images des expériences PLA sont acquises avec un microscope à fluorescence et sont analysées à l’aide du logiciel d’analyse ImageJ. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
1. Exposition à la culture cellulaire et au stress de cisaillement des fluides
REMARQUE: Les CE de la veine ombilicale humaine (HUVEC) ont été utilisés comme exemple pour étudier l’interaction induite par la SS des SMAD. Le protocole décrit ci-dessous peut être appliqué à chaque type de cellule réactive SS.
- Enduire la lame à 6 canaux avec 0,1% de gélatine pour la peau porcine dans du PBS pendant 30 min à 37 °C.
- Ensemencez des HUVEC dans des lames pré-enduites à 6 canaux à une densité de 2,5 x 106 cellules par mL dans 30 μL de milieu complet M199.
REMARQUE : Pour plus d’informations sur la façon d’ensemencer des cellules dans la diapositive de flux, reportez-vous à la référence24. - Laisser les cellules adhérer pendant 1 h à 37 °C dans un incubateur humidifié.
- Ajouter 60 μL de milieu plein M199 préchauffé à chacun des réservoirs.
- Culture pendant 2 jours, avec un échange moyen doux une fois par jour, à 37 °C dans un incubateur humidifié.
- Aspirer complètement les réservoirs, ajouter 120 μL de milieu complet M199 préchauffé dans l’un des réservoirs et aspirer de l’autre côté.
- Ajouter 60 μL de milieu complet M199 préchauffé aux deux réservoirs.
- Assemblez et démarrez la configuration du flux comme indiqué dans la référence25.
- Monter des tubes sur des unités fluidiques. Ici, des tubes en silicone d’un diamètre de 0,8 mm et 1,6 mm sont utilisés pour appliquer une contrainte de cisaillement de 1 dyn/cm2 et 30 dyn/cm2, respectivement.
REMARQUE: Le matériau et la longueur du tube doivent rester constants, car les changements pourraient influencer la contrainte de cisaillement qui en résulte. En général, d’autres combinaisons de systèmes de pompe et de tubes peuvent être utilisées, à condition que la contrainte de cisaillement résultante soit connue et que la pompe crée un flux laminaire régulier. - Remplir les réservoirs avec une quantité appropriée de milieu plein M199 préchauffé (minimum 10 mL).
- Connectez les unités fluidiques avec le tube au système de pompe et effectuez un pré-fonctionnement sans cellules pour équilibrer le fluide et éliminer tout air restant25.
- Connectez en série les canaux uniques de la glissière à 6 canaux les uns aux autres à l’aide d’un tube de connexion série. Le premier et le dernier canal de la glissière seront connectés au tube assemblé au point 1.6.1 (voir la figure 1A pour un schéma). Veillez à ne pas introduire d’air dans le système, car cela pourrait nuire gravement aux cellules. Vous trouverez de plus amples informations sur la connexion série dans la référence26.
- Pour l’exposition des cellules à des niveaux élevés de contrainte de cisaillement (>20 dyn/cm2), utilisez une phase de rampe, c’est-à-dire augmentez la contrainte de cisaillement par étapes avec des phases d’adaptation. Les étapes peuvent être définies par incréments de 5 dyn/cm2 par 30 min.
- Monter des tubes sur des unités fluidiques. Ici, des tubes en silicone d’un diamètre de 0,8 mm et 1,6 mm sont utilisés pour appliquer une contrainte de cisaillement de 1 dyn/cm2 et 30 dyn/cm2, respectivement.
2. Fixation
- Détachez les glissières des pompes après l’exposition au fluide SS. Utilisez des pinces sur le tube lors du détachement, pour éviter le déversement de milieu dans l’incubateur.
- Le flux de transfert immédiat glisse sur la glace, tandis que le tube restant est détaché séquentiellement. Lors du retrait du tube des réservoirs, le réservoir de l’autre côté doit être maintenu fermé avec un doigt pour éviter de piéger les bulles d’air dans le canal. Cela peut interférer avec les étapes de fixation.
- En gardant les cellules sur la glace, aspirez soigneusement le milieu des réservoirs, mais pas du canal où résident les cellules. Par la suite, laver les échantillons avec du PBS stérile à froid (4 °C) avec trois fois le volume du canal (90 μL). Ajouter PBS dans un réservoir et aspirer soigneusement de l’autre réservoir. Répétez cette étape dans chacun des 6 canaux par diapositive.
REMARQUE: Pour toutes les étapes de lavage et d’incubation, la solution respective est ajoutée dans l’un des réservoirs, ce qui conduit à un échange de solutions dans le canal. Pour permettre la substitution complète des solutions dans le canal, la solution excédentaire est ensuite aspirée à partir de l’autre réservoir. La solution sur le dessus des cellules dans le canal n’est pas enlevée. Les cellules ne doivent pas sécher à tout moment. Par conséquent, il est important de laver soigneusement sans aucune insertion de bulle d’air dans les glissières. - Fixez les cellules en ajoutant 90 μL de solution tamponnée de PFA à 4% dans le même réservoir où le PBS a été ajouté au préalable et aspirez de la même manière le liquide de l’autre réservoir. Répétez cette étape dans chaque couche de chaque diapositive. Après l’ajout de la solution de PFA, transférer les échantillons de la glace à température ambiante (RT) et incuber pendant 20 min.
ATTENTION : Le PFA est toxique et doit être manipulé avec précaution. Utilisez des gants et travaillez sous une hotte aspirante. - Lavez les cellules 3x avec PBS (RT) en l’ajoutant dans un réservoir et en aspirant soigneusement de l’autre réservoir. Videz uniquement les réservoirs, en veillant à ne pas assécher le canal. Répétez cette étape pour chacune des 6 couches par diapositive.
- Éteindre la fixation du PFA en ajoutant 90 μL de chlorure d’ammonium ambiant de 50 mM dans le PBS dans l’un des réservoirs. Aspirer l’excès de solution de l’autre réservoir. Répétez l’opération pour chaque couche de la diapositive. Incuber les échantillons pendant 10 min à TA.
- Laver comme décrit à l’étape 2.5.
REMARQUE: À ce stade, les échantillons peuvent être conservés à 4 ° C pendant la nuit, ou le protocole peut être immédiatement poursuivi avec le blocage et l’incubation d’anticorps primaires (voir l’étape 3).
3. Blocage et incubation d’anticorps primaires
- Pour perméabiliser les cellules, ajouter 90 μL de Triton-X-100 à 0,3 % dans le PBS dans un réservoir vidé et aspirer de l’autre réservoir pour chaque canal. Incuber pendant 10 min à RT.
- Laver comme décrit à l’étape 2.5.
- Ajouter 90 μL de solution stérile bloquant le PLA dans un réservoir d’un canal et aspirer de l’autre côté. Répétez cette étape pour chaque canal. Bloquer pendant 1 h à 37 °C dans une chambre humidifiée.
- Pour faire une chambre humidifiée, utilisez un plat de 10 cm avec du tissu humide scellé avec un film de cire et placez le plat dans l’incubateur. Alternativement, d’autres formats de chambre d’humidité peuvent être utilisés qui fournissent une atmosphère humide.
REMARQUE: Alternativement, une solution de blocage auto-fabriquée peut être utilisée (par exemple, 3% (w / v) BSA dans PBS, filtré stérile).
- Pour faire une chambre humidifiée, utilisez un plat de 10 cm avec du tissu humide scellé avec un film de cire et placez le plat dans l’incubateur. Alternativement, d’autres formats de chambre d’humidité peuvent être utilisés qui fournissent une atmosphère humide.
- Préparer des anticorps primaires (1:100) dans un diluant d’anticorps PLA. Préparer 30 μL de la solution par canal. Ajouter les deux anticorps primaires simultanément et vortex.
REMARQUE: Alternativement, un diluant d’anticorps auto-fabriqué peut être utilisé (par exemple, 1% (p / v) BSA dans PBS). Les anticorps utilisés ici sont des combinaisons de SMAD1-SMAD2/3, SMAD2/3-SMAD4 et phospho-SMAD1/5-SMAD4. Des informations détaillées peuvent être trouvées dans la Table des matériaux. - Avant l’application d’anticorps primaires, aspirez la solution bloquante des réservoirs et, également, soigneusement du canal. Pipette 30 μL de la solution d’anticorps primaire immédiatement dans le canal vide en inclinant le canal tout en ajoutant la solution.
REMARQUE: Effectuez le retrait de la solution bloquante et l’ajout de la solution d’anticorps canal par canal pour vous assurer que les cellules ne sèchent pas entre les deux. - Incuber des échantillons avec les anticorps primaires pendant la nuit dans des chambres humidifiées à 4 °C.
REMARQUE: L’incubation peut également être effectuée pendant 1 h à température ambiante, si vous souhaitez poursuivre les étapes suivantes le même jour.
4. Incubation de sonde PLA
REMARQUE: Pour toutes les étapes de la section 4.1-7.3, les tampons de lavage A et B sont stockés à 4 °C et doivent être chauffés à RT avant l’utilisation.
- Diluer les sondes PLA (+)-souris et (-)-lapin à 1:5 dans une solution de diluant diluant d’anticorps PLA (ou 1% BSA). Préparer 30 μL par canal.
- Laver les échantillons 2x pendant 5 min en utilisant 90 μL du tampon de lavage A à RT en l’ajoutant dans l’un des réservoirs et en aspirant soigneusement de l’autre réservoir. Répétez cette étape pour chacune des 6 couches par diapositive.
- Aspirer soigneusement le tampon de lavage A et ajouter 30 μL de solution de sonde pla (préparée à l’étape 4.1), de la même manière que l’ajout d’anticorps primaires à l’étape 3.5.
- Incuber les échantillons pendant 1 h à 37 °C dans une chambre humidifiée.
5. Ligature
- Laver les échantillons 2x pendant 5 min en utilisant 90 μL du tampon de lavage A à RT, comme décrit au point 4.2.
- Préparer une dilution 1:5 du tampon de ligature dans de l’eau désionisée. Utilisez ce tampon pour diluer l’enzyme ligase à 1:40 (sur glace). Utilisez 30 μL par canal.
- Aspirer complètement le tampon de lavage A et ajouter la solution de ligature comme décrit au point 3.5.
- Incuber les échantillons pendant 30 min à 37 °C dans une chambre humidifiée.
6. Amplification
- Laver les échantillons 2x pendant 2 min en utilisant 90 μL de tampon de lavage A à RT, comme décrit au point 4.2.
- Préparez le tampon d’amplification en le diluant à 1:5 dans de l’eau désionisée et utilisez-le pour diluer l’enzyme polymérase à 1:80 (sur glace). Protéger de la lumière. Préparer 30 μL par canal.
- Aspirer le tampon de lavage A complètement et immédiatement ajouter la solution d’amplification préparée dans le canal vide, comme décrit au point 3.5. Incuber les échantillons pendant 100 min à 37 °C dans une chambre humidifiée.
7. Montage
- Laver les échantillons 2x pendant 10 min en utilisant 90 μL de tampon de lavage B à RT comme décrit au point 4.2. Ajouter le DAPI (1:500) à partir d’une solution mère de 1 mg/mL (dans de l’eau désionisée) dans le premier lavage pour tacher les noyaux. Ne séchez pas le canal.
- Diluer le tampon de lavage B dans de l’eau désionisée (1:10) et laver 1x avec 90 μL de solution tampon B 0,1x comme décrit au point 4.2.
- Aspirer complètement le tampon de lavage B et ajouter immédiatement 2-3 gouttes du milieu de montage liquide dans un réservoir. Distribuez-le dans le canal en inclinant la diapositive. Conserver les échantillons à 4 °C dans un environnement humidifié jusqu’à l’imagerie.
8. Acquisition d’images
- Acquérir des images à l’aide d’un microscope à fluorescence. Assurez-vous que les filtres respectifs équipant les sondes PLA fluorescentes sont disponibles.
REMARQUE: Il est avantageux d’utiliser un microscope confocal, si possible, car les taches de PLA obtenues sont plus définies. Cela prend également en charge le traitement d’images et l’analyse de données.
9. Analyse et quantification d’images à l’aide d’ImageJ/FIJI
- Traitez les images exportées (.tiff) avec un programme de traitement d’image, tel que ImageJ27.
REMARQUE: Tous les scripts utilisés dans cette étude et qui sont nécessaires pour le comptage automatique des événements cellulaires, nucléaires et de tous les événements PLA (par cellule) peuvent être trouvés dans un référentiel GitHub: https://github.com/Habacef/Proximity-Ligation-Assay-analysis. Effectuer une analyse statistique à l’aide de tout programme ou outil approprié.
Résultats
Nous avons déjà utilisé le PLA pour détecter les interactions de différentes protéines SMAD3 et analysé les changements induits par le stress de cisaillement dans la phosphorylation SMAD28.
Ici, les deux méthodes ont été combinées avec le protocole décrit ci-dessus. Les HUVEC ont été soumis à une contrainte de cisaillement de 1 dyn/cm2 et 30 dyn/cm2 et analysés pour les interactions des facteurs de transcript...
Discussion
Le protocole basé sur le PLA décrit ici offre un moyen efficace de déterminer la proximité de deux protéines (par exemple, leur interaction directe) dans les CE exposés à une contrainte de cisaillement avec une résolution quantitative et spatiale. En utilisant des lames d’écoulement avec plusieurs canaux parallèles, plusieurs interactions protéiques différentes peuvent être examinées en même temps dans des cellules dans des conditions mécaniques identiques. En revanche, les systèmes de chambre d’éco...
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Nous remercions le Dr Maria Reichenbach et le Dr Christian Hiepen pour leur soutien sur le système de mise en place du flux et Eleanor Fox et Yunyun Xiao pour la lecture critique du manuscrit. P-L.M. a été financé par l’école internationale de recherche Max Planck IMPRS-Biology and Computation (IMPRS-BAC). PK a reçu un financement de la DFG-SFB1444. La figure 1 a été créée à l’aide de BioRender.
matériels
| Name | Company | Catalog Number | Comments |
| µ-Slide VI 0.4 | ibidi | 80606 | 6-channel slide |
| Ammonium Chloride | Carl Roth | K298.1 | Quenching |
| Bovine Serum Albumin | Carl Roth | 8076.4 | Blocking |
| DAPI | Sigma Aldrich/ Merck | D9542 | Stain DNA/Nuclei |
| DPBS | PAN Biotech | P04-53500 | PBS |
| Duolink In Situ Detection Reagents Green | Sigma Aldrich/ Merck | DUO92014 | PLA kit containing Ligase, ligation buffer, polymerase and amplification buffer (with green labeled oligonucleotides) |
| Duolink In Situ PLA Probe Anti-Mouse MINUS | Sigma Aldrich/ Merck | DUO92004 | MINUS probe |
| Duolink In Situ PLA Probe Anti-Rabbit PLUS | Sigma Aldrich/ Merck | DUO92002 | PLUS probe |
| Duolink In Situ Wash Buffers, Fluorescence | Sigma Aldrich/ Merck | DUO82049 | PLA wash buffers A and B |
| Endothelial Cell Growth Supplement | Corning | supplement for medium (ECGS) | |
| Fetal calf Serum | supplement for medium | ||
| FIJI | Image Analysis software | ||
| Formaldehyde solution 4% buffered | KLINIPATH/VWR | VWRK4186.BO1 | PFA |
| Full medium | M199 basal medium +20 % FCS +1 % P/S + 2 nM L-Glu + 25 µg/mL Hep + 50 µg/mL ECGS | ||
| Gelatin from porcine skin, Type A | Sigma Aldrich | G2500 | Use 0.1% in PBS for coating of flow channels |
| GraphPad Prism v.7 | GarphPad | Statistical Program used for the Plots and statistical calculations | |
| Heparin sodium salt from porcine intestinal mucosa | Sigma Aldrich | H4784-250MG | supplement for medium (Hep) |
| HUVECs | |||
| ibidi Mounting Medium | ibidi | 50001 | Liquid mounting medium |
| ibidi Pump System | ibidi | 10902 | pneumatic pump |
| Leica TCS SP8 | Leica | confocal microscope | |
| L-Glutamin 200mM | PAN Biotech | P04-80100 | supplement for medium (L-Glu) |
| Medium 199 | Sigma Aldrich | M2154 | Base medium |
| mouse anti- SMAD1 Antibody | Abcam | ab53745 | Suited for PLA |
| mouse anti- SMAD2/3 Antibody | BD Bioscience | 610843 | Not suited for PLA in combination with CST 9515 |
| mousee anti- SMAD4 Antibody | Sanata Cruz Biotechnology | sc-7966 | Suited for PLA |
| Penicillin 10.000U/ml /Streptomycin 10mg/ml | PAN Biotech | P06-07100 | supplement for medium (P/S) |
| Perfusion Set WHITE | ibidi | 10963 | Tubings used for 1 dyn/cm2 |
| Perfusion Set YELLOW and GREEN | ibidi | 10964 | Tubings used for 30 dyn/cm2 |
| rabbit anti- phospho SMAD1/5 Antibody | Cell Signaling Technologies | 9516 | Suited for PLA |
| rabbit anti- SMAD2/3 XP Antibody | Cell Signaling Technologies | 8685 | Suited for PLA |
| rabbit anti- SMAD4 Antibody | Cell Signaling Technologies | 9515 | Not suited for PLA in combination with BD 610843 |
| Serial Connector for µ-Slides | ibidi | 10830 | serial connection tubes |
| Triton X-100 | Carl Roth | 6683.1 | Permeabilization |
Références
- Yadin, D., Knaus, P., Mueller, T. D. Structural insights into BMP receptors: Specificity, activation and inhibition. Cytokine and Growth Factor Reviews. 27, 13-34 (2016).
- Sieber, C., Kopf, J., Hiepen, C., Knaus, P. Recent advances in BMP receptor signaling. Cytokine and Growth Factor Reviews. 20 (5-6), 343-355 (2009).
- Hiepen, C., et al. BMPR2 acts as a gatekeeper to protect endothelial cells from increased TGFβ responses and altered cell mechanics. PLoS Biology. 17 (12), 3000557 (2019).
- Hildebrandt, S., et al. ActivinA induced SMAD1/5 Signaling in an iPSC derived EC model of Fibrodysplasia Ossificans Progressiva (FOP) can be rescued by the drug candidate saracatinib. Stem Cell Reviews and Reports. , (2021).
- Goumans, M. J., et al. Balancing the activation state of the endothelium via two distinct TGF-beta type I receptors. The EMBO Journal. 21 (7), 1743-1753 (2002).
- Goumans, M. J., et al. Activin receptor-like kinase (ALK)1 is an antagonistic mediator of lateral TGFbeta/ALK5 signaling. Molecular Cell. 12 (4), 817-828 (2003).
- Daly, A. C., Randall, R. A., Hill, C. S. Transforming growth factor beta-induced Smad1/5 phosphorylation in epithelial cells is mediated by novel receptor complexes and is essential for anchorage-independent growth. Molecular and Cellular Biology. 28 (22), 6889-6902 (2008).
- Ramachandran, A., et al. TGF-β uses a novel mode of receptor activation to phosphorylate SMAD1/5 and induce epithelial-to-mesenchymal transition. eLife. 7, 31756 (2018).
- Flanders, K. C., et al. Brightfield proximity ligation assay reveals both canonical and mixed transforming growth factor-β/bone morphogenetic protein Smad signaling complexes in tissue sections. The Journal of Histochemistry and Cytochemistry : The Official Journal of The Histochemistry Society. 62 (12), 846-863 (2014).
- Miyazono, K., Maeda, S., Imamura, T., Dijke, P. T., Heldin, C. -. H. . Smad Signal Transduction: Smads in Proliferation, Differentiation and Disease. , 277-293 (2006).
- Goumans, M. J., Zwijsen, A., Ten Dijke, P., Bailly, S. Bone morphogenetic proteins in vascular homeostasis and disease. Cold Spring Harbor Perspectives in Biology. 10 (2), 031989 (2018).
- Cai, J., Pardali, E., Sánchez-Duffhues, G., ten Dijke, P. BMP signaling in vascular diseases. FEBS Letters. 586 (14), 1993-2002 (2012).
- Cunha, S. I., Magnusson, P. U., Dejana, E., Lampugnani, M. G. Deregulated TGF-β/BMP signaling in vascular malformations. Circulation research. 121 (8), 981-999 (2017).
- MacCarrick, G., et al. Loeys-Dietz syndrome: a primer for diagnosis and management. Genetics in Medicine : An Official Journal of the American College of Medical Genetics. 16 (8), 576-587 (2014).
- Baeyens, N., Bandyopadhyay, C., Coon, B. G., Yun, S., Schwartz, M. A. Endothelial fluid shear stress sensing in vascular health and disease. The Journal of Clinical Investigation. 126 (3), 821-828 (2016).
- Min, E., et al. Activation of Smad 2/3 signaling by low shear stress mediates artery inward remodeling. bioRxiv. , 691980 (2019).
- Zhou, J., et al. BMP receptor-integrin interaction mediates responses of vascular endothelial Smad1/5 and proliferation to disturbed flow. Journal of Thrombosis and Haemostasis. 11 (4), 741-755 (2013).
- Zhou, J., et al. Force-specific activation of Smad1/5 regulates vascular endothelial cell cycle progression in response to disturbed flow. Proceedings of the National Academy of Sciences of the United States of America. 109 (20), 7770-7775 (2012).
- van Dijk, R. A., et al. Visualizing TGF-β and BMP signaling in human atherosclerosis: A histological evaluation based on Smad activation. Histology and Histopathology. 27 (3), 387-396 (2012).
- Derwall, M., et al. Inhibition of bone morphogenetic protein signaling reduces vascular calcification and atherosclerosis. Arteriosclerosis, Thrombosis, and Vascular Biology. 32 (3), 613-622 (2012).
- Fredriksson, S., et al. Protein detection using proximity-dependent DNA ligation assays. Nature Biotechnology. 20 (5), 473-477 (2002).
- Söderberg, O., et al. Direct observation of individual endogenous protein complexes in situ by proximity ligation. Nature Methods. 3 (12), 995-1000 (2006).
- Alam, M. S. Proximity Ligation Assay (PLA). Current Protocols in Immunology. 123 (1), 58 (2018).
- Application Note 03: Growing Cells in µ-Channels. ibidi Available from: https://ibidi.com/img/cms/support/AN/AN03_Growing_cells.pdf (2012)
- Application Note 13: HUVECs under perfusion. ibidi Available from: https://ibidi.com/img/cms/support/AN/AN13_HUVECs_under_perfusion.pdf (2019)
- ibidi. Application Note 31: Instructions µ-Slide VI 0.4. ibidi. , (2013).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Reichenbach, M., et al. Differential impact of fluid shear stress and YAP/TAZ on BMP/TGF-β induced osteogenic target genes. Advanced Biology. 5 (2), 2000051 (2021).
- Hiepen, C., Mendez, P. L., Knaus, P. It takes two to tango: Endothelial TGFβ/BMP signaling crosstalk with mechanobiology. Cells. 9 (9), 1965 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon