È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Visualizzazione e quantificazione della segnalazione TGFβ/BMP/SMAD in diverse condizioni di sforzo di taglio del fluido utilizzando il test di prossimità-legatura
In questo articolo
Riepilogo
Qui, stabiliamo un protocollo per visualizzare e analizzare simultaneamente più complessi SMAD utilizzando il test di legatura di prossimità (PLA) in cellule endoteliali esposte a condizioni patologiche e fisiologiche di stress da taglio del fluido.
Abstract
La segnalazione del fattore di crescita trasformante β (TGFβ) / proteina morfogenetica ossea (BMP) è strettamente regolata ed equilibrata durante lo sviluppo e l'omeostasi del sistema vascolare Pertanto, la deregolazione in questa via di segnalazione provoca gravi patologie vascolari, come l'ipertensione dell'arteria polmonare, la teleangectasia emorragica ereditaria e l'aterosclerosi. Le cellule endoteliali (CE), come strato più interno dei vasi sanguigni, sono costantemente esposte allo stress da taglio fluido (SS). Modelli anormali di SS fluida hanno dimostrato di migliorare la segnalazione TGFβ / BMP, che, insieme ad altri stimoli, induce l'aterogenesi. In relazione a questo, l'ateroprone, SS a basso laminare è stato trovato per migliorare la segnalazione TGFβ / BMP mentre l'ateroprotettivo, SS laminare elevato, diminuisce questa segnalazione. Per analizzare in modo efficiente l'attivazione di questi percorsi, abbiamo progettato un flusso di lavoro per studiare la formazione di complessi di fattori di trascrizione in condizioni di SS a basso laminare e SS laminare elevato utilizzando un sistema di pompe pneumatiche disponibile in commercio e un test di legatura di prossimità (PLA).
La segnalazione attiva TGFβ/BMP richiede la formazione di complessi trimerici SMAD costituiti da due SMED regolatori (R-SMAD); SMAD2/3 e SMAD1/5/8 per la segnalazione TGFβ e BMP, rispettivamente) con un mediatore comune SMAD (co-SMAD; SMAD4). Utilizzando il PLA mirato a diverse subunità del complesso trimerico SMAD, cioè R-SMAD / co-SMAD o R-SMAD / R-SMAD, la formazione di complessi di fattori di trascrizione SMAD attivi può essere misurata quantitativamente e spazialmente utilizzando la microscopia a fluorescenza.
L'utilizzo di guide di flusso con 6 piccoli canali paralleli, collegabili in serie, consente di studiare la formazione complessa del fattore di trascrizione e di inserire i necessari controlli.
Il flusso di lavoro qui spiegato può essere facilmente adattato per studi mirati alla vicinanza degli SMED ad altri fattori di trascrizione o a complessi di fattori di trascrizione diversi dagli SMAD, in diverse condizioni di SS fluida. Il flusso di lavoro qui presentato mostra un modo rapido ed efficace per studiare la segnalazione TGFβ/BMP indotta da SS fluidi nelle CE, sia quantitativamente che spazialmente.
Introduzione
Le proteine della superfamiglia dei fattori di crescita trasformanti beta (TGFβ) sono citochine pleiotropiche con una varietà di membri, tra cui TGFβs, proteine morfogenetiche ossee (BMP) e Activins1,2. Il legame del ligando induce la formazione di oligomeri recettoriali che portano alla fosforilazione e, quindi, all'attivazione dello SMAD citosolico regolatorio (R-SMAD). A seconda della sottofamiglia di ligandi, vengono attivati diversi R-SED1,2. Mentre TGFβs e Activins inducono principalmente fosforilazione di SMAD2/3, BMP inducono fosforilazione SMAD1/5/8. Tuttavia, ci sono prove crescenti che BMP e TGFβs / Activins attivano anche R-SMAD della rispettiva altra sottofamiglia, in un processo definito come "segnalazione laterale" 3,4,5,6,7,8 e che esistono complessi SMAD misti costituiti da entrambi, SMAD1/5 e SMAD2/3, members3,9 . Due R-SMAD attivati formano successivamente complessi trimerici con il mediatore comune SMAD4. Questi complessi di fattori di trascrizione sono quindi in grado di traslocare nel nucleo e regolare la trascrizione dei geni bersaglio. Gli SED possono interagire con una varietà di diversi co-attivatori e co-repressori trascrizionali, portando alla diversificazione delle possibilità di regolare i geni bersaglio10. La deregolamentazione della segnalazione SMAD ha gravi implicazioni in una varietà di malattie. In linea con questo, la segnalazione Squilibrata di TGFβ/BMP può portare a gravi patologie vascolari, come ipertensione dell'arteria polmonare, teleangectasia emorragica ereditaria o aterosclerosi3,11,12,13,14.
Le cellule endoteliali (CE) formano lo strato più interno dei vasi sanguigni e sono, quindi, esposte allo stress da taglio (SS), una forza di attrito esercitata dal flusso viscoso del sangue. È interessante notare che le CE che risiedono nelle parti della vascolarizzazione, che sono esposte ad alti livelli di SS uniforme e laminare, sono mantenute in uno stato omeostatico e quiescente. Al contrario, le CE che sperimentano SS basse e non uniformi, ad esempio a biforcazioni o la minore curvatura dell'arco aortico, sono proliferative e attivano le vie infiammatorie15. A loro volta, i siti di EC disfunzionali sono inclini a sviluppare aterosclerosi. È interessante notare che gli EC in queste aree di ateropatrone mostrano livelli aberrantemente elevati di SMAD2/3 e SMAD1/516,17,18 attivati. In questo contesto, la segnalazione TGFβ/BMP potenziata è risultata essere un evento precoce nello sviluppo di lesioni aterosclerotiche19 e l'interferenza con la segnalazione BMP è risultata ridurre notevolmente l'infiammazione vascolare, la formazione di ateroma e la calcificazione associata20.
Proximity Ligation Assay (PLA) è una tecnica biochimica per studiare le interazioni proteina-proteina in situ21,22. Si basa sulla specificità di anticorpi di diverse specie che possono legare proteine bersaglio di interesse, consentendo il rilevamento altamente specifico di interazioni proteiche endogene a livello di singola cellula. Qui, gli anticorpi primari devono legarsi al loro epitopo bersaglio a una distanza inferiore a 40 nm per consentire il rilevamento23. Pertanto, il PLA è molto utile rispetto ai tradizionali approcci di co-immunoprecipitazione, in cui sono necessari diversi milioni di cellule per rilevare le interazioni proteiche endogene. Nel PLA, gli anticorpi secondari specie-specifici, legati covalentemente ai frammenti di DNA (chiamati sonde Plus e Minus), legano gli anticorpi primari e se le proteine di interesse interagiscono, le sonde Plus e Minus si avvicinano. Il DNA viene legato nella fase successiva e l'amplificazione del cerchio rotolante del DNA circolare è resa possibile. Durante l'amplificazione, oligonucleotidi complementari marcati fluorescenti si legano al DNA sintetizzato, consentendo a queste interazioni proteiche di essere visualizzate mediante microscopia a fluorescenza convenzionale.
Il protocollo qui descritto consente agli scienziati di confrontare quantitativamente il numero di complessi di trascrizione SMAD attivi in condizioni ateroprotettive e ateroprotroniche SS in vitro utilizzando PLA. SS è generato tramite un sistema di pompe pneumatiche programmabili che è in grado di generare flusso unidirezionale laminare di livelli definiti e consente aumenti graduali delle portate. Questo metodo consente il rilevamento di interazioni tra SMAD1/5 o SMAD2/3 con SMAD4, nonché complessi misti-R-SMAD. Può essere facilmente espanso per analizzare le interazioni degli SMAD con i co-regolatori trascrizionali o con complessi di fattori di trascrizione diversi dagli SMAD. La Figura 1 mostra i passaggi principali del protocollo presentati di seguito.
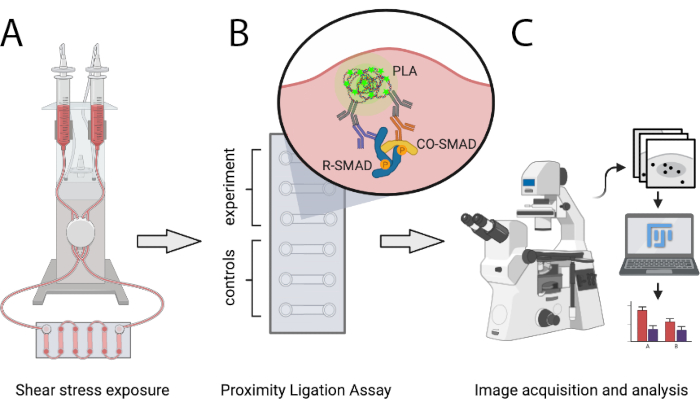
Figura 1: Rappresentazione schematica del protocollo descritto. (A) Le celle seminate in vetrini a 6 canali sono esposte a sollecitazioni di taglio con un sistema di pompe pneumatiche. (B) Le celle fisse sono utilizzate per esperimenti PLA o per condizioni di controllo. (C) Le immagini degli esperimenti PLA vengono acquisite con un microscopio a fluorescenza e vengono analizzate utilizzando il software di analisi ImageJ. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocollo
1. Coltura cellulare ed esposizione allo stress da taglio dei fluidi
NOTA: le EC delle vene ombelicali umane (HUVEC) sono state utilizzate come esempio per studiare l'interazione indotta da SS di SMED. Il protocollo descritto di seguito può essere applicato a ogni tipo di cella reattiva SS.
- Rivestire il vetrino a 6 canali con gelatina cutanea suina allo 0,1% in PBS per 30 minuti a 37 °C.
- HUVEC di semi in vetrini preverniciati a 6 canali ad una densità di 2,5 x 106 celle per mL in 30 μL di M199 full medium.
NOTA: per ulteriori informazioni su come eseguire il seeding delle celle nella diapositiva di flusso, vedere reference24. - Lasciare che le cellule aderiscano per 1 ora a 37 °C in un incubatore umidificato.
- Aggiungere 60 μL di M199 full medium preriscaldato a ciascuno dei serbatoi.
- Coltura per 2 giorni, con un delicato scambio medio una volta al giorno, a 37 °C in un incubatore umidificato.
- Aspirare completamente i serbatoi, aggiungere 120 μL di mezzo completo M199 preriscaldato in uno dei serbatoi e aspirare dall'altro lato.
- Aggiungere 60 μL di M199 full medium preriscaldato a entrambi i serbatoi.
- Assemblate e avviate la configurazione del flusso come descritto nel riferimento25.
- Montare tubi su unità fluidiche. Qui, i tubi in silicone con un diametro di 0,8 mm e 1,6 mm vengono utilizzati per applicare sollecitazioni di taglio rispettivamente di 1 dyn / cm2 e 30 dyn / cm2.
NOTA: il materiale e la lunghezza del tubo devono rimanere costanti, poiché le modifiche potrebbero influenzare la tensione di taglio risultante. In generale, è possibile utilizzare altre combinazioni di sistemi di pompaggio e tubi, purché lo stress di taglio risultante sia noto e la pompa crei un flusso laminare costante. - Riempire i serbatoi con una quantità appropriata di M199 full medium preriscaldato (minimo 10 ml).
- Collegare le unità fluidiche con il tubo al sistema di pompaggio ed eseguire una pre-corsa senza celle per equilibrare il mezzo e rimuovere l'eventuale aria rimanente25.
- Collegare in serie i singoli canali sulla diapositiva a 6 canali l'uno all'altro utilizzando tubi di connessione seriale. Il primo e l'ultimo canale della slitta saranno collegati al tubo assemblato in 1.6.1 (vedere la Figura 1A per uno schema). Fare attenzione a non introdurre aria nel sistema in quanto ciò potrebbe danneggiare gravemente le cellule. Ulteriori informazioni sulla connessione seriale sono disponibili in reference26.
- Per l'esposizione delle cellule ad alti livelli di sforzo di taglio (>20 dyn/cm2), utilizzare una fase di rampa, ovvero aumentare lo sforzo di taglio gradualmente con fasi di adattamento. I passi possono essere impostati con incrementi di 5 dyn/cm2 per 30 min.
- Montare tubi su unità fluidiche. Qui, i tubi in silicone con un diametro di 0,8 mm e 1,6 mm vengono utilizzati per applicare sollecitazioni di taglio rispettivamente di 1 dyn / cm2 e 30 dyn / cm2.
2. Fissazione
- Staccare le diapositive dalle pompe dopo l'esposizione al fluido SS. Utilizzare morsetti sul tubo durante il distacco, per evitare la fuoriuscita media nell'incubatore.
- Il flusso di trasferimento istantaneo scorre sul ghiaccio, mentre il tubo rimanente viene staccato sequenzialmente. Quando si rimuove il tubo dai serbatoi, il serbatoio sull'altro lato deve essere tenuto chiuso con un dito per evitare di intrappolare bolle d'aria nel canale. Ciò potrebbe interferire con i passaggi di fissazione.
- Mantenendo le cellule sul ghiaccio, aspirare accuratamente il mezzo dai serbatoi ma non dal canale in cui risiedono le cellule. Successivamente, lavare i campioni con PBS sterile freddo (4 °C) con tre volte il volume del canale (90 μL). Aggiungere PBS in un serbatoio e aspirare con attenzione dall'altro serbatoio. Ripetere questo passaggio in ciascuno dei 6 canali per diapositiva.
NOTA: Per tutte le fasi di lavaggio e incubazione la rispettiva soluzione viene aggiunta in uno dei serbatoi che porta ad uno scambio di soluzioni nel canale. Per consentire la sostituzione completa delle soluzioni nel canale, la soluzione in eccesso viene quindi aspirata dall'altro serbatoio. La soluzione sulla parte superiore delle celle nel canale non viene rimossa. Le cellule non dovrebbero asciugarsi in nessun momento. Pertanto, è importante lavare accuratamente senza alcun inserimento di bolle d'aria nelle diapositive. - Fissare le celle aggiungendo 90 μL di soluzione tamponata al 4% di PFA nello stesso serbatoio in cui è stato aggiunto il PBS in anticipo e allo stesso modo aspirare il liquido dall'altro serbatoio. Ripetere questo passaggio in ogni canale di ogni diapositiva. Dopo l'aggiunta della soluzione di PFA, trasferire i campioni dal ghiaccio alla temperatura ambiente (RT) e incubare per 20 minuti.
ATTENZIONE: il PFA è tossico e deve essere maneggiato con attenzione. Usa i guanti e lavora sotto un cappuccio di fumo. - Lavare le celle 3x con PBS (RT) aggiungendolo in un serbatoio e aspirando con attenzione dall'altro serbatoio. Svuotare solo i serbatoi, assicurandosi di non asciugare il canale. Ripetere questo passaggio per ciascuno dei 6 canali per diapositiva.
- Estinguere la fissazione del PFA aggiungendo 90 μL di cloruro di ammonio ambiente da 50 mM in PBS in uno dei serbatoi. Aspirare la soluzione in eccesso dall'altro serbatoio. Ripetere l'operazione per ogni canale della diapositiva. Incubare i campioni per 10 minuti a RT.
- Lavare come descritto nel passaggio 2.5.
NOTA: A questo punto, i campioni possono essere conservati a 4 °C durante la notte, oppure il protocollo può essere immediatamente continuato con il blocco e l'incubazione di anticorpi primari (vedere fase 3).
3. Blocco e incubazione di anticorpi primari
- Per permeabilizzare le celle, aggiungere 90 μL di Triton-X-100 allo 0,3% in PBS in un serbatoio svuotato e aspirare dall'altro serbatoio per ciascun canale. Incubare per 10 minuti a RT.
- Lavare come descritto nel passaggio 2.5.
- Aggiungere 90 μL di soluzione bloccante pla sterile in un serbatoio di un canale e aspirare dall'altro lato. Ripetere questo passaggio per ogni canale. Blocco per 1 ora a 37 °C in camera umidificata.
- Per realizzare una camera umidificata, utilizzare un piatto di 10 cm con tessuto bagnato sigillato con pellicola di cera e posizionare il piatto nell'incubatrice. In alternativa, è possibile utilizzare altri formati di camera di umidità che forniscono un'atmosfera umida.
NOTA: In alternativa, è possibile utilizzare una soluzione di blocco autoprodotta (ad esempio, 3% (p / v) BSA in PBS, filtrato sterile).
- Per realizzare una camera umidificata, utilizzare un piatto di 10 cm con tessuto bagnato sigillato con pellicola di cera e posizionare il piatto nell'incubatrice. In alternativa, è possibile utilizzare altri formati di camera di umidità che forniscono un'atmosfera umida.
- Preparare gli anticorpi primari (1:100) nel diluente anticorpale PLA. Preparare 30 μL della soluzione per canale. Aggiungere entrambi gli anticorpi primari contemporaneamente e vortice.
NOTA: In alternativa, è possibile utilizzare un diluente anticorpale autoprodotto (ad esempio, 1% (p/v) BSA in PBS). Gli anticorpi utilizzati qui sono combinazioni di SMAD1-SMAD2/3, SMAD2/3-SMAD4 e fosfo-SMAD1/5-SMAD4. Informazioni dettagliate sono disponibili nella Tabella dei materiali. - Prima dell'applicazione di anticorpi primari, aspirare la soluzione bloccante dai serbatoi e, anche, con attenzione dal canale. Pipettare 30 μL della soluzione anticorpale primaria immediatamente nel canale vuoto inclinando il canale mentre si aggiunge la soluzione.
NOTA: Eseguire la rimozione della soluzione bloccante e l'aggiunta della soluzione anticorpale canale per canale per garantire che le cellule non si secchino nel mezzo. - Incubare campioni con gli anticorpi primari durante la notte in camere umidificate a 4 °C.
NOTA: L'incubazione può essere eseguita anche per 1 ora a temperatura ambiente, se interessati a continuare con i seguenti passaggi nello stesso giorno.
4. Incubazione della sonda PLA
NOTA: per tutte le fasi di cui al punto 4.1-7.3, i tamponi di lavaggio A e B sono conservati a 4 °C e devono essere riscaldati a RT prima dell'uso.
- Diluire le sonde PLA (+)-mouse e (-)-coniglio a 1:5 in soluzione di diluente anticorpale PLA (o BSA all'1%). Preparare 30 μL per canale.
- Lavare i campioni 2 volte per 5 minuti utilizzando 90 μL del tampone di lavaggio A a RT aggiungendolo in uno dei serbatoi e aspirando attentamente dall'altro serbatoio. Ripetere questo passaggio per ciascuno dei 6 canali per diapositiva.
- Aspirare accuratamente il tampone di lavaggio A e aggiungere 30 μL di soluzione di sonda PLA (preparata nella fase 4.1), simile all'aggiunta di anticorpi primari nella fase 3.5.
- Incubare i campioni per 1 ora a 37 °C in una camera umidificata.
5. Legatura
- Lavare i campioni 2 volte per 5 minuti utilizzando 90 μL del tampone di lavaggio A a RT, come descritto al punto 4.2.
- Preparare una diluizione 1:5 del tampone di legatura in acqua deionizzata. Utilizzare questo tampone per diluire l'enzima ligasi a 1:40 (su ghiaccio). Utilizzare 30 μL per canale.
- Aspirare completamente il tampone di lavaggio A e aggiungere la soluzione di legatura come descritto al punto 3.5.
- Incubare i campioni per 30 minuti a 37 °C in una camera umidificata.
6. Amplificazione
- Lavare i campioni 2 volte per 2 minuti utilizzando 90 μL di tampone di lavaggio A a RT, come descritto al punto 4.2.
- Preparare il tampone di amplificazione diluendolo 1:5 in acqua deionizzata e utilizzarlo per diluire l'enzima polimerasi a 1:80 (su ghiaccio). Proteggere dalla luce. Preparare 30 μL per canale.
- Aspirare completamente il tampone di lavaggio A e aggiungere immediatamente la soluzione di amplificazione preparata nel canale vuoto, come descritto al punto 3.5. Incubare i campioni per 100 minuti a 37 °C in una camera umidificata.
7. Montaggio
- Lavare i campioni 2 volte per 10 minuti utilizzando 90 μL di wash buffer B a RT come descritto al punto 4.2. Aggiungere DAPI (1:500) da 1 mg/mL di soluzione madre (in acqua deionizzata) al primo lavaggio per colorare i nuclei. Non asciugare il canale.
- Diluire il tampone di lavaggio B in acqua deionizzata (1:10) e lavare 1x con 90 μL di soluzione tampone B 0,1x come descritto al punto 4.2.
- Aspirare completamente il tampone di lavaggio B e aggiungere immediatamente 2-3 gocce del mezzo di montaggio del liquido in un serbatoio. Distribuiscilo nel canale inclinando la diapositiva. Conservare i campioni a 4 °C in un ambiente umidificato fino all'imaging.
8. Acquisizione di immagini
- Acquisire immagini utilizzando un microscopio a fluorescenza. Assicurarsi che i rispettivi filtri che si adattano alle sonde fluorescenti pla siano disponibili.
NOTA: È utile utilizzare un microscopio confocale, se possibile, poiché le macchie di PLA ottenute sono più definite. Ciò supporta anche un'ulteriore elaborazione delle immagini e l'analisi dei dati.
9. Analisi e quantificazione delle immagini utilizzando ImageJ/FIJI
- Elaborare le immagini esportate (.tiff) con un programma di elaborazione delle immagini, ad esempio ImageJ27.
NOTA: tutti gli script utilizzati in questo studio e necessari per il conteggio automatico di eventi cellulari, nucleari e tutti gli eventi PLA (per cella) sono disponibili in un repository GitHub: https://github.com/Habacef/Proximity-Ligation-Assay-analysis. Eseguire analisi statistiche utilizzando qualsiasi programma o strumento adatto.
Risultati
In precedenza abbiamo utilizzato il PLA per rilevare le interazioni di diverse proteine SMAD3 e analizzato i cambiamenti indotti dallo stress di taglio nella fosforilazione SMAD28.
Qui, entrambi i metodi sono stati combinati con il protocollo sopra descritto. Gli HUVEC sono stati sottoposti a stress di taglio di 1 dyn/cm2 e 30 dyn/cm2 e analizzati per le interazioni dei fattori di trascrizione SMAD. Mostriamo che, rispetto a...
Discussione
Il protocollo basato sul PLA qui descritto offre un modo efficiente per determinare la stretta vicinanza di due proteine (ad esempio, la loro interazione diretta) nelle CE esposte allo stress di taglio con risoluzione quantitativa e spaziale. Utilizzando diapositive di flusso con più canali paralleli, diverse interazioni proteiche possono essere esaminate contemporaneamente in cellule in condizioni meccaniche identiche. Al contrario, i sistemi di camere di flusso costruiti su misura spesso utilizzano un singolo canale c...
Divulgazioni
Gli autori non dichiarano alcun conflitto di interessi.
Riconoscimenti
Ringraziamo la dott.ssa Maria Reichenbach e il dott. Christian Hiepen per il loro supporto sul sistema di impostazione del flusso e Eleanor Fox e Yunyun Xiao per aver letto criticamente il manoscritto. P-L.M. è stato finanziato dalla Scuola di Ricerca internazionale Max Planck IMPRS-Biology and Computation (IMPRS-BAC). PK ha ricevuto finanziamenti dal DFG-SFB1444. La Figura 1 è stata creata utilizzando BioRender.
Materiali
| Name | Company | Catalog Number | Comments |
| µ-Slide VI 0.4 | ibidi | 80606 | 6-channel slide |
| Ammonium Chloride | Carl Roth | K298.1 | Quenching |
| Bovine Serum Albumin | Carl Roth | 8076.4 | Blocking |
| DAPI | Sigma Aldrich/ Merck | D9542 | Stain DNA/Nuclei |
| DPBS | PAN Biotech | P04-53500 | PBS |
| Duolink In Situ Detection Reagents Green | Sigma Aldrich/ Merck | DUO92014 | PLA kit containing Ligase, ligation buffer, polymerase and amplification buffer (with green labeled oligonucleotides) |
| Duolink In Situ PLA Probe Anti-Mouse MINUS | Sigma Aldrich/ Merck | DUO92004 | MINUS probe |
| Duolink In Situ PLA Probe Anti-Rabbit PLUS | Sigma Aldrich/ Merck | DUO92002 | PLUS probe |
| Duolink In Situ Wash Buffers, Fluorescence | Sigma Aldrich/ Merck | DUO82049 | PLA wash buffers A and B |
| Endothelial Cell Growth Supplement | Corning | supplement for medium (ECGS) | |
| Fetal calf Serum | supplement for medium | ||
| FIJI | Image Analysis software | ||
| Formaldehyde solution 4% buffered | KLINIPATH/VWR | VWRK4186.BO1 | PFA |
| Full medium | M199 basal medium +20 % FCS +1 % P/S + 2 nM L-Glu + 25 µg/mL Hep + 50 µg/mL ECGS | ||
| Gelatin from porcine skin, Type A | Sigma Aldrich | G2500 | Use 0.1% in PBS for coating of flow channels |
| GraphPad Prism v.7 | GarphPad | Statistical Program used for the Plots and statistical calculations | |
| Heparin sodium salt from porcine intestinal mucosa | Sigma Aldrich | H4784-250MG | supplement for medium (Hep) |
| HUVECs | |||
| ibidi Mounting Medium | ibidi | 50001 | Liquid mounting medium |
| ibidi Pump System | ibidi | 10902 | pneumatic pump |
| Leica TCS SP8 | Leica | confocal microscope | |
| L-Glutamin 200mM | PAN Biotech | P04-80100 | supplement for medium (L-Glu) |
| Medium 199 | Sigma Aldrich | M2154 | Base medium |
| mouse anti- SMAD1 Antibody | Abcam | ab53745 | Suited for PLA |
| mouse anti- SMAD2/3 Antibody | BD Bioscience | 610843 | Not suited for PLA in combination with CST 9515 |
| mousee anti- SMAD4 Antibody | Sanata Cruz Biotechnology | sc-7966 | Suited for PLA |
| Penicillin 10.000U/ml /Streptomycin 10mg/ml | PAN Biotech | P06-07100 | supplement for medium (P/S) |
| Perfusion Set WHITE | ibidi | 10963 | Tubings used for 1 dyn/cm2 |
| Perfusion Set YELLOW and GREEN | ibidi | 10964 | Tubings used for 30 dyn/cm2 |
| rabbit anti- phospho SMAD1/5 Antibody | Cell Signaling Technologies | 9516 | Suited for PLA |
| rabbit anti- SMAD2/3 XP Antibody | Cell Signaling Technologies | 8685 | Suited for PLA |
| rabbit anti- SMAD4 Antibody | Cell Signaling Technologies | 9515 | Not suited for PLA in combination with BD 610843 |
| Serial Connector for µ-Slides | ibidi | 10830 | serial connection tubes |
| Triton X-100 | Carl Roth | 6683.1 | Permeabilization |
Riferimenti
- Yadin, D., Knaus, P., Mueller, T. D. Structural insights into BMP receptors: Specificity, activation and inhibition. Cytokine and Growth Factor Reviews. 27, 13-34 (2016).
- Sieber, C., Kopf, J., Hiepen, C., Knaus, P. Recent advances in BMP receptor signaling. Cytokine and Growth Factor Reviews. 20 (5-6), 343-355 (2009).
- Hiepen, C., et al. BMPR2 acts as a gatekeeper to protect endothelial cells from increased TGFβ responses and altered cell mechanics. PLoS Biology. 17 (12), 3000557 (2019).
- Hildebrandt, S., et al. ActivinA induced SMAD1/5 Signaling in an iPSC derived EC model of Fibrodysplasia Ossificans Progressiva (FOP) can be rescued by the drug candidate saracatinib. Stem Cell Reviews and Reports. , (2021).
- Goumans, M. J., et al. Balancing the activation state of the endothelium via two distinct TGF-beta type I receptors. The EMBO Journal. 21 (7), 1743-1753 (2002).
- Goumans, M. J., et al. Activin receptor-like kinase (ALK)1 is an antagonistic mediator of lateral TGFbeta/ALK5 signaling. Molecular Cell. 12 (4), 817-828 (2003).
- Daly, A. C., Randall, R. A., Hill, C. S. Transforming growth factor beta-induced Smad1/5 phosphorylation in epithelial cells is mediated by novel receptor complexes and is essential for anchorage-independent growth. Molecular and Cellular Biology. 28 (22), 6889-6902 (2008).
- Ramachandran, A., et al. TGF-β uses a novel mode of receptor activation to phosphorylate SMAD1/5 and induce epithelial-to-mesenchymal transition. eLife. 7, 31756 (2018).
- Flanders, K. C., et al. Brightfield proximity ligation assay reveals both canonical and mixed transforming growth factor-β/bone morphogenetic protein Smad signaling complexes in tissue sections. The Journal of Histochemistry and Cytochemistry : The Official Journal of The Histochemistry Society. 62 (12), 846-863 (2014).
- Miyazono, K., Maeda, S., Imamura, T., Dijke, P. T., Heldin, C. -. H. . Smad Signal Transduction: Smads in Proliferation, Differentiation and Disease. , 277-293 (2006).
- Goumans, M. J., Zwijsen, A., Ten Dijke, P., Bailly, S. Bone morphogenetic proteins in vascular homeostasis and disease. Cold Spring Harbor Perspectives in Biology. 10 (2), 031989 (2018).
- Cai, J., Pardali, E., Sánchez-Duffhues, G., ten Dijke, P. BMP signaling in vascular diseases. FEBS Letters. 586 (14), 1993-2002 (2012).
- Cunha, S. I., Magnusson, P. U., Dejana, E., Lampugnani, M. G. Deregulated TGF-β/BMP signaling in vascular malformations. Circulation research. 121 (8), 981-999 (2017).
- MacCarrick, G., et al. Loeys-Dietz syndrome: a primer for diagnosis and management. Genetics in Medicine : An Official Journal of the American College of Medical Genetics. 16 (8), 576-587 (2014).
- Baeyens, N., Bandyopadhyay, C., Coon, B. G., Yun, S., Schwartz, M. A. Endothelial fluid shear stress sensing in vascular health and disease. The Journal of Clinical Investigation. 126 (3), 821-828 (2016).
- Min, E., et al. Activation of Smad 2/3 signaling by low shear stress mediates artery inward remodeling. bioRxiv. , 691980 (2019).
- Zhou, J., et al. BMP receptor-integrin interaction mediates responses of vascular endothelial Smad1/5 and proliferation to disturbed flow. Journal of Thrombosis and Haemostasis. 11 (4), 741-755 (2013).
- Zhou, J., et al. Force-specific activation of Smad1/5 regulates vascular endothelial cell cycle progression in response to disturbed flow. Proceedings of the National Academy of Sciences of the United States of America. 109 (20), 7770-7775 (2012).
- van Dijk, R. A., et al. Visualizing TGF-β and BMP signaling in human atherosclerosis: A histological evaluation based on Smad activation. Histology and Histopathology. 27 (3), 387-396 (2012).
- Derwall, M., et al. Inhibition of bone morphogenetic protein signaling reduces vascular calcification and atherosclerosis. Arteriosclerosis, Thrombosis, and Vascular Biology. 32 (3), 613-622 (2012).
- Fredriksson, S., et al. Protein detection using proximity-dependent DNA ligation assays. Nature Biotechnology. 20 (5), 473-477 (2002).
- Söderberg, O., et al. Direct observation of individual endogenous protein complexes in situ by proximity ligation. Nature Methods. 3 (12), 995-1000 (2006).
- Alam, M. S. Proximity Ligation Assay (PLA). Current Protocols in Immunology. 123 (1), 58 (2018).
- Application Note 03: Growing Cells in µ-Channels. ibidi Available from: https://ibidi.com/img/cms/support/AN/AN03_Growing_cells.pdf (2012)
- Application Note 13: HUVECs under perfusion. ibidi Available from: https://ibidi.com/img/cms/support/AN/AN13_HUVECs_under_perfusion.pdf (2019)
- ibidi. Application Note 31: Instructions µ-Slide VI 0.4. ibidi. , (2013).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Reichenbach, M., et al. Differential impact of fluid shear stress and YAP/TAZ on BMP/TGF-β induced osteogenic target genes. Advanced Biology. 5 (2), 2000051 (2021).
- Hiepen, C., Mendez, P. L., Knaus, P. It takes two to tango: Endothelial TGFβ/BMP signaling crosstalk with mechanobiology. Cells. 9 (9), 1965 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon