É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Visualização e Quantificação de TGFβ/BMP/SMAD Sinalizando em diferentes condições de estresse da tesoura de fluidos usando o ensaio de proximidade-ligação-assay
Neste Artigo
Resumo
Aqui, estabelecemos um protocolo para visualizar e analisar simultaneamente múltiplos complexos de SMAD utilizando o ensaio de ligadura de proximidade (PLA) em células endoteliais expostas a condições patológicas e fisiológicas de estresse da tesoura de fluidos.
Resumo
Transformando fator de crescimento β (TGFβ)/Proteína Morfogenética Óssea (BMP) a sinalização é fortemente regulada e equilibrada durante o desenvolvimento e homeostase do sistema de vasculatura Portanto, a desregulamentação nesta via de sinalização resulta em patologias vasculares graves, como hipertensão arterial pulmonar, hemorrha pneumética hereditária e atherosclerosis. As células endoteliais (CE), como a camada mais interna dos vasos sanguíneos, estão constantemente expostas ao estresse da tesoura de fluidos (SS). Padrões anormais de SS fluido têm sido mostrados para melhorar a sinalização TGFβ/BMP, que, juntamente com outros estímulos, induzem aterogenese. Em relação a isso, atheroprone, baixo laminar SS foi encontrado para melhorar a sinalização TGFβ/BMP enquanto atheroprotetonte, alto laminar SS, diminui essa sinalização. Para analisar eficientemente a ativação dessas vias, projetamos um fluxo de trabalho para investigar a formação de complexos de fatores de transcrição sob ss de baixo laminar e condições de SS de alta laminar usando um sistema de bomba pneumática comercialmente disponível e ensaio de ligadura de proximidade (PLA).
A sinalização ativa de TGFβ/BMP requer a formação de complexos SMAD trimericos constituídos por dois SMADs regulatórios (R-SMAD); SMAD2/3 e SMAD1/5/8 para sinalização TGFβ e BMP, respectivamente) com um mediador comum SMAD (co-SMAD; SMAD4). Utilizando PLA visando diferentes subunidades do complexo trimerico SMAD, ou seja, R-SMAD/co-SMAD ou R-SMAD/R-SMAD, a formação de complexos ativos de fator de transcrição SMAD pode ser medida quantitativa e espacialmente usando microscopia de fluorescência.
O uso de slides de fluxo com 6 pequenos canais paralelos, que podem ser conectados em série, permite a investigação do fator de transcrição formação e inclusão dos controles necessários.
O fluxo de trabalho aqui explicado pode ser facilmente adaptado para estudos que visam a proximidade de SMADs a outros fatores de transcrição ou para complexos de fator de transcrição que não sejam SMADs, em diferentes condições de SS fluidos. O fluxo de trabalho aqui apresentado mostra uma maneira rápida e eficaz de estudar a sinalização TGFβ/BMP induzida pelo fluido em CEs, tanto quantitativa quanto espacialmente.
Introdução
Proteínas dos fatores transformadores de crescimento beta (TGFβ) superfamília são citocinas pleiotrópicos com uma variedade de membros, incluindo TGFβs, proteínas morfogenéticas ósseas (BMPs) e Activins1,2. A ligação ligante induz a formação de oligômeros receptores que levam à fosforilação e, assim, ativação do SMAD regulatório citomúmico (R-SMAD). Dependendo da subfamo de ligantes, diferentes R-SMADs são ativados1,2. Enquanto TGFβs e Activins induzem principalmente fosforilação de SMAD2/3, os BMPs induzem a fosforilação SMAD1/5/8. No entanto, há evidências acumuladas de que os BMPs e TGFβs/Activins também ativam R-SMADs da respectiva outra subfamo, em um processo denominado como 'sinalização lateral'3,4,5,6,7,8 e que existem complexos SMAD mistos compostos por ambos, SMAD1/5 e SMAD2/3, membros3,9 . Dois R-SMADs ativados formam posteriormente complexos triméricos com o mediador comum SMAD4. Esses complexos de fator de transcrição são então capazes de translocar para o núcleo e regular a transcrição dos genes-alvo. Os SMADs podem interagir com uma variedade de diferentes co-ativadores e co-repressores transcricionais, levando à diversificação das possibilidades de regular genes-alvo10. A desregulamentação da sinalização SMAD tem sérias implicações em uma variedade de doenças. De acordo com isso, a sinalização TGFβ/BMP desequilibrada pode levar a patologias vasculares graves, como hipertensão arterial pulmonar, telangiectasia hemorrágica hereditária ou aterosclerose3,11,12,13,14.
As células endoteliais (CE) formam a camada mais interna dos vasos sanguíneos e são, portanto, expostas ao estresse da cisalhamento (SS), uma força de atrito exercida pelo fluxo viscoso do sangue. Curiosamente, os CEs residentes nas partes da vasculatura, que são expostos a altos níveis de uniforme, laminar SS, são mantidos em um estado homeostático e quiescente. Em contraste, as CES que experimentam SS baixas e não uniformes, por exemplo, em bifurcações ou na menor curvatura do arco aórtico, são proliferativas e ativam vias inflamatórias15. Por sua vez, os locais de ECs disfuncionais são propensos a desenvolver aterosclerose. Curiosamente, os CES nessas áreas de atheroprone exibem níveis aberrantemente altos de SMAD2/3 ativados e SMAD1/516,17,18. Nesse contexto, verificou-se que a sinalização TGFβ/BMP aprimorada foi um evento inicial no desenvolvimento de lesões ateroscleróticas19 e a interferência na sinalização de BMP foi encontrada para reduzir significativamente a inflamação vascular, a formação de atheroma e calcificação associada20.
O Ensaio de Ligadura de Proximidade (PLA) é uma técnica bioquímica para estudar interações proteína-proteína em situ21,22. Ele se baseia na especificidade de anticorpos de diferentes espécies que podem ligar proteínas-alvo de interesse, permitindo detecção altamente específica de interações proteicas endógenas a um nível unicelular. Aqui, os anticorpos primários têm que se ligar ao seu epítope alvo a uma distância inferior a 40 nm para permitir a detecção23. Portanto, o PLA é muito benéfico em relação às abordagens tradicionais de co-imunoprecipitação, onde vários milhões de células são necessárias para detectar interações proteicas endógenas. No PLA, anticorpos secundários específicos da espécie, covalentemente ligados a fragmentos de DNA (denominados sondas Plus e Minus), ligam os anticorpos primários e se as proteínas de interesse interagirem, as sondas Plus e Minus se aproximam. O DNA é ligado na etapa seguinte e a amplificação do círculo de rolamento do DNA circular é possível. Durante a amplificação, oligonucleotídeos complementares rotulados fluorescente se ligam ao DNA sintetizado, permitindo que essas interações proteicas sejam visualizadas pela microscopia de fluorescência convencional.
O protocolo descrito aqui permite que os cientistas comparem quantitativamente o número de complexos ativos de transcrição SMAD em condições de SS atheroprotetor e atheroprone in vitro usando PLA. O SS é gerado através de um sistema de bomba pneumática programável que é capaz de gerar fluxo direcional laminar de níveis definidos e permite aumentos stepwise das taxas de fluxo. Este método permite a detecção de interações entre SMAD1/5 ou SMAD2/3 com SMAD4, bem como complexos mistos-R-SMAD. Ele pode ser facilmente expandido para analisar interações de SMADs com co-reguladores transcricionais ou para complexos de fatores de transcrição que não sejam SMADs. A Figura 1 mostra os principais passos do protocolo apresentado abaixo.
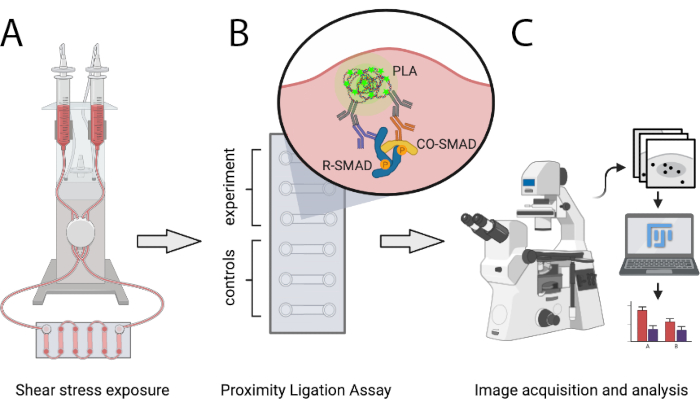
Figura 1: Representação esquemática do protocolo descrito. (A) As células semeadas em slides de 6 canais são expostas ao estresse da cisalhamento com um sistema de bomba pneumática. (B) As células fixas são utilizadas para o experimento PLA ou para condições de controle. (C) As imagens dos experimentos pla são adquiridas com um microscópio de fluorescência e são analisadas por meio do software de análise ImageJ. Clique aqui para ver uma versão maior desta figura.
Protocolo
1. Cultura celular e exposição ao estresse da tesoura fluida
NOTA: Os CES de veia umbilical humana (HUVECs) foram utilizados como exemplo para estudar a interação induzida por SS de SMADs. O protocolo descrito abaixo pode ser aplicado a todos os tipos de células responsivas SS.
- Cubra slide de 6 canais com 0,1% de gelatina de pele suína em PBS por 30 min a 37 °C.
- HuVECs de sementes em slides pré-revestidos de 6 canais a uma densidade de 2,5 x 106 células por mL em 30 μL de m199 meio completo.
NOTA: Para obter mais informações sobre como semear células no slide de fluxo, consulte referência24. - Deixe as células aderirem por 1h a 37 °C em uma incubadora umidificada.
- Adicione 60 μL de M199 médio completo pré-aquecido a cada um dos reservatórios.
- Cultura por 2 dias, com uma troca média suave uma vez por dia, a 37 °C em uma incubadora umidificada.
- Aspire completamente os reservatórios, adicione 120 μL de m199 médio completo pré-aquecido em um dos reservatórios, e aspire do outro lado.
- Adicione 60 μL de m199 médio completo pré-aquecido para ambos os reservatórios.
- Monte e inicie a configuração de fluxo conforme detalhado na referência25.
- Montar tubos em unidades fluidas. Aqui, tubos de silicone com diâmetro de 0,8 mm e 1,6 mm são utilizados para aplicar estresse de tesoura de 1 dyn/cm2 e 30 dyn/cm2, respectivamente.
NOTA: O material e o comprimento do tubo devem permanecer constantes, pois as mudanças podem influenciar o estresse resultante da tesoura. Em geral, outras combinações de sistemas de bomba e tubos podem ser usadas, desde que o estresse resultante da cisalhamento seja conhecido, e a bomba crie um fluxo laminar constante. - Encha os reservatórios com uma quantidade adequada de meio completo M199 pré-aquecido (mínimo de 10 mL).
- Conecte unidades fluidas com o tubo ao sistema de bomba e realize um pré-funcionamento sem células para equilibrar o meio e remover qualquer ar restante25.
- Conecte serialmente os canais únicos no slide de 6 canais um ao outro usando tubos de conexão serial. O primeiro e o último canal no slide serão conectados à tubulação montada em 1.6.1 (ver Figura 1A para um esquema). Tenha cuidado para não introduzir qualquer ar no sistema, pois isso pode prejudicar severamente as células. Mais informações sobre a conexão serial podem ser encontradas em referência26.
- Para a exposição das células a altos níveis de estresse de corte (>20 dyn/cm2), use uma fase de rampa, ou seja, aumentar o estresse da tesoura stepwise com fases de adaptação. As etapas podem ser definidas em incrementos de 5 dyn/cm2 por 30 min.
- Montar tubos em unidades fluidas. Aqui, tubos de silicone com diâmetro de 0,8 mm e 1,6 mm são utilizados para aplicar estresse de tesoura de 1 dyn/cm2 e 30 dyn/cm2, respectivamente.
2. Fixação
- Desprender slides das bombas após a exposição do fluido SS. Use grampos na tubulação ao se desprender, para evitar o derramamento médio na incubadora.
- Transfira imediatamente as lâminas de fluxo no gelo, enquanto o tubo restante é separado sequencialmente. Ao remover a tubulação dos reservatórios, o reservatório do outro lado deve ser mantido fechado com um dedo para evitar a captura de bolhas de ar no canal. Isso pode interferir com as etapas de fixação.
- Mantendo as células no gelo, aspire o meio cuidadosamente dos reservatórios, mas não do canal onde as células residem. Posteriormente, lave amostras com PBS estéril a frio (4 °C) com três vezes o volume do canal (90 μL). Adicione PBS em um reservatório e aspire cuidadosamente do outro reservatório. Repita esta etapa em cada um dos 6 canais por slide.
NOTA: Para todas as etapas de lavagem e incubação, a respectiva solução é adicionada em um dos reservatórios que leva a uma troca de soluções no canal. Para permitir a substituição completa de soluções no canal, a solução em excesso é então aspirada do outro reservatório. A solução na parte superior das células do canal não é removida. As células não devem secar em nenhum momento. Por isso, é importante lavar cuidadosamente sem qualquer inserção de bolha de ar nos slides. - Fixar as células adicionando 90 μL de solução pfa de 4% tamponada no mesmo reservatório onde o PBS foi adicionado antecipadamente e aspirar o líquido do outro reservatório. Repita esta etapa em cada canal em cada slide. Após a adição da solução PFA, transfira as amostras do gelo para a temperatura ambiente (RT) e incubar por 20 minutos.
ATENÇÃO: O PFA é tóxico e deve ser manuseado com cuidado. Use luvas e trabalhe sob um capô de fumaça. - Lave as células 3x com PBS (RT) adicionando-a em um reservatório e aspirando cuidadosamente do outro reservatório. Esvazie apenas os reservatórios, garantindo não secar o canal. Repita esta etapa para cada um dos 6 canais por slide.
- Sacie a fixação de PFA adicionando 90 μL de cloreto de amônio ambiente de 50 mM em PBS em um dos reservatórios. Aspirar solução em excesso do outro reservatório. Repita para cada canal no slide. Incubar as amostras por 10 minutos na RT.
- Lave como descrito na etapa 2.5.
NOTA: Neste ponto, as amostras podem ser armazenadas a 4 °C durante a noite, ou o protocolo pode ser imediatamente continuado com bloqueio e incubação de anticorpos primários (ver passo 3).
3. Bloqueio e incubação primária de anticorpos
- Para permeabilizar as células, adicione 90 μL de 0,3% Triton-X-100 em PBS em um reservatório esvaziado, e aspire do outro reservatório para cada canal. Incubar por 10 min na RT.
- Lave como descrito na etapa 2.5.
- Adicione 90 μL de solução de bloqueio PLA estéril em um reservatório de um canal e aspire do outro lado. Repita esta etapa para cada canal. Bloqueie por 1h a 37 °C em uma câmara umidificada.
- Para fazer uma câmara umidificada, use um prato de 10 cm com tecido molhado selado com filme de cera e coloque o prato na incubadora. Alternativamente, outros formatos de câmara de umidade podem ser usados que fornecem uma atmosfera úmida.
NOTA: Alternativamente, pode-se utilizar solução de bloqueio autodidato (por exemplo, 3% (w/v) BSA no PBS, filtrado estéril).
- Para fazer uma câmara umidificada, use um prato de 10 cm com tecido molhado selado com filme de cera e coloque o prato na incubadora. Alternativamente, outros formatos de câmara de umidade podem ser usados que fornecem uma atmosfera úmida.
- Prepare anticorpos primários (1:100) em diluente de anticorpos PLA. Prepare 30 μL da solução por canal. Adicione ambos os anticorpos primários simultaneamente e vórtice.
NOTA: Alternativamente, pode-se utilizar diluente de anticorpos auto-fabricados (por exemplo, 1% (w/v) BSA na PBS). Os anticorpos utilizados aqui são combinações de SMAD1-SMAD2/3, SMAD2/3-SMAD4 e phospho-SMAD1/5-SMAD4. Informações detalhadas podem ser encontradas na Tabela de Materiais. - Antes da aplicação de anticorpos primários, aspire a solução de bloqueio dos reservatórios e, também, cuidadosamente do canal. Pipeta 30 μL da solução de anticorpos primário imediatamente no canal vazio, inclinando o canal enquanto adiciona a solução.
NOTA: Realize a remoção da solução de bloqueio e a adição da solução de anticorpos canal a canal para garantir que as células não sequem no meio. - Incubar amostras com os anticorpos primários durante a noite em câmaras umidificadas a 4 °C.
NOTA: A incubação também pode ser realizada por 1h em temperatura ambiente, se estiver interessada em continuar com as seguintes etapas no mesmo dia.
4. Incubação da sonda PLA
NOTA: Para todas as etapas da seção 4.1-7.3, os tampões de lavagem A e B são armazenados a 4 °C e precisam ser aquecidos ao RT antes do uso.
- Diluir as sondas PLA (+)-mouse e (-)-rabbit para 1:5 na solução diluída de anticorpos PLA (ou 1% BSA). Prepare 30 μL por canal.
- Lave amostras 2x por 5 min usando 90 μL do tampão de lavagem A na RT, adicionando-a em um dos reservatórios e aspirando cuidadosamente do outro reservatório. Repita esta etapa para cada um dos 6 canais por slide.
- Aspire o tampão de lavagem A cuidadosamente e adicione 30 μL de solução de sonda PLA (preparada na etapa 4.1), semelhante à adição de anticorpos primários na etapa 3.5.
- Incubar amostras por 1h a 37 °C em uma câmara umidificada.
5. Ligadura
- Lave amostras 2x por 5 min usando 90 μL do tampão de lavagem A na RT, conforme descrito em 4.2.
- Prepare uma diluição de 1:5 do tampão de ligadura em água deionizada. Use este tampão para diluir a enzima ligase para 1:40 (no gelo). Use 30 μL por canal.
- Aspire completamente o tampão de lavagem A e adicione a solução de ligadura conforme descrito em 3.5.
- Incubar amostras por 30 min a 37 °C em uma câmara umidificada.
6. Amplificação
- Lave amostras 2x por 2 min usando 90 μL de tampão de lavagem A na RT, conforme descrito em 4.2.
- Prepare o tampão de amplificação diluindo-o 1:5 em água deionizada e use-o para diluir a enzima polimerase para 1:80 (no gelo). Proteja-se contra a luz. Prepare 30 μL por canal.
- Aspire o tampão de lavagem A adicione completamente e imediatamente a solução de amplificação preparada no canal vazio, conforme descrito em 3.5. Incubar amostras por 100 min a 37 °C em uma câmara umidificada.
7. Montagem
- Lave amostras 2x por 10 min usando 90 μL de Wash Buffer B em RT, conforme descrito em 4.2. Adicionar DAPI (1:500) a partir de 1 mg/mL solução de estoque (em água deionizada) na primeira lavagem para coloração de núcleos. Não seque o canal.
- Diluir o tampão de lavagem B em água desionizada (1:10) e lavar 1x com 90 μL de solução B tampão de 0,1x conforme descrito em 4.2.
- Aspire o tampão de lavagem B completamente e imediatamente adicione 2-3 gotas do meio de montagem líquida em um reservatório. Distribua-o no canal inclinando o slide. Armazene amostras a 4 °C em um ambiente umidificado até a imagem.
8. Aquisição de imagens
- Adquira imagens usando um microscópio de fluorescência. Certifique-se de que os respectivos filtros que se encaixam nas sondas PLA fluorescentes estejam disponíveis.
NOTA: É benéfico fazer uso de um microscópio confocal, se possível, pois as manchas PLA obtidas são mais definidas. Isso também suporta mais processamento de imagens e análise de dados.
9. Análise e quantificação de imagens usando ImageJ/FIJI
- Processar imagens exportadas (.tiff) com um programa de processamento de imagens, como ImageJ27.
NOTA: Todos os scripts usados neste estudo e necessários para a contagem automática de eventos celulares, nucleares e todos os PLA (por célula) podem ser encontrados em um repositório do GitHub: https://github.com/Habacef/Proximity-Ligation-Assay-analysis. Realize análises estatísticas utilizando qualquer programa ou ferramenta adequada.
Resultados
Anteriormente, usamos o PLA para detectar interações de diferentes proteínas SMAD3 e analisamos alterações induzidas pelo estresse da tesoura na fosforilação SMAD28.
Aqui, ambos os métodos foram combinados com o protocolo descrito acima. Os HUVECs foram submetidos ao estresse de tesoura de 1 dyn/cm2 e 30 dyn/cm2 e analisados para interações de fatores de transcrição do SMAD. Mostramos que, quando comparado ao alt...
Discussão
O protocolo baseado em PLA descrito aqui oferece uma maneira eficiente de determinar a proximidade de duas proteínas (por exemplo, sua interação direta) em CEs expostas ao estresse de tesoura com resolução quantitativa e espacial. Usando slides de fluxo com múltiplos canais paralelos, várias interações proteicas diferentes podem ser examinadas ao mesmo tempo em células sob condições mecânicas idênticas. Em contraste, os sistemas de câmara de fluxo de construção personalizada geralmente fazem uso de um ú...
Divulgações
Os autores não declaram conflito de interesses.
Agradecimentos
Agradecemos à Dra. Maria Reichenbach e ao Dr. Christian Hiepen pelo apoio ao sistema de escoamento e Eleanor Fox e Yunyun Xiao por lerem criticamente o manuscrito. P-L.M. foi financiado pela International Max Planck Research School IMPRS-Biology and Computation (IMPRS-BAC). PK recebeu financiamento pelo DFG-SFB1444. A Figura 1 foi criada usando BioRender.
Materiais
| Name | Company | Catalog Number | Comments |
| µ-Slide VI 0.4 | ibidi | 80606 | 6-channel slide |
| Ammonium Chloride | Carl Roth | K298.1 | Quenching |
| Bovine Serum Albumin | Carl Roth | 8076.4 | Blocking |
| DAPI | Sigma Aldrich/ Merck | D9542 | Stain DNA/Nuclei |
| DPBS | PAN Biotech | P04-53500 | PBS |
| Duolink In Situ Detection Reagents Green | Sigma Aldrich/ Merck | DUO92014 | PLA kit containing Ligase, ligation buffer, polymerase and amplification buffer (with green labeled oligonucleotides) |
| Duolink In Situ PLA Probe Anti-Mouse MINUS | Sigma Aldrich/ Merck | DUO92004 | MINUS probe |
| Duolink In Situ PLA Probe Anti-Rabbit PLUS | Sigma Aldrich/ Merck | DUO92002 | PLUS probe |
| Duolink In Situ Wash Buffers, Fluorescence | Sigma Aldrich/ Merck | DUO82049 | PLA wash buffers A and B |
| Endothelial Cell Growth Supplement | Corning | supplement for medium (ECGS) | |
| Fetal calf Serum | supplement for medium | ||
| FIJI | Image Analysis software | ||
| Formaldehyde solution 4% buffered | KLINIPATH/VWR | VWRK4186.BO1 | PFA |
| Full medium | M199 basal medium +20 % FCS +1 % P/S + 2 nM L-Glu + 25 µg/mL Hep + 50 µg/mL ECGS | ||
| Gelatin from porcine skin, Type A | Sigma Aldrich | G2500 | Use 0.1% in PBS for coating of flow channels |
| GraphPad Prism v.7 | GarphPad | Statistical Program used for the Plots and statistical calculations | |
| Heparin sodium salt from porcine intestinal mucosa | Sigma Aldrich | H4784-250MG | supplement for medium (Hep) |
| HUVECs | |||
| ibidi Mounting Medium | ibidi | 50001 | Liquid mounting medium |
| ibidi Pump System | ibidi | 10902 | pneumatic pump |
| Leica TCS SP8 | Leica | confocal microscope | |
| L-Glutamin 200mM | PAN Biotech | P04-80100 | supplement for medium (L-Glu) |
| Medium 199 | Sigma Aldrich | M2154 | Base medium |
| mouse anti- SMAD1 Antibody | Abcam | ab53745 | Suited for PLA |
| mouse anti- SMAD2/3 Antibody | BD Bioscience | 610843 | Not suited for PLA in combination with CST 9515 |
| mousee anti- SMAD4 Antibody | Sanata Cruz Biotechnology | sc-7966 | Suited for PLA |
| Penicillin 10.000U/ml /Streptomycin 10mg/ml | PAN Biotech | P06-07100 | supplement for medium (P/S) |
| Perfusion Set WHITE | ibidi | 10963 | Tubings used for 1 dyn/cm2 |
| Perfusion Set YELLOW and GREEN | ibidi | 10964 | Tubings used for 30 dyn/cm2 |
| rabbit anti- phospho SMAD1/5 Antibody | Cell Signaling Technologies | 9516 | Suited for PLA |
| rabbit anti- SMAD2/3 XP Antibody | Cell Signaling Technologies | 8685 | Suited for PLA |
| rabbit anti- SMAD4 Antibody | Cell Signaling Technologies | 9515 | Not suited for PLA in combination with BD 610843 |
| Serial Connector for µ-Slides | ibidi | 10830 | serial connection tubes |
| Triton X-100 | Carl Roth | 6683.1 | Permeabilization |
Referências
- Yadin, D., Knaus, P., Mueller, T. D. Structural insights into BMP receptors: Specificity, activation and inhibition. Cytokine and Growth Factor Reviews. 27, 13-34 (2016).
- Sieber, C., Kopf, J., Hiepen, C., Knaus, P. Recent advances in BMP receptor signaling. Cytokine and Growth Factor Reviews. 20 (5-6), 343-355 (2009).
- Hiepen, C., et al. BMPR2 acts as a gatekeeper to protect endothelial cells from increased TGFβ responses and altered cell mechanics. PLoS Biology. 17 (12), 3000557 (2019).
- Hildebrandt, S., et al. ActivinA induced SMAD1/5 Signaling in an iPSC derived EC model of Fibrodysplasia Ossificans Progressiva (FOP) can be rescued by the drug candidate saracatinib. Stem Cell Reviews and Reports. , (2021).
- Goumans, M. J., et al. Balancing the activation state of the endothelium via two distinct TGF-beta type I receptors. The EMBO Journal. 21 (7), 1743-1753 (2002).
- Goumans, M. J., et al. Activin receptor-like kinase (ALK)1 is an antagonistic mediator of lateral TGFbeta/ALK5 signaling. Molecular Cell. 12 (4), 817-828 (2003).
- Daly, A. C., Randall, R. A., Hill, C. S. Transforming growth factor beta-induced Smad1/5 phosphorylation in epithelial cells is mediated by novel receptor complexes and is essential for anchorage-independent growth. Molecular and Cellular Biology. 28 (22), 6889-6902 (2008).
- Ramachandran, A., et al. TGF-β uses a novel mode of receptor activation to phosphorylate SMAD1/5 and induce epithelial-to-mesenchymal transition. eLife. 7, 31756 (2018).
- Flanders, K. C., et al. Brightfield proximity ligation assay reveals both canonical and mixed transforming growth factor-β/bone morphogenetic protein Smad signaling complexes in tissue sections. The Journal of Histochemistry and Cytochemistry : The Official Journal of The Histochemistry Society. 62 (12), 846-863 (2014).
- Miyazono, K., Maeda, S., Imamura, T., Dijke, P. T., Heldin, C. -. H. . Smad Signal Transduction: Smads in Proliferation, Differentiation and Disease. , 277-293 (2006).
- Goumans, M. J., Zwijsen, A., Ten Dijke, P., Bailly, S. Bone morphogenetic proteins in vascular homeostasis and disease. Cold Spring Harbor Perspectives in Biology. 10 (2), 031989 (2018).
- Cai, J., Pardali, E., Sánchez-Duffhues, G., ten Dijke, P. BMP signaling in vascular diseases. FEBS Letters. 586 (14), 1993-2002 (2012).
- Cunha, S. I., Magnusson, P. U., Dejana, E., Lampugnani, M. G. Deregulated TGF-β/BMP signaling in vascular malformations. Circulation research. 121 (8), 981-999 (2017).
- MacCarrick, G., et al. Loeys-Dietz syndrome: a primer for diagnosis and management. Genetics in Medicine : An Official Journal of the American College of Medical Genetics. 16 (8), 576-587 (2014).
- Baeyens, N., Bandyopadhyay, C., Coon, B. G., Yun, S., Schwartz, M. A. Endothelial fluid shear stress sensing in vascular health and disease. The Journal of Clinical Investigation. 126 (3), 821-828 (2016).
- Min, E., et al. Activation of Smad 2/3 signaling by low shear stress mediates artery inward remodeling. bioRxiv. , 691980 (2019).
- Zhou, J., et al. BMP receptor-integrin interaction mediates responses of vascular endothelial Smad1/5 and proliferation to disturbed flow. Journal of Thrombosis and Haemostasis. 11 (4), 741-755 (2013).
- Zhou, J., et al. Force-specific activation of Smad1/5 regulates vascular endothelial cell cycle progression in response to disturbed flow. Proceedings of the National Academy of Sciences of the United States of America. 109 (20), 7770-7775 (2012).
- van Dijk, R. A., et al. Visualizing TGF-β and BMP signaling in human atherosclerosis: A histological evaluation based on Smad activation. Histology and Histopathology. 27 (3), 387-396 (2012).
- Derwall, M., et al. Inhibition of bone morphogenetic protein signaling reduces vascular calcification and atherosclerosis. Arteriosclerosis, Thrombosis, and Vascular Biology. 32 (3), 613-622 (2012).
- Fredriksson, S., et al. Protein detection using proximity-dependent DNA ligation assays. Nature Biotechnology. 20 (5), 473-477 (2002).
- Söderberg, O., et al. Direct observation of individual endogenous protein complexes in situ by proximity ligation. Nature Methods. 3 (12), 995-1000 (2006).
- Alam, M. S. Proximity Ligation Assay (PLA). Current Protocols in Immunology. 123 (1), 58 (2018).
- Application Note 03: Growing Cells in µ-Channels. ibidi Available from: https://ibidi.com/img/cms/support/AN/AN03_Growing_cells.pdf (2012)
- Application Note 13: HUVECs under perfusion. ibidi Available from: https://ibidi.com/img/cms/support/AN/AN13_HUVECs_under_perfusion.pdf (2019)
- ibidi. Application Note 31: Instructions µ-Slide VI 0.4. ibidi. , (2013).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Reichenbach, M., et al. Differential impact of fluid shear stress and YAP/TAZ on BMP/TGF-β induced osteogenic target genes. Advanced Biology. 5 (2), 2000051 (2021).
- Hiepen, C., Mendez, P. L., Knaus, P. It takes two to tango: Endothelial TGFβ/BMP signaling crosstalk with mechanobiology. Cells. 9 (9), 1965 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados