Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Isolement des cellules musculaires lisses aortiques primaires spécifiques au patient et mesures semi-quantitatives de contraction en temps réel in vitro
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Cet article décrit une méthode basée sur la culture d’explants pour l’isolement et la culture de cellules musculaires lisses aortiques humaines primaires spécifiques au patient et de fibroblastes dermiques. En outre, une nouvelle méthode est présentée pour mesurer la contraction cellulaire et l’analyse ultérieure, qui peut être utilisée pour étudier les différences spécifiques au patient dans ces cellules.
Résumé
Les cellules musculaires lisses (SMC) sont le type de cellule prédominant dans le milieu aortique. Leur machinerie contractile est importante pour la transmission de la force dans l’aorte et régule la vasoconstriction et la vasodilatation. Les mutations dans les gènes codant pour les protéines de l’appareil contractile SMC sont associées à des maladies de l’aorte, telles que les anévrismes de l’aorte thoracique. Mesurer la contraction de la SMC in vitro est difficile, en particulier de manière à haut débit, ce qui est essentiel pour le dépistage du matériel du patient. Les méthodes actuellement disponibles ne conviennent pas à cette fin. Cet article présente une nouvelle méthode basée sur la détection d’impédance de substrat de cellule électrique (ECIS). Tout d’abord, un protocole d’explantation est décrit pour isoler les SMC primaires humains spécifiques au patient à partir de biopsies aortiques et de fibroblastes dermiques primaires humains spécifiques au patient pour l’étude des anévrismes de l’aorte. Ensuite, une description détaillée d’une nouvelle méthode de contraction est donnée pour mesurer la réponse contractile de ces cellules, y compris l’analyse ultérieure et la suggestion de comparer différents groupes. Cette méthode peut être utilisée pour étudier la contraction des cellules adhérentes dans le cadre d’études translationnelles (cardiovasculaires) et d’études de dépistage de patients et de médicaments.
Introduction
Les cellules musculaires lisses (SMC) sont le type de cellule prédominant dans la couche médiale aortique, la couche la plus épaisse de l’aorte. À l’intérieur du mur, ils sont orientés radialement et sont impliqués, entre autres fonctions, dans la vasoconstriction et la vasodilatation1. La machine contractile SMC est impliquée dans la transmission de la force dans l’aorte à travers le lien fonctionnel avec la matrice extracellulaire2. Des mutations dans les gènes codant pour les protéines de l’appareil contractile SMC, telles que la chaîne lourde de la myosine du muscle lisse (MYH11) et l’actine du muscle lisse (ACTA2), ont été liées à des cas d’anévrismes de l’aorte thoracique familiale, soulignant la pertinence de la contraction du SMC dans le maintien de l’intégrité structurelle et fonctionnelle de l’aorte 1,2 . En outre, des mutations dans la voie de signalisation TGFβ sont également associées à des anévrismes de l’aorte, et leurs effets sur la physiopathologie de l’anévrisme de l’aorte peuvent également être étudiés dans les fibroblastes cutanés3.
La mesure à haut débit de la contraction SMC in vitro est un défi. Comme la contractilité SMC ne peut pas être mesurée in vivo chez l’homme, les essais in vitro sur des cellules humaines présentent une alternative réalisable. De plus, le développement de l’anévrisme de l’aorte abdominale (AAA) dans les modèles animaux est soit induit chimiquement par, par exemple, la perfusion d’élastase, soit causé par une mutation spécifique. Par conséquent, les données animales ne sont pas comparables au développement de l’AAA chez l’homme, qui a principalement une cause multifactorielle, comme le tabagisme, l’âge et / ou l’athérosclérose. In vitro Jusqu’à présent, la contractilité du SMC a été principalement mesurée par microscopie à force de traction 4,5, quantification des flux de calcium intracellulaire de fluorescence Fura-26 et essais de rides au collagène7. Bien que la microscopie à force de traction fournisse un aperçu numérique inestimable des forces générées par une seule cellule, elle ne convient pas au criblage à haut débit en raison du traitement complexe des données mathématiques et de l’analyse d’une cellule à la fois, ce qui signifie qu’il est très long de mesurer un nombre représentatif de cellules par donneur. Les tests de teinture Fura-2 et de rides au collagène permettent la détermination superficielle de la contraction et ne donnent pas une sortie numérique précise, ce qui les rend moins adaptés à la discrimination des différences spécifiques au patient. Une altération de la contraction de la SMC dans les cellules dérivées de l’aorte des patients atteints d’anévrisme de l’aorte abdominale a été démontrée pour la première fois en optimisant une nouvelle méthode de mesure de la contraction de la SMC in vitro8. Cela a été fait en réorientant la méthode de détection d’impédance de la cellule électrique et du substrat (ECIS). ECIS est un test en temps réel à débit moyen pour la quantification du comportement et de la contraction des cellules adhérentes 9,10,11 tels que la croissance et le comportement SMC dans les tests de cicatrisation et de migration des plaies 12,13,14. La méthode exacte est décrite dans la section protocole. De cette manière optimisée, l’ECIS peut également être utilisé pour étudier la contraction des fibroblastes en raison de leur taille et de leur morphologie similaires.
L’objectif de cet article est de fournir une description par étapes de la méthode de mesure de la contraction SMC in vitro à l’aide d’ECIS8 et de comparer la contraction entre les SMC témoins et patients. Tout d’abord, l’isolement et la culture des SMC primaires à partir de biopsies aortiques de contrôle et de patients sont expliqués, ce qui peut être utilisé pour la mesure de la contraction. Deuxièmement, les mesures et l’analyse de la contraction, ainsi que la vérification de l’expression des marqueurs SMC, sont décrites. En outre, cet article décrit la méthode d’isolement des fibroblastes dermiques spécifiques au patient dont la contraction peut être mesurée à l’aide de la même méthodologie. Ces cellules peuvent être utilisées pour des études spécifiques au patient axées sur l’anévrisme de l’aorte ou d’autres pathologies cardiovasculaires15 ou des études pronostiques utilisant un protocole de transdifférenciation qui permet la mesure de la contraction avant la chirurgie de l’anévrisme16.
Access restricted. Please log in or start a trial to view this content.
Protocole
REMARQUE: Des biopsies de l’aorte ont été obtenues lors de la réparation d’anévrisme ouvert dans les centres médicaux universitaires d’Amsterdam, le centre médical universitaire VU, Amsterdam, Zaans Medisch Centrum, Zaandam et l’hôpital Dijklander, Hoorn, Pays-Bas. Le tissu aortique témoin a été obtenu à partir du morceau de l’aorte attaché à l’artère rénale prélevé pour les greffes de rein. Seuls les patients âgés de plus de 18 ans ont été inclus et tous les patients ont donné leur consentement éclairé pour participer à l’étude. Tout le matériel a été collecté conformément aux règlements de la Déclaration de l’AMM d’Helsinki et aux directives institutionnelles du Comité d’éthique médicale du Centre médical VU. Toutes les expériences et les protocoles expérimentaux ont été réalisés conformément aux directives institutionnelles et approuvés par le Comité d’éthique médicale du Centre médical VU. Pour obtenir des renseignements complets sur les lignées cellulaires de contrôle et de patients utilisées, reportez-vous à la section 8.
1. Isoler les SMC humains primaires des biopsies aortiques
REMARQUE: Effectuez les étapes suivantes sous une hotte à écoulement laminaire de culture tissulaire stérile. Portez des gants et utilisez des techniques aseptiques standard lorsque vous manipulez des échantillons de sang et de tissus humains. Les SMC sont cultivés dans 231 cellules musculaires lisses vasculaires humaines milieu basal complété par 100 U / mL de pénicilline, 100 μg / mL de streptomycine et un supplément de croissance musculaire lisse appelé milieu SMC.
- Stérilisez deux paires de pinces chirurgicales et un scalpel en les immergeant dans de l’éthanol à 70% et en les essuyant ensuite à sec.
- Pipette 2 mL de milieu SMC dans une boîte de Petri dans laquelle la dissection tissulaire sera effectuée.
- Pipeter 2,5 mL de milieu SMC dans deux flacons T25. Faites tourbillonner les flacons de sorte que le petit volume de milieu recouvre toute la surface.
- Transporter la biopsie de la paroi aortique humaine récoltée de la salle d’opération au laboratoire sur de la glace dans un tube en plastique stérile avec une solution de transfert de tissu froide et stérile (voir le tableau des matériaux) ou 0,9% de NaCl.
- Ouvrez le tube avec le tissu à l’intérieur de la hotte de culture tissulaire. Sortez la biopsie du tube à l’aide de la pince stérilisée et placez-la dans la boîte de Pétri (Figure 1A).
- Inspectez visuellement la biopsie pour identifier les trois couches aortiques, la tunique intima (interne), le milieu (milieu) et l’adventice (couche externe). Recherchez la présence de plaques d’athérosclérose du côté intima et de tissu conjonctif visqueux du côté adventice (Figure 1B) pour distinguer les couches.
- Pour isoler les SMC du support, supprimez les deux autres couches. Placez d’abord le tissu avec la plaque d’intima vers le haut (Figure 1B). Retirez la plaque solide en l’éloignant du tissu avec une pince tout en poussant le tissu vers le bas avec l’autre paire de pinces. Retirez les couches suivantes de plaque jusqu’à ce que la couche médiale rose et uniforme soit visible.
- Retournez le tissu (Figure 1C). Répétez la même procédure, comme à l’étape 1.7, en retirant la couche adventice (Figure 1D). Assurez-vous de retirer toutes les parties visibles en autant de tentatives que nécessaire, car ce calque ne se détachera pas facilement du support.
REMARQUE: Il est essentiel que les couches intimales et adventices soient enlevées correctement pour obtenir une population SMC aussi propre que possible. - Une fois la couche médiale isolée, coupez le tissu en cubes d’environ 1 mm x 1 mm x 1 mm. Appuyez sur le support avec une pince et coupez le tissu dans une direction à l’aide du scalpel. Ne coupez pas d’avant en arrière; faire des coupes propres et unidirectionnelles pour minimiser les dommages. Essayez de faire autant de cubes que possible, compte tenu de la taille de la biopsie (Figure 1E).
- Placez les morceaux de tissu dans le quart supérieur de la fiole T25 à l’aide de la pince. Placez 10 à 20 cubes par flacon si la quantité de matériau le permet (Figure 1F).
REMARQUE: Utilisez des pinces lisses pour minimiser l’adhérence du tissu aux côtes de la pince et détachez facilement le tissu dans la fiole. - Incuber les cubes de tissu dans les flacons T25 pendant environ 10 jours à 5 % de CO2 à 37 °C dans un incubateur.
REMARQUE: La première excroissance cellulaire est attendue à ce moment-là. Les cellules qui migrent initialement peuvent sembler plus petites que les SMC normaux. - Une fois la croissance cellulaire observée, ajoutez 2,5 mL de milieu à la fiole, en veillant à ne pas la pipeter sur les morceaux de tissu pour les empêcher de se détacher.
- Après environ 5 jours de plus, lorsque des grappes de cellules sont observées autour des morceaux de tissu, changez le milieu de culture. Si des morceaux de tissu se détachent, retirez-les car ils ne se rattacheront pas.
- Une fois que les cellules sont confluentes à 80-90%, transférez-les dans une fiole T75 et continuez à cultiver dans ce format.
REMARQUE: Un rapport de division de 1: 2 ou 1: 3 est recommandé. Congelez une sauvegarde des cellules à un passage précoce. Les cellules conservent généralement leurs propriétés jusqu’au passage 10; les passages ultérieurs ne doivent pas être utilisés pour des expériences.
2. Isoler les fibroblastes dermiques primaires des biopsies cutanées
REMARQUE: Effectuez les étapes suivantes sous une hotte à écoulement laminaire de culture tissulaire stérile. Portez des gants et utilisez des techniques aseptiques standard lorsque vous manipulez des échantillons de sang et de tissus humains. Les fibroblastes sont cultivés dans un milieu basal complété par 10% de sérum bovin fœtal, 100 U / mL de pénicilline et 100 μg / mL de streptomycine, appelée milieu fibroblastique.
- Stériliser deux paires de pinces chirurgicales et un scalpel en les trempant dans de l’éthanol à 70 % et en les essuyant ensuite à sec.
- Pipette 2 mL de milieu fibroblastique dans une boîte de Petri dans laquelle la dissection tissulaire sera effectuée.
- Pipeter 2,5 mL de milieu fibroblastique dans deux flacons T25. Faites tourbillonner les flacons de sorte que le petit volume de milieu recouvre toute la surface.
- Transporter la biopsie cutanée récoltée de la salle d’opération au laboratoire sur de la glace dans une solution de transfert de tissu stérile froide (voir la table des matériaux) ou 0,9% de NaCl dans un tube en plastique stérile.
- Ouvrez le tube avec le tissu à l’intérieur de la hotte de culture tissulaire. Sortez la biopsie du tube à l’aide de la pince stérilisée et placez-la dans la boîte de Pétri (Figure 2A).
- Inspectez visuellement la biopsie pour identifier les trois couches de la peau, l’épiderme, le derme et la graisse sous-cutanée. Recherchez une surface de peau reconnaissable, parfois avec des cheveux visibles, pour identifier l’épiderme. De l’autre côté, recherchez la graisse sous-cutanée, qui est souvent jaune et visqueuse. Identifier la couche entre l’épiderme et la graisse sous-cutanée comme le derme - la source de fibroblastes viables (Figure 2B).
- Pour isoler les fibroblastes du derme, retirez les deux autres couches et placez le tissu sur le côté afin que les trois couches soient visibles.
REMARQUE: Contrairement au tissu aortique, les couches de la peau ne peuvent pas être séparées les unes des autres; par conséquent, ils doivent être coupés. Le tissu est également plus caoutchouteux, ce qui le rend plus difficile à couper. Utilisez un scalpel pointu. - Maintenez le tissu vers le bas avec une pince. Couper parallèlement à la frontière entre l’épiderme et le derme. Coupez tout l’épiderme. Essayez de couper en une ligne propre et ne bougez pas d’avant en arrière pour éviter les dommages aux tissus.
- Retournez le tissu. Répétez la même procédure qu’à l’étape 2.8 ; cette fois, couper dans le derme parallèlement à la bordure avec la graisse sous-cutanée.
- Une fois le derme isolé, coupez le tissu en cubes d’environ 1 x 1 x 1 mm3. Appuyez sur le tissu avec une pince et coupez le tissu dans une direction à l’aide du scalpel. Ne coupez pas d’avant en arrière; faire des coupes propres et unidirectionnelles pour minimiser les dommages. Essayez de faire autant de cubes que possible, compte tenu de la taille de la biopsie (Figure 2C).
- Placez les morceaux de tissu dans le quart supérieur de la fiole T25 à l’aide de la pince. Placez 10 à 20 cubes par flacon si la quantité de matériau le permet (Figure 2D).
REMARQUE: Utilisez une pince lisse pour minimiser l’adhérence du tissu aux côtes de la pince et détachez facilement le tissu dans la fiole. - Incuber les cubes de tissu dans les flacons T25 pendant environ 10 jours à 5 % de CO2 à 37 °C dans un incubateur.
REMARQUE: La première excroissance cellulaire est attendue à ce moment-là. Les cellules qui migrent initialement peuvent sembler plus petites que les fibroblastes normaux. - Une fois la croissance cellulaire observée, ajoutez 2,5 mL de milieu à la fiole, en veillant à ne pas la pipeter sur les morceaux de tissu pour les empêcher de se détacher.
- Après environ 5 jours de plus, lorsque des grappes de cellules peuvent être observées autour des morceaux de tissu, changez le milieu de culture. Si des morceaux de tissu se détachent, retirez-les car ils ne se rattacheront pas.
- Une fois que les cellules sont confluentes à 80-90%, transférez-les dans une fiole T75 et continuez à cultiver dans ce format.
REMARQUE: Un rapport de division de 1: 2 ou 1: 3 est recommandé. Congelez une sauvegarde des cellules à un passage précoce. Les cellules conservent généralement leurs propriétés jusqu’au passage 10; les passages ultérieurs ne doivent pas être utilisés pour des expériences.
3. Mesure de la contraction (exemples de SMC)
- Préparez un réseau ECIS 96w10e (voir la figure 3A pour une image représentative du réseau avec des électrodes agrandies et des cellules ensemencées sur les électrodes).
REMARQUE: Effectuez les étapes suivantes sous une hotte à écoulement laminaire de culture tissulaire stérile.- Enduisez un réseau stérile de 96w10e avec 200 μL de L-cystéine de 10 mM par puits pendant 30 min à température ambiante.
- Lavez la plaque deux fois avec de l’eau stérile. Laissez la plaque sécher pendant la nuit dans la hotte de culture tissulaire avec le couvercle légèrement ouvert.
REMARQUE: Le revêtement de la plaque avec de la L-cystéine n’est nécessaire que lors de la première utilisation de la plaque. - Le lendemain, stériliser les UV de la plaque et du couvercle pendant 30 min. Tournez le couvercle vers le haut pour que l’intérieur soit stérilisé. Une fois la plaque stérilisée, n’ouvrez pas la plaque à l’extérieur de la hotte d’écoulement.
- Ensemencement de cellules sur le réseau ECIS
- Préchauffer 1% solution de gélatine stérile au bain-marie pendant 30 min.
REMARQUE: La solution est stockée dans le réfrigérateur et peut se solidifier, ce qui rend difficile la pipette. - Par la suite, recouvrir les puits de 100 μL de la solution de gélatine à 1 % par puits et incuber la plaque à 37 °C pendant au moins 1 h.
- Aspirer la gélatine des puits.
- Comptez les cellules à l’aide d’un compteur de cellules automatisé et ensemencez les SMC en trois fois à une densité d’ensemencement de 30 000 cellules/puits dans 200 μL de milieu SMC.
- Placez la plaque avec les SMC dans le support de puits ECIS 96 dans l’incubateur de culture cellulaire. Double-cliquez sur le logiciel ECIS Applied Biophysics pour ouvrir le programme et appuyez sur le bouton Configuration .
- Vérifiez si toutes les électrodes sont en contact avec le support (vert; rouge si aucune connexion) dans le panneau inférieur gauche intitulé Configuration du puits. Si les électrodes ne sont pas correctement connectées, ajustez la plaque dans le support avant de commencer la mesure.
- Sélectionnez le type de plaque (tableau de 96 puits) dans le même panneau en cliquant sur Type de tableau.
- Dans le panneau supérieur droit Configuration de la collecte de données, cliquez sur une seule fréquence et modifiez la valeur d’impédance à 4000 Hz et l’intervalle à 8 s.
- Cliquez sur le bouton Démarrer pour lancer les mesures. Attendez qu’un nouveau panneau s’ouvre, où le fichier ECIS peut être enregistré.
- Laissez les cellules s’attacher et établir une monocouche pendant 48 h.
REMARQUE : La fixation et l’étalement des cellules sur les électrodes génèrent une valeur de résistance de base (Figure 3B).
- Préchauffer 1% solution de gélatine stérile au bain-marie pendant 30 min.
- Stimulation des cellules pour induire la contraction
- Induire la contraction SMC en stimulant les cellules avec 10 μg/mL d’ionophore de calcium, l’ionomycine.
REMARQUE: L’ionomycine induira l’afflux de Ca2+ extracellulaire, activant la cascade contractile; voir la figure 4A pour des images représentatives de cellules contractées non stimulées et stimulées. - Diluer 1 mg de poudre d’ionomycine dans 100 μL de diméthylsulfoxyde pour obtenir une solution mère de 10 mg/mL, et conserver 10 μL d’aliquotes à -20 °C. Avant utilisation, diluer la solution d’ionomycine 1:10 dans un milieu SMC pour préparer la solution de travail à ajouter aux cellules.
- Effectuez la stimulation cellulaire pendant que le réseau est encore dans le support du réseau à l’intérieur de l’incubateur de culture cellulaire et que les électrodes sont attachées au système.
- Pour stimuler les cellules, retirez le couvercle et placez-le sur une surface stérile à l’intérieur de l’incubateur. Préparer deux pipettes, réglées à 2 μL et 150 μL.
- Avant de commencer la stimulation, appuyez sur Marquer dans le logiciel et placez un commentaire.
REMARQUE: Cela facilitera la recherche du moment exact de la stimulation lors de l’analyse des données. - Pipette 2 μL de la solution de travail d’ionomycine dans chaque puits le plus rapidement possible. Une fois que toutes les cellules ont été stimulées, mélanger le milieu dans les puits à l’aide de la deuxième pipette.
REMARQUE: En raison des effets rapides, il n’est pas nécessaire de changer les pointes entre différentes lignées cellulaires et conditions. Travaillez rapidement tout en stimulant et en mélangeant car l’ionomycine a un effet aigu. Lorsque vous stimulez une assiette pleine, stimulez un maximum de 3 colonnes à la fois. Après chaque stimulation, attendez au moins 30 minutes jusqu’à la prochaine stimulation pour laisser les cellules se remettre de la température et du changement de CO2 causés par l’ouverture de la porte de l’incubateur. - Directement après la stimulation, appuyez à nouveau sur Marquer pour ajouter un commentaire indiquant que la stimulation est terminée.
- Terminer l’expérience de stimulation
- Enregistrez les valeurs d’impédance pendant environ 5 minutes après la stimulation à l’ionomycine. Terminez l’enregistrement en appuyant sur Terminer.
- Réutilisez les réseaux ECIS jusqu’à trois fois : lavez les puits deux fois avec de l’eau stérile et incubez la plaque à 37 °C pendant 2-3 h avec de la trypsine ou un réactif similaire. Répétez les étapes 3.1.2 et 3.1.3.
- Induire la contraction SMC en stimulant les cellules avec 10 μg/mL d’ionophore de calcium, l’ionomycine.
- Exportation des données
- Pour exporter les données, cliquez sur Fichier | Exporter des données | Vers Excel (Sélectionné).. Sélectionnez un dossier pour enregistrer le fichier.
- Cliquez sur Séparer lorsque le programme demande à combiner ou à séparer des feuilles.
- Analyse de la sortie contractile
- Pour calculer la réponse contractile, utilisez l’équation suivante (1), où C indique la contraction (exprimée en % de la variation par rapport à la ligne de base), la préstimulation (PrS) indique la valeur de résistance (exprimée en Ω) juste avant la stimulation à l’ionomycine et la post-stimulation (PoS) indique la résistance (exprimée en Ω) 2 min après la fin de la stimulation.
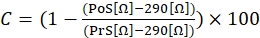 (1)
(1)
REMARQUE: Comme le montre l’équation (1), la valeur d’impédance d’un puits rempli de milieu de culture sans cellules attachées (valeur de 290 Ω) est soustraite de tous les résultats du calcul final. Il est recommandé de mesurer les réponses contractiles en trois exemplaires dans chaque expérience et d’effectuer trois expériences indépendantes avec la même lignée cellulaire pour tenir compte de toute variation inter- et intra-expérimentale. - Une fois les trois expériences indépendantes réalisées, regroupez les données en calculant la moyenne des trois mesures, y compris l’écart-type (ET).
- Pour calculer la réponse contractile, utilisez l’équation suivante (1), où C indique la contraction (exprimée en % de la variation par rapport à la ligne de base), la préstimulation (PrS) indique la valeur de résistance (exprimée en Ω) juste avant la stimulation à l’ionomycine et la post-stimulation (PoS) indique la résistance (exprimée en Ω) 2 min après la fin de la stimulation.
- Comparaison de différents groupes
- Pour définir la plage de contraction normale et étudier les questions de recherche spécifiques ultérieures, utilisez le groupe témoin pour définir la « contraction normale » et la comparer à la réponse contractile des patients ou des groupes de traitement.
- Calculer la moyenne et la DS de l’ensemble du groupe témoin, c’est-à-dire des valeurs finales pour toutes les lignées cellulaires, comme décrit à l’étape 3.5.2.
- Utilisez la plage de la moyenne ± 2SD pour identifier les cellules de l’autre groupe qui se situent en dehors de cette plage, indiquant qu’elles ont une réponse contractile altérée.
REMARQUE: Le mécanisme derrière la réponse contractile modifiée est sujet à des questions de recherche spécifiques et dépend de la cellule et de la condition et ne sera pas discuté dans ce protocole.
4. Détection de la présence de marqueurs spécifiques au SMC
- Isolement de l’ARN
- Ensemencez les mêmes lignées cellulaires spécifiques au patient utilisées pour les mesures de contraction dans des plaques de 6 puits, un puits par lignée cellulaire. Comptez les cellules à l’aide d’un compteur de cellules automatisé et ensemencez les cellules à une densité d’ensemencement de 200 000 cellules / puits dans un milieu SMC et laissez-les se fixer pendant la nuit.
- Lavez les cellules une fois avec du PBS stérile.
- Lysez les cellules et isolez les cellules selon les instructions du fabricant.
- Synthèse de l’ADNc
- Effectuer la synthèse de l’ADNc dans 20 μL d’un mélange réactionnel de transcription inverse. Diluer les concentrations d’ARN isolé total conformément aux instructions fournies par le fabricant.
- qPCR
- Mesurez l’expression génique des gènes marqueurs SMC, par exemple ACTA2, CNN1, SMTN et TAGLN pour confirmer que les cellules isolées sont bien des SMC et détecter les marqueurs SMC. Vérifiez la corrélation des résultats avec la sortie contractile.
- Utilisez au moins deux gènes d’entretien ménager pour normaliser les données, par exemple , YWHAZ et TBP.
REMARQUE: L’exécution et l’analyse qPCR peuvent être effectuées à l’aide de différentes machines PCR et de logiciels compatibles, en fonction de ce qui est disponible et optimisé en laboratoire. Voir le tableau supplémentaire S1 pour les séquences d’amorces.
Access restricted. Please log in or start a trial to view this content.
Résultats
Pour tester la reproductibilité de cette méthode, la méthode a d’abord été validée en utilisant uniquement des SMC de contrôle. Pour déterminer la reproductibilité des mesures interexpérimentaires, deux mesures indépendantes de toutes les lignées cellulaires de contrôle et de patients incluses ont été tracées sous la forme d’un diagramme de Bland-Altman (figure 3B). Le graphique a démontré que cette méthode ne montre pas de variabilité en dehors de l’intervalle de c...
Access restricted. Please log in or start a trial to view this content.
Discussion
Cet article présente une méthode pour mesurer la contraction de la SMC in vitro, basée sur les changements d’impédance et d’occupation de surface. Tout d’abord, l’isolement, la culture et l’expansion des SMC et des fibroblastes cutanés humains primaires spécifiques au patient sont décrits, suivis de la façon de les utiliser pour les mesures de contraction.
Une limitation de l’étude est liée à l’obtention des cellules par le biais d’un protocole d’explanta...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à déclarer.
Remerciements
Nous tenons à remercier Tara van Merrienboer, Albert van Wijk, Jolanda van der Velden, Jan D. Blankensteijn, Lan Tran, Peter L. Hordijk, l’équipe PAREL-AAA et tous les chirurgiens vasculaires de l’UMC d’Amsterdam, du Zaans Medisch Centrum et de l’hôpital Dijklander pour avoir fourni du matériel et du soutien à cette étude.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 96-well Array | Applied Biophysics | 96W10idf PET | Array used to measure contraction in the ECIS setup |
| Custodiol | Dr. Franz Höhler Chemie GmbH | RVG 12801 | Solution used to transfer tissue in from surgery room to laboratorium |
| Dimethyl sulfoxide | Sigma-Aldrich | 472301 | Solution used to dilute ionomycin |
| Fetal Bovine Serum | Gibco | 26140079 | Addition to cell culture medium |
| Ham's F-10 Nutrient Mix | Gibco | 11550043 | Medium used to culture skin fibroblasts |
| Human Vascular Smooth Muscle Cell Basal Medium (formerly ''Medium 231'') | Gibco | M231500 | Medium used to culture smooth muscle cells |
| Invitrogen countess II | Thermo Fisher Scientific | AMQAX1000 | Automated cell counter |
| Ionomycin calcium salt from Streptomyces conglobatus | Sigma-Aldrich | I0634-1MG | Compound used for contraction stimulation |
| NaCl 0.9% | Fresenius Kabi | B230561 | Solution used to transfer tissue in from surgery room to laboratorium |
| Penicillin-Streptomycin | Gibco | 15140122 | Antibiotics used for cell culture medium |
| Phospathe buffered saline | Gibco | 10010023 | Used to wash cells |
| Quick-RNA Miniprep Kit | Zymo Research | R1055 | Kit used for RNA isolation |
| Smooth Muscle Growth Supplement (SMGS) | Gibco | S00725 | Supplement which is added to smooth muscle cell culture medium |
| SuperScript VILO cDNA Synthesis Kit | Thermo Fisher Scientific | 11754250 | Kit used for cDNA synthesis |
| SYBR Green PCR Master Mix | Thermo Fisher Scientific | 4309155 | Reagent for qPCR |
| Trypsin-EDTA | Gibco | 15400-054 | Used to trypsinize cells |
| ZTheta | Applied Biophysics | ZTheta | ECIS instrument used for contraction measurements |
Références
- Milewicz, D. M., et al. Genetic basis of thoracic aortic aneurysms and dissections: focus on smooth muscle cell contractile dysfunction. Annual Review of Genomics and Human Genetics. 9, 283-302 (2008).
- Milewicz, D. M., et al. Altered smooth muscle cell force generation as a driver of thoracic aortic aneurysms and dissections. Arteriosclerosis, Thrombosis, and Vascular Biology. 37 (1), 26-34 (2017).
- Groeneveld, M. E., et al. Betaglycan (TGFBR3) up-regulation correlates with increased TGF-β signaling in Marfan patient fibroblasts in vitro. Cardiovascular Pathology. 32, 44-49 (2018).
- Chen, J., Li, H., SundarRaj, N., Wang, J. H. C. Alpha-smooth muscle actin expression enhances cell traction force. Cell Motility and the Cytoskeleton. 64 (4), 248-257 (2007).
- Peyton, S. R., Putnam, A. J. Extracellular matrix rigidity governs smooth muscle cell motility in a biphasic fashion. Journal of Cellular Physiology. 204 (1), 198-209 (2005).
- Williams, D. A., Fogarty, K. E., Tsien, R. Y., Fay, F. S. Calcium gradients in single smooth muscle cells revealed by the digital imaging microscope using Fura-2. Nature. 318 (6046), 558-561 (1985).
- Wu, D., et al. NLRP3 (nucleotide oligomerization domain-like receptor family, pyrin domain containing 3)-caspase-1 inflammasome degrades contractile proteins: implications for aortic biomechanical dysfunction and aneurysm and dissection formation. Arteriosclerosis, Thrombosis, and Vascular Biology. 37 (4), 694-706 (2017).
- Bogunovic, N., et al. Impaired smooth muscle cell contractility as a novel concept of abdominal aortic aneurysm pathophysiology. Scientific Reports. 9 (1), 1-14 (2019).
- Hurst, V., Goldberg, P. L., Minnear, F. L., Heimark, R. L., Vincent, P. A. Rearrangement of adherens junctions by transforming growth factor-β1: role of contraction. American Journal of Physiology-Lung Cellular and Molecular Physiology. 276 (4), 582-595 (1999).
- Hu, N., et al. Comparison between ECIS and LAPS for establishing a cardiomyocyte-based biosensor. Sensors and Actuators B: Chemical. 185, 238-244 (2013).
- Peters, M. F., Lamore, S. D., Guo, L., Scott, C. W., Kolaja, K. L. Human stem cell-derived cardiomyocytes in cellular impedance assays: bringing cardiotoxicity screening to the front line. Cardiovascular Toxicology. 15 (2), 127-139 (2015).
- Zhang, S., Yang, Y., Kone, B. C., Allen, J. C., Kahn, A. M. Insulin-stimulated cyclic guanosine monophosphate inhibits vascular smooth muscle cell migration by inhibiting Ca/calmodulin-dependent protein kinase II. Circulation. 107 (11), 1539-1544 (2003).
- Halterman, J. A., Kwon, H. M., Zargham, R., Bortz, P. D. S., Wamhoff, B. R. Nuclear factor of activated T cells 5 regulates vascular smooth muscle cell phenotypic modulation. Arteriosclerosis, Thrombosis, and Vascular Biology. 31 (10), 2287-2296 (2011).
- Bass, H. M., Beard, R. S., Cha, B. J., Yuan, S. Y., Nelson, P. R. Thrombomodulin induces a quiescent phenotype and inhibits migration in vascular smooth muscle cells in vitro. Annals of Vascular Surgery. 30, 149-156 (2016).
- Burger, J., et al. Molecular phenotyping and functional assessment of smooth muscle like-cells with pathogenic variants in aneurysm genes ACTA2, MYH11, SMAD3 and FBN1. Human Molecular Genetics. , (2021).
- Yeung, K. K., et al. Transdifferentiation of human dermal fibroblasts to smooth muscle-like cells to study the effect of MYH11 and ACTA2 mutations in aortic aneurysms. Human Mutation. 38 (4), 439-450 (2017).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon