É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Isolamento de células musculares lisas aórticas específicas do paciente primário e medidas de contração em tempo real semiquantitativas in vitro
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Este artigo descreve um método baseado em cultura explant para o isolamento e cultivo de células musculares humanas aórticas humanas primárias e específicas do paciente e fibroblastos dérmicos. Além disso, é apresentado um novo método para medir a contração celular e a análise subsequente, que pode ser usado para estudar diferenças específicas do paciente nessas células.
Resumo
As células musculares lisas (SMCs) são o tipo de célula predominante na mídia aórtica. Sua máquina de fundição é importante para a transmissão da força na aorta e regula a vasoconstrição e vasodilatação. Mutações em genes codificados para as proteínas do aparelho contítil do SMC estão associadas a doenças aórticas, como aneurismas de aoórtico torácico. Medir a contração de SMC in vitro é desafiador, especialmente de forma de alto rendimento, o que é essencial para a triagem do material do paciente. Atualmente, os métodos disponíveis não são adequados para este fim. Este artigo apresenta um novo método baseado no sensor de impedância de células elétricas (ECIS). Primeiro, um protocolo de explant é descrito para isolar os SMCs primários humanos específicos do paciente de biópsias aórticas e fibroblastos dérmicos humanos específicos do paciente para o estudo de aneurismas àórtica. Em seguida, é dada uma descrição detalhada de um novo método de contração para medir a resposta contratil dessas células, incluindo a análise subsequente e sugestão para comparar diferentes grupos. Este método pode ser utilizado para estudar a contração de células aderentes no contexto de estudos translacionais (cardiovasculares) e estudos de triagem de pacientes e medicamentos.
Introdução
As células musculares lisas (SMCs) são o tipo de célula predominante na camada medial aórtica, a camada mais grossa da aorta. Dentro da parede, eles são radialmente orientados e estão envolvidos, entre outras funções, vasoconstrição e vasodilatação1. O maquinário contratado da SMC está envolvido na transmissão de força na aorta através do vínculo funcional com a matriz extracelular2. Mutações em genes codificados para as proteínas do aparelho contítil SMC, como a cadeia pesada de miosina muscular lisa (MYH11) e a actina muscular lisa (ACTA2), têm sido relacionadas a casos de aneurismas aórticos torácicos familiares, ressaltando a relevância da contração do SMC na manutenção da integridade estrutural e funcional da aorta 1,2 . Além disso, mutações na via de sinalização TGFβ também estão associadas a aneurismas de aoórtico, e seus efeitos na fisiopatologia aneurisma aórtica também podem ser estudados em fibroblastos de pele3.
A medição de alta produtividade da contração SMC in vitro é desafiadora. Como a contratude do SMC não pode ser medida in vivo em humanos, ensaios in vitro em células humanas apresentam uma alternativa viável. Além disso, o desenvolvimento do aneurisma de aoórtico abdominal (AAA) em modelos animais é quimicamente induzido por, por exemplo, perfusão elastase, ou causada por uma mutação específica. Portanto, os dados animais não são comparáveis ao desenvolvimento de AAA em humanos, que tem principalmente uma causa multifatorial, como tabagismo, idade e/ou aterosclerose. In vitro A contratilidade da SMC tem sido medida principalmente pela microscopia de forçade tração 4,5, quantificação dos fluxos de cálcio intracelular6 da Fluorescência Fura-2 e pelos ensaios de enrugamento de colágeno7. Embora a microscopia de força de tração forneça uma visão numérica inestimável das forças geradas por uma única célula, ela não é adequada para a triagem de alto rendimento devido ao complexo processamento de dados matemáticos e à análise de uma célula de cada vez, o que significa que é muito demorado medir um número representativo de células por doador. Os ensaios de enrugação de corante e colágeno fura-2 permitem a determinação superficial da contração e não dão uma saída numérica precisa, tornando-os menos adequados para discriminar diferenças específicas do paciente. A contração prejudicada do SMC em células derivadas da aorta de pacientes com aneurisma de aorta abdominal foi demonstrada pela primeira vez pela otimização de um novo método para medir a contração de SMC in vitro8. Isso foi feito redefinindo o método de sensor de sensoriamento de impedância de células elétricas (ECIS). O ECIS é um ensaio de média-produção em tempo real para quantificação do comportamento celular aderente e contração 9,10,11, como o crescimento e o comportamento do SMC na cicatrização de feridas e ensaios migratórios 12,13,14. O método exato está descrito na seção de protocolo. Dessa forma otimizada, o ECIS também pode ser usado para estudar a contração do fibroblasto devido ao seu tamanho e morfologia semelhantes.
O objetivo deste artigo é fornecer uma descrição passo a passo do método de medição da contração de SMC in vitro usando ECIS8 e comparando a contração entre controle e SMCs do paciente. Em primeiro lugar, explica-se o isolamento e o cultivo de SMCs primários a partir do controle e biópsias aórticas do paciente, que podem ser utilizadas para a medição da contração. Em segundo lugar, são descritas medidas de contração e análise, juntamente com a verificação da expressão do marcador SMC. Além disso, este artigo descreve o método de isolamento de fibroblastos dérmicos específicos do paciente cuja contração pode ser medida utilizando a mesma metodologia. Essas células podem ser usadas para estudos específicos do paciente focados em aneurisma de aorta ou outras patologias cardiovasculares15 ou estudos prognósticos usando um protocolo de transdiferenciação que permite a medição da contração antes da cirurgia de aneurisma16.
Protocolo
NOTA: Biópsias aórticas foram obtidas durante reparo de aneurisma aberto em Amsterdam University Medical Centers, VU University Medical center, Amsterdam, Zaans Medisch Centrum, Zaandam e Dijklander hospital, Hoorn, holanda. O tecido aórtico de controle foi obtido a partir do pedaço da aorta ligado à artéria renal colhida para transplantes renais. Apenas pacientes acima de 18 anos foram incluídos, e todos os pacientes deram seu consentimento informado para participar do estudo. Todo o material foi coletado de acordo com as normas da Declaração de Helsinque da WMA e as diretrizes institucionais do Comitê de Ética Médica do Centro Médico da VU. Todos os experimentos e protocolos experimentais foram realizados de acordo com as diretrizes institucionais e aprovados pelo Comitê de Ética Médica do Centro Médico da VU. Para obter informações completas sobre o controle e as linhas celulares do paciente utilizadas, consulte 8.
1. Isolando SMCs humanos primários de biópsias aórticas
NOTA: Execute as seguintes etapas sob uma capa de fluxo laminar de cultura de tecido estéril. Use luvas e use técnicas assépticas padrão ao manusear amostras de sangue humano e tecido humano. As SMCs são cultivadas em 231 Músculos Lisos Vasculares Humanos Médio Basal complementado com penicilina de 100 U/mL, 100 μg/mL estreptomicina e Suplemento de Crescimento Muscular Liso referido como meio SMC.
- Esterilize dois pares de fórceps cirúrgicos e um bisturi, imergindo-os em 70% de etanol e, posteriormente, limpando-os secos.
- Pipeta 2 mL de meio SMC em uma placa de Petri na qual será realizada a dissecção tecidual.
- Pipeta 2,5 mL de meio SMC em dois frascos T25. Gire os frascos ao redor para que o pequeno volume de meio cubra toda a superfície.
- Transporte a biópsia de parede aórtica humana colhida da sala de cirurgia para o laboratório em gelo em um tubo plástico estéril com solução de transferência de tecido frio e estéril (ver a Tabela de Materiais) ou 0,9% NaCl.
- Abra o tubo com o tecido dentro da capa da cultura tecidual. Tire a biópsia do tubo usando os fórceps esterilizados e coloque-a na placa de Petri (Figura 1A).
- Inspecione visualmente a biópsia para identificar as três camadas aórticas, a tunica intima (interior), mídia (meio) e adventitia (camada externa). Procure a presença de placas ateroscleróticas no lado intimado e tecido conjuntivo viscoso no lado advento (Figura 1B) para distinguir as camadas.
- Para isolar os SMCs da mídia, remova as outras duas camadas. Coloque o tecido com o lado da placa intima primeiro (Figura 1B). Remova a placa sólida puxando-a para longe do tecido com fórceps enquanto empurra o tecido para baixo com o outro par de fórceps. Remova as camadas subsequentes da placa até que a camada medial rosa e uniforme seja visível.
- Vire o tecido (Figura 1C). Repita o mesmo procedimento, como na etapa 1.7, puxando a camada aventurra (Figura 1D). Certifique-se de remover todas as partes visíveis em quantas tentativas for necessário, pois esta camada não se desprende facilmente da mídia.
NOTA: É essencial que as camadas intimais e aventurárias sejam removidas adequadamente para obter o mais limpo possível uma população de SMC. - Uma vez que a camada medial esteja isolada, corte o tecido em cubos de aproximadamente 1 mm x 1 mm x 1 mm. Pressione a mídia para baixo com fórceps e corte o tecido em uma direção usando o bisturi. Não corte para frente e para trás; fazer cortes limpos e unidirecionais para minimizar os danos. Tente fazer o máximo de cubos possível, dado o tamanho da biópsia (Figura 1E).
- Coloque os pedaços de tecido no quarto superior do frasco T25 usando os fórceps. Coloque 10-20 cubos por frasco se a quantidade de material permitir (Figura 1F).
NOTA: Use fórceps lisos para minimizar a adesão do tecido às costelas dos fórceps e desprender facilmente o tecido no frasco. - Incubar os cubos de tecido nos frascos T25 por aproximadamente 10 dias a 5% de CO2 a 37 °C em uma incubadora.
NOTA: O crescimento da primeira célula é esperado por volta desse tempo. As células que migram inicialmente podem parecer menores do que os SMCs normais. - Uma vez observado o crescimento celular, adicione 2,5 mL de médio ao frasco, certificando-se de não pipetá-lo sobre as peças de tecido para evitar que se desprendem.
- Após aproximadamente mais 5 dias, quando são observados aglomerados de células ao redor dos pedaços de tecido, mude o meio de cultura. Se os pedaços de tecido se soltarem, remova-os, pois não se recolocarão.
- Uma vez que as células são 80-90% confluentes, transfira-as para um frasco T75 e continue a culminar neste formato.
NOTA: Recomenda-se uma relação de divisão de 1:2 ou 1:3. Congele um backup das células em uma passagem antecipada. As células geralmente mantêm suas propriedades até a passagem 10; passagens posteriores não devem ser usadas para experimentos.
2. Isolando fibroblastos dérmicos primários de biópsias cutâneas
NOTA: Execute as seguintes etapas sob uma capa de fluxo laminar de cultura de tecido estéril. Use luvas e use técnicas assépticas padrão ao manusear amostras de sangue humano e tecido humano. Os fibroblastos são cultivados em Meio Basal suplementado com soro bovino fetal de 10%, penicilina U/mL de 100 u/mL e 100 μg/mL estreptomicina, referida como meio fibroblasto.
- Esterilize dois pares de fórceps cirúrgicos e um bisturi mergulhando-os em 70 % de etanol e, posteriormente, limpando-os secos.
- Pipeta 2 mL de meio fibroblasto em uma placa de Petri na qual será realizada a dissecção tecidual.
- Pipeta 2,5 mL de meio fibroblasto em dois frascos T25. Gire os frascos ao redor para que o pequeno volume de meio cubra toda a superfície.
- Transporte a biópsia da pele colhida da sala de cirurgia para o laboratório em gelo em solução fria e estéril de transferência de tecido (ver a Tabela de Materiais) ou 0,9% NaCl em um tubo plástico estéril.
- Abra o tubo com o tecido dentro da capa da cultura tecidual. Tire a biópsia do tubo usando os fórceps esterilizados e coloque-a na placa de Petri (Figura 2A).
- Inspecione visualmente a biópsia para identificar as três camadas de pele, a epiderme, a derme e a gordura subcutânea. Procure uma superfície de pele reconhecível, às vezes com cabelo visível, para identificar a epiderme. No lado oposto, procure a gordura subcutânea, que muitas vezes é amarela e viscosa. Identifique a camada entre a epiderme e a gordura subcutânea como a derme - a fonte de fibroblastos viáveis (Figura 2B).
- Para isolar os fibroblastos da dermis, remova as outras duas camadas e coloque o tecido de lado para que todas as três camadas sejam visíveis.
NOTA: Ao contrário do tecido aórtico, as camadas de pele não podem ser separadas umas das outras; portanto, eles devem ser cortados. O tecido também é mais emborrachado, tornando mais difícil cortar. Use um bisturi afiado. - Segure o tecido com fórceps. Corte paralelamente à fronteira entre a epiderme e a derme. Corte toda a epiderme. Tente cortar em uma linha limpa e não se mova para frente e para trás para evitar danos teciduais.
- Vire o tecido. Repetir o mesmo procedimento da etapa 2.8; desta vez, corte dentro da dermis paralela à fronteira com a gordura subcutânea.
- Uma vez que a dermis esteja isolada, corte o tecido em cubos aproximadamente 1 x 1 x 1 mm3. Pressione o tecido para baixo com fórceps e corte o tecido em uma direção usando o bisturi. Não corte para frente e para trás; fazer cortes limpos e unidirecionais para minimizar os danos. Tente fazer o máximo de cubos possível, dado o tamanho da biópsia (Figura 2C).
- Coloque os pedaços de tecido no quarto superior do frasco T25 usando os fórceps. Coloque 10-20 cubos por frasco se a quantidade de material permitir (Figura 2D).
NOTA: Use fórceps lisos para minimizar a adesão do tecido às costelas dos fórceps e desprender facilmente o tecido no frasco. - Incubar os cubos de tecido nos frascos T25 por aproximadamente 10 dias a 5% de CO2 a 37 °C em uma incubadora.
NOTA: O primeiro crescimento da célula é esperado por volta de então. As células que migram inicialmente podem parecer menores do que os fibroblastos normais. - Uma vez observado o crescimento celular, adicione 2,5 mL de médio ao frasco, certificando-se de não pipetá-lo sobre as peças de tecido para evitar que se desprendem.
- Após aproximadamente mais 5 dias, quando grupos de células podem ser observados ao redor dos pedaços de tecido, mude o meio de cultura. Se os pedaços de tecido se soltarem, remova-os, pois não se recolocarão.
- Uma vez que as células são 80-90% confluentes, transfira-as para um frasco T75 e continue a culminar neste formato.
NOTA: Recomenda-se uma relação de divisão de 1:2 ou 1:3. Congele um backup das células em uma passagem antecipada. As células geralmente mantêm suas propriedades até a passagem 10; passagens posteriores não devem ser usadas para experimentos.
3. Medição da contração (exemplo SMCs)
- Prepare uma matriz ECIS 96w10e (ver Figura 3A para uma imagem representativa da matriz com eletrodos ampliados e células semeadas nos eletrodos).
NOTA: Execute as seguintes etapas sob uma capa de fluxo laminar de cultura de tecido estéril.- Cubra uma matriz estéril de 96w10e com 200 μL de 10 mM L-cisteína por poço por 30 min a temperatura ambiente.
- Lave a placa duas vezes com água estéril. Deixe a placa secar durante a noite na capa da cultura tecidual com a tampa ligeiramente aberta.
NOTA: O revestimento da placa com L-cisteína só é necessário ao usar a placa pela primeira vez. - No dia seguinte, placa esterilizada UV e tampa por 30 minutos. Gire a tampa para cima para que o interior seja esterilizado. Uma vez que a placa esteja esterilizada, não abra a placa fora do capô de fluxo.
- Células de semeadura na matriz ECIS
- Solução de gelatina pré-quente 1% estéril no banho de água por 30 minutos.
NOTA: A solução é armazenada na geladeira e pode se solidificar, dificultando a pipetria. - Posteriormente, cubra os poços com 100 μL da solução de gelatina de 1 % por poço e incubar a placa a 37 °C por pelo menos 1 h.
- Aspire a gelatina dos poços.
- Conte as células usando um contador celular automatizado e semee os SMCs em triplicado a uma densidade de semeadura de 30.000 células/poço em 200 μL de meio SMC.
- Coloque a placa com os SMCs no suporte de poçoS ECIS 96 na incubadora de cultura celular. Clique duas vezes no software de biofísica aplicada ECIS para abrir o programa e pressionar o botão Configuração .
- Verifique se todos os eletrodos têm contato com o suporte (verde; vermelho se não houver conexão) no painel inferior esquerdo rotulado de Configuração de Poço. Se os eletrodos não estiverem conectados corretamente, ajuste a placa no suporte antes de iniciar a medição.
- Selecione o tipo de placa (matriz de 96 poços) no mesmo painel clicando no tipo Array.
- Na configuração de coleta de dados do painel superior direito, clique em uma única frequência e altere o valor de impedância para 4000 Hz e o intervalo para 8 s.
- Clique no botão Iniciar para iniciar as medições. Aguarde a abertura de um novo painel, onde o arquivo ECIS possa ser salvo.
- Permita que as células se conectem e estabeleçam uma monocamada por 48 h.
NOTA: O acessório e a disseminação de células nos eletrodos geram um valor de resistência da linha de base (Figura 3B).
- Solução de gelatina pré-quente 1% estéril no banho de água por 30 minutos.
- Estimulação das células para induzir contração
- Induzir a contração do SMC estimulando as células com 10 μg/mL do ionophore de cálcio, ionomicina.
NOTA: A ionomicina induzirá o influxo de Ca2+extracelular, ativando a cascata contractiêile; ver Figura 4A para imagens representativas de células contraídas não estimuladas e estimuladas. - Diluir 1 mg de pó de ionomicina em 100 μL de dimetilsulfoxida para fazer uma solução de estoque de 10 mg/mL, e armazenar 10 alíquotas de μL a -20 °C. Antes de usar, diluir a solução de ionomicina 1:10 em meio SMC para preparar a solução de trabalho a ser adicionada às células.
- Realize a estimulação celular enquanto a matriz ainda está no suporte de matriz dentro da incubadora de cultura celular e os eletrodos estão ligados ao sistema.
- Para estimular as células, remova a tampa e coloque-a em uma superfície estéril dentro da incubadora. Prepare duas pipetas, fixas em 2 μL e 150 μL.
- Antes de iniciar a estimulação, pressione Mark no software e coloque um comentário.
NOTA: Isso facilitará a localização do tempo exato da estimulação ao analisar os dados. - Pipeta 2 μL da solução de trabalho de ionomicina em cada um, bem o mais rápido possível. Uma vez estimuladas todas as células, misture o meio nos poços usando a segunda pipeta.
NOTA: Devido aos efeitos rápidos, não é necessário alterar dicas entre diferentes linhas celulares e condições. Trabalhe rapidamente enquanto estimula e mistura porque a ionomicina tem um efeito agudo. Ao estimular uma placa completa, estimule um máximo de 3 colunas ao mesmo tempo. Após cada estimulação, espere pelo menos 30 minutos até a próxima estimulação para deixar as células se recuperarem da temperatura e da troca de CO2 causada pela abertura da porta da incubadora. - Logo após o estímulo ser feito, pressione Mark novamente para adicionar um comentário de que a estimulação é feita.
- Terminando o experimento de estimulação
- Regisso dos valores de impedância por aproximadamente 5 minutos após a estimulação da ionomicina. Termine a gravação pressionando 'Concluir'.
- Reutilize as matrizes ECIS até três vezes: lave os poços duas vezes com água estéril e incuba a placa a 37 °C por 2-3 h com trippsina ou um reagente similar. Repetir as etapas 3.1.2 e 3.1.3.
- Induzir a contração do SMC estimulando as células com 10 μg/mL do ionophore de cálcio, ionomicina.
- Exportando os dados
- Para exportar os dados, clique em Arquivo | Dados de exportação | Para Excel (Selecionado). Selecione uma pasta para salvar o arquivo.
- Clique em Separar quando o programa pedir para combinar ou separar folhas.
- Analisando a saída de contrato
- Para calcular a resposta contratil, utilize a seguinte equação (1), onde C indica contração (expressa em % de alteração em relação à linha de base), a pré-estimulação (RS) indica o valor de resistência (expresso em Ω) pouco antes da estimulação da ionomicina e pós-estimulação (SD) indica a resistência (expressa em Ω) 2 minutos após o término da estimulação.
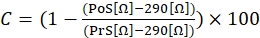 (1)
(1)
NOTA: Como mostrado na equação (1), o valor de impedância de um poço cheio de meio de cultura sem qualquer célula anexada (valor de 290 Ω) é subtraído de todos os resultados no cálculo final. Recomenda-se medir as respostas contratuais em triplicados em cada experimento e realizar três experimentos independentes com a mesma linha celular para explicar qualquer variação inter e intra-experimental. - Uma vez realizados os três experimentos independentes, agrupar os dados calculando a média das três medidas, incluindo o desvio padrão (SD).
- Para calcular a resposta contratil, utilize a seguinte equação (1), onde C indica contração (expressa em % de alteração em relação à linha de base), a pré-estimulação (RS) indica o valor de resistência (expresso em Ω) pouco antes da estimulação da ionomicina e pós-estimulação (SD) indica a resistência (expressa em Ω) 2 minutos após o término da estimulação.
- Comparando diferentes grupos
- Para definir o intervalo de contração normal e investigar questões específicas subsequentes de pesquisa, utilize o grupo controle para definir a "contração normal" e compará-la com a resposta contratil de pacientes ou grupos de tratamento.
- Calcule a média e a SD de todo o grupo controle, ou seja, dos valores finais para todas as linhas celulares, conforme descrito na etapa 3.5.2.
- Use a faixa da média ± 2SD para identificar células do outro grupo que estão fora dessa faixa, indicando que elas têm uma resposta contraitil alterada.
NOTA: O mecanismo por trás da resposta contraída alterada está sujeito a questões específicas de pesquisa e é dependente de células e condições e não será discutido neste protocolo.
4. Detectando a presença de marcadores específicos do SMC
- Isolamento do RNA
- Sementes as mesmas linhas celulares específicas do paciente usadas para as medidas de contração em placas de 6 poços, uma bem por linha celular. Conte as células usando um contador celular automatizado e semee as células a uma densidade de semeadura de 200.000 células/poço no meio SMC e permita que elas se conectem durante a noite.
- Lave as células uma vez com PBS estéril.
- Lise as células e isole as células de acordo com as instruções do fabricante.
- Síntese cDNA
- Realize a síntese de cDNA em 20 μL de uma mistura de reação de transcrição reversa. Diluir as concentrações do RNA total isolado de acordo com as instruções fornecidas pelo fabricante.
- qPCR
- Meça a expressão genética dos genes marcadores SMC, por exemplo, ACTA2, CNN1, SMTN e TAGLN para confirmar que as células isoladas são de fato SMCs e detectam marcadores SMC. Verifique se há correlação dos resultados com a saída de contrato.
- Use pelo menos dois genes de limpeza para normalizar os dados, por exemplo, YWHAZ e TBP.
NOTA: A execução e análise do qPCR podem ser realizadas utilizando diferentes máquinas PCR e software compatível, dependendo do que estiver disponível e otimizado em laboratório. Consulte a tabela suplementar S1 para obter sequências de primer.
Resultados
Para testar a reprodutibilidade deste método, o método foi validado apenas utilizando SMCs de controle. Para determinar a reprodutibilidade da medição interexperimental, duas medidas independentes de todas as linhas de controle e células do paciente foram traçadas como um enredo Bland-Altman (Figura 3B). O enredo demonstrou que este método não mostra variabilidade fora do intervalo de confiança, exceto por uma linha celular outlier. Além disso, esses resultados demonstraram que doi...
Discussão
Este artigo apresenta um método para medir a contração do SMC in vitro, com base nas mudanças de impedância e ocupação superficial. Em primeiro lugar, descreve-se o isolamento, a culminação e a expansão de SMCs humanos primários específicos do paciente e fibroblastos de pele, seguidos de como usá-los para medidas de contração.
Uma limitação do estudo está relacionada à obtenção das células através de um protocolo de explant. As células que se proliferam da bi?...
Divulgações
Os autores não têm conflitos de interesse para declarar.
Agradecimentos
Gostaríamos de reconhecer com gratidão Tara van Merrienboer, Albert van Wijk, Jolanda van der Velden, Jan D. Blankensteijn, Lan Tran, Peter L. Hordijk, a equipe DO PAREL-AAA, e todos os cirurgiões vasculares do UmC de Amsterdã, Zaans Medisch Centrum e Dijklander por fornecerem materiais e apoio para este estudo.
Materiais
| Name | Company | Catalog Number | Comments |
| 96-well Array | Applied Biophysics | 96W10idf PET | Array used to measure contraction in the ECIS setup |
| Custodiol | Dr. Franz Höhler Chemie GmbH | RVG 12801 | Solution used to transfer tissue in from surgery room to laboratorium |
| Dimethyl sulfoxide | Sigma-Aldrich | 472301 | Solution used to dilute ionomycin |
| Fetal Bovine Serum | Gibco | 26140079 | Addition to cell culture medium |
| Ham's F-10 Nutrient Mix | Gibco | 11550043 | Medium used to culture skin fibroblasts |
| Human Vascular Smooth Muscle Cell Basal Medium (formerly ''Medium 231'') | Gibco | M231500 | Medium used to culture smooth muscle cells |
| Invitrogen countess II | Thermo Fisher Scientific | AMQAX1000 | Automated cell counter |
| Ionomycin calcium salt from Streptomyces conglobatus | Sigma-Aldrich | I0634-1MG | Compound used for contraction stimulation |
| NaCl 0.9% | Fresenius Kabi | B230561 | Solution used to transfer tissue in from surgery room to laboratorium |
| Penicillin-Streptomycin | Gibco | 15140122 | Antibiotics used for cell culture medium |
| Phospathe buffered saline | Gibco | 10010023 | Used to wash cells |
| Quick-RNA Miniprep Kit | Zymo Research | R1055 | Kit used for RNA isolation |
| Smooth Muscle Growth Supplement (SMGS) | Gibco | S00725 | Supplement which is added to smooth muscle cell culture medium |
| SuperScript VILO cDNA Synthesis Kit | Thermo Fisher Scientific | 11754250 | Kit used for cDNA synthesis |
| SYBR Green PCR Master Mix | Thermo Fisher Scientific | 4309155 | Reagent for qPCR |
| Trypsin-EDTA | Gibco | 15400-054 | Used to trypsinize cells |
| ZTheta | Applied Biophysics | ZTheta | ECIS instrument used for contraction measurements |
Referências
- Milewicz, D. M., et al. Genetic basis of thoracic aortic aneurysms and dissections: focus on smooth muscle cell contractile dysfunction. Annual Review of Genomics and Human Genetics. 9, 283-302 (2008).
- Milewicz, D. M., et al. Altered smooth muscle cell force generation as a driver of thoracic aortic aneurysms and dissections. Arteriosclerosis, Thrombosis, and Vascular Biology. 37 (1), 26-34 (2017).
- Groeneveld, M. E., et al. Betaglycan (TGFBR3) up-regulation correlates with increased TGF-β signaling in Marfan patient fibroblasts in vitro. Cardiovascular Pathology. 32, 44-49 (2018).
- Chen, J., Li, H., SundarRaj, N., Wang, J. H. C. Alpha-smooth muscle actin expression enhances cell traction force. Cell Motility and the Cytoskeleton. 64 (4), 248-257 (2007).
- Peyton, S. R., Putnam, A. J. Extracellular matrix rigidity governs smooth muscle cell motility in a biphasic fashion. Journal of Cellular Physiology. 204 (1), 198-209 (2005).
- Williams, D. A., Fogarty, K. E., Tsien, R. Y., Fay, F. S. Calcium gradients in single smooth muscle cells revealed by the digital imaging microscope using Fura-2. Nature. 318 (6046), 558-561 (1985).
- Wu, D., et al. NLRP3 (nucleotide oligomerization domain-like receptor family, pyrin domain containing 3)-caspase-1 inflammasome degrades contractile proteins: implications for aortic biomechanical dysfunction and aneurysm and dissection formation. Arteriosclerosis, Thrombosis, and Vascular Biology. 37 (4), 694-706 (2017).
- Bogunovic, N., et al. Impaired smooth muscle cell contractility as a novel concept of abdominal aortic aneurysm pathophysiology. Scientific Reports. 9 (1), 1-14 (2019).
- Hurst, V., Goldberg, P. L., Minnear, F. L., Heimark, R. L., Vincent, P. A. Rearrangement of adherens junctions by transforming growth factor-β1: role of contraction. American Journal of Physiology-Lung Cellular and Molecular Physiology. 276 (4), 582-595 (1999).
- Hu, N., et al. Comparison between ECIS and LAPS for establishing a cardiomyocyte-based biosensor. Sensors and Actuators B: Chemical. 185, 238-244 (2013).
- Peters, M. F., Lamore, S. D., Guo, L., Scott, C. W., Kolaja, K. L. Human stem cell-derived cardiomyocytes in cellular impedance assays: bringing cardiotoxicity screening to the front line. Cardiovascular Toxicology. 15 (2), 127-139 (2015).
- Zhang, S., Yang, Y., Kone, B. C., Allen, J. C., Kahn, A. M. Insulin-stimulated cyclic guanosine monophosphate inhibits vascular smooth muscle cell migration by inhibiting Ca/calmodulin-dependent protein kinase II. Circulation. 107 (11), 1539-1544 (2003).
- Halterman, J. A., Kwon, H. M., Zargham, R., Bortz, P. D. S., Wamhoff, B. R. Nuclear factor of activated T cells 5 regulates vascular smooth muscle cell phenotypic modulation. Arteriosclerosis, Thrombosis, and Vascular Biology. 31 (10), 2287-2296 (2011).
- Bass, H. M., Beard, R. S., Cha, B. J., Yuan, S. Y., Nelson, P. R. Thrombomodulin induces a quiescent phenotype and inhibits migration in vascular smooth muscle cells in vitro. Annals of Vascular Surgery. 30, 149-156 (2016).
- Burger, J., et al. Molecular phenotyping and functional assessment of smooth muscle like-cells with pathogenic variants in aneurysm genes ACTA2, MYH11, SMAD3 and FBN1. Human Molecular Genetics. , (2021).
- Yeung, K. K., et al. Transdifferentiation of human dermal fibroblasts to smooth muscle-like cells to study the effect of MYH11 and ACTA2 mutations in aortic aneurysms. Human Mutation. 38 (4), 439-450 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados