Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Tomographie cardiaque à émission de positons à haute résolution/tomodensitométrie pour petits animaux
Dans cet article
Résumé
Nous présentons ici un protocole d’imagerie expérimentale pour la quantification de la fonction cardiaque et de la morphologie à l’aide de la tomographie par émission de positons à haute résolution / tomodensitométrie pour les petits animaux. Les souris et les rats sont pris en compte, discutant des différentes exigences des agents de contraste par tomodensitométrie pour les deux espèces.
Résumé
La tomographie par émission de positons (TEP) et la tomodensitométrie (TDM) sont parmi les techniques d’imagerie diagnostique les plus utilisées, et les deux servent à comprendre la fonction cardiaque et le métabolisme. Dans la recherche préclinique, des scanners dédiés avec une sensibilité élevée et une résolution spatio-temporelle élevée sont utilisés, conçus pour faire face aux exigences technologiques exigeantes posées par la petite taille du cœur et la fréquence cardiaque très élevée des souris et des rats. Dans cet article, un protocole bimodal d’imagerie TEP/TDM cardiaque pour des modèles expérimentaux de maladies cardiaques chez la souris et/ou le rat est décrit, de la préparation animale et de l’acquisition et de la reconstruction d’images au traitement et à la visualisation d’images.
En particulier, le 18 fluorodésoxyglucose marqué F ([18F]FDG)-PET permet de mesurer et de visualiser le métabolisme du glucose dans les différents segments du ventricule gauche (VG). Les cartes polaires sont des outils pratiques pour afficher ces informations. La partie CT consiste en une reconstruction 3D résolue dans le temps de l’ensemble du cœur (4D-CT) à l’aide d’un déclenchement rétrospectif sans électrocardiographie (ECG), permettant l’évaluation morphofonctionnelle du VG et la quantification ultérieure des paramètres les plus importants de la fonction cardiaque, tels que la fraction d’éjection (FE) et le volume de l’AVC (SV). À l’aide d’un scanner TEP/TDM intégré, ce protocole peut être exécuté dans la même induction d’anesthésie sans qu’il soit nécessaire de repositionner l’animal entre différents scanners. Par conséquent, la TEP / TDM peut être considérée comme un outil complet pour l’évaluation morphofonctionnelle et métabolique du cœur dans plusieurs modèles animaux de maladies cardiaques.
Introduction
Les modèles de petits animaux sont extrêmement importants pour faire progresser la compréhension des maladies cardiovasculaires 1,2. Les outils d’imagerie diagnostique non invasifs ont révolutionné la façon dont nous envisageons la fonction cardiaque au cours des dernières décennies, tant dans les milieux cliniques que précliniques. En ce qui concerne les modèles de maladies cardiaques chez les petits animaux, des outils d’imagerie spécifiques ont été développés avec une très haute résolution spatio-temporelle. Ainsi, de tels instruments peuvent répondre au besoin de quantification précise des paramètres métaboliques et cinétiques du myocarde pertinents sur les cœurs très petits et très rapides de souris et de rats dans des modèles de maladies spécifiques, tels que l’insuffisance cardiaque (IC)3 ou l’infarctus du myocarde (IM)4. Plusieurs modalités sont disponibles à cet effet, chacune avec ses propres forces et faiblesses. L’imagerie par ultrasons (US) est la modalité la plus utilisée en raison de sa grande flexibilité, de sa très haute résolution temporelle et de son coût relativement faible. L’adoption de l’imagerie cardiaque américaine chez les petits animaux a considérablement augmenté depuis l’avènement des systèmes utilisant des sondes à ultra-haute fréquence5,6, avec des résolutions spatiales inférieures à 50 μm.
L’un des principaux inconvénients de l’US pour l’imagerie cardiaque entièrement 3D est la nécessité de balayages linéaires le long de l’axe cardiaque en montant la sonde sur une platine de translation motorisée pour créer une pile complète d’images dynamiques en mode B de l’ensemble du cœur7. Finalement, cette procédure donne lieu (après enregistrement spatial et temporel précis des images acquises dans chaque position de sonde) à une image 4D avec des résolutions spatiales différentes entre les directions dans le plan et hors plan. Le même problème de résolution spatiale non uniforme se produit dans l’IRM cardiaque (RMC),8 qui représente toujours l’étalon-or dans l’imagerie fonctionnelle du cœur. L’imagerie 3D isotrope réelle peut être obtenue à l’aide de la tomodensitométrie (TDM) et de la tomographie par émission de positons (TEP)9. Le PET fournit un outil très sensible en termes de signal d’image par quantité de sonde injectée (dans la gamme nanomolaire), même s’il souffre d’une résolution spatiale réduite par rapport à CT, MR ou US. Le principal avantage de la TEP est sa capacité à afficher les mécanismes cellulaires et moléculaires sous-jacents à la physiopathologie de l’organe. Par exemple, une TEP après l’injection de [18F]FDG permet la reconstruction d’une carte 3D du métabolisme du glucose dans le corps. En combinant cela avec l’acquisition de données dynamiques (c’est-à-dire résolues dans le temps), la modélisation cinétique du traceur peut être utilisée pour calculer des cartes paramétriques des taux métaboliques d’absorption du glucose (MRGlu), qui fourniront des informations importantes sur la viabilité du myocarde10.
La TDM nécessite des volumes importants d’agents de contraste externes (AC) à des concentrations élevées (jusqu’à 400 mg d’iode par mL) pour fournir une amélioration mesurable des composants tissulaires pertinents (p. ex. sang vs muscle), mais elle excelle dans la résolution spatiale et temporelle, en particulier lors de l’utilisation de micro-tomodensitomètres de pointe conçus pour l’imagerie des petits animaux. 11 Un modèle typique de maladie dans lequel la TEP/TDM cardiaque peut être appliquée est l’évaluation expérimentale de l’infarctus du myocarde et de l’insuffisance cardiaque et de la réponse connexe au traitement. Une façon courante d’induire l’IM chez les petits animaux consiste à procéder à une ligature chirurgicale de l’artère coronaire descendante antérieure gauche (DAL)12,13, puis à évaluer longitudinalement la progression de la maladie et le remodelage cardiaque dans les jours suivants4. Néanmoins, l’évaluation morphofonctionnelle quantitative du cœur chez les petits animaux est largement applicable également à d’autres modèles de maladies, tels que l’évaluation de l’effet du vieillissement sur la fonction cardiaque14 ou l’expression altérée des récepteurs dans des modèles d’obésité15. Le protocole d’imagerie présenté n’est pas limité à un modèle de maladie donné et, par conséquent, pourrait présenter le plus grand intérêt dans plusieurs contextes de recherche préclinique sur de petits rongeurs.
Dans cet article, nous présentons un protocole expérimental du début à la fin pour l’imagerie cardiaque utilisant la TEP/TDM intégrée aux petits animaux. Même si le protocole présenté est conçu pour un scanner intégré bimodal spécifique, les parties TEP et CT de la procédure décrite pourraient être effectuées indépendamment sur des scanners distincts de différents fabricants. Dans le scanner TEP/TDM utilisé, la séquence des opérations est organisée dans un flux de travail préprogrammé. Les branches principales de chaque flux de travail sont un ou plusieurs protocoles d’acquisition ; Chaque protocole d’acquisition peut avoir une ou plusieurs branches pour des protocoles de prétraitement spécifiques, et à son tour, chaque protocole de prétraitement peut avoir une ou plusieurs branches pour des protocoles de reconstruction spécifiques. La préparation de l’animal sur le lit d’imagerie et la préparation des agents externes à injecter pendant les procédures d’imagerie sont décrites. Une fois la procédure d’acquisition d’images terminée, des exemples de procédures d’analyse quantitative d’images basées sur des outils logiciels couramment disponibles sont fournis. Le protocole principal est spécialement conçu pour les modèles de souris ; Même si la souris reste l’espèce la plus utilisée dans ce domaine, nous montrons également une adaptation du protocole pour l’imagerie du rat à la fin du protocole principal. Des résultats représentatifs sont présentés pour les souris et les rats, démontrant le type de résultat auquel on pourrait s’attendre avec les procédures décrites. Une discussion approfondie est faite à la fin de ce document pour souligner les avantages et les inconvénients de la technique, les points critiques, ainsi que la façon dont différents radiotraceurs TEP pourraient être utilisés sans presque aucune modification des étapes préparatoires et d’acquisition / reconstruction.
Protocole
Les expérimentations sur les animaux ont été réalisées conformément aux recommandations du Guide pour le soin et l’utilisation des animaux de laboratoire des Directives internationales sur la manipulation des animaux de laboratoire, exigées par la directive européenne (directive 86/609/CEE de 1986 et directive 2010/63/UE) et les lois italiennes (D.Lgs. 26/2014).
1. Configuration des protocoles d’imagerie TEP/TDM et du flux de travail
REMARQUE: Le protocole présenté ici est spécialement conçu pour l’imagerie cardiaque des modèles murins. Travailler avec des rats peut impliquer certaines modifications du protocole actuel, principalement en raison de la plus grande taille de l’animal (environ 10 fois plus lourd). Les modifications pour l’imagerie du rat sont spécifiquement mentionnées dans les étapes; Si aucune modification n’est mentionnée, les mêmes étapes pour l’imagerie de la souris peuvent être utilisées pour les rats.
- Ouvrez l’interface utilisateur graphique (GUI) du scanner TEP/TDM (voir Tableau des matériaux) et créer un ensemble de nouveaux protocoles (y compris des paramètres pour l’acquisition de données, le prétraitement et la reconstruction d’images): (i) a TEP dynamique, ii) un tomodensitométrie à faible dose pour la correction d’atténuation (Le) sans agent de contraste, et iii) un Ciné-tomodensitométrie à contraste amélioré.
REMARQUE : La création de nouveaux protocoles (c.-à-d. des instructions logicielles spécifiques pour le tomographe) pour les phases d’acquisition, de prétraitement et de reconstruction est un processus simple; en cas de problème, l’utilisateur peut trouver des informations plus détaillées dans le manuel d’utilisation de l’interface graphique.- Pour la TEP, ouvrez l’onglet Protocole du scanner (GUI) et créez trois nouveaux protocoles (pour l’acquisition, le prétraitement et la reconstruction) avec les paramètres suivants :
- Pour le protocole d’acquisition : définissez le temps de balayage total de 3 600 s et la position du lit simple. Enregistrez ce protocole avec un nom propre pour l’importation ultérieure dans le workflow. Faites de même pour tous les protocoles suivants dans les points suivants.
- Pour le protocole de prétraitement pour souris : sélectionnez une fenêtre d’énergie (EW) de 250-750 keV et activez les corrections suivantes : désintégration radioactive, coïncidences aléatoires et temps mort. Définissez le protocole de cadrage (c’est-à-dire le fractionnement dynamique des données brutes) comme suit : 8 x 5 s, 8 x 10 s, 3 x 40 s, 2 x 60 s, 2 x 120 s, 10 x 300 s (= 3 600 s). Pour le rat, sélectionnez une fenêtre d’énergie (EW) de 350-750 keV, en utilisant le même cadrage que pour le protocole de la souris.
- Pour le protocole de reconstruction : sélectionnez l’algorithme 3D-OSEM-MC (3D Ordered Subset Expectations Maximization) de haute qualité, basé sur Monte Carlo, avec 8 sous-ensembles et 8 itérations, avec la normalisation, la correction quantitative et la correction d’atténuation CT activées.
- Pour la tomodensitométrie à faible dose pour la correction de l’atténuation (CTAC), utilisez les paramètres suivants :
- Pour le protocole d’acquisition : image unique, position du lit unique, balayage complet ; réglages du tube : 80 kV, faible courant (faible dose) ; 576 vues à 360°, avec 34 ms de temps d’exposition par vue (temps de balayage de 20 s) ; Type de rotation: continu, mode de sensibilité: haute sensibilité.
- Pour le protocole de prétraitement : taille voxel 240 μm, FOV transversale : Rat, FOV axial : 100%.
- Pour le protocole de reconstruction : fenêtre de filtre: lisse, taille du voxel: standard , activer le durcissement du faisceau et la précorrection de l’anneau, désactiver la correction de post-artefact de l’anneau.
- Pour la tomodensitométrie à grille à contraste amélioré, créez trois nouveaux protocoles (pour l’acquisition, le prétraitement et la reconstruction) avec les paramètres suivants :
- Pour le protocole d’acquisition pour souris : réglage d’une seule image, position du lit simple, balayage complet ; réglages du tube : 65 kV, courant plein (faible bruit) ; 8 000 vues à plus de 360°, avec 15 ms de temps d’exposition par vue (temps de balayage de 120 s) ; type de rotation : continu, mode de sensibilité : haute sensibilité. Pour le rat, définissez les paramètres du protocole d’acquisition comme suit : tension du tube de 80 kV, 16 000 vues sur 360°, avec un temps d’exposition de 12 ms par vue (temps de balayage de 192 s).
- Pour le protocole de prétraitement pour souris : sélectionnez une taille de voxel de 120 μm ; champ de vision transversal (FOV) : souris ; FOV axial : 50 %. Pour rat, sélectionnez une taille de voxel de 240 μm; champ de vision transversal (FOV): Rat; FOV axial: 50%.
- Pour le protocole de reconstruction : fenêtres filtrantes: lisse, taille du voxel: standard; Activez le durcissement du faisceau et la précorrection de l’anneau, désactivez la post-correction de l’artefact de l’anneau.
- Ouvrez l’onglet Workflow dans l’interface graphique et créez un nouveau workflow, en ajoutant les protocoles qui viennent d’être créés : étapes 1.1.1.1-1.1.1.3 pour PET, étapes 1.1.2.1. -1.1.2.3. pour CTAC et les étapes 1.1.3.1. -1.1.3.3. pour les CT fermés, dans l’ordre donné. Dans les deux cas, assurez-vous que les protocoles sont imbriqués dans l’ordre suivant : Acquisition | Prétraitement | La reconstruction.
REMARQUE: Des images TEP dynamiques d’une durée de <5 s pour mieux capturer le pic de la fonction d’entrée artérielle au début de la TEP sont possibles mais ne sont pas recommandées car cela peut conduire à des images bruyantes avec une précision quantitative réduite. Dans l’étape 1.1.2.2, nous avons utilisé la taille « Rat » pour le champ de vision transversal. Ceci est couramment utilisé pour les rats et les souris dans CTAC.
- Pour la TEP, ouvrez l’onglet Protocole du scanner (GUI) et créez trois nouveaux protocoles (pour l’acquisition, le prétraitement et la reconstruction) avec les paramètres suivants :
2. Préparation de l’animal pour l’imagerie TEP/TDM
NOTE: Pour le présent protocole, tous les animaux ont été à jeun pendant la nuit.
- Anesthésier la souris avec 3%-4% (v/v) d’isoflurane dans une chambre d’induction initialement, puis maintenir avec 1%-2% (v/v) isoflurane.
- Pesez la souris et mesurez la glycémie basale pour surveiller l’état de l’animal. Pour prélever l’échantillon de sang requis, utilisez des ciseaux tranchants et faites une petite coupure au bout de la queue, puis massez doucement la queue pour recueillir une gouttelette de sang (~ 1 μL) directement sur la bandelette réactive.
- Procéder à l’insertion d’un accès veineux au niveau de la veine caudale à l’aide d’un papillon de 29 G pour la souris et de 24 G pour le rat.
- Pour effectuer la technique de canulation, utilisez un chauffage simultané (généralement, sous une lampe chauffante) et une désinfection du point où l’aiguille est insérée pour la vasodilatation de la veine. Après la canulation, fixez le papillon avec un ruban de soie à la queue pour le maintenir en place pendant la procédure.
NOTE: Le jeûne est requis pour [18études F]FDG. Différents traceurs peuvent impliquer différentes préparations animales, mais une discussion approfondie sur ce sujet n’entre pas dans le cadre du présent protocole. En ce qui concerne [18F]FDG, éviter le jeûne conduit à une biodistribution du traceurtrès différente 16.
- Pour effectuer la technique de canulation, utilisez un chauffage simultané (généralement, sous une lampe chauffante) et une désinfection du point où l’aiguille est insérée pour la vasodilatation de la veine. Après la canulation, fixez le papillon avec un ruban de soie à la queue pour le maintenir en place pendant la procédure.
- Allumez le système d’anesthésie (isoflurane 1%-2%, 0,8 L/min O 2 pour la souris et 1-1,2 L/min pour le rat) connecté au scanner TEP-CT et transférez la souris au lit.
- Placez la souris en décubitus dorsal, la tête la première, sur le lit scanner du tomographe TEP-CT, en mettant son nez dans le masque nasal pour l’anesthésie et en bloquant doucement la tête de la souris au masque avec du ruban adhésif.
- Fixez les membres supérieurs et inférieurs de la souris sur le lit du scanner pour éviter tout mouvement involontaire pendant les procédures d’imagerie, qui peut entraîner des artefacts de mouvement.
- Surveillez la température corporelle et la fréquence respiratoire à l’aide d’une sonde rectale et d’un oreiller respiratoire, respectivement.
3. Préparation de la dose de traceur TEP
- Pour les souris, prélever 10 MBq de [18F]FDG dans un volume de 100-150 μL avec une seringue à insuline (1 mL). Pour les rats, prélever une dose plus élevée de 15 MBq dans 0,20-0,25 mL.
REMARQUE: Évitez une activité plus élevée car le scanner TEP décrit dans ce protocole a une sensibilité très élevée et nécessite juste une quantité modeste d’activité pour obtenir des images de haute qualité. - Si la concentration initiale du traceur dans le flacon est trop élevée, utiliser une solution physiologique (0,9 % p/v de NaCl) pour diluer la dose du traceur à une concentration de 50 à 100 MBq/mL.
- Utilisez le calibrateur de dose TEP pour mesurer l’activité réelle dans la seringue. Annotez l’activité de préinjection et le temps de mesure car ces valeurs seront utilisées ultérieurement à l’aide de modules d’entrée spécifiques de l’interface graphique du scanner TEP.
4. Préparation de l’agent de contraste CT
- Prélever 0,2 mL par 20 g de poids de souris d’agent de contraste en émulsion lipidique iodée dans une seringue de 1 mL. Limiter le volume d’injection à 0,5 mL d’AC pour les souris plus lourdes. Si vous utilisez l’ioméprol, réglez le débit d’injection pour les souris à 10 mL/h (~0,17 mL/min) et limitez le volume d’injection à 0,5 mL.
- Pour les rats, aspirer 2,3 à 3 mL d’ioméprol, dilué à une concentration de 200 mg/mL, dans une seringue de 5 mL.
REMARQUE : Si l’émulsion lipidique pour petits animaux CA n’est pas disponible, l’ioméprol par injection continue au moyen d’une pompe à seringue peut être utilisé, comme indiqué ci-dessous. - Connectez la seringue à la pompe à seringue et réglez la pompe en fonction de la taille et du diamètre réels de la seringue.
- Connectez la seringue à la tubulure et à l’aiguille CA et préremplissez la tubulure avec l’AC.
- Régler le débit d’injection à 24 mL/h (= 0,4 mL/min), en limitant l’injection à un volume maximal de 2 mL.
REMARQUE: L’utilisation d’AC de pool sanguin à base d’émulsion lipidique iodée est également possible chez le rat, malgré le coût relativement élevé de cette procédure en raison du volume plus important d’une seule injection. Si cette option est préférée (par exemple, pour simplifier le protocole en évitant la pompe à seringue), la procédure suivante peut être utilisée:
- Pour les rats, aspirer 2,3 à 3 mL d’ioméprol, dilué à une concentration de 200 mg/mL, dans une seringue de 5 mL.
- Prélever 7,5 mL par kg de poids corporel d’agent de contraste en émulsion lipidique iodée dans une seringue de 5 mL. Limiter le volume d’injection à 2 mL d’AC également pour les rats plus lourds.
5. Alignement des animaux et opérations préliminaires avant imagerie
- Lors de l’immobilisation de l’animal sur le lit d’imagerie, créer une nouvelle étude sur l’interface graphique du tomographe. Ajoutez un identificateur de nom d’étude dans le module Nom de l’étude et sélectionnez le flux de travail d’imagerie précédemment enregistré dans le menu déroulant.
- Sélectionnez la partie anatomique appropriée avec les informations sur l’animal / le spécimen | Partie anatomique | Positionnement cardiaque et animal par information sur les animaux/échantillons | Positionnement | Couché sur le dos/La tête la première. Annoter le poids de l’animal en grammes pour le module correspondant : Informations sur les animaux/spécimens | Poids de l’animal.
REMARQUE: Toutes les autres informations de cette section sont facultatives, mais il est utile de fournir autant que possible les informations demandées pour les trouver dans l’en-tête DICOM des images de reconstruction, facilitant ainsi l’interrogation ultérieure des données. - Sélectionnez le radionucléide dans les informations TEP | F18 pour les études [18 F]FDG et 18autres composés marqués F; modifier si d’autres traceurs (p. ex., [13N]NH3) sont utilisés. Écrivez également le nom du traceur dans les informations de TEP | Module de nom de traceur car ce nom sera signalé dans l’en-tête DICOM à la fin de la reconstruction de l’image.
REMARQUE: Les informations sur le temps d’injection, l’activité et le volume du traceur sont obligatoires, mais pourraient être fournies plus tard lors de l’acquisition du TEP. - Dans les informations de tomodensitométrie, écrivez toutes les informations disponibles concernant l’agent de contraste.
REMARQUE: Toutes ces informations sont facultatives mais pourraient faciliter l’interrogation ultérieure des données si elles sont fournies. - Appuyez sur Effectuer l’analyse et attendez qu’un autre onglet de l’interface graphique s’ouvre, ce qui permet le positionnement de l’animal et la spécification d’autres options de numérisation.
- Sélectionnez le type d’étalonnage CT dans l’étalonnage CT | Utilisez l’étalonnage CT par défaut.
- Dans la section Préparation de l’étude , sélectionnez chaque protocole d’analyse dans le menu déroulant et cochez la case Attendre la confirmation de l’utilisateur avant cette analyse .
REMARQUE: Cette étape est très importante, car elle mettra le scanner en veille en attendant l’entrée de l’utilisateur avant de commencer la phase d’acquisition correspondante. Pour le PET scan, cela permettra la synchronisation de l’injection du traceur et le démarrage réel de la TEP; pour le CTAC scan, il permettra à l’utilisateur de fermer le couvercle (blindage) avant l’émission de rayons X pendant le scanner (l’étude sera automatiquement interrompue si le couvercle est ouvert avant le début de la tomodensitométrie); pour le Ciné-TDM, cette pause permettra à l’utilisateur de lancer le protocole de perfusion d’AC et le balayage des données CT avec le délai requis. - Pour le positionnement des animaux, activez le module de commande du moteur à l’aide du commutateur situé dans le volet gauche de l’interface graphique.
REMARQUE: Cela activera les lasers de centrage sur le lit de l’animal et activera les boutons d’alignement manuel du lit placés sur le côté du scanner. - Utilisez les boutons manuels d’alignement du lit pour déplacer la poitrine de l’animal sur les marques laser. Vérifiez soigneusement l’alignement longitudinal et vertical de l’animal.
- Une fois que l’animal est placé dans la bonne position en fonction du laser de centrage, appuyez sur Éteindre le laser pour enregistrer la position actuelle marquée par laser, qui doit être déplacée au centre des scanners TEP et CT pendant les phases d’acquisition correspondantes. Ensuite, éteignez le module de commande du moteur.
6. TEP
- Appuyez sur Démarrer l’acquisition pour déplacer l’animal sur le scanner TEP FOV. La queue et la canule resteront à l’extérieur du champ de vision pour permettre l’injection de traceurs. Le scanner restera inactif jusqu’à ce que l’utilisateur appuie sur le bouton Continuer .
- Préparez la seringue avec la dose de traceur TEP étalonnée.
- Démarrez l’acquisition en appuyant sur le bouton Continue (Continuer ) et commencez à injecter le traceur dans la canule dans les 5 s suivant le début de l’analyse (Figure 1).
REMARQUE: La durée d’injection sera de ~20-25 s. - Placez la seringue dans le calibrateur de dose PET pour mesurer l’activité résiduelle dans la seringue. Annotez l’activité réelle et l’heure de mesure.
- Dans l’onglet Moniteur matériel de l’interface graphique du scanner, utilisez le bouton Mettre à jour les informations du traceur PET pour insérer l’heure, l’activité et le volume d’injection réels.
- Pendant l’examen, vérifiez périodiquement les paramètres physiologiques de l’animal.
- Pendant l’examen, mesurez la glycémie comme expliqué à l’étape 2.2 aux points temporels suivants : 5 min, 20 min, 40 min et 60 min après le début de la TEP.
- Après la mesure de la glycémie, placez la bandelette de test dans le compteur gamma et effectuez la mesure de l’activité pendant 60 s. Enregistrer l’heure réelle à laquelle la mesure de l’activité a été effectuée et corriger la désintégration radioactive, en prenant le temps d’injection du traceur comme temps de référence. Convertissez les valeurs d’activité enregistrées en concentration d’activité (Bq/mL) en considérant un volume sanguin moyen de 1 μL dans la bandelette de test glycémique (c.-à-d. en utilisant l’équation [1]) :
C sang(t) = Asang(t)/0,001 mL [Bq/mL] (1)
où A sang(t) est l’activité mesurée corrigée de la désintégration de l’échantillon desang dans la bandelette réactive, exprimée en Bq.
REMARQUE: Le démarrage de la TEP et l’injection du traceur peuvent être effectués par le même opérateur à l’aide du dispositif de commande mobile du tomographe placé sur la table latérale du scanner près du site de l’opérateur pendant l’injection. Des délais plus longs entre le début de l’analyse et le début de l’injection sont autorisés, mais certaines images reconstruites au début de la séquence dynamique resteront vides. Il est recommandé d’éviter les retards supérieurs à 10 s (c.-à-d. conduisant à deux images vides avec le protocole actuel).

Figure 1 : Injection du traceur TEP. Cette opération est effectuée juste après le démarrage de la TEP. L’animal se trouve à l’intérieur du champ de vision du PET (tête la première, avec sa queue visible du côté de l’opérateur). Abréviation : TEP = tomographie par émission de positrons. Veuillez cliquer ici pour voir une version agrandie de cette figure.
7. CT scans
- Avant d’injecter l’agent de contraste CT, lancez le scanner CTAC juste après avoir fermé le couvercle du scanner et appuyé sur le bouton Continue (Continuer ) de l’interface graphique. À la fin de cette très courte acquisition, utilisez les procédures suivantes pour assurer une bonne amélioration du pool sanguin en injectant l’AC avant l’acquisition en utilisant le même accès vasculaire utilisé pour l’injection du traceur TEP.
- Émulsion lipidique iodée CA:
- Une fois l’analyse CTAC terminée, injecter l’émulsion lipidique iodée CA à l’aide de la canule déjà connectée à la veine de la queue de la souris. La durée typique de l’injection est de l’ordre de 30-60 s.
- Commencez l’imagerie juste après la fin de l’injection. Appuyez sur Continuer sur l’interface graphique du scanner pour lancer l’acquisition de Cine-CT.
- Iomeprol/pompe à seringue :
- Si une AC à rayons X normale est utilisée, telle que l’ioméprol, utilisez une pompe à seringue permettant une injection lente à un taux constant.
- Pour les souris, régler le débit d’injection de l’AC à 10 mL/h (~0,17 mL/min) en limitant le volume d’injection à 0,5 mL. Avec ce réglage, arrêtez l’injection après ~3 min. Pour les rats, régler la pompe à un débit de 24 mL/h (= 0,4 mL/min) et limiter le volume d’injection à 2 mL. Avec ce réglage, arrêtez l’injection après 5 min.
- Connectez l’aiguille attachée à la tubulure CA à la canule de la veine de la queue, en veillant à ce que la tubulure et l’aiguille soient préremplies de CA.
- Commencez l’injection. Fermez le couvercle du scanner et préparez-vous pour le Cine-CT scan.
- Appuyez sur le bouton Continuer de l’interface graphique du tomographe 60 s après le début de l’injection pour les souris et après 90 s après le début de l’injection pour les rats, afin que l’acquisition Cine-CT soit lancée. L’injection d’AC cessera à peu près en même temps que la fin de la scintigraphie Cine-CT pour les souris et à la fin pour les rats.
- Émulsion lipidique iodée CA:
- Une fois le cinétogramme terminé, débranchez l’animal du système de surveillance physiologique et retirez la canule de la veine de la queue. Selon le protocole réel, les animaux sont récupérés ou euthanasiés après la procédure d’imagerie décrite. Dans le premier cas, les animaux sont réveillés dans leurs cages dans un environnement chaud sous une lampe infrarouge. Ils sont surveillés jusqu’au réveil complet, prenant 15/30 min après l’anesthésie gazeuse. Dans le cas de protocoles exigeant, par exemple, le prélèvement de tissus à la fin de la procédure d’imagerie, les animaux sont euthanasiés en utilisant un surdosage anesthésique dans une chambre d’induction (isoflurane à 5%), conformément à l’annexe VI du D.Lgs. 26/2014.
NOTA : Dans le cas de 18radionucléides à base F, comme il est indiqué dans le présent protocole, 24 heures après l’injection du traceur sont suffisantes pour atteindre un niveau de radioactivité résiduelle sur le corps de l’animal qui est sans danger pour toutes les fins pratiques.
8. Reconstruction des images 4DCT cardiaques à l’aide d’un gating cardiorespiratoire intrinsèque
REMARQUE: À la fin de l’étude d’imagerie, la reconstruction TEP et CT standard est automatiquement effectuée. Néanmoins, la reconstruction de la séquence cardio CT 4D (Cine) doit être effectuée manuellement et nécessite une certaine interaction de l’utilisateur. Ce type particulier de reconstruction, obligatoire pour l’analyse cardiaque morpho-fonctionnelle ultérieure, est discuté dans cette section.
- Ouvrez le module de contrôle cardiaque de l’interface graphique du tomogragh et sélectionnez l’étude d’imagerie à analyser.
- Sélectionnez une région d’intérêt (ROI) sur les radiographies de l’animal affiché (Figure 2) pour construire une courbe de mouvement cardiaque dépendante du temps, représentant le signal de déclenchement - le kymogramme. Déplacez verticalement le ROI rectangulaire prédessiné de manière à sélectionner à la fois l’apex du cœur et le diaphragme. Ensuite, sélectionnez Analyse du signal de gating. L’interface utilisateur affichera désormais le signal de déclenchement à la fois sur le domaine temporel et sur le domaine fréquentiel.
- Dans le premier graphique du domaine fréquentiel, sélectionnez la bande de fréquence respiratoire en mettant en surbrillance le premier groupe de pics du spectre de fréquences (voir la figure 3 pour un exemple de spectre).
- Dans le deuxième graphique du domaine fréquentiel, sélectionnez la bande de fréquence du mouvement cardiaque, en mettant en surbrillance le deuxième pic le plus net.
- Dans la phase suivante, observez le signal de déclenchement dans le domaine temporel avec des marqueurs de couleur (points) superposés, montrant les pics respiratoires et les pics de contraction cardiaque identifiés. Si les positions des marqueurs correspondent bien aux pics respiratoires et cardiaques du signal de déclenchement initial, passez à la phase suivante. Autrement:
- Si la forme du signal de déclenchement est trop différente de celle affichée à la figure 3, revenez à l’étape 8.2 et sélectionnez un autre retour sur investissement.
- Si la forme du signal de déclenchement est raisonnablement similaire à celle illustrée à la figure 3, revenez aux étapes 8.3 et 8.4 et sélectionnez différentes bandes de fréquences sur le spectre du signal de déclenchement.
- Dans la phase suivante, sélectionnez un minimum de quatre portes cardiaques.
REMARQUE: La reconstruction Cine-CT typique consiste en 8 à 12 portes cardiaques. - Sélectionnez la fenêtre respiratoire appropriée à l’aide du menu déroulant : Fenêtre respiratoire | 20%-80%.
NOTE: Cela permettra de conserver 60% des données acquises dans la reconstruction, à l’exclusion de la phase d’inspiration maximale et, ainsi, d’améliorer la netteté des parois myocardiques reconstruites à chaque phase cardiaque. - Effectuez une reconstruction pour convertir les images Cine-CT rétrospectivement fermées au format DICOM, prêtes à être importées dans le logiciel pour une analyse fonctionnelle ultérieure.
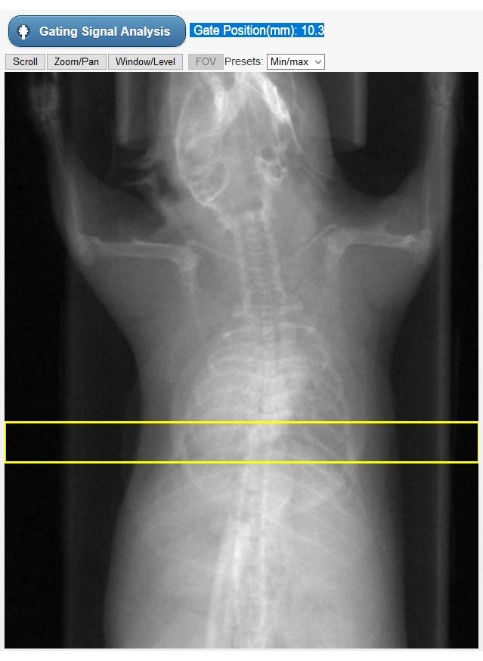
Figure 2 : Outil de sélection du retour sur investissement pour les contrôles intrinsèques. Cette image est montrée dans l’interface graphique du tomographe pendant la phase de reconstruction Ciné-CT. L’utilisateur doit sélectionner la position du ROI (rectangle jaune) sur lequel le signal de déclenchement intrinsèque (kymogramme) est obtenu à partir des projections CT brutes. L’objet de forme circulaire superposé à la poitrine de l’animal est l’oreiller respiratoire utilisé uniquement pour le suivi physiologique pendant l’étude. Abréviations : ROI = région d’intérêt; CT = tomodensitométrie; GUI = interface utilisateur graphique. Veuillez cliquer ici pour voir une version agrandie de cette figure.
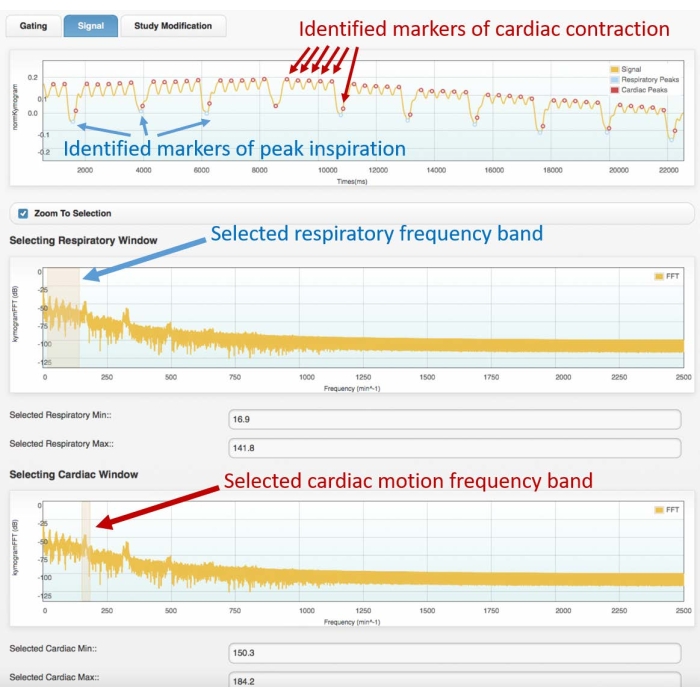
Figure 3 : Exemple de signal de déclenchement (image supérieure) et spectre de fréquences correspondant (centre et bas). Images obtenues avec le module de contrôle cardiaque du logiciel Atrium. L’utilisateur doit sélectionner les bandes de fréquences appropriées pour les mouvements respiratoires (cadre central) et cardiaques (cadre inférieur). Cela permettra d’identifier les marqueurs respiratoires et cardiaques sur le signal de déclenchement, qui doivent être vérifiés par l’utilisateur avant de procéder à la reconstruction 4D. Une mauvaise identification des pics ou une mauvaise affectation (p. ex., respiratoire à cardiaque, ou vice versa) entraînera une reconstruction incorrecte. Les données présentées ont été obtenues à partir de l’analyse d’un ciné-tomodensitométrie 4D d’un rat Wistar mâle adulte en bonne santé (507 g) injecté avec 2 mL d’ioméprol, 200 mg/mL, à raison de 0,4 mL/min pendant 5 min (le graphique en haut est zoomé sur les 22 premières secondes d’acquisition pour permettre une meilleure visualisation des mouvements cardiaques et respiratoires identifiés). Abréviation : CT = tomodensitométrie. Veuillez cliquer ici pour voir une version agrandie de cette figure.
9. Analyse cardiaque TEP
REMARQUE : Cette section montre comment effectuer une analyse cinétique des données dynamiques [18F]FDG du ventricule gauche du petit animal. L’analyse est basée sur le logiciel Carimas. Les instructions ci-dessous ne sont pas destinées à remplacer le manuel d’utilisationdu logiciel 17. La procédure présentée ci-dessous est basée sur l’analyse graphique Patlak des données TEP dynamiques18. Reportez-vous à la section Discussion pour plus de détails concernant cette analyse.
- Ouvrez les images DICOM de la TEP dynamique.
- Sélectionnez le module HeartPlugin .
- Zoomez sur l’image sur le cœur de souris / rat et sélectionnez la dernière période (ou de manière équivalente, la somme des trois à cinq dernières périodes) pour laquelle la majeure partie de l’activité de la piscine de sang a déjà été éliminée.
- Suivez les instructions à l’écran pour réorienter l’image le long de l’axe principal du cœur de l’animal (axe court, axe vertical et axe long horizontal). Pour ce faire, déplacez les marqueurs affichés pour la base du cœur et l’apex (Figure 4).
- Sélectionnez l’outil Segmentation .
Remarque : Par défaut, la segmentation automatique est activée, ce qui donne des résultats fiables dans la plupart des cas. - Si le résultat de la segmentation automatique n’est pas acceptable, affinez la forme du myocarde segmenté et/ou de la cavité VG en activant le mode manuel (recherche ROI désactivée).
- Dans l’outil de modélisation , sélectionnez le modèle cinétique approprié à utiliser pour l’analyse TEP dynamique. Dans ce cas, sélectionnez Graphique | Patlak pour permettre l’analyse du diagramme de Patlak pour le calcul du taux métabolique d’absorption du glucose (MRGlu) pour chaque secteur cardiaque.
- Dans l’outil polarmap , sélectionnez le nombre correct de segments cardiaques affichés. Dans ce cas, sélectionnez 17 segments.
- Maintenant, appuyez sur le bouton Fit pour effectuer la procédure d’ajustement de l’analyse Patlak.
- À la fin de la procédure d’ajustement, observez la carte polaire affichée des valeurs Ki (c’est-à-dire la pente de la régression linéaire exprimée en mL/[mL × min]).
- En utilisant les valeurs Ki pour chaque secteur indiqué dans un tableau, calculer la MRGlu à l’aide de l’équation (2):
MR Glu = (Ki × PGlu)/LC (2)
où PGlu est une valeur dérivée de l’échantillon sanguin de la concentration de glucose plasmatique (mmol/L), et la constante groupée (CL) est un coefficient empirique utilisé pour compenser la différence d’absorption entre le glucose normal et le FDG. Voir, par exemple, Ng et coll.22 pour les valeurs typiques de la constante groupée dans diverses conditions expérimentales.
REMARQUE: Avant de commencer l’analyse TEP, il est recommandé d’inspecter visuellement la séquence dynamique des volumes TEP dans l’outil logiciel d’analyse TEP. Ceci est nécessaire pour exclure le mouvement macroscopique de l’animal entre les périodes au cours de l’étude. Si un mouvement est présent, l’enregistrement approprié de l’image (en dehors du champ d’application de ce protocole) doit être effectué avant l’analyse, si possible.
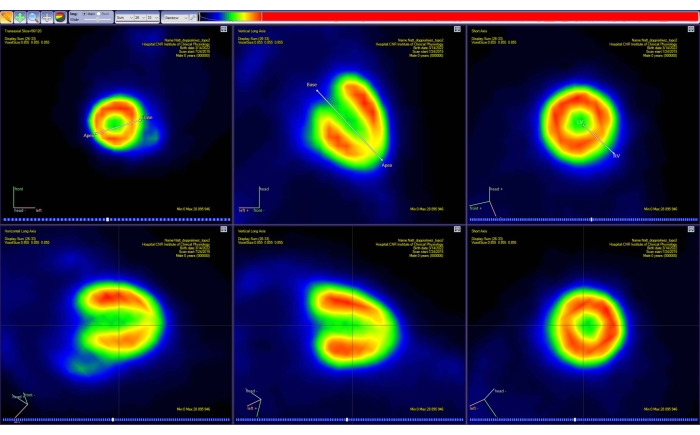
Figure 4 : Outil de réorientation du logiciel d’analyse TEP. La projection de deux segments de droite simples dans l’espace 3D est représentée sur chacun des trois plans standard (transaxial, coronal et sagittal). Le premier segment permet à l’utilisateur de sélectionner la base du cœur et l’apex, tandis que le second permet de sélectionner les côtés gauche et droit du cœur. Cette étape aboutit à une nouvelle image PET (interpolée) (rangée du bas), avec le cœur réorienté le long de la représentation AHA standard. Des images ont été obtenues avec Carimas à partir d’une souris CD-1 mâle adulte en bonne santé pesant 51 g et injectée avec 10 MBq de [18F]FDG. Abréviations : TEP = tomographie par émission de positrons; AHA = American Heart Association; FDG = fluorodésoxyglucose. Veuillez cliquer ici pour voir une version agrandie de cette figure.
10. Analyse cardiaque Ciné-TDM
REMARQUE: Cette section montre comment effectuer une analyse quantitative de l’image cardiaque Ciné-CT pour collecter des données quantitatives globales de la fonction cardiaque. L’analyse est basée sur le logiciel Osirix MD. Les instructions ci-dessous ne sont pas destinées à remplacer le manuel d’utilisation Osirix24.
- Chargez les images DICOM du Cine-CT scan dans le logiciel.
- Ouvrez le jeu de données dynamique avec la visionneuse 4D intégrée.
- À l’aide de l’outil de réforme multiplanaire 3D (MPR), réorientez les données d’image le long de l’axe court (Figure 5).
- Exportez les données réorientées vers DICOM, en veillant à ce que l’ensemble des données 4D soient exportées, avec une épaisseur de tranche préservée (identique à l’original) et une profondeur de bits d’image (16 bits par voxel)
- Ouvrez les images MPR 4D exportées à l’aide de la visionneuse 4D.
- Sélectionnez une période correspondant à la diastole de fin. Parcourez toutes les périodes avec le curseur temporel dans la barre d’outils principale pour vous assurer que la phase cardiaque correcte est sélectionnée.
- Sur cette période, choisissez l’outil d’annotation de polygone fermé et délimitez manuellement la paroi endocardique du LV.
- Faites de même pour 10-20 tranches de la base au sommet, en vous assurant que tous les ROI ont le même nom (par exemple, LVENDO).
- Dans le menu ROI, sélectionnez Volume ROI | Générez les ROI manquants pour générer les ROI sur toutes les tranches d’axe court par interpolation des ROI dessinés manuellement.
- Dans le menu ROI, sélectionnez Volume ROI | Compute Volume pour calculer le volume du groupe ROI portant le même nom de ROI.
- Parcourez les délais et sélectionnez une phase correspondant au système final (volume BT plus petit) et répétez les étapes 10.7-10.10 ci-dessus.
- Calculez le volume de course (SV) et la fraction d’éjection à l’aide des équations (3) et (4) :
SV = VDE - ESV[mL] (3)
FE = 100 × SV/EDV [%] (4)
où EDV est le volume diastolique final et ESV est le volume systolique final.
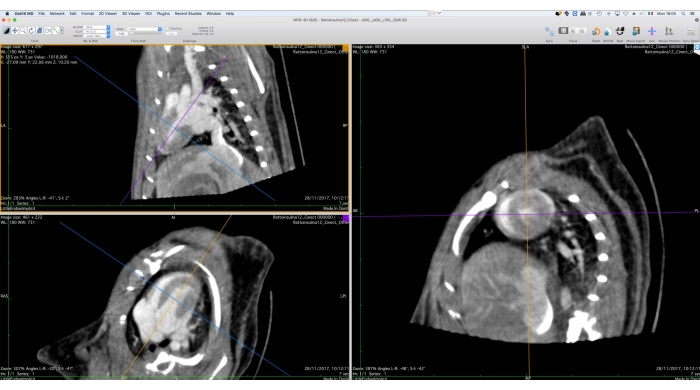
Figure 5 : Interface graphique de l’outil de reformation multiplanaire. Cet outil est utilisé pour la réorientation des données Ciné-CT pour une analyse fonctionnelle ultérieure. L’utilisateur doit faire pivoter et traduire les axes de référence sur le côté gauche de l’écran de manière à ce que la vue du cœur sur l’axe court soit affichée à droite. À la fin de cette procédure, l’utilisateur peut exporter les images réorientées sous la forme d’un ensemble de fichiers DICOM. Les images ont été obtenues avec Osirix MD et se réfèrent à un rat Wistar mâle adulte en bonne santé (507 g) injecté avec 2 mL d’ioméprol, 200 mg/mL, à un débit de 0,4 mL/min pendant 5 min, reconstruit avec rétroprojection filtrée avec une taille de voxel de 0,24 mm3. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Résultats
Dans cette section, les résultats typiques sont présentés pour l’analyse TEP et CT suivant les procédures décrites jusqu’à présent. La figure 6 montre les résultats de la segmentation automatique du myocarde et de la cavité VG de la TEP [18F]FDG d’une souris témoin (saine) CD-1. Même si le ventricule droit n’est pas toujours visible dans les images reconstruites, les axes d’orientation basés sur l’en-tête DICOM peuvent être utilisés pour distinguer corre...
Discussion
Le protocole présenté dans cet article se concentre sur une procédure expérimentale typique pour la recherche cardiovasculaire translationnelle sur de petits modèles animaux de lésions cardiaques en utilisant l’imagerie TEP/TDM à haute résolution. Les résultats présentés sont révélateurs de la valeur quantitative et qualitative élevée des images TEP et Ciné-CT, fournissant des informations fonctionnelles et structurelles de l’ensemble du cœur concernant son métabolisme du glucose, sa forme et la dyn...
Déclarations de divulgation
Daniele Panetta a reçu des subventions pour la R&D de l’instrumentation micro-CT d’Inviscan Sas.
Remerciements
Cette recherche a été soutenue en partie par le projet « gutmom » JPI-HDHL-INTIMIC : Obésité maternelle et dysfonctionnement cognitif chez la progéniture : rôle cause-effet du GUT MicrobiOMe et prévention diététique précoce (projet no. INTIMIC-085, décret universitaire et de recherche n° 946/2019 du ministère italien de l’Éducation, de l’Université et de la Recherche).
matériels
| Name | Company | Catalog Number | Comments |
| 0.9% sterile saline | Fresenius Kabi | 0.9% sodium chloride for injection | |
| 1025L Physiological Monitoring | Small Animal Instruments | Physiological monitoring system for small animal imaging | |
| 5 mL syringes | Artsana | Syringes with needle for injection of PET tracer | |
| Atomlab 500 | Else Nuclear | PET Dose calibrator | |
| Atrium software | Inviscan | Version 1.5.5 | PET/CT operating software |
| Butterfly catheters | Delta Med | 27.5 G needle | |
| Carimas software | Turku PET Center | Version 2.10 | Image analysis software |
| Fenestra VC | Medilumine | Lipid emulsion iodinated contrast agent for small animals | |
| Heat lamp | Heat lamp with clamp and switch | ||
| Insulin syringes | Artsana | Syringes with needle for injection of CT CA | |
| Iomeron 400 mgI/mL | Bracco | Iomeprol, vascular contrast agent | |
| IRIS PET/CT | Inviscan | PET/CT scanner for small animals | |
| Isoflurane | Zoetis | Inhalation anesthetic, 250 mL | |
| OneTouch Glucometer | Johnson&Johnson Medical | Glucose meter kit | |
| Osirix MD software | Pixmeo | Version 11 | Image analysis software |
| Oxygen | Air liquide | Compressed gas | |
| Rectal probe for 1025L | Small Animal Instruments | Rectal probe with cable for SAII 1025L systems | |
| Respiratory sensor for 1025L | Small Animal Instruments | Respiratory pillow with tubings for SAII 1025L systems | |
| TJ-3A syringe pump | Longer | Motorized syringe pump for CT CA injection |
Références
- Zaragoza, C. Animal models of cardiovascular diseases. Journal of Biomedicine and Biotechnology. 2011, 497841 (2011).
- Russell, J. C., Proctor, S. D. Small animal models of cardiovascular disease: Tools for the study of the roles of metabolic syndrome, dyslipidemia, and atherosclerosis. Cardiovascular Pathology. 15 (6), 318-330 (2006).
- Riehle, C., Bauersachs, J. Small animal models of heart failure. Cardiovascular Research. 115 (13), 1838-1849 (2019).
- Menichetti, L., et al. MicroPET/CT imaging of αvß3 integrin via a novel 68Ga-NOTA-RGD peptidomimetic conjugate in rat myocardial infarction. European Journal of Nuclear Medicine and Molecular Imaging. 40 (8), 1265-1274 (2013).
- Zhou, H., et al. Development of a micro-computed tomography-based image-guided conformal radiotherapy system for small animals. International Journal of Radiation Oncology, Biology, Physics. 78 (1), 297-305 (2010).
- Di Lascio, N., Kusmic, C., Stea, F., Faita, F. Ultrasound-based pulse wave velocity evaluation in mice. Journal of Visualized Experiments. (120), e54362 (2017).
- Dann, M. M., et al. Quantification of murine myocardial infarct size using 2-D and 4-D high-frequency ultrasound. American Journal of Physiology-Heart and Circulatory Physiology. 322 (3), 359-372 (2022).
- Espe, E. K. Novel insight into the detailed myocardial motion and deformation of the rodent heart using high-resolution phase contrast cardiovascular magnetic resonance. Journal of Cardiovascular Magnetic Resonance. 15 (1), 82 (2013).
- Vanhove, C., et al. Accurate molecular imaging of small animals taking into account animal models, handling, anaesthesia, quality control and imaging system performance. EJNMMI Physics. 2 (1), 31 (2015).
- Garcia, M. J., et al. State of the art: Imaging for myocardial viability: A scientific statement from the American Heart Association. Circulation: Cardiovascular Imaging. 13 (7), 000053 (2020).
- Panetta, D., et al. Cardiac computed tomography perfusion: Contrast agents, challenges and emerging methodologies from preclinical research to the clinics. Academic Radiology. 28 (1), 1-18 (2020).
- Kusmic, C. Up-regulation of heme oxygenase-1 after infarct initiation reduces mortality, infarct size and left ventricular remodeling: experimental evidence and proof of concept. Journal of Translational Medicine. 12 (1), 89 (2014).
- Muthuramu, I., Lox, M., Jacobs, F., De Geest, B. Permanent ligation of the left anterior descending coronary artery in mice: A model of post-myocardial infarction remodelling and heart failure. Journal of Visualized Experiments. (94), e52206 (2014).
- Fischer, M., et al. Comparison of metabolic and functional parameters using cardiac 18F-FDG-PET in early to mid-adulthood male and female mice. EJNMMI Research. 11 (1), 7 (2021).
- Valenta, I., et al. Feasibility evaluation of myocardial cannabinoid type 1 receptor imaging in obesity: A translational approach. JACC: Cardiovascular Imaging. 11 (2), 320-332 (2018).
- Fueger, B. J., et al. Impact of animal handling on the results of 18F-FDG PET studies in mice. Journal of Nuclear Medicine. 47 (6), 999-1006 (2006).
- . Carimas User Manual Available from: https://turkupetcentre.fl/carimas/files/archive/Html/a1.html (2022)
- Peters, A. M. Graphical analysis of dynamic data: The Patlak-Rutland plot. Nuclear Medicine Communications. 15 (9), 669-672 (1994).
- Choi, Y., et al. Parametric images of myocardial metabolic rate of glucose generated from dynamic cardiac PET and 2-[18F]fluoro-2-deoxy-d-glucose studies. Journal of Nuclear Medicine. 32 (4), 733-738 (1991).
- Laffon, E., Marthan, R. Is Patlak y-intercept a relevant metrics. European Journal of Nuclear Medicine and Molecular Imaging. 48 (5), 1287-1290 (2021).
- Flores, J. E., McFarland, L. M., Vanderbilt, A., Ogasawara, A. K., Williams, S. -. P. The effects of anesthetic agent and carrier gas on blood glucose and tissue uptake in mice undergoing dynamic FDG-PET imaging: Sevoflurane and isoflurane compared in air and in oxygen. Molecular Imaging and Biology. 10 (4), 192-200 (2008).
- Ng, C. K. Sensitivity of myocardial fluorodeoxyglucose lumped constant to glucose and insulin. American Journal of Physiology-Heart and Circulatory Physiology. 260 (2), 593-603 (1991).
- Shoghi, K. I., Welch, M. J. Hybrid image and blood sampling input function for quantification of small animal dynamic PET data. Nuclear Medicine and Biology. 34 (8), 989-994 (2007).
- Heuberger, J., Pixmeo, S., Rosset, A. OsiriX User Manual. Blurb. , (2017).
- Cerqueira, M. D., et al. Standardized myocardial segmentation and nomenclature for tomographic imaging of the heart. A statement for healthcare professionals from the Cardiac Imaging Committee of the Council on Clinical Cardiology of the American Heart Association. Circulation. 105 (4), 539-542 (2002).
- Kolanowski, T. J., et al. Multiparametric evaluation of post-MI small animal models using metabolic ([18F]FDG) and perfusion-based (SYN1) heart viability tracers. International Journal of Molecular Sciences. 22 (22), 12591 (2021).
- Guiducci, L., et al. Contribution of organ blood flow, intrinsic tissue clearance and glycaemia to the regulation of glucose use in obese and type 2 diabetic rats: A PET study. Nutrition Metabolism and Cardiovascular Diseases. 21 (9), 726-732 (2011).
- Tadinada, S. M., et al. Functional resilience of C57BL/6J mouse heart to dietary fat overload. American Journal of Physiology-Heart and Circulatory Physiology. 321 (5), 850-864 (2021).
- Dreyfuss, A. D., et al. A novel mouse model of radiation-induced cardiac injury reveals biological and radiological biomarkers of cardiac dysfunction with potential clinical relevance. Clinical Cancer Research. 27 (8), 2266-2276 (2021).
- Hsu, B. PET tracers and techniques for measuring myocardial blood flow in patients with coronary artery disease. Journal of Biomedical Research. 27 (6), 452-459 (2013).
- Dinkel, J., et al. Intrinsic gating for small-animal computed tomography. Circulation: Cardiovascular Imaging. 1 (3), 235-243 (2008).
- Kuntz, J., et al. Fully automated intrinsic respiratory and cardiac gating for small animal CT. Physics in Medicine and Biology. 55 (7), 2069-2085 (2010).
- Li, Y., Zhang, W., Wu, H., Liu, G. Advanced tracers in PET imaging of cardiovascular disease. BioMed Research International. 2014, 504532 (2014).
- Kim, D. -. Y., Cho, S. -. G., Bom, H. -. S. Emerging tracers for nuclear cardiac PET imaging. Nuclear Medicine and Molecular Imaging. 52 (4), 266-278 (2018).
- Maddahi, J., Packard, R. R. S. Cardiac PET perfusion tracers: Current status and future directions. Seminars in Nuclear Medicine. 44 (5), 333-343 (2014).
- Bentourkia, M. Kinetic modeling of PET data without blood sampling. IEEE Transactions on Nuclear Science. 52 (3), 697-702 (2005).
- Lammertsma, A. A. Forward to the past: The case for quantitative PET imaging. Journal of Nuclear Medicine. 58 (7), 1019-1024 (2017).
- Nahrendorf, M., et al. High-resolution imaging of murine myocardial infarction with delayed-enhancement cine micro-CT. American Journal of Physiology-Heart and Circulatory Physiology. 292 (6), 3172-3178 (2007).
- Badea, C. T., Fubara, B., Hedlund, L. W., Johnson, G. A. 4-D micro-CT of the mouse heart. Molecular Imaging. 4 (2), 110-116 (2005).
- Technical Resources. MediLumine Available from: https://www.medilumine.com/technical-resources (2019)
- Nebuloni, L., Kuhn, G. A., Müller, R. A Comparative analysis of water-soluble and blood-pool contrast agents for in vivo vascular imaging with micro-CT. Academic Radiology. 20 (10), 1247-1255 (2013).
- Panetta, D., et al. Performance evaluation of the CT component of the IRIS PET/CT preclinical tomograph. Nuclear Instruments & Methods in Physics Research Section A: Accelerators Spectrometers Detectors and Associated Equipment. 805, 135-144 (2016).
- Gu, J., et al. At what dose can total body and whole abdominal irradiation cause lethal intestinal injury among C57BL/6J mice. Dose-Response. 18 (3), 1559325820956783 (2020).
- Amirrashedi, M., Zaidi, H., Ay, M. R. Advances in preclinical PET instrumentation. PET Clinics. 15 (4), 403-426 (2020).
- Clark, D. P., Badea, C. T. Advances in micro-CT imaging of small animals. Physica Medica. 88, 175-192 (2021).
- Belcari, N., Del Guerra, A., Panetta, D., Grupen, C., Buvat, I. High-Resolution and Animal Imaging Instrumentation and Techniques. Handbook of Particle Detection and Imaging. , 1497-1535 (2021).
- Wang, G., Rahmim, A., Gunn, R. N. PET Parametric imaging: Past, present, and future. IEEE Transactions on Radiation and Plasma Medical Sciences. 4 (6), 663-675 (2020).
- Befera, N. T., Badea, C. T., Johnson, G. A. Comparison of 4D-microSPECT and microCT for murine cardiac function. Molecular Imaging and Biology. 16 (2), 235-245 (2014).
- van Deel, E., Ridwan, Y., van Vliet, J. N., Belenkov, S., Essers, J. In vivo quantitative assessment of myocardial structure, function, perfusion and viability using cardiac micro-computed tomography. Journal of Visualized Experiments. (108), e53603 (2016).
- Lee, C. -. L., et al. Assessing cardiac injury in mice with dual energy-microCT, 4D-microCT and microSPECT imaging following partial-heart irradiation. International Journal of Radiation Oncology, Biology, Physics. 88 (3), 686-693 (2014).
- Harms, H., et al. Comparison of clinical non-commercial tools for automated quantification of myocardial blood flow using oxygen-15-labelled water PET/CT. European Heart Journal - Cardiovascular Imaging. 15 (4), 431-441 (2013).
- Nesterov, S. V., et al. Myocardial perfusion quantitation with 15O-labelled water PET: High reproducibility of the new cardiac analysis software (CarimasTM). European Journal of Nuclear Medicine and Molecular Imaging. 36 (10), 1594-1602 (2009).
- Nesterov, S. V., et al. Myocardial perfusion quantification with Rb-82 PET: Good interobserver agreement of Carimas software on global, regional, and segmental levels. Annals of Nuclear Medicine. 36, 507-514 (2022).
- Nesterov, S. V., et al. One-tissue compartment model for myocardial perfusion quantification with N-13 ammonia PET provides matching results: A cross-comparison between Carimas, FlowQuant, and PMOD. Journal of Nuclear Cardiology. , (2021).
- Thackeray, J. T., Kuntner-Hannes, C., Haemisch, Y. Preclinical Multimodality Imaging and Image Fusion in Cardiovascular Disease. Image Fusion in Preclinical Applications. , 161-181 (2019).
- Vohra, R., Batra, A., Forbes, S. C., Vandenborne, K., Walter, G. A. Magnetic resonance monitoring of disease progression in mdx mice on different genetic backgrounds. The American Journal of Pathology. 187 (9), 2060-2070 (2017).
- Baehr, A., et al. Agrin promotes coordinated therapeutic processes leading to improved cardiac repair in pigs. Circulation. 142 (9), 868-881 (2020).
- Lalwani, K., et al. Contrast agents for quantitative microCT of lung tumors in mice. Comparative Medicine. 63 (6), 482-490 (2013).
- Bertoldo, A., et al. Evaluation of compartmental and spectral analysis models of [18F]FDG kinetics for heart and brain studies with PET. IEEE Transactions on Bio-medical Engineering. 45 (12), 1429-1448 (1998).
- Li, Y., Kundu, B. K. An improved optimization algorithm of the three-compartment model with spillover and partial volume corrections for dynamic FDG PET images of small animal hearts in vivo. Physics in Medicine and Biology. 63 (5), 055003 (2018).
- Mabrouk, R., Dubeau, F., Bentourkia, M., Bentabet, L. Extraction of time activity curves from gated FDG-PET images for small animals' heart studies. Computerized Medical Imaging and Graphics. 36 (6), 484-491 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon