Method Article
Un modèle d’intestin sur puce pour étudier l’axe microbiome intestinal-système nerveux
Dans cet article
Résumé
neuroHuMiX est un modèle avancé d’intestin sur puce permettant d’étudier les interactions des cellules bactériennes, épithéliales et neuronales dans des conditions de co-culture proximales et représentatives. Ce modèle permet de démêler les mécanismes moléculaires sous-jacents à la communication entre le microbiome intestinal et le système nerveux.
Résumé
Le corps humain est colonisé par au moins le même nombre de cellules microbiennes que de cellules humaines, et la plupart de ces micro-organismes sont situés dans l’intestin. Bien que l’interaction entre le microbiome intestinal et l’hôte ait fait l’objet d’études approfondies, la façon dont le microbiome intestinal interagit avec le système nerveux entérique reste largement inconnue. À ce jour, il n’existe pas de modèle in vitro physiologiquement représentatif pour étudier les interactions entre le microbiome intestinal et le système nerveux.
Pour combler cette lacune, nous avons développé le modèle d’intestin sur puce de diaphonie humaine-microbienne (HuMiX) en introduisant des neurones entériques induits dérivés de cellules souches pluripotentes dans le dispositif. Le modèle qui en résulte, « neuroHuMiX », permet la co-culture de cellules bactériennes, épithéliales et neuronales à travers des canaux microfluidiques, séparés par des membranes semi-perméables. Malgré la séparation des différents types de cellules, les cellules peuvent communiquer entre elles par le biais de facteurs solubles, ce qui permet simultanément d’étudier chaque type de cellule séparément. Cette configuration permet de mieux comprendre comment le microbiome intestinal affecte les cellules neuronales entériques. Il s’agit d’une première étape cruciale dans l’étude et la compréhension de l’axe microbiome intestin-système nerveux humain.
Introduction
Le microbiome intestinal humain joue un rôle crucial dans la santé et la maladie humaines. Il a fait l’objet d’études approfondies au cours de la dernière décennie et demie, et son rôle potentiel dans la modulation de la santé et de la maladie est maintenant établi1. Une perturbation du microbiome conduisant à une communauté microbienne déséquilibrée (dysbiose) a été postulée comme étant impliquée dans la pathogenèse de nombreux troubles chroniques, tels que l’obésité, les maladies inflammatoires de l’intestin et le cancer colorectal, ou même des maladies neurodégénératives telles que la maladie de Parkinson 2,3.
Bien que le microbiome intestinal humain ait été associé à des troubles neurologiques, on ne sait toujours pas comment le microbiome intestinal communique avec le système nerveux entérique et l’affecte. Comme le système nerveux entérique humain n’est pas facilement accessible pour une étude immédiate, des modèles animaux ont été utilisés dans des expériences jusqu’à présent4. Cependant, compte tenu des différences apparentes entre les modèles animaux et les humains5, le développement de modèles in vitro imitant l’intestin humain est d’un intérêt immédiat. Dans ce contexte, l’essor et l’avancée du domaine des cellules souches pluripotentes induites humaines (CSPi) nous a permis d’obtenir des neurones entériques (EN) représentatifs6. Les EN dérivés des CSPi permettent d’étudier le système nerveux entérique dans des modèles de culture in vitro, tels que des inserts de culture cellulaire, des organoïdes ou des organes sur puce 7,8.
Le modèle de diaphonie humain-microbien (HuMiX) est un modèle d’intestin sur puce imitant l’intestin humain9. Le modèle HuMiX initial (ci-après dénommé le dispositif initial) accueillait des cellules épithéliales (Caco-2) et des cellules bactériennes10,11. Cependant, pour étudier le lien entre le microbiome intestinal et le système nerveux, desEN6 dérivés d’iPSC ont également été introduits dans le système (Figure 1). La co-culture proximale de cellules neuronales, épithéliales et bactériennes permet d’analyser individuellement les différents types de cellules et d’étudier les interactions entre les différents types de cellules dans un environnement mimant celui de l’intestin humain.
Au cours des dernières années, des progrès ont été réalisés dans le développement de modèles permettant d’étudier les organes de manière plus représentative sur le plan physiologique en utilisant des modèles d’organes sur puce (p. ex., intestin sur puce). Ces modèles sont plus représentatifs de l’environnement intestinal humain en raison de l’apport constant de nutriments et de l’élimination des déchets, ainsi que de la surveillance en temps réel, par exemple, des niveaux d’oxygène ou de l’intégrité de la barrière 8,12. Ces modèles permettent spécifiquement d’étudier les effets des bactéries intestinales sur les cellules hôtes. Cependant, pour pouvoir utiliser des organes sur puce pour étudier les interrelations entre le microbiome intestinal et le système nerveux, les cellules neuronales doivent être intégrées dans de tels systèmes. Par conséquent, l’objectif du développement de HuMiX et de la mise en place du système neuroHuMiX (ci-après dénommé le dispositif) était de développer un modèle d’intestin sur puce, qui comprend des cellules neuronales entériques en co-culture proximale avec des cellules épithéliales intestinales et des bactéries.
Protocole
1. Culture et tri cellulaire
- Neurones entériques induits dérivés de cellules souches pluripotentes
REMARQUE : Cultivez les cellules iPS 6 semaines avant le début d’un cycle. Le protocole de différenciation des EN a été adapté de Fattahi et al.6.- Cultivez les cellules iPS sur une plaque matricielle à 6 puits recouverte de gel dans 2 mL/puits de milieu de culture iPSC complété par 1 % de pénicilline-streptomycine (P/S).
- À une confluence de 80 % à 90 %, faire passer les cellules, semer dans une plaque matricielle enduite de gel en utilisant le même milieu, incuber à 37 °C, 5 % de CO2 et 90 % d’humidité relative (HR).
- Pour la dérivation des CSPi, après deux passages après décongélation, à une confluence de 80 % à 90 %, ensemencer les cellules dans une plaque matricielle enduite de gel à 6 puits à une densité de 100 000 cellules/puits. Incuber dans le milieu décrit ci-dessus + inhibiteur ROCK Y-27632 (1 :2 000) pendant 24 h à 37 °C.
- Après 24 h, remplacer le milieu surnageant par le milieu du jour 0, conformément au tableau 1. Inclure un puits de contrôle à utiliser comme témoin négatif lors du tri des cellules pour le processus de dérivation. Gardez le contrôle bien dans la composition du média du jour 0 pendant toute la période de dérivation. Changez le milieu tous les deux jours du jour 2 au jour 10, selon le tableau 1.
- Au jour 11, triez les cellules pour les cellules CD49d positives, qui seront utilisées dans les étapes suivantes de ce protocole.
- Regroupez les cellules à trier en fonction des cellules CD49d positives, ainsi que du puits témoin. Centrifuger les cellules pendant 3 min à 300 × g. Remettre en suspension la pastille cellulaire dans de l’albumine sérique bovine (BSA) à 2 % + 1 % P/S dans une solution saline tamponnée au phosphate (PBS) 1x.
- Divisez chaque lot de cellules (regroupées et témoins) en deux : colorer une fraction avec un anticorps anti-CD49d humain et l’autre fraction avec un anticorps témoin isotype.
- Gate la population cellulaire principale, puis les cellules individuelles, sur la base desquelles les cellules présentant CD49d à leur surface seront sélectionnées. Collectez ces cellules pour l’étape suivante de la différenciation.
REMARQUE : Habituellement, 30 à 40 % des cellules triées sont CD49d positives.
- Transférer deux à quatre millions de cellules triées sur une plaque à 6 puits d’une plaque de fixation ultra-basse dans un milieu N2B27 (voir tableau 2) + FGF2 + CHIR pendant 4 jours pour permettre la formation de sphéroïdes.
- Le jour 15, replaquer les sphéroïdes sur une plaque à 6 puits recouverte de poly L-ornithine/laminine/fibronectine (P/L/F) dans 2 mL/puits de milieu N2B27 avec du GDNF et de l’acide ascorbique (AA). Remplacez le milieu tous les 2-3 jours.
REMARQUE : Rapport de revêtement : poly L-ornithine, 15 μg/mL ; laminine, 2 μg/mL ; fibronectine, 2 μg/mL dans 1x PBS. - Après 3 semaines, utilisez les cellules pour l’inoculation dans l’appareil.
- Caco-2
REMARQUE : Décongeler les cellules Caco-2 au moins 1 semaine avant une course.- Ensemencer une fiole T75 avec 1 × 106 cellules Caco-2 dans un supplément de glutamine RPMI 1640 + HEPES + 10% de sérum de veau fœtal (FBS) ; incuber à 37 °C, 5 % de CO2 et 90 % d’humidité relative.
REMARQUE : Pour ces expériences, utilisez idéalement Caco-2 lors de leur premier ou deuxième passage après la décongélation.
- Ensemencer une fiole T75 avec 1 × 106 cellules Caco-2 dans un supplément de glutamine RPMI 1640 + HEPES + 10% de sérum de veau fœtal (FBS) ; incuber à 37 °C, 5 % de CO2 et 90 % d’humidité relative.
2. Préparation de la course HuMiX
- Préparation et revêtement de couvercles en polycarbonate
- Autoclaver une paire de couvercles en polycarbonate (PC) (Figure 2) avec quatre vis en utilisant le cycle d’instrument initial de l’appareil (voir le tableau 3 pour plus de détails).
- Dans une enceinte de sécurité biologique, insérez quatre vis dans chaque coin du couvercle inférieur du PC (voir Figure 3) et transférez-les dans une boîte de Pétri carrée stérile pour un processus d’enrobage en deux étapes.
- Le premier jour, ajoutez 2 mL de poly L-ornithine à 1,5 % dans du PBS (1x) au milieu du couvercle du PC. Incuber pendant la nuit à 37 °C, 5 % de CO2 et 90 % d’humidité relative.
- Le lendemain, retirez la solution et remplacez-la par une solution contenant 0,2 % de laminine et 0,2 % de fibronectine dans le PBS (1x) pour couvrir la surface de la chambre cellulaire neuronale. Incuber le couvercle pendant la nuit à 37 °C, 5 % de CO2 et 90 % d’humidité relative.
- Après l’incubation, conservez la solution d’enrobage sur le couvercle du PC et fermez la boîte de Pétri avec un film d’étanchéité. Placez-les à 4 °C pour les conserver jusqu’à leur utilisation.
- Avant utilisation, retirez la solution de revêtement et séchez-la à l’air libre dans une enceinte de sécurité biologique pendant 30 minutes jusqu’à ce que le revêtement soit complètement sec.
- Préparation et revêtement des joints
REMARQUE : Cette section décrit comment préparer les joints en collant des membranes semi-perméables sur des joints en silicone. Après une première étape d’autoclavage, les joints sont enduits de collagène ou de mucine pour permettre l’adhésion des cellules épithéliales intestinales ou des bactéries. La membrane recouverte de mucine imite la barrière muqueuse présente dans l’intestin humain.- Fixez une membrane en polycarbonate de 1 μm au joint de collagène et une membrane en polycarbonate de 50 μm entre les joints sandwich inférieurs et supérieurs. Autoclaver les joints avec les membranes fixées en utilisant le cycle initial de l’instrument de l’appareil (voir le tableau 3).
- Pour enrober les membranes, placez chaque joint dans une boîte carrée stérile.
REMARQUE : Le revêtement est effectué dans des conditions stériles dans une enceinte de sécurité biologique.- Pour le joint de collagène, ajouter 3 mL de collagène (50 μg/mL) à la membrane et incuber pendant 3 h à 37 °C.
REMARQUE : Veillez à ne pas toucher la membrane avec l’embout de la pipette lors de l’ajout du collagène pour éviter d’endommager la membrane. - Pour l’enrobage de mucine, placez le joint sandwich avec le côté supérieur du joint vers le haut dans une boîte de Pétri pour enduire le côté supérieur de la membrane de 3 mL de mucine (0,025 mg/mL). Incuber les joints à 37 °C pendant 1 h.
- Pour le joint de collagène, ajouter 3 mL de collagène (50 μg/mL) à la membrane et incuber pendant 3 h à 37 °C.
- Après le temps d’incubation, aspirez les solutions de collagène et de mucine de leurs joints respectifs et séchez la membrane à l’air libre dans une enceinte de sécurité biologique pendant 30 min. Fermez la vaisselle, scellez avec un film d’étanchéité de laboratoire et conservez les joints à 4 °C jusqu’à utilisation.
- Assemblage de tubes
REMARQUE : Cette section décrit comment assembler le tube pour la perfusion de l’appareil. Avant l’assemblage, autoclez toutes les pièces et/ou achetez celles qui sont emballées individuellement dans un emballage stérile. Placez tous les composants dans une enceinte de sécurité biologique.- Utilisez trois tubes pour chaque appareil et pour chaque tube, un morceau de tuyau de pompe, deux morceaux de tube Marprene long (20 cm), deux morceaux de tube Marprene court (8 cm), une aiguille de 40 mm pour le flacon de sortie, une aiguille de 120 mm pour les flacons d’entrée de 250 ml ou une aiguille de 80 mm pour les flacons d’entrée plus petits, trois connecteurs Luer vers cannelure mâles et sept femelles, trois adaptateurs Luer et deux vannes d’arrêt à trois voies.
- Assemblez les conduites de tubulure de gauche à droite à l’aide des composants autoclavés et stérilisés énumérés ci-dessus (Figure 4).
REMARQUE : Vaporisez chaque composant avec de l’éthanol à 70% entre chaque étape de connexion.
- Préparation des supports
REMARQUE : Cette section décrit comment préparer le milieu pour les différents types de cellules dans le dispositif, ainsi que comment transférer le milieu dans des flacons de sérum de manière stérile.- Complétez le RPMI 1640 avec 10 % de FBS filtré à 0,22 μm et inactivé par la chaleur. Complétez le support N2B27 avec du GDNF et de l’AA.
- Transférez le milieu dans des flacons de sérum dans des conditions stériles. Transférez 200 mL de média RPMI 1640 dans des bouteilles de 250 mL et 30 mL de média N2B27 dans des flacons de 50 mL.
- Préparer le milieu bactérien (RPMI 1640 + 10 % FBS + 5 % de milieu de culture De Man, Rogosa et Shapre [MRS]) à un stade ultérieur, car cela n’est nécessaire que pendant les dernières 24 heures de l’expérience.
- Fermez les flacons à l’aide d’un septum, sertissez-les en aluminium et autoclavez.
- Pour transférer le support, retirez le sertissage en aluminium à l’aide d’un décapsuleur approprié (diamètre = 20 mm). Retirez également le septum. Versez délicatement le milieu préparé dans les flacons de sérum sans toucher le flacon.
- Pour stériliser, enflammez l’ouverture du biberon à l’aide d’un bec Bunsen portable. Scellez les bouteilles en pulvérisant le septum avec 70 % d’éthanol, en l’ajoutant à la bouteille et en la fermant avec un sertissage en aluminium à l’aide d’une sertisseuse à joint.
- Placez les flacons fermés dans l’incubateur à 37 °C, 5 % de CO2 pendant 24 h pour réchauffer le milieu à 37 °C, ainsi que pour vous assurer qu’il n’y a pas de signes visibles de contamination avant d’utiliser des flacons de milieu pour l’appareil.
3. Démarrage de HuMiX
- Assemblage NeuroHuMiX
REMARQUE : Cette section explique comment assembler l’appareil. En bref, le système de serrage est stérilisé et resserré, après quoi le couvercle inférieur du PC est placé sur la base de la pince. Ensuite, les joints revêtus et les couvercles supérieurs en PC sont empilés les uns sur les autres, suivis de la partie supérieure de la pince. Enfin, la pince est serrée pour comprimer les joints et rendre le système étanche et sans fuite. La figure 4 illustre les différentes pièces nécessaires à l’assemblage.- Autoclaver les pinces et les deux couvercles en PC (haut et bas) avant l’assemblage.
- Serrez les vis utilisées pour assembler les parties supérieure et inférieure de la pince (Figure 4C,D). Ouvrez les vis avant le processus d’autoclavage et resserrez-les plus tard.
- Transférez le couvercle inférieur du PC enduit et séché (étape 2.2.3) (Figure 4 A1) de la boîte de Pétri carrée vers le haut de la base de la pince en maintenant les quatre vis dans les coins du couvercle du PC.
REMARQUE : Évitez de toucher le couvercle du PC pour réduire le risque de contamination. - Placez le joint de la chambre épithéliale vers le haut (c’est-à-dire avec la membrane sur le dessus) sur le couvercle du PC à l’aide d’une pince à épiler stérile. Utilisez les vis pour aligner le joint et le couvercle du PC afin de vous assurer que les orifices d’entrée et de sortie dans les coins du couvercle sont alignés avec les ouvertures du joint (Figure 3).
- À l’aide de la pince à épiler stérile, placez le joint sandwich sur le joint de collagène, avec le côté supérieur vers le haut.
- Placez le couvercle supérieur du PC sur le joint sandwich. Pour réduire le risque de contamination, faites-le en ne touchant que les bords du couvercle (ne touchez pas la partie supérieure ou inférieure du couvercle). Assurez-vous que les cannelures du couvercle de la pince sont alignées avec l’ouverture d’entrée et de sortie de l’assemblage de la membrane et que les vis du couvercle inférieur du PC s’insèrent dans l’ouverture du couvercle supérieur du PC.
- Pour fermer l’appareil, placez le couvercle de la pince (la partie supérieure de la pince) sur le couvercle du PC. Assurez-vous que les ouvertures du couvercle coïncident avec les cannelures d’entrée et de sortie du couvercle supérieur du PC. Fermez les loquets. Maintenez le dispositif fermé (Figure 5) sous le capot pour le raccorder aux conduites de tubulure amorcées (étape 3.2.6).
- Amorçage des conduites de tubes
REMARQUE : Cette section décrit comment connecter le tube aux bouteilles d’entrée et de sortie, ainsi qu’à la pompe, puis amorcer le tube avec un milieu pour éliminer tout produit résiduel utilisé pendant le nettoyage et la stérilisation, et s’assurer qu’aucune bulle n’est présente dans le tube.- L’amorçage des conduites de tubulure est effectué dans l’enceinte de sécurité biologique. Insérez des aiguilles d’aération avec des filtres dans le septum de chaque bouteille d’entrée et de sortie. À l’aide d’une pince à épiler propre et stérile, insérez les aiguilles de 120 mm dans les flacons de sérum de 250 ml. Insérez les aiguilles de 80 mm dans les flacons de sérum de 50 ml.
- Insérez les aiguilles de 40 mm, à l’extrémité de chaque ligne de tubulure, dans un flacon de sérum à écouler. Par appareil, il y a deux bouteilles de sortie pour les trois lignes de tuyaux. Les aiguilles de 40 mm des tubulures épithéliales et neuronales sont reliées à la même bouteille de sortie pour une mise au rebut moyenne. La ligne de tube bactérien va à la deuxième bouteille de sortie.
- Insérez les conduites de tubulure de la pompe dans les cassettes de la pompe. Assurez-vous que les robinets d’arrêt à trois voies des conduites de tubes sont tous ouverts.
- Au début, réglez la pompe péristaltique pour diriger le fluide de l’entrée vers la bouteille de sortie à une vitesse de 5 tr/min (voir le tableau 4).
REMARQUE : Pendant l’amorçage de la ligne de tube, la vitesse peut être augmentée jusqu’à 10 tr/min pour accélérer le processus. - Démarrez la pompe en appuyant sur le bouton de démarrage et assurez-vous que le sens de l’action de pompage est dans le sens des aiguilles d’une montre. Une fois que le fluide tombe dans les bouteilles de sortie, assurez-vous qu’il n’y a pas de fuites et qu’il ne reste pas de bulles d’air dans les conduites de tubulure et les points de raccordement. Éliminez les bulles restantes en tapotant le tube et les connecteurs ou en augmentant la vitesse de la pompe.
- Lorsque toutes les conduites de tube tombent dans les bouteilles de sortie, réglez le débit sur 2 tr/min pour connecter l’appareil initial.
- Amorçage de l’appareil
REMARQUE : Cette section décrit comment amorcer le dispositif avec un milieu de culture cellulaire pour s’assurer que toutes les chambres sont remplies et pré-enrobées de milieu, afin qu’aucune bulle ne reste dans le système et que les cellules puissent facilement adhérer pendant l’inoculation.- L’amorçage de l’appareil est effectué dans l’enceinte de sécurité biologique dans des conditions stériles. Pour connecter l’appareil, commencez par connecter la ligne neuronale, la chambre inférieure de l’appareil.
- Pour connecter une conduite, fermez les vannes du robinet d’arrêt à trois voies (Figure 5C), en commençant d’abord par le côté sortie, où le tube court est déconnecté du connecteur Luer femelle et connecté à l’orifice de sortie de l’appareil, en poussant le tube sur l’ardillon. Pour assurer une bonne connexion et réduire les risques de fuites et/ou de contamination, assurez-vous que le tube est en contact avec le couvercle en le poussant complètement sur le connecteur. Raccordez le tube d’entrée de la même manière. Une fois qu’une ligne est complètement connectée à l’appareil (entrée et sortie), ouvrez les robinets d’arrêt à trois voies.
- Répétez l’étape précédente avec la ligne épithéliale, puis avec la ligne bactérienne. Maintenez la pompe à un débit de 2 tr/min. Augmentez la vitesse de la pompe à 2,5 tr/min, mais pas plus, afin d’éviter les fuites dues à l’accumulation de pression. Laissez la pompe amorcer les chambres de l’appareil.
- Surveillez les flacons de sérum sortants à tout moment. Si des bulles d’air se coincent dans les chambres, les conduites ou le connecteur, une conduite de tube avec une bulle d’air à l’intérieur ne tombera plus dans la chambre de sortie. Pour vous débarrasser des bulles d’air, laissez d’abord les appareils fonctionner pendant quelques minutes. Si cela ne résout pas le problème, fermez l’une des autres conduites qui tombent pendant un court laps de temps en fermant le robinet d’arrêt à trois voies de l’écoulement.
- Une fois que toutes les chambres de l’appareil sont complètement amorcées - toutes les chambres sont remplies de milieu de culture cellulaire et qu’il ne reste aucune bulle dans l’appareil - réduisez la vitesse de la pompe à 0,5 tr/min. L’appareil est maintenant prêt pour l’inoculation cellulaire.
4. Préparation et inoculation des cellules
REMARQUE : Cette section décrit comment préparer les différents types de cellules nécessaires à l’inoculation du dispositif, ainsi que comment les inoculer dans le dispositif de manière stérile et sans introduire de bulles d’air. De plus, il décrit comment effectuer un rafraîchissement du milieu pour les cellules neuronales et comment préparer le milieu pour la culture bactérienne dans le dispositif.
- Cellules épithéliales
- Détacher les cellules Caco-2 de la fiole à l’aide de l’acide trypsine-éthylènediaminetétraacétique (EDTA), les remettre en suspension dans RPMI 1640 + 10 % FBS et les compter dans un compteur de cellules Neubauer à l’aide du test d’exclusion du bleu de trypan. Centrifuger la suspension cellulaire Caco-2 (3 min, 300 × g) et jeter le surnageant pour éliminer la trypsine-EDTA restante. Remettre en suspension les cellules Caco-2 dans RMPI 1640 + 10% FBS pour obtenir une suspension de 350 000 cellules/mL. Pour chaque appareil, un volume de 1,5 ml est nécessaire.
- Transférez 1,5 mL de la suspension cellulaire Caco-2 dans une seringue stérile de 2 mL et retirez l’air ou les bulles qui restent dans la seringue.
- Fermez les vannes d’arrêt à trois voies de la tubulure des chambres bactériennes et neuronales. Débranchez les tubes des chambres bactériennes et neuronales de la pompe en retirant les cassettes avec les tubes respectifs du rotor.
- Ouvrez le bouchon de la vanne d’arrêt à trois voies de la tubulure d’entrée menant à la chambre épithéliale et tournez la vanne d’arrêt à trois voies pour rediriger le débit de fluide de l’appareil vers le « connecteur ouvert » (Figure 5C). Laissez le fluide s’écouler jusqu’à ce qu’une goutte de fluide apparaisse à l’extrémité ouverte de la vanne d’arrêt à trois voies.
- Insérez la seringue contenant les cellules épithéliales dans le « connecteur ouvert » en utilisant la méthode de connexion goutte-goutte pour permettre l’insertion de la seringue dans le connecteur sans introduction de bulles d’air. Tournez la vanne du robinet d’arrêt à trois voies pour arrêter l’écoulement du fluide du flacon d’entrée (via la pompe) et pour permettre l’écoulement de la seringue connectée vers le dispositif initial. Débranchez le canal épithélial de la pompe.
- Appuyez lentement sur la seringue pour inoculer la chambre épithéliale avec la suspension cellulaire. Assurez-vous qu’environ une goutte toutes les 3 s tombe dans la bouteille de sortie.
- Ajouter 1,5 mL de suspension de cellule, puis fermer la vanne du robinet d’arrêt à trois voies de sortie. Débranchez la seringue et fermez l’extrémité ouverte du robinet d’arrêt à trois voies avec le bouchon. Gardez la chambre fermée pendant au moins 2 h. Pendant ce temps, inoculez les cellules neuronales.
- Cellules neuronales
- Détachez les cellules neuronales du puits en remettant en suspension les cellules dans le milieu des puits respectifs à l’aide d’une pipette. Inoculer chaque dispositif avec des cellules entièrement confluentes à partir d’un puits (9,6 cm2) d’une plaque à 6 puits.
- Remettre en suspension la pastille cellulaire dans le volume moyen N2B27 correspondant (1,5 mL de milieu par dispositif) + 1 μL de fibronectine/mL de suspension cellulaire. Transférez 1,5 mL de la suspension cellulaire remise en suspension dans une seringue de 2 mL et retirez les bulles d’air qui restent dans la seringue. Placez la seringue remplie dans un tube conique stérile de 50 ml et transférez-la dans l’enceinte de sécurité biologique contenant le dispositif HuMiX amorcé.
- Suivez le même processus d’inoculation que celui décrit précédemment pour l’inoculation de la chambre épithéliale, à l’exception du changement de ligne de tube. Ici, débranchez les conduites des tubes bactériens et épithéliaux de la pompe.
- Après l’inoculation des cellules neuronales, fermez tous les robinets d’arrêt à trois voies des conduites de tube et débranchez-les de la pompe. Placez l’appareil dans l’incubateur à 37 °C et 5 % de CO2. Gardez tous les canaux fermés pendant 2 h pour permettre aux cellules de se fixer.
- Après 2 h, connectez les canaux bactériens et épithéliaux à la pompe et ouvrez les robinets d’arrêt à trois voies d’entrée et de sortie des deux lignes. Gardez la chambre neuronale fermée pendant les 14 jours suivant l’exécution initiale de l’appareil, sauf pendant le changement de milieu.
- Pendant la course de 14 jours, changez le milieu de la chambre neuronale tous les 3-4 jours. Pour un rafraîchissement moyen, préparez 3 mL de milieu N2B27 frais par dispositif et transférez-le dans un flacon de sérum stérile de 20 mL. Fermez le flacon de sérum avec du milieu à l’aide d’un septum stérile et d’un joint à sertir en aluminium.
- Insérez une aiguille d’aération et de 80 mm dans le septum du nouveau flacon. Remplacez l’ancien flacon de support par le nouveau dans l’incubateur en déconnectant le Luer mâle de l’aiguille de l’ancien biberon à l’aiguille du nouveau biberon.
- Connectez la cassette avec le tube de la pompe de la chambre neuronale à la pompe et ouvrez les robinets d’arrêt à trois voies. Laisser le fluide s’écouler à 0,5 tr/min pendant 2 h avant de fermer les robinets d’arrêt à trois voies de la tubulure neuronale et de se déconnecter de la pompe, jusqu’au prochain échange de fluide.
5. Culture bactérienne et inoculation
NOTE : Dans cette étude, au jour 12, une culture liquide de la souche F275 de Limosilactobacillus reuteri a été réanimée à partir d’un stock de glycérol. En fonction des besoins ou des plans d’étude, d’autres espèces bactériennes peuvent être utilisées.
- Préparer trois tubes avec 5 mL de bouillon MRS : un tube témoin stérile et deux pour l’inoculation de L. reuteri. À l’aide d’une anse d’inoculation, grattez le haut du stock de glycérol et transférez-le dans un tube. Répétez l’opération avec un deuxième tube d’inoculation. Incuber les tubes à 37 °C, 170 tr/min pendant la nuit.
REMARQUE : Ne laissez pas le bouillon de glycérol décongeler. - Préparez un milieu frais pour les dispositifs initiaux en mélangeant Roswell Park Memorial Institute (RPMI) 1640 + 10 % FBS avec 5 % de bouillon MRS. Préparer 25 ml par dispositif et transférer dans des flacons de sérum de 100 ml dans des conditions stériles. Fermez les flacons à l’aide d’un septum stérile et d’un sertissage en aluminium. Placez les bouteilles dans l’incubateur à 37 °C, 5% de CO2 pendant la nuit.
- Avant de raccorder les flacons à la ligne de tubes bactériens, ajoutez une aiguille d’aération et une aiguille de 80 mm à chaque flacon de média RPMI 1640/MRS.
- Préparez de nouveaux tubes, chacun avec 3 mL de bouillon RPMI 1640 + 10 % FBS + 5 % MRS. Préparez au moins deux tubes, un tube de contrôle et un tube par appareil. Inoculer 15 μL de la culture de nuit de L. reuteri (densité optique [OD] > 2) dans deux des tubes nouvellement préparés.
- Incuber les tubes pendant 1 h à 37 °C, 170 tr/min, après quoi on atteint un diamètre extérieur de 0,05 à 0,10, ce qui correspond à ~1 × 107 unités formant colonie (UFC)/mL. Transférez 1,5 mL dans une seringue de 2 mL (une par dispositif). Utilisez le reste de la suspension bactérienne pour le placage CFU et la coloration vivante/morte.
- Pour l’inoculation bactérienne du dispositif, suivez la même procédure que celle mentionnée à l’étape 4.1, sauf qu’ici les lignes du tube neuronal et épithélial sont fermées.
- Après l’inoculation, fermez également la conduite du tube bactérien en fermant les vannes et en débranchant la pompe pendant 30 minutes. Connectez les lignes de tube épithélial et bactérien et ouvrez-les à nouveau. Laissez les appareils fonctionner pendant encore 24 h à 37 °C et 0,5 tr/min.
6. Ouverture et échantillonnage de HuMiX
REMARQUE : La section ci-dessous décrit l’échantillonnage de différents types de cellules. Par exemple, les pastilles de cellules neuronales sont utilisées pour l’extraction de l’ARN et la réaction en chaîne par polymérase quantitative (qPCR) subséquente, les pastilles bactériennes pour l’extraction de l’ADN et le séquençage du gène de l’ARNr 16S, et les surnageants pour les tests immuno-enzymatiques (ELISA) et d’autres tests (par exemple, le test du lactate).
- Le jour 14, le jour de l’ouverture, fermez tous les robinets d’arrêt à trois voies, débranchez les tubes de la pompe et sortez le dispositif de l’incubateur sur la paillasse de laboratoire.
REMARQUE : Lorsque vous déplacez l’appareil, assurez-vous de garder l’appareil à l’horizontale. - Retirez les conduites de tubulure connectées à l’appareil avant d’ouvrir lentement la pince et retirez le couvercle de la pince. Avec précaution, retirez le couvercle supérieur du PC, collectez le milieu dans un tube de microcentrifugation de 1,5 ml et placez-le sur de la glace.
- Retirez délicatement le joint sandwich, tout en recueillant le milieu de la chambre épithéliale ; Veillez à ne pas toucher la couche cellulaire. Placez le joint sandwich dans une boîte de Pétri carrée et ajoutez une solution stérile de NaCl à 0,9 % dans H2O dans la chambre bactérienne jusqu’à ce que la chambre soit complètement recouverte (environ 1 ml).
- Retirez lentement le joint de collagène tout en recueillant le milieu de la chambre neuronale et en le transférant dans un tube de microcentrifugation. Placez tous les tubes de média sur de la glace. Placez le joint de collagène dans un plat carré et ajoutez doucement quelques millilitres de 1x PBS à la couche de Caco-2 jusqu’à ce que la couche cellulaire soit entièrement recouverte.
- Placez le couvercle inférieur du PC dans une boîte de Pétri carrée et ajoutez doucement environ 2 mL de PBS 1x sur les cellules neuronales, afin qu’elles ne se dessèchent pas pendant le processus d’échantillonnage.
- Centrifuger le tube de milieu bactérien à 5 000 × g pendant 5 min à 4 °C. Centrifuger les tubes épithéliaux et neuronaux à 300 × g pendant 5 min à 4 °C. Après la centrifugation, transférez le surnageant de chaque tube dans un nouveau tube de microcentrifugation et placez-le immédiatement sur de la glace carbonique.
- Cellules épithéliales
- Retirez délicatement le PBS du joint et collectez-le dans un tube conique de 15 ml. Ajouter 2 mL de trypsine aux cellules et incuber le joint pendant 5 min à 37 °C, 5 % de CO2. Ajouter 2 mL de RPMI 1640 dans le joint après incubation, remettre les cellules en suspension et recueillir dans un autre tube conique de 15 mL.
- Centrifuger les deux tubes pendant 5 min à 300 × g. Jeter le surnageant. Remettre en suspension la pastille du lavage PBS dans 300 μL de PBS et la pastille cellulaire dans 1 mL de PBS.
- Transférez 50 μL de chaque tube dans un tube de microcentrifugation de 0,5 mL pour le compteur cellulaire automatisé et le comptage cellulaire d’exclusion au bleu trypan. Transvaser la pastille de cellules remise en suspension (1 ml) dans un tube de 1,5 mL et centrifuger à 300 × g pendant 5 min.
- Retirer le surnageant et remettre en suspension la pastille cellulaire dans 250 μL de tampon de lyse + 1 % de bêta-mercaptoéthanol et placer le tube sur de la glace carbonique.
- Cellules neuronales
- Prenez la boîte de Pétri carrée, avec le couvercle inférieur en PC transparent avec des cellules neuronales de l’étape précédente (6.5) dans un microscope à contraste de phase inversée.
REMARQUE : Lorsque vous déplacez le couvercle du PC avec PBS dessus, soyez très doux, car le réseau neuronal se détache très facilement. - Effectuer un dernier contrôle de qualité avant l’inoculation en observant la morphologie et la densité cellulaire des cellules neuronales à l’aide de la microscopie à fond clair. Vérifiez que les cellules ont migré des sphéroïdes et qu’un réseau neuronal s’est formé. Le réseau neuronal doit être confluent à environ 90 %. Pour plus de détails, voir Fattahi et al.6.
- À la paillasse, remettre les cellules en suspension dans du PBS et les recueillir dans un tube de 1,5 ml. Centrifuger le tube à 300 × g pendant 3 min. Jeter le surnageant, remettre en suspension la pastille cellulaire dans 250 μL de tampon de lyse + 1 % de bêta-mercaptoéthanol et placer sur de la glace sèche.
REMARQUE : La coloration par immunofluorescence (IF) peut être effectuée sur les cellules neuronales sur le couvercle du PC. Si la coloration IF est le test préféré afin de ne pas détruire le réseau neuronal, les cellules doivent être fixées sur le couvercle du PC avec 4% de paraformaldéhyde (PFA), ce qui empêche la réutilisation du couvercle du PC dans de futures expériences.
- Prenez la boîte de Pétri carrée, avec le couvercle inférieur en PC transparent avec des cellules neuronales de l’étape précédente (6.5) dans un microscope à contraste de phase inversée.
- Cellules bactériennes
- Remettre en suspension les cellules bactériennes attachées à la membrane dans une solution de NaCl à 0,9 %. Si les cellules sont fermement attachées, utilisez doucement un grattoir cellulaire pour détacher les cellules bactériennes de la membrane.
REMARQUE : L’utilisation d’un grattoir peut endommager les cellules, augmentant la probabilité de voir plus de cellules mortes. - Recueillir la suspension cellulaire dans un tube conique de 15 mL. Ajouter la pastille restante du milieu précédemment collecté dans le tube conique de 15 ml. Centrifuger à 5 000 × g pendant 5 min à 4 °C. Jeter le surnageant et remettre en suspension la pastille cellulaire dans 1 mL de solution de NaCl à 0,9 %.
- Divisez ce volume en trois parties : une pour la congélation d’une pastille bactérienne pour l’extraction d’acides nucléiques (650 μL), une pour le placage CFU (50 μL) sur des plaques MRS et une pour la coloration vivante/morte (300 μL). Pour la préparation de la pastille pour l’extraction des acides nucléiques, transférer la suspension cellulaire dans un tube de microcentrifugation, centrifuger à 5 000 × g pendant 5 min à 4 °C et jeter le surnageant. Placez la pastille de cellule sur de la glace carbonique.
- À la fin de l’échantillonnage, transvaser tous les tubes sur de la glace carbonique dans un congélateur à -80 °C pour les stocker en vue d’analyses ultérieures en aval.
REMARQUE : Le surnageant peut également être utilisé pour l’analyse par chromatographie en phase gazeuse et spectrométrie de masse (GC-MS). De plus, lors de l’ouverture, la membrane enrobée de collagène peut être divisée en différentes parties pour différentes analyses - une moitié pour être utilisée pour la coloration de l’occlusine IF, une autre partie pour l’extraction de l’ARN pour une analyse plus approfondie de l’expression génique et une autre partie pour le comptage cellulaire.
- Remettre en suspension les cellules bactériennes attachées à la membrane dans une solution de NaCl à 0,9 %. Si les cellules sont fermement attachées, utilisez doucement un grattoir cellulaire pour détacher les cellules bactériennes de la membrane.
Résultats
Dans neuroHuMiX, nous avons co-cultivé trois types de cellules différents : des cellules bactériennes, épithéliales et neuronales (Figure 1). Pour nous assurer que les cellules étaient toutes viables, nous avons effectué différents tests sur les différents types de cellules. Par exemple, nous avons effectué des comptages d’UFC sur les cellules bactériennes, des tests de comptage cellulaire et de viabilité cellulaire sur les cellules épithéliales, tandis que les cellules neuronales ont été évaluées par des analyses microscopiques.
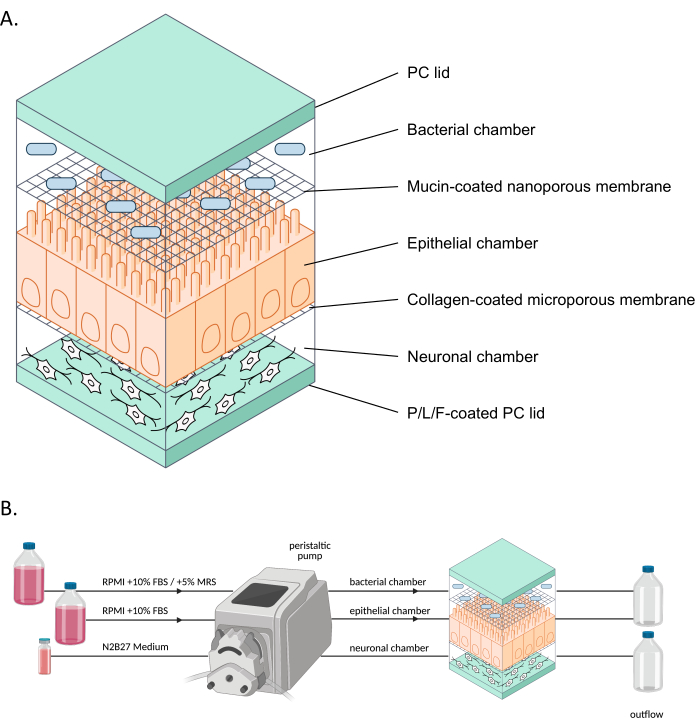
Figure 1 : Représentation schématique de neuroHuMiX et de son dispositif expérimental. (A) Les trois chambres sont maintenues entre deux couvercles PC pour les maintenir fermés. Chaque chambre est remplie d’un milieu spécifique pour les cellules cultivées à l’intérieur. Les différentes chambres sont séparées par des membranes semi-perméables permettant la communication cellulaire via des facteurs solubles traversant les membranes. (B) Représentation de la configuration de neuroHuMiX. Chaque chambre est reliée à différentes bouteilles de média. Pour la chambre bactérienne, pendant les 12,5 premiers jours, la chambre est connectée à RPMI + 10 % FBS, avant d’être changée pendant les 36 dernières heures en RPMI + 10 % FBS + 5 % MRS. Abréviations : PC = polycarbonate ; P/L/F = poly L-ornithine/laminine/fibronectine ; RPMI = milieu de culture cellulaire du Roswell Park Memorial Institute ; MRS = milieu de culture De Man, Rogosa et Shapre. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Pour déterminer si les cellules étaient correctement fixées, lors de l’ouverture des dispositifs, nous avons évalué la formation d’une couche cellulaire sur la membrane recouverte de collagène (Figure 6A). Pour s’assurer que les cellules de l’appareil étaient viables, un comptage automatisé des cellules (Figure 6B) et un comptage cellulaire d’exclusion au bleu trypan ont été effectués (Figure 6C). Les tests ont été effectués sur des cellules Caco-2 provenant de trois configurations HuMiX différentes : (i) Caco-2 en culture avec des EN, (ii) Caco-2 en culture avec L. reuteri, et (iii) le dispositif impliquant la co-culture des trois types de cellules. Les tests statistiques à l’aide d’une ANOVA à un facteur n’ont pas révélé de différences significatives entre les types de cellules, ce qui suggère que les cellules Caco-2 sont restées viables dans toutes les configurations et conditions initiales de l’appareil testées dans cette étude. Cela souligne le fait que la densité bactérienne atteinte lors de la co-culture de L. reuteri et les deux types de cellules humaines n’ont pas d’effets cytotoxiques sur les cellules humaines.
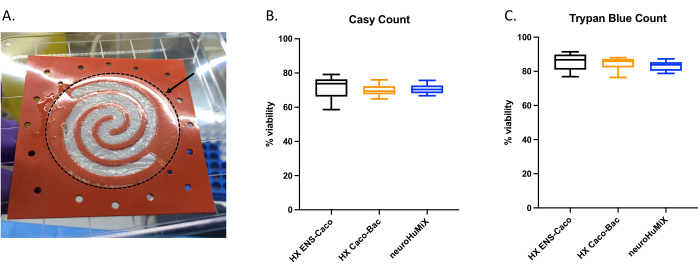
Figure 6 : Évaluation des cellules Caco-2 sur la membrane enrobée de collagène. (A) Couche de cellules Caco-2 sur la membrane enrobée de collagène après ouverture. La flèche indique la membrane recouverte de collagène, qui est entourée d’un cercle en pointillés. Les cellules Caco-2 se développaient en forme de spirale sur la membrane. Viabilité cellulaire des cellules Caco-2 après 14 jours dans HuMiX. Les numérations cellulaires ont été obtenues à l’aide (B) du compteur cellulaire automatisé et (C) de la numération cellulaire par test d’exclusion du bleu trypanique. Le nombre de cellules Caco-2 a été déterminé à partir de différentes configurations de culture dans le dispositif initial : co-culture avec des neurones entériques (EN) (noir), co-culture avec L. reuteri (orange) et dans le dispositif (ENs et L. reuteri) (bleu). Une ANOVA à un facteur a été réalisée, montrant qu’il n’y a pas de différence significative entre les différentes configurations de culture (ANOVA à un facteur, p = 0,1234 [ns] ; les barres d’erreur indiquent l’erreur type). Veuillez cliquer ici pour voir une version agrandie de cette figure.
Pour pouvoir cultiver L. reuteri avec des cellules de mammifères, nous avons d’abord optimisé et adapté le milieu de culture pour l’utiliser dans l’appareil. Nous avons constaté qu’un mélange de 5 % de MRS dans RPMI 1640 (complété par 10 % de FBS) était parfaitement adapté à la croissance de L. reuteri, tout en n’étant pas cytotoxique pour les cellules de mammifères utilisées dans ces essais. Par la suite, un comptage de l’UFC a été effectué pour évaluer la croissance de L. reuteri lorsqu’il a été cultivé dans le dispositif pendant 24 h. Le nombre d’UFC a été évalué pour deux configurations initiales différentes du dispositif (Figure 7) - L. reuteri co-cultivé avec Caco-2 et L. reuteri dans le dispositif. Dans les deux configurations, le nombre d’UFC était significativement différent de celui de l’inoculum HuMiX et des cellules récoltées (ANOVA à un facteur, p = 0,0002), ce qui indique une croissance des cellules bactériennes à l’intérieur du dispositif initial.
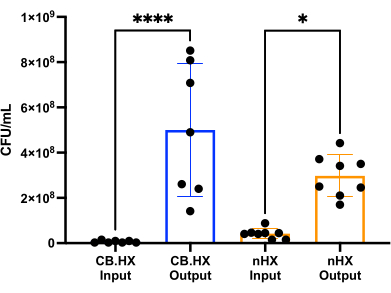
Figure 7 : Comptage en UFC de Limosilactobacillus reuteri de l’inoculum (dilué 1 :100 000) et après 24 h dans HuMiX. Deux configurations différentes : des cellules Caco-2 en co-culture avec L. reuteri et le dispositif. Une ANOVA à un facteur montre une différence significative (p = 0,0002 [***]) entre l’inoculum et les cellules récoltées, ce qui signifie que les bactéries se développent à l’intérieur de HuMiX. Les barres d’erreur indiquent l’erreur type. Abréviations : CB. HX = Bactérie Caco-2 HuMiX ; nHX = neuroHuMiX. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Afin d’évaluer si la culture des EN à l’intérieur du dispositif modifierait le phénotype des cellules, la morphologie macroscopique des EN a été observée à l’aide d’un microscope à contraste de phase inversé. Au cours de cette étape, la confluence et la morphologie EN ont été évaluées. La mise en place d’un réseau neuronal confluent a indiqué que les cellules s’étaient bien fixées sur le couvercle du PC du dispositif enrobé. Il est important de noter que cela met en évidence l’idée qu’ils ont grandi en co-culture avec Caco-2 et L. reuteri. Le bord entre le réseau neuronal confluent et la spirale délimitée par le joint était clairement apparent (Figure 8).
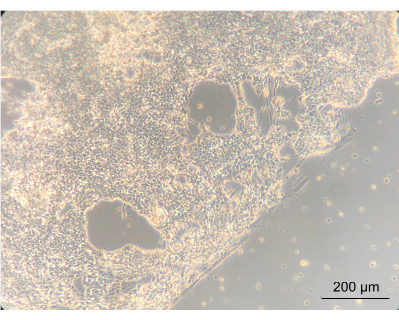
Figure 8 : Neurones entériques après 14 jours de culture dans le dispositif. Sur le côté gauche de l’image, les neurones se sont développés en une couche confluente sur la spirale. Le bord, entre la couche neuronale et l’espace sans cellules, est le bord de la spirale ; Grossissement 10x, barre d’échelle = 200 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Couvercles utilisés dans l’appareil. Les images montrent les couvercles en haut (à gauche) et en bas (à droite) du PC. Chaque côté du couvercle du PC mesure 6,4 cm. Abréviation : PC = polycarbonate. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Joint de la chambre épithéliale sur le couvercle inférieur du PC. Vue de dessus du joint de la chambre épithéliale placé sur le couvercle inférieur du PC (à gauche) et vue du bas (à droite) montrant l’alignement du joint de la chambre épithéliale avec les entrées et les sorties du couvercle inférieur du PC. Chaque côté des joints, ainsi que le couvercle en PC, mesure 6,4 cm. Abréviation : PC = polycarbonate. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Assemblage de l’appareil. (A) Différentes pièces pour l’assemblage de HuMiX : (1) couvercle inférieur du PC ; (2) joint avec membrane microporeuse recouverte de collagène, qui est placé au-dessus de (1) ; (3) joint sandwich avec une membrane nanoporeuse recouverte de mucine entre les deux et placé au-dessus de (2) ; (4) couvercle supérieur du PC placé au-dessus de (3). Chaque côté des joints et des couvercles en PC mesure 6,4 cm. (B) Toutes les pièces de (A) sont placées ensemble. (C,D) Vue de dessus (à gauche) et de côté (à droite) de l’appareil assemblé. B est placé dans le système de serrage pour fermer le système. (C) Chaque côté de la pince supérieure mesure 8 cm. Abréviation : PC = polycarbonate. Veuillez cliquer ici pour voir une version agrandie de cette figure.
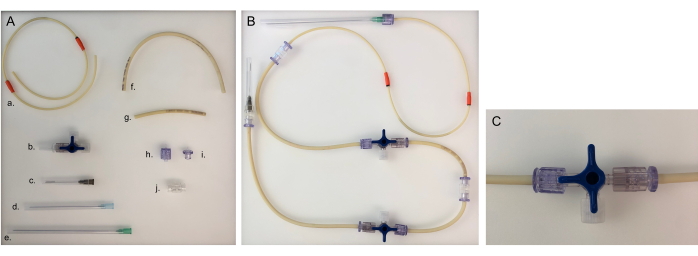
Figure 5 : Pièces nécessaires pour la ligne de tubes et la ligne de tubes assemblés pour une chambre. (A) Différentes pièces pour construire une ligne de tubes : a. ligne de tubes de pompe ; b. robinet d’arrêt à trois voies ; c. aiguille de 40 mm ; d. aiguille de 80 mm ; e. aiguille de 120 mm ; f. longue ligne de tube (20 cm) ; g. ligne de tube courte (8 cm) ; h. Luer mâle ; i. femelle Luer ; J. Adaptateur. (B) Ligne de tubulure assemblée pour la chambre bactérienne ou épithéliale. Pour la chambre neuronale, l’aiguille de 120 mm devrait être remplacée par une aiguille de 80 mm. (C) Vanne d’arrêt à trois voies tournée pour rediriger le débit de fluide de l’appareil vers le « connecteur ouvert » et pour fermer la chambre. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Jour | 0 | 2 | 4 | 6 | 8 | 10 |
| Composition des médias | 100% E6 | 100% E6 | 75 % E6 | 50 % E6 | 25 % E6 | 100% N2 |
| + LDN | + LDN | 25 % de N2 | 50 % de N2 | 75 % de N2 | + LDN | |
| + SB | + SB | + LDN | + LDN | + LDN | + SB | |
| + CHIR | + SB | + SB | + SB | + CHIR | ||
| + CHIR | + CHIR | + CHIR | + RA | |||
| + RA | + RA | |||||
| Molécule | [concentration] | |||||
| LDN (en anglais seulement) | 100 nM | |||||
| SB | 10 μM | |||||
| CHIR (en anglais seulement) | 3 μM | |||||
| Acide rétinoïque (PR) | 1 μM |
Tableau 1 : Composition des supports.
| Média | Composants (concentrations indiquées dans le tableau des matériaux) | Volume (mL) |
| Média N2 (50 mL) | DMEM-F12 | 48 |
| Supplément de N2 | 0.5 | |
| L-Glutamine | 0.5 | |
| Pénicilline/Streptomycine | 0.5 | |
| NEAA (en anglais seulement) | 0.5 | |
| Média N2B27/ENS (50 mL) | Neurobasale | 48 |
| Supplément de N2 | 0.5 | |
| L-Glutamine | 0.5 | |
| Pénicilline/Streptomycine | 0.5 | |
| B27-A | 0.5 |
Tableau 2 : Recettes de supports.
| Température de stérilisation (°C) | 116 |
| Temps de stérilisation (min) | 20 |
| Temps de séchage (min) | 10 |
| Légumes secs | 3 |
| Température finale (°C) | 99 |
Tableau 3 : Fonctionnement de l’autoclave HuMiX.
| Rotations par minute (tr/min) | Débit moyen (μL/min) |
| 0.5 | 13 |
| 2 | 79 |
| 5 | 180 |
Tableau 4 : Débits de la pompe péristaltique.
Discussion
Il est maintenant établi que le microbiome intestinal humain influence la santé et la maladie de l’hôte. Malgré les connaissances suggérant l’importance de notre microbiome, en particulier dans les troubles neurologiques tels que la maladie d’Alzheimer ou la maladie de Parkinson 3,13, on ignore encore largement comment le microbiome intestinal interagit avec le système nerveux entérique et, par conséquent, avec le cerveau.
Jusqu’à présent, il n’existait pas de modèle représentatif permettant d’étudier les interactions entre le microbiome intestinal et le système nerveux. Les études concernant l’axe intestin-cerveau ont traditionnellement été réalisées à l’aide de modèles murins13. Les souris et les humains partagent 85 % de leurs séquences génomiques14, mais il existe des différences significatives à prendre en compte lorsque l’on compare les souris aux humains. En ce qui concerne l’intestin, il est important de noter que, par rapport aux humains, les souris sont exclusivement herbivores. En conséquence, leur tractus gastro-intestinal diffère par sa longueur et ses caractéristiques, telles que la « vidange gastrique»14. Les cerveaux murins présentent également des différences importantes, la structure globale entre les souris et les humains étant différente15. Il est important de noter que les humains ont des temps de cycle cellulaire plus longs pour les progéniteurs neuronaux15. Par conséquent, il est important de développer des modèles représentatifs qui incluent des cellules d’origine humaine, y compris des cellules intestinales et neuronales5. Dans ce contexte, le développement de modèles de recherche in vitro plus reproductibles réduit le besoin d’utiliser des modèles animaux et améliore la reproductibilité.
neuroHuMiX est une version avancée du précédent modèle HuMiX9. HuMiX est un modèle d’intestin sur puce permettant des co-cultures proximales et représentatives de cellules épithéliales et bactériennes. La communication intercellulaire est possible grâce à la co-culture proximale et à la diffusion des facteurs sécrétés et des métabolites via des membranes semi-perméables. Cependant, pour étendre l’utilité de l’appareil initial à l’étude de l’environnement intestinal humain, l’introduction d’un type de cellule supplémentaire est nécessaire. Pour remédier à ce problème, neuroHuMiX, développé avec l’introduction d’ENs dérivés d’iPSC, permet une co-culture proximale de bactéries, de cellules épithéliales intestinales et d’ENs. Le modèle in vitro qui en résulte nous permet d’aborder des questions concernant le microbiome intestinal humain en relation avec le système nerveux humain. La co-culture de différents types de cellules, en particulier les co-cultures de cellules de mammifères et de bactéries, présente plusieurs défis, notamment la perte de viabilité, une mauvaise adhérence et une perte globale de confluence16. Ici, nous avons démontré qu’au sein de ce dispositif, nous sommes capables de co-cultiver trois types de cellules différents au sein d’un même système tout en maintenant la viabilité cellulaire élevée.
Une étape cruciale du protocole consiste à s’assurer de la confluence des cellules neuronales (80 % à 90 % de confluence et de viabilité cellulaires) avant d’inoculer le dispositif. Étant donné qu’il n’est pas possible d’évaluer la croissance cellulaire pendant l’exécution, il est de la plus haute importance de s’assurer que les cellules sont confluentes et se développent bien avant de les introduire dans le modèle. Bien qu’il puisse s’agir d’un facteur limitant, la viabilité globale et la confluence observées dans l’appareil sont généralement élevées.
L’appareil est relié par des conduites tubulaires à une pompe péristaltique. Chaque chambre cellulaire a sa ligne de tubulure spécifique. La tubulure comprend une tubulure de pompe qui permet l’utilisation d’une pompe péristaltique pour la perfusion du milieu, ainsi qu’une tubulure reliant la tubulure de la pompe à l’appareil et une tubulure reliant l’appareil aux bouteilles de sortie/déchets. Des orifices d’échantillonnage sont inclus avant et après l’appareil, pour permettre l’inoculation et l’échantillonnage du milieu d’écoulement. Chaque chambre peut être connectée à un milieu différent, ce qui permet d’obtenir les meilleures conditions de culture pour chaque type de cellule. Chaque chambre peut être ouverte ou fermée en fonction des besoins spécifiques d’approvisionnement en fluide. Dans l’appareil, la chambre neuronale reste fermée pendant la majeure partie de l’expérience, tandis que les chambres bactériennes et épithéliales sont ouvertes en permanence, ce qui signifie qu’elles reçoivent un milieu frais tout au long de l’expérience. Pour s’assurer que le fluide s’écoule sans interruption, il est crucial de ne pas laisser d’air dans les tuyaux, les connecteurs ou dans l’appareil. Par conséquent, il est important de laisser d’abord les appareils fonctionner pendant quelques minutes à l’étape d’amorçage. Cela résout souvent le problème. Si ce n’est pas le cas, l’une des autres conduites qui tombent peut être fermée pendant une courte période en fermant le robinet d’arrêt à trois voies de l’écoulement. Cela redirige le fluide vers la ligne avec la bulle d’air, résolvant ainsi le problème en poussant la bulle vers l’extérieur à travers le tube.
Pour toute expérience de culture cellulaire, le milieu est un élément clé, où chaque type de cellule a son milieu respectif. Dans une installation de co-culture, le milieu doit être compatible non seulement avec le type de cellule qui s’y développe, mais aussi avec les autres types de cellules au sein de la co-culture. Ce n’est pas différent pour l’appareil, ce qui pose un défi supplémentaire car nous avons trois compartiments différents avec trois types de cellules différents à l’intérieur : cellules bactériennes, épithéliales et neuronales. Nous avons cependant montré qu’en modifiant le milieu bactérien - avec l’ajout de 5% de MRS au RPMI 1640 avec 10% de FBS - tous les types de cellules, en particulier les cellules bactériennes et épithéliales, peuvent être co-cultivés avec succès dans le système. Cependant, dans le dispositif, différents types de cellules sont co-cultivés à proximité et ne sont donc pas en contact direct les uns avec les autres. Même si cela n’est pas tout à fait représentatif du contact direct entre les cellules de l’intestin humain, et donc une limitation, la condition de co-culture proximale et représentative est un atout pour les analyses en aval. Échange de facteurs solubles entre les différentes chambres et types de cellules ; Par conséquent, les cellules interagissent toujours les unes avec les autres. De plus, le fait que les types de cellules puissent être récoltés et analysés séparément nous permet d’étudier l’effet d’un microbiome sain et/ou malade sur différents types de cellules (y compris les cellules neuronales) et ainsi de déterminer/récupérer des lectures spécifiques au type de cellule. Une autre limite est que la morphologie des cellules ne peut pas être suivie pendant le cycle expérimental, car le dispositif ne peut être ouvert et les cellules vérifiées qu’à la fin de chaque expérience.
À notre connaissance, neuroHuMiX est le premier modèle d’intestin sur puce incluant des EN. Il s’agit d’une étape vers l’élucidation de la communication entre le microbiote intestinal et le système nerveux entérique. Il s’agit d’un modèle permettant d’étudier l’interaction entre une espèce bactérienne, une couche épithéliale et les EN. Sa conception nous permet d’étudier l’échange de facteurs solubles sécrétés par les différents types de cellules et leur effet les uns sur les autres. À l’avenir, il serait important d’avoir non seulement des NE dérivés d’iPSC, mais aussi des cellules épithéliales dérivées d’iPSC à l’intérieur de l’appareil, afin de faire la transition de l’appareil vers un modèle personnalisé. Il est important de noter que ce modèle personnalisé pourrait être utilisé pour tester des prébiotiques, des probiotiques et des synbiotiques 10,11 et potentiellement développer des approches thérapeutiques et de dépistage personnalisées17. Personalized neuroHuMiX pourrait éventuellement faire la lumière sur la « matière noire » du microbiome intestinal humain et ses interactions avec le système nerveux le long de l’axe microbiome intestinal-système nerveux, ouvrant la voie à des évaluations thérapeutiques et à des interventions.
Nous pouvons conclure que la possibilité d’avoir un intestin sur puce incluant le système neuronal entérique est cruciale pour progresser dans l’étude et la compréhension des interactions le long de l’axe microbiome intestinal-système nerveux. NeuroHuMiX nous permet d’étudier les effets des espèces bactériennes sur les cellules hôtes et nous fournit une bonne base pour améliorer encore le modèle d’une manière encore plus représentative physiologiquement.
Déclarations de divulgation
P.W. déclare être inscrit en tant qu’inventeur sur les brevets PCT/EP2013/056607, PCT/EP2016/062024, PCT/US2017/061602 et PCT/EP2019/081424. P.W., C.S. et L.G. déclarent être inscrits en tant qu’inventeurs sur LU503075 de brevets.
Remerciements
Les auteurs tiennent à remercier le Dr Jared Sterneckert de nous avoir fourni les cellules de la lignée K7. Nous tenons également à remercier les collaborateurs de longue date, le Dr Frederic Zenhausern et Matthew W. Barret de l’Université de l’Arizona, pour leur aide en matière d’ingénierie. Nous tenons également à remercier la Dre Valentina Galata pour son aide dans la conception de la représentation schématique de neuroHuMiX. Ce projet a reçu un financement du Conseil européen de la recherche (ERC) dans le cadre du programme de recherche et d’innovation Horizon 2020 de l’Union européenne (convention de subvention 863664). La figure 1 a été partiellement créée à l’aide de Biorender.com.
matériels
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol | Sigma Aldrich | 10712 | |
| Aeration cannula (length: 1.10 diameter: 30 mm) | VWR (B.Braun) | BRAU4190050 | |
| Agar-agar | Merck Millipore | 1.01614.1000 | |
| Aluminium Crimp | Glasgerätebau Ochs | 102050 | |
| Ascorbic acid | Sigma Aldrich | A4544 | |
| B-27 Supplement Minus Vitamin A (50x) | Gibco | 12587-010 | |
| Bacterial Cell Membrane, pore size: 1 µm | VWR (Whatman) | 515-2084 | |
| Caco-2 cells | DSMZ | ACC169 | |
| Cell Counter & Analyzer CASY | OMNI Life Sceince | ||
| CHIR | Axon Mechem BV | CT99021 | |
| Collagen I, Rat Tail | Invitrogen | A1048301 | |
| Costar 6-well Clear Flat Bottom Ultra-Low Attachment Plates | Corning | 3471 | |
| Difco Lactobacilli MRS Broth | BD Biosciences | 288130 | |
| Discofix 3-way stopcock | B. Braun | BRAU40951111 | |
| DMEM/F12, no glutamine | Thermofisher Scientific | 21331020 | |
| Dulbecco's Phosphate-Buffered Saline, D-PBS | Sigma Aldrich | 14190-169 | |
| Essential 6 Medium | Thermofisher Scientific | A1516401 | |
| Essential 8 Medium | Thermofisher Scientific | A1517001 | |
| Female Luer Lock to Barb Connector | Qosina | 11733 | |
| FGF2 | R&D Systems | 233-FB | |
| Fibronectin | Sigma Aldrich | F1141 | |
| Foetal Bovine Serum, FBS | Thermofisher Scientific | 10500-064 | |
| GDNF | PeproTech | 450-10 | |
| Human Cell Membrane, pore size: 50 nm | Sigma Aldrich (GE Healthcare) | WHA111703 | |
| HuMiX Gasket Collagen | Auer Precision | 216891-003 | |
| HuMiX Gasket Sandwich Bottom | Auer Precision | 216891-002 | |
| HuMiX Gasket Sandwich Top | Auer Precision | 216891-001 | |
| iPSC | Max Planck Institute for Molecular Biomedicine | K7 line | |
| L-Glutamine (200 mM) | Gibco | 25030081 | |
| Laminin from Engelbreth-Holmswarm | Sigma Aldrich | L2020 | |
| LDN193189 | Sigma Aldrich | SML0559 | |
| Limosilactobacillus reuteri | ATCC | 23272 | |
| Live/Dead BacLight Bacterial Viability kit | Thermofisher Scientific | L7012 | |
| Male Luer with Spin Lock to Barb | Qosina | 11735 | |
| Marprene tubing (0.8 mm x 1.6 mm) | Watson-Marlow | 902.0008.J16 | |
| Matrigel hESC-qualified matrix | Corning | 354277 | |
| Mucin, from porcine stomach | Sigma Aldrich | T3924 | |
| N2 Supplement (100x) | Gibco | 17502048 | |
| NEAA | Thermofisher Scientific | 11140050 | |
| Needle (length: 120 mm; diameter: 0.80 mm) | B.Braun (color code: green) | 466 5643 | |
| Needle (length: 40 mm; diameter: 0.70 mm) | Henke Sass Wolf (color code: black) | 4710007040 | |
| Needle (length: 80 mm; diameter: 0.60 mm) | B.Braun (color code: blue) | 466 5635 | |
| Neurobasal Medium | Gibco | 21103049 | |
| PE/Cy7 anti-human CD49d antibody | Biolegend | 304314 | |
| Penicillin-Streptomycin | Sigma Aldrich | P0781 | |
| Peristaltic pump | Watson-Marlow | 205CA | |
| Poly-L-ornithine Hydrobromide | Sigma Aldrich | P3655 | |
| Polycarbonate lids (HuMiX) | University of Arizona | HuMiX 1.0 / 2.0 | |
| Retinoic Acid | Sigma Aldrich | R2625 | |
| RLT Buffer (RNeasy Minikit) | Qiagen | 74104 | |
| RPMI 1640 Medium | Thermofisher Scientific | 72400-021 | |
| SB431542, ALK inhibitor | Abcam | ab120163 | |
| Serum bottles | Glasgerätebau Ochs | 102091 | |
| Syringe | BD Biosciences | 309110 | |
| Trypsin-EDTA solution | Sigma Aldrich | T3924 | |
| Y-27632 Dihydrochloride | R&D Systems | 1254 |
Références
- Heintz-Buschart, A., Wilmes, P. Human gut microbiome: function matters. Trends in Microbiology. 26 (7), 563-574 (2018).
- Toor, D., et al. Dysbiosis disrupts gut immune homeostasis and promotes gastric diseases. International Journal of Molecular Sciences. 20 (10), 2432 (2019).
- Braak, H., de Vos, R. A. I., Bohl, J., Del Tredici, K. Gastric α-synuclein immunoreactive inclusions in Meissner's and Auerbach's plexuses in cases staged for Parkinson's disease-related brain pathology. Neuroscience Letters. 396 (1), 67-72 (2006).
- Schmit, K. J., et al. Dietary fibre deprivation and bacterial curli exposure shift gut microbiome and exacerbate Parkinson's disease-like pathologies in an alpha-synuclein-overexpressing mouse. bioRxiv. , (2022).
- Fritz, J. V., Desai, M. S., Shah, P., Schneider, J. G., Wilmes, P. From meta-omics to causality: experimental models for human microbiome research. Microbiome. 1 (1), 14 (2013).
- Fattahi, F., et al. Deriving human ENS lineages for cell therapy and drug discovery in Hirschsprung disease. Nature. 531 (7592), 105-109 (2016).
- Wu, Q., et al. Organ-on-a-chip: Recent breakthroughs and future prospects. BioMedical Engineering Online. 19 (1), 9 (2020).
- May, S., Evans, S., Parry, L. Organoids, organs-on-chips and other systems, and microbiota. Emerging Topics in Life Sciences. 1 (4), 385-400 (2017).
- Shah, P., et al. A microfluidics-based in vitro model of the gastrointestinal human-microbe interface. Nature Communications. 7, 11535 (2016).
- Greenhalgh, K., et al. Integrated in vitro and in silico modeling delineates the molecular effects of a synbiotic regimen on colorectal-cancer-derived cells. Cell Reports. 27 (5), 1621-1632 (2019).
- Mao, J. H., et al. Genetic and metabolic links between the murine microbiome and memory. Microbiome. 8 (1), 53 (2020).
- Moysidou, C. M., Owens, R. M. Advances in modelling the human microbiome-gut-brain axis in vitro. Biochemical Society Transactions. 49 (1), 187-201 (2021).
- Kim, S., et al. Transneuronal propagation of pathologic α-synuclein from the gut to the brain models Parkinson's disease. Neuron. 103 (4), 627-641 (2019).
- Hugenholtz, F., de Vos, W. M. Mouse models for human intestinal microbiota research: a critical evaluation. Cellular and Molecular Life Sciences. 75 (1), 149-160 (2018).
- Marshall, J. J., Mason, J. O. Mouse vs man: Organoid models of brain development & disease. Brain Research. 1724, 146427 (2019).
- Goers, L., Freemont, P., Polizzi, K. M. Co-culture systems and technologies: taking synthetic biology to the next level. Journal of the Royal Society Interface. 11 (96), 20140065 (2014).
- Sedrani, C., Wilmes, P. Toward hypothesis-driven, personalized microbiome screening. Cell Reports Methods. 2 (1), 100139 (2022).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon