Method Article
Culture à long terme de coupes organotypiques de moelle épinière de souris comme plate-forme de validation de la transplantation cellulaire dans les lésions de la moelle épinière
Dans cet article
Résumé
Dans cet article, nous proposons une méthode reproductible pour générer et maintenir à long terme des coupes organotypiques de la moelle épinière transplantées avec des cellules souches neurales comme modèle ex vivo pour tester des thérapies de remplacement cellulaire.
Résumé
Il n’existe toujours pas de remèdes résolutifs pour les lésions de la moelle épinière (LME) en raison de la physiopathologie complexe. L’une des approches régénératives les plus prometteuses est basée sur la greffe de cellules souches pour remplacer les tissus perdus et favoriser la récupération fonctionnelle. Cette approche devrait être mieux explorée in vitro et ex vivo pour en assurer l’innocuité et l’efficacité avant de procéder à des essais sur des animaux, qui sont plus coûteux et prennent plus de temps. Dans ce travail, nous montrons la mise en place d’une plateforme à long terme basée sur des coupes organotypiques de moelle épinière (CS) de souris transplantées avec des cellules souches neurales humaines pour tester des thérapies de remplacement cellulaire pour les LME.
Les cultures organotypiques SC standard sont maintenues pendant environ 2 ou 3 semaines in vitro. Nous décrivons ici un protocole optimisé pour la maintenance à long terme (≥30 jours) jusqu’à 90 jours. Le milieu utilisé pour la culture à long terme des tranches SC a également été optimisé pour la transplantation de cellules souches neurales dans le modèle organotypique. Des cellules souches neuroépithéliales humaines dérivées de SC (h-SC-NES) portant un rapporteur de protéine fluorescente verte (GFP) ont été transplantées dans des tranches de SC de souris. Trente jours après la greffe, les cellules présentent toujours une expression de GFP et un faible taux d’apoptotique, ce qui suggère que l’environnement optimisé a soutenu leur survie et leur intégration à l’intérieur du tissu. Ce protocole représente une référence solide pour tester efficacement les thérapies de remplacement cellulaire dans le tissu SC. Cette plateforme permettra aux chercheurs d’effectuer un pré-dépistage ex vivo de différentes thérapies de transplantation cellulaire, ce qui les aidera à choisir la stratégie la plus appropriée avant de procéder à des expériences in vivo .
Introduction
Les lésions traumatiques de la moelle épinière (LME) ont des conséquences physiques, psychologiques et économiques dévastatrices pour les patients et les soignants1. De nombreuses tentatives ont été faites pour favoriser la régénération axonale dans les lésions médullaires avec différentes approches 2,3,4 et certains effets bénéfiques ont été démontrés par la formation de relais neuronaux entre les neurones proximaux et distaux dans le site de la lésion par le biais de thérapies de remplacement cellulaire. L’intérêt pour les thérapies cellulaires ne cessede croître 5 car les cellules transplantées peuvent jouer de nombreux rôles, notamment en fournissant un soutien trophique, une modulation immunitaire, en régénérant les circuits neuronaux perdus par induction de la plasticité, en remplaçant les cellules et en remyélinisant des axones6.
Récemment, l’effort principal dans le domaine s’est concentré sur les cellules souches/progénitrices neurales humaines (NSC/NPC)7. Plusieurs études suggèrent que les NSC/NPC modulent la réponse astrocytaire8, favorisent la sécrétion de facteurs prorégénératifs 9,10 et remplacent les cellules neuronales manquantes dans les lésions médullaires11,12. Cependant, les études qui soutiennent la différenciation des cellules transplantées en neurones fonctionnels sont encore médiocres. De plus, la survie et la différenciation des cellules transplantées dans la moelle épinière lésée (SC) sont faibles13, peut-être parce que les cellules transplantées mettent plusieurs semaines, voire des mois, à se différencier in vivo. De plus, les études actuelles n’ont pas complètement élucidé de nombreux aspects biochimiques, moléculaires, cellulaires et fonctionnels des thérapies de remplacement cellulaire. Dans ce contexte, des modèles simples, rapides et rentables sont nécessaires pour étudier les mécanismes de la greffe cellulaire, la capacité des cellules greffées à proliférer, à se différencier en types spécifiques ou sous-populations de cellules et à former des synapses avec les neurones résidents.
L’intégration des études histologiques dans l’enregistrement électrophysiologique et le profilage du transcriptome et du protéome est nécessaire pour une compréhension complète de la cascade moléculaire qui se produit après la greffe de cellule. Cela accélérera certainement la conception et la validation de nouvelles thérapies de remplacement cellulaire dans des modèles précliniques et des études cliniques. En effet, à ce jour, l’utilisation de rongeurs, de grands animaux et de primates non humains a permis d’élucider de nombreux processus cellulaires après transplantation14. Cependant, en raison de leur coût élevé, de leur impact éthique élevé et de la complexité de l’organisme, leur utilisation n’est souvent pas simple ou insuffisante pour démêler les processus biochimiques et moléculaires. De plus, ils peuvent présenter de nombreux inconvénients corrélés à des différences biologiques, à la fois inter-espèces (métabolisme) et intra-espèces (sexe, âge). Ces facteurs, ainsi que des facteurs externes tels que des situations stressantes, pourraient modifier le résultat d’une expérience et leur prévisibilité en termes d’application thérapeutique chez l’homme 15,16,17.
Ainsi, de nombreux groupes utilisent la culture cellulaire in vitro 2D et des coupes organotypiques ex vivo (cultures ex vivo) en plus des modèles animaux. La culture cellulaire 2D est le système le plus couramment utilisé pour étudier des processus biologiques spécifiques au niveau d’une cellule unique et/ou d’une population cellulaire. Néanmoins, les cultures cellulaires monocouches ne reflètent pas la complexité que l’on trouve dans un organisme entier. L’absence de structures tissulaires et d’environnement physiologique ne permet pas aux systèmes de culture 2D d’imiter complètement les principaux aspects structurels, morphologiques et fonctionnels des tissus étudiés 18,19,20.Les cultures organotypiques peuvent surmonter certains de ces problèmes. Les modèles organotypiques sont basés sur l’explantation d’un fragment d’un tissu ou d’un organe et son maintien ex vivo pendant une période limitée21,22. En particulier, des tranches de tissu explanté sont générées avec une épaisseur précise qui permet aux nutriments d’atteindre facilement presque toutes les cellules des tranches. Ils peuvent être générés à partir de diverses régions du système nerveux central, telles que l’hippocampe, l’hypothalamus, le cervelet, le thalamus, le cortex cérébral, la substance noire et le striatum, et la moelle épinière23. Les cultures organotypiques conservent l’architecture tissulaire, la distribution spatiale des cellules, la diversité cellulaire et l’environnement (c’est-à-dire la composition de la matrice extracellulaire) de l’organe d’origine. De plus, ils préservent l’activité neuronale d’origine, les connexions entre les cellules, et en particulier, les circuits à courte distance après l’explantation.
Ces aspects offrent certains avantages pour les cultures ex vivo par rapport aux cultures monocouches et aux modèles animaux. Ils conservent les principales caractéristiques tissulaires trouvées in vivo, mais avec la réduction des coûts et la possibilité de réaliser différents types d’expériences moléculaires, cellulaires et fonctionnelles avec une régulation précise des paramètres environnementaux de la culture 24,25,26,27,28,29. Les coupes organotypiques peuvent également être exploitées pour développer des modèles pour différents troubles neurologiques en ressemblant à des caractéristiques histopathologiques clés de conditions spécifiques30. De plus, la conservation de l’environnement tissulaire multicellulaire d’origine en fait des plateformes appropriées pour le criblage de médicaments et pour tester des molécules et des matériaux neuroprotecteurs et neuro-régénératifs.
Dans ce travail, nous proposons l’utilisation de cultures organotypiques SC comme modèle pour optimiser les greffes de NSC. Ce n’est pas anodin puisque des conditions de culture optimales sont nécessaires pour garantir la survie de l’hôte (tissu SC) et de la greffe (CSN) pendant des semaines. Différents groupes de recherche greffés dans des cultures organotypiques, dérivées du cerveau et dérivées de SC, divers types de cellules. La plupart des travaux ont montré la transplantation de cellules souches mésenchymateuses31,32,33, de cellules olfactives34, ou NSCs 35,36,37,38,39,40 et ont évalué les interactions des cellules greffées avec les cellules hôtes, la survie de l’ensemble du système et si les cellules transplantées se différenciaient en neurones ou en cellules neuronales à l’intérieur de l’environnement tissulaire ex vivo 32,33,41. Certains d’entre eux ont évalué le potentiel de régénération des cellules après la greffe, en observant leur croissance axonale à l’intérieur du tissu 37,40,41, la capacité myélinisante des précurseurs greffés d’oligodendrocytes 42, la migration des cellules greffées dans le tissu hôte43, et si les cellules transplantées libéraient des facteurs poussant vers un environnement prorégénératif31. L’une des limites des études actuelles est qu’elles n’explorent pas la greffe sur une période à long terme.
Étant donné que les NSC semblent nécessiter plusieurs semaines pour se différencier in vivo44,45, cette étude se concentre sur la façon de générer et de maintenir à long terme (≥30 jours) des tranches de CS de souris jusqu’à 90 jours. On a constaté que les coupes conservaient leur structure anatomique d’origine et maintenaient un taux d’apoptose faible et stable au fil du temps et une viabilité cellulaire élevée. Nous avons observé une expression diffuse des marqueurs neuronaux de liaison à l’ARN fox-1 homologue 3 (RBFOX3) et de la chaîne légère des neurofilaments (NFL), ces dernières montrant une tendance croissante à la germination axonale autour des tranches au fil du temps, attestant de leur état de santé. De plus, nous avons transplanté avec succès dans les tranches SC des cellules souches neuroépithéliales humaines dérivées de SC (H-SC-NES) exprimant la GFP aux premiers stades de la différenciation neuronale. Le greffon de NSC a été maintenu pendant 30 jours après la greffe et les cellules ont montré une expression de la GFP pendant toute la période de culture. Le taux d’apoptose des cellules le jour suivant la greffe (DPT) 30 s’est également avéré être conforme à la valeur du taux d’apoptose observée à la DPT 7 dans les mêmes cellules40. Les cellules semblaient se greffer dans l’environnement tissulaire et survivaient jusqu’à plusieurs semaines.
En résumé, nos données démontrent qu’il est possible de maintenir en culture des tranches organotypiques SC pendant 3 mois sans compromettre leur cytoarchitecture d’origine et l’environnement tissulaire. Plus important encore, ils peuvent être exploités pour tester des thérapies cellulaires avant de procéder à une expérience in vivo , réduisant ainsi les coûts et le temps expérimental. Ici, nous illustrons en détail tous les passages pour générer des tranches organotypiques de SC de souris et comment les maintenir pendant de longues périodes (≥30 jours). De plus, nous expliquons en profondeur comment effectuer la transplantation de NPC dans les tranches et comment les maintenir pour l’analyse en aval.
Protocole
Les procédures sur les animaux ont été effectuées dans le strict respect des protocoles approuvés par le ministère italien de la Santé publique et le comité d’éthique local de l’Université de Pise, conformément à la directive 2010/63/UE (licence de projet n° 39E1C. N.5Q7 publié le 30/10/2021). Les souris C57BL/6J ont été maintenues dans un environnement réglementé (23 ± 1 °C, 50 ± 5 % d’humidité) avec un cycle lumière-obscurité de 12 h avec de la nourriture et de l’eau à volonté.
Tous les travaux liés aux cellules h-SC-NES ont été effectués conformément aux directives des NIH pour l’acquisition et la distribution de tissus humains à des fins de recherche biomédicale et avec l’approbation des comités d’investigation humaine et des comités d’éthique institutionnels de chaque institut d’où des échantillons ont été obtenus. L’approbation finale a été obtenue de la Commission de bioéthique de l’Université de Pise (Revue n° 29/2020). Des échantillons humains anonymisés ont été fournis par la subvention conjointe MRC/Wellcome Trust (099175/Z/12/Z), Human Developmental Biology Resource (www.hdbr.org). Un consentement éclairé approprié a été obtenu, et tous les renseignements non identificatoires disponibles ont été consignés pour chaque échantillon. Les tissus ont été manipulés conformément aux directives éthiques et aux réglementations pour l’utilisation des tissus cérébraux humains à des fins de recherche, établies par le NIH (http://bioethics.od.nih.gov/humantissue. html) et la Déclaration d’Helsinki de l’AMM (http://www.wma.net/en/30publications/10policies/b3/index.html).
1. Préparation de solutions et d’équipements pour l’isolement et la culture de la moelle épinière
- Solution de revêtement pour inserts de membrane
- Préparez la solution d’enrobage (tableau 1) : une solution aqueuse avec 0,1 mg mL-1 de collagène, 0,01 mg mL-1 de poly-L-lysine et 0,01 mg mL-1 de laminine.
- Placez chaque insert à membrane dans un plat de 35 mm ou une plaque à 6 puits.
- Ajouter sur le dessus de la membrane 1 mL de solution d’enrobage : incuber la solution pendant 4 h à température ambiante (RT) ; ensuite, retirez-le et laissez l’insert de membrane sécher pendant la nuit (ON). Stockez les inserts membranaires à 4 °C jusqu’à leur utilisation.
REMARQUE : Tous les passages doivent être effectués dans des conditions stériles. Le revêtement de la membrane doit être conservé pendant un maximum de 1 semaine à 4 °C avant utilisation afin d’éviter la dégradation des protéines et l’adhésion sous-optimale de la tranche à la membrane.
- Préparation des milieux : milieu organotypique, milieu de dissection, milieu de greffe
- Préparer le milieu organotypique (MO, Tableau 1).
- Préparez le produit de dissection comme décrit dans le tableau 1.
- Préparez le milieu de greffe (GM, optimisé d’après Onorati et al.46, Tableau 1).
REMARQUE : Les solutions doivent être préparées dans des conditions stériles et juste avant leur utilisation (1 jour avant ou le jour même de l’expérience).
- Préparation du matériel pour la chirurgie
- Dans l’enceinte de biosécurité, gardez les éléments suivants prêts à l’emploi : le stéréomicroscope de dissection et les instruments chirurgicaux : deux paires de micro-ciseaux, deux paires de pinces à épiler droites et deux paires de pinces à épiler courbes.
- Dans l’enceinte de biosécurité, préparez l’instrument hachoir en l’équipant d’une lame pour couper le SC en tranches. Tournez une vis du hachoir pour soulever le bras métallique où la lame doit être placée. Placez la lame à l’endroit désigné, abaissez le bras métallique avec la lame jusqu’à ce qu’il entre en contact avec le plateau de coupe et fixez-le en serrant la vis de sécurité avec une clé hexagonale jusqu’à ce que la lame soit fermement fixée. Faites pivoter la vis micrométrique jusqu’à l’épaisseur de tranche souhaitée (généralement 350 μm). Vérifiez si la lame est placée précisément perpendiculairement au plateau de coupe.
REMARQUE : Un placement précisément perpendiculaire de la lame par rapport au plateau de coupe est nécessaire pour effectuer la coupe correctement. - Préparez dans l’enceinte de biosécurité : deux pipettes Pasteur en plastique (nécessaires pour déplacer les SC isolés et les tranches), au moins quatre plats de 35 mm et deux de 60 mm, et une boîte de glace fraîche.
- Stérilisez tous les instruments avec de l’éthanol à 70% et des UV (un cycle de 20 min) juste avant leur utilisation pour préserver la stérilité de la culture.
2. Isolation de la souris SC et génération de tranches
- Isolation de la souris SC
- Sacrifiez des bébés souris postnatals du jour 3 (P3) selon la licence du projet.
- À l’aide d’une laparotomie médiane à l’aide des micro-ciseaux, isolez la région lombaire de la colonne vertébrale du reste du corps de la souris et placez-la dans un milieu de dissection froid dans une boîte de 35 mm.
- À l’aide d’un stéréomicroscope de dissection et des micro-ciseaux, coupez la colonne vertébrale le long de l’axe sagittal et utilisez une pince à épiler droite pour retirer délicatement le SC de la cavité de la colonne vertébrale.
- Décollez soigneusement les méninges de la région lombaire isolée du SC à l’aide d’une pince à épiler droite.
- Transférez et incubez la région lombaire SC isolée dans un milieu de dissection froid et frais pendant 10 à 15 minutes avant de passer à l’étape suivante.
- Génération de tranches
- À l’aide d’une pipette Pasteur en plastique, prélevez la région lombaire SC isolée du milieu de dissection et placez-la sur le plateau de coupe de l’instrument hachoir, perpendiculairement à la lame.
REMARQUE : Le positionnement correct du SC par rapport à la lame (perpendiculaire) est essentiel pour générer correctement des tranches SC. - Retirez le milieu de dissection résiduel sur le pont autour du SC à l’aide de la pipette Pasteur et du papier absorbant stérile. Procéder au sectionnement automatisé SC.
- Une fois les tranches générées, placez un peu de milieu de dissection frais avec une pipette Pasteur sur le plateau de coupe avec les tranches. Ensuite, rassemblez les tranches dans un plat de 35 mm avec un milieu de dissection frais et incubez-les pendant 15 min.
- Pendant l’incubation des tranches, laver la surface des inserts membranaires 3 fois avec OM à l’aide d’une pipette Pasteur en plastique. Ensuite, laissez 1 mL de MO au bas de chaque insert de membrane.
- Vérifiez les coupes sous le stéréomicroscope de dissection. Ensemencez le nombre désiré de tranches sur les inserts de membrane conditionnés en les transférant avec une pipette Pasteur en plastique.
- Déplacez les tranches dans l’orientation et la position souhaitées sur les inserts de membrane à l’aide de la pince à épiler droite. Retirer l’excédent de milieu à l’aide d’une pipette Pasteur pour permettre aux tranches de mieux adhérer à la surface de la membrane.
REMARQUE : Le déplacement et l’orientation des tranches avec la pince à épiler droite doivent être effectués en douceur pour éviter d’endommager les tissus ou les inserts de membrane. - Après 30 min d’incubation à 37 °C, transférez l’insert dans une nouvelle boîte de Pétri.
REMARQUE : Touchez l’anneau en plastique mais pas les membranes lors du changement de fluide. - Ajouter 1 mL de MO fraîche complétée par du facteur neurotrophique dérivé de la lignée cellulaire gliale (GDNF) 100 ug mL-1 au bas de l’insert de la membrane.
- Incuber les tranches à 37 °C. Se référer au premier jour en culture comme jour ex vivo (DEV) 0.
- À l’aide d’une pipette Pasteur en plastique, prélevez la région lombaire SC isolée du milieu de dissection et placez-la sur le plateau de coupe de l’instrument hachoir, perpendiculairement à la lame.
3. Culture à long terme de tranches organotypiques
- Maintenir les tranches en culture à 37 °C jusqu’aux points de temps souhaités.
- Remplacez le support par un MO frais au niveau DEV 1 comme décrit aux étapes 2.2.7-2.2.8.
- À DEV 3, passez le milieu au GM afin de créer l’environnement approprié pour la transplantation de cellules souches le lendemain. Remplacez le milieu par du GM frais toutes les 48 h (par exemple, DEV 5, DEV 7...).
- Ajouter du GDNF frais (concentration finale de 100 μg mL-1) au milieu tous les jours jusqu’à DEV 7. Après DEV 7, ajoutez-le uniquement lorsque le support est changé (étape 3.3).
4. Culture de cellules h-SC-NES
REMARQUE : Les cellules h-SC-NES sont maintenues en culture en présence de facteurs de croissance (milieu NES, étape 4.1.1). Avant la transplantation, les cellules sont mises en état de prédifférenciation pendant 7 jours en retirant les facteurs de croissance du milieu (milieu de prédifférenciation : milieu NES sans facteur de croissance des fibroblastes 2 (FGF-2) et facteur de croissance épidermique (EGF), étape 4.1.2). Ensuite, les cellules sont plaquées dans des conditions de différenciation (milieu de différenciation, étape 4.1.3) pendant 2 jours avant la transplantation. La différenciation est soutenue par l’ajout de suppléments neurotrophiques (facteur neurotrophique dérivé du cerveau, BDNF) au milieu de différenciation. L’entretien, la division, la pré-différenciation et la différenciation des cellules h-SC-NES12,46 sont décrits en détail ci-dessous.
- Préparation des milieux : NES, prédifférenciation et milieux de différenciation
- Préparez le milieu d’entretien de la cellule h-SC-NES (milieu NES, tableau 1).
- Préparer le milieu de prédifférenciation cellulaire h-SC-NES (milieu de pré-différenciation, tableau 1).
- Préparer le milieu de différenciation cellulaire h-SC-NES (Milieu de différenciation, Tableau 1). Ajoutez du BDNF fraîchement lorsque le milieu est changé ou lorsque les cellules sont plaquées pour la première fois dans la condition de différenciation.
REMARQUE : Tous les milieux doivent être préparés dans des conditions stériles et doivent être filtrés avec des filtres de 0,22 μm.
- Solution de revêtement pour cellules h-SC-NES
REMARQUE : les cellules h-SC-NES sont maintenues dans des supports de culture enrobés de POLFN (POLFN = Poly-L-Ornithine, Laminin, Fibronectin).- Préparez la solution d’enrobage dans un tube : une solution de Poly-L-Ornithine avec de la laminine (5 μg mL−1) et de la fibronectine (1 μg mL−1).
- Transférez la solution d’enrobage préparée dans le support de culture cellulaire, en prenant soin d’en ajouter suffisamment pour couvrir toute la surface du support de culture cellulaire. Incuber l’enrobage pendant 1 h à 37 °C ou une nuit à 4 °C.
- Retirer la solution d’enrobage des supports de culture cellulaire.
REMARQUE : La solution POLFN peut être recyclée deux fois de plus, mais la laminine et la fibronectine doivent être ajoutées fraîches à chaque fois. - Lavez l’enrobage 3 fois avec de l’eau stérile de qualité culture cellulaire. Stockez les revêtements à 4 °C ou utilisez-les.
REMARQUE : Les revêtements doivent être utilisés dans un délai de 1 semaine. Après cela, les revêtements sont considérés comme périmés en raison des processus de dégradation des protéines ajoutées.
- Maintenance de la cellule h-SC-NES
- Maintenir les cellules h-SC-NES en culture dans un milieu NES. Vérifiez les cellules tous les jours au microscope pour savoir quand elles atteignent la confluence.
- Changez la moitié du milieu tous les 2 jours : retirez la moitié du milieu conditionné et ajoutez le frais (comptez un taux d’évaporation de 20%).
- Si les cellules atteignent la confluence, procédez à la séparation, comme décrit à l’étape 4.4.
- Passage de la cellule h-SC-NES
REMARQUE : Les cellules sont divisées comme suit12 :- Retirez le milieu conditionné et lavez les cellules une fois avec la solution saline tamponnée au phosphate de Dulbecco (DPBS) sans Ca2+/Mg2+.
- Retirez le DPBS et ajoutez une solution de trypsine/EDTA aux cellules pour effectuer un détachement enzymatique. Incuber les cellules à 37 °C pendant 30 s à 1 min.
- Après l’incubation, vérifiez les cellules au microscope : si elles ne sont pas détachées, tapotez légèrement le support de culture cellulaire pour effectuer un détachement mécanique et incuberez-les à 37 °C pendant 30 s de plus.
- Après l’incubation, inactiver la trypsine/EDTA en ajoutant 4 volumes de solution de DPBS/sérum de veau fœtal (FBS) (10% vol/vol) au support de culture cellulaire avec des cellules et de la trypsine/EDTA. Pipetez doucement la solution sur la surface du support de culture cellulaire de haut en bas pour aider toutes les cellules à se détacher. Recueillir la suspension cellulaire dans un tube.
- Centrifuger la suspension cellulaire à 200 × g pendant 3 min. Jetez le surnageant et remettez la pastille en suspension dans un milieu NES frais.
- Compter les cellules et les déposer sur chaque nouveau support de culture enrobé de POLFN à une densité de ̴0,5-1 × 105 cellules/cm2.
- Ajouter l’Y-27632 (10 μM) et placer les cellules à 37 °C. Vérifiez-les tous les jours jusqu’à la confluence, puis divisez-les à nouveau pour la maintenance/l’expansion ou la banque de cellules.
- Prédifférenciation cellulaire h-SC-NES
- Divisez les cellules comme décrit à l’étape 4.4.
- Plaquez les cellules sur les supports de culture cellulaire revêtus de POLFN à une densité de ̴0,5-1 × 105 cellules/cm2 dans un milieu de prédifférenciation. Ajouter Y-27632 (10 μM) après la séparation. Appelez le premier jour de la journée de prédifférenciation in vitro (DIV) 0.
- Changez la moitié du milieu tous les 2-3 jours (voir étape 4.3.2).
- Maintenez les cellules en état de prédifférenciation jusqu’à la DIV 7, puis passez à l’étape 4.6.
- Différenciation cellulaire h-SC-NES
- À la DIV 7 de la prédifférenciation, divisez les cellules comme décrit à l’étape 4.4.
- Plaquez les cellules sur des supports de culture cellulaire recouverts de POLFN à une densité de ̴1-1,5 × 105cellules/cm2dans un milieu de différenciation. Ajouter de l’Y-27632 (10 μM) et du BDNF (30 ng mL−1) après la séparation.
- Après 2 jours de différenciation (DIV 10), divisez les cellules pour la transplantation en tranches.
5. Transduction cellulaire h-SC-NES avec des vecteurs lentiviraux porteurs de GFP
REMARQUE : La transduction des cellules est effectuée pendant la phase de maintenance des cellules h-SC-NES. Lorsque les cellules sont correctement transduites, celles-ci peuvent être dilatées et les protocoles de prédifférenciation et de différenciation décrits précédemment sont appliqués (étapes 4.5 et 4.6).
- Préparation du milieu de transduction cellulaire
REMARQUE : Le milieu de transduction cellulaire est préparé en mélangeant un volume spécifique de milieu NES et un volume précis de stock de vecteurs lentiviraux (LVS) en fonction de différents paramètres : le MOI souhaité (multiplicité de l’infection = rapport entre le nombre de particules virales et le nombre de cellules hôtes dans un milieu d’infection donné) ; le nombre de cellules plaquées ; la concentration initiale de la préparation EFV (=EFV PFU, unité formant des plaques) ; la surface du récipient de culture utilisé.- Calculer le volume correct de préparation d’EFV à ajouter au milieu NES, en fonction du MOI choisi à l’aide des équations (1) et (2).
(n cellules par plaque/cm2) × cm2 de support de culture cellulaire × MOI = LVS PFU pour le MOI choisi (1)
LVS PFU : Tot Initial LVS Vol (μL) = LVS PFU pour MOI : LVS Vol à ajouter au moyen (μL)(2)
REMARQUE : Le PFU LVS (PFU initial du LVS) et le volume initial total du LVS sont indiqués par le fabricant. L’EFV PFU pour le MMŒ choisi est calculé comme décrit dans l’équation (1). Ainsi, nous pouvons obtenir le volume de préparation de LVS qui doit être ajouté au volume total de milieu NES (basé sur le support de culture cellulaire) pour le MOI choisi, comme décrit dans l’équation (2).
Exemple : Nous avons utilisé le MOI 3, sur la base de l’expérience de laboratoire précédente (le MOI peut varier en fonction de la lignée cellulaire utilisée et de la préparation virale). Si le moment d’inertie souhaité est de 3, que le nombre de cellules à plaquer est de 0,5 × 105/cm2 et que le support de culture est de 1 puits-MW24 (2 cm2), en supposant que le LVS PFU/TU (unité de formation de plaques/unité de transduction) initial est de 25 × 106 PFU dans 1 mL (1 000 μL = volume initial LVS), les calculs sont les suivants :
Cellules plaquées dans 1 puits-MW24 (2 cm2) = 0,5 × 105 cellules × 2 cm2 = 1 × 105 cellules
1 × 105 cellules × 3 (MOI) = 3 × 105 PFU = LVS PFU pour MOI 3
25 × 106 UFP : 1 000 μL = 3 × 105 UFP : x μL
x μL = 12 μL = volume LVS à ajouter au milieu
Ainsi, pour transduire les cellules avec un milieu de transduction cellulaire avec un MOI 3 dans 1 puits-MW24, ajouter 12 μL de préparation initiale de LVS au milieu NES (238 μL) préparé pour 1 puits de MW24. Le volume total final est de 250 μL.
REMARQUE : Le milieu est généralement préparé fraîchement le jour de la transduction dans des conditions stériles.
- Calculer le volume correct de préparation d’EFV à ajouter au milieu NES, en fonction du MOI choisi à l’aide des équations (1) et (2).
- Protocole de transduction h-SC-NES
- Plaquez les cellules h-SC-NES à faible passage sur une plaque 24-multi puits revêtue de POLN (ou dans tout autre support de culture) à une densité de 0,5 × 105/cm2 en milieu NES.
- Le lendemain, récupérez le milieu NES conditionné dans les puits où les cellules ont été placées. Selon le support de culture choisi, ajouter aux cellules le plus petit volume de milieu de transduction de cellules fraîches nécessaire pour couvrir uniformément la surface d’ensemencement (p. ex., 250 μL/puits d’une plaque à 24 puits multiples).
- Ensuite, incubez les cellules h-SC-NES pendant 6 h à 37 °C. Après cela, ajoutez le milieu conditionné précédemment collecté dans les cellules (200 μL/puits d’une plaque à 24 puits multiples) et incubez les cellules ON à 37 °C.
- Le lendemain, lavez une fois les cellules h-SC-NES avec du DPBS et effectuez un changement total de milieu (milieu NES).
- Les jours suivants, vérifiez les cellules au microscope à fluorescence pour observer l’expression de la GFP.
- Développer les cellules h-SC-NES pour la banque et la transplantation de cellules.
6. Transplantation cellulaire dans des tranches SC et co-culture
- Préparation des micro-aiguilles de verre
- À l’aide d’un extracteur, obtenir de fines aiguilles à partir de capillaires en verre borosilicaté. Réglez l’extracteur comme suit : HEAT 990, PULL 350.
REMARQUE : À partir d’un capillaire, il est possible d’obtenir deux aiguilles fines.
- À l’aide d’un extracteur, obtenir de fines aiguilles à partir de capillaires en verre borosilicaté. Réglez l’extracteur comme suit : HEAT 990, PULL 350.
- Préparation cellulaire pour la transplantation
- Divisez les cellules comme décrit à l’étape 4.4.
REMARQUE : Si les cellules n’expriment pas de rapporteur fluorescent, marquez-les avec un colorant de suivi cellulaire pour les surveiller à l’aide d’un microscope à fluorescence après la transplantation et pendant la culture à long terme. Suivez le protocole du fabricant choisi pour l’étape d’étiquetage. - Comptez les cellules après la séparation et centrifugez à 200 × g pendant 3 min. Suspendre la pastille obtenue avec un milieu frais + Y-27632 (10 μM) pour avoir la concentration souhaitée de cellules (généralement une plage comprise entre 30 000 et 50 000 cellules μL-1).
- Transférez la suspension cellulaire dans un tube de 500 μL ou 1,5 mL et placez-la sur de la glace. Les cellules sont prêtes pour la transplantation.
- Divisez les cellules comme décrit à l’étape 4.4.
- Transplantation cellulaire en tranches organotypiques
REMARQUE : Effectuez des greffes de cellules h-SC-NES dans les tranches organotypiques SC de souris à l’aide d’un micro-injecteur d’air et de micro-aiguilles en verre.- Chargez une micro-aiguille de verre avec 4 μL de suspension cellulaire à l’aide d’une micropipette et d’embouts de microchargeur.
REMARQUE : Évitez la formation de bulles d’air dans l’aiguille car cela pourrait entraver le processus de micro-injection. Si des bulles se forment, retirez-les à l’aide de la micropipette. - Placez l’aiguille dans le support assigné du micro-injecteur et cassez la pointe de l’aiguille à l’aide de la pince à épiler droite.
REMARQUE : Cassez l’aiguille de verre plus près de la pointe pour éviter la formation de gros trous. - Avant de transplanter dans les tranches, réglez les paramètres de micro-injection. Réglez la pression à 10 psi.
REMARQUE : La valeur de pression peut être modifiée en fonction du micro-injecteur et des observations de l’opérateur : la pression doit être suffisante pour micro-injecter la suspension cellulaire, évitant ainsi d’endommager les tissus. - Sur une lame de verre calibrée, déposez une goutte d’huile minérale à l’aide d’une pipette Pasteur et microinjectez la suspension cellulaire dans la goutte. Le diamètre de la sphère de suspension cellulaire obtenue dans la goutte d’huile est corrélé à un volume de micro-injection spécifique. Modifier les paramètres de micro-injection au besoin pour atteindre un diamètre de la sphère de suspension de la cellule de 0,2 mm pour l’injection de 4nL.
- Après avoir réglé le bon volume, micro-injectez rapidement la suspension cellulaire dans les tranches. Vérifiez au stéréomicroscope à fluorescence la présence de cellules dans les coupes pour vérifier que la microinjection/greffe a réussi.
REMARQUE : La suspension cellulaire peut parfois obstruer l’aiguille : dans ce cas, essayez de supprimer le bouchon de la suspension cellulaire en modifiant les paramètres d’injection ou chargez une nouvelle aiguille avec une suspension cellulaire fraîche. - Après la transplantation, placez les tranches à 37 °C et 5 % de CO2jusqu’au point temporel souhaité et effectuez un changement de milieu tous les deux jours comme décrit aux étapes 2.2.7-2.2.8.
- Chargez une micro-aiguille de verre avec 4 μL de suspension cellulaire à l’aide d’une micropipette et d’embouts de microchargeur.
7. Coloration par immunofluorescence
- Jour 1
- Retirez le milieu du bas de la membrane d’insertion et lavez les tranches 3x avec du DPBS préchauffé.
- Fixez les tranches avec du formaldéhyde (FA) préchauffé à 4 % : retirez le DPBS et ajoutez 1,5 mL de FA à 4 % au bas de l’insert de la membrane avec les tranches. Après 15 min d’incubation à RT, ajouter 1 mL de plus de 4 % d’AF sur la surface supérieure de l’insert membranaire et incuber pendant 15 min à RT. Temps de fixation total : 30 min à RT
- Retirez l’AF 4% et lavez les tranches pendant 3 x 10 min avec du DPBS.
- Coupez la membrane de l’insert sur la circonférence avec un couteau chirurgical, séparez la membrane avec les tranches du composant plastique de l’insert et procédez aux étapes d’immunofluorescence.
REMARQUE : Après cette étape, la membrane avec les tranches flotte dans DPBS dans le plat. - Perméabiliser avec 1 mL/membrane d’une solution contenant 0,7 % de Triton dans le DPBS pendant 10 min à RT.
- Retirer la solution de perméabilisation et incuber les échantillons pendant 4 h à 4 °C avec 1 mL/membrane de solution bloquante composée de 0,5 % de Triton et de 10 % de FBS dans du DPBS.
- Retirer la solution bloquante et ajouter aux tranches les anticorps primaires à leur dilution de travail, par exemple, anticorps anti-neurofilaments de souris (NFL), 1:500 ; lapin anti-NFL, 1:500 ; anticorps anti-RBFOX3 (NeuN) chez le lapin, 1:400 ; caspase-3 anti-active pour lapin (aCASP3), 1:400 ; noyaux anti-humains de souris, (Hu-Nu), 1:400 ; lapin anti-Hu-Nu, 1:400 ; anti-GFP de souris, 1:400 (comme indiqué dans le tableau 1) dans 1 mL de solution d’anticorps composée de 0,5 % de Triton, 1 % de FBS dans le DPBS. Incuber à 4 °C.
- Jour 2
- Lavez les membranes pendant 3 x 10 min avec 1 à 2 mL de DPBS.
- Incuber la membrane avec des anticorps secondaires (p. ex., anticorps secondaire anti-IgG de souris (H+L) de chèvre, Alexa Fluor 488, 1:500 ; Anticorps secondaire anti-IgG (H+L) de chèvre, Alexa Fluor 568, 1:500 ; anticorps secondaire anti-IgG de souris (H+L) de chèvre, Alexa Fluor 647, 1:500 ; Anticorps secondaires anti-IgG (H+L) de chèvre, Alexa Fluor 647, 1:500 comme indiqué dans le tableau 1) et Hoechst/DAPI pour les noyaux dilués dans 1 mL/membrane de solution d’anticorps (Triton 0,5 % + FBS 1 % dans DPBS) pendant 3 h à RT.
REMARQUE : Protégez soigneusement les échantillons de la lumière pour éviter le blanchiment secondaire des anticorps pendant l’incubation et dans les étapes suivantes. - Retirer la solution d’anticorps et laver pendant 3 x 10 min avec du DPBS (1-2 mL).
- Remplacez le DPBS par du DPBS frais et conservez-le à 4 °C dans des conditions protégées de la lumière.
- A la fin du protocole d’immunofluorescence, monter les membranes sur des lames de verre. Déposer une goutte de 200 μL de solution de montage sur une lame de verre. À l’aide d’une pince à épiler droite, transférez la membrane flottante de la parabole de 35 mm sur une lamelle, puis transférez la membrane sur la lame de verre avec la solution de montage.
- Mettez une goutte de 100 μL de solution de montage sur une nouvelle lamelle et recouvrez la membrane avec celle-ci, en la fixant sur la lame de verre. Laissez-le sécher toute la nuit sous le capot chimique dans des conditions protégées de la lumière.
- Stockez les échantillons à 4 °C dans l’obscurité ou effectuez une analyse d’imagerie.
8. Essai vivant/mort
- Préparer la solution de travail en aliquotant 700 μL par plat de milieu frais et ajouter, à la dilution de travail correcte, le Sytox (p. ex., composant B, 1:2 000) et le Calcein AM (p. ex., composant A, 1:2 000).
REMARQUE : Comme les réactifs sont sensibles à la lumière, protégez la solution de travail de la lumière. - Évaluer le volume moyen au fond de la membrane et ajouter le Sytox et le Calcein AM à la même dilution de travail décrite à l’étape 8.1.
- Ajouter 2 gouttes de 30 μL chacune sur le dessus de chaque tranche de la solution de travail préparée à l’étape 8.1.
REMARQUE : Protégez le plat de la lumière en le plaçant dans des conditions sombres. - Incuber les tranches pendant 30 min à RT.
- Après l’incubation, découpez la membrane circonférentiellement de l’insert à l’aide d’un couteau chirurgical : après cela, la membrane avec les tranches flotte dans la solution de travail.
- Placez la membrane sans la solution de montage à l’envers sur une lamelle à l’aide d’une pince à épiler droite et ajoutez 100 μL de DPBS sur les membranes pour les maintenir hydratées.
- Acquérez des images en direct à l’aide du microscope confocal le plus rapidement possible.
REMARQUE : Ajoutez 2 gouttes de 40 μL chacune de DPBS sur le dessus de la membrane toutes les 30 minutes pendant l’acquisition de l’image pour éviter que la membrane ne se dessèche.
9. Imagerie
- Imagerie confocale d’échantillons fixes
- Pour l’analyse qualitative, acquérez des images à l’aide d’un microscope confocal avec les paramètres d’acquisition suivants : réglez l’option grande image (choisissez : 4 x 4), utilisez un objectif 10x, sans empilement et une résolution de 3 634 x 3 634 pixels.
- Pour l’analyse quantitative (aCASP3, Calcein et Sytox pour les coupes et aCASP3 pour les cellules), acquérez des images à l’aide du microscope confocal avec les paramètres d’acquisition suivants : objectif 20x, résolution de 1 024 x 1 024 pixels avec un pas Z de 3 μm.
- Imagerie en direct de coupes transplantées à l’aide du stéréomicroscope
REMARQUE : Capturez des images à l’aide du stéréomicroscope en modes fond clair et épifluorescence.- À l’aide du réglage fond clair , acquérez des images des tranches (objectif 1 x avec zoom 3x utilisé ici).
REMARQUE : Modifiez la lumière en fonction du microscope utilisé et utilisez les fibres optiques si nécessaire. - À l’aide du réglage de fluorescence , acquérez des images des cellules transplantées avec le même objectif et le même zoom que ceux utilisés pour les coupes (voir étape 9.2.1). Utilisez les paramètres suivants pour l’acquisition : gain 1, exposition 200-500 ms, décalage -10.
- À l’aide du réglage fond clair , acquérez des images des tranches (objectif 1 x avec zoom 3x utilisé ici).
- Imagerie en direct après dosage sous tension/mort à l’aide du microscope confocal
- Acquérez des images à l’aide d’un microscope confocal avec les paramètres d’acquisition suivants : objectif 20x, résolution 1 024 x 1 024 pixels avec un pas Z de 3 μm.
10. Analyse d’images par ImageJ
- Analyse de zone NFL, RBFOX3 et DAPI
- Ouvrez le logiciel ImageJ (https://imagej.net/software/imagej/).
- Ouvrez l’image du fichier en cliquant sur Fichier | Ouvrir | Sélectionner un fichier | Ouvrir.
- Dans la fenêtre contextuelle, sélectionnez Affichage de la pile | Hyperstack et mode couleur | Par défaut (avec mise à l’échelle automatique).
- Dans la barre d’outils, sélectionnez Image | Couleur | Divisez les chaînes.
- Choisissez les canaux souhaités à analyser : canal vert pour NFL (marqueur axonal), canal rouge pour RBFOX 3 (marqueur neuronal) et canal bleu pour DAPI (coloration nucléaire).
- Pour l’analyse NFL, procédez comme suit : dans la barre d’outils, sélectionnez Image | Ajuster | Seuil | Sélectionnez les paramètres (fond sombre, algorithme, par exemple, par défaut) et déplacez le curseur sur la barre de valeurs (sous/au-dessus) pour couvrir et circonscrire toute la zone des neurites (les neurites sont surlignées en blanc sur un fond sombre) | Ensemble | Postuler.
- Sélectionnez dans la barre d’outils l’outil de traçage Wand et utilisez-le pour définir automatiquement la zone blanche couverte par NFL. Presse Analyser | Mesurer | Valeur de surface en μm2.
- Pour l’analyse DAPI et RBFOX3, procédez comme suit : dans la barre d’outils, sélectionnez Image | Ajuster | Seuil | Sélectionnez les paramètres (fond blanc, algorithme, par exemple, par défaut) et déplacez le curseur sur la barre de valeurs (sous/au-dessus) pour couvrir et circonscrire toute la zone RBFOX3 ou DAPI | Ensemble | Postuler.
- Dans la barre d’outils, sélectionnez Traiter | FFT | Filtre passe-bande. Utilisez la barre de valeurs de seuil pour ajuster la zone blanche couverte par RBFOX3 ou DAPI, correspondant à leur signal de fluorescence.
- Dans la barre d’outils, sélectionnez l’outil de traçage Wand et utilisez-le pour définir automatiquement la zone couverte par RBFOX3 ou DAPI. Presse Analyser | Mesurer | Valeur de surface en μm2.
- Analyse de l’apoptose par ImageJ
- Ouvrez le logiciel ImageJ (https://imagej.net/software/imagej/).
- Ouvrez l’image du fichier Z-stack en cliquant sur Fichier | Ouvrir | Sélectionner un fichier | Ouvrir.
- Dans la fenêtre contextuelle, sélectionnez Affichage de la pile | Hyperstack et mode couleur | Par défaut (avec mise à l’échelle automatique).
- Dans la barre d’outils, sélectionnez Image | Couleur | Divisez les chaînes.
- Choisissez les canaux souhaités : canal rouge pour aCASP3 (marqueur d’apoptose à analyser) et bleu ou cyan pour DAPI ou Hu-Nu pour les noyaux. Ensuite, superposez les canaux en sélectionnant dans la barre d’outils Image | Couleur | Fusionner des canaux | Créer un composite.
- Faites glisser la barre Z en bas de l’image pour parcourir la pile Z de l’image et identifier les piles dans la région centrale des tranches avec une positivité aCASP3.
- Dans la barre d’outils, sélectionnez Plug-ins | Analyser | Compteur de cellules.
- Dans la fenêtre contextuelle ouverte, sélectionnez Initialiser pour préparer l’image pour le comptage ; ensuite, sélectionnez un type de compteur (par exemple, Type 1) et renommez-le en tant qu’objet à compter (par exemple, cellules aCASP3+ ). Renommez d’autres types de compteurs comme décrit ci-dessus pour compter d’autres objets (par exemple, cellules DAPI+ ou Hu-Nu+ pour le nombre total de cellules).
- Dans la fenêtre contextuelle, sélectionnez le type de compteur correspondant à l’objet à compter (par exemple, cellules aCASP3+ ), puis sélectionnez l’outil Point dans la barre d’outils et commencez à compter manuellement le nombre de cellules apoptotiques, positives pour aCASP3, en cliquant sur chaque cellule positive dans l’image ouverte.
- Sélectionnez un autre type de compteur dans la fenêtre du compteur de cellules et commencez à compter le nombre total de cellules (cellules DAPI+ , pour les tranches ; cellules Hu-Nu+ , pour les cellules transplantées).
- Analyse du dosage vivant/mort par ImageJ
- Ouvrez le logiciel ImageJ (https://imagej.net/software/imagej/).
- Ouvrez l’image du fichier Z-stack en cliquant sur Fichier | Ouvrir | sélectionner un fichier | ouvrir.
- Dans la fenêtre contextuelle, sélectionnez Affichage de la pile | Hyperstack et mode couleur | Par défaut (avec mise à l’échelle automatique).
- Dans les barres d’outils, sélectionnez Image | Couleur | Divisez les chaînes.
- Choisissez les canaux souhaités : canal vert pour Calcein (marqueur de vitalité à analyser) et canal cyan pour Sytox (marqueur mort). Ensuite, superposez les canaux en sélectionnant dans la barre d’outils Image | Couleur | Fusionner des canaux | Sélectionnez Créer un composite.
- Faites glisser la barre Z en bas de l’image pour parcourir la pile Z de l’image et identifier les piles dans la région centrale des tranches avec une positivité Calcein et Sytox.
- Dans la barre d’outils, sélectionnez Plug-ins | Analyser | Compteur de cellules.
- Dans la fenêtre contextuelle ouverte, sélectionnez Initialiser pour préparer l’image pour le comptage ; puis sélectionnez un type de compteur (par exemple, Type 1) et renommez-le en tant qu’objet à compter (par exemple, cellules Calcein+ ). Renommez les autres types de compteurs comme décrit ci-dessus s’il est nécessaire de compter un autre objet (par exemple, les cellules Sytox+ ).
- Dans la fenêtre contextuelle, sélectionnez le type de compteur correspondant à l’objet à compter ; puis sélectionnez l’outil Point dans la barre d’outils et commencez à compter manuellement le nombre de cellules Calcein+ , en cliquant sur chaque élément positif de l’image ouverte.
- Sélectionnez un autre type de compteur dans la fenêtre du compteur de cellules et comptez les cellules Sytox+ comme décrit pour Calcein.
11. Graphiques et analyses statistiques
- Effectuez toutes les analyses statistiques et tracez des graphiques à l’aide du logiciel de votre choix.
Résultats
Les méthodes décrites permettent l’établissement de coupes organotypiques SC à partir de souris au stade P3 et leur maintien en culture pendant une période prolongée dans des conditions saines. De plus, nous montrons un protocole pour la transplantation de cellules dans les tranches et pour leur co-culture jusqu’à 30 jours (Figure 1). Dans un premier temps, nous montrons l’optimisation des conditions de culture et un protocole adapté à la culture prolongée des tranches SC avec des cellules transplantées (Figure 2A). Les tranches sont générées et gérées à partir de DEV 0 jusqu’à DEV 2 dans le MO, qui a été proposé à l’origine comme un support optimal pour la maintenance des tranches SC47. Cependant, en raison de la présence de protéines sériques, ce milieu pourrait être sous-optimal pour soutenir la différenciation neuronale et la maturation des cellules précurseurs neurales transplantées. En effet, à DEV 3, nous avons testé le passage de l’OM au GM, une formulation contenant du Neurobasal plus B27, qui soutient la survie neuronale, et sans sérum, qui inhibe la différenciation neuronale correcte, favorisant à la place un destin glial48,49.
La figure 2B montre les résultats obtenus en changeant le milieu au DEV 3 de OM à GM, par rapport aux tranches SC ne recevant pas le commutateur (les tranches de contrôle ont été cultivées en MO). Nous avons utilisé la distribution du signal NFL à l’intérieur des tranches comme marqueur de l’intégrité neuronale (Figure 2B,C). Les coupes de DEV 7 étaient saines dans les deux conditions de culture, montrant la distribution diffuse de neurofilaments (NFL, en vert) à l’intérieur. À DEV 10, les tranches cultivées en GM semblaient être plus saines par rapport aux tranches témoins cultivées en OM, comme documenté par la distribution de coloration NFL. Nous avons également estimé la surface NFL+ (% NFL+ Area/DAPI+ Area) des tranches montrées dans les images représentatives de la figure 2B. La zone NFL+ estimée est représentée dans les histogrammes de la figure 2C, confirmant que le signal NFL est distribué de manière diffuse dans les tranches à DEV 7 dans les deux conditions. Cependant, à DEV 10, la zone estimée couverte par la coloration NFL diminue pour la condition de culture OM.
Ces données suggèrent que le passage à la MG à DEV 3 est bien toléré pour la culture prolongée de tranches SC (DEV 10). Comme étape suivante, nous avons testé GM à des moments plus prolongés : DEV 30, DEV 60 et DEV 90. Comme le montrent les figures 3A et B, les tranches ont été maintenues saines en culture jusqu’à DEV 90. La coloration NFL a été trouvée largement présente dans les tranches à chaque point temporel, avec une germination diffuse autour des tranches de neurites partant de la région centrale. En effet, nous avons estimé la surface NFL+ des tranches montrées à la figure 3A et elle a augmenté au fil du temps comme le montrent les histogrammes de la figure 3B. Nous avons également observé une positivité au marqueur neuronal RBFOX3, fournissant une autre preuve de la différenciation neuronale des tranches. À chaque point temporel, nous avons également vérifié le taux d’apoptose en évaluant dans différentes tranches le nombre de cellules positives à aCASP3 (Figure 4A,B). L’analyse a été effectuée conformément à la section 10.2 du protocole. Le taux d’apoptose (% de cellules aCASP3+/nombre total de cellules DAPI+) s’est avéré très faible à chaque point temporel (0,85 ± 0,52 %, 0,71 ± 0,27 %, 0,66 ± 0,45 % pour DEV 30, 60 et 90, respectivement), sans différences significatives entre les trois points temporels considérés (valeur p > 0,05, figure 4B). Ces données suggèrent que le taux d’apoptose associé à aCASP3 reste stable au cours du temps et, avec la large distribution de NFL dans les coupes (Figure 4A), confirment la survie des tranches à chaque point temporel.
À l’appui des données précédentes, nous avons également effectué un test vivant/mort pour évaluer la viabilité des coupes aux trois points temporels différents. Nous avons utilisé Calcein (coloration verte) pour marquer les cellules viables et métaboliquement actives et Sytox (coloration cyan) pour évaluer la mort cellulaire. Comme le montrent les histogrammes de la figure 4C, le pourcentage de cellules métaboliquement actives augmente légèrement de DEV 30 à DEV 90 (93,17 ± 5,21 %, 96,43 ± 3,02 %, 96,33 ± 3,10 % pour DEV 30, 60 et 90, respectivement), se stabilisant entre les deux derniers points temporels (DEV 30 vs DEV 60 valeur p = 0,018 ; DEV 30 vs DEV 90 : valeur p = 0,027 ; DEV 60 vs DEV 90 valeur p = 0,99). Nous avons constaté de faibles niveaux de mort cellulaire qui ont diminué au fil du temps (6,83 ± 5,21 %, 3,57 ± 3,02 %, 3,66 ± 3,10 % pour DEV 30, 60 et 90, respectivement) et une différence significative a été observée entre DEV 30 et les points temporels ultérieurs, DEV 60 et DEV 90 (DEV 30 vs DEV 60 valeur p = 0,018 ; DEV 30 vs DEV 90 : valeur p = 0,027 ; DEV 60 vs DEV 90 valeur p = 0,99). Ces données, associées au taux d’apoptose, confirment la survie des tranches au fil du temps et soutiennent l’efficacité du protocole de culture à long terme effectué.
Une fois que la faisabilité d’une culture prolongée des tranches SC a été établie, nous avons défié le système par la transplantation de cellules h-SC-NES aux premiers stades de la différenciation neuronale. Nous avons testé les cellules h-SC-NES car elles ont montré des résultats prometteurs pour le traitement des lésions médullaires12. La procédure de transplantation de cellules h-SC-NES dans des tranches SC de souris est décrite dans la section 6 du protocole. Les coupes SC et les cellules h-SC-NES transplantées ont été maintenues jusqu’à DPT 30. Les cellules ont été greffées à la DIV 10 de différenciation (stade précurseur neural) en tranches organotypiques DEV 4, comme le montre le schéma de protocole de la figure 5A. L’expression de la GFP en culture a permis de surveiller l’expression de la GFP dans les cellules transplantées pendant une période allant jusqu’à 30 jours. La figure 5B montre des images en direct représentatives, à différents DPT, d’une tranche SC avec des cellules GFP+ transplantées. L’expression stable de la GFP au fil du temps (figures 5B et 6A) suggère que les cellules ont survécu dans le tissu SC dans les conditions de culture précédemment optimisées. Nous avons également vérifié le taux d’apoptose des cellules transplantées comme décrit dans la section 10.2 du protocole. Le taux d’apoptose (% de cellules aCASP3+ /nombre total de cellules Hu-Nu+ ) s’est avéré très faible (0,44 ± 0,34 %) après 30 DPT (Figure 6B). De plus, le taux d’apoptose à DPT 30 s’est avéré être conforme à celui trouvé pour le même type de cellules à DPT 7, comme précédemment rapporté40, documentant que les cultures se stabilisent au fil du temps.
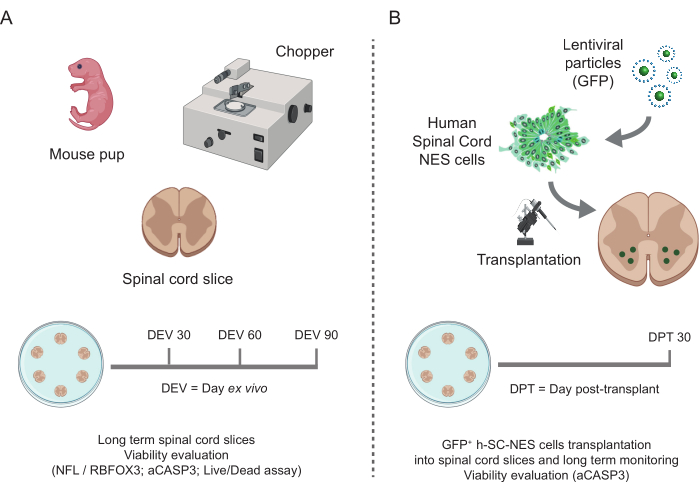
Figure 1 : Flux de travail du protocole. Schéma représentatif montrant le flux de travail général du protocole effectué. (A) À gauche, un schéma résumant la génération de tranches SC de souris à partir de SC isolés de petits de souris à P3 et la culture à long terme de tranches organotypiques SC. (B) À droite, un schéma résumant la transplantation de cellules h-SC-NES exprimant la GFP dans des coupes organotypiques de SC de souris. Les cellules greffées sont conservées pendant 30 jours après la greffe. Abréviations : h-SC-NES = tige neuroépithéliale dérivée de la moelle épinière humaine ; GFP = protéine fluorescente verte ; DEV = jour ex vivo ; DTC = jour après la greffe ; NFL = chaîne légère de neurofilaments ; RBFOX3 = ARN liant l’homologue 3 du fox-1 ; aCASP3 = caspase-3 active ; SC = moelle épinière. Veuillez cliquer ici pour voir une version agrandie de cette figure.
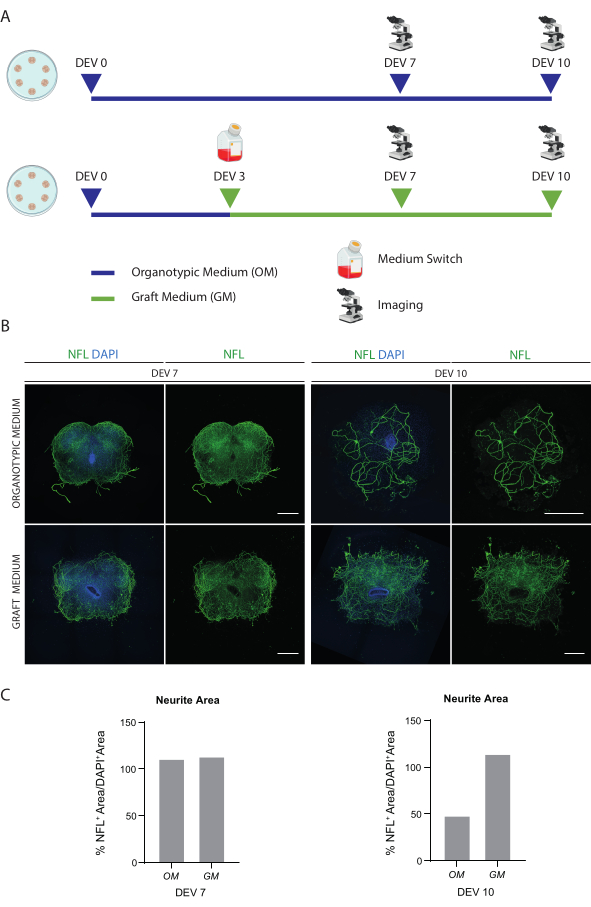
Figure 2 : Optimisation des conditions d’élevage à long terme. (A) Schéma représentatif du protocole d’essai OM et GM. OM est maintenu jusqu’au DEV 7-10 pour le groupe témoin. Le milieu est basculé sur le GM à DEV 3 pour les tranches traitées ; ensuite, ils sont fixés à DEV 7-10 pour comparaison avec les commandes. (B) Images représentatives comparant des coupes organotypiques de souris SC à DEV 7 et 10 cultivées dans des conditions différentes. Les tranches sont colorées pour le neurofilament marqueur du cytosquelette (NFL, vert). La large distribution de la coloration NFL dans les tranches cultivées avec des OGM suggère une survie et une différenciation globales. Les noyaux sont contre-colorés avec du DAPI. Barre d’échelle = 500 μm. (C) Histogrammes représentatifs de l’estimation de la zone couverte par NFL dans les tranches illustrées à la figure 1B. À DEV 10, la surface de NFL diminue dans les conditions de culture de la MO. Abréviations : DEV = jour ex vivo ; DAPI = 4',6-diamidino-2-phénylindole ; NFL = chaîne légère de neurofilaments. ; OM = milieu organotypique ; GM = milieu de greffe ; SC = moelle épinière. Veuillez cliquer ici pour voir une version agrandie de cette figure.
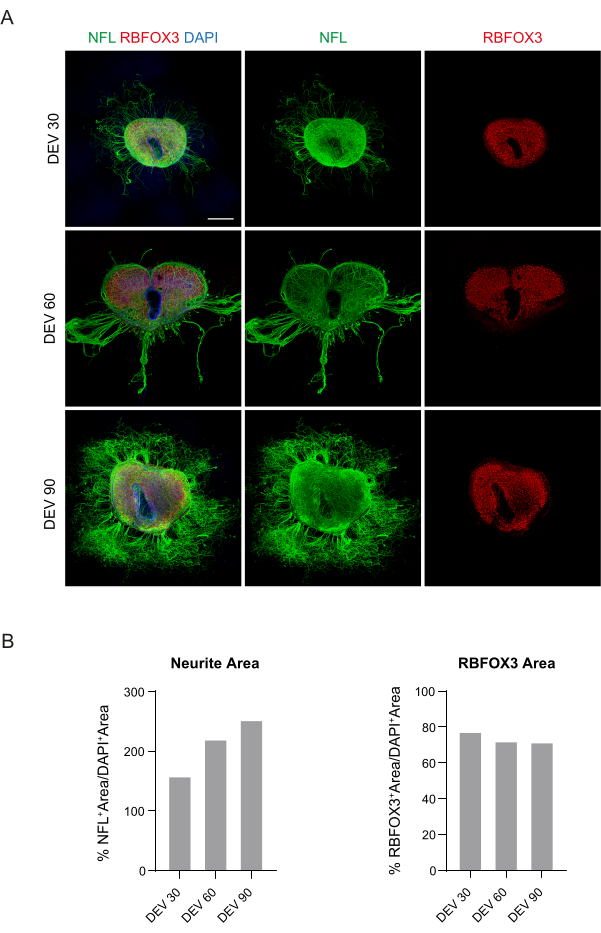
Figure 3 : Coupes organotypiques de souris cultivées à long terme. (A) Les tranches sont maintenues en culture jusqu’à DEV 90. Le test d’immunofluorescence révèle une large distribution du neurofilament marqueur cytosquelettique (NFL, vert) et du marqueur neuronal nucléaire RBFOX3 (rouge), attestant de leur état de santé et de leur identité neuronale après une culture à long terme. Il est à noter que les axones NFL+ poussent de manière diffuse autour des tranches au fil du temps. Les noyaux sont contre-colorés avec du DAPI. Barre d’échelle = 500 μm. (B) Histogrammes représentatifs de l’estimation de la zone et du temps NFL+ et de l’aire RBFOX3+ des coupes montrées dans le panneau A. La surface neurite NFL+ augmente avec le temps. Abréviations : DEV = jour ex vivo ; DAPI = 4',6-diamidino-2-phénylindole ; NFL = chaîne légère de neurofilaments ; SC = moelle épinière ; RBFOX3 = Homologue 3 de l’ARN reliant l’ARN-1. Veuillez cliquer ici pour voir une version agrandie de cette figure.
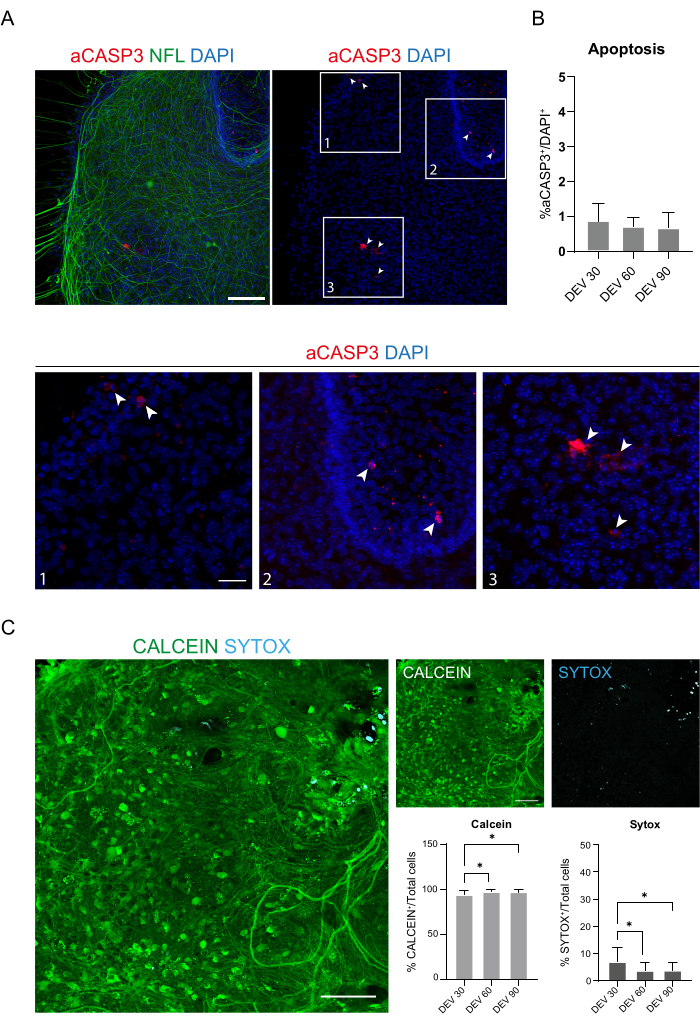
Figure 4 : Évaluation de la viabilité cellulaire dans les coupes SC au cours du temps. (A) Images représentatives de coupes organotypiques à DEV 60 colorées pour aCASP3 (rouge) et NFL (vert). Barre d’échelle = 100 μm. NFL montre un motif diffus. Des cellules rares sont positives pour le marqueur apoptotique aCASP3 (Inserts : 1-2-3). (B) Analyse du taux d’apoptose en tranches à différents points temporels. Moyenne ± écart-type, N (répétitions) = 6 tranches, n (cellules totales) > 1 000 pour chaque tranche, test de Kruskal-Wallis, comparaison multiple, valeur p > 0,05. Le taux d’apologie est stable dans le temps. Dans les encarts 1-2-3 du panneau A, il est possible d’observer le détail des cellules positives pour aCASP3 (coloration rouge, flèches blanches). De petits points rouges marquent les débris cellulaires et les noyaux pycnotiques. Barre d’échelle = 50 μm. (C) Images représentatives du test vivant/mort réalisé sur des coupes SC à DEV 90 : les cellules métaboliquement actives sont marquées en vert avec Calcein, tandis que les cellules mortes et endommagées sont marquées en bleu clair (cyan) avec Sytox. Les deux histogrammes montrent le % de cellules positives pour Calcein (à gauche) et Sytox (à droite) sur le nombre total de cellules. Pour les deux moyennes ± écarts-types, N (répétitions) = 6 tranches, n (cellules totales) > 1 000 pour chaque tranche, test de Kruskal-Wallis, comparaison multiple, valeur p DEV 30 vs DEV 60 = 0,018 ; DEV 30 vs DEV 90 : valeur p = 0,027 ; DEV 60 vs DEV 90 : la valeur p > 0,99. Abréviations : DEV = jour ex vivo ; DAPI = 4',6-diamidino-2-phénylindole ; NFL = chaîne légère de neurofilaments ; SC = moelle épinière ; aCASP3 = caspase-3 active. Veuillez cliquer ici pour voir une version agrandie de cette figure.
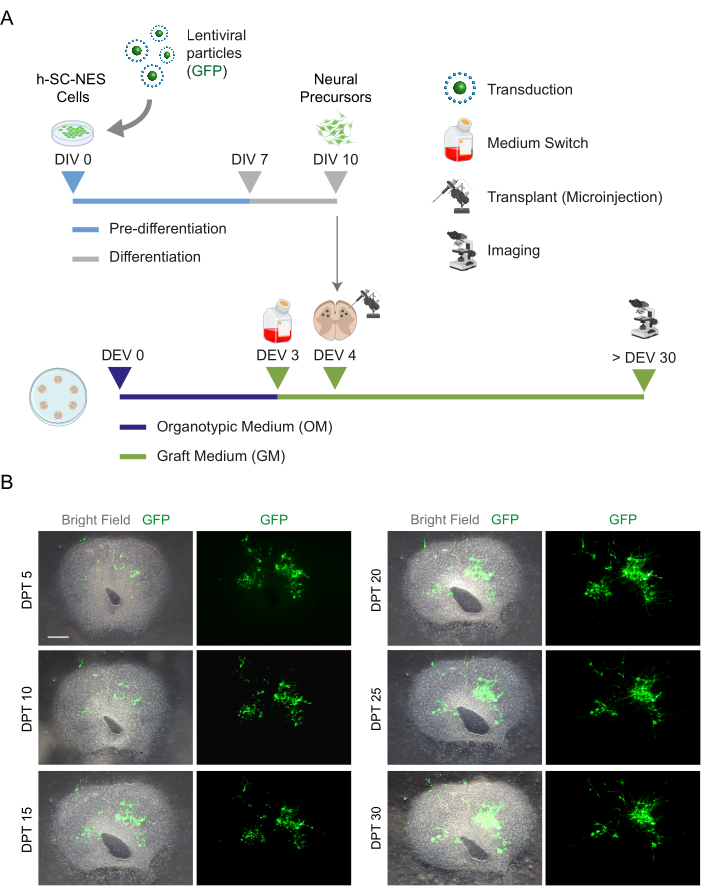
Figure 5 : Transplantation de cellules h-SC-NES dans des coupes organotypiques de souris. (A) Schéma représentatif du protocole de transplantation. Les cellules sont transplantées en tant que précurseurs neuraux à la DIV 10 de la différenciation en tranches organotypiques DEV 4. (B) Images représentatives de coupes organotypiques de souris transplantées avec des cellules h-SC-NES exprimant la GFP au fil du temps jusqu’à DPT 30. Les cellules sont transduites à l’aide d’un vecteur lentiviral porteur du gène GFP. L’expression de la GFP au fil du temps confirme leur viabilité et leur adaptation à l’environnement de la tranche. Barre d’échelle = 500 μm. Abréviations : DIV = premier jour de pré-différenciation ; h-SC-NES = tige neuroépithéliale dérivée de la moelle épinière humaine ; GFP = protéine fluorescente verte ; DEV = jour ex vivo ; OM = milieu organotypique ; GM = milieu de greffe ; DTC = jours après la greffe. Veuillez cliquer ici pour voir une version agrandie de cette figure.
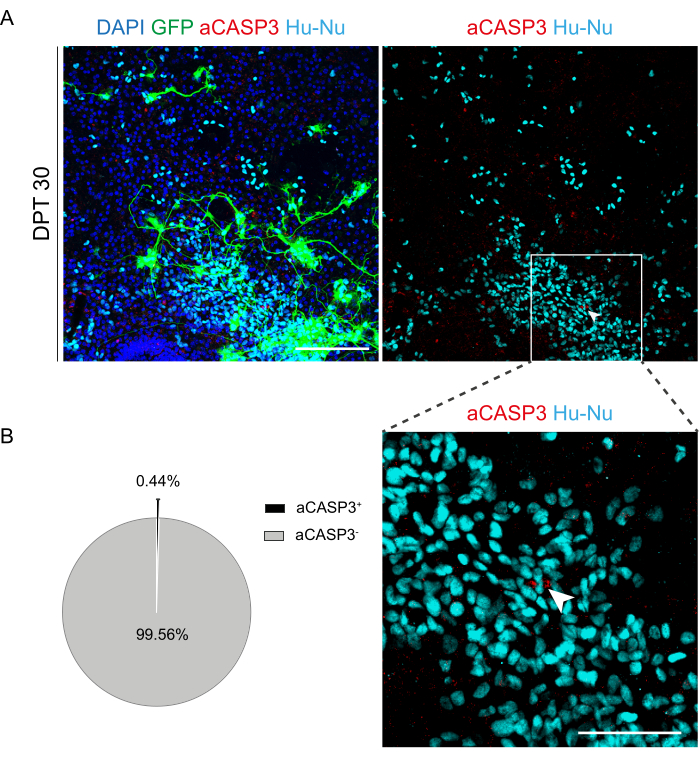
Figure 6 : Évaluation du taux d’apoptose des cellules h-SC-NES transplantées 30 jours après la greffe. (A) Image représentative d’une tranche organotypique de souris transplantée avec des cellules h-SC-NES exprimant la GFP. Les cellules sont transduites à l’aide d’un vecteur lentiviral porteur du gène GFP pour les surveiller dans les tranches après la transplantation. L’expression de la GFP au fil du temps confirme leur viabilité et leur adaptation à l’environnement de la tranche. Le point temporel indiqué est DPT 30 ; Les cellules sont colorées pour les noyaux humains (cyan) et aCASP3 (rouge). Barre d’échelle = 150 μm. (B) À gauche, graphique circulaire représentatif de l’analyse de l’apoptose des cellules transplantées en coupes à DPT 30 (N (répétitions) = 5 tranches, n (cellules) = 5 000), et à droite, un encart de cellules Hu-Nu+ et le détail d’une cellule positive à aCASP3 (flèche blanche). Barre d’échelle = 75 μm. De petits points rouges marquent les débris cellulaires et les noyaux pycnotiques. Abréviations : h-SC-NES = tige neuroépithéliale dérivée de la moelle épinière humaine ; GFP = protéine fluorescente verte ; DTC = jour après la greffe ; DAPI = 4',6-diamidino-2-phénylindole ; NFL = chaîne légère de neurofilaments ; aCASP3 = caspase-3 active ; Hu-Nu = noyaux humains. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 1 : Composition des solutions utilisées dans ce protocole. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
Il n’existe toujours pas de traitement efficace pour les patients atteints de LME. Différentes approches ont été testées et l’une des plus prometteuses est basée sur une stratégie régénérative de remplacement cellulaire. À l’heure actuelle, les progrès dans le domaine de la médecine régénérative nécessitent de nouvelles plateformes pour tester l’efficacité et l’innocuité des greffes de cellules, seules ou en combinaison avec d’autres approches. Leur validation préclinique est essentielle pour poursuivre les études cliniques. Les cultures organotypiques SC constituent une plate-forme utile pour étudier différents aspects de la neurodégénérescence, de la régénération neuronale et du neurodéveloppement, et pour étudier l’efficacité de nouvelles approches thérapeutiques23. En particulier, des caractéristiques spécifiques des cultures organotypiques telles que le maintien de l’histoarchitecture originale et de la composition cellulaire et microenvironnementale sont avantageuses pour démêler la dynamique de la transplantation, telle que la greffe, l’intégration, la différenciation et la maturation cellulaires.
Conformément aux protocoles publiés, les coupes organotypiques SC peuvent être maintenues en culture pendant environ 2 à 3 semaines dans des conditions saines, ce qui limite leur utilisation pour les investigations à long terme et le dépistage fonctionnel requis pour tester les schémas de thérapie cellulaire. L’exploration de processus importants tels que la différenciation et la maturation vers le destin correct des cellules transplantées à l’intérieur du tissu SC nécessite une surveillance à long terme. Ces processus cellulaires sont essentiels lors des greffes courantes dans les modèles animaux. La disponibilité d’un système ex vivo qui imite de nombreuses caractéristiques présentes in vivo serait utile dans la phase de dépistage préclinique.
Pour cette raison, dans ce travail, nous proposons une méthode de culture organotypique SC optimale à long terme (≥30 jours) qui permet de maintenir des tranches SC viables jusqu’à 90 jours, triplant ainsi leur délai de culture habituel. De plus, nous montrons une greffe stable de cellules h-SC-NES à l’intérieur des tranches SC et le maintien de la culture de transplantation jusqu’à 30 jours. Nous avons suivi l’enracinement cellulaire au fil du temps en observant l’expression de la GFP pour vérifier la survie cellulaire jusqu’à DPT 30. Après 30 DPT, nous avons évalué le taux d’apoptose cellulaire. Dans la littérature, l’évaluation de l’apoptose des cellules h-SC-NES transplantées dans des coupes SC à 7 DPT a été rapportée40. Ici, nous avons étendu l’analyse de l’apoptose cellulaire à DPT 30 pour comparer le taux d’apoptose par rapport au point temporel précédent (DPT 7). Nous avons découvert que nos données sont conformes à la littérature, ce qui suggère que les cellules h-SC-NES transplantées survivent également à un moment ultérieur si elles sont maintenues dans les conditions de culture optimisées dans notre travail. Cette plateforme ex vivo améliorée à long terme, seule et dans la configuration de la transplantation, aidera les chercheurs dans le dépistage préclinique des greffes de cellules souches pour les lésions médullaires. Cela leur permettra d’identifier la meilleure cellule candidate pour d’autres études in vivo favorisant le succès des greffes. De plus, après un criblage initial, les coupes organotypiques SC pourraient également être utilisées en parallèle des études in vivo pour confirmer et corroborer la dynamique cellulaire à long terme et les comportements observés dans les modèles animaux ou pour soutenir les études mécanistes.
Notre protocole décrit en détail comment générer ce modèle organotypique à long terme, mais certaines étapes critiques doivent également être discutées. En ce qui concerne la génération des cultures organotypiques SC, il y a quelques défis lors de la chirurgie et des premières étapes de la culture. Une intervention chirurgicale bien réalisée est essentielle pour générer des coupes qui maintiennent l’histoarchitecture d’origine. Si le SC est ruiné pendant l’isolement, les tranches peuvent perdre leur structure anatomique typique et des lésions tissulaires peuvent induire une agression pro-inflammatoire excessive entraînant des conditions malsaines et la mort cellulaire. La phase la plus difficile de la chirurgie est l’extraction du SC de la colonne vertébrale et l’ablation des méninges du SC isolé. La réussite de ces étapes dépend de l’expérience de l’opérateur ; Par conséquent, une période de formation avant de commencer les expériences est recommandée.
La section coronale du SC à travers un hacheur est également une phase difficile. Le SC isolé doit être placé sur le plateau de coupe exactement perpendiculairement à la lame. L’opérateur doit également placer la lame perpendiculairement au plateau de coupe. Ces précautions sont nécessaires pour assurer la génération de coupes reproductibles entre les mêmes expériences et les différentes expériences. Un autre point important est que le temps de l’opération est limité : l’ensemble de la procédure de génération de tranches doit prendre ~30 min. Si l’opérateur passe plus de temps sur la chirurgie et la coupe, le tissu SC en souffrira, ce qui peut nuire au succès de la culture et aux prochaines étapes de l’expérience.
Une fois les tranches placées sur la membrane de culture, il est important de les nourrir correctement. Le GDNF est nécessaire pour soutenir la récupération et la survie des tissus. La coupe avec un hachoir est traumatisante pour le tissu et, pour cette raison, les tranches sont placées peu de temps après la coupe dans un milieu de dissection glacé pour nettoyer l’excès de molécules pro-inflammatoires et favorisant la mort. Ensuite, les tranches sont placées sur les membranes de culture (inserts de culture cellulaire) avec un milieu frais modifié avec du GDNF pour favoriser une récupération plus rapide et une adhésion de la tranche à la membrane. Le GDNF doit être ajouté au milieu tous les jours pendant la première semaine de culture en raison de sa courte demi-vie50,51. Nous avons observé que les tranches ont besoin de la présence continue de GDNF pendant les premiers jours de culture pour favoriser la récupération et la viabilité des tissus. Dans tous les cas, comme la présence de GDNF est importante pendant toute la période de culture, il est fortement déconseillé d’interrompre l’administration de GDNF à d’autres moments.
Au cours de la première semaine de culture, il est également important de vérifier les coupes macroscopiquement à l’œil nu et au microscope. Le tissu translucide et la transparence des bords sont des signes de bonne adhérence des tranches à la membrane et de tissu viable. Le tissu nécrotique apparaîtra extrêmement blanc à la première vue macroscopique et les zones nécrotiques apparaîtront gris foncé au microscope. Après quelques semaines de culture, la morphologie des tissus peut changer : les mouvements cellulaires et l’adhésion des tissus à la membrane peuvent influencer ce processus. Nous avons observé, par exemple, la perte de la lumière centrale dans certaines tranches remplies de cellules et la perte de la morphologie de la corne dorsale et ventrale. Cela se produit principalement avec des tranches plus petites, alors que la plupart d’entre elles conserveront une structure anatomique proche de celle d’origine. Les coupes sont généralement générées à partir des régions lombaires ou thoraciques, car de cette façon, elles peuvent avoir la taille appropriée pour maintenir leur histoarchitecture d’origine dans le temps : si elles sont trop petites, elles perdent leur architecture tandis que, si elles sont trop grandes, la région centrale peut subir une nécrose. Ainsi, nous avons utilisé la région lombaire des petits de souris pour générer des tranches de la taille appropriée pour une culture optimale à long terme, mais, en principe, d’autres segments peuvent être envisagés. De plus, nous avons choisi d’utiliser la région lombaire, car les régions ventrale et dorsale sont plus distinctes l’une de l’autre. De plus, cette région présente des zones tissulaires avec un pourcentage plus élevé de motoneurones et de matière grise, qui sont des sites d’intérêt pour les thérapies de remplacement cellulaire dans les LME. En ce qui concerne la transplantation de cellules dans les tranches, le principal problème est lié à la rupture de la pointe de la micro-aiguille de verre. Si le trou pour le passage des cellules est trop grand, il peut endommager le tissu SC lors de la microinjection. S’il est trop petit, l’empilement de cellules peut obstruer l’aiguille, entravant le processus de transplantation. La procédure de transplantation doit être terminée dans un délai de 1 h pour minimiser la souffrance et la mort des cellules.
Le protocole proposé fournit un outil optimal et polyvalent pour différents types d’enquêtes. Ici, nous appliquons notre plateforme à long terme pour valider la transplantation de cellules h-SC-NES aux premiers stades de la différenciation à l’intérieur du tissu SC de souris pendant 30 jours. La principale nouveauté de l’approche proposée est l’optimisation du protocole de co-culture. Les composants de GM maintiennent la survie neuronale à long terme des tranches SC et des cellules h-SC-NES transplantées. En effet, la GM, étant un milieu sans sérum, soutient la différenciation des cellules transplantées vers le destin neuronal par rapport au milieu précédemment utilisé pour la culture en tranches organotypiques47.
En ce qui concerne les modèles proposés pour les lésions médullaires, les expériences sont généralement réalisées sur des souris adultes. Jusqu’à présent, les différences les plus importantes entre les cellules souches néonatales et adultes sont liées au potentiel de régénération plus élevé observé chez les souris néonatales par rapport aux souris adultes52. Cependant, ces différences n’ont pas d’impact sur le type de protocole que nous proposons, car nous nous concentrons ici sur la réponse des cellules greffées à l’environnement du tissu hôte plutôt qu’aux capacités de régénération des neurones résidents. Une autre différence entre les souris néonatales et adultes après une lésion médullaire est liée à la formation de la cicatrice gliale qui se produit chez les adultes. Cet aspect n’est pas pris en compte dans le modèle proposé, qui ne prend pas en compte les processus physiopathologiques complexes résultant de lésions primaires et secondaires.
En ce qui concerne les applications, la plateforme pourrait également être utilisée pour étudier l’intégration entre les cellules transplantées et les circuits résidents présents dans le modèle organotypique SC. Des outils de génie génétique étaient déjà utilisés dans le SNC pour évaluer la connectivité synaptique et pouvaient être exploités à cet égard 53,54,55. En particulier, l’intégration pourrait être étudiée et validée en évaluant la formation de synapses entre les cellules greffées et le tissu SC ex vivo. Ces cultures organotypiques à long terme pourraient également être exploitées pour tester des agents neuroprotecteurs et neurorégénératifs ou de nouvelles molécules/matériaux ou pour étudier des troubles neurodégénératifs impliquant le CS. Pour étudier des troubles neurodégénératifs spécifiques, le protocole doit être adapté à la culture de coupes SC générées à partir de modèles pertinents, tels que des souris transgéniques porteuses de mutations spécifiques associées à une pathologie, au stade pertinent de la pathologie (c’est-à-dire néonatale, juvénile, adulte). En conclusion, notre protocole et les cultures organotypiques en général, étant des explants d’un organe spécifique, présentent des caractéristiques qui comblent le fossé entre les cultures cellulaires 2D et les modèles in vivo, les confirmant comme un outil inestimable pour la recherche fondamentale et les essais précliniques.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à déclarer.
Remerciements
L’étude a été soutenue par la Fondation Wings for Life (WFL-IT-20/21), le plan national pour la reprise et la résilience (NRRP) de l’Union européenne Next-Generation EU-mission 4 composante 2, l’investissement n° 1.4-CUP N. B83C22003930001 (Tuscany Health Ecosystem-THE, Spoke 8) et Marina Romoli Onlus. Ce manuscrit ne reflète que les points de vue et opinions des auteurs, ni l’Union européenne ni la Commission européenne ne peuvent en être tenus responsables. Les données et les métadonnées sont disponibles sur Zenodo 10.5281/zenodo.10433147. Les images ont été générées avec Biorender https://www.biorender.com/.
matériels
| Name | Company | Catalog Number | Comments |
| anti-cleaved Caspase-3, (Asp175) (5A1E) (Rabbit) | Cell Signaling Technology | 9661S | 1:400 |
| anti-GFP (Mouse) - monoclonal | Sigma/Merck | G6539 | 1:400 |
| anti-Human Nuclei (Mouse) - monoclonal, clone 235-1 | Sigma/Merck | MAB1281 | 1:400 |
| anti-Human Nuclei (Rabbit) | NeoBiotechnologies | RBM5-346-P1 | 1:400 |
| anti-NeuN (RBFOX3) (Rabbit) - polyclonal | Sigma/Merck | ABN78 | 1:400 |
| anti-NFL (Mouse) | Sigma/Merck | MAB1615 | 1:400 |
| anti-NFL H-Phospho (Rabbit) -polyclonal | Biologend | 840801 | 1:500 |
| Aqua Polymount | Poly-sciences | 18606-20 | |
| B-27 | Gibco | 17504-044 | |
| BDNF | Gibco | PHC7074 | |
| Blades | Leica | 118364227 | |
| Cell culture graded water | Sigma/Merck | W3500-500ML | |
| Collagen from rat tail | Sigma/Merck | C7661 | |
| Confocal microscope - A1 Confocal Microscope (Eclipse Ti) | Nikon | ||
| D(+)-Glucose | Sigma/Merck | G7021 | |
| Dissecting Forceps | World Precision Instruments | 15915 | |
| DMEM/F12 | Gibco | 31330 | |
| DPBS | Sigma/Merck | D8537 | |
| EGF | Sigma/Merck | gf144 | |
| FBS | Gibco | 10270-106 | |
| FGF-2 | Stemgent | 03-0002 | |
| GDNF | Sigma/Merck | SRP3200 | |
| Glass capillaries, 3.5" | Drummond Scientific Company | 3-000-203-G/X | |
| Glutamax | Gibco | 35050-038 | |
| Goat-anti Mouse IgG Alexa Fluor 488 | Thermo Fisher Scientific | A11029 | |
| Goat-anti Mouse IgG Alexa Fluor 647 | Thermo Fisher Scientific | A21236 | 1:500 |
| Goat-anti Rabbit IgG Alexa Fluor 568 | Thermo Fisher Scientific | A11011 | 1:500 |
| Goat-anti Rabbit IgG Alexa Fluor 647 | Thermo Fisher Scientific | A21244 | 1:500 |
| Graph Pad-Prism | Dotmatics | Software for Statistical Analysis | |
| HBSS | Gibco | 14025-050 | 1:500 |
| HEPES | Gibco | 15630-056 | |
| Hoechst 33342 | Thermo Fisher Scientific | H3570 | |
| Horse Serum | Gibco | 16050-122 | |
| Insulin | Sigma/Merck | I9278 | |
| Laminin | Sigma/Merck | L2020 | |
| Lentiviral prep | Addgene | 17446-LV | |
| L-Glutamine | Thermo Fisher Scientific | 25030024 | |
| LIVE/DEAD Viability/Cytotoxicity assay kit | Thermo Fisher Scientific | L32250 | |
| McIlwain Tissue Chopper | World Precision Instruments | ||
| MEM | Gibco | 11090-081 | |
| Microloader tips | Eppendorf | 5242956003 | to load cells in the needle for transplantation |
| Microscope slides | VWR | 631-0909 | |
| Millicell cell culture membrane | Sigma/Merck | PICM0RG50 | |
| Miscroscope cover glasses | VWR | ECN 631-1572 | |
| N-2 | Gibco | 17502-048 | |
| Neurobasal | Gibco | 21103-049 | |
| Penicillin/Streptomycin | Thermo Fisher Scientific | 15140122 | |
| Petri dish (35mm) | VWR | 734-2317 | |
| PFA | Sigma/Merck | P6148-500G | |
| Plastic pasteur pipette | Sarstedt | 86.1171.010 | |
| Pneumatic PicoPump | World Precision Instruments | PV830 | Microinjector for transplantation |
| Poly-L-lysine | Sigma/Merck | P4707 | |
| Scalpel blade No 10 Sterile Stainless Steel | VWR International | SWAN3001 | |
| Scalpel handle #3 | World Precision Instruments | 500236 | |
| Spring Scissors | World Precision Instruments | 501235 | |
| Stereomicroscope for imaging and acquisition | Nikon | SMZ18 | |
| Stereomicroscope for surgery | VWR | ||
| Triton X-100 | Merck | T8787 | |
| Tweezers-Dumont #5-inox | World Precision Instruments | 501985 | |
| Vannas Scissors, 8.5 cm | World Precision Instruments | 500086 | |
| Vertical micropipette puller | Shutter Instrument | P-30 | |
| Y-27632 | R&D Systems | 1254/50 |
Références
- Ding, W., et al. Spinal cord injury: The global incidence, prevalence, and disability from the Global Burden of Disease Study 2019. Spine. 47 (21), Phila Pa. 1532-1540 (2022).
- Yang, B., et al. Strategies and prospects of effective neural circuits reconstruction after spinal cord injury. Cell Death Dis. 11 (6), 439(2020).
- Liu, K., et al. PTEN deletion enhances the regenerative ability of adult corticospinal neurons. Nat Neurosci. 13 (9), 1075-1081 (2010).
- Anderson, M. A., et al. Required growth facilitators propel axon regeneration across complete spinal cord injury. Nature. 561 (7723), 396-400 (2018).
- de Freria, C. M., Van Niekerk, E., Blesch, A., Lu, P. Neural stem cells: promoting axonal regeneration and spinal cord connectivity. Cells. 10 (12), 3296(2021).
- Badner, A., Siddiqui, A. M., Fehlings, M. G. Spinal cord injuries: how could cell therapy help. Expert Opin Biol Ther. 17 (5), 529-541 (2017).
- Assinck, P., Duncan, G. J., Hilton, B. J., Plemel, J. R., Tetzlaff, W. Cell transplantation therapy for spinal cord injury. Nat Neurosci. 20 (5), 637-647 (2017).
- Ishii, K., et al. Neutralization of ciliary neurotrophic factor reduces astrocyte production from transplanted neural stem cells and promotes regeneration of corticospinal tract fibers in spinal cord injury. J Neurosci Res. 84 (8), 1669-1681 (2006).
- Zhang, Y. W., Denham, J., Thies, R. S. Oligodendrocyte progenitor cells derived from human embryonic stem cells express neurotrophic factors. Stem Cells Dev. 15 (6), 943-952 (2006).
- Faulkner, J., Keirstead, H. S. Human embryonic stem cell-derived oligodendrocyte progenitors for the treatment of spinal cord injury. Transpl Immunol. 15 (2), 131-142 (2005).
- Kadoya, K., et al. Spinal cord reconstitution with homologous neural grafts enables robust corticospinal regeneration. Nat Med. 22 (5), 479-487 (2016).
- Dell' Anno, M. T., et al. Human neuroepithelial stem cell regional specificity enables spinal cord repair through a relay circuit. Nat Commun. 9 (1), 3419(2018).
- Wu, S., FitzGerald, K. T., Giordano, J. On the viability and potential value of stem cells for repair and treatment of central neurotrauma: overview and speculations. Front Neurol. 9, 602(2018).
- Nardone, R., et al. Rodent, large animal and non-human primate models of spinal cord injury. Zoology. 123, 101-114 (2017).
- Hartung, T. Thoughts on limitations of animal models. Parkinsonism Relat Disord. 14, SUPPL.2 (2008).
- Shanks, N., Greek, R., Greek, J. Are animal models predictive for humans. Philosophy, Ethics, and Humanities in Medicine. 4 (1), 2(2009).
- Dawson, T. M., Golde, T. E., Lagier-Tourenne, C. Animal models of neurodegenerative diseases. Nat Neurosci. 21 (10), 1370-1379 (2018).
- Hayden, P. J., Harbell, J. W. Special review series on 3D organotypic culture models: Introduction and historical perspective. In Vitro Cell Dev Biol Anim. 57 (2), 95(2021).
- Jensen, C., Teng, Y. Is it time to start transitioning from 2D to 3D cell culture. Front Mol Biosci. 7, 33(2020).
- Mirbagheri, M., et al. Advanced cell culture platforms: a growing quest for emulating natural tissues. Materials Horizons. 6 (1), 45-71 (2019).
- Gähwiler, B. H. Organotypic monolayer cultures of nervous tissue. J Neurosci Methods. 4 (4), 329-342 (1981).
- Stoppini, L., Buchs, P. -A., Muller, D. A simple method for organotypic cultures of nervous tissue. J Neurosci Methods. 37 (2), 173-182 (1991).
- Pandamooz, S., Nabiuni, M., Miyan, J., Ahmadiani, A., Dargahi, L. Organotypic spinal cord culture: a proper platform for the functional screening. Mol Neurobiol. 53 (7), 4659-4674 (2016).
- Fuller, L., Dailey, M. E. Preparation of rodent hippocampal slice cultures. CSH Protoc. 2007, (2007).
- Gertz, C. C., Lui, J. H., LaMonica, B. E., Wang, X., Kriegstein, A. R. Diverse behaviors of outer radial glia in developing ferret and human cortex. J Neurosci. 34 (7), 2559-2570 (2014).
- Ballerini, L., Galante, M. Network bursting by organotypic spinal slice cultures in the presence of bicuculline and/or strychnine is developmentally regulated. Eur J Neurosci. 10 (9), 2871-2879 (1998).
- Avossa, D., Rosato-Siri, M. D., Mazzarol, F., Ballerini, L. Spinal circuits formation: a study of developmentally regulated markers in organotypic cultures of embryonic mouse spinal cord. Neuroscience. 122 (2), 391-405 (2003).
- Lossi, L., Merighi, A. The use of ex vivo rodent platforms in neuroscience translational research with attention to the 3RS philosophy. Front Vet Sci. 5, 164(2018).
- Nogueira, G. O., Garcez, P. P., Bardy, C., Cunningham, M. O., Sebollela, A. Modeling the human brain with ex vivo slices and in vitro organoids for translational neuroscience. Front Neurosci. 16, 838594(2022).
- Qi, X. R., et al. Human brain slice culture: a useful tool to study brain disorders and potential therapeutic compounds. Neurosci Bull. 35 (2), 244(2019).
- Park, H. W., et al. Human mesenchymal stem cell-derived Schwann cell-like cells exhibit neurotrophic effects, via distinct growth factor production, in a model of spinal cord injury. Glia. 58 (9), 1118-1132 (2010).
- Charrière, K., Risold, P. Y., Fellmann, D. In vitro interactions between bone marrow stromal cells and hippocampal slice cultures. C R Biol. 333 (8), 582-590 (2010).
- Jeong, D. K., Taghavi, C. E., Song, K. J., Lee, K. B., Kang, H. W. Organotypic human spinal cord slice culture as an alternative to direct transplantation of human bone marrow precursor cells for treating spinal cord injury. World Neurosurg. 75 (3-4), 533-539 (2011).
- Riggio, C., et al. Generation of magnetized olfactory ensheathing cells for regenerative studies in the central and peripheral nervous tissue. Int J Mol Sci. 14 (6), 10852-10868 (2013).
- Kamei, N., et al. Neural progenitor cells promote corticospinal axon growth in organotypic co-cultures. Neuroreport. 15 (17), 2579-2583 (2004).
- Kamei, N., et al. NGF released from transplanted neural progenitor cells promote corticospinal axon growth in organotypic cocultures. Spine. 32 (12), Phila Pa. 1272-1278 (2007).
- Hamasaki, T., et al. Magnetically labeled neural progenitor cells, which are localized by magnetic force, promote axon growth in organotypic cocultures. Spine. 32 (21), Phila Pa. 2300-2305 (2007).
- Kim, H. M., Lee, H. J., Lee, M. Y., Kim, S. U., Kim, B. G. Organotypic spinal cord slice culture to study neural stem/progenitor cell microenvironment in the injured spinal cord. Exp Neurobiol. 19 (2), 106-113 (2010).
- Liu, X., Chu, T. H., Su, H., Guo, A., Wu, W. Neural progenitor cell apoptosis and differentiation were affected by activated microglia in spinal cord slice culture. Neurol Sci. 35 (3), 415-419 (2014).
- De Vincentiis, S., et al. Low forces push the maturation of neural precursors into neurons. Small. 19 (30), 2205871(2023).
- Abouelfetouh, A., Kondoh, T., Ehara, K., Kohmura, E. Morphological differentiation of bone marrow stromal cells into neuron-like cells after co-culture with hippocampal slice. Brain Res. 1029 (1), 114-119 (2004).
- Sypecka, J., Koniusz, S., Kawalec, M., Sarnowska, A. The organotypic longitudinal spinal cord slice culture for stem cell study. Stem Cells Int. 2015, 471216(2015).
- Tanvig, M., et al. A brain slice culture model for studies of endogenous and exogenous precursor cell migration in the rostral migratory stream. Brain Res. 1295, 1-12 (2009).
- Tennstaedt, A., et al. Human neural stem cell intracerebral grafts show spontaneous early neuronal differentiation after several weeks. Biomaterials. 44, 143-154 (2015).
- Vogel, S., et al. The in vivo timeline of differentiation of engrafted human neural progenitor cells. Stem Cell Res. 37, 101429(2019).
- Onorati, M., et al. Zika virus disrupts phospho-TBK1 localization and mitosis in human neuroepithelial stem cells and radial glia. Cell Rep. 16 (10), 2576-2592 (2016).
- Vyas, A., et al. An in vitro model of adult mammalian nerve repair. Exp Neurol. 223 (1), 112-118 (2010).
- Brewer, G. J., Torricelli, J. R., Evege, E. K., Price, P. J. Optimized survival of hippocampal neurons in B27-supplemented neurobasal, a new serum-free medium combination. J Neurosci Res. 35 (5), 567-576 (1993).
- De Vries, G. H., Boullerne, A. I. Glial cell lines: an overview. Neurochem Res. 35 (12), 1978-2000 (2010).
- Ziv-Polat, O., et al. The role of neurotrophic factors conjugated to iron oxide nanoparticles in peripheral nerve regeneration: in vitro studies. Biomed Res Int. 2014, 267808(2014).
- Mesa-Infante, V., Afonso-Oramas, D., Salas-Hernández, J., Rodríguez-Núñez, J., Barroso-Chinea, P. Long-term exposure to GDNF induces dephosphorylation of Ret, AKT, and ERK1/2, and is ineffective at protecting midbrain dopaminergic neurons in cellular models of Parkinson's disease. Mol Cell Neurosci. 118, 103684(2022).
- Montero, A. M., Huang, A. H. The regenerative capacity of neonatal tissues. Development. 149 (12), (2022).
- Feng, L., Kwon, O., Lee, B., Oh, W. C., Kim, J. Using mammalian GFP reconstitution across synaptic partners (mGRASP) to map synaptic connectivity in the mouse brain. Nat Protoc. 9 (10), 2425-2437 (2014).
- Il Choi, D., Kaang, B. -K. Interrogating structural plasticity among synaptic engrams. Curr Opin Neurobiol. 75, 102552(2022).
- Choi, J. -H., et al. Interregional synaptic maps among engram cells underlie memory formation. Science. 360 (6387), 430-435 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon