A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
טיהור של מ magneticum מסננים השגריר-1 Magnetosome חלבון Associated MamAΔ41
In This Article
Summary
אמא הוא חלבון ייחודי הקשור Magnetosome אשר הוצגה להיות מעורב ההפעלה magnetosome. כאן אנו מציגים את הפרוטוקול טיהור של אמא המחיקה מוטציה (MamAΔ41) מ מ magneticum השגריר-1.
Abstract
חיידקים Magnetotactic מהווים קבוצה מגוונת של מיקרואורגניזמים ימיים המסוגלים להתמצא עצמם לאורך שדות geomagnetic. התנהגות זו הוא האמין סיוע בחיפוש אחר בסביבה מתאימה
Protocol
1. שיבוט וביטוי של אמא ג'ין ב E. coli
הגן המוטנטי mamAΔ41 היה מוגבר באמצעות שרשרת התגובה פולימראז (PCR) של הדנ"א הגנומי של Magnetospirillum magneticum השגריר-1, עם primers: 5'-3-GCATTACGCATATGGACGACATCCGCCAGGTG 'ו 5'-GCGCGGCAGCCATA-TGGCATACG-3 ". בשנת שברי דנ"א מוגבר, אתר NcoI הוצג בבית קודון התחלת ATG ואת קודון סיום הוחלף באתר ScoI. שברי היו מתעכל עם NcoI ו SacI ו משובטים לתוך האתרים השונים של pET52b (+), והוליד pET52bMamAΔ41-AMB1. ב לבנות זו, הגן mamAΔ41 היה התמזגו בתוך מסגרת עם תג-10 שלו בתחנה הסופית-C. הפלסמיד היה electroporated לתוך זן החיידק BL21. החיידק זן BL21 מחסה pET52bMamAΔ41 היה גדל אינדוקציה אוטומטי (15) בינוני המכיל אמפיצילין (50 מ"ג / מ"ל) 310 מעלות קלווין במשך 3 שעות. הטמפרטורה טיפוח הועבר אז מ ° 310-300 ° K ומתוחזק עבור 48 שעות נוספות על 300 ° K. התאים נקצרו על ידי צנטריפוגה ב 5465g במשך 10 דקות ב 277 קלווין 8 ליטר תרבות הפיק 60 גרם של התא גלולה רטוב.
2. ביואינפורמטיקה חישובים
חישוב משקל מולקולרי (MW), על פי רצף חומצות אמינו הקליטה חזה של 1mg/1ml החלבון 280nm, באמצעות שרת ProtParam (http://www.expasy.ch/tools/protparam.html). עבור 10His-Tag-MamAΔ41 MW הוא 22,529 Da (240 חומצות אמינו), ועל MamAΔ41 (עם 9 חומצות אמינו שמאל לאחר ההסרה שלו, תג על ידי טרומבין) Mw הוא 20596.5Da (187 חומצות אמיניות). הקליטה חזה של 1mg/1ml החלבון 280nm עבור 10His-Tag-MamAΔ41 הוא 0.595 עבור MamAΔ41 הוא 0.579. בנוסף, את רצף החומצות האמיניות אינו מכיל שאריות ציסטאין. לכן, סוכני צמצום אינם נדרשים במהלך תהליך טיהור.
3. טיהור MamAΔ41
- להכנת פתרון המניות, לשקול את ולהכין (200 מ"ל כל אחד) בריכוזים הבאים: Imidazole (MW 228.2 g / mol) פתרון 4M, NaCl (Mw 58.44 g / mol) פתרון 5M, טריס-HCl (Mw 121.3 g / mol ) פתרון 1M, להתאים את ה-pH = 8. הוסף מים מזוקקים כפול (DDW) ומערבבים בעזרת מערבולת עד הפתרון הוא ברור. פתרונות סינון עם מסנן 0.22μm לשמור בטמפרטורת החדר.
- הכן ולסנן 6 מאגרים טריים פתרונות מניות (טבלה 1).
- מדולל ו להשעות תאים חיץ נפח יחס 01:02, גרם תאים למאגר (מ"ל).
- הוסף 10μl עבור כל 10gr של חיידקים של DNase אני (1 מ"ג / מ"ל) כדי לשבור את שברי DNA במדגם. הוסף 1ml עבור כל 20gr של חיידקים של קוקטייל EDTA חינם מעכב פרוטאז. דגירה של 20 דקות על הקרח.
- שיבשו תאים על ידי שני מחזורים של העיתונות הצרפתית 25,000 psi. בניגוד sonication, בעיתונות הצרפתית נועדה בנפח גבוה (10 מ"ל>) שחול התא ידי אילוץ התאים בלחץ גבוה דרך פתח צר. הבוכנה צריך להיות מקורר על קרח למשך עשר דקות לפני השימוש בו והרכבה עקב הצטברות חום.
- העברת התאים lysed לתוך צינורות ultracentrifuge 25ml ולאזן אותם בדיוק (50 מ"ג) עם חיץ מדגם מגיע עד קו הצוואר של הצינור.
- תא פסולת נפרדת על ידי צנטריפוגה Ultra ב 270.000 גרם במשך שעה 1 ב 277 קלווין
- הכן הכבידה תוצרת בית Ni נ.ת. ע עמודה (2.5cm קוטר) על ידי הוספת 4 מ"ל של ניקל נ.ת. ע שרף, אשר שומרת באתנול 20%, על הטור. תנו טיפה נוזל לחלוטין, ולאחר מכן לשטוף עם 40 מ"ל של DDW ושמטה את נוזל לחלוטין. טרום equilibrated שרף בטור עם 40 מ"ל של חיץ א
- החלת חלק מן מסיסים צנטריפוגה אולטרה צינורות על עמודה הכבידה Ni נ.ת. ע. תנו טיפה נוזל, לאסוף flow-through (חלבונים מאוגד) ולשמור על 277K.
- איסוף דגימת גלולה מן צנטריפוגה אולטרה צינורות באמצעות טיפ קטן לבטל את גלולה. מערבבים את הטיפ של 5% 50μl β-mercaptoethanol ב 2X SDS-PAGE חיץ המדגם.
- לשטוף את העמודה עם 100 מ"ל חוצץ ב תנו טיפה נוזל, זרימה דרך לאסוף ולשמר ב 277K.
- הכן 15 מבחנות מסומנות Eppendorf (1.5ml).
- Elution - שסתום טור קרוב ולאחר מכן לטעון 1ml של חיץ C על הטור, דגירה 3 דקות לאחר מכן על ידי פתיחת השסתום זרימה דרך האוסף. חזור על שלב זה שלוש פעמים.
- שאר Elute ההשעיה ב 1 מ"ל במרווחים חיץ C ולסגור את הברז.
- מדידת OD ב 280nm של שברים ולהעריך את מיקומו של השיא חלבון, אם OD עדיין> 0.3, מדגמים נוספים נדרשים.
- SDS-PAGE ניתוח של שברים שנאספו. ניתוח כזה יש לבצע גם עם דגימות הבאים, חלבונים מאוגד זרימה דרך, לשטוף זרימה דרך ultra-צנטריפוגה מדגם גלולה. מערבבים 10μl ממדגם כל אחד עם 5% 10μl של β-mercaptoethanol ב 2X SDS-PAGE חיץ המדגם. טען את דגימות על 15% SDS-PAGE ולרוץ 45 דקות ב 180 וולט.
- הכתם ג'ל 15 מ"ל של תמיסת InstantBlue, ללחוץ במשך שעה 1. ודא כי תיבת פלסטיק מכוסה ומוגן מפני אור.
- הערכת ג'ל, אם ביטוי חלבון גבוה וספציפי על פי להקות מעיד, מיזוג שברים eluted שנבחר.
- על מנת להסיר 10-התג שלו, להוסיף שור תרומבין (10 יחידות / μl), 10μl תרומבין חלבון לכל 1 מ"ג, על פי המלצת היצרן המדגם המשולב.
- דיאליזה משמש להסרת Imidazole עודף לירידה בריכוז NaCl רמות צורך במהלך מחייב חילוף יונים. עבור דיאליזה, לחתוך לאורך הרצוי של 7 משקל מולקולרי KD מנותקים (MWCO) צינורות דיאליזה. לשטוף עם DDW ולעשות קשר בקצה אחד (או לאטום עם קליפים). העברת פתרון חלבון הממוזגת על התיק תוך השארת קצת מקום בראש לצבוט למעלה.
- לצלול דיאליזה צינורות במאגר ליטר 4 דיאליזה מצונן (Buffer D). מערבבים את הפתרון באיטיות על לילה 277 קלווין
- קח את צינורות דיאליזה מתוך כוס, לעשות חתך קטן ולהעביר את החלבון שפופרת 50 מ"ל.
- יון מטבע כרומטוגרפיה: להרכיב מראש ארז MonoQ טור 4.6/100 PE על כרומטוגרפיה נוזלית מהיר ביצועים (FPLC). לשטוף את העמודה עם 5 כרכים עמודה (CV) של מים מסוננים DDW. טור Equilibrated עם קורות חיים של 5 חיץ ד
- לשטוף את הלולאה עם זריקה DDW (שני כרכים לולאה) וחזור עם חיץ ד טען את החלבון הלולאה ההזרקה.
- להזריק את החלבון מדגם (קצב זרימה של 1 מ"ל / דקה) ולהתחיל באיסוף השברים לגילוי SDS-PAGE של חלבונים בלתי מוגבל.
- אם נפח חלבון ראשוני (שלב 3.23) הוא גדול יותר מאשר לולאה הזרקה, חזור על שלבים 3.25-3.26, מבלי לשטוף את הלולאה, עד המדגם כולו נטען על עמודה.
- לשטוף את העמודה עם 3 קורות חיים של חיץ C על מנת להסיר חלבונים גבולות.
- Elute את החלבון עם שיפוע של 60 דקות ליניארי של 40 2000mM NaCl בין מאגרים D & E (כאשר E חיץ 100% הסוף של שיפוע), קצב הזרימה 1 דקות מ"ל / (למרות ספיקה גבוהה יותר ניתן להשתמש העלות של כרכים גדולים יותר, אך פחות מרוכז, חלבון פסגות). איסוף פסגות חלבונים כפי שהם מופיעים chromatogram 280nm.
- טור נקי ארוז מראש על פי המלצה ייצור.
- SDS-PAGE ניתוח של שברים שנאספו. ניתוח כזה יש לבצע עם דגימות הבאים, חלבונים גבולות זרימה דרך וחלבון פסגות זרימה דרך. הפעל את SDS-PAGE ו כתם כמו להזכיר בצעדים 3.17-3.18.
- הערכת ג'ל לקיום חלבון להקות ב Mw החזוי לפי הסמן חלבון מראש מוכתם. מיזוג שברים elution נבחרים המכילים את החלבונים הרלוונטיים.
- Dialyze דגימות הממוזג, על מנת להפחית ריכוזי NaCl הדרושים כרומטוגרפיה הדרה גודל. ביצוע דיאליזה כאמור צעדים 3.21-3.23 חוצץ נגד פ
- תתרכז המדגם חלבון באמצעות Vivaspin-15 10000 MWCO עד 8 מ"ג / מ"ל בצנטריפוגה שולחן, 4000 סל"ד. אמת ריכוז רצון ידי מדידת OD ב 280nm ב קובט קוורץ. אם חלבון מרוכז מדי מדללים אותו עם F המאגר ולמדוד שוב.
- להרכיב הדרה גודל ארוז מראש הטור, HiLoad 26/60 Superdex 200, על FPLC; לשטוף את העמודה עם קורות חיים של 3 מים מסוננים DDW. טור Equilibrated עם קורות חיים של 3 חיץ פ
- לשטוף עם לולאה הזרקת DDW 5 מ"ל (שני כרכים לולאה) ואחריו נפח זהה עם חיץ פ טען מ"ל לא יותר מ 4 המדגם חלבון בלולאה את הזריקה.
- להזריק את החלבון מדגם (קצב הזרימה של 3.5 mL / min) ולהתחיל באיסוף חלבונים שברים שיא כפי שהם מופיעים זה chromatogram 280nm. תן את זרימת חיץ עד שהוא מגיע בנפח העמודה.
- אם נפח חלבון ראשוני (שלב 3.23) הוא גדול יותר מאשר לולאה הזרקה, חזור על שלבים 3.25-3.26, מבלי לשטוף את הלולאה, עד המדגם כולו נטען על עמודה.
- טור נקי ארוז מראש על פי המלצת ייצור.
- SDS-PAGE ניתוח של שברים שנאספו. ניתוח כזה יש לבצע עם דגימות פסגות חלבון. הפעל את SDS-PAGE ו כתם כמו להזכיר בצעדים 3.17-3.18.
- הערכת ג'ל: אם פסגות חלבון ספציפי, Mw ניבא נכונה לפי הסמן חלבון מראש מוכתם ורק אחד הלהקה גלוי המיזוג הנבחר שברים eluted של החלבון הרלוונטי.
- תתרכז מדגם חלבון באמצעות-15 Vivaspin 10,000 MWCO אל> סל"ד 20 מ"ג / מ"ל בצנטריפוגה שולחן 4000,. אמת ריכוז רצון ידי מדידת OD ב 280nm. MamAΔ41 מטוהרים התרכזה אז 26.5 מ"ג / מ"ל של התגבשות.
- אשר טוהר מדגם זיהוי חלבון באמצעות מטריקס בסיוע לייזר desorption / יינון (MALDI-TOF).
- אם חלבון מטוהרים פי ספקטרומטר מסה SDS-PAGE, לחלק את MamAΔ41 מרוכז מראש המסומנים צינורות Eppendorf: 25-50μl חלבון כל אחד.
- פלאש קפואים בחנקן נוזלי חנות ב 193 קלווין
4. נציג תוצאות
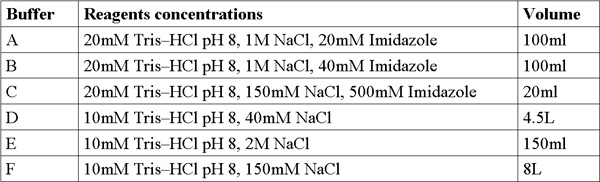
כאשר פרוטוקול זה הוא נעשה בצורה נכונה אחד צריך לקבל מטוהרים, הומוגנית ומרוכזת גודל דגימות חלבון. אלה הן דוגמאות חלבון אז מוכן ניסויים התגבשות כמו גם מחקרים ביוכימיים, כגון קינטיקה האנזים, זיקה מחייבת ועוד. המתוארות כאן הן תוצאות נציג שלבים עיקריים בפרוטוקול טיהור. SDS-PAGE ניתוח של פרופיל elution הטור זיקה Ni-נ.ת. ע צריך לחשוף מאוד על חלבון מתבטא השבר מסיס ב MW המתאים ~ 22kDa (איור 1). זה ניתוח SDS-PAGE צריך גם לחשוף את המינימום אם בכלל חלבון החלבונים גבולות זרימה דרך, לשטוף זרימה דרך אולטרה צנטריפוגה מדגם גלולה. אם להקות גדולות של חלבון המתאים מופיעות דוגמאות אלה יש לשקול הכנה חיץ רע, בעיות הפרעה התא או בעיות אוטומטי אינדוקציה התנאים תרבות וצמיחה.
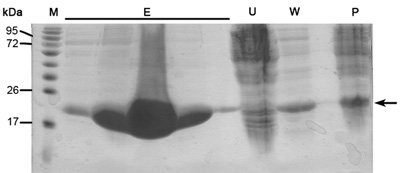
חילוף יונים כרומטוגרפיה הוא preformed על מנת להפריד בין חלבון רצוי שלנו חלבונים אחרים E.coli שהיו מאוגדים שרף ניקל (עקב אינטראקציות אלקטרוסטטיות או היסטידין / שלילי חומצות אמינו בחלבון עשיר לולאות) וחלבון נימול Tagged שלו רצוי. טור זה מפריד חלבונים לפי זיקה מחייבת שלהם שרף מטען חשמלי חיובי תחת NaCl בריכוז הולך וגובר. חלבון מאוד טעונים שלילית יהיה elute NaCl בריכוז גבוה appose למתן אלה טעונים שלילית. היתרונות של טור זה קצב זרימה גבוה ויכולת מחייב. Chromatogram חילוף יונים (איור 2) מגלה הפרדה טובה בין האוכלוסיות 3 חלבונים בריכוז NaCl גוברת. SDS-PAGE ניתוח נחוץ על מנת לקבוע מגוואט כל האוכלוסייה, לבודד את הערכה רצוי והאם השלבים לטיהור נוספים נדרשים. האוכלוסייה הראשונה היא MamAΔ41 (~ 20 KDA) ואילו האוכלוסייה השנייה היא MamAΔ41 + תג שלו (~ 22kDa) כי לא היה ביקע ידי תרומבין שור לבין האוכלוסייה השלישית היא לגילוי ב-SDS PAGE בשל ריכוז נמוך. אם הפסגות חלבונים לא מופרדים בבירור אחד צריך לשקול הכנה חיץ טועה או שינוי שיפוע שיפוע NaCl.
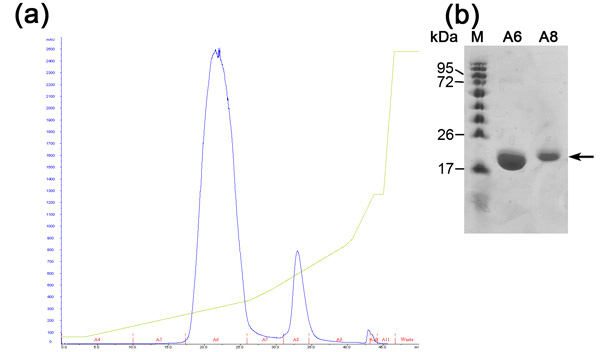
הדרה גודל כרומטוגרפיה הוא preformed על מנת להפריד בין חלבון רצוי שלנו חלבונים אחרים E.coli התא שהיו מאוגדים שרף Ni-נ.ת. ע ולא הופרדו במהלך חילוף יונים כרומטוגרפיה. עמודה זו מפרידה חלבונים על פי גודלם. חלבונים גדולים יהיה elute בכמויות קטנות elution לעומת הקטנים אשר יהיה eluted בנפחים גדולים יותר. Chromatogram עמודה (איור 3) מגלה הפרדה טובה של חלבון רצוי האוכלוסייה העיקרית. האוכלוסייה נראה elute בגודל המתאים מונומר עם MW של ~ 20 kDa. הנוכחות של הלהקה 80 ~ kDa (חלבון E.coli טיפוסי שקושר Ni-נ.ת. ע שרף) הוא זוהה-SDS PAGE לפני טעינת העמודה נעלם במהלך הריצה הזאת. מאז הריכוז של חלבון זה 80 ~ kDa נמוך מלכתחילה, אוכלוסייתה אינה מופיעה chromatogram בשל דילול שלה SDS-PAGE ניתוח על מנת לקבוע את יעילות הטיהור. אנו ממליצים טעינת מדגם בדילול מלא ומרוכז של שיא הראשי דף SDS כך אפשר בהחלט לקבוע נוכחות של חלבון טהור MW המתאים. בשלב הזה החלבון הוא מטוהרים ההומוגניות שלה צריך להיות מוערך על ידי MALDI-TOF. ניתוח זה חשף החלבון של 20,347 מגה וואט דה ואחד Mw של 10,176 Da (איור 4). ~ 10 חלבון kDa הוא ~ 20 חלבון kDa הוכפל טעונה. MW החזוי של MamAΔ41 עם 9 חומצות אמינו שמאל לאחר ההסרה שלו-Tag היה 20596.5 דא. ידי השוואתה המתקבל MALDI-TOF MW מצאנו כי הם 248 דה בנפרד. שוני זה יכול לנבוע MALDI-TOF שגיאות מדידה ו / או בשל השפלה המשותף של מתיונין הראשון, גליצין השני. לסיכום, החלבון הוא מטוהרים והוא יכול לשמש עבור ניסויים נוספים.
טבלה 1. ניסוחים חוצץ.
באיור 1. נציג SDS-PAGE ניתוח של טיהור Ni-נ.ת. ע עמודה מימין לשמאל:. P-אולטרה צנטריפוגה גלולה (3.11 צעד פרוטוקול), W-לשטוף (3.12 צעד פרוטוקול), U-גבולות חלבונים (3.10 צעד פרוטוקול), E- חמשת המסלולים של elution samples (3.14 צעד פרוטוקול), M - סמן חלבון (המספרים מציינים MW). חץ מציין MamAΔ41.
איור 2. ניתוח של צעד יון כרומטוגרפיה החליפין (א) יון החליפין (טור MonoQ) chromatogram; כחול - 280nm הקליטה, מייצג ריכוז חלבון, Green-NaCl בריכוז, אדום - מעיד על שברים שנאספו. (ב) נציג SDS-PAGE ניתוח של הצעד טיהור חילוף יונים. משמאל לימין: M - סמן חלבון (המספרים מציינים MW), A6 - שבר שיא הראשון A8 השני שבר שיא.

איור 3. הדרה גודל כרומטוגרפיה ניתוח (א) הדרה גודל (Superdex 200 טור) chromatogram; כחול - 280nm הקליטה, מייצג ריכוז חלבון, סימן אדום של שברים שנאספו. (ב) נציג SDS-PAGE ניתוח צעד הדרה גודל טיהור. משמאל לימין: M-חלבון סמן (המספרים מציינים MW), PreI-מראש מוזרק מדגם, A4-6 חלק בשילוב שנאספו השיא הראשון, A4-6d-מדולל חלק שנאספו השיא.
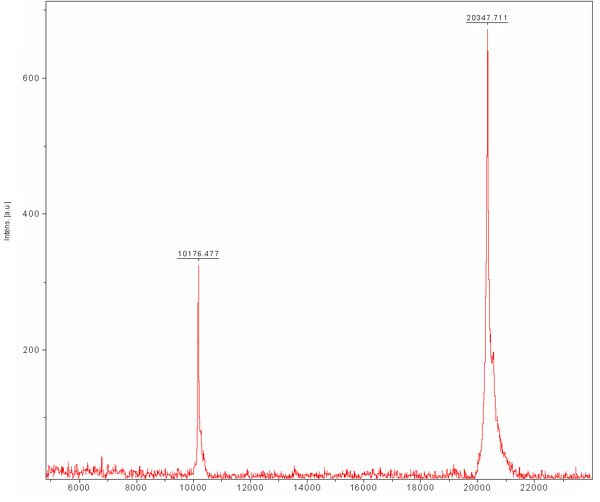
איור 4. מטריקס בסיוע לייזר desorption / יינון (MALDI-TOF) ספקטרום המונית של מטוהרים MamAΔ41. מטריצה היא חומצה Sinapic (SA). MamAΔ41 הראו על 20,347 דא, הראה גם הוא הכפיל של מינים טעונים MamAΔ41 ב 10,176 Da.
Discussion
טיהור חלבון היא הצעד העיקרי כל החלבונים או הביוכימיות מחקרים מבניים. מאז החלבון הוא ייחודי עם ההתנהגות שלו, צריך להגדיר את המאפיינים שלו ולשנות טיהור שלה בהתאם. היעד חלבון יש לנתח כצעד ראשון לקראת טיהור שימוש בכלים לביואינפורמטיקה. הם משמשים כדי לחשב את נקודת iso חשמל?...
Acknowledgements
אנו מכירים ד"ר אמיר אהרוני על תמיכתו וגאולה דוידוב, נועם גרימברג חן גוטמן עצה והערותיהם.
Materials
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| French Press | Equipment | Thermo scientific | FA-078A | |
| Pressure cell | Equipment | Thermo scientific | FA-032 | |
| Ultra-centrifuge | Equipment | Sorvall | Discovery 90SE | |
| Rottor | Equipment | Beckman | Ti60 | |
| Ultra-centrifuge tubes; PC-Bottle+Cap Assay 26.3ml | Equipment | Beckman | BC-355618 | |
| 2.5cm diameter, Glass Econo-Column Chromatography Columns | Equipment | BioRad | 737-2521 | |
| Ni-NTA His Bind resin | Equipment | Novagen | M0063428 | |
| Spectrophotometer | Equipment | Amersham Biosiences | Ultraspec 2100 pro | |
| Quartz cuvette | Equipment | Hellma | 104-QS | |
| Fast Performance Liquid Chromatography- AKTA purifier 10 | Equipment | GE Healthcare Biosciences | 28-4062-64 | |
| Ion exchange column – MonoQ 4.6/100 PE | Equipment | GE Healthcare Biosciences | 10025543 | |
| Size exclusion pre-packed column-HiLoad 26/60 Superdex 200 | Equipment | GE Healthcare Biosciences | 17-1071-01 | |
| Centricon - Vivaspin15 – 10,000 MWCO | Equipment | Sartorius Stedim Biotech GmbH | VS1501 | |
| Table centrifuge | Equipment | Thermo scientific | IEC CL30R | |
| MALDI-TOF | Equipment | Bruker Daltonics | Reflex IV | |
| Tris-HCl (hydrotymethyl) aminomethane | Reagent | BioLab | 20092391 | |
| Sodium Chloride | Reagent | FRUTROM | 235553470 | |
| Imidazole | Reagent | Alfa Aesar | 288-32-4 | |
| EDTA free protease inhibitors cocktail | Reagent | Sigma | P-8849 | |
| Dnase I (Deoxyribonuclease I) | Reagent | Sigma | DN-25 | |
| Bovine Thrombin | Reagent | Fisher BioReagents | BP25432 | |
| Glycine | Reagent | BioLab | 07132391 | |
| Soudim Dodecyl Sulfate (SDS) | Reagent | BioLab | 19822391 | |
| Beta-mercaptoethanol | Reagent | Sigma | M-3148 | |
| InstantBlue | Reagent | Expedeon | 1SB01L | |
| PageRuler Prestained Protein Ladder | Reagent | Fermentas | SM0671 |
References
- Faivre, D., Schuler, D. Magnetotactic Bacteria and Magnetosomes. Chem Rev. 108, 4875-4898 (2008).
- D'Andrea, L. D., Regan, L. TPR proteins: the versatile helix. Trends Biochem Sci. 28, 655-662 (2003).
- Young, J. C., Barral, J. M., Hartl, U. l. r. i. c. h., F, . More than folding: localized functions of cytosolic chaperones. Trends Biochem Sci. 28, 541-547 (2003).
- Brocard, C., Hartig, A. Peroxisome targeting signal 1: is it really a simple tripeptide?. Biochim Biophys Acta. 1763, 1565-1573 (2006).
- Fransen, M., Amery, L., Hartig, A., Brees, C., Rabijns, A., Mannaerts, G. P., Van Veldhoven, P. P. Comparison of the PTS1- and Rab8b-binding properties of Pex5p and Pex5Rp/TRIP8b. Biochim Biophys Acta. 1783, 864-873 (2008).
- Baker, M. J., Frazier, A. E., Gulbis, J. M., Ryan, M. T. Mitochondrial protein-import machinery: correlating structure with function. Trends Cell Biol. 17, 456-464 (2007).
- Mirus, O., Bionda, T., von Haeseler, A., Schleiff, E. Evolutionarily evolved discriminators in the 3-TPR domain of the Toc64 family involved in protein translocation at the outer membrane of chloroplasts and mitochondria. J Mol Model. 15, 971-982 (2009).
- Gatsos, X., Perry, A. J., Anwari, K., Dolezal, P., Wolynec, P. P., Likic, V. A., Purcell, A. W., Buchanan, S. K., Lithgow, T. Protein secretion and outer membrane assembly in Alphaproteobacteria. FEMS Microbiol Rev. 32, 995-1009 (2008).
- Tiwari, D., Singh, R. K., Goswami, K., Verma, S. K., Prakash, B., Nandicoori, V. K. Key residues in Mycobacterium tuberculosis protein kinase G play a role in regulating kinase activity and survival in the host. J Biol Chem. 284, 27467-27479 (2009).
- Edqvist, P. J., Broms, J. E., Betts, H. J., Forsberg, A., Pallen, M. J., Francis, M. S. Tetratricopeptide repeats in the type III secretion chaperone, LcrH: their role in substrate binding and secretion. Mol Microbiol. 59, 31-44 (2006).
- Grunberg, K., Muller, E. C., Otto, A., Reszka, R., Linder, D., Kube, M., Reinhardt, R., Schuler, D. Biochemical and proteomic analysis of the magnetosome membrane in Magnetospirillum gryphiswaldense. Appl Environ Microbiol. 70, 1040-1050 (2004).
- Komeili, A., Vali, H., Beveridge, T. J., Newman, D. K. Magnetosome vesicles are present before magnetite formation, and MamA is required for their activation. Proc Natl Acad Sci USA. 101, 3839-3843 (2004).
- Okuda, Y., Fukumori, Y. Expression and characterization of a magnetosome-associated protein, TPR-containing MAM22, in Escherichia coli. FEBS Lett. 491, 169-173 (2001).
- Taoka, A., Asada, R., Sasaki, H., Anzawa, K., Wu, L. F., Fukumori, Y. Spatial localizations of Mam22 and Mam12 in the magnetosomes of Magnetospirillum magnetotacticum. J Bacteriol. 188, 3805-3812 (2006).
- Studier, F. W. Protein production by auto-induction in high density shaking cultures. Protein Expression and Purification. 41, 207-234 (2005).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved