Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Purification de la M. magneticum Strain AMB-1 Protein magnétosome Associated MamAΔ41
Dans cet article
Résumé
Mama est un unique protéines associées magnétosome qui a été montré pour être impliqués dans l'activation magnétosome. Nous présentons ici le protocole de purification de Mama mutant de délétion (MamAΔ41) de M. magneticum AMB-1.
Résumé
Bactéries magnétotactique constituent un groupe diversifié de micro-organismes aquatiques qui sont capables de s'orienter le long des champs géomagnétiques. Ce comportement est considéré comme une aide leur recherche d'environnements adaptés
Protocole
1. Clonage et expression des gènes dans les mama E. coli
Le gène mutant mamAΔ41 été amplifié en utilisant la réaction en chaîne polymérase (PCR) à partir d'ADN génomique de Magnetospirillum magneticum AMB-1, avec des amorces: 5'-GCATTACGCATATGGACGACATCCGCCAGGTG-3 'et 5'-GCGCGGCAGCCATA-TGGCATACG-3'. Dans les fragments d'ADN amplifié, un site Ncol a été introduit au codon d'initiation ATG et le codon de terminaison a été remplacé par un site SCOI. Les fragments ont été digérés avec Ncol et Sacl et clone dans les sites respectifs de pET52b (+), donnant lieu à pET52bMamAΔ41-amb1. Dans cette construction, le gène a été mamAΔ41 fusionnés dans le cadre avec le 10-His à la partie C-terminale. Le plasmide a été électroporés dans la souche E. coli BL21. E. coli souche BL21 hébergeant pET52bMamAΔ41 a été cultivé dans une auto-induction (15) support contenant de l'ampicilline (50 mg / ml) 310 ° K pendant 3 heures. La température de culture a ensuite été déplacé de 310 ° à 300 ° K et maintenu pour une période supplémentaire de 48 heures à 300 ° K. Les cellules ont été récoltées par centrifugation à 5465g pendant 10 min à 277 ° K. 8 litres de culture produit 60 grammes de granulés cellule humide.
2. Les calculs de bioinformatique
Calculs de poids moléculaire (MW), selon la séquence des acides aminés et l'absorption prévue de 1mg/1ml de protéines dans 280nm, en utilisant le serveur ProtParam (http://www.expasy.ch/tools/protparam.html). Pour 10His-Tag-MamAΔ41 l'MW 22529 Da (240 acides aminés) et pour MamAΔ41 (avec 9 acides aminés à gauche après son enlèvement par tag-thrombine), le Mw est 20596.5Da (187 acides aminés). L'absorption prévue de 1mg/1ml de protéines dans 280nm pour les 10His-Tag-MamAΔ41 est 0,595 et 0,579 MamAΔ41 est. En outre, la séquence d'acides aminés ne contient pas de résidus cystéine. Par conséquent, les agents réducteurs ne sont pas nécessaires pendant le processus de purification.
3. Purification de MamAΔ41
- Pour la préparation de la solution mère, peser et préparer (200 ml chacune) dans les concentrations suivantes: imidazole (228,2 MW g / mol) solution 4M, NaCl (Mw 58,44 g / mol) solution de 5M, Tris-HCl (Mw 121,3 g / mol solution) 1M, ajuster le pH = 8. Ajouter l'eau bidistillée (DDW) et mélanger en utilisant un vortex jusqu'à la solution est claire. Solutions de filtration avec filtre à 0.22μm et garder à température ambiante.
- Préparer et filtre 6 nouveaux tampons à partir des solutions mères (tableau 1).
- Diluer et suspendre les cellules dans le tampon A volume en rapport 1:2, grammes de cellules à mémoire tampon (ml).
- Ajouter 10 ul pour chaque 10gr de bactéries de DNase I (1 mg / ml) afin de briser des fragments d'ADN dans l'échantillon. Ajouter 1ml de chaque 20gr de bactéries sans EDTA cocktail inhibiteur de protéase. Incuber pendant 20 min sur la glace.
- Perturbé les cellules en deux cycles de presse française à 25.000 psi. Contrairement à la sonication, la presse française est destiné à un volume élevé (10ml>) d'extrusion cellulaire en forçant les cellules sous haute pression par un orifice mince. Le piston doit être refroidi sur la glace pendant dix minutes avant son utilisation et de montage en raison de l'accumulation de température.
- Transférer les cellules lysées dans des tubes de 25ml et ultracentrifugeuse les équilibrer avec précision (50mg) avec le tampon A jusqu'à ce que l'échantillon atteint la ligne du cou le tube s.
- Les débris cellulaires par centrifugation séparés Ultra à 270 000 g pendant 1 heure à 277 ° K.
- Préparer une maison gravité Ni NTA colonne (2,5 cm de diamètre) en ajoutant 4 ml de Ni NTA résine, qui sont conserves dans l'éthanol à 20%, sur la colonne. Laissez la chute du liquide complètement, puis laver avec 40 ml de DDW et laisser tomber le liquide complètement. Pré-équilibrée de la résine dans la colonne avec 40 ml de tampon A.
- Appliquer la fraction soluble de l'ultra-centrifugation des tubes sur la gravité Ni NTA colonne. Laissez la chute de fluide, de recueillir à écoulement continu (protéines non) et de préserver au 277K.
- Recueillir un échantillon de granulés à partir des tubes ultra-centrifugation en utilisant un petit pourboire à la ferraille le culot. Mélangez du bout de 50 pl de 5% β-mercaptoéthanol dans le tampon d'échantillon 2X SDS-PAGE.
- Laver la colonne avec 100 ml de tampon B. Laissez la chute de fluide, de recueillir accréditives et de préserver au 277K.
- Préparer 15 tubes Eppendorf marqués (1,5 ml).
- Élution - vanne colonne serrée, puis charger 1ml de tampon C sur la colonne; incuber pendant 3 min, puis en ouvrant la vanne et le débit-à travers la collecte. Répétez cette étape trois fois.
- Eluer reste de la suspension au incrément de 1 ml de tampon C et fermer la vanne.
- Mesurer DO à 280nm des fractions et d'évaluer l'emplacement du pic de protéine, et si la DO est toujours> 0,3, les échantillons supplémentaires sont nécessaires.
- Analyse SDS-PAGE des fractions collectées. Une telle analyse devrait être effectuée aussi avec les échantillons suivants, protéines non accréditives, lavez accréditives et ultra-centrifugation de l'échantillon à granulés. Mélanger 10 ul de chaque échantillon avec 10 ul de 5% β-mercaptoéthanol dans le tampon d'échantillon 2X SDS-PAGE. Charger les échantillons sur 15% SDS-PAGE et courir pendant 45 min à 180 volts.
- Tache le gel dans 15 ml de solution InstantBlue, agiter pendant 1 heure. Assurez-vous que la boîte en plastique est recouvert et protégé de la lumière.
- Évaluer un gel, si l'expression de protéines est élevé et spécifique selon les bandes indicatif, fusion sélectionnées fractions éluées.
- Afin de supprimer les 10-His-tag, ajouter la thrombine bovine (10 unités / ul), 10 ul thrombine par 1 mg de protéine, selon la recommandation du fabricant pour l'échantillon combiné.
- La dialyse est utilisée pour l'enlèvement imidazole excès et à diminuer les concentrations de NaCl pour les niveaux nécessaires pendant l'échange d'ions contraignant. Pour la dialyse, couper la longueur désirée de 7 kDa de poids moléculaire coupé tube à dialyse (MWCO). Laver avec DDW et faire un noeud à une extrémité (ou le sceau avec des clips). Transfert solution de protéines fusionnées sur le sac, tout en laissant peu d'espace en haut et en pince en place.
- Immerger le tube de dialyse en mémoire tampon de 4 litres de dialyse frais (tampon D). Incorporer lentement la solution pendant la nuit à 277 ° K.
- Sortez le tube de dialyse du bécher, faire une petite coupure et le transfert à la protéine de tube 50ml.
- Chromatographie échangeuse d'ions: assembler pré-emballés MonoQ 4.6/100 PE colonne chromatographie liquide rapide (FPLC). Laver la colonne avec 5 volumes de colonne (CV) d'eau filtrée DDW. Équilibrée de la colonne avec 5 CV de tampon D.
- Laver la boucle d'injection avec DDW (deux volumes en boucle) et de répéter avec le tampon D. Charge de la protéine dans la boucle d'injection.
- Injecter l'échantillon de protéine (débit de 1 ml / min) et commencer à recueillir des fractions pour SDS-PAGE de détection des protéines non borné.
- Si le volume protéine initiale (étape 3.23) est plus grande que la boucle d'injection, répétez les étapes 3.25 à 3.26, sans se laver la boucle, jusqu'à ce que tout l'échantillon est chargé sur la colonne.
- Laver la colonne avec 3 CV de tampon C afin d'éliminer les protéines non borné.
- Eluer la protéine avec un gradient linéaire de 60 min 40 2000mm NaCl entre les tampons de D & E (lorsque E tampon de 100% est la fin de la pente), débit 1 ml / min (bien que des débits plus élevés peuvent être utilisés dans le coût de la volumes plus importants, mais moins concentré pics de protéine,). Recueillir des pics de protéines tels qu'ils apparaissent dans le chromatogramme 280nm.
- Nettoyer pré-emballés pour la fabrication de colonnes selon recommandation.
- Analyse SDS-PAGE des fractions collectées. Une telle analyse doit être effectuée avec les échantillons suivants, sans limite d'écoulement des protéines et des protéines grâce à des pics accréditives. Exécutez le SDS-PAGE et coloration que mentionné dans les étapes 3.17 à 3.18.
- Évaluer un gel de protéines des bandes existence à la Mw prédite selon la protéine pré-teint marqueur. Fusionner les fractions d'élution sélectionnées qui contiennent la protéine d'intérêt.
- Dialyser les échantillons fusionnés, afin de réduire les concentrations de NaCl nécessaire pour la chromatographie d'exclusion stérique. Effectuer la dialyse comme mentionné dans les étapes 3.21 à 3.23 contre le tampon F.
- Concentrer l'échantillon de protéines en utilisant un Vivaspin-15 10 000 MWCO à 8 mg / ml dans une centrifugeuse de table, 4000 rpm. Valider la concentration en mesurant le désir de DO à 280nm au Quartz cuvette. Si la protéine est trop concentré, il diluer avec le tampon F et de mesurer à nouveau.
- Assemblez l'exclusion de taille pré-emballés colonne, HiLoad 26/60 Superdex 200, le FPLC; laver la colonne avec 3 CV de l'eau filtrée DDW. Équilibrée de la colonne avec 3 CV de tampon F.
- Laver boucle d'injection avec DDW 5ml (deux volumes en boucle), suivi par le même volume avec le tampon F. Ne chargez pas plus de 4 ml de l'échantillon de protéine dans la boucle d'injection.
- Injecter l'échantillon de protéine (débit de 3,5 ml / min) et commencer à recueillir des fractions de protéines de pointe, tels qu'ils apparaissent sur le chromatogramme on 280nm. Laissez les flux de tampon jusqu'à ce qu'il atteigne le volume de la colonne.
- Si le volume protéine initiale (étape 3.23) est plus grande que la boucle d'injection, répétez les étapes 3.25 à 3.26, sans se laver la boucle, jusqu'à ce que tout l'échantillon est chargé sur la colonne.
- Nettoyer pré-emballés colonne selon la recommandation de fabrication.
- Analyse SDS-PAGE des fractions collectées. Une telle analyse devrait être effectuée avec des pics de protéines des échantillons. Exécutez le SDS-PAGE et coloration que mentionné dans les étapes 3.17 à 3.18.
- Évaluer un gel: si les pics de protéines spécifiques, dans le Mw correcte prédite selon la protéine pré-teint marqueur et une seule bande est visible de fusion sélectionnées fractions éluées de la protéine pertinents.
- Concentré de protéines en utilisant un échantillon de Vivaspin-15 10 000 MWCO à> 20 mg / ml dans une centrifugeuse de table, 4000 rpm. Valider la concentration en mesurant le désir de DO à 280nm. Le MamAΔ41 purifiée est ensuite concentré à 26,5 mg / ml pour la cristallisation.
- Confirmer la pureté de l'échantillon et l'identification de protéines en utilisant Matrix-assisted laser desorption / ionisation (MALDI-TOF).
- Si la protéine est purifiée selon le spectromètre de masse et SDS-PAGE, divisez le MamAΔ41 concentré pour pré-marqués des tubes Eppendorf, 25-50 pl protéine dans chaque.
- Flash congelés dans l'azote liquide et conserver à 193 ° K.
4. Les résultats représentatifs
Lorsque ce protocole est fait correctement on devrait obtenir hautement purifiée, des échantillons de taille homogène et concentrée en protéines. Ces échantillons de protéines sont alors prêts pour des essais de cristallisation ainsi que des études biochimiques, tels que la cinétique enzymatique, l'affinité de liaison et plus encore. Décrites ici sont des résultats représentatifs des principales étapes dans le protocole de purification. SDS-PAGE analyse du profil d'élution de Ni-NTA colonne d'affinité devraient révéler hautement plus protéine exprimée dans la fraction soluble à MW appropriée de ~ 22 kDa (figure 1). Cette analyse SDS-PAGE devrait également révéler minimum si aucune protéine dans les protéines non borné accréditives, lavez accréditives et ultra-centrifugation de l'échantillon à granulés. Si de grandes bandes de la protéine appropriée apparaissent dans ces échantillons on devrait envisager la préparation mauvais tampon, les problèmes de rupture des cellules ou des problèmes dans la culture d'auto-induction des conditions et de croissance.
Chromatographie échangeuse d'ions est préformé de façon à séparer entre nos protéines souhaitable à d'autres protéines de E. coli qui étaient liés à la résine de nickel (due à des interactions électrostatiques ou histidine / négatif aminés des boucles acides riches en protéines) et non coupés His-tag protéines souhaitable. Cette colonne sépare les protéines en fonction de leur affinité de liaison à la résine chargée positivement dans l'augmentation des concentrations de NaCl. Protéine hautement chargées négativement vont éluer des concentrations plus élevées de NaCl apposer à modérée celles chargées négativement. Les avantages de cette colonne sont haut débit et capacité de liaison. Le chromatogramme d'échange d'ions (Figure 2) révèle une bonne séparation entre les populations des protéines 3 en augmentant la concentration de NaCl. Analyse SDS-PAGE est nécessaire afin de déterminer MW de chaque population, d'isoler le souhaitable et l'évaluation si étapes de purification sont nécessaires. La première population est MamAΔ41 (~ 20 kDa) alors que la deuxième population est MamAΔ41 + Son Tag (~ 22 kDa) qui n'était pas clivé par la thrombine bovine et la troisième population est indétectable dans SDS-PAGE en raison de la faible concentration. Si les pics de protéines ne sont pas clairement séparés on devrait envisager la préparation mauvais tampon ou de changer la pente du gradient de NaCl.
Chromatographie d'exclusion stérique est préformé de façon à séparer entre nos protéines souhaitable à d'autres protéines de la cellule de E. coli qui étaient liés à la résine Ni-NTA et n'ont pas été séparés pendant chromatographie échangeuse d'ions. Cette colonne sépare les protéines selon leur taille. Protéines de grande taille seront éluer dans les plus petits volumes d'élution par rapport aux petits qui seront élues dans des volumes plus importants. Le chromatogramme colonne (figure 3) révèle une bonne séparation de la protéine souhaitable car une population principale. La population semble éluer en taille avec un monomère approprié MW de ~ 20 kDa. La présence de la bande ~ 80 kDa (E.coli protéine typique qui lie résine Ni-NTA) est détecté en SDS-PAGE avant le chargement de la colonne et disparaît au cours de cette course. Puisque la concentration de cette protéine ~ 80 kDa est faible au départ, sa population ne figure pas dans le chromatogramme en raison de sa dilution et analyse SDS-PAGE est nécessaire pour déterminer l'efficacité de la purification. Nous recommandons de charger un échantillon dilué et concentré sur le pic principal de la SDS-PAGE ainsi on pourrait sûrement déterminer la présence d'une protéine pure dans la MW appropriée. A ce stade, la protéine est purifiée et son homogénéité doivent être évaluées par MALDI-TOF. Cette analyse a révélé une protéine en MW de 20347 Da et une autre dans Mw de 10 176 Da (Figure 4). La protéine ~ 10 kDa est la protéine ~ 20 kDa doublé facturés. Le MW prévue de MamAΔ41 avec les 9 acides aminés à gauche après son enlèvement a été Tag-20596.5 Da. En la comparant à la MALDI-TOF obtenu MW nous avons constaté qu'ils sont 248 Da à part. Cette dissemblance peut entraîner des erreurs de mesure MALDI-TOF et / ou en raison de la dégradation commune de la première méthionine et la seconde la glycine. Pour conclure, la protéine est hautement purifié et peut être utilisé pour d'autres expériences.
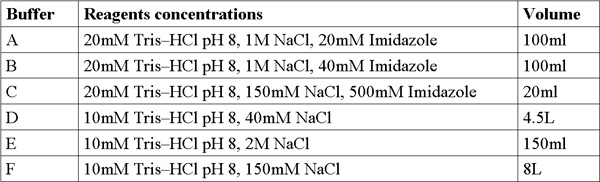
Tableau 1. Formulations tampon.
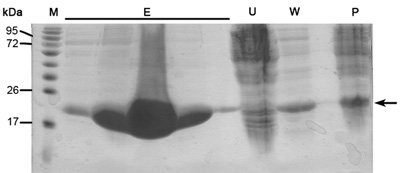
Figure 1. Représentant analyse SDS-PAGE de la purification sur colonne Ni-NTA De droite à gauche;. P-ultra-centrifugeuse granulés (3,11 étape du protocole), W-lavage (3,12 étape du protocole), U-sans limite protéines (3,10 étape du protocole), E- cinq voies d'élution SAMPLes (3,14 étape du protocole), M - marqueur protéique (les chiffres indiquent MW). La flèche indique MamAΔ41.
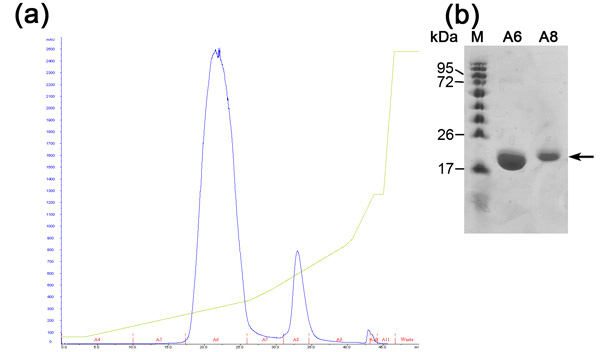
Figure 2. Analyse de la chromatographie échangeuse d'ions étape (a) L'échange d'ions (colonne MonoQ) chromatogramme; Bleu - 280nm absorption, représente la concentration de protéines, vert-de NaCl de concentration, Rouge - indication des fractions collectées. (B) Représentant analyse SDS-PAGE de l'étape de purification par échange d'ions. De gauche à droite, M - marqueur protéique (les chiffres indiquent Mw), A6 - fraction premier pic, fraction du pic A8-seconde.

Figure 3. Analyse par chromatographie d'exclusion de taille (a) l'exclusion Taille (colonne Superdex 200) chromatogramme; Bleu - 280nm absorption, représente la concentration de protéines, l'indication rouge de fractions collectées. (B) Représentant analyse SDS-PAGE d'étape de purification d'exclusion de taille. De gauche à droite; M-protéine marqueur (les chiffres indiquent MW), Prei-pré-injecté l'échantillon, A4-6 fraction combinée recueillies auprès de la première crête, A4-6D-dilué fraction recueillie à partir de la pointe.
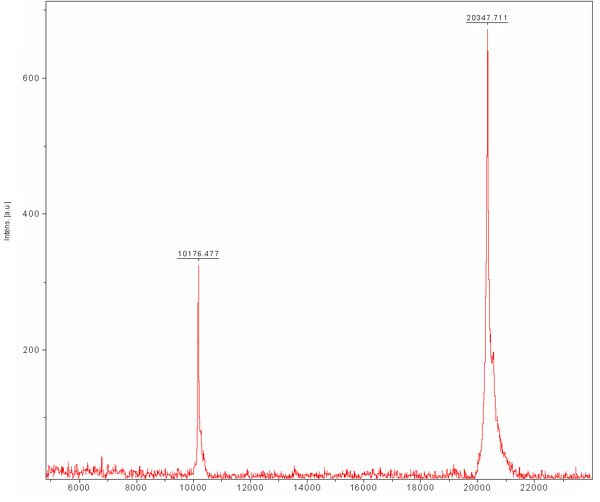
Figure 4. Matrix-assisted laser desorption / ionisation (MALDI-TOF) spectre de masse de purifier MamAΔ41. La matrice est acide sinapique (SA). MamAΔ41 montré à 20347 Da, également représentée est l'espèce doublé chargé de MamAΔ41 à 10176 Da.
Discussion
Purification des protéines est l'étape principale dans toutes les protéines biochimiques ou des études structurales. Comme chaque protéine est unique avec son propre comportement, on a besoin de définir ses propriétés et de modifier en conséquence sa purification. Protéine cible doit être analysé comme une première étape vers la purification à l'aide d'outils bioinformatiques. Ils sont utilisés pour calculer la cible point isoélectrique, d'évaluer son besoin de réduction / oxydation en...
Remerciements
Nous reconnaissons le Dr Amir Aharoni pour son soutien et Geula Davydov, Noam Grimberg et Chen Guttman, pour leurs conseils et commentaires.
matériels
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| French Press | Equipment | Thermo scientific | FA-078A | |
| Pressure cell | Equipment | Thermo scientific | FA-032 | |
| Ultra-centrifuge | Equipment | Sorvall | Discovery 90SE | |
| Rottor | Equipment | Beckman | Ti60 | |
| Ultra-centrifuge tubes; PC-Bottle+Cap Assay 26.3ml | Equipment | Beckman | BC-355618 | |
| 2.5cm diameter, Glass Econo-Column Chromatography Columns | Equipment | BioRad | 737-2521 | |
| Ni-NTA His Bind resin | Equipment | Novagen | M0063428 | |
| Spectrophotometer | Equipment | Amersham Biosiences | Ultraspec 2100 pro | |
| Quartz cuvette | Equipment | Hellma | 104-QS | |
| Fast Performance Liquid Chromatography- AKTA purifier 10 | Equipment | GE Healthcare Biosciences | 28-4062-64 | |
| Ion exchange column – MonoQ 4.6/100 PE | Equipment | GE Healthcare Biosciences | 10025543 | |
| Size exclusion pre-packed column-HiLoad 26/60 Superdex 200 | Equipment | GE Healthcare Biosciences | 17-1071-01 | |
| Centricon - Vivaspin15 – 10,000 MWCO | Equipment | Sartorius Stedim Biotech GmbH | VS1501 | |
| Table centrifuge | Equipment | Thermo scientific | IEC CL30R | |
| MALDI-TOF | Equipment | Bruker Daltonics | Reflex IV | |
| Tris-HCl (hydrotymethyl) aminomethane | Reagent | BioLab | 20092391 | |
| Sodium Chloride | Reagent | FRUTROM | 235553470 | |
| Imidazole | Reagent | Alfa Aesar | 288-32-4 | |
| EDTA free protease inhibitors cocktail | Reagent | Sigma | P-8849 | |
| Dnase I (Deoxyribonuclease I) | Reagent | Sigma | DN-25 | |
| Bovine Thrombin | Reagent | Fisher BioReagents | BP25432 | |
| Glycine | Reagent | BioLab | 07132391 | |
| Soudim Dodecyl Sulfate (SDS) | Reagent | BioLab | 19822391 | |
| Beta-mercaptoethanol | Reagent | Sigma | M-3148 | |
| InstantBlue | Reagent | Expedeon | 1SB01L | |
| PageRuler Prestained Protein Ladder | Reagent | Fermentas | SM0671 |
Références
- Faivre, D., Schuler, D. Magnetotactic Bacteria and Magnetosomes. Chem Rev. 108, 4875-4898 (2008).
- D'Andrea, L. D., Regan, L. TPR proteins: the versatile helix. Trends Biochem Sci. 28, 655-662 (2003).
- Young, J. C., Barral, J. M., Hartl, U. l. r. i. c. h., F, . More than folding: localized functions of cytosolic chaperones. Trends Biochem Sci. 28, 541-547 (2003).
- Brocard, C., Hartig, A. Peroxisome targeting signal 1: is it really a simple tripeptide?. Biochim Biophys Acta. 1763, 1565-1573 (2006).
- Fransen, M., Amery, L., Hartig, A., Brees, C., Rabijns, A., Mannaerts, G. P., Van Veldhoven, P. P. Comparison of the PTS1- and Rab8b-binding properties of Pex5p and Pex5Rp/TRIP8b. Biochim Biophys Acta. 1783, 864-873 (2008).
- Baker, M. J., Frazier, A. E., Gulbis, J. M., Ryan, M. T. Mitochondrial protein-import machinery: correlating structure with function. Trends Cell Biol. 17, 456-464 (2007).
- Mirus, O., Bionda, T., von Haeseler, A., Schleiff, E. Evolutionarily evolved discriminators in the 3-TPR domain of the Toc64 family involved in protein translocation at the outer membrane of chloroplasts and mitochondria. J Mol Model. 15, 971-982 (2009).
- Gatsos, X., Perry, A. J., Anwari, K., Dolezal, P., Wolynec, P. P., Likic, V. A., Purcell, A. W., Buchanan, S. K., Lithgow, T. Protein secretion and outer membrane assembly in Alphaproteobacteria. FEMS Microbiol Rev. 32, 995-1009 (2008).
- Tiwari, D., Singh, R. K., Goswami, K., Verma, S. K., Prakash, B., Nandicoori, V. K. Key residues in Mycobacterium tuberculosis protein kinase G play a role in regulating kinase activity and survival in the host. J Biol Chem. 284, 27467-27479 (2009).
- Edqvist, P. J., Broms, J. E., Betts, H. J., Forsberg, A., Pallen, M. J., Francis, M. S. Tetratricopeptide repeats in the type III secretion chaperone, LcrH: their role in substrate binding and secretion. Mol Microbiol. 59, 31-44 (2006).
- Grunberg, K., Muller, E. C., Otto, A., Reszka, R., Linder, D., Kube, M., Reinhardt, R., Schuler, D. Biochemical and proteomic analysis of the magnetosome membrane in Magnetospirillum gryphiswaldense. Appl Environ Microbiol. 70, 1040-1050 (2004).
- Komeili, A., Vali, H., Beveridge, T. J., Newman, D. K. Magnetosome vesicles are present before magnetite formation, and MamA is required for their activation. Proc Natl Acad Sci USA. 101, 3839-3843 (2004).
- Okuda, Y., Fukumori, Y. Expression and characterization of a magnetosome-associated protein, TPR-containing MAM22, in Escherichia coli. FEBS Lett. 491, 169-173 (2001).
- Taoka, A., Asada, R., Sasaki, H., Anzawa, K., Wu, L. F., Fukumori, Y. Spatial localizations of Mam22 and Mam12 in the magnetosomes of Magnetospirillum magnetotacticum. J Bacteriol. 188, 3805-3812 (2006).
- Studier, F. W. Protein production by auto-induction in high density shaking cultures. Protein Expression and Purification. 41, 207-234 (2005).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon