Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Die Reinigung des M. magneticum Stamm AMB-1 Magnetosom assoziierten Protein MamAΔ41
In diesem Artikel
Zusammenfassung
Mama ist eine einzigartige Magnetosom assoziierten Protein, das nachweislich in Magnetosomen Aktivierung beteiligt sein sollte. Hier präsentieren wir die Aufreinigungsprotokolls von Mama Deletionsmutante (MamAΔ41) aus M. magneticum AMB-1.
Zusammenfassung
Magnetotaktische Bakterien umfassen eine heterogene Gruppe von aquatischen Mikroorganismen, die in der Lage, sich entlang geomagnetischen Feldern orientieren werden. Dieses Verhalten wird angenommen, dass die Suche nach geeigneten Umgebungen Hilfe
Protokoll
1. Klonierung und Expression von Mama Gene in E. coli
Das mutierte Gen mamAΔ41 wurde unter Verwendung der Polymerase-Kettenreaktion (PCR) aus genomischer DNA von Magnetospirillum magneticum AMB-1 mit Primer: 5'-GCATTACGCATATGGACGACATCCGCCAGGTG-3 'und 5'-GCGCGGCAGCCATA-TGGCATACG-3'. In der amplifizierten DNA-Fragmente wurde eine NcoI-Stelle am ATG eingeführt und die Terminationscodon wurde mit einem ScoI Ort ersetzt. Die Fragmente wurden mit NcoI und SacI verdaut und kloniert in den jeweiligen Standorten der pET52b (+), was zu pET52bMamAΔ41-AMB1. In diesem Konstrukt wurde die mamAΔ41 Gen in-frame mit dem 10-His-Tag am C-Terminus fusioniert. Das Plasmid wurde in E. coli-Stamm BL21 elektroporiert. E.-coli-Stamm BL21 beherbergen pET52bMamAΔ41 in auto-Induktion wurde angebaut (15)-Medium mit Ampicillin (50 mg / ml) 310 ° K für 3 Stunden. Der Anbau Temperatur wurde dann von 310 ° bis 300 ° K verschoben und gewartet für weitere 48 h bei 300 ° K. Die Zellen wurden durch Zentrifugation bei 5465g für 10 min bei 277 ° K. geerntet 8 Liter Kultur produziert 60 Gramm feuchten Zellpellet.
2. Bioinformatics Berechnungen
Berechnungen von Molekulargewicht (MW), nach Aminosäuren-Sequenz und die vorhergesagte Aufnahme von 1mg/1ml von Protein in 280nm, mit dem ProtParam Server (http://www.expasy.ch/tools/protparam.html). Für 10His-Tag-MamAΔ41 der MW ist 22529 Da (240 Aminosäuren) und für MamAΔ41 (mit 9 Aminosäuren nach His-tag Entfernung von Thrombin links) Mw ist 20596.5Da (187 Aminosäuren). Der vorhergesagte Aufnahme von 1mg/1ml von Protein in 280nm für 10His-Tag-MamAΔ41 ist 0,595 und für MamAΔ41 ist 0,579. Darüber hinaus bedeutet die Aminosäure-Sequenz enthalten keine Cysteinreste. Daher sind Reduktionsmittel nicht während der Reinigung benötigt.
3. Reinigung von MamAΔ41
- Für Stammlösung Vorbereitung, abwiegen und vorbereiten (200 ml) in den folgenden Konzentrationen: Imidazol (MW 228,2 g / mol) 4M Lösung, NaCl (Mw 58,44 g / mol) 5M-Lösung, Tris-HCl (Mw 121,3 g / mol ) 1M-Lösung, pH-Wert einstellen = 8. Fügen Sie doppelt destilliertem Wasser (DDW) und mischen mit einem Vortex, bis die Lösung klar ist. Filter-Lösungen mit 0,22 &mgr; m-Filter und halten bei Raumtemperatur.
- Vorbereiten und Filter 6 frischen Puffer aus Stammlösungen (Tabelle 1).
- Verdünnen und Suspend-Zellen in Puffer A im Verhältnis 1:2, Gramm Zellen Puffervolumen (ml).
- Add 10 &mgr; l für jede 10gr von Bakterien der DNase I (1 mg / ml), um DNA-Fragmente in der Probe zu brechen. 1ml für jede 20gr von Bakterien EDTA frei Protease-Inhibitor-Cocktail. Inkubieren für 20 min auf Eis.
- Unter aufgeschlossenen Zellen in zwei Zyklen von Französisch Presse bei 25.000 psi. Im Gegensatz zu Ultraschall ist die Französisch drücken bei hoher Lautstärke (> 10ml) Zelle Extrusion, indem sie den Zellen unter hohem Druck durch eine dünne Öffnung gerichtet. Der Kolben sollte auf Eis 10 Minuten vor ihrer Verwendung und Montage durch Temperatur Aufbau gekühlt werden.
- Übertragen Sie die lysierten Zellen in 25ml Ultrazentrifuge Rohre und Balance ihnen genau (50mg) mit Puffer A, bis die Probe erreicht das Rohr s Ausschnitt.
- Separate Zelltrümmer durch Ultra-Zentrifugation bei 270.000 g für 1 Stunde bei 277 ° K.
- Bereiten Sie eine hausgemachte Schwerkraft Ni NTA-Säule (2,5 cm Durchmesser) durch Zugabe von 4 ml Ni NTA-Harz, das in 20% Ethanol konserviert sind, auf die Säule. Lassen Sie die Flüssigkeit fallen komplett, dann mit 40 ml DDW waschen und lassen Sie die Flüssigkeit fallen vollständig aus. Voräquilibriert das Harz in der Säule mit 40 ml Puffer A
- Übernehmen der löslichen Fraktion von ultra-Zentrifugenröhrchen auf die Schwerkraft Ni NTA-Säule. Lassen Sie die Flüssigkeit fallen, sammeln Flow-Through-(ungebundene Proteine) und erhalten bei 277K.
- Sammeln Sie Pellet Probe aus dem ultra-Zentrifugenröhrchen mit einem kleinen Tipp, um das Pellet Schrott. Mix der Spitze in 50 ul 5% β-Mercaptoethanol in 2X SDS-PAGE-Probenpuffer.
- Die Säule wird mit 100 ml Puffer B Lassen Sie die Flüssigkeit fallen, sammeln Flow-Through und erhalten bei 277K.
- Bereiten Sie 15 markierte Eppendorf-Röhrchen (1,5 ml).
- Elution - in der Nähe Spalte Ventil und laden Sie dann 1 ml Puffer C auf die Säule; inkubieren 3 min von Öffnungs-und Flow-Through-Auflistung. Wiederholen Sie diesen Schritt dreimal.
- Eluieren Rest der Suspension bei 1 ml Puffer C-Schritten und Schließen des Ventils.
- Messen der OD bei 280 nm der Fraktionen und bewerten Ort der Protein-Peak, wenn die OD ist> 0,3, zusätzliche Proben notwendig sind.
- SDS-PAGE-Analyse der gesammelten Fraktionen. Eine solche Analyse sollte auch mit den folgenden Proben durchgeführt werden, ungebundenen Proteine Flow-Through-, Wasch-Flow-Through-und uLTRA-Zentrifugationspellet Probe. Mix 10 &mgr; l von jeder Probe mit 10 &mgr; l 5% β-Mercaptoethanol in 2X SDS-PAGE-Probenpuffer. Laden Sie die Proben auf 15% SDS-PAGE und führen Sie für 45 min in 180 Volt.
- Stain das Gel in 15 ml InstantBlue Lösung für 1 Stunde geschüttelt. Stellen Sie sicher, dass die Kunststoff-Box bedeckt ist und vor Licht geschützt.
- Bewerten Gel, wenn Proteinexpression ist hoch und spezifische gemäß der indikativen Bands, zusammenführen ausgewählt eluierten Fraktionen.
- Um 10-His-Tag zu entfernen, fügen Rinderthrombin (10 Einheiten / ul), 10 &mgr; l Thrombin pro 1 mg Protein, entsprechend der Empfehlung des Herstellers der kombinierten Probe.
- Die Dialyse ist für überschüssige Imidazol Entfernung verwendet und NaCl-Konzentrationen für die Stufen beim Ionenaustausch Bindung benötigt verringern. Für die Dialyse, schneiden die gewünschte Länge von 7 kD molecular weight cut off (MWCO) Dialyseschlauch. Waschen mit DDW und machen einen Knoten an einem Ende (oder Siegel mit Clips). Transfer fusionierte Protein-Lösung auf den Beutel während Verlassen etwas Platz am oberen und Klemmung.
- Tauchen Sie die Dialyseschlauch in 4 Liter gekühltes Dialyse-Puffer (Buffer D). Rühren Sie die Lösung langsam über Nacht bei 277 ° K.
- Nehmen Sie den Dialyseschlauch aus dem Becherglas, machen einen kleinen Schnitt und den Transfer des Proteins auf 50ml Tube.
- Ionenaustausch-Chromatographie: Montage vorverpackten MonoQ 4.6/100 PE Spalte auf Fast Performance Liquid Chromatography (FPLC). Die Säule wird mit 5 Säulenvolumen (CV) von DDW gefiltertes Wasser. Equilibrated die Säule mit 5 SV Puffer D.
- Waschen Sie die Probenschleife mit DDW (zwei Loop-Volumes) und wiederholen Sie mit Puffer D. Laden des Proteins in der Probenschleife.
- Spritzen Sie die Proteinprobe (Durchfluss von 1 ml / min) und sammeln Fraktionen für SDS-PAGE Detektion von Proteinen unbegrenzt.
- Wenn das erste Protein Volumen (Schritt 3,23) größer ist als Injektionsschleife, wiederholen Sie die Schritte 3,25-3,26, ohne Waschen der Schleife, bis die gesamte Probe auf der Säule geladen wird.
- Die Säule wird mit 3 CV des Puffers C, um grenzenlose Proteine zu entfernen.
- Eluieren des Proteins mit einer 60 min linearer Gradient von 40 2000mm NaCl zwischen Puffer D & E (bei 100% Puffer E das Ende der Steigung), Fließgeschwindigkeit 1 ml / min (obwohl höhere Strömungsgeschwindigkeiten in den Kosten genutzt werden können größere Volumina, aber weniger konzentriert, Protein-Peaks). Sammeln Proteine Gipfel, wie sie in der 280nm-Chromatogramm erscheinen.
- Reinigen Sie vor Füllkörperkolonne nach Empfehlung herzustellen.
- SDS-PAGE-Analyse der gesammelten Fraktionen. Eine solche Analyse sollte mit den folgenden Proben durchgeführt werden, unbounded Proteine Flow-Through-und Protein-Peaks Durchströmung. Führen Sie das SDS-PAGE und Färbung wie in den Schritten von 3,17 bis 3,18 zu nennen.
- Bewerten Gel für Proteinbanden Existenz der vorhergesagten Mw nach dem Protein vor-gefärbten Marker. Merge ausgewählt Elutionsfraktionen, dass die entsprechenden Proteine enthalten.
- Dialysieren der fusionierten Proben, um NaCl-Konzentrationen für Größenausschlusschromatographie nötig zu senken. Führen Sie die Dialyse wie in den Schritten 3,21-3,23 gegen Puffer F. genannten
- Konzentrieren Sie sich das Protein Probe mit einem Vivaspin-15 10.000 MWCO zu 8 mg / ml in einer Tischzentrifuge, 4000 Umdrehungen pro Minute. Validate Wunsch Konzentration durch Messung der OD bei 280 nm in Quarz-Küvette. Wenn das Protein zu konzentriert zu verdünnt ist, mit Puffer F und messen Sie erneut.
- Montieren Größenausschlusschromatographie vorverpackten Spalte HiLoad 26/60 Superdex 200, auf FPLC; Die Säule wird mit 3 CV von DDW gefiltertes Wasser. Equilibrated die Säule mit 3 SV Puffer F.
- Wash Injektionsschleife mit 5 ml DDW (zwei Loop-Volumes) durch das gleiche Volumen mit Puffer F. gefolgt Laden nicht mehr als 4 ml der Proteinprobe in die Probenschleife.
- Spritzen Sie die Proteinprobe (Flussrate von 3,5 ml / min) und sammeln Proteine Peak-Fraktionen, wie sie es dem 280nm-Chromatogramm erscheinen. Lassen Sie den Puffer fließen, bis es Säulenvolumen erreicht.
- Wenn das erste Protein Volumen (Schritt 3,23) größer ist als Injektionsschleife, wiederholen Sie die Schritte 3,25-3,26, ohne Waschen der Schleife, bis die gesamte Probe auf der Säule geladen wird.
- Reinigen Sie vor Füllkörperkolonne nach der Herstellung Empfehlung.
- SDS-PAGE-Analyse der gesammelten Fraktionen. Eine solche Analyse sollte mit dem Protein-Peaks Proben durchgeführt werden. Führen Sie das SDS-PAGE und Färbung wie in den Schritten von 3,17 bis 3,18 zu nennen.
- Bewerten Gel: Wenn Protein-Peaks spezifisch sind, in die richtige vorhergesagt Mw nach dem Protein vor-gefärbten Marker und nur eine Bande sichtbar ist verschmelzen ausgewählte eluierten Fraktionen des entsprechenden Proteins.
- Konzentrieren Sie sich Proteinprobe mit einem Vivaspin-15 10.000 MWCO bis> 20 mg / ml in einer Tischzentrifuge, 4000 Umdrehungen pro Minute. Validate Wunsch Konzentration durch Messung der OD bei 280nm. Das gereinigte MamAΔ41 wurde dann auf 26,5 mg / ml für die Kristallisation konzentriert.
- Bestätigen Probe Reinheit und Identifizierung von Proteinen mittels Matrix-Assisted Laser Desorption / Ionisation (MALDI-TOF).
- Wenn das Protein nach dem Massenspektrometer und SDS-PAGE gereinigt wird, teilen Sie die konzentrierte MamAΔ41 vor, markierte Eppendorf-Röhrchen, 25-50 ul Protein in jeder.
- Flash in flüssigem Stickstoff und lagern bei 193 ° K. eingefroren
4. Repräsentative Ergebnisse
Wenn dieses Protokoll korrekt ausgeführt wird sollte man sich hochreine, Größe homogen und konzentrierte Protein-Proben. Diese Protein-Proben werden dann zur Kristallisation Studien sowie biochemische Untersuchungen, wie Enzymkinetik, Bindungsaffinität und mehr bereit. Hier beschriebenen repräsentativen Ergebnisse der wichtigsten Schritte bei der Reinigung Protokoll. SDS-PAGE-Analyse von Elutionsprofil Ni-NTA-Affinitätssäule sollte hoch offenbaren über exprimierten Proteins in der löslichen Fraktion an geeigneten MW ~ 22kDa (Abbildung 1). Das SDS-PAGE-Analyse sollte auch zeigen, Minimum, wenn jedes Protein in der grenzenlosen Proteine Flow-Through-, Wasch-Flow-Through-und ultra-Zentrifugationspellet Probe. Wenn große Bands des entsprechenden Proteins in diesen Proben nicht erscheinen sollte man überlegen, falsch Puffer Vorbereitung, Probleme in Zellaufschluss oder Probleme in der Kultur-induzierenden Bedingungen und Wachstum.
Die Ionenaustauschchromatographie ist vorgeformt, um zwischen unseren wünschenswert Protein zu anderen E. coli-Proteine, die an die Nickel-Harz (zB aufgrund elektrostatischer Wechselwirkungen oder Histidin / negative Aminosäuren reiches Protein-Schleifen) und uncut His-Tagged wünschenswert Protein gebunden waren getrennt. Diese Spalte trennt Proteine nach ihrer Affinität zu positiv geladenen Harz unter zunehmendem NaCl-Konzentrationen. Stark negativ geladenen Proteine werden in höheren NaCl-Konzentrationen eluiert appose zu negativ geladenen moderieren. Die Vorteile dieser Spalte sind hohe Strömungsgeschwindigkeit und Bindungsfähigkeit. Die Ionenaustausch-Chromatogramm (Abbildung 2) zeigt eine gute Trennung zwischen 3 Proteine Populationen in zunehmenden NaCl-Konzentration. SDS-PAGE-Analyse ist notwendig, um MW jeder Population zu bestimmen, die Isolierung des wünschenswertes und Bewertung, ob weitere Reinigungsschritte notwendig sind. Die erste Bevölkerung MamAΔ41 (~ 20 kDa), während die zweite Population ist MamAΔ41 + His Tag (~ 22kDa), die nicht von bovinem Thrombin gespalten und die dritte Bevölkerung ist in SDS-PAGE aufgrund der geringen Konzentration nicht nachweisbar. Wenn die Proteine Peaks nicht getrennt sind klar sollte man überlegen, falsch Puffer Vorbereitung oder Änderung der Steigung der NaCl-Gradienten.
Größenausschlußchromatographie ist vorgeformt, um zwischen unseren wünschenswert Protein zu anderen E. coli-Zelle Proteine, die das Ni-NTA-Harz gebunden waren und nicht während der Ionenaustauschchromatographie getrennt zu trennen. Diese Spalte trennt Proteine nach ihrer Größe. Große Proteine werden in kleineren Elutionsvolumen im Gegensatz zu kleinen, die in größeren Mengen eluiert werden eluieren. Die Spalte Chromatogramm (Abbildung 3) zeigt eine gute Trennung der erwünschten Proteins als eine Haupt Bevölkerung. Die Bevölkerung scheint in geeigneten Monomer Größe mit einem Molekulargewicht von ~ 20 kDa eluieren. Die Anwesenheit von ~ 80 kDa-Bande (E.coli typische Protein, das Ni-NTA-Harz bindet) in SDS-PAGE vor Spalte Laden erkannt und verschwindet während dieses Laufes. Da die Konzentration dieser ~ 80 kDa-Protein ist niedrig zu beginnen, ist die Bevölkerung nicht in dem Chromatogramm durch ihre Verdünnung und SDS-PAGE-Analyse ist notwendig, um die Reinigungsleistung zu bestimmen scheinen. Wir empfehlen das Laden einer verdünnten und konzentrierten Probe der Hauptgipfel der SDS-PAGE so dass man sicher bestimmen konnte das Vorhandensein eines reinen Proteins in der entsprechenden MW. Zu diesem Zeitpunkt wird das Protein gereinigt und seine Homogenität sollte durch MALDI-TOF ausgewertet werden. Diese Analyse zeigte, ein Protein in MW 20347 Da und ein weiteres in Mw von 10176 Da (Abbildung 4). Die ~ 10 kDa-Protein ist die ~ 20 kDa Protein berechnet verdoppelt. Die prognostizierte MW MamAΔ41 mit den 9 Aminosäuren nach His-Tag Entfernung links war 20596,5 Da. Durch Vergleich mit den erhaltenen MALDI-TOF MW fanden wir, dass sie 248 Da voneinander entfernt sind. Diese Verschiedenheit kann von MALDI-TOF Messfehler und / oder durch gemeinsamen Abbau der ersten Methionin und Glycin die zweite Folge. Zum Schluss wird das Protein hoch gereinigt und kann für weitere Experimente verwendet werden.
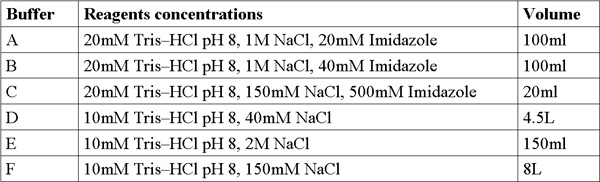
Tabelle 1. Buffer Formulierungen.
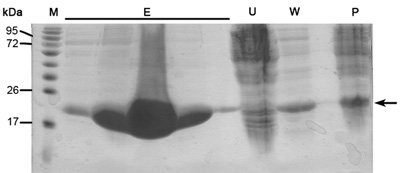
Abbildung 1. Vertreter SDS-PAGE-Analyse von Ni-NTA-Säule Reinigung Von rechts nach links;. P-Ultrazentrifuge Pellet (3,11 Protokoll Schritt), W-wash (3,12 Protokoll Schritt), U-unbounded Proteine (3,10 Protokoll Schritt), E- fünf Bahnen der Elution samples (3,14 Protokoll Schritt), M - Protein-Marker (Zahlen geben MW). Der Pfeil zeigt MamAΔ41.
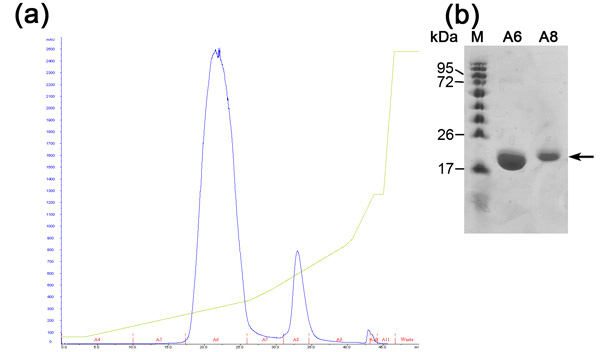
Abbildung 2. Die Analyse der Ionenaustauschchromatographie Schritt (a) Ionenaustauscher (MonoQ Spalte) Chromatogramm; Blue - Absorption 280 nm, stellt Proteinkonzentration, Green-NaCl-Konzentration, Red - Anzeige der Fraktionen gesammelt. (B) Repräsentative SDS-PAGE-Analyse der Ionenaustausch Reinigungsschritt. Von links nach rechts; M - Protein-Marker (Zahlen geben Mw), A6 - erste Peakfraktion, A8-Sekunden-Peak-Fraktion.

Abbildung 3. Größenausschlußchromatographie Analyse (a) Größenausschlußchromatographie (Superdex 200-Säule) Chromatogramm; Blue - Absorption 280 nm, stellt Proteinkonzentration, Red Angabe der Fraktionen gesammelt. (B) Repräsentative SDS-PAGE-Analyse von Size Exclusion Reinigungsschritt. Von links nach rechts; M-Protein-Marker (Zahlen geben MW), Prei-pre-injizierten Probe, A4-6 kombiniert Fraktion aus der ersten Spitze gesammelt, A4-6D-verwässert-Fraktion aus der Spitze gesammelt.
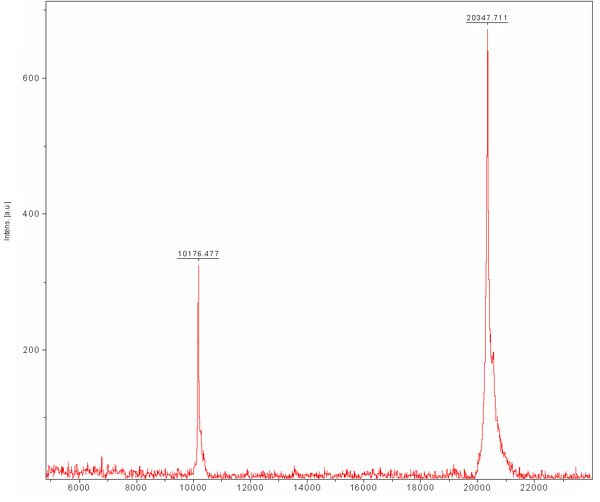
Abbildung 4. Matrix-Assisted Laser Desorption / Ionisation (MALDI-TOF)-Massenspektrum des gereinigten MamAΔ41. Die Matrix ist Sinapinsäure (SA). MamAΔ41 bei 20347 Da gezeigt, auch gezeigt, ist das doppelt geladene Spezies von MamAΔ41 bei 10176 Da.
Diskussion
Proteinreinigung ist der wichtigste Schritt in jedem Proteine biochemische oder strukturelle Untersuchungen. Da jedes Protein ist einzigartig mit seinen eigenen Verhaltens, braucht man, um seine Eigenschaften zu definieren und ändern ihre Reinigung entsprechend. Zielprotein sollte als ein erster Schritt zur Reinigung mit bioinformatischen Methoden analysiert werden. Sie werden verwendet, um das Ziel iso-elektrischen Punkt zu berechnen, bewerten ihre Notwendigkeit für reduzierende / oxidierende Umwelt und ihrer N...
Danksagungen
Wir danken Dr. Amir Aharoni für seine Unterstützung und Geula Dawydow, Noam Grimberg und Chen Guttman für ihre Ratschläge und Kommentare.
Materialien
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| French Press | Equipment | Thermo scientific | FA-078A | |
| Pressure cell | Equipment | Thermo scientific | FA-032 | |
| Ultra-centrifuge | Equipment | Sorvall | Discovery 90SE | |
| Rottor | Equipment | Beckman | Ti60 | |
| Ultra-centrifuge tubes; PC-Bottle+Cap Assay 26.3ml | Equipment | Beckman | BC-355618 | |
| 2.5cm diameter, Glass Econo-Column Chromatography Columns | Equipment | BioRad | 737-2521 | |
| Ni-NTA His Bind resin | Equipment | Novagen | M0063428 | |
| Spectrophotometer | Equipment | Amersham Biosiences | Ultraspec 2100 pro | |
| Quartz cuvette | Equipment | Hellma | 104-QS | |
| Fast Performance Liquid Chromatography- AKTA purifier 10 | Equipment | GE Healthcare Biosciences | 28-4062-64 | |
| Ion exchange column – MonoQ 4.6/100 PE | Equipment | GE Healthcare Biosciences | 10025543 | |
| Size exclusion pre-packed column-HiLoad 26/60 Superdex 200 | Equipment | GE Healthcare Biosciences | 17-1071-01 | |
| Centricon - Vivaspin15 – 10,000 MWCO | Equipment | Sartorius Stedim Biotech GmbH | VS1501 | |
| Table centrifuge | Equipment | Thermo scientific | IEC CL30R | |
| MALDI-TOF | Equipment | Bruker Daltonics | Reflex IV | |
| Tris-HCl (hydrotymethyl) aminomethane | Reagent | BioLab | 20092391 | |
| Sodium Chloride | Reagent | FRUTROM | 235553470 | |
| Imidazole | Reagent | Alfa Aesar | 288-32-4 | |
| EDTA free protease inhibitors cocktail | Reagent | Sigma | P-8849 | |
| Dnase I (Deoxyribonuclease I) | Reagent | Sigma | DN-25 | |
| Bovine Thrombin | Reagent | Fisher BioReagents | BP25432 | |
| Glycine | Reagent | BioLab | 07132391 | |
| Soudim Dodecyl Sulfate (SDS) | Reagent | BioLab | 19822391 | |
| Beta-mercaptoethanol | Reagent | Sigma | M-3148 | |
| InstantBlue | Reagent | Expedeon | 1SB01L | |
| PageRuler Prestained Protein Ladder | Reagent | Fermentas | SM0671 |
Referenzen
- Faivre, D., Schuler, D. Magnetotactic Bacteria and Magnetosomes. Chem Rev. 108, 4875-4898 (2008).
- D'Andrea, L. D., Regan, L. TPR proteins: the versatile helix. Trends Biochem Sci. 28, 655-662 (2003).
- Young, J. C., Barral, J. M., Hartl, U. l. r. i. c. h., F, . More than folding: localized functions of cytosolic chaperones. Trends Biochem Sci. 28, 541-547 (2003).
- Brocard, C., Hartig, A. Peroxisome targeting signal 1: is it really a simple tripeptide?. Biochim Biophys Acta. 1763, 1565-1573 (2006).
- Fransen, M., Amery, L., Hartig, A., Brees, C., Rabijns, A., Mannaerts, G. P., Van Veldhoven, P. P. Comparison of the PTS1- and Rab8b-binding properties of Pex5p and Pex5Rp/TRIP8b. Biochim Biophys Acta. 1783, 864-873 (2008).
- Baker, M. J., Frazier, A. E., Gulbis, J. M., Ryan, M. T. Mitochondrial protein-import machinery: correlating structure with function. Trends Cell Biol. 17, 456-464 (2007).
- Mirus, O., Bionda, T., von Haeseler, A., Schleiff, E. Evolutionarily evolved discriminators in the 3-TPR domain of the Toc64 family involved in protein translocation at the outer membrane of chloroplasts and mitochondria. J Mol Model. 15, 971-982 (2009).
- Gatsos, X., Perry, A. J., Anwari, K., Dolezal, P., Wolynec, P. P., Likic, V. A., Purcell, A. W., Buchanan, S. K., Lithgow, T. Protein secretion and outer membrane assembly in Alphaproteobacteria. FEMS Microbiol Rev. 32, 995-1009 (2008).
- Tiwari, D., Singh, R. K., Goswami, K., Verma, S. K., Prakash, B., Nandicoori, V. K. Key residues in Mycobacterium tuberculosis protein kinase G play a role in regulating kinase activity and survival in the host. J Biol Chem. 284, 27467-27479 (2009).
- Edqvist, P. J., Broms, J. E., Betts, H. J., Forsberg, A., Pallen, M. J., Francis, M. S. Tetratricopeptide repeats in the type III secretion chaperone, LcrH: their role in substrate binding and secretion. Mol Microbiol. 59, 31-44 (2006).
- Grunberg, K., Muller, E. C., Otto, A., Reszka, R., Linder, D., Kube, M., Reinhardt, R., Schuler, D. Biochemical and proteomic analysis of the magnetosome membrane in Magnetospirillum gryphiswaldense. Appl Environ Microbiol. 70, 1040-1050 (2004).
- Komeili, A., Vali, H., Beveridge, T. J., Newman, D. K. Magnetosome vesicles are present before magnetite formation, and MamA is required for their activation. Proc Natl Acad Sci USA. 101, 3839-3843 (2004).
- Okuda, Y., Fukumori, Y. Expression and characterization of a magnetosome-associated protein, TPR-containing MAM22, in Escherichia coli. FEBS Lett. 491, 169-173 (2001).
- Taoka, A., Asada, R., Sasaki, H., Anzawa, K., Wu, L. F., Fukumori, Y. Spatial localizations of Mam22 and Mam12 in the magnetosomes of Magnetospirillum magnetotacticum. J Bacteriol. 188, 3805-3812 (2006).
- Studier, F. W. Protein production by auto-induction in high density shaking cultures. Protein Expression and Purification. 41, 207-234 (2005).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten