Method Article
פריימר ללא aptamer לבחירה באמצעות ספריית ה-DNA אקראיים
In This Article
Summary
פרוטוקולים Selex מהווים סבבים רבים של ברירה, שכל אחד מהם דורש התחדשות של ligands כבול, אשר בתורו דורשים רצפים פריימר קבוע איגוף האזורים ספריה אקראי. רצפים אלה תחל קבוע יכול להפריע בתהליך הבחירה (חיוביות שגויות תשלילים). כאן אנו מציגים פריימר ללא פרוטוקול.
Abstract
Aptamers oligonucleotides הם מובנים מאוד (DNA או RNA) שיכולות להיקשר מטרות עם זיקות להשוות נוגדנים 1. הם מזוהים באמצעות תהליך שנקרא אבולוציה במבחנה בחירה שיטתית של ligands על ידי העשרה מעריכי (Selex) להכיר מגוון רחב של מטרות, החל מולקולות קטנות לחלבונים מקרומולקולות אחרים 2-4. Aptamers יש תכונות כי הם גם מתאים in vivo אבחון ו / או יישומים רפואיים: מלבד סגוליות זיקה טובה, הם מסונתזים בקלות, לשרוד בתנאים עיבוד קפדני יותר, הם immunogenic גרוע, ואת גודל קטן יחסית שלהם יכול לגרום לחדירה של קליל רקמות.
Aptamers המזוהות בתהליך Selex רגיל בדרך כלל מהווים ~ 80 נוקלאוטידים (NT), שכן הם נבחרו בדרך כלל מספריות חומצות גרעין עם ~ 40 אזורים nt אקראי ארוך בנוסף לאתרי פריימר קבוע של ~ 20 NT בכל צד. רצפים פריימר קבוע ולכן ניתן מהווים כמעט 50% ~ של רצפי הספרייה, ולכן עשוי באופן חיובי או שלילי פשרה זיהוי aptamers בתהליך הבחירה 3, למרות הגישות ביואינפורמטיקה מראים כי רצפים קבועים שאינם תורמים באופן משמעותי למבנה aptamer לאחר בחירת 5 . כדי לענות על צרכים אלה בעיות פוטנציאליות, רצפים פריימר נחסמו על ידי oligonucleotides משלימים או עבר באמצע רצפים שונים במהלך סבבי Selex 6, או שהם כבר קיזזתי 6-9 nt 7, 8. וון גריי 9 עיצב פריימר ללא Selex שיטה גנומי, שבו רצפי פריימר הוסרו לחלוטין מהספריה לפני הבחירה היו אז מחדש כדי לאפשר הגברה של שברי גנומית שנבחר. עם זאת, כדי להעסיק את הטכניקה, ספרייה גנומית ייחודי שיוקם, אשר ברשותה מגוון מוגבל, התחדשות אחרי סיבובי בחירה מסתמך על צעד reamplification ליניארי. לחלופין, המאמצים כדי לעקוף בעיות הנגרמות על ידי רצפי פריימר קבוע באמצעות מחיצות יעילות גבוהה הם נפגשו עם בעיות לגבי הגברה PCR 10.
פיתחנו פריימר חינם (PF) שיטת הבחירה משמעותית מפשט הליכים Selex וביעילות מבטלת פריימר התערבות בעיות 11, 12. פרוטוקולים העבודה בצורה ישירה. האזור אקראי המרכזי של הספרייה הוא מטוהרים ללא רצפים איגוף זרים והוא מחויב ליעד מתאים (למשל לחלבון מטוהרים או תערובות מורכבות כגון שורות תאים). ואז רצפים מחויב מתקבלים, להתאחד עם רצפים איגוף, מחדש מוגבר ליצור נבחר תת ספריות. כדוגמה, כאן בחרנו aptamers אל S100B, סמן חלבון למלנומה. מבחני איגוד הראה Kd s ב -10 -7 - 10 -8 M טווח אחרי כמה סיבובים של הבחירה, ואנו מראים כי aptamers לתפקד ביעילות בפורמט כריך מחייב.
Protocol
1. תיאור קצר של פריימר ללא הפרוטוקולים Selection
ספריה פעמיים גדילי דנ"א נבנה באמצעות PCR עם oligonucleotides המקביל (Figure1 ו -2, שלב א), אשר מכילים תחום אקראי המרכזי של 30 nt, בין שני האזורים פריימר. שני פרוטוקולים שונים במקצת "פריימר חופשי" (PF) פותחו. בספריית dsDNA, האזור 5'-מכיל endonuclease "שסחב" האתר עבור Nt.BstNBI endonuclease; אנזים זה מזהה dsDNA cleaves אבל רק אחד גדיל DNA של המצע. האזור 3'של הספרייה מכילה dsDNA אחר "שסחב" באתר, עבור endonuclease Nt.BbvCI כי גם מכיר dsDNA אבל cleaves גדיל אחד בלבד, והשאיר CC ב 3'end (PF 1), וכן BspMI הגבלת אתר endonuclease, אשר cleaves שני הגדילים עוזב נוקלאוטידים מס '3 "נוספים (PF 2). בדרך כלל אנחנו מפעילים את פרוטוקול PF 1 בהתחלה (כי קל יותר שלה שברי נפרד מיוצר בתהליך הבחירה; ראו להלן), ולאחר מכן להעסיק את פרוטוקול PF 2 עבור סיבובים הבאים של הבחירה.
32 NT של "שבר (המיועד 32 +, שבר) לבין 30 nt של 5'-pN30-3" 5'-pN30-CC-3 שבר (המיועד 30 +, שבר) נוצרו בהתאמה ידי Nt.BbvCI / NT . BstNBI או BspMI / Nt.BstNBI המחשוף של הספרייה dsDNA, ואת ג'ל לטיהור. 32 +, שבר מכיל את תחום 30 nt אקראי, עם רצף CC איגוף בסוף 3'-(PF 1). 30 +, שבר (PF 2) כולל רק את רצף 30 nt תחום אקראית. "העצמי גשר" 66 - שבר (המכיל את האזור אקראי N30 עם 5 'ו 3' רצפים איגוף) הושג על ידי טיהור ג'ל (Nt.BbvCI או Nt.BstNBI חתכים רק העליון "+" גדיל). זו עצמית גשר מתקבל ישירות בפרוטוקול 1 PF, או יכול להיווצר ומבודדים לאחר חיתוך ה-DNA ספריה עם NtBbv.CI בלבד או Nt.BstNBI עבור 2 PF פרוטוקול (איור 1 ו -2, שלב ב). 32 + - או 32 +, שברי הודגרו עם חלבונים מטוהרים או בתאי מלנומה בתרבית כדי לאפשר השברים להתחבר לחלבונים או התאים, ולאחר מכן את שברי מאוגד נשטפו (איור 1 ו -2, שלב ג). שברי כבול או שנבחרו שימשו לייצור מחדש של האזורים פריימר.
בתגובה הכלאה / קשירת, את עצמי גשר הופק מטוהרים באותו זמן כמו 32 + - 30 + ו-שבר טיהור; 5'-end פריימר היה זהה לזה של בניית הספרייה 3'סוף primers הוחלפו עם התאמת primers שהכיל גם האמרגן נוספים SP6 שעתוק בסוף 3'-(איור 1 ו -2, שלב ד). המוצרים התגובה הכלאה / קשירת שימשו ואז ב שעתוק במבחנה רנ"א (איור 1 ו -2, דואר שלב). בעקבות תעתיק הרנ"א, DNAs ligated (כולל שבר העצמי גשר נבחר DNA) היו מעוכל על ידי DNase אני להסרת רצפי רקע מפריעים. שעתוק לאחור (RT)-PCR הנתונים הראו כי תחל מחדש, המוצרים היו reamplified ביעילות, המהווה "עגול" של בחירה (איור 1 ו - 2, שלב ו). הגברה הרקע שמוצג על המספרים מחזור גבוה לא-RT-PCR השליטה ניתן להסיר לחלוטין על ידי עיכול נוסף אני DNase, ואינו לזיהוי מספרי לאחר מחזור נמוך יותר, אשר אנו מעסיקים באופן שגרתי.
לאחר 7 סיבובים של הבחירה, aptamers התאפיינו עבור מאפייני מחייב. Kd של כולם היו בטווח M 10 -7 10 -8 (איור 3). בכל מבחני הכריכה, זוגות שונים של aptamers הראה מחייב כתוסף, המציין כי הם יעד לאתרים ייחודיים על החלבון S100B. לפיכך, אנו נבחן זוגות aptamers מבחני "כריך" מחייב, הן על microarrays זכוכית (שקופיות Codelink) באמצעות aptamers second fluorescently-tagged, ועל nanowires זהב derivatized עם aptamers second מצמידים את חלקיקי זהב 50 ננומטר (AuNPs; איור 3). בשני המקרים, וספציפיות מחייב היה גבוה: ב microarrays Codelink, לא מחייב כריך נצפתה עם aptamers אשר לא הראו כתוסף מחייב קביעת Kd. עם nanowires derivatized ראינו כמעט שום מחייב למקד שאינם חלבונים, מתחמי כריך בודדים ניתן לצפות באמצעות AuNPs aptamer מצמידים (איור 3).
2. חומרים
2.1. הדור של ספריית ה-DNA PF ו Reamplification של שברים Bound
פרטים מתוארים פאן קלוסון, 2009; פאן et al, 2008..
2.2. מטוהרים לבחירה חלבון בהתבסס
- 20 מ"מ טריס-HCl, pH 7.4
- 32 +, 30 + שבר ו-Fragment
- בחירת מאגר (2.5 מ"מ CaCl 2, 5 מ"מ MgCl 2 ב פוספט 1X בופר, pH 7.4, Gibco)
- Ni-agarose חרוזים נ.ת. ע (Qiagen)
- טור פוליפרופילן (Qiagen)
- מאגר הכבילה (50 mM Na 2HPO 4-לאא 2 PO 4, pH7.2, 150 mM NaCl)
- לידי ביטוי, מטוהרים חלבון S100B ב הצפת הכבילה (5 מיקרוגרם / μL)
- פנול: ChCl3: רשות העתיקות (pH 7.9, Ambion)
- 3 M NaAc, pH 5.2
- 100%, אתנול 70%
2.3. Topo שיבוט וניתוח רצף של ספריות נבחרות dsDNA
- בסיבוב ה 7 נבחר מוצרי ה-PCR (סיבובים נוספים ניתן לבצע כדי להמשיך אופטימיזציה של aptamer מחייב)
- pCR2.1-Topo וקטור (Invitrogen)
- DH5 רכיב תאים (Invitrogen)
- פלסמיד Mini Kit (Qiagen)
- M13 קדימה פריימר
- Informax של וקטור NTI (Invitrogen)
2.4. מאפייני עקידת Aptamers
- Aptamers ושליטה oligos (N 30 CC ו-N 30, IDT)
- Polynucleotide קינאז T4 (ניו אינגלנד Biolabs)
- γ-32-P-ATP (3000 Ci / mmol; 10 μCi / ml)
- מטוהרים S100B ב הצפת הכבילה (5 מיקרוגרם / μL)
2.5. כריך עם חלבון מבחני עקידת S100B מטוהרים Aptamers נבחרים
- 1 st aptamer היה מצמידים או א) Codelink שקופיות או microarray ב) nanowires Au.
- מטוהרים S100B חלבון, מדולל או על 0.125 מיקרומטר μl 70 עבור א) או 1 מיקרומטר עבור μl 50 ב).
- Nd 2 aptamer סומן או עם א) Alexafluor 546 עבור השקופיות מערך Codelink, או ב) מצמידים את AuNPs 50 ננומטר של מחקרים עם derivatized nanowires Au.
3. שיטות
3.1. הדור של PF-DNA הספרייה
שיטות מפורט הפרוטוקולים לבחירה aptamer ניתן למצוא פאן קלוסון, 2009; פאן et al, 2008..
3.2. Aptamer לבחירה באמצעות חלבון מטוהרים S100B
האדם S100 מחייב סידן חלבון B (S100B. ID Gene # 6285) שימש כמטרה. 6-tagged שלו חלבון S100B (98 חומצות אמינו אורך) באה לידי ביטוי ו מטוהרים באמצעות מערכת QIAexpressionist. (QIAGEN). אם תרצה או ציינו את שלו 6-התג ניתן להסיר enzymatically. במהלך סיבובי הבחירה, 1 מ מדגם μL כל צעד נשמרה עבור נצנץ נוזלי ספירה כדי לקבוע את היעילות מחייב הכוללת.
3.2.1. הכנת הספרייה קטעים PF-DNA
- Re-להשעות את + 32 ו - 30 + שברי ב μL 40 של 20 מ"מ טריס-HCl, pH 7.4.
- מחממים 3 דקות ב 85 מעלות צלזיוס, קריר עד 37 מעלות צלזיוס במשך 3 דקות באינקובטור של 37 ° C.
- הוסף μL 760 של חיץ הבחירה (2.5 מ"מ CaCl 2, 5 מ"מ MgCl 2 ב פוספט 1X בופר, pH 7.4, GIBCO) ו דגירה 3 דקות ב 37 מעלות צלזיוס, ולאחר מכן לשמור בטמפרטורת החדר (RT) במשך 10 דקות.
- לעבור עמודה הכלול Ni-agarose נ.ת. ע, חרוזים (QIAGEN) מראש נשטף עם חיץ הבחירה, ואז לשמור על RT עד לשימוש.
3.2.2. הכנת S100B Ni-agarose נ.ת. ע, ביד Bound ב RT
- ספין למטה 400 μL של Ni-נ.ת. ע חרוזים agarose במשך 3 שניות וזורקים supernatant.
- שטפו את החרוזים עם μL 400 של חיץ מחייבים (50 mM Na 2 HPO 4-לאא 2 ת.ד. 4, pH7.2, 150 mM NaCl) על ידי בעדינות pipetting 5 פעמים, ואז ספין למטה להתעלמות supernatant.
- חזור על שלב 2 פעמיים.
- הוספת 400 μL של מטוהרים S100B (5 מיקרוגרם / μL, תלוי חיץ מחייב) בעדינות פיפטה 5 פעמים בכל 3 דקות עבור סכום כולל של 15 דקות.
- ספין למטה להתעלמות supernatant.
- שטפו את S100B חרוז הנכנס עם μL 400 של חיץ מחייב ידי בעדינות pipetting 3 פעמים, ואז ספין למטה להתעלמות supernatant.
- חזור על שלב 6 פעמיים.
3.2.3. מבחר S100B הנכנס Aptamers
- + מעבירים את 32 ו - 30 +, שברי מן 3.2.1 כדי S100B חרוז הנכנס.
- דגירה במשך 15 דקות ומערבבים בעדינות כל 3 דקות על ידי בעדינות pipetting.
- שטפו את מורכבות S100B-DNA עם μL 800 של חיץ מחייב ידי בעדינות pipetting, אז ספין למטה להתעלמות supernatant.
- חזור על צעד 3 פעמיים.
3.2.4. שחזור של S100B-נבחרות Aptamers
- הוספת 200 μL 20 מ"מ טריס-HCl (pH7.4), 3 דקות על חום 85 מעלות, דקות 1 מערבולת ספין למטה, ולאחר מכן להעביר את supernatant לצינור טריים.
- חזור על שלב 1 פעם אחת, לשלב את supernates.
- לטהר את נבחרת ה-DNA כמו שברי צעדים לכל 9-12 מתוך פרסומים קודמים (11, 12).
3.2.5. Topo שיבוט וניתוח סידור עבור זיהוי רצפים aptamer הקונצנזוס
- Clone את הסיבוב ה 10 מוצרים נבחרים PCR לתוך pCR2.1-Topo וקטור (מ Invitrogen). נוסףשיבוט יכול להיעשות כמו בחירות נוספות מבוצעות.
- 40-50 רצף מושבות יחיד עם פריימר M13 קדימה (אנו משתמשים מתקן Core לגנטיקה מולקולרית בבית החולים הרשי מרכז).
- יישר את רצפי נבחרים באמצעות וקטור Informax של NTI (Invitrogen).
- קבע את רצפי הקונצנזוס מבוסס על מערכים.
- Aptamers הקונצנזוס נרכשו אז מ טכנולוגיות DNA משולב.
3.3. סנדוויץ מבחני עקידת עם זוגות של נבחרות Aptamers
3.3.1. פורמט microarray באמצעות שכותרתו fluorescently aptamers 2 nd.
- 1 הקונצנזוס Aptamers רח' היו מסונתז עם מחצית 5'-amineC6. הם היו תלויים חיץ הדפסה לריכוז סופי של 15 מיקרומטר ו הבחין על שקופיות Codelink הופעל (GE Healthcare / Amersham Biosciences) באמצעות תגליות Apogent MicroGrid Arrayer ב-DNA microarray מתקן PSU, אוניברסיטת Park), בעקבות פרוטוקולי Codelink מומלץ. כל שקופית הודפסה עם 12 מערכים.
- הכלאה בוצע 2 x 8 קלטת microarray הכלאה הפורמט (מערך זה, TeleChem הבינלאומי). קלטת microarray נחתם באמצעות nuclease ללא איטום דבק נייר (AlumaSeal השנייה, מחקר מוצרים הבינלאומי) כדי למנוע אידוי.
- חלבון S100B היה מדולל PBS לריכוז המתאים μl 70 ו להחיל את הבארות של הקלטת microarray. מחייב את aptamers מודפס בשקופית Codelink היה במשך שעה 1 בטמפרטורת החדר. בבארות של קלטת היו אז שטף בנפרד 3X עם PBS + 5 mM MgCl 2 (PBSM), וכן חוצץ לשטוף עודף נמחק. Aptamers שכותרתו fluorescently היו מדולל PBSM ל 0.125 מיקרומטר μl 70, ולאחר מכן מחומם ל 85 מעלות צלזיוס במשך 3 דקות ואחריו 3 דקות על הקרח. Aptamers יושמו הבארות של הקלטת microarray, וטופחו במשך שעה אחת בטמפרטורת החדר. וולס היו שוב שטף בנפרד 3X עם PBSM. הקלטת צולמה בנפרד ואז, המודעה את השקופית כולה ושטף PBSM. השקופית אז מיובש לחלוטין על ידי צנטריפוגה, וסקר באמצעות סריקה Packard Biosciences ScanArray 4000XL (פרקין אלמר).
3.3.2. פורמט Derivatized Nanowire באמצעות 2 nd aptamers מצמידים את AuNPs 50 ננומטר
זהב NWS (~ 5 מיקרומטר אורך, 320 ננומטר בקוטר) היו מסונתז על ידי electrodeposition galvanostatic בתוך ממברנות אלומינה נקבובי הבאים הפרוטוקולים שפורסמו בעבר (13, 14). עם פירוקה של קרום נקבובי, nanowires היו resuspended באתנול 1 מ"ל.
כאמור, ה-DNA נרכש טכנולוגיות DNA משולב. חלקיקי זהב (50 ננומטר) נרכשו מ טד פלה. ה-DNA Thiolated היה ביקע עם DTT 100 מ"מ 0.1 pH 8.3 סודיום פוספט M במשך שעה אחת ואחר כך מטוהרים בעמודה 10 Centri ספין.
Aptamer המצורף NWS ו NPS זהב
Aliquot 50 μL של nanowires Au הושם לתוך צינור 0.5 שאינו מקל מ"ל צנטריפוגות ושטף למאגר 10 mM פוספט, 300 mM NaCl, pH 7.4. ה-DNA thiolated ב 'בסוף (עם spacer 10 T ב 5' 5 בסוף) נוספה בריכוז סופי של 0.4 מיקרומטר על חוטים. המדגם vortexed למשך 30 דקות ושטף מכן על ידי צנטריפוגה (8,100 גרם) שלוש פעמים עם חיץ 10 mM פוספט, 300 mM NaCl, pH 7.4 ושלוש פעמים עם חיץ פוספט 50 מ"מ, 5 מ"מ MgCl 2, pH 7.2. חוטי ה-DNA מצופה היו resuspended במאגר 100 μL לדלל בחצי.
ה-DNA derivatized AuNPs הוכנו על ידי הוספת 50 μl 100 mM 2 nd aptamer (עם spacer של 10 של T בסוף 5'-) ל 1 מ"ל AuNPs 50 ננומטר מחומם על 37 מעלות צלזיוס במשך שעה אחת. לאחר החימום, 10 mM נתרן זרחתי חיץ, NaCl 1M, pH 7.4 נוספה (25 μL שתי פעמים, ואחריו μL 100, 150 μL, ו 128 μL) ב 0.5 מרווחי שעה. Conjugates נותרו על בלוק 37 מעלות צלזיוס חום לילה לפני השימוש. דוגמאות היו שטופים 3 פעמים עם חיץ פוספט 50 מ"מ, 5 מ"מ MgCl 2, pH 7.2. Conjugates היו resuspended במאגר 120 μL.
סנדוויץ הכלאה על NWS
חוטי ה-DNA מצופה, 1 μL בדילול מלא, נוספו למאגר ב צינורות PCR. חלבון S100B HtrA1 או חלבון בקרה (1 מיקרוגרם / μL) נוספה ריכוז סופי של 1 מיקרומטר חיץ 50 μL (~ 10-12 מולקולות חלבון נוסף לכל ננומטר מרובע על פני nanowire Au). דוגמאות היו vortexed עבור ~ 2 שעות. חוטים היו שטופים על ידי צנטריפוגה (8,100 גרם) שלוש פעמים עם חיץ פוספט 50 מ"מ, 5 מ"מ MgCl 2, pH 7.2 והיו כל resuspended ב 20 AuNP / DNA conjugates μL. החוטים היו vortexed ב conjugates עבור שעה 2, ואחר כך היו שטופים 5x ידי צנטריפוגה (1,300 גרם) עם חיץ פוספט 50 מ"מ, 5 מ"מ MgCl 2, pH 7.2 כדי להסיר את חלקיקי עודף. הדגימות resuspended ב20 חיץ μL מיובשים על פרוסות Au סי מצופה לניתוח FE-SEM.
FE-SEM תמונות של nanowires הושגו באמצעות פליטה 1530 ליאו שדה סריקת אלקטרונים מיקרוסקופ באמצעות מקור פליטת שדה שוטקי אלקטרונים בבית מתח 5.00 ההפעלה kV.
4. נציג תוצאות
באיור 1. פריימר 5'-Free (PF 1) ו-Free פריימר (PF 2) הפרוטוקולים.
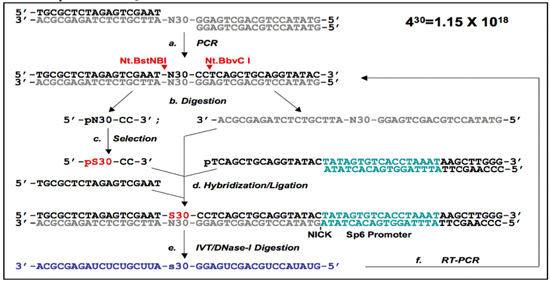
(א) 1 PF-DNA aptamer פרוטוקול הבחירה. Oligonucleotides הספרייה הם annealed יחד, נתון הגברה PCR להניב ספריה פעמיים גדילי דנ"א (א). 5'-end ללא פריימר (PF 1) באזור אקראי מן חד גדילי ספריות PF-DNA הוא הוכן על ידי Nt.BstNBI / Nt.BbvCI digestions (אתרי מחשוף מסומנים ראשי חץ אדום) ג'ל purifications, ואת עצמי גשר (שחור חץ) מבודד על ידי טיהור ג'ל (ב). לאחר הבחירה (ג), רצפים הנבחר (המיועד pS30-CC) הם הכלאה עם oligomers המקביל שלהם העצמי גשר ligated כדי ליצור מחדש את האזורים פריימר הוסר בעבר. בשלב זה, primers מוצגים המכילים מקדם SP6 בסוף 3'-(ד). זה משמש אז לתמלל RNA המכיל את אזורים נבחרים (ה). לבסוף, RNA הוא אז באופן בלעדי מחדש מוגבר (ה-DNA תבנית מתעכל) באמצעות RT-PCR (ו), ואת נבחרת תת ספרייה מוכן לסיבוב הבא של הבחירה.
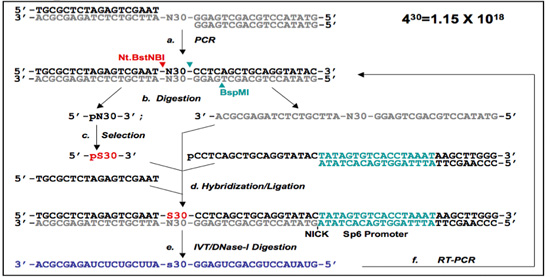
(ב) 2 PF-DNA aptamer פרוטוקול הבחירה. Oligonucleotides הספרייה הם annealed יחד נתון הגברה PCR להניב ספריה פעמיים גדילי דנ"א (א). האזור פריימר חינם (PF 2) אקראי מספריית חד גדילי PF-DNA הוא הוכן על ידי Nt.BstNBI / BspMI digestions (אתרי לחתוך מסומנים עם ראשי חץ) וטיהור ג'ל. העצמי גשר מבודד או מתוך פרוטוקול 1 PF ו / או על ידי עיכול יחיד של הספרייה החל Nt.BstNBI (ב), יחד עם טיהור ג'ל. לאחר הבחירה (ג), רצפים הנבחר (המיועד pS30) הם הכלאה עם oligomers המקביל שלהם העצמי ברידג', ligated להתחדש האזורים פריימר הוסר בעבר. כמו PF 1, primers מוצגים המכילים מקדם SP6 בסוף-3'(ד). זה משמש אז לתמלל RNA המכיל את אזורים נבחרים (ה). RNA היא אז reamplified בלעדי באמצעות RT-PCR (ו), ואת נבחרת תת ספרייה מוכן לסיבוב הבא של הבחירה.
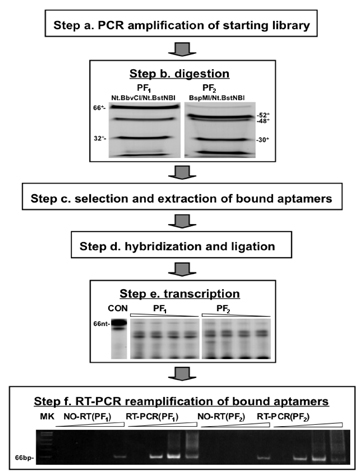
איור 2. ייצוג סכמטי ואת תוצאות הניסוי / הפחתה לתרגל של פרוטוקול הבחירה PF. צעדים המיועדים מתאימות אלה מתואר באיור 1. לאחר העיכול של הספרייה PF נבנה על ידי PCR (שלב א) עם endonucleases הגבלה (כפי שצוין), מוצרי מתעכל הופרדו על ידי ג'ל על 10% בתנאים denaturing. שברי המקביל שהושג עם PF PF 1 ו 2 פרוטוקולים מבחר מוצגים (שלב ב). לאחר הבחירה (שלב ג), aptamers כבול הם הכלאה עם oligomers המקביל שלהם העצמי ברידג', ligated להתחדש האזורים פריימר הוסר בעבר. בסוף-3', primers מוצגים המכילים מקדם SP6 בסוף-3'(שלב ד). RNAs P-32 שכותרתו תועתקו שימוש במוצרים ligated של 1, 0.5, 0.25, 0.125 מ μl 5 תגובות μl, והיו מופרדים על ידי עמוד ג'לים 6% בתנאים denaturing (ה שלב). תמלילי טופלו אז עם DNase כדי להסיר את האזורים שלא נבחרו באופן אקראי דנ"א, עיבד הפוך לתוך cDNA ו reamplified ואז מחזורים 7, 14, 21 או 28 PCR. היו מוצרים נפרדים על ידי ג'לים 8% מתחת אי denaturing תנאים. קטע 66 nt מייצג את המוצר באורך מלא המכיל את נבחרת pS30-CC ו pS30 רצפים כמו מחדש מוטבע בתוך רצף 5 'ו 3' איגוף. הגירה של סמנים PhiX174/HinfI הוא מופיע בצד שמאל (שלב ו). בקרת כללו השמטה של הצעד ההפוך שעתוק (RT). כמות קטנה של מוצר בדגימות כאלה ניתן היה לראות את המספרים מחזור גבוה, זה כנראה ניתן לחסל לחלוטין עם הצעד השני DNase (או ממושך) העיכול.
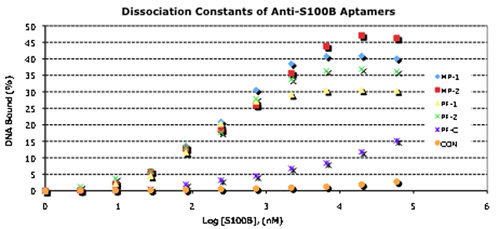
איור 3. מאפייני עקידת Aptamers נבחרים השתמש מותאמים שלהם בפורמט סנדוויץ'.
ריכוז תלויי א 32 P-aptamer מחייב קביעת Kd. Aptamers היו בסוף 5'-שכותרתו באמצעות g-32-P-ATP (3000Ci/mmol, ~ 10 MCI) ו - קינאז Polynucleotide T4 (ניו אינגלנד Biolabs), ו - ווא מחייבזה נקבע כאמור.
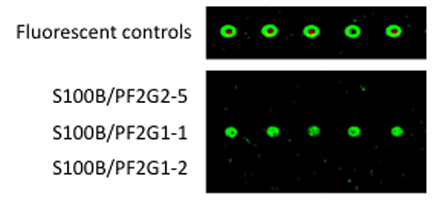
ב 5'-אמין, derivatized 1 aptamers רח' היו מצמידים את Codelink microarrays. חלבונים מטוהרים S100B היה מאוגדים 1 st aptamer, מגלשות היו שטופים היטב, ואת AlexaFluor546 שכותרתו 2 nd aptamer היה מאוגדים 1 st aptamer: מתחמי S100B. לאחר השטיפה דרך, הקרינה היה לכמת באמצעות סורק ScanArray.

ג 5'-תיאול derivatized 1 aptamers רח' היו מצמידים את nanowires Au באמצעות כימיה תיאול סטנדרטי. 2 nd aptamers היו מצמידים את AuNPs 50 ננומטר באותו אופן. מטוהרים חלבון S100B (משמאל) או מטוהרים חלבון HtrA1 השליטה (מימין) היה כרוך אז לראשונה את nanowires derivatized. לאחר שטיפה יסודית, 2 nd aptamer-50 ננומטר AuNPs היו כרוכים בהמשך ל 1 st-aptamer-nanowire: מתחמי S100B. לאחר השטיפה דרך, מתחמי כריך מחויב היו דמיינו באמצעות פליטת שדה מיקרוסקופיית אלקטרונים. ברים סולם = 1 מיקרומטר.
Disclosures
Acknowledgements
אנו מודים קרייג פול במתקן Core microarray עזרתו. עבודה זו נתמכה על ידי NIH / NCI מענק # CA118591 מהתוכנית IMAT. SLD גם מודה שטח פנסילבניה גרנט אחוות Consortium לתמיכה כספית. פרסום זו נתמכה גם על ידי אוניברסיטת פנסילבניה חומרים מכון המחקר ננו ייצור רשת הקרן הלאומית למדע הסכם שיתופית מס '0335765, התשתיות הלאומיות רשת ננוטכנולוגיה, עם אוניברסיטת קורנל.
Materials
| Name | Company | Catalog Number | Comments |
| Tris-HCl | |||
| HotMaster TAQ DNA Polymerase | 5 PRIME | ||
| dNTP Mix | Sigma-Aldrich | ||
| Acrylamide | Fisher Scientific | ||
| UltraPure 10X TBE Buffer | Invitrogen | ||
| Ammonium Persulfate | Bio-Rad | ||
| TEMED | Fisher Scientific | ||
| PhiX174 DNA/Hinf I DNA Marker | Promega Corp. | ||
| Ethidium Bromide | |||
| α-32P-dCTP | |||
| Phenol:ChCl3:IAA | Ambion | ||
| NaCl | |||
| Ethanol | |||
| Restriction Enzymes | New England Biolabs | ||
| Urea | Fisher Scientific | ||
| Photographic Film | ECE Scientific | ||
| PBS | GIBCO, by Life Technologies | ||
| CaCl2 | GIBCO, by Life Technologies | ||
| MgCl2 | GIBCO, by Life Technologies | ||
| Ni-NTA Agarose Beads | Qiagen | ||
| Polypropylene Column | Qiagen | ||
| NaHPO4 | |||
| NaAc | |||
| Detroit-551 cells | |||
| SK-MEL-31 cells | |||
| MEM | |||
| TrypLE Express | GIBCO, by Life Technologies | ||
| T4 DNA Ligase | New England Biolabs | ||
| Dimethyl Sulfoxide | Promega Corp. | ||
| Sp6 RNA Polymerase | Promega Corp. | ||
| RNase-Free DNase I | Promega Corp. | ||
| RNase Inhibitor | Promega Corp. | ||
| SensiScript Reverse Transcriptase | Qiagen | ||
| pCR-2.1-TOPO Vector | Invitrogen | ||
| DH5α Compotent Cells | Invitrogen | ||
| Plasmid Mini Kit | Qiagen | ||
| Vector NTI | Invitrogen | ||
| T4 Polynucleotide Kinase | New England Biolabs | ||
| γ-32P-ATP |
References
- Kulbachinskiy, A. V. Methods for selection of aptamers to protein targets. Biochemistry (Moscow). 72, 1505-1518 (2006).
- Mairal, T. Aptamers: molecular tools for analytical applications. Anal Bioanal Chem. 390, 989-1007 (2008).
- Stoltenburg, R., Reinemann, C., Strehlitz, B. SELEX -- A (r)evolutionary method to generate high-affinity nucleic acid ligands. Biomolec Engineering. 24, 381-403 (2007).
- Que-Gewirth, N. S., Sullenger, B. A. Gene therapy progress and prospects: RNA aptamers. Gene Ther. 14, 283-2891 (2007).
- Cowperthwaite, M. C., Ellington, A. D. Bioinformatic analysis of the contribution of primer sequences to aptamer structures. J Mol Evol. 67, 95-102 (2008).
- Shtatland, T., Gill, S. C., Javornik, B. E. Interactions of Escherichia coli RNA with bacteriophage MS2 coat protein: genomic SELEX. Nucleic Acids Res. 28, E93-E93 (2000).
- Jarosch, F., Buchner, K., Klussmann, S. In vitro selection using a dual RNA library that allows primerless selection. Nucleic Acids Res. 34, E86-E86 (2006).
- Vater, A., Jarosch, F., Buchner, K., Klussmann, S. Short bioactive Spiegelmers to migraine-associated calcitonin gene-related peptide rapidly identified by a novel approach: tailored-SELEX. Nucleic Acids Res. 31, E130-E130 (2003).
- Wen, J. D., Gray, D. M. Selection of genomic sequences that bind tightly to Ff gene 5 protein: primer-free genomic SELEX. Nucleic Acids Res. 32, E182-E182 (2004).
- Musheev, M. U., Krylov, S. N. Selection of aptamers by systematic evolution of ligands by exponential enrichment: addressing the polymerase chain reaction issue. Anal Chim Acta. 564, 91-916 (2006).
- Pan, W. H., Clawson, G. A. Primer-free aptamer selection using a random DNA library. Methods Mol Biol. , (2009).
- Pan, W., Xin, P., Clawson, G. A. Minimal-Primer and Primer-Free SELEX Protocols for Selection of Aptamers from Random DNA Libraries. BioTechniques. 44, 351-360 (2008).
- Nicewarner-Pena, S., Freeman, G., Reiss, B. Submicrometer metallic barcodes. Science. 294, 137-141 (2001).
- JC, H. u. l. t. e. e. n., CR, M. a. r. t. i. n. A general template-based method for the preparation of nanomaterials. J Materials Chem. 7, 1075-1087 (1997).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved