Method Article
Primer-Свободное выделение аптамер Использование случайных библиотеки ДНК
В этой статье
Резюме
SELEX протоколы составляют несколько раундов отбора, каждый из которых требует регенерации связанных лигандов, которые, в свою очередь, потребует фиксированные последовательности праймера фланговые случайных регионах библиотеки. Эти фиксированные последовательности праймера может вмешиваться в процесс отбора (ложных тревог). Здесь мы представляем грунтовка без протокола.
Аннотация
Aptamers are highly structured oligonucleotides (DNA or RNA) that can bind to targets with affinities comparable to antibodies 1. They are identified through an in vitro selection process called Systematic Evolution of Ligands by EXponential enrichment (SELEX) to recognize a wide variety of targets, from small molecules to proteins and other macromolecules 2-4. Aptamers have properties that are well suited for in vivo diagnostic and/or therapeutic applications: Besides good specificity and affinity, they are easily synthesized, survive more rigorous processing conditions, they are poorly immunogenic, and their relatively small size can result in facile penetration of tissues.
Aptamers that are identified through the standard SELEX process usually comprise ~80 nucleotides (nt), since they are typically selected from nucleic acid libraries with ~40 nt long randomized regions plus fixed primer sites of ~20 nt on each side. The fixed primer sequences thus can comprise nearly ~50% of the library sequences, and therefore may positively or negatively compromise identification of aptamers in the selection process 3, although bioinformatics approaches suggest that the fixed sequences do not contribute significantly to aptamer structure after selection 5. To address these potential problems, primer sequences have been blocked by complementary oligonucleotides or switched to different sequences midway during the rounds of SELEX 6, or they have been trimmed to 6-9 nt 7, 8. Wen and Gray 9 designed a primer-free genomic SELEX method, in which the primer sequences were completely removed from the library before selection and were then regenerated to allow amplification of the selected genomic fragments. However, to employ the technique, a unique genomic library has to be constructed, which possesses limited diversity, and regeneration after rounds of selection relies on a linear reamplification step. Alternatively, efforts to circumvent problems caused by fixed primer sequences using high efficiency partitioning are met with problems regarding PCR amplification 10.
We have developed a primer-free (PF) selection method that significantly simplifies SELEX procedures and effectively eliminates primer-interference problems 11, 12. The protocols work in a straightforward manner. The central random region of the library is purified without extraneous flanking sequences and is bound to a suitable target (for example to a purified protein or complex mixtures such as cell lines). Then the bound sequences are obtained, reunited with flanking sequences, and re-amplified to generate selected sub-libraries. As an example, here we selected aptamers to S100B, a protein marker for melanoma. Binding assays showed Kd s in the 10-7 - 10-8 M range after a few rounds of selection, and we demonstrate that the aptamers function effectively in a sandwich binding format.
протокол
1. Краткое описание Primer-Free протоколы выбора
Двухцепочечной ДНК библиотеки было построено с использованием ПЦР с соответствующими олигонуклеотидов (рисунок 1 и 2, Step), которые содержат центральную случайной области от 30 п, в окружении двух регионах грунт. Два незначительно различающихся "грунт-свободные" (PF) протоколы были разработаны. В библиотеке двухцепочечной ДНК, 5'-область содержит эндонуклеазы "уменьшение поперечного сечения" сайт для эндонуклеазы Nt.BstNBI, это фермент узнает двухцепочечной ДНК, но расщепляет только одной нити ДНК субстрата. 3'-область двухцепочечной ДНК библиотека содержит еще один "уменьшение поперечного сечения" сайта, для эндонуклеазы Nt.BbvCI, что также признает двухцепочечной ДНК, но расщепляет только одной нити, оставляя СС в 3'(PF 1), а также BspMI рестриктазы сайт, который расщепляет обе нити не оставляя никаких дополнительных 3 'нуклеотиды (PF 2). Как правило, мы используем PF 1 протокол изначально (потому что его легче отделить фрагментов в процессе отбора, см. ниже), а затем использовать PF 2 протокол для последующих раундах отбора.
32 П 5'-pN30-CC-3 'фрагмента (назначенный 32 +-фрагмент) и 30 нт 5'-pN30-3' фрагмента (назначенный 30 +-фрагмент) были, соответственно, порожденные Nt.BbvCI / NT . BstNBI или BspMI / Nt.BstNBI расщепления библиотеку двухцепочечной ДНК, и гель для очистки. 32 +-фрагмент содержит 30 нт случайной области, с CC фланговые последовательности на 3'-конце (PF 1). 30 +-фрагмент (PF 2) состоит только 30 п случайной последовательности домена. "Само-мост» 66 - фрагмент (содержащий случайные N30 региона с 5 'и 3' фланговые последовательности) был получен гель очистки (Nt.BbvCI или Nt.BstNBI льготы только верхние "+" нить). Это само-мост получается непосредственно в ПФ 1 протокол, или могут быть созданы и изолированных после резки библиотеки ДНК только с NtBbv.CI или Nt.BstNBI для PF 2 протокола (рис. 1 и 2, пункт Б). 32 + - или 32 +-фрагменты инкубировали с очищенных белков или культивируемых клеток меланомы, чтобы фрагменты связываются с белками или клетки, а затем несвязанных фрагментов были смыты (рис. 1 и 2, шаг с). Связаны или выбранных фрагментов были использованы для повторной генерации грунтовки регионах.
В гибридизации / лигирования реакции, самостоятельного моста был подготовлен и очищенный в то же время, как 32 + - и 30 +-фрагмент очистки; 5'-концу праймера был таким же, как строительство библиотеки и 3'-концу праймера были заменены с соответствующими праймерами, которые также содержатся дополнительные Sp6 транскрипции промоутер на 3'-конце (рис. 1 и 2, Шаг г). Продукты гибридизации / лигирования реакции были затем использованы для экстракорпорального РНК транскрипции (рис. 1 и 2, Шаг е). После транскрипции РНК, ДНК лигируют (включая невыбранные самостоятельного моста фрагмент ДНК) были переваривается ДНКазы я удалить вмешательства последовательности фоне. Обратной транскрипции (RT)-ПЦР данные показали, что грунт-регенерировать продукты эффективно реамплифицировали, составляющих "круглый" отбора (рис. 1 и 2, Шаг е). Фоне усиления показано при больших числах цикла в не-RT контроль ПЦР могут быть полностью удалены дополнительные пищеварения ДНКазы I, и не прощупывается после нижнем цикле числа, которые мы регулярно использовать.
После 7 туров отбора, аптамеры были характерны для привязки свойств. Kd были всего в 10 -7 10 -8 М диапазоне (рис. 3). В обязательные анализы, различные пары аптамеры показал добавка обязательными, указывая, что они ориентированы на различные сайты на S100B белка. Поэтому мы протестированы пар аптамеры "сэндвич" обязательные анализы, как на стекле микрочипов (Codelink слайдов) с помощью флуоресцентной меткой-вторых аптамеры, а также на производные нанопроводов золота со вторым аптамеры связан с 50 нм наночастиц золота (AuNPs; рис. 3). В обоих случаях специфичность связывания была высокой: на Codelink микрочипы, не бутерброд обязательным наблюдалось с аптамеры, которые не показывают добавка обязательными в Kd определений. С производные нанопроводов мы наблюдали практически без привязки к нецелевых белков, и отдельные комплексы сэндвич можно наблюдать через аптамер связью AuNPs (рис. 3).
2. Материалы
2.1. Генерация PF библиотеки ДНК и Reamplification связанных Фрагменты
Подробности описаны в Пан и Клоусон, 2009; Пан и др., 2008..
2.2. Очищенного белка Отбор
- 20 мМ Трис-HCl, рН 7,4
- 32 +-фрагмент и 30 +-Фрагмент
- Выбор буфера (2,5 мМ CaCl 2, 5 мМ MgCl 2 в 1X фосфатно-солевым буфером, рН 7,4, Gibco)
- Ni-NTA агарозы бусы (Qiagen)
- Полипропилен Колонка (Qiagen)
- Связывание буфера (50 мМ Na 2HPO 4-NaH 2 4 PO, pH7.2, 150 мМ NaCl)
- Выраженный, очищенная S100B белка в связывании буфера (5 мкг / мкл)
- Фенол: CHCl3: IAA (рН 7,9, Амбион)
- 3 М NaAc, рН 5,2
- 100%, и 70% этанола
2.3. TOPO клонирование и секвенирование Анализ Выбранный Библиотеки двухцепочечной ДНК
- 7-го тура выбран продуктов ПЦР (дополнительные раунды могут быть выполнены, чтобы продолжить оптимизацию аптамер переплет)
- pCR2.1-TOPO Vector (Invitrogen)
- DH5 компонентов клетки (Invitrogen)
- Плазмиды Mini Kit (Qiagen)
- M13 прямого праймера
- Informax в Vector NTI (Invitrogen)
2.4. Связывание Характеристики Аптамеры
- Аптамеры и контроля олиго (N 30 CC и N 30, IDT)
- Т4 полинуклеотидкиназы (Новая Англия Биолабс)
- γ-32 Р-АТФ (3000 Ки / ммоль, 10 мкКи / мл)
- Очищенная S100B в связывающем буфере (5 мкг / мкл)
2.5. Сэндвич Связывание Анализы с очищенного белка S100B и отдельных Аптамеры
- 1-й аптамер был связан либо а) Codelink микрочипов слайдов или б) Аи нанопроводов.
- Очищенная S100B белка, разбавляют до или 0,125 мкМ в 70 мкл для) или 1 мкМ в 50 мкл для б).
- 2-й аптамер был либо помечены a.) Alexafluor 546 для слайдов Codelink массив, или б) в сочетании с 50 AuNPs нм для исследования с производное нанопроводов Au.
3. Методы
3.1. Генерация PF ДНК библиотеки
Подробные методы и протоколы аптамер подборку можно найти в Пан и Клоусон, 2009; Пан и др., 2008..
3.2. Аптамер Выбор с помощью очищенного белка S100B
Человек S100 кальций связывающий белок B (S100B. Гена ID # 6285) был использован в качестве мишени. Его 6-меткой S100B белка (98 аминокислот в длину) была высказана и очищенная с помощью QIAexpressionist системы. (QIAGEN). При желании или не указано, его 6-теги могут быть удалены ферментативно. Во время раунда отбора, 1 мкл пробы из каждого шага была спасена для жидкого сцинтилляционного счета, чтобы определить общие обязательные эффективности.
3.2.1. Подготовка PF-библиотека фрагментов ДНК
- Повторное приостановить 32 + и 30 + фрагментов в 40 мкл 20 мМ Трис-HCl, рН 7,4.
- Нагрейте 3 мин при 85 ° С, и охлаждают до 37 ° С в течение 3 мин в инкубатор на 37 ° C.
- Добавить 760 мкл буфера выбора (2,5 мМ CaCl 2, 5 мМ MgCl 2 в 1X фосфатно-солевым буфером, рН 7,4, GIBCO) и инкубировать в течение 3 мин при 37 ° С, то держать при комнатной температуре (RT) в течение 10 мин.
- Пройдите через колонку, содержащихся Ni-NTA агарозы-бусы (QIAGEN) предварительно промывают выбора буфера, а затем сохранить при комнатной температуре до использования.
3.2.2. Подготовка Ni-NTA агарозы-Bead Связанные S100B при комнатной температуре
- Спином вниз 400 мкл Ni-NTA агарозы бисером на 3 секунды и отбросить супернатант.
- Вымойте бусин с 400 мкл буфера для связывания (50 мМ Na 2 HPO 4-NaH 2 4 PO, pH7.2, 150 мМ NaCl), аккуратно пипеткой 5 раз, то спином вниз и отбросить супернатант.
- Повторите шаг 2 в два раза.
- Добавить 400 мкл очищенных S100B (5 мкг / мкл, взвешенных в связывающем буфере) и осторожно пипеткой 5 раз каждые 3 мин в течение всего 15 мин.
- Спином вниз и отбросить супернатант.
- Вымойте бусинка-S100B связан с 400 мкл буфера для связывания, осторожно пипетки 3 раза, то спином вниз и отбросить супернатант.
- Повторите шаг 6 в два раза.
3.2.3. Выбор S100B связанного Аптамеры
- Передача 32 + - и 30 +-фрагменты из 3.2.1, чтобы шарик-S100B связаны.
- Инкубируйте в течение 15 мин и аккуратно перемешать каждые 3 мин, осторожно пипеткой.
- Вымойте S100B-комплекса ДНК с 800 мкл буфера для связывания, осторожно пипеткой, то спином вниз и отбросить супернатант.
- Повторите шаг 3, в два раза.
3.2.4. Восстановление S100B-Выбранный Аптамеры
- Добавить 200 мкл 20 мМ Трис-HCl (pH7.4), тепло 3 мин при 85 ° C, вихревые 1 мин и со спином вниз, а затем перенести супернатант в новую пробирку.
- Повторите шаг 1 раз, и объединить supernates.
- Purify выбранный ДНК-фрагментов в соответствии с шагами 9-12 из предыдущих публикаций (11, 12).
3.2.5. TOPO клонирование и секвенирование анализ для идентификации последовательностей консенсуса аптамер
- Клон 10-го тура выбран продуктов ПЦР в pCR2.1-TOPO вектор (от Invitrogen). ДополнительныйКлонирование может быть сделано как дополнительный выбор выполняются.
- Последовательность 40-50 одной колонии с M13 прямого праймера (мы используем молекулярной генетики Основной фонд в Hershey Medical Center).
- Выравнивание выбранных последовательностей с помощью Informax в Vector NTI (Invitrogen).
- Определите консенсуса последовательностей на основе выравнивания.
- Консенсус аптамеры затем были приобретены у интегрированные технологии ДНК.
3.3. Сэндвич Связывание с Анализы Пары Выбранный Аптамеры
3.3.1. Microarray формате с использованием флуоресцентно помеченных 2-й аптамеры.
- Консенсус 1-й Аптамеры были синтезированы с 5'-amineC6 части. Они были приостановлены в печать буфера конечной концентрации 15 мкМ и с пятнами на Codelink Слайды активированный (GE Healthcare / Amersham Biosciences) с использованием Apogent Открытия Microgrid Arrayer в ДНК-микрочипов объекта блока питания, University Park), после Codelink рекомендуется протоколов. Каждый слайд был напечатан с 12 массивов.
- Гибридизация проводилась в 2 х 8 формате Microarray Гибридизация кассеты (Array Это, TeleChem International). Кассета Microarray была решена использованием нуклеазы без клея уплотнения фольги (AlumaSeal II, научный Products International), чтобы предотвратить испарение.
- S100B белка растворяли в PBS до соответствующей концентрации в 70 мкл и наносили на скважинах кассету микрочипов. Привязка к аптамеры напечатаны на слайд Codelink был в течение 1 часа при комнатной температуре. Скважинах кассеты были тогда в отдельности промывается 3X с PBS + 5 мМ MgCl 2 (PBSM), а избыток промывочного буфера был уничтожены. Флуоресцентно меченных аптамеры разводили в PBSM до 0,125 мкМ в 70 мкл, а затем нагревают до 85 ° С в течение 3 минут, затем 3 минуты на льду. Аптамеры были применены к скважинам кассету микрочипов, и инкубировали в течение одного часа при комнатной температуре. Уэллс снова индивидуально мыть 3X с PBSM. Кассета была затем разобрали, объявление целом слайд промывали в PBSM. Слайд был тщательно просушить с помощью центрифугирования, и отсканированный с помощью сканирования Packard Biosciences ScanArray 4000XL (Perkin Elmer).
3.3.2. Производные формате нанопроволоки с использованием 2-й аптамеры связан с 50 нм AuNPs
Золото ННК (~ 5 мкм в длину, 320 нм в диаметре) были синтезированы гальваностатическом электроосаждения в пористого оксида алюминия в мембранах следующих ранее опубликованных протоколов (13, 14). После растворения пористых мембран, нанопроводов ресуспендировали в 1 мл этанола.
Как уже отмечалось, ДНК был приобретен у интегрированные технологии ДНК. Наночастицы золота (50 нм) были куплены у Теда Пелла. Тиолированного ДНК расщепляют 100 мМ DTT в 0,1 М фосфат натрия рН 8,3 в течение одного часа и затем очищается в Centri-спинового 10 столбцов.
Аптамер привязанность к Золотой ННК и национальных парков
50 мкл нанопроводов Au был помещен в 0,5 мл антипригарным центрифуге трубки и промывали в 10 мМ фосфатного буфера, 300 мМ NaCl, рН 7,4. ДНК тиолированного на 5 'конце (с 10 T Spacer на 5' конце) был добавлен в конечной концентрации 0,4 мкМ до проводов. Образец перемешивали в течение 30 минут, а затем промыть путем центрифугирования (8100 г) три раза с 10 мМ фосфатного буфера, 300 мМ NaCl, рН 7,4 и в три раза с 50 мМ фосфатного буфера, 5 мМ MgCl 2, рН 7,2. ДНК покрытием провода ресуспендировали в 100 мкл буфера для разбавления вдвое.
ДНК-производные AuNPs готовили добавлением 50 мкл 100 мМ 2-й аптамер (с сепаратором 10-Т на 5'-конец) до 1 мл 50 AuNPs нм и нагревают при 37 ° С в течение одного часа. После нагрева, 10 мМ натрий-фосфатного буфера, 1M NaCl, рН 7,4 был добавлен (25 мкл в два раза, со 100 мкл, 150 мкл и 128 мкл) в 0,5-часовыми интервалами. Конъюгаты были оставлены на 37 ° С тепла блока ночь перед использованием. Образцы были промыты 3 раза с 50 мМ фосфатного буфера, 5 мМ MgCl 2, рН 7,2. Конъюгаты ресуспендировали в 120 мкл буфера.
Сэндвич гибридизации на ННК
ДНК покрытием провода, 1 мкл разбавленного, были добавлены в буфер в ПЦР пробирок. S100B белка или HtrA1 контроль белка (1 мкг / мкл) было добавлено для конечной концентрации 1 мкМ в 50 мкл буфера (~ 10-12 молекул белка добавил на квадратный нанометр от поверхности Au нанопроволоки). Пробы перемешивали в течение ~ 2 часов. Провода промывали путем центрифугирования (8100 г) три раза с 50 мМ фосфатного буфера, 5 мМ MgCl 2, рН 7,2 и были друг ресуспендировали в 20 мкл AuNP / ДНК конъюгатов. Провода были встряхивали в конъюгатов в течение 2 часов, а затем промывали 5 раз путем центрифугирования (1300 г) с 50 мМ фосфатного буфера, 5 мМ MgCl 2, рН 7,2, чтобы удалить избыток наночастиц. Образцы ресуспендировали в20 мкл буфера и сушат на Au покрытие пластин кремния для FE-SEM анализа.
FE-SEM изображения нанопроводов были получены с использованием Лео 1530 по полевой эмиссии сканирующего электронного микроскопа использованием Шоттки автоэмиссионные источника электронов на рабочее напряжение 5,00 кВ.
4. Представитель Результаты
Рисунок 1. 5'-премьер-Free (PF 1) и Primer-Free (PF 2) протоколов.
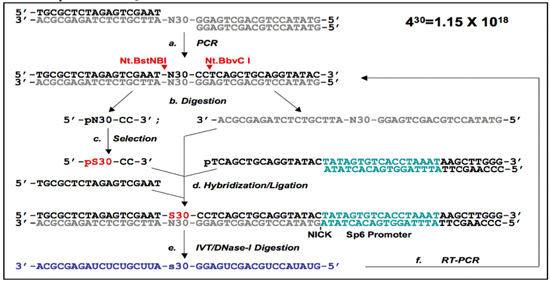
(А) PF 1 ДНК-аптамеров выбор протокола. Библиотека олигонуклеотиды отожженных вместе, и подвергают ПЦР для получения двухцепочечной ДНК библиотеки (). 5'-концу праймера, свободного (PF 1) случайные региона от PF одноцепочечных ДНК библиотеки подготовлена Nt.BstNBI / Nt.BbvCI пищеварения (расщепление сайтов отмечены красными стрелками) и гель-очищения, и само мост (черная стрелка) выделяют гель очистки (б). После выбора (с), подобранных последовательностей (назначенные pS30-CC) гибридизуются с соответствующими олигомеров и сам мост и лигировали перегенерировать ранее удалили регионах грунт. На данном этапе, грунтовки вводят которые содержат Sp6 промоутер на 3'-конце (г). , Который затем используется для транскрипции РНК-содержащих отдельных регионах (е). Наконец, РНК, то исключительно повторно усиливается (матричной ДНК переваривается) с использованием RT-PCR (F), и выбранный под-библиотеку готов к следующему раунду отбора.
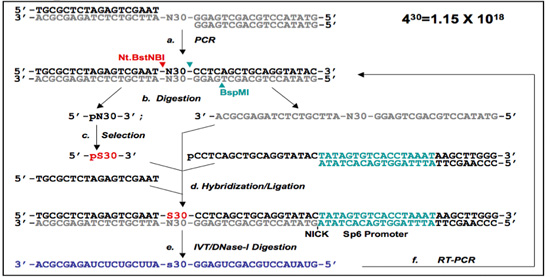
(Б) П. Ф. 2 ДНК-аптамеров выбор протокола. Библиотека олигонуклеотиды отожженных вместе и подвергнуты ПЦР для получения двухцепочечной ДНК библиотеки (). Грунтовка без (PF 2) случайные региона от PF одноцепочечных ДНК библиотеки подготовлена Nt.BstNBI / BspMI пищеварения (вырезать участки обозначены стрелками) и гель очистки. Самостоятельно мост изолирован ни от PF 1 протокол и / или с помощью одного переваривания начиная библиотека с Nt.BstNBI (б), в сочетании с гелем очистки. После выбора (с), подобранных последовательностей (назначенные pS30) гибридизуются с соответствующими олигомеров и сам мост, и лигируют для восстановления ранее удаленного регионах грунт. Как и в PF 1, грунтовки вводят которые содержат Sp6 промоутер на 3'-конце (г). , Который затем используется для транскрипции РНК-содержащих отдельных регионах (е). РНК, то исключительно реамплифицировали использованием RT-PCR (F), и выбранный под-библиотеку готов к следующему раунду отбора.
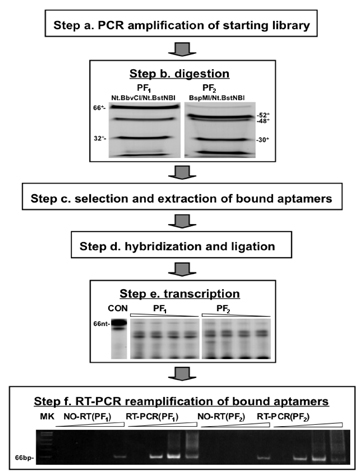
Рисунок 2. Схематическое изображение и экспериментальные результаты / снижение на практике PF протоколе отбора. Назначенные шаги соответствуют тем, которые изображены на рисунке 1. После переваривания PF библиотеки построено методом ПЦР (шаг) с рестриктаз (как указано), усваиваются продукты были разделены Страница на 10% гель в денатурирующих условиях. Соответствующие фрагменты, полученные с PF 1 и PF 2 протоколы отбора показано (стадия б). После выбора (Шаг с), связанный аптамеры гибридизуются с соответствующими олигомеров и сам мост, и лигируют к регенерации ранее удалили регионах грунт. На 3'-конце, грунтовки вводят которые содержат Sp6 промоутер на 3'-конце (шаг г). 32 Р-меченых РНК были расшифрованы использованием лигируют продукты 1, 0,5, 0,25, 0,125 мкл от 5 мкл реакции, и были разделенных Страница на 6% гелей в денатурирующих условиях (шаг д). Стенограммы были затем обрабатывают, чтобы удалить ДНКазы невыбранные случайных участков ДНК, обратной транскрипции в кДНК, а затем реамплифицировали на 7, 14, 21 или 28 циклов ПЦР. Продукты были разделены на страницу на 8% гели, не связанных с денатурирующих условиях. 66 п фрагмент представляет полнометражный продукт, содержащий выбранные pS30-CC и pS30 последовательностей повторно внедрены в 5 'и 3' фланговые последовательности. Миграция маркеры PhiX174/HinfI это показано на рисунке слева (Шаг е). Управление включены бездействие обратной транскрипции (RT) шаг. Небольшое количество продукта в таких образцах можно было увидеть на большом количестве циклов, это предположительно могут быть полностью устранены с секунду (или длительное) шаг пищеварения ДНКазы.
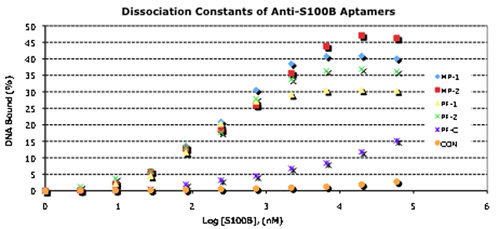
Рисунок 3. Связывание Характеристики Выбранный Аптамеры и их парные Использование в сэндвич Format.
А. зависит от концентрации 32 Р-аптамеров для связывания Kd Определение. Аптамеры были 5'-конце помечены использовании г-32 Р-АТФ (3000Ci/mmol, ~ 10 мКи) и Т4 полинуклеотидкиназы (Новая Англия Биолабс), и обязательным вас определяется как описано.
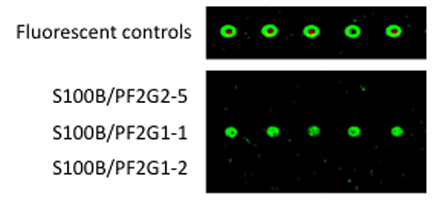
Б. 5'-амин-производного 1-й аптамеры были связаны с Codelink микрочипов. Очищенная S100B белок должен был 1-й аптамеров, слайды были промыты основательно, и AlexaFluor546 меченных 2-й аптамер должен был 1-й аптамер: S100B комплексов. После через полоскание, флуоресценция была количественно, используя ScanArray сканера.

С. 5'-тиол производные 1-го аптамеры были связаны с Au нанопроводов с использованием стандартных тиоловых химии. 2-й аптамеры были связаны с 50 нм AuNPs в том же порядке. Очищенная S100B белка (слева) или очищенные HtrA1 контроль белка (справа) был связан первый производные нанопроводов. После тщательной промывки, 2-й аптамер-50 AuNPs нм впоследствии были привязаны к 1-й-аптамеров-нанопроволоки: S100B комплексов. После через полоскание, связанные комплексы сэндвич были визуализированы использованием автоэмиссионных сканирующей электронной микроскопии. Шкала баров = 1 мкм.
Раскрытие информации
Благодарности
Мы благодарим Крейг Павла в фонд Microarray Основные его о помощи. Эта работа была поддержана NIH / NCI грант № CA118591 от ИСАТ программы. SLD также признает, штат Пенсильвания космических грантов консорциума стипендий за финансовую поддержку. Эта публикация была также поддержана Университета штата Пенсильвания Материалы научно-исследовательский институт Nano Изготовление сети и Национальный научный фонд Совместная соглашение № 0335765, Национальной нанотехнологической инфраструктуры сети, с Корнельского университета.
Материалы
| Name | Company | Catalog Number | Comments |
| Tris-HCl | |||
| HotMaster TAQ DNA Polymerase | 5 PRIME | ||
| dNTP Mix | Sigma-Aldrich | ||
| Acrylamide | Fisher Scientific | ||
| UltraPure 10X TBE Buffer | Invitrogen | ||
| Ammonium Persulfate | Bio-Rad | ||
| TEMED | Fisher Scientific | ||
| PhiX174 DNA/Hinf I DNA Marker | Promega Corp. | ||
| Ethidium Bromide | |||
| α-32P-dCTP | |||
| Phenol:ChCl3:IAA | Ambion | ||
| NaCl | |||
| Ethanol | |||
| Restriction Enzymes | New England Biolabs | ||
| Urea | Fisher Scientific | ||
| Photographic Film | ECE Scientific | ||
| PBS | GIBCO, by Life Technologies | ||
| CaCl2 | GIBCO, by Life Technologies | ||
| MgCl2 | GIBCO, by Life Technologies | ||
| Ni-NTA Agarose Beads | Qiagen | ||
| Polypropylene Column | Qiagen | ||
| NaHPO4 | |||
| NaAc | |||
| Detroit-551 cells | |||
| SK-MEL-31 cells | |||
| MEM | |||
| TrypLE Express | GIBCO, by Life Technologies | ||
| T4 DNA Ligase | New England Biolabs | ||
| Dimethyl Sulfoxide | Promega Corp. | ||
| Sp6 RNA Polymerase | Promega Corp. | ||
| RNase-Free DNase I | Promega Corp. | ||
| RNase Inhibitor | Promega Corp. | ||
| SensiScript Reverse Transcriptase | Qiagen | ||
| pCR-2.1-TOPO Vector | Invitrogen | ||
| DH5α Compotent Cells | Invitrogen | ||
| Plasmid Mini Kit | Qiagen | ||
| Vector NTI | Invitrogen | ||
| T4 Polynucleotide Kinase | New England Biolabs | ||
| γ-32P-ATP |
Ссылки
- Kulbachinskiy, A. V. Methods for selection of aptamers to protein targets. Biochemistry (Moscow). 72, 1505-1518 (2006).
- Mairal, T. Aptamers: molecular tools for analytical applications. Anal Bioanal Chem. 390, 989-1007 (2008).
- Stoltenburg, R., Reinemann, C., Strehlitz, B. SELEX -- A (r)evolutionary method to generate high-affinity nucleic acid ligands. Biomolec Engineering. 24, 381-403 (2007).
- Que-Gewirth, N. S., Sullenger, B. A. Gene therapy progress and prospects: RNA aptamers. Gene Ther. 14, 283-2891 (2007).
- Cowperthwaite, M. C., Ellington, A. D. Bioinformatic analysis of the contribution of primer sequences to aptamer structures. J Mol Evol. 67, 95-102 (2008).
- Shtatland, T., Gill, S. C., Javornik, B. E. Interactions of Escherichia coli RNA with bacteriophage MS2 coat protein: genomic SELEX. Nucleic Acids Res. 28, E93-E93 (2000).
- Jarosch, F., Buchner, K., Klussmann, S. In vitro selection using a dual RNA library that allows primerless selection. Nucleic Acids Res. 34, E86-E86 (2006).
- Vater, A., Jarosch, F., Buchner, K., Klussmann, S. Short bioactive Spiegelmers to migraine-associated calcitonin gene-related peptide rapidly identified by a novel approach: tailored-SELEX. Nucleic Acids Res. 31, E130-E130 (2003).
- Wen, J. D., Gray, D. M. Selection of genomic sequences that bind tightly to Ff gene 5 protein: primer-free genomic SELEX. Nucleic Acids Res. 32, E182-E182 (2004).
- Musheev, M. U., Krylov, S. N. Selection of aptamers by systematic evolution of ligands by exponential enrichment: addressing the polymerase chain reaction issue. Anal Chim Acta. 564, 91-916 (2006).
- Pan, W. H., Clawson, G. A. Primer-free aptamer selection using a random DNA library. Methods Mol Biol. , (2009).
- Pan, W., Xin, P., Clawson, G. A. Minimal-Primer and Primer-Free SELEX Protocols for Selection of Aptamers from Random DNA Libraries. BioTechniques. 44, 351-360 (2008).
- Nicewarner-Pena, S., Freeman, G., Reiss, B. Submicrometer metallic barcodes. Science. 294, 137-141 (2001).
- JC, H. u. l. t. e. e. n., CR, M. a. r. t. i. n. A general template-based method for the preparation of nanomaterials. J Materials Chem. 7, 1075-1087 (1997).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены