Method Article
Primer-Free Aptamer Selection mit einem zufälligen DNA-Bibliothek
In diesem Artikel
Zusammenfassung
SELEX-Protokolle umfassen mehrere Runden der Selektion, von denen jeder Regeneration von Liganden, die wiederum erfordern feste Primersequenzen flankierenden die zufällige Bibliothek Regionen erfordern. Diese festen Primersequenzen können mit der Auswahl (false positive und negative) stören. Hier präsentieren wir eine Grundierung-free-Protokoll.
Zusammenfassung
Aptamere sind hoch strukturierte Oligonukleotiden (DNA oder RNA), die Ziele mit vergleichbaren Affinitäten Antikörper 1 binden kann. Sie sind durch ein in-vitro-Selektion Prozess namens Systematische Evolution von Liganden durch EXponentielle Anreicherung (SELEX) für eine Vielzahl von Zielen zu erkennen, von kleinen Molekülen an Proteine und andere Makromoleküle 2-4 identifiziert. Aptamere haben Eigenschaften, die auch für in vivo diagnostische und / oder therapeutische Anwendungen geeignet sind: Neben einer guten Spezifität und Affinität, sie leicht synthetisiert werden, überleben strengere Verarbeitungsbedingungen, sie sind schwach immunogen und ihrer relativ geringen Größe können in einfache Penetration des Ergebnisses Gewebe.
Aptamere, die über die Standard-SELEX-Prozess identifiziert werden üblicherweise ~ 80 Nukleotiden (nt), da sie typischerweise von Nukleinsäure-Bibliotheken mit ~ 40 nt langen randomisierten Regionen plus feste Grundierung Websites von ~ 20 nt auf jeder Seite ausgewählt werden. Die feste Primersequenzen können somit fast umfassen ca. 50% der Bibliothek Sequenzen, und deshalb kann positiv oder negativ beeinträchtigen Identifizierung von Aptameren in der Auswahl 3, obwohl schlagen Bioinformatik Ansätze, die den festen Sequenzen nicht wesentlich dazu beitragen, Aptamer-Struktur nach Auswahl 5 . Um diese möglichen Probleme haben Primersequenzen von komplementären Oligonukleotiden blockiert oder ausgeschaltet, um unterschiedliche Sequenzen auf halbem Weg in die Runde der SELEX 6, oder sie haben auf 6-9 nt 7, 8 getrimmt. Wen und Gray 9 ausgebildet einem Primer-freie genomische SELEX-Verfahren, in denen die Primersequenzen waren völlig aus der Bibliothek vor der Auswahl entfernt und wurden dann regeneriert werden, um Verstärkung des ausgewählten genomischen Fragmenten zu ermöglichen. Doch die Technik zu beschäftigen, hat eine einzigartige Genbank konstruiert werden, bei dem sich nur die Vielfalt und die Regeneration nach dem Runden der Selektion beruht auf einer linearen Reamplifikation Schritt. Alternativ sind auch Bemühungen um die Probleme, die durch feste Primersequenzen mit hohem Wirkungsgrad Partitionierung verursacht umgehen mit Problemen in Bezug auf die PCR-Amplifikation 10 erfüllt.
Wir haben ein Primer-free (PF) Auswahl-Verfahren deutlich vereinfacht SELEX Verfahren und beseitigt effektiv Primer-Störungsprobleme 11, 12 entwickelt. Die Protokolle der Arbeit in einer einfachen Weise. Die zentrale random Region der Bibliothek ist ohne fremde flankierenden Sequenzen gereinigt und ist ein geeignetes Ziel (zum Beispiel, um ein gereinigtes Protein oder komplexen Gemischen wie Zell-Linien) gebunden. Dann werden die gebundenen Sequenzen erhalten werden, wieder mit flankierenden Sequenzen, und wieder verstärkt auf ausgewählte Sub-Bibliotheken zu generieren. Als ein Beispiel, hier sind wir selektierten Aptamere zu S100B, ein Protein Marker für Melanome. Binding Assays zeigte Kd s in den 10 -7 - 10 -8 M Reihe nach ein paar Runden der Selektion, und wir zeigen, dass die Aptamere effektiv funktionieren in einer Sandwich-Bindung Format.
Protokoll
1. Kurze Beschreibung der Primer-Freie Auswahl Protokolle
Ein doppelsträngige DNA-Bibliothek wurde unter Verwendung PCR mit den entsprechenden Oligonukleotiden (Abbildung 1 und 2, Schritt a), die eine zentrale random Domain von 30 nt enthalten, die von zwei Primer flankiert. Zwei etwas andere "Primer-free" (PF)-Protokolle entwickelt wurden. In der dsDNA-Bibliothek enthält die 5'-Region eine Endonuklease "nicking" Standort für die Endonuklease Nt.BstNBI; dieses Enzym erkennt dsDNA sondern spaltet nur einen Strang der DNA-Substrat. Die 3'-Region der dsDNA-Bibliothek enthält eine weitere "nicking" site, für die Endonuklease Nt.BbvCI, dass erkennt auch dsDNA sondern spaltet nur einem Strang, so dass eine CC am 3'-Ende (PF 1), sowie eine BspMI Endonuklease Restriktionsstelle, die beide Stränge so dass keine zusätzlichen 3 'Nukleotide (PF 2) spaltet. Wir verwenden im allgemeinen die PF-1-Protokoll zunächst (weil die leichter zu trennen Fragmente bei der Auswahl-Verfahren hergestellt, siehe unten), und dann setzen die PF-2-Protokoll für die nachfolgenden Runden der Selektion.
Die 32 nt der 5'-PN30-CC-3 'Fragment (benannt 32 +-Fragment) und die 30 nt der 5'-PN30-3-Fragment (benannt 30 +-Fragment) wurden jeweils durch Nt.BbvCI / Nt generiert . BstNBI oder BspMI / Nt.BstNBI Spaltung der dsDNA-Bibliothek, und das Gel-Reinigung. Die 32 +-Fragment enthält die 30 nt zufällige Domain, mit einer CC-flankierende Sequenz am 3'-Ende (PF 1). Die 30 +-Fragment (PF 2) besteht nur die 30 nt zufällige Domain-Sequenz. Die "self-Brücke" 66 - Fragment (mit den zufälligen N30 Region mit dem 5 'und 3' flankierenden Sequenzen) wurde durch Gel-Reinigung (Nt.BbvCI oder Nt.BstNBI schneidet nur der obere "+"-Strang) erhalten. Diese Selbst-Brücke ist direkt in der PF-1-Protokoll erhalten, oder können erzeugt und isoliert werden nach dem Schneiden der Bibliothek DNA mit nur NtBbv.CI oder Nt.BstNBI für den PF-2-Protokoll (Abbildung 1 und 2, Schritt b). Die 32 + - oder 32 +-Fragmente wurden mit dem gereinigten Proteine oder die kultivierten Melanomzellen inkubiert, damit die Fragmente binden an die Proteine oder die Zellen, und dann die ungebundenen Fragmente wurden entfernt (Abbildung 1 und 2, Schritt c) gewaschen. Die gebundene bzw. ausgewählte Fragmente wurden zur Wiederwahl Generation der Primer Regionen.
In der Hybridisierung / Ligationsreaktion wurde die Selbst-Brücke hergestellt und gereinigt zur gleichen Zeit wie die 32 + - und 30 +-Fragment Reinigung; 5'-Ende-Primer war dieselbe wie die Bibliothek Bau-und 3'-Ende Primer wurden ersetzt mit passenden Primern, enthielt auch eine zusätzliche Sp6 Transkriptionspromotor am 3'-Ende (Abbildung 1 und 2, Schritt d). Die Produkte der Hybridisierung / Ligationsreaktion wurden dann in vitro RNA-Transkription (Abbildung 1 und 2, Schritt e) verwendet. Nach der RNA-Transkription wurden die ligierten DNAs (einschließlich der nicht ausgewählten Selbst-Brücke DNA-Fragment) durch DNase verdaut ich störende Hintergrund-Sequenzen zu entfernen. Die reverse Transkription (RT)-PCR-Daten haben gezeigt, dass die Primer-regeneriert Produkte effizient waren reamplifiziert und eine "Runde" der Selektion (Abbildung 1 und 2, Schritt f). Der Hintergrund Verstärkung bei hohen Taktzahlen in der no-RT-PCR gezeigt Kontrolle kann vollständig durch eine zusätzliche DNase I-Verdau entfernt werden, und nicht nach Unterstufe Zahlen, die wir bisher routinemäßig eingesetzt nachweisbar.
Nach 7 Runden der Selektion wurden Aptamere für die Bindung Eigenschaften charakterisiert. Kd ist wurden alle in den 10 -7 10 -8 M-Bereich (Abbildung 3). In Bindungsassays, zeigten verschiedene Paare von Aptameren additive Bindung, was darauf hinweist, dass sie verschiedene Standorte auf der S100B Zielprotein. Deshalb getestet Paare von Aptameren in "Sandwich"-Bindungs-Assays, sowohl auf Glas-Microarrays (CodeLink Dias) mit Fluoreszenz-markierten zweiten Aptamere, und auf derivatisierten Gold-Nanodrähte mit dem zweiten Aptamere gekoppelt zu 50 nm Gold-Nanopartikel (AuNPs; Abbildung 3). In beiden Fällen wurde Bindungsspezifität hoch: CodeLink Microarrays, kein Sandwich-Bindung wurde mit Aptameren, die keinerlei additive Bindung in Kd Bestimmungen beobachtet. Mit derivatisiert Nanodrähte beobachteten wir praktisch keine Bindung an Nicht-Ziel-Proteine und einzelne Sandwich-Komplexe können über die Aptamer-gekoppelten AuNPs (Abbildung 3) beobachtet werden.
2. Materialien
2.1. Generierung von PF DNA-Bibliothek und Reamplifikation gebundenen Fragmente
Details sind in Pan und Clawson, 2009 beschrieben; Pan et al, 2008..2.2. Gereinigtes Protein Based Selection
- 20 mM Tris-HCl, pH 7,4
- 32 +-Fragment und 30 +-Fragment
- Selection Buffer (2,5 mM CaCl 2, 5 mM MgCl 2 in 1X Phosphate Buffered Saline, pH 7,4, Gibco)
- Ni-NTA Agarose Beads (Qiagen)
- Polypropylen-Säule (Qiagen)
- Binding Buffer (50 mM Na 2 HPO 4 -NaH 2 PO 4, pH 7,2, 150 mM NaCl)
- Exprimiert, gereinigt S100B-Protein in Binding Buffer (5 ug / ul)
- Phenol: CHCl3: IAA (pH 7,9, Ambion)
- 3 M NaAc, pH 5,2
- 100% und 70% Ethanol
2.3. TOPO Klonierung und Sequenzierung Analyse der ausgewählten dsDNA Bibliotheken
- 7. Runde ausgewählten PCR-Produkte (weitere Runden durchgeführt werden, um die Optimierung von Aptamer-Bindung fortgesetzt werden)
- pCR2.1-TOPO Vektor (Invitrogen)
- DH5 Component Cells (Invitrogen)
- Plasmid Mini Kit (Qiagen)
- M13 Vorwärts-Primer
- Informax der Vector NTI (Invitrogen)
2.4. Bindungseigenschaften der Aptamere
- Aptamere und Kontrolle Oligos (N 30 CC und N 30, IDT)
- T4-Polynukleotid-Kinase (New England Biolabs)
- γ-32 P-ATP (3000 Ci / mmol, 10 Ci / ml)
- Gereinigtes S100B in Binding Buffer (5 ug / ul)
2.5. Sandwich Bindungsstudien mit gereinigtem S100B-Protein und selektierten Aptamere
- Der 1. Aptamer wurde entweder a. gekoppelt) CodeLink Microarray Slides oder b.) Au-Nanodrähte.
- Gereinigtes S100B-Protein, verdünnt, um entweder 0,125 um in 70 ul für a) oder 1 uM in 50 ul für b).
- Die 2 nd Aptamer entweder mit a. wurde beschriftet) AlexaFluor 546 für die CodeLink Array Folien oder b.) auf 50 nm AuNPs für die Studien mit derivatisierten Au-Nanodrähte gekoppelt.
3. Methoden
3.1. Generierung von PF DNA-Bibliothek
Pan et al, 2008;. Detaillierte Beschreibung der Methoden und Protokolle für die Aptamer-Auswahl kann in Pan und Clawson, 2009 gefunden werden.
3.2. Aptamer Auswahl unter Verwendung von gereinigten Protein S100B
Menschliche S100 Kalzium bindenden Proteins B (S100B. Gene ID # 6285) wurde als Ziel verwendet. Die His 6-Tag S100B-Protein (98 Aminosäuren lang) wurde exprimiert und gereinigt, indem Sie die QIAexpressionist System. (QIAGEN). Falls gewünscht oder angegeben, kann der His 6-tag enzymatisch entfernt werden. Während der Runden der Selektion wurde 1 ul Probe von jeder Schritt für Flüssigszintillationszählung die Allgemeinverbindlichkeit Effizienz bestimmen gespeichert.
3.2.1. Vorbereitung der PF-Bibliothek-DNA-Fragmenten
- Re-suspend die 32 + und 30 +-Fragmente in 40 ul 20 mM Tris-HCl, pH 7,4.
- Heat 3 min bei 85 ° C und Abkühlung auf 37 ° C für 3 min in einem Brutschrank bei 37 ° C.
- Add 760 ul der Auswahl-Puffer (2,5 mM CaCl 2, 5 mM MgCl 2 in 1X Phosphate Buffered Saline, pH 7,4, GIBCO) und Inkubation für 3 min bei 37 ° C, dann bei Raumtemperatur (RT) halten für 10 min.
- Pass durch eine Säule enthaltenen Ni-NTA-Agarose-Perlen (QIAGEN) vorgewaschen mit Auswahl-Puffer, dann bei RT halten bis zum Gebrauch.
3.2.2. Vorbereitung der Ni-NTA Agarose-Bead Bound S100B bei RT
- Spin down 400 ul Ni-NTA Agarose-Beads für 3 sec und den Überstand verwerfen.
- Waschen Sie die Perlen mit 400 ul Bindungspuffer (50 mM Na 2 HPO 4-NaH 2 PO 4, pH 7,2, 150 mM NaCl) durch vorsichtiges Pipettieren 5 mal, dann Spin-Down und den Überstand verwerfen.
- Wiederholen Sie Schritt 2 zweimal.
- Add 400 ul gereinigten S100B (5 ug / ul, suspendiert in Bindungspuffer) und sanft Pipette 5 mal alle 3 min für insgesamt 15 min.
- Spin nach unten und den Überstand verwerfen.
- Waschen Sie die Bead-gebundenen S100B mit 400 ul Bindungspuffer durch vorsichtiges Pipettieren 3 mal, dann Spin-Down und den Überstand verwerfen.
- Wiederholen Sie Schritt 6 zweimal.
3.2.3. Auswahl der S100B-gebundenen Aptamere
- Übertragen Sie die 32 + - und 30 +-Fragmente aus der 3.2.1 auf die Bead-gebundenen S100B.
- Inkubieren für 15 min und vorsichtig mischen alle 3 min durch vorsichtiges Pipettieren.
- Waschen Sie die S100B-DNA-Komplex mit 800 ul Bindungspuffer durch vorsichtiges Pipettieren, dann Spin-Down und den Überstand verwerfen.
- Wiederholen Sie Schritt 3 zweimal.
3.2.4. Wiederherstellung der S100B-selektierten Aptamere
- Geben Sie 200 ul 20 mM Tris-HCl (pH 7,4), Hitze 3 min bei 85 ° C, 1 min vortexen und Spin-Down, dann überweisen Sie den Überstand in ein frisches Röhrchen.
- Wiederholen Sie Schritt 1 einmal, und kombinieren Sie die Überstände.
- Purify ausgewählten DNA-Fragmenten, die als pro Schritte 9-12 aus früheren Veröffentlichungen (11, 12).
3.2.5. TOPO Klonierung und Sequenzierung zur Identifikation von Consensus Aptamersequenzen
- Clone der 10. Runde ausgewählten PCR-Produkte inicht pCR2.1-TOPO Vektor (von Invitrogen). Zusätzliche Klonen kann getan werden als weitere Selektionen durchgeführt werden.
- Sequence 40-50 einzelne Kolonien mit M13 Vorwärts-Primer (wir benutzen das Molecular Genetics Core Facility am Hershey Medical Center).
- Richten Sie die ausgewählten Sequenzen mit Informax der Vector NTI (Invitrogen).
- Bestimmen Sie die Konsensus-Sequenzen auf die Ausrichtungen basieren.
- Consensus-Aptamere wurden dann von Integrated DNA Technologies erworben.
3.3. Sandwich Binding Assays mit Paaren von selektierten Aptamere
3.3.1. Microarray-Format mit fluoreszierend 2. Aptamere bezeichnet.
- Consensus 1 M. Aptamere wurden mit einem 5'-amineC6 Einheit synthetisiert. Sie wurden im Druck-Puffer zu einer Endkonzentration von 15 uM suspendiert und entdeckte auf CodeLink Activated Slides (GE Healthcare / Amersham Biosciences) mit einem Apogent Entdeckungen MicroGrid Arrayer in der DNA Microarray Facility PSU, University Park), nach CodeLink empfohlen Protokolle. Jeder Objektträger wurde mit 12 Arrays gedruckt.
- Hybridisierung erfolgte in einem 2 x 8-Format Microarray Hybridisierung Cassette (Array It, Telechem International) durchgeführt. Die Microarray-Kassette wurde versiegelt mit Nuklease-freiem Kleber Dichtfolie (AlumaSeal II, Research Products International), um Verdunstung zu verhindern.
- S100B-Protein wurde in PBS auf die geeignete Konzentration in 70 ul verdünnt und auf die Vertiefungen der Microarray-Kassette. Die Bindung an die Aptamere auf der CodeLink schieben gedruckt wurde für 1 Stunde bei Raumtemperatur. Die Brunnen der Kassette wurden dann einzeln mit PBS gewaschen + 5 mM MgCl 2 (PBSM) 3X, und die überschüssige Waschpuffer wurde ausgelöscht. Die fluoreszenzmarkierten Aptamere wurden in PBSM auf 0,125 uM in 70 ul verdünnt und dann auf 85 ° C für 3 Minuten mit 3 Minuten auf Eis. Aptamere wurden in die Vertiefungen der Microarray-Kassette aufgetragen und inkubiert für eine Stunde bei Raumtemperatur. Wells wurden wieder einzeln gewaschen 3X mit PBSM. Die Kassette wurde dann zerlegt, ad die gesamte Folie wurde in PBSM gespült. Die Folie wurde dann gründlich durch Zentrifugation getrocknet und gescannt mit einem Scanning Packard Biosciences ScanArray 4000XL (Perkin Elmer).
3.3.2. Derivatisiert Nanodraht-Format mit 2 nd Aptamere gekoppelt zu 50 nm AuNPs
Gold-Nanodrähte (~ 5 uM in der Länge, 320 nm im Durchmesser) wurden von galvanostatische Elektroabscheidung in porösen Aluminiumoxid-Membranen nach früher veröffentlichten Protokollen (13, 14) synthetisiert. Bei Auflösung der porösen Membran, wurden die Nanodrähte in 1 ml Ethanol resuspendiert.
Wie bereits erwähnt, wurde die DNA von Integrated DNA Technologies erworben. Gold-Nanopartikel (50 nm) wurden von Ted Pella gekauft. Thiolierten DNA wurde mit 100 mM DTT in 0,1 M Natriumphosphat pH 8,3 für 1 Stunde gespalten und anschließend gereinigt in einer Centri-Spin-10-Säule.
Aptamer Bindung an Gold-Nanodrähte und NPs
Eine 50 ul-Aliquot der Au-Nanodrähte wurde in ein 0,5 ml Antihaft-Zentrifugenröhrchen gegeben und gespült in 10 mM Phosphat-Puffer, 300 mM NaCl, pH 7,4. DNA am 5'-Ende (mit einem 10 T spacer am 5'-Ende) thiolierten wurde bei einer Endkonzentration von 0,4 M bis die Drähte aufgenommen. Die Probe wurde für 30 min gevortext und anschließend durch Zentrifugation (8100 g) dreimal mit dem 10 mM Phosphat-Puffer, 300 mM NaCl, pH 7,4 und dreimal mit 50 mM Phosphatpuffer, 5 mM MgCl 2, pH 7,2 gespült. Die DNA-Drähte wurden in 100 ul Puffer um die Hälfte zu verdünnen resuspendiert.
DNA-derivatisierten AuNPs wurden durch Zugabe von 50 ul 100 mM 2 nd Aptamer (mit einem Abstandhalter von 10 T ist am 5'-Ende) auf 1 mL 50 nm AuNPs und beheizt bei 37 ° C für eine Stunde vorbereitet. Nach dem Erhitzen wurde 10 mM Natrium-Phosphat-Puffer, 1M NaCl, pH 7,4 hinzugefügt (25 uL zwei Mal mit 100 ul, 150 ul und 128 ul folgte) nach 0,5 Stunden Intervallen. Die Konjugate wurden auf einem 37 ° C Heizblock über Nacht vor dem Gebrauch überlassen. Die Proben wurden 3 mal mit 50 mM Phosphatpuffer, 5 mM MgCl 2, pH 7,2 gespült. Konjugate wurden in 120 ul Puffer resuspendiert.
Sandwich-Hybridisierung auf Nanodrähte
DNA beschichtete Drähte, 1 ul verdünnt, wurden hinzugefügt, um in PCR-Röhrchen Puffer. S100B-Protein oder HtrA1 Kontroll-Protein (1 pg / mL) wurde für eine endgültige Konzentration von 1 nM in 50 ul Puffer (~ 10-12 Proteinmoleküle Wertschöpfung pro Quadratnanometer der Au-Nanodraht-Oberfläche) aufgenommen. Die Proben wurden für ca. 2 Std. verwirbelt. Drähte wurden durch Zentrifugation (8100 g) dreimal mit 50 mM Phosphatpuffer, 5 mM MgCl 2, pH 7,2 gespült und wurden jeweils in 20 ul AuNP / DNA-Konjugate resuspendiert. Die Drähte wurden in der Konjugate für 2 h gevortext und anschließend gespült 5x mit 50 mM Phosphatpuffer, 5 mM MgCl 2, pH durch Zentrifugation (1300 g)7,2 bis das überschüssige Nanopartikel zu entfernen. Die Proben wurden in 20 ul Puffer resuspendiert und trocknet über Au beschichteten Si-Wafern für die FE-SEM-Analyse.
FE-REM-Aufnahmen der Nanodrähte wurden mit einem LEO-1530-Field Emission Scanning Electron Microscope mit einer Schottky Feldemissions-Elektronenquelle auf 5,00 kV Betriebsspannung.
4. Repräsentative Ergebnisse
Abbildung 1. Der 5'-Primer-Free (PF 1) und Primer-Free (PF 2) Protokolle.
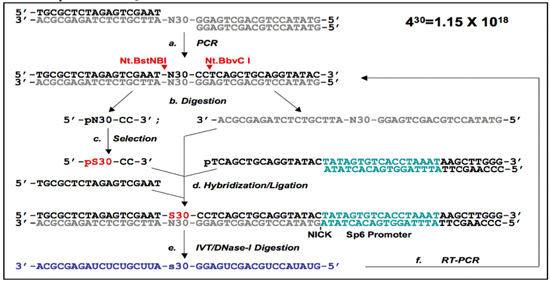
(A) Die PF 1 DNA-Aptamer-Auswahl-Protokoll. Die Bibliothek Oligonukleotiden zusammen, geglüht und einer PCR-Amplifikation zu einem doppelsträngigen DNA-Bibliothek Ausbeute (a). Das 5'-Ende Primer-free (PF 1) random Region aus der PF Einzelstrang-DNA-Bibliotheken wird durch Nt.BstNBI / Nt.BbvCI Aufschlüsse (Spaltstellen sind mit rot Pfeilspitzen markiert) und Gel-Reinigungen vorbereitet, und das Selbst -Brücke (schwarzer Pfeil) wird durch Gel-Reinigung (b) isoliert. Nach der Auswahl (c), werden die ausgewählten Sequenzen (benannt PS30-CC) mit den entsprechenden Oligomere und das Selbst-Brücke hybridisiert und ligiert, um re-generieren die zuvor entfernten Primer Regionen. In diesem Stadium werden Primer eingeführt, enthalten einen SP6-Promotor am 3'-Ende (d). Dieser wird dann verwendet, um RNA mit den ausgewählten Regionen (e) zu transkribieren. Schließlich wird die RNA dann ausschließlich erneut verstärkt (die Template-DNA verdaut wird) mittels RT-PCR (f), und die ausgewählten Sub-Bibliothek ist bereit für die nächste Runde der Selektion.
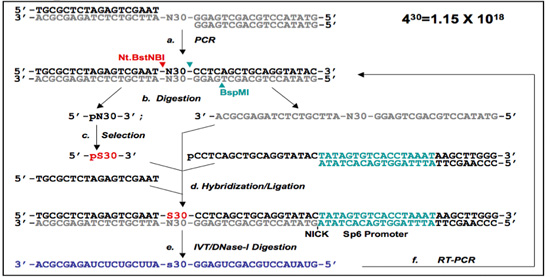
(B) Die PF 2 DNA-Aptamer-Auswahl-Protokoll. Die Bibliothek Oligonukleotiden zusammen und geglüht unterzogen PCR-Amplifikation zu einem doppelsträngigen DNA-Bibliothek Ausbeute (a). Die Primer-free (PF 2) random Region aus der PF Einzelstrang-DNA-Bibliothek wird von Nt.BstNBI / BspMI Aufschlüsse (cut Seiten sind mit Pfeilspitzen markiert) und Gelreinigung vorbereitet. Die Selbst-Brücke wird entweder aus dem PF-1-Protokoll und / oder durch Einzel-Verdauung des Ausgangsmaterials Bibliothek mit Nt.BstNBI (b) isoliert, gepaart mit Gelreinigung. Nach der Auswahl (c), werden die ausgewählten Sequenzen (benannt PS30) mit den entsprechenden Oligomere und das Selbst-Brücke hybridisiert und ligiert, die zuvor entfernt Primer Regionen zu regenerieren. Wie in PF 1 werden Primer eingeführt, enthalten einen SP6-Promotor am 3'-Ende (d). Dieser wird dann verwendet, um RNA mit den ausgewählten Regionen (e) zu transkribieren. Die RNA wird dann ausschließlich reamplifiziert mittels RT-PCR (f), und die ausgewählten Sub-Bibliothek ist bereit für die nächste Runde der Selektion.
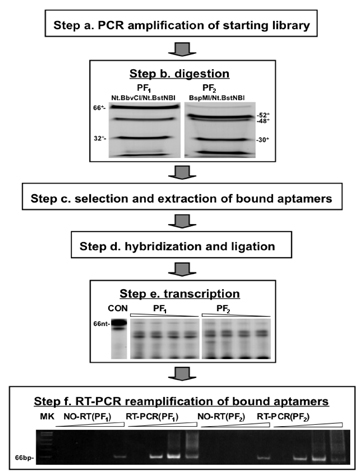
Abbildung 2. Schematische Darstellung und experimentelle Ergebnisse / Reduktion auf der PF Auswahl Protokoll Praxis. Designated Schritte, um die in Abbildung 1 dargestellt entsprechen. Nach der Verdauung des PF-Bibliothek durch PCR (Schritt a) mit Restriktionsendonukleasen (wie angegeben) konstruiert wurden die verdauten Produkte von PAGE auf einem 10% Gel unter denaturierenden Bedingungen getrennt. Die entsprechenden Fragmente mit dem PF 1 und PF 2 Auswahl Protokolle erhalten werden angezeigt (Schritt b). Nach der Auswahl (Schritt c) werden die gebundenen Aptamere mit ihren entsprechenden Oligomere und das Selbst-Brücke hybridisiert und ligiert, die zuvor entfernt Primer Regionen zu regenerieren. Am 3'-Ende werden Primer eingeführt, enthalten einen SP6-Promotor am 3'-Ende (Schritt d). 32 P-markierten RNAs transkribiert wurden mit ligierten Produkte von 1, 0,5, 0,25, 0,125 ul von 5 ul Reaktionen und wurden getrennt durch PAGE auf 6% Gele unter denaturierenden Bedingungen (Schritt e). Die Transkripte wurden dann mit DNase behandelt, um die nicht ausgewählten entfernt zufällige DNA-Regionen, reverse in cDNA umgeschrieben und dann für 7, 14, 21 oder 28 PCR-Zyklen reamplifiziert. Die Produkte wurden getrennt durch PAGE auf 8% Gelen unter nicht-denaturierenden Bedingungen. Die 66 nt-Fragment stellt den full-length Produkt mit dem ausgewählten PS30-CC und PS30-Sequenzen als in der 5 'und 3' flankierenden Sequenzen re-integriert. Migration von PhiX174/HinfI Marker ist links abgebildet (Schritt f). Kontrollen umfassten Wegfall der reversen Transkription (RT) Schritt. Eine kleine Menge des Produktes in solchen Proben konnten bei hohen Taktzahlen zu sehen, das vermutlich vollständig mit einer zweiten (oder längere) DNase-Verdau Schritt beseitigt werden.
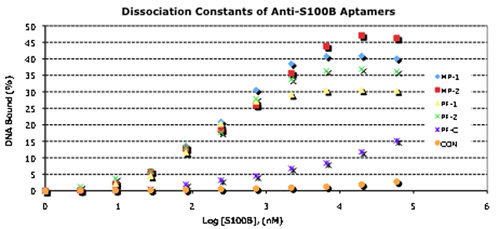
Abbildung 3. Bindungseigenschaften von selektierten Aptamere und ihre Paired Verwendung in einer Sandwich-Format.
A. konzentrationsabhängige 32 P-Aptamer-Bindung für Kd Bestimmung. Aptamere wurden 5'-Ende markiert mit g-32 P-ATP (3000ci /mmol, war ~ 10 mCi) und T4 Polynukleotid-Kinase (New England Biolabs), und die Bindung wie beschrieben bestimmt.
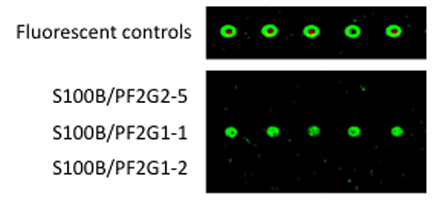
B. 5'-Amin-derivatisierten 1 M. Aptamere gekoppelt waren, um Microarrays CodeLink. Gereinigtes S100B-Protein in die 1. Aptamer gebunden war, wurden die Objektträger gründlich gespült und AlexaFluor546-Bezeichnung 2 nd Aptamer, das der 1. Aptamer gebunden war: S100B-Komplexen. Nach über Spülen wurde Fluoreszenz quantifiziert mit einem ScanArray Scanner.

C. 5'-thiol derivatisiert 1 M. Aptamere wurden Au-Nanodrähte mit Standard-Thiol-Chemie gekoppelt. 2. Aptamere wurden auf 50 nm AuNPs in der gleichen Weise gekoppelt. Gereinigtes S100B-Protein (links) oder gereinigt HtrA1 Kontroll-Protein (rechts) wurde dann gebunden ersten bis zum derivatisierten Nanodrähte. Nach gründlichem Spülen, 2 nd Aptamer-50 nm AuNPs wurden anschließend auf die 1 st-Aptamer-Nanodraht gebunden: S100B-Komplexen. Nach über Spülen wurden die gebundenen Sandwichkomplexe visualisiert Feldemissions-Rasterelektronenmikroskopie. Maßstabsbalken = 1 um.
Offenlegungen
Danksagungen
Wir danken Craig Paul an der Microarray Core Facility für seine Hilfe. Diese Arbeit wurde vom NIH / NCI gewähren # CA118591 aus dem IMAT Programm unterstützt. SLD erkennt auch der Pennsylvania Raum Stipendium Consortium Fellowship für finanzielle Unterstützung. Diese Veröffentlichung wurde auch von der Pennsylvania State University Materials Research Institute Nano Fabrication Network und der National Science Foundation Cooperative Agreement Nr. 0335765, National Nanotechnology Infrastructure Network, mit der Cornell University unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Tris-HCl | |||
| HotMaster TAQ DNA Polymerase | 5 PRIME | ||
| dNTP Mix | Sigma-Aldrich | ||
| Acrylamide | Fisher Scientific | ||
| UltraPure 10X TBE Buffer | Invitrogen | ||
| Ammonium Persulfate | Bio-Rad | ||
| TEMED | Fisher Scientific | ||
| PhiX174 DNA/Hinf I DNA Marker | Promega Corp. | ||
| Ethidium Bromide | |||
| α-32P-dCTP | |||
| Phenol:ChCl3:IAA | Ambion | ||
| NaCl | |||
| Ethanol | |||
| Restriction Enzymes | New England Biolabs | ||
| Urea | Fisher Scientific | ||
| Photographic Film | ECE Scientific | ||
| PBS | GIBCO, by Life Technologies | ||
| CaCl2 | GIBCO, by Life Technologies | ||
| MgCl2 | GIBCO, by Life Technologies | ||
| Ni-NTA Agarose Beads | Qiagen | ||
| Polypropylene Column | Qiagen | ||
| NaHPO4 | |||
| NaAc | |||
| Detroit-551 cells | |||
| SK-MEL-31 cells | |||
| MEM | |||
| TrypLE Express | GIBCO, by Life Technologies | ||
| T4 DNA Ligase | New England Biolabs | ||
| Dimethyl Sulfoxide | Promega Corp. | ||
| Sp6 RNA Polymerase | Promega Corp. | ||
| RNase-Free DNase I | Promega Corp. | ||
| RNase Inhibitor | Promega Corp. | ||
| SensiScript Reverse Transcriptase | Qiagen | ||
| pCR-2.1-TOPO Vector | Invitrogen | ||
| DH5α Compotent Cells | Invitrogen | ||
| Plasmid Mini Kit | Qiagen | ||
| Vector NTI | Invitrogen | ||
| T4 Polynucleotide Kinase | New England Biolabs | ||
| γ-32P-ATP |
Referenzen
- Kulbachinskiy, A. V. Methods for selection of aptamers to protein targets. Biochemistry (Moscow). 72, 1505-1518 (2006).
- Mairal, T. Aptamers: molecular tools for analytical applications. Anal Bioanal Chem. 390, 989-1007 (2008).
- Stoltenburg, R., Reinemann, C., Strehlitz, B. SELEX -- A (r)evolutionary method to generate high-affinity nucleic acid ligands. Biomolec Engineering. 24, 381-403 (2007).
- Que-Gewirth, N. S., Sullenger, B. A. Gene therapy progress and prospects: RNA aptamers. Gene Ther. 14, 283-2891 (2007).
- Cowperthwaite, M. C., Ellington, A. D. Bioinformatic analysis of the contribution of primer sequences to aptamer structures. J Mol Evol. 67, 95-102 (2008).
- Shtatland, T., Gill, S. C., Javornik, B. E. Interactions of Escherichia coli RNA with bacteriophage MS2 coat protein: genomic SELEX. Nucleic Acids Res. 28, E93-E93 (2000).
- Jarosch, F., Buchner, K., Klussmann, S. In vitro selection using a dual RNA library that allows primerless selection. Nucleic Acids Res. 34, E86-E86 (2006).
- Vater, A., Jarosch, F., Buchner, K., Klussmann, S. Short bioactive Spiegelmers to migraine-associated calcitonin gene-related peptide rapidly identified by a novel approach: tailored-SELEX. Nucleic Acids Res. 31, E130-E130 (2003).
- Wen, J. D., Gray, D. M. Selection of genomic sequences that bind tightly to Ff gene 5 protein: primer-free genomic SELEX. Nucleic Acids Res. 32, E182-E182 (2004).
- Musheev, M. U., Krylov, S. N. Selection of aptamers by systematic evolution of ligands by exponential enrichment: addressing the polymerase chain reaction issue. Anal Chim Acta. 564, 91-916 (2006).
- Pan, W. H., Clawson, G. A. Primer-free aptamer selection using a random DNA library. Methods Mol Biol. , (2009).
- Pan, W., Xin, P., Clawson, G. A. Minimal-Primer and Primer-Free SELEX Protocols for Selection of Aptamers from Random DNA Libraries. BioTechniques. 44, 351-360 (2008).
- Nicewarner-Pena, S., Freeman, G., Reiss, B. Submicrometer metallic barcodes. Science. 294, 137-141 (2001).
- JC, H. u. l. t. e. e. n., CR, M. a. r. t. i. n. A general template-based method for the preparation of nanomaterials. J Materials Chem. 7, 1075-1087 (1997).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten