Method Article
משופר מופחת ייצוג Bisulfite רצף להערכה של DNA מתילציה ברזולוציה בסיסי זוג
* These authors contributed equally
In This Article
Summary
Enhanced Reduced Representation Bisulfite Sequencing is a method for the preparation of sequencing libraries for DNA methylation analysis based on restriction enzyme digestion combined with cytosine bisulfite conversion. This protocol requires 50 ng of starting material and yields base pair resolution data at GC-rich genomic regions.
Abstract
מיפוי תבנית המתילציה של הדנ"א הוא למד בכבדות ברקמות בריאות וחולות. מגוון רחב של שיטות הוקמו כדי לחקור את דפוסי מתילציה ציטוזין בתאים. ייצוג מופחת של רצף הגנום כולו bisulfite פותח כדי לזהות דפוסי מתילציה ציטוזין הרזולוציה זוג בסיס הכמותי בלוקוסים הגנומי GC-עשיר. המטרה זו מושגת על ידי שילוב של השימוש באנזים הגבלה ואחרי המרת bisulfite. משופר ייצוג מופחת Bisulfite רצף (ERRBS) מגדיל את הלוקוסים הגנומי רלוונטיים מבחינה ביולוגית המכוסים ומאז משמש לפרופיל מתילציה ציטוזין בDNA מאדם, עכבר ואורגניזמים אחרים. ERRBS יוזם עם עיכול אנזים הגבלה של DNA כדי ליצור שברי משקל מולקולריים נמוכים לשימוש בהכנת ספרייה. שברים אלה נתונים לבניית ספרייה סטנדרטית עבור סידור הדור הבא. המרת Bisulfite של cytosines unmethylated לפני amplificati הסופיבצעד מאפשר רזולוציה בסיס כמותי של רמות מתילציה ציטוזין בלוקוסים הגנומי מכוסים. הפרוטוקול ניתן להשלים בתוך ארבעה ימים. למרות מורכבות נמוכות בשלושת הבסיסים הראשונים רצף, ספריות ERRBS להניב נתונים באיכות גבוהה בעת שימוש בנתיב שליטת רצף מיועד. ניתוח מיפוי וביואינפורמטיקה מתבצע לאחר מכן ונתוני תשואות שניתן לשלב בקלות עם מגוון רחב של פלטפורמות הגנום. ERRBS יכול לנצל כמויות חומר הזנה קטנות שהופך אותו אפשרי על מנת לעבד דגימות קליניות בבני אדם וישימים במגוון רחב של יישומי מחקר. הסרטון שהופק מדגים שלבים קריטיים של פרוטוקול ERRBS.
Introduction
מתילציה DNA בציטוזין (5-methylcytosine) היא סימן אפיגנטיים קריטי בתאי יונקים עבור מגוון רחב של תהליכים ביולוגיים, כולל אך לא מוגבל להחתמה, איון כרומוזום X, פיתוח, ורגולציה של ביטוי גני 1-8. המחקר של דפוסי מתילציה DNA בממאירים ואחרות הפרעות קבע דפוסים מסוימים מחלה ותרם להבנה של היווצרות מחלה ותגליות סמן ביולוגי פוטנציאליות 9-17. יש פרוטוקולים רבים שחקרו את epigenome למעמד מתילציה DNA. אלה יכולים להיות מחולקים הלמבוססים על זיקה, המבוסס אנזים הגבלה, ומבחנים מבוססי המרת bisulfite לנצל פלטפורמות microarray או רצף במורד הזרם. יתר על כן, יש כמה פרוטוקולים שלגשר קטגוריות הכלליות אלה כוללים, אך לא מוגבלים ל, ניתוח הגבלת Bisulfite משולב 18 וצמצום ייצוג Bisulfite רצף (RRBS 19).
RRBS תואר במקור על ידי et al מייסנר. 19,20. הפרוטוקול הציג צעד להעשיר אזורים הגנומי GC-העשיר ואחריו רצף bisulfite, שהביא לנתונים ברזולוציה בסיס-זוג כמותי שהוא עלות אפקטיבית 21,22. אזורי GC העשיר ממוקדים על ידי אנזים הגבלת MspI (C ^ CGG), ומתילציה ציטוזין נפתרה על ידי המרת bisulfite של cytosines (דיאמינציה של cytosines ללא שינוי לאורציל), ואחריו תגובת השרשרת של פולימראז הגברה (PCR). RRBS כיסה את רוב יזמי גן ואיי CPG בשבריר של הרצף הדרוש לגנום שלם; היה לי עם זאת RRBS כיסוי מוגבל של חופי CPG ואזורים חוץ-אחרים רלוונטיים ביולוגי. כמה קבוצות פרסמו מעודכנות פרוטוקולי RRBS מאז הדוח המקורי שלשפר את המתודולוגיה וכיסוי כתוצאה של אזורים הגנומי אלה 23-25. משופר ייצוג מופחת Bisulfite Sequencing (ERRBS) כולל שינויי ספריית הכנה ויישור נתונים חלופי גישה 26 בהשוואה לRRBS. ERRBS הביא מספר גבוה יותר של CpGs המיוצג בנתונים שנוצרו וגדל כיסוי של כל האזורים הגנומי נחקר 26. שיטה זו נעשתה שימוש כדי לפתור דפוסי מתילציה DNA במטופל אדם ודגימות בעלי חיים אחרים 26-30.
פרוטוקול ERRBS תאר פרטי הצעות על כל צורך להשלמה ולנתונים שנוצרו באמצעות DNA האנושי נציג הצעדים (דגימות נלקחו מדגימות שדווחו בעבר, מזוהה de חולה 31, ודגימת מוח CD34 + עצם מתורם אנושי נורמלי). הפרוטוקול כולל תהליך אוטומטי גודל בחירה, אשר מפחית את זמן עיבוד לדגימה ומאפשר לדיוק מוגבר בבחירת גודל ספרייה. הפרוטוקול משלב סדרה של טכניקות ביולוגיה מולקולריות הוקמו. DNA משקל המולקולרי הגבוה מתעכל wה- i-אנזים הגבלת מתילציה רגישה (MspI) ואחריו הסוף-תיקון, A-עוקב, וקשירה של מתאמים מפוגלים. בחירת גודל של שברי GC-העשיר ואחריו המרה bisulfite והגברת PCR לפני הריצוף. המרת Bisulfite כבר בעבר תיארה 32 וסקירה מפורטת של ניתוח נתונים ויישומים היא מעבר להיקף של מאמר זה, עם זאת המלצות והפניות כלולות לשימושם של הקוראים. הפרוטוקול יכול להתבצע על פני ארבעה ימים, והוא ניתן לקלט קטן (50 ng או פחות) בסכומים מהותיים. הפרוטוקול כנתונים תשואות מתוארות עם כיסוי גבוה לאתר CPG מספיק לא רק לקביעות אתר מתילציה ואזור ההפרש אלא גם לזיהוי פולימורפיזם אפיגנטיים כפי שתואר על ידי לנדן, et al. 33.
Protocol
כל הנהלים מבוצעים מאושרים על ידי בית הספר לרפואה של אוניברסיטת אינדיאנה מוסדית טיפול בבעלי חי ועדת שימוש ופעלו מכון הלאומי לבריאות הנחיות.
1. טכניקה כירורגית
- לשמור על טכניקת aseptic במהלך הליך זה על ידי שימוש בכפפות סטריליות, מכשירים, ותחום כירורגית סטרילית על פי הנחיות NIH 25. לעקר כלים לפני תחילת ניתוח על ידי המעוקר (ראה טבלה של חומרים כימיים / ציוד ספציפי לרשימה מלאה). השתמש מעקר חרוז זכוכית לעקר כלים במהלך המבצע.
2. הרדמה והכנה
- להרדים את העכבר בתיבת הרדמה בתערובת של 0.9 L חמצן / דקה וisoflurane 2.5% באמצעות מערכת אידוי isoflurane וטרינרים. ודא שהעכבר אינו מגיב לשינויים בתנוחת גוף לפני הסרתו מהתיבה.
- החל משחת עיניים לmouעיניו של se כדי להגן עליהם מהתייבשות.
- לעבור את זרימת הגז מהתיבה לחרטום. מניחים את העכבר ישירות על צדו השמאלי על משטח לוהט מכוסה עם כרית כירורגית ונייר סופג ספסל עם האף שלה ופה בתוך חרוט. לפקח באופן רציף קצב הנשימה של העכבר ושיעור ולהתאים את רמות isoflurane לפי צורך (בין 2.5 - 3% isoflurane) כדי לשמור על רמה נאותה של הרדמה, ולהשתמש ברפלקס קמצוץ הבוהן כדי לאשר הרגעה כוללת.
3. גישה ניתוחית
- יישר ולהתמקד stereoscope עם את שדה הניתוח. התאם את החרטום וקלט אותו כך שהוא נמצא בעמדה בשולי שדה הראייה.
- עם העכבר שוכב על צידו השמאלי, תדביק את קצה אוזן ימין לחרטום, וחושף את האזור שמאחורי האוזן שבו החתך ייעשה. ודא שוריד אוזן האחורי נוסע אופקי על פני האוזן. שימו לב כי המיקום הנכון של tהוא חיה ומקליטה של האוזן הן קריטיות כדי למצוא עצב הפנים במהירות.
- להרטיב את הפרווה ובמאחורי האוזן עם 70% אתנול ו לגלח את האתר כירורגית באמצעות סכין גילוח או אזמל. טרום להרטיב את הפרווה הופך גילוח קל יותר במיקום האנטומי הזה.
- לנקות את העור עם תמיסת יוד, כגון לשפשף פולידין כירורגית (7.5% povidone- יוד), ואחריו אתנול 70%. חזור על ניקוי זה פעמים נוספות כדי לחטא את האזור ביסודיות.
- כדי לקבוע היכן לבצע את החתך, לעקוב אחר וריד אוזן האחורי מהאוזן caudally לאזור האחורי לבליטת האוזן. בעזרת מספריים האביב, לעשות חתך 4 מ"מ - 2 מ"מ אחורי 3 לבליטה.
- לנתח באמצעות השומן תת עורי וfascia באמצעות לנתיחה בוטה. הימנע חיתוך ישיר עם המספריים כי כלי דם או רקמת שריר יכולים להיפגע בקלות.
- אם דימום מתרחש, להפעיל לחץ לאתר כירורגית עם מקלון צמר גפן סטרילילפחות 30 שניות. אם אובדן נוזלים משמעותי מתרחש, להזריק את העכבר intraperitoneally עם עד 0.5 מיליליטר של תמיסת מלח סטרילית 0.9% באמצעות 25 או 27 מחט G.
- השתמש בכמה ציוני דרך מרכזיות, עצב אבזר השדרה, תעלת אוזן, וקדמיים שריר הדו-בטנים (שיפורט להלן), כדי לאתר את עצב הפנים. לנתח סביב ציוני הדרך הללו עד הסניפים של עצב הפנים הם דמיינו. העצב יופיע כמבנה לבן מוצק משמעותי כאשר הוא נחשף ושכבת fascia שומרת אותו למבנים שבבסיס.
- מצא את עצב אבזר השדרה, אשר נוסע מחלק הזנב של הגולגולת למעצבב את שריר הטרפז, ברגע שהשומן תת עורי וfascia כבר גזור. עצב הפנים עמוק לעצב אבזר השדרה.
- מצא את תעלת אוזן הסחוסים שנראית פנינים לבנה וניתן לראות מקורי לעצב הפנים.
- מצא את שריר הבטן של השריר הדו-הבטנים הקדמית שנמצאת על גבי וגaudal לעצב הפנים.
- כאשר הענפים העיקריים של עצב הפנים הם דמיינו, לעקוב אחר אותם dorsally למצוא מוצאם מנקב stylomastoid. באמצעות קנס היטה מלקחיים דומון # 5/45 להחזיק אתר כירורגית פתוח, לקדם את הטיפים מספריים האביב הבא הנתיב של העצב, ולאחר מכן להעביר את המלקחיים dorsally לשמור על האזור המתקדם החדש פתוחים.
- דמיינו את תא המטען של עצב הפנים עם הזיגומטית, buccal, וסניפים בלסת תחתונים, שוליים בנקודה זו.
הערה: הסניף הזמני יימצא קרוב יותר לforamen. ענפי עצב הלסת תחתונים, השוליים לחלקיו העליונים ותחתונים קרובים ללסת, וכך בענפי עצב אלה לא יהיו גלויים ברמה זו.- אם ביצוע חיתוך רוחב עצב, לייצב את העצב בעדינות עם המלקחיים טיפ הנאה וחתך את העצב עם המספריים באביב. הימנע מהפעלה יותר מדי מתיחה בעצב עם המלקחיים כדי למנוע avulsing העצבי מהמוח. Pushהגדמים אחד מהשני, או לחתוך ולהסיר חלק מהעצב הדיסטלי כדי לוודא ששום חיבור מחדש יכול להתרחש.
- אם ביצוע פציעה למעוך, להשתמש דומון # 5/45 במלקחיים כדי לדחוס את העצב למשך 30 שניות באמצעות לחץ מתמיד לנתק את כל האקסונים, ולאחר מכן לחזור למחוץ זה בזווית שנייה בניצב לאתר למעוך הראשון. הימנע מהפעלת כמויות משתנות של לחץ במהלך למעוך 30 שניות, אחרת הפציעה תהיה עקבית בין בעלי החיים.
4. סגירה ושחזור
- למקם מחדש את השומן ושרירים על מבני היסוד.
- בקירוב את הקצוות של החתך ולסגור את הפצע באמצעות קליפ פצע 7.5 מ"מ. תפרים או דבק מקובלים גם לסגירת פצעים. יכולים להיות מסופקים על משככי כאבים לאחר ניתוח בשלב זה.
- הסר את הסרט מאוזנו של העכבר. כבה את זרימת isoflurane ולאפשר העכבר כדי לנשום חמצן טהור למשך 30 שניות לדקות 1. Plאס העכבר בכלוב ריק ללא מצע להתאושש מהרדמה.
- כאשר העכבר הוא התאושש, לבחון את ההתנהגות שלה בחיפוש אחר סימנים לאישוש של שיתוק פנים. השפם יהיה משותק והזווית חזרה לכיוון הלחי, האף יהיה חרג, והעין לא למצמץ בתגובה למשב אוויר.
- בעלי חיים במשותף הבית לאחר ניתוח אם הם ממין נקבה. הימנע דיור עכברי זכרים במשותף כי הם אגרסיביים יותר ונוטים להעביר בכפיית קליפים הפצע של cagemate, מה שמוביל לזיהום. לספק משככי כאבים לאחר ניתוח בשלב זה, במידת צורך.
- לפקח על העכברים פעם ביום במשך כמה ימים לאחר הניתוח כדי לוודא ששום זיהום או סיבוך אחר מתרחש לאחר הניתוח. הסר קליפים פצע 7-10 ימים לאחר הניתוח, אם הם לא נפלו החוצה בכוחות עצמם.
- החל הסיכה משחה העין לעין המושפעת מדי יום כדי למנוע סיבוכים בקרנית, או עד שרפלקס מצמוץ העין הוא מחדשמכוסה או עד המתת חסד.
תוצאות
איור 1 מספק סקירה של ERRBS, הדגשת שלבים עיקריים, אשר הסבירו בכל הפרוטוקול המתואר. ספריות ERRBS הוכנו באמצעות DNA קלט 50 ng.
להעריך את האיכות של הספריות מוכנות. ייצור הספרייה מניב באופן שיגרתי בגדלים שבריר של 150-250 נקודות בסיס ו250-400 נ"ב (איור 3 א-ג). הבדלים קלים בהפצות גודל ספרייה בין הדגימות צפויים. שים לב כי בשני שברים הספרייה נמוכים וגבוהים יש גדלי DNA מאוד אינטנסיביים, מעידים על ההעשרה של רצף מסוים. תוצאות עיכול MspI בהעשרה של משפחה של DNA חוזר על עצמו רצפי נוכחים בגנום האנושי ב 190 נ"ב, 250 נ"ב ו 310 נ"ב בספריות ERRBS. שלוש חזרות אלה מייצגות חתימה אופיינית לספריית ERRBS 20 (ראה איורים 3 א-C ו3G). ספריות נציג היו רצף ברצף של הדור הבאr חד-סוף השימוש קורא. בעת הטעינה בריכוז הספרייה המומלצת על רצף Illumina HiSeq 2500, צפיפות מקבץ של 500,000-700,000 למ"מ 2 צפויות. בצפיפות זה קיבוץ באשכולות, 81.6% ± 3.14% (n = 81) של האשכולות לעבור סינון (איור 4 א). בשל מורכבות של הסוף הנמוך מוסיף הספרייה (אתר ההכרה MspI: C ^ CGG), ערכי עוצמה וציוני איכות שנרשמו ברצף הם משתנים מאוד בשלושת הבסיסים הראשונים (איור 4 ב-ג), לעומת זאת, אם נתיב שליטה עצמאי כלול (ראה דיון), 85% מבסיסים יהיו ציוני איכות של 30 או יותר (ערכי Q30; איור 4D).
יישור נתונים ונחישות מתילציה ציטוזין כפי שמתואר בנתונים ברזולוציה תשואות פרוטוקול בסיס-זוג (לוח 7). לגנום האנושי, ריצת 51-מחזור קריאה יחידה רצף של ספריית ERRBS בנתיב אחד של HiSeq 2500 במצב תפוקה גבוהה רגילly מייצר 153194882 ± 12918302 כולל קורא כי לאחר סינון איכותי ומתאם זמירה תשואות קוראים 152231183 ± 13189678 לקלט לתוך צינור הניתוח. יעילות המיפוי הממוצעת לספריית ERRBS היא בדרך כלל 62.95% ± 5.92% עם ייצוג של 3183594 ± 713547 CpGs עם כיסוי מינימאלי לCPG של 10x וכיסוי לCPG ממוצע של 84.94 ± 16.29 (n = 100).
פרוטוקול ERRBS ניתן לריבוב (ראה קובץ נוסף 1: הסתגלות פרוטוקול עבור סידור ריבוב). נתונים מרצף נציג הרץ מסוכמים באיור 5 נתונים מריצות רצף ריבוב (לקריאה יחידה ריצת רצף 51-מחזור;. N = 128 לשתי ספריות לכל נתיב; n = 11 לשלוש ספריות לכל נתיב; n = 11 לארבע ספריות לכל נתיב) היו בהשוואה לריצוף נתיב מלא של ספריית ERRBS (ריצות רצף קריאה יחידה 51-מחזור; n = 100), כמו גם downsampling נתיב יחיד לsimulatדואר 50%, 33% ו -25% מקורא לכל נתיב (2, 3, ו -4 ריבוב מדגם לכל נתיב בהתאמה; n = 3). ככל שמספר קורא לדגימה יורד עם גורם הריבוב, מספר CpGs המכוסה בכיסוי מינימאלי של 10x והכיסוי לCPG יורד גם כן (איור 5 ולוח 8). שיעורי המרה ממוצעות של אתרים שאינם CPG הצפויים הם 99.85% ± 0.04% (n = 400). שיעורי המרה נמוכים מ -99% עשויים להצביע על פחות מהמרת bisulfite אופטימלית שיכול לגרום לשיעור גבוה של רמות מתילציה שווא.
נתונים מספריית ERRBS הוכנה מהדנ"א הגנומי אנושי נציג נותחו בR 2.15.2 45 באמצעות חבילת methylKit 26 (ראה קובץ קוד נוסף 1 לפרטים פקודה). ניתן דמיינו נתונים בדפדפנים נפוצים הגנום (איור 6 א). נתונים מתילציה ציטוזין נגזר באופן שווה משני הגדילים (איור 6) ונעים כלספקטרום של רמות פוטנציאל מתילציה ציטוזין (איור 6 ג). ניתוח טכני משכפל מגבוהה התאמה תשואות דגימת DNA האנושיות נציג בין תוצאות נתונים (איור 6 ד) ומכסה CpGs בקשת רחבה של לוקוסים הגנומי (איור 6-ו-ו 'וכפי שתואר לעיל 26). בעוד העתקים טכניים יניבו גבוה R 2 ערכים (יותר מ 97%), העתקים ביולוגיים יניבו R 2 ערכים הנעים 0.92-0.96 26, והשוואה בין סוגים שונים של תאי אדם יניב R 2 ערכים נמוכים יותר מאשר 0.86 (מידע לא מוצג).
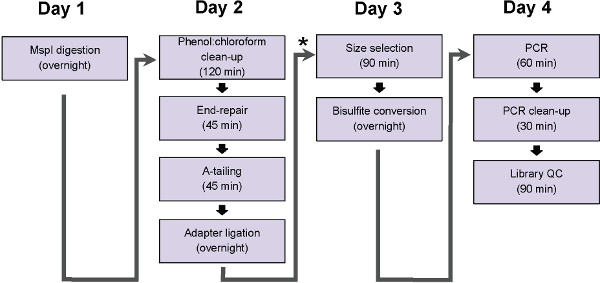
איור 1: תרשים זרימה של שלבי פרוטוקול ERRBS. תרשים מייצג את שלבים, שיכול להסתיים ביום עבודה מסורתי. * מציין נקודת הפסקה פוטנציאלית (מייד מעקב קשירת ing לנקות ולפני בחירת גודל, צעד פרוטוקול 5) שביכולות להיות קפוא דגימות ב -20 ° C לפני שתמשכנה עם את משך הזמן של פרוטוקול.
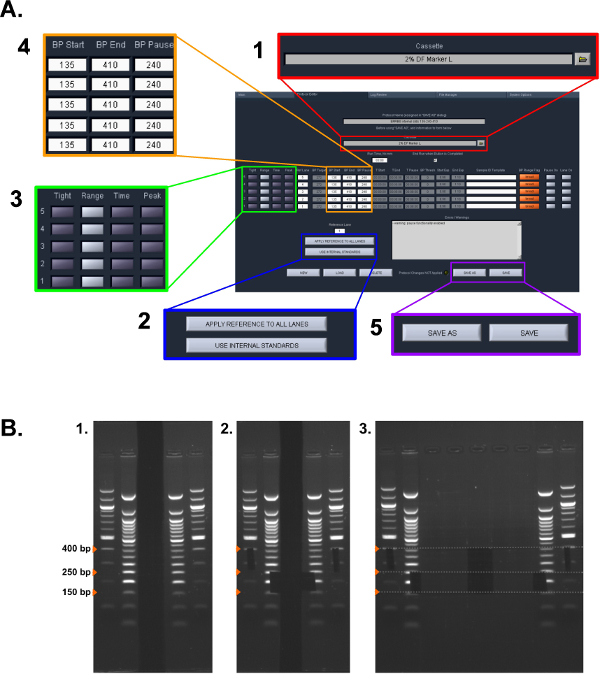
איור 2: פרוטוקול בחירת גודל. (א) צילום מסך של הגדרות המשמשות בפרוטוקול ERRBS פיפין Prep (ראה סעיף 5.1.2 פרוטוקול - 5.1.6): (1) סוג הקלטת בחר. (2) הסטנדרטי בחר לשמש. (3) בחר את מצב הגבייה לכל מסלול. (4) הזן את הטווחים נ"ב אוסף. (5) שמור את פרוטוקול שלבים (ב) לחילוץ ג'ל המדריך לשימוש בסעיף 5.2 פרוטוקול:. (1) סולמות ג'ל דמיינו. (2) מסומן גדלים לבחירת גודל באמצעות סכין גילוח. (3) תמונה של דגימות נכרתה (חלק תחתון: 150-250 נקודות בסיס וחלק גבוה יותר: 250-400 נ"ב)."> לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
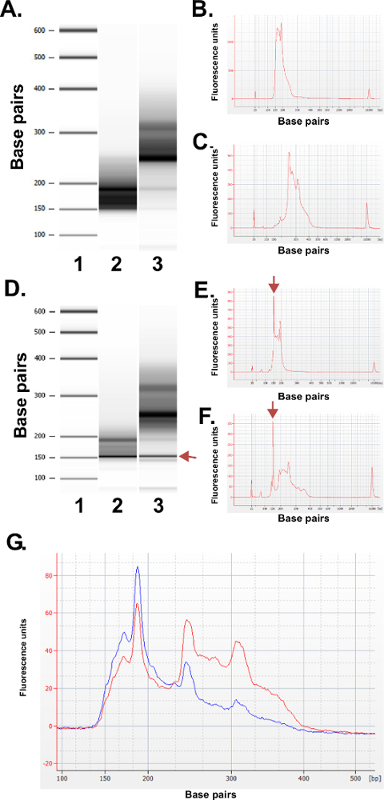
איור 3: תוצאות בקרת איכות לספריות ERRBS נציג הוכנו מדגימות DNA אנושיות באמצעות מכונה bioanalyzer. (א) תמונה דמוית ג'ל מראה סולם סטנדרטי (1), חלק תחתון ספרייה (135-240 נ"ב חלק מפיפין Prep); 2) ושבר ספרייה גבוה יותר (240-410 נ"ב חלק מפיפין Prep); . 3) (electropherogram B) Bioanalyzer של החלק תחתון הספרייה הצפויה (C) electropherogram Bioanalyzer של שבריר הספרייה גבוה יותר הצפוי D -.. F) נציגי נתונים מהכנת ספרייה באיכות ירודה. תמונה כמו ג'ל (D) של הסולם הסטנדרטי (1), חלק תחתון ספרייה (2) ושבר הספרייה גבוה יותר (3). הלהקה ב -150 נ"ב מסומנת בarrow מציין כמויות עודפות של מתאם. Electropherogram של נמוך (E) ושברים גבוהים ספרייה (F) עם פסגות המתאם העודפות ב 150 נ"ב (מסומן בחיצים). Electropherogram (G) Bioanalyzer של ספריית ERRBS נקוותה לסידור. עקבות אדומות מייצגת ספרייה ונקוותה באיכות גבוהה עם ייצוג שווה של שברים גבוהים יותר ונמוכים יותר. עקבות כחולות מייצגת ספרייה במאגר לא מתאים לסידור בשל היעדר הייצוג שווה של שברים גבוהים יותר ונמוכים יותר. לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
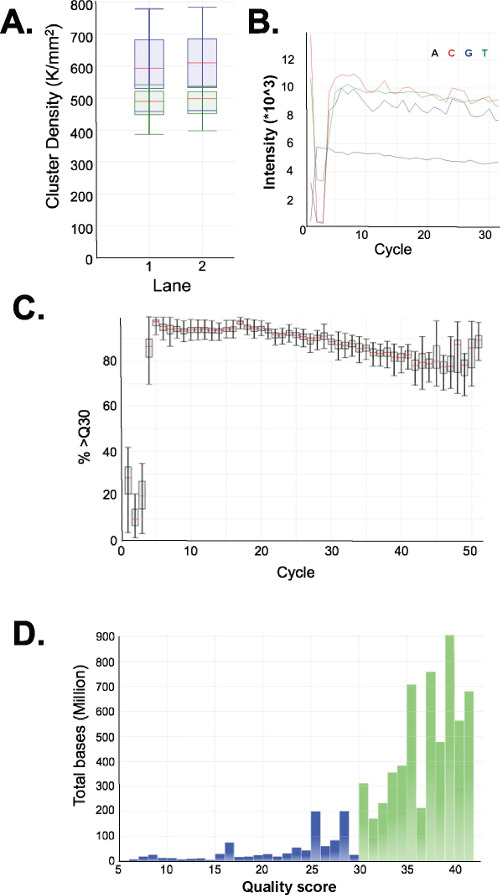
איור 4: רצף תרשימים לנציג ERRBS ריצת רצף קריאה יחיד 51-מחזור ברצף HiSeq 2500 בתפוקה גבוההמצב. (א) צפיפות אשכול (K / מ"מ 2 = 1,000 אשכולות לכל מילימטר מרובעים; כחולה). וצפיפויות אשכול עוברות סינון (ירוק) בשני מסלולים עם ספריות ERRBS (B) עוצמות אופייניות ראו ב -30 המחזורים הראשונים במסלול עם ספריית ERRBS. שים לב לחתימת CGG מעיכול MspI בעוצמות של שלושה המחזורים הראשונים. אחוז (C) של בסיסים עם ציון איכות של 30 ומעלה (%> Q30) לכל מחזור בנתיב ERRBS אחד. חלוקת ציון איכות (ד ') ל כל המחזורים בנתיב ERRBS אחד. כחול = פחות מ Q30, ירוק = גדול או שווה לQ30. במסלול זה, 84.7% מבסיסים היו ציוני איכות של 30 ומעלה.
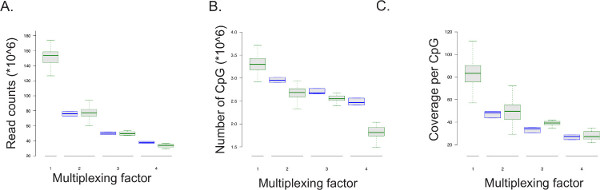
איור 5: רצף תוצאות פלט. חלקות תיבה של נתוני ניסוי ממדגם מרובב ויחיד לכל רצף נתיב ru ns (מוצג כקופסות ירוקות) ושל נתונים שהופקו על ידי downsampling המדומה מריצות רצף של שלוש ERRBS ספריות (מוצג כקופסות כחולות; נדגמו חמש פעמים לכל ריצת רצף) מ -51 מחזור רצף קריאה יחיד פועל גורם הריבוב מתאים. מספר ספריות ERRBS רצף לכל נתיב. 1 = כל נתיב או 100% מקורא ומייצג נתונים מספריית ERRBS אחת לכל נתיב; 2 = 50% מנתיב ומייצגים נתונים משני ERRBS ספריות לכל נתיב; 3 = 33% מנתיב ומייצגים נתונים משלוש ERRBS ספריות לכל נתיב; ו, 4 = 25% מנתיב ומייצגים נתונים מארבע ERRBS ספריות לכל נתיב. (א) סעיפי הקריאה, או מספר הרצפים ניתחו, לגורם ריבוב. (ב) מספר CPG של מכוסה בנתוני הרצף לריבוב גורם. (ג) הכיסוי הממוצע לCPG לגורם ריבוב._blank "> לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
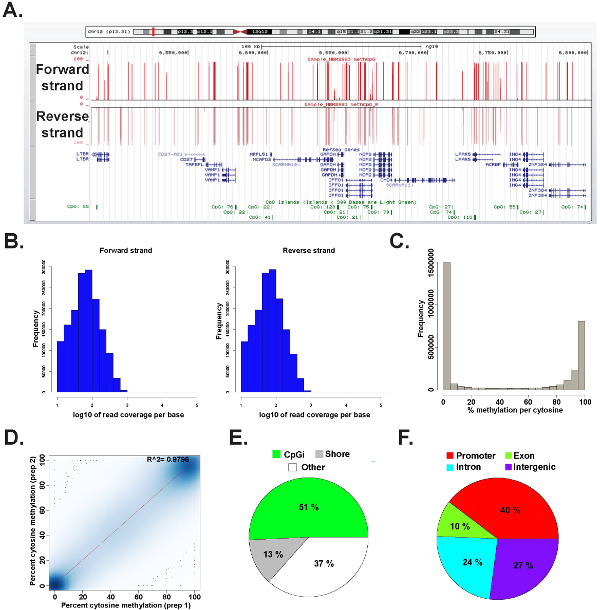
איור 6: נציגי נתונים מספריית ERRBS הוכנה מהדנ"א הגנומי אנושי () אוניברסיטת קליפורניה, סנטה קרוז (UCSC) דפדפן הגנום 43 תמונה של נתונים נציג מנתיב רצף ERRBS.. סרגל קנה המידה ציר y מייצג מתילציה% 0-100 בכל ציטוזין מכוסה עם מינימום של 10x. המסלול המותאם אישית העליון מייצג את הגדיל קדימה והמסלול המותאם אישית התחתון מייצג את הגדיל ההפוך. מוצג היא chr12:.. 6,489,523-6,802,422 (hg19) כולל של גני refseq ואיי CPG בתוך אזור גנומי זה היסטוגרמות הפצה (ב) לכיסוי CPG לאורך קדימה לאחור גדילים במדגם CD34 אדם + מח עצם נציג היסטוגרמה (C) הפצהרמות מתילציה CPG לאורך שני הגדילים במדגם CD34 אדם + מח עצם נציג. (ד) עלילת מתאם של רמות מתילציה CPG מהעתק טכני נציג של דגימת DNA אנושית. תרשים (E) פאי הממחיש את הפרופורציות של CpGs המכוסה בERRBS ש מבואר לאיי CPG (אור ירוק), CPG חופים (אפור) ואזורים אחרים (לבן) במדגם מייצג שהוכן מהדנ"א הגנומי אנושי. תרשים (F) פאי הממחיש את הפרופורציות של CpGs המכוסה בERRBS שמבואר ליזמי גן (אדום ), אקסונים (ירוק), אינטרונים (כחול) ואזורים חוץ-(סגולים). אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
| מֵגִיב | נפח | תגובה |
| 10x אנזים T4 DNA | 10 μl | |
| אדנוזין Deoxynucleotide (dNTP) הפתרון Mix | 4 μl | לערבב של 10 מ"מ של כל נוקלאוטיד |
| T4 DNA פולימראז | 5 μl | 3,000 יחידות / מיליליטר |
| קטע DNA פולימראז אני גדול (Klenow) | 1 μl | 5,000 יחידות / מיליליטר |
| T4 polynucleotide קינאז | 5 μl | 10,000 יחידות / מיליליטר |
| ללא מים DNase | 45 μl |
טבלת 1:. ריאגנטים סוף תגובת תיקון שמות מגיב וכמויות המשמשים בתגובת תיקון הסוף (שלב פרוטוקול 2.1).
| מֵגִיב | נפח | תגובה |
| מאגר תגובת 10x | 5 μl | למשל, NEBuffer 2 |
| 1 מ"מ 2'-deoxyadenosine 5'-טריפוספט (dATP) | 10 μl | |
| Klenow שבר (3 '→ 5' exo-) | 3 μl | 5,000 יחידות / מיליליטר |
טבלת 2:. ריאגנטים-עוקב תגובת שמות מגיב וכמויות המשמשים בתגובה עוקב-A (צעד פרוטוקול 3.1).
| מֵגִיב | נפח | תגובה |
| 15 מיקרומטר מתאמים מרותקים במים ללא DNase | 3 μl | מתאם 1.0 וPE מתאם PE 2.0; ראה טבלה 4 לרצפים והתייחסות |
| מאגר תגובת 10x אנזים T4 DNA | 581; l | |
| אנזים T4 DNA | 1 μl | 2,000,000 יחידות / מיליליטר |
| ללא מים DNase | 31 μl |
לוח 3: ריאגנטים מתאם קשירת תגובת שמות מגיב וכמויות המשמשות בתגובת קשירת מתאם (צעד פרוטוקול 4.2)..

לוח 4:. Oligos שימוש בפרוטוקול ERRBS רשימה של oligos בשימוש בכל פרוטוקול ERRBS בתגובת קשירה (צעד פרוטוקול 4) וצעדי PCR הגברה (צעד פרוטוקול 7).
| מֵגִיב | נפח | תגובה |
| wi מאגר תגובת 10x FastStart היי פידליטיה -18 מגנזיום כלוריד מ"מ | 20 μl | |
| 10 מ"מ dNTP פתרון Mix | 5 μl | |
| 25 מיקרומטר פריימר PCR PE 1.0 | 4 μl | ראה טבלת 4 |
| 25 מיקרומטר פריימר PCR PE 2.0 | 4 μl | ראה טבלת 4 |
| FastStart נאמנות גבוהה אנזים | 2 μl | 5 יחידות / μl FastStart תקי DNA פולימראז |
| ללא מים DNase | 125 μl |
לוח 5: ריאגנטים PCR תגובת שמות מגיב וכמויות המשמשות בתגובת PCR ההגברה (צעד פרוטוקול 7.1)..
| צעד פרוטוקול | מגיב / פירוט פרוטוקול | כמות ה- DNA קלט | ||
| 5-10 ng | 25 ng | 50 ng | ||
| 1 | אנזים MspI | 1 μl | 2 μl | 2 μl |
| MspI לעכל נפח תגובה | 50 | 100 | 100 | |
| 4 | מתאמים בתגובת קשירה | 1 μl | 2 μl | 3 μl |
| קשירת נפח תגובה | 20 μl | 25 μl | 50 μl | |
| 5 | פרוטוקול בחירת גודל | ג'ל הידני בלבד | פיפין Prep או ג'ל מדריך ל | פיפין Prep או ג'ל מדריך ל |
| 7 | ריכוז פריימר PCR | 25 מיקרומטר | 25 מיקרומטר | 10 מיקרומטר במשך 14 מחזורים; 25 מיקרומטר במשך 18 מחזורים |
| מספר מחזורי PCR | 18 | 18 | 14-18 | |
לוח 6:. שינויי פרוטוקול צעד לכמויות חומר הזנה החל 5-50 ng כמה צעדים לאורך כל הפרוטוקול מחייבים שינוי של כמויות מגיב משמשות ליצירת ספריות באיכות גבוהות מכמויות שונות של חומרי מוצא. שינויים בכמויות מגיב מפתח כלולים כאן. התאם חיץ ונפחי מים בתגובות בהתאם.
| Chr | בסיס | Strand | כיסוי | freqC | freqT |
| chr1 | 10564 | R | 366 | 85.52 | 14.48 |
| chr1 | 10,571 | F | 423 | 91.25 | 8.75 |
| chr1 | 10542 | F | 432 | 91.2 | 8.8 |
| chr1 | 10563 | F | 429 | 94.64 | 5.36 |
| chr1 | 10572 | R | 366 | 96.99 | 3.01 |
| chr1 | 10590 | R | 370 | 88.11 | 11.89 |
| chr1 | 10,526 | R | 350 | 92 | 8 |
| chr1 | 10543 | R | 368 | 92.93 | 7.07 |
| chr1 | 10525 | F | 433 | 91.92 | 8.08 |
| chr1 | 10497 | F | 435 | 88.74 | 11.26 |
לוח 7: נתונים ERRBS נציג. לאחר יישור נתונים ונחישות מתילציה ציטוזין, נתונים זוג הבסיס מתקבל למכוסה כל CPG, פרוטוקול היישור כפי שתואר יקבע לתאם (עמודות: chr = כרומוזום, בסיס וStrand) הגנומי., שיעור הכיסוי של המוקד הספציפי (סיקור ), ושיעור ציטוזין זיהוי לעומת thymidine כאחוז (freqC וfreqT בהתאמה).
| מספר ספריות ERRBS לכל נתיב | אומר מספר מיושר באופן ייחודי קורא | אומר מספר CpGs המכוסה | אומר כיסוי לCPG |
| 1 | 152231184 ± 13189678 | 3183594 ± 713547 | 85 ± 16 |
| 2 | 77680837 ± 7657058 | 2674823± 153494 | 49 ± 9 |
| 3 | 49938156 ± 2436865 | 2552186 ± - 76624 | 39 ± 2 |
| 4 | 34457208 ± 4441686 | 1814461 ± 144339 | 28 ± 4 |
לוח 8:. פרמטרים נציג מרצף ספריות ERRBS אחת ומרובבות מוצג נתונים לכל נתיב מריצות רצף קריאה אחת 51 מחזור: ממוצע וסטיית התקן של מיושר באופן ייחודי קורא, מספר CpGs מכוסה וכיסוי לכל אתר CPG מתקבל מרצף אחד ספריות ERRBS לכל נתיב (n = 100), שתי ERRBS ספריות לכל נתיב (n = 128), שלוש ERRBS ספריות לכל נתיב (n = 11), וארבע ERRBS ספריות לכל נתיב (n = 11).
Discussion
נתונים שהוצג בפרוטוקול החלטת תשואות בסיס-זוג מתילציה ציטוזין באזורים הגנומי ביולוגי-רלוונטיים. הפרוטוקול כפי שנכתב הוא מותאם עבור 50 ng של חומר מוצא, לעומת זאת, זה יכול להיות מותאם לטיפול במגוון רחב של חומר הזנה (ng 5 או יותר) 26. זה ידרוש התאמות של חלק מצעדי הפרוטוקול כפי שניתן לראות בטבלה 6. ספריות ERRBS נוחות לסידור הסוף לזווג וכיסוי הגנומי נוסף יכול גם להתבצע על ידי רצף קוראת יותר מ 51 מחזורים. רצף ריבוב יציע פרוטוקול עלות נמוך יותר לדגימה, לעומת זאת, זה יגרום לכיסוי מופחת לאתר CPG המיוצג בנתונים (איור 5 ולוח 8), ולא יניב עומק מספק של כיסוי לביצוע ניתוחים אשר דורשים כיסוי גבוה ל אתר CPG (למשל כפי שתואר על ידי הנדן et al. 33). לבסוף, פרוטוקול זה (או כל protoco מבוסס bisulfiteיב) לא יכול להבחין בין מתיל-ציטוזין וhydroxymethyl-ציטוזין 46,47. עם זאת, הנתונים שנוצרו יכול להיות משולב עם פרוטוקול אחר תוצאות 48,49 להתוות שינויים השונים, ושינויי ציטוזין אחרים דיווחו לאחרונה 50, הם צריכים להיות עניין.
ספריות באיכות גבוהות תופיע כפי שמוצג באיור 3 א-C, וברגע שאסף עבור סידור מניב עקבות כפי שמוצג באיור 3G (זכר אדום) המייצג את התרומות טוחנות שווה משני השברים הספרייה. כישלון הכנת ספרייה יכול לנבוע מכל צעד במהלך ההליך. אם DNA המושפל מעובד זה יגרום לספריות שאינם מועשרים שבברי MspI ומכאן בכיסוי CPG הנמוך באמצעות פרמטרי הרצף המתוארים בפרוטוקול זה. אם אנזים הוא שאינם פונקציונלי או לא נכלל בטעות מאחד מהתגובות, הפרוטוקול לא יניב הספרייה הצפויה. אם המחשבה קשירהction אינו יעיל, המתאמים נמצאים בריכוז גבוה מהצפוי, ו / או ריכוז פריימרים המשמש הוא מגיב להגבלת צעדי ההגברה הסופיות, כישלון ספרייה יכול להתרחש. מתאמים עודפים (נתפסו כפסגות בנ"ב ~ 150 בתוצאות bioanalyzer; איור 3D-F) בספרייה גם תפריע רצף בשל האשכולות חסרי הבחנה של שתי הספרייה ומתאמים עודפים. בעוד ספרייה כזו עשויה רצף כנראה בדרך כלל, חלק משמעותי מקורא יהיו רצפים רק מתאם. אם מתאמים עודפים הם נצפו בספרייה, דרך טובה ביותר הוא לחזור על הכנת הספרייה אם חומר נגיש באמצעות חומר הזנה אופטימלי למתאם יחס כמויות. לבסוף, על מנת להבטיח הגברה PCR יעילה של הספריות, השברים ספרייה הנמוכים וגבוהים נשמרים כדגימות נפרדות ברחבי המרת bisulfite וצעדי העשרת PCR. אם לא יעשה זאת מניבה יעילות ההפרש של הגברה בPתגובת CR של שברים גבוהים יותר ונמוכים יותר (כפי שניתן לראות באיור עקבות כחולות 3G) והפוטנציאל לייצוג שוויוני של הלוקוסים הגנומי בהתאמה מכוסות בכל שבריר ספרייה במהלך רצף. המשתמש יכול לבחור לכלול צעד PCR כמותי מייד לאחר המרת bisulfite לטיטרציה נוספת של מחזורי PCR אופטימליים הנחוצים כדי להגביר את הספריות שנוצרו.
יש פרוטוקול הכנת ספריית ERRBS כמה שלבים עיקריים שבי חומרים כימיים מסוימים מומלצים. בשלב הסוף-תיקון, השימוש בתערובת dNTP ארבעה נוקליאוטידים מאפשרת לקצה תיקון של כל מוצרים שאינו מכילים את הסככה CG, כגון אלה הנובעים מפעילות האנזימטית כוכב MspI ושברי DNA טעון בדגימת DNA המקורית. התוצאה היא ייצוג CPG שיפור בתוצאות. בשלב קשירה זה קריטי להשתמש האנזים ריכוז גבוה (2,000,000 יחידות / מיליליטר) ומתאמים מפוגלים כדי להבטיח שReacti קשירהבהוא יעיל ושמרת bisulfite אינה משפיעה על רצפי המתאם חיוניים ליישור נתונים מדויק. בצעד PCR, באמצעות פולימראז מסוגל הגברת שברי DNA GC-עשיר שטופל bisulfite הוא הכרחי עבור סגוליות גבוהה. לבסוף, כדי להבטיח חיסול מתאמים ופריימרים עודפים, טיהור SPRI חרוז (לדוגמא: Agencourt AMPure XP) מומלצת ולא מבוססים מבחני טור לקשירה ובידודי מוצר ה- PCR.
על מנת להפיק נתונים באיכות גבוהה, חשוב להבטיח המרת bisulfite יעילה. השליטה מוצגת מציעה למשתמש את היכולת לקבוע את יעילות המרה לפני הרצף. כחלופה, DNA שאינו בני אדם כמו למבדה DNA יכול לשמש כבקרה פנימית (ספייק-ב). בשל ההבדלים במין, זה סוג של שליטה יכול להיכלל ישירות ברצף במורד הזרם (למשל כפי שYu, et al. 34). עם זאת, אם אני ספייק-בs מנוצל, זה לא יכול לשמש כדי לקבוע את יעילות המרה לפני רצף ספרייה, אלא אם כן מוגבר באופן ייחודי ועצמאי רצף לפני רצף ספרייה. שיעורי ההמרה נקבעו מבוססים על מעמד מתילציה באתרים לא CPG. זה לא יכול להיות מתאים לשימוש בהקשר של מתילציה ציטוזין הגבוהה שבאינו CPG הקשר (למשל בתאי גזע עובריים) ודוגמאות מקבילות או באמצעים אחרים של הערכה ליעילות המרה יכולה להיות מנוצל למטרה זו.
יש כמה אזהרות לכתובת זו הנן ייחודיות לרצף של ספריות ERRBS. שלושה הבסיסים הראשונים של השברים ספריית רצף כמעט אחיד שאינו אקראי בשל אתר MspI הכרת החתך (C ^ CGG, ראו איור 4, C). תוצאות זה בפוטנציאל לאובדן נתונים משמעותי בשל איכות נמוכה קורא כתוצאה מלוקליזציה אשכול עני למרות צפיפות אשכול גבוהה לכאורה ברצף. כדי להתגבר על המכשול הזה,כולל ספריית מורכבות גבוהה בנתיב עצמאי (שליטת PhiX או סוג ספרייה אחר) כנתיב שליטה ייעודי. יש ספריות מורכבות גבוהות קצוות המכילים ייצוג מאוזן של A, C, T ו- G בארבעת הבסיסים הראשונים רצף. נתיבי בקרה נאותים כוללים ספריות כגון RNA-seq, שבב seq, רצף הגנום כולו, או שליטה המוצעת על ידי היצרן המכונה רצף (למשל PhiX בקרת v3). כאשר יועד כנתיב שליטה לריצת הרצף המתאים, היא יכולה לשמש כבסיס לדור המטריצה המנוצל בארבעת הבסיסים הראשונים של רצף כדי לזהות עמדות אשכול. האיכות גבוהה יותר קוראה נתפסה תעלה את ממוצע הכיסוי לכל אתר על ידי CPG 5.2 (n = 4). לחלופין, קושי טכני זה יכול להיות גם להתגבר באמצעות גישת רצף כהה כפי שתואר קודם לכן 23. קריטריונים לקביעת רצף אחרים בצעו נהלי עבודה לפרוטוקולים של היצרן. לבסוף, הכיסוי לCPG גחוסן לניתוח נתונים ותונחה על ידי המשתמש ובחלקו על ידי השאלות הביולוגיות של עניין. סף כיסוי 10x מאפשר גישת ניתוח כיסוי גבוה, אולם סף זה יכול להיות הוריד צריך שיהיה העניין.
דיון מלא בניתוח נתונים ERRBS הוא מעבר להיקף של מאמר זה, עם זאת, באופן דיפרנציאלי cytosines ואזורים מפוגלים ניתן לקבוע באמצעות כלים קוד פתוח 31,51-53. שיקולי ניתוח נוספים וגישות שתוארו היטב 54,55, והקורא מוזמן לחפש הספרות בכלים המתאימים ביותר לניתוח המתוכנן.
בהשוואה לשיטות אחרות שפורסמו, ERRBS מציע פרוטוקול בן ארבעה ימים שכאשר הופיע כתשואות מתוארות שיעור גבוה של שחזור. שיטה זו הוכחה בהשוואה לזהב הסטנדרטי MassARRAY EpiTYPER 26, הוא חסכוני לנתוני כיסויים גבוהים, וניתן להתאמה לחומר הזנה שונותכמויות (נוחים לסוגי עיבוד מדגם קליני ותאים אחרים של תדירות נמוכה) וגישות רצף. הוא מציע רזולוציה בסיסית-זוג בלוקוסים רלוונטיים מבחינה ביולוגית וניתן להשתמש בו באינטגרטיבית מנתח עם טכניקות אחרות פרופיל גורם הגנום שעתוק מחייב, שיפוץ הכרומטין, סימנים אפיגנטיים ושינויי ציטוזין אחרים בעלי עניין. שימוש בנתוני ERRBS במחקרים מסוג זה יכול לתרום לגישה מולקולרית מקיפה ולאפשר לממדים גבוהים ניתוחים במחקר של מודלים ביולוגיים ומחלות של בני אדם.
Disclosures
יש לי המחברים אין ניגודי אינטרסים לחשוף.
Acknowledgements
We thank all the authors of the original ERRBS report. We thank Mame Fall for technical assistance. We acknowledge the Weill Cornell Medical College Epigenomics Core for technical services and assistance. The work was supported by a Sass Foundation Judah Folkman Fellowship, an NCI K08CA169055 and ASH-AMFDP12005 to FGB, NIH R01HG006798 and R01NS076465, funding from the Irma T. Hirschl and Monique Weill-Caulier Charitable Trusts and STARR Consortium (I7-A765) to CEM, and an LLS SCORE grant (7006-13) to AMM.
Materials
| Name | Company | Catalog Number | Comments |
| MspI | New England Biolabs | R0106M | 100,000 units/ml |
| NEBuffer 2 | New England Biolabs | B7002S | Reaction buffer for MspI enzyme; protocol step 1.2 |
| Phenol solution | Sigma-Aldrich | P4557 | Equilibrated with 10 mM Tris HCl, pH 8.0; see safety and handling instructions at http://www.sigmaaldrich.com/catalog/product/sigma/p4557 |
| Chloroform | Sigma-Aldrich | C2432 | See safety and handling instructions at http://www.sigmaaldrich.com/catalog/product/sial/c2432 |
| Glycogen | Sigma-Aldrich | G1767 | 19-22 mg/ml |
| NaOAc | Sigma-Aldrich | S7899 | 3 M, pH 5.2 |
| Ethanol | Sigma-Aldrich | E7023 | 200 proof, for molecular biology |
| Buffer EB | Qiagen | 19086 | 10 mM Tris-Cl, pH 8.5 |
| tris(hydroxymethyl)aminomethane (Tris) | Sigma-Aldrich | T1503 | prepare a 1 M, pH 8.5 solution |
| Tris- Ethylenediaminetetraacetic acid (TE) | Sigma-Aldrich | T9285 | Dilute to 1x buffer solution per manufacturer's recommendations |
| T4 DNA Ligase Reaction Buffer | New England Biolabs | B0202S | 10x concentration |
| Deoxynucleotide triphosphate (dNTP) Solution Mix | New England Biolabs | N0447L | 10 mM each nucleotide |
| T4 DNA Polymerase | New England Biolabs | M0203L | 3,000 units/ml |
| DNA Polymerase I, Large (Klenow) Fragment | New England Biolabs | M0210L | 5,000 units/ml |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | 10,000 units/ml |
| QIAquick PCR Purification Kit | Qiagen | 28104 | Used for DNA product purification in protocol step 2.3 |
| 2'-deoxyadenosine 5'-triphosphate (dATP) | Promega | U1201 | 100 mM |
| Klenow Fragment (3'→5' exo-) | New England Biolabs | M0212L | 5,000 units/ml |
| MinElute PCR Purification Kit | Qiagen | 28004 | Used for DNA product purification in protocol step 3.3 |
| T4 DNA Ligase | New England Biolabs | M0202M | 2,000,000 units/ml |
| Methylation Adapter Oligo Kit | Illumina | ME-100-0010 | |
| Agencourt AMPure XP | Beckman Coulter | A63881 | Used in protocol sections that implement magnetic bead purification steps (steps 4.3 and 8.2). Equilibrate to room temperature before use. |
| Pippin Prep Gel Cassettes, 2% Agarose, dye-free | Sage Science | CDF2010 | with internal standards |
| Certified Low Range Ultra Agarose | Bio-Rad | 161-3106 | |
| Tris-Borate-EDTA (TBE) buffer | Sigma-Aldrich | T4415 | |
| Ethidium bromide solution | Sigma-Aldrich | E1510 | 10 mg/ml |
| 50 bp DNA Ladder | NEB | N3236S | |
| 100 bp DNA Ladder | NEB | N3231S | |
| Gel Loading Dye, Orange (6x) | NEB | B7022S | |
| Scalpel Blade No. 11 | Fisher Scientific | 3120030 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| EZ DNA Methylation Kit | Zymo Research | D5001 | Used in protocol step 6.2 |
| EZ DNA Methylation-Lightning Kit | Zymo Research | D5030 | Alternative for step 6.2 |
| Universal Methylated Human DNA Standard | Zymo Research | D5011 | Used as bisulfite conversion control |
| FastStart High Fidelity PCR System | Roche | 03553426001 | |
| Qubit dsDNA High Sensitivity Assay Kit | Life Technologies | Q32854 | A fluorescence-based DNA quantitation assay; used in protocol steps 1.1, 9.1 and 10.1 |
| DynaMag-2 Magnet | Life Technologies | 12321D | |
| High Sensitivity DNA Kit | Agilent Technologies | 5067-4626 | |
| 2100 Bioanalyzer | Agilent Technologies | ||
| PhiX Control v3 | Illumina | FC-110-3001 | |
| HiSeq 2500 | Illumina | ||
| Pippin Prep | Sage Science | ||
| Qubit 2.0 Fluorometer | Life Technologies | Q32872 | |
| TruSeq SR Cluster Kit v3-cBot-HS | Illumina | GD-401-3001 | |
| TruSeq SBS Kit v3-HS | Illumina | FC-401-3002 | |
| TruSeq RNA Sample prep | Illumina | RS-122-2001 | Barcoded adapters used for multiplexing libraries; See Supplemental file for multiplexing protocol. |
| Microcentrifuge | |||
| Vortex Mixer | |||
| Dry Block Heater | |||
| Thermal Cycler | |||
| Water Bath | |||
| Gel electrophoresis system | |||
| Electrophoresis power supply | |||
| Gel doc | |||
| UV or blue light transilluminator |
References
- Jones, P. A. Functions of DNA methylation: islands, start sites, gene bodies and beyond. Nat Rev Genet. 13 (7), 484-492 (2012).
- Barlow, D. P. Genomic imprinting: a mammalian epigenetic discovery model. Annual Review Of Genetics. 45, 379-403 (2011).
- Thiagarajan, R. D., Morey, R., Laurent, L. C. The epigenome in pluripotency and differentiation. Epigenomics. 6 (1), 121-137 (2014).
- Reik, W. Stability and flexibility of epigenetic gene regulation in mammalian development. Nature. 447 (7143), 425-432 (2007).
- Hartnett, L., Egan, L. J. Inflammation, DNA methylation and colitis-associated cancer. Carcinogenesis. 33 (4), 723-731 (2012).
- Smith, Z. D., Meissner, A. DNA methylation: roles in mammalian development. Nat Rev Genet. 14 (3), 204-220 (2013).
- Li, E., Bestor, T. H., Jaenisch, R. Targeted mutation of the DNA methyltransferase gene results in embryonic lethality. Cell. 69 (6), 915-926 (1992).
- Okano, M., Bell, D. W., Haber, D. A., Li, E. DNA methyltransferases Dnmt3a and Dnmt3b are essential for de novo methylation and mammalian development. Cell. 99 (3), 247-257 (1999).
- Feinberg, A. P. Phenotypic plasticity and the epigenetics of human disease. Nature. 447 (7143), 433-440 (2007).
- Bock, C. Epigenetic biomarker development. Epigenomics. 1 (1), 99-110 (2009).
- Laird, P. W. The power and the promise of DNA methylation markers. Nat Rev Cancer. 3 (4), 253-266 (2003).
- How Kit, A., Nielsen, H. M., Tost, J. DNA methylation based biomarkers: practical considerations and applications. Biochimie. 94 (11), 2314-2337 (2012).
- Mikeska, T., Bock, C., Do, H., Dobrovic, A. DNA methylation biomarkers in cancer: progress towards clinical implementation. Expert Review Of Molecular Diagnostics. 12 (5), 473-487 (2012).
- Gyparaki, M. T., Basdra, E. K., Papavassiliou, A. G. DNA methylation biomarkers as diagnostic and prognostic tools in colorectal cancer. Journal of Molecular Medicine. 91 (11), 1249-1256 (2013).
- Figueroa, M. E., et al. DNA methylation signatures identify biologically distinct subtypes in acute myeloid leukemia. Cancer Cell. 17 (1), 13-27 (2010).
- Heyn, H., Mendez-Gonzalez, J., Esteller, M. Epigenetic profiling joins personalized cancer medicine. Expert review of Molecular Diagnostics. 13 (5), 473-479 (2013).
- Kulis, M., Esteller, M. DNA methylation and cancer. Advances in Genetics. 70, 27-56 (2010).
- Xiong, Z., Laird, P. W. COBRA: a sensitive and quantitative DNA methylation assay. Nucleic Acids Res. 25 (12), 2532-2534 (1997).
- Meissner, A., et al. Reduced representation bisulfite sequencing for comparative high-resolution DNA methylation analysis. Nucleic Acids Res. 33 (18), 5868-5877 (2005).
- Gu, H., et al. Preparation of reduced representation bisulfite sequencing libraries for genome-scale DNA methylation profiling. Nat Protoc. 6 (4), 468-481 (2011).
- Bock, C., et al. Quantitative comparison of genome-wide DNA methylation mapping technologies. Nat Biotechnol. 28 (10), 1106-1114 (2010).
- Harris, R. A., et al. Comparison of sequencing-based methods to profile DNA methylation and identification of monoallelic epigenetic modifications. Nat Biotechnol. 28 (10), 1097-1105 (2010).
- Boyle, P., et al. Gel-free multiplexed reduced representation bisulfite sequencing for large-scale DNA methylation profiling. Genome Biol. 13 (10), R92 (2012).
- Chatterjee, A., Rodger, E. J., Stockwell, P. A., Weeks, R. J., Morison, I. M. Technical considerations for reduced representation bisulfite sequencing with multiplexed libraries. Journal of Biomedicine & Biotechnology. 2012, 741542 (2012).
- Lee, Y. K., et al. Improved reduced representation bisulfite sequencing for epigenomic profiling of clinical samples. Biological Procedures Online. 16 (1), 1 (2014).
- Akalin, A., et al. Base-pair resolution DNA methylation sequencing reveals profoundly divergent epigenetic landscapes in acute myeloid leukemia. PLoS Genet. 8 (6), e1002781 (2012).
- Hatzi, K., et al. A Hybrid Mechanism of Action for BCL6 in B Cells Defined by Formation of Functionally Distinct Complexes at Enhancers and Promoters. Cell Reports. 4 (3), 578-588 (2013).
- Will, B., et al. Satb1 regulates the self-renewal of hematopoietic stem cells by promoting quiescence and repressing differentiation commitment. Nature Immunology. 14 (5), 437-445 (2013).
- Lu, C., et al. Induction of sarcomas by mutant IDH2. Genes Dev. 27 (18), 1986-1998 (2013).
- Kumar, R., et al. AID stabilizes stem-cell phenotype by removing epigenetic memory of pluripotency genes. Nature. 500 (7460), 89-92 (2013).
- Li, S., et al. An optimized algorithm for detecting and annotating regional differential methylation. BMC Bioinformatics. 14, S10 (2013).
- Patterson, K., Molloy, L., Qu, W., Clark, S. DNA methylation: bisulphite modification and analysis. Journal of Visualized Experiments. (56), 3170 (2011).
- Landan, G., et al. Epigenetic polymorphism and the stochastic formation of differentially methylated regions in normal and cancerous tissues. Nat Genet. 44 (11), 1207-1214 (2012).
- Yu, M., et al. Tet-assisted bisulfite sequencing of 5-hydroxymethylcytosine. Nat Protoc. 7 (12), 2159-2170 (2012).
- Goecks, J., Nekrutenko, A., Taylor, J., Galaxy, T. Galaxy: a comprehensive approach for supporting accessible, reproducible, and transparent computational research in the life sciences. Genome Biol. 11 (8), R86 (2010).
- Dorff, K. C., et al. GobyWeb: simplified management and analysis of gene expression and DNA methylation sequencing data. PLoS One. 8 (7), e69666 (2013).
- Roehr, J. T., Dodt, M., Ahmed, R., Dieterich, C. Flexbar − flexible barcode and adapter processing for next-generation sequencing platforms. MDPI Biology. 1 (3), 895-905 (2012).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal, North America. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Needleman, S. B., Wunsch, C. D. A general method applicable to the search for similarities in the amino acid sequence of two proteins. J Mol Biol. 48 (3), 443-453 (1970).
- Krueger, F., Andrews, S. R. Bismark: a flexible aligner and methylation caller for Bisulfite-Seq applications. Bioinformatics. 27 (11), 1571-1572 (2011).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Kent, W. J., et al. The human genome browser at UCSC. Genome Res. 12 (6), 996-1006 (2002).
- Thorvaldsdottir, H., Robinson, J. T., Mesirov, J. P. Integrative Genomics Viewer (IGV): high-performance genomics data visualization and exploration. Briefings in Bioinformatics. 14 (2), 178-192 (2013).
- Team, R. C. R. A language and environment for statistical computing. R Foundation for Statistical Computing. Vienna, Austria. ISBN 3-900051-07-0, (2012).
- Nestor, C., Ruzov, A., Meehan, R., Dunican, D. Enzymatic approaches and bisulfite sequencing cannot distinguish between 5-methylcytosine and 5-hydroxymethylcytosine in DNA. BioTechniques. 48 (4), 317-319 (2010).
- Huang, Y., et al. The behaviour of 5-hydroxymethylcytosine in bisulfite sequencing. PLoS One. 5 (1), e8888 (2010).
- Yu, M., et al. Base-resolution analysis of 5-hydroxymethylcytosine in the mammalian genome. Cell. 149 (6), 1368-1380 (2012).
- Song, C. X., et al. Genome-wide profiling of 5-formylcytosine reveals its roles in epigenetic priming. Cell. 153 (3), 678-691 (2013).
- Ito, S., et al. Tet proteins can convert 5-methylcytosine to 5-formylcytosine and 5-carboxylcytosine. Science. 333 (6047), 1300-1303 (2011).
- Akalin, A., et al. methylKit: a comprehensive R package for the analysis of genome-wide DNA methylation profiles. Genome Biol. 13 (10), R87-1186 (2012).
- Stockwell, P. A., Chatterjee, A., Rodger, E. J., Morison, I. M. DMAP: Differential Methylation Analysis Package for RRBS and WGBS data. Bioinformatics. 30 (13), 1814-1822 (2014).
- Sun, D., et al. MOABS: model based analysis of bisulfite sequencing data. Genome Biol. 15 (2), R38 (2014).
- Bock, C. Analysing and interpreting DNA methylation data. Nat Rev Genet. 13 (10), 705-719 (2012).
- Rivera, C. M., Ren, B. Mapping human epigenomes. Cell. 155 (1), 39-55 (2013).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved