Method Article
Enhanced приведенного представления Бисульфитное секвенирование по оценке метилирования ДНК в паре оснований резолюции
* Эти авторы внесли равный вклад
В этой статье
Резюме
Enhanced Reduced Representation Bisulfite Sequencing is a method for the preparation of sequencing libraries for DNA methylation analysis based on restriction enzyme digestion combined with cytosine bisulfite conversion. This protocol requires 50 ng of starting material and yields base pair resolution data at GC-rich genomic regions.
Аннотация
ДНК отображение метилирование сильно учился в нормальных и больных тканей. Разнообразие методов были созданы, чтобы допросить шаблоны цитозин метилирования в клетках. Снижение представление целого генома бисульфита последовательности была разработана, чтобы обнаружить количественные пар оснований узоры разрешение цитозин метилирования в GC-богатых локусов генома. Это достигается путем объединения использование фермента рестрикции с последующим бисульфита преобразования. Улучшенная Снижение Представительство бисульфит Секвенирование (ERRBS) увеличивает биологически соответствующий локусов генома закрытых и был использован в профиль цитозин метилирование ДНК от человека, мыши и других организмов. ERRBS инициирует с рестриктазой ДНК, чтобы генерировать низкомолекулярных фрагментов для использования в библиотеке препарата. Эти фрагменты подвергаются стандартной конструкции библиотеки для следующего поколения секвенирования. Бисульфит превращение неметилированных цитозинов до окончательного amplificatiна этапе позволяет количественно базовой разрешением цитозина уровнях метилирования в крытых локусов генома. Протокол может быть завершена в течение четырех дней. Несмотря на низкую сложности в течение первых трех баз секвенированных, ERRBS библиотеки дают высокое качество данных при использовании назначенным контролировать выполнение последовательности полосу. Картирование и биоинформатики Анализ затем выполняется, и данные дает, что может быть легко интегрирован с различными генома платформ. ERRBS могут использовать небольшие вход количества материалов делает возможным обрабатывать клинические образцы человека и могут применяться в различных научно-исследовательских приложений. Полученное видео демонстрирует важные шаги протокола ERRBS.
Введение
Метилирование ДНК в цитозина (5-метилцитозина) является критическим эпигенетическое знак в клетках млекопитающих для различных биологических процессах, в том числе, но не ограничиваясь импринтинга, инактивации Х-хромосомы, развития и регуляции экспрессии генов 1-8. Исследование метилирования ДНК в злокачественных и других расстройств определяется по определенному заболеванию образцы и вклад в понимание патогенеза заболевания и потенциальных биомаркеров открытий 9-17. Есть большое количество протоколов, допросить эпигеном для статуса метилирования ДНК. Они могут быть разделены на основе сродства, рестриктазы основе, и бисульфит преобразования на основе анализов, которые используют микрочипов или секвенирования платформы вниз по течению. Кроме того, есть несколько протоколов, которые преодолеть все эти общие категории, включая, но не ограничиваясь, в сочетании бисульфита анализа Ограничение 18 и приведенного представления Бисульфитное секвенирование (RRBS 19).
RRBS первоначально был описан Мейснер и др. 19,20. Протокол введены шаг обогащать GC-богатых областей генома с последующим бисульфита последовательности, в результате чего с количественным разрешения данных пар оснований, что является экономически эффективным 21,22. ГХ-богатых областей ориентированы на MspI (С ^ CGG) рестриктазы и цитозин метилирование разрешается бисульфита преобразования цитозина (дезаминирование немодифицированных цитозина в урацил), а затем с помощью полимеразной цепной реакции (ПЦР). RRBS охватывает большинство промоторов генов и CpG островов в фракции секвенирования, необходимого для целого генома; однако RRBS имели ограниченный охват CpG берегов и других межгенных биологического актуальность. Несколько групп опубликовала обновленные RRBS протоколы с оригинальной докладе, что улучшить его методологии и, как следствие освещения этих областей генома 23-25. Улучшенная Снижение Представительство бисульфит Sequencing (ERRBS) включает в себя библиотеки модификации подготовки и альтернативный выравнивание данных подход 26 по сравнению с RRBS. ERRBS привело к увеличению числа CpGs, представленных в данных, полученных и увеличение охвата всех областей генома допрашивали 26. Этот метод был использован для решения метилирования ДНК в населенных пациента и других образцов животных 26-30.
Протокол ERRBS описано предложения подробную информацию о всех шагов, необходимых для завершения и данные были получены с использованием репрезентативных ДНК человека (образцы были получены из сообщалось ранее, обезличенной образцов пациентов 31 и CD34 + костного образца костного мозга от нормального человеческого донора). Протокол включает в себя автоматический процесс выбора размера, что уменьшает время обработки на образец и позволяет повысить точность в библиотеке выбора размера. Протокол сочетает в себе ряд установленных методов молекулярной биологии. Высокомолекулярной ДНК вес переваривается WIth метилирования без учета регистра рестриктазой (МГПИ), а затем к концу ремонта, A-хвостов и перевязки метилированных адаптеров. Выбор Размер GC-богатых фрагментов с последующим бисульфита преобразования и ПЦР-амплификации до секвенирования. Бисульфит преобразования был описан ранее 32 и подробный обзор анализ и его приложения данных выходит за рамки данной статьи, однако рекомендации и ссылки включены для использования читателей. Протокол может быть выполнена в течение четырех дней и поддается небольшой вход (50 нг или менее) Материал количествах. Протокол, как описывалось урожайности данных с высоким уровнем охвата в CpG сайт достаточном не только для дифференциальных метилирование сайта и области определения, но и для эпигенетической обнаружения полиморфизма, как описано Landan и др. 33.
протокол
Все процедуры выполняются утверждаются университета Индианы школы медицины Уходу за животными и использованию комитета и следуйте Национальный институт инструкций по охране.
1. Хирургическая техника
- Поддерживать асептики во время этой процедуры с помощью стерильных перчаток, инструментов и стерильных операционного поля в соответствии с NIH руководящих принципов 25. Стерилизовать инструменты перед началом операции по стерилизации в автоклаве их (см таблицу специфических реагентов / Оборудование для полного списка). Используйте стеклянная бусина стерилизатора для стерилизации инструментов во время операции.
2. Анестезия и подготовка
- Обезболить мышь в анестезии поле со смесью 0,9 л / мин кислорода и 2,5% изофлуран использованием ветеринарной системы ИФ испаритель. Убедитесь, что мышь не реагируют на изменения положения тела, прежде чем снимать его из коробки.
- Применить глазной мази на муглаза SE, чтобы защитить их от высыхания.
- Переключение потока газа из коробки с носовым конусом. Наведите прямо на его левой стороне на нагретой площадки, покрытой с хирургической площадки и абсорбирующим скамейке бумаги с его носа и рта внутри конуса. Непрерывный мониторинг ритм дыхания мышки и скорость и отрегулировать изофлурановой уровней по мере надобности (от 2,5 - 3% ИФ), чтобы поддерживать адекватный уровень анестезии, а также использовать ног щепотку рефлекс, чтобы подтвердить общее успокоение.
3. Хирургическая тактика
- Выравнивание и сосредоточиться стереоскоп с операционного поля. Регулировка носовой конус и прикрепить ее вниз так, чтобы она располагалась по краю поля зрения.
- С мыши лежа на левом боку, лента край правого уха к носу конуса, обнажая область за ухом, где разрез будет сделан. Убедитесь, что задней ушной вены проходит горизонтально через ухо. Следует отметить, что правильное размещение тон животное и лентой уха имеют решающее значение для того, чтобы быстро найти лицевого нерва.
- Смочите шерсть на и за ухом с 70% этанола и брить хирургического сайт, используя бритву или лезвие скальпеля. Предварительное смачивание мех делает бритье легче в этом анатомического расположения.
- Протереть кожу раствором йода, такие как Бетадин хирургических скраб (7,5% повидон-йод), а затем с помощью 70% этанола. Повторите этот чистка еще два раза, чтобы тщательно дезинфицировать области.
- Чтобы определить, где сделать надрез, проследить задней ушной вены из уха каудально к области боковых зубов в ухо выступа. Использование пружинных ножниц, сделать 4 мм разрез 2 - мм кзади 3 к выпуклости.
- Проанализируйте через подкожно-жировой клетчатки и фасции с использованием тупым. Избегайте прямого Резка ножницами, потому что кровеносные сосуды или в мышечную ткань может быть легко повреждены.
- Если кровотечение происходит, оказать давление на участке хирургического вмешательства с помощью стерильного ватного тампонав течение по меньшей мере 30 сек. Если происходит существенная потеря жидкости, вводят мыши внутрибрюшинно до 0,5 мл стерильной 0,9% солевого раствора с использованием 25 или 27 G иглу.
- Используйте несколько ключевых достопримечательностей, спинного нерва, наружный слуховой проход, а впереди двубрюшная мышцы (как описано ниже), чтобы найти лицевого нерва. Проанализируйте вокруг этих ориентиров, пока ветви лицевого нерва не визуализируется. Нерв появляется в качестве существенного твердого белого структуры, когда это показано, и слой прилипает фасции его основных структур.
- Найти спинного нерва, который проходит от каудальной части черепа для иннервации трапециевидной мышцы, как только подкожный жир и фасции были расчленены. Лицевой нерв является глубоко, чтобы спинного нерва.
- Найти хрящевой ушной канал, который выглядит жемчужно-белая, и можно увидеть ростральнее лицевого нерва.
- Найти мышц живота в передней двубрюшной мышцы, которая находится в верхней части и сaudal лицевого нерва.
- Когда основные ветви лицевого нерва визуализировать, отслеживать их спинки, чтобы найти свое происхождение от stylomastoid отверстия. Использование тонких наконечником Дюмон щипцы # 5/45 провести хирургическую сайт открыт, продвижения весной ножницами кончики следующие пути нерва, а затем перейти щипцы дорсально, чтобы вновь передовые область открыта.
- Представьте ствол лицевого нерва с скуловой, щечной и предельные нижней челюсти ветви в этой точке.
ПРИМЕЧАНИЕ: височной ветви будут найдены ближе к отверстию. Краевые нижнечелюстного нерва разветвляется на его верхней и нижней частей ближе к челюсти, таким образом, эти нервные ветви не будет видна на этом уровне.- Если выполнение пересечения нерва, стабилизировать нерв аккуратно с помощью тоненького кончика пинцетом и перерезать нерв с помощью пружинных ножниц. Избегайте применения слишком много сцепления к нерву с помощью пинцета, чтобы предотвратить avulsing нерв из мозга. От Себяпни друг от друга, или вырезать и удалить часть дистального нерва, чтобы гарантировать, что не может произойти повторное подключение.
- При выполнении травмы раздавить, использовать Дюмон # 5/45 щипцы для сжатия нерва в течение 30 сек с использованием постоянного давления разорвать все аксоны, а затем повторите этот влюблен под вторым углом перпендикулярно к первой раздавить сайте. Избегайте применения различное количество давления при 30 сек давке, в противном случае травмы будут несовместимы между животными.
4. Закрытие и восстановление
- Повторно жир и мышцы над базовых структур.
- Приблизительная края разреза и закрыть рану с помощью 7,5 мм, свернутой клип. Швы или клей также являются приемлемыми для закрытия раны. Послеоперационные анальгетики могут быть предоставлены в это время.
- Удалите ленту от уха мыши. Выключите поток ИФ и позволяют мышь, чтобы дышать чистым кислородом в течение 30 сек до 1 мин. Plтуз мышь в пустую клетку, не постельные принадлежности, чтобы оправиться от наркоза.
- Когда мышь восстанавливается, изучить его поведение подтверждающих признаков паралича лицевого нерва. Усы будет парализована и под углом назад по направлению к щеке, нос сместятся, и глаз не будет мигать в ответ на облаке воздуха.
- Дом животные совместно после операции, если они являются женщинами. Избегайте жилье самцов мышей совместно, потому что они более агрессивны и склонны к принудительной высылке раны клипы их cagemate, которая приводит к инфекции. Обеспечить послеоперационные анальгетиков в это время, если это необходимо.
- Монитор мышей раз в день в течение нескольких дней после операции, чтобы гарантировать, что никакая инфекция или другое осложнение не происходит после операции. Удалить раны клипы 7 - 10 дней после операции, если они не упали на свои собственные.
- Применить смазочных глазную мазь, чтобы пораженный глаз ежедневно, чтобы предотвратить роговицы осложнений, либо до тех пор, моргание глазом рефлекс не будет сноване покрыты или пока эвтаназии.
Результаты
Рисунок 1 представляет собой обзор ERRBS, выделяя основные этапы, которые объясняются в течение протоколу, описанному. ERRBS библиотеки были подготовлены с использованием входного ДНК 50 нг.
Оцените качество библиотек, полученных. Библиотека производство обычно дает размеры фракции 150-250 б.п. и 250-400 б.п. (3А-С). Небольшие различия в библиотеке распределения по размерам между образцами ожидается. Следует отметить, что в обоих нижних и высших фракций библиотеки есть очень интенсивные размеры ДНК, что свидетельствует о обогащение определенной последовательности. МГПИ пищеварения приводит к обогащению семейства повторяющихся последовательностей ДНК в человеческом геноме в 190 б.п., 250 б.п. и 310 б.п. в ERRBS библиотек. Эти три повторы представляют собой характерный подпись библиотеке ERRBS 20 (см 3А-C и 3G). Типичные библиотеки секвенировали на последовательности следующего поколенияR с помощью одного конца читает. При загрузке в рекомендуемой концентрации библиотеки на Ан Illumina HiSeq 2500 секвенсор, кассетные плотности 500,000-700,000 на мм 2 ожидается. При такой плотности кластеров, 81,6% ± 3,14% (N = 81) кластеров Фильтр низких частот (рис 4а). Из-за низкой конце сложность библиотеки вставками (сайт узнавания MspI: C ^ CGG), значения интенсивности и оценки качества записанных во время секвенирования весьма разнообразны в течение первых трех оснований (рис 4B-C), однако, если независимый контроль пер включен (см обсуждения), 85% из баз будет иметь качество набрали 30 или более (значения Q30; рис 4D).
Выравнивание данных и определение цитозин метилирование, как описано в данных разрешением дает протокола пар оснований (таблица 7). Для человеческого генома, 51-цикл одного чтения последовательности выполнения из библиотеки ERRBS в одну из полос HiSeq 2500 в режиме высокой выходной регулярноLY генерирует 153194882 ± 12918302 Общее говорится, что после того, как качество фильтрации и адаптер обрезки урожайность 152231183 ± 13189678 читает для ввода в трубопровод анализа. Средняя эффективность отображение для ERRBS библиотеки, как правило, 62,95% ± 5,92% с представлением 3183594 ± 713547 CpGs с минимальным покрытием в CpG 10х и средней покрытия на ЦГ 84,94 ± 16,29 (п = 100).
Протокол ERRBS поддается мультиплексирования (см Дополнительный файл 1: Протокол приспособление для мультиплексной последовательности). Данные представительства последовательности бежит приводится на рисунке 5 данные из уплотненных работает секвенирования (51 цикла одного чтения секвенирования перспективе;. N = 128 для двух библиотек на одну полосу движения, N = 11 для трех библиотеках, на одну полосу движения, N = 11 для четырех библиотек на полосу) по сравнению с полной полосы секвенирования с ERRBS библиотеки (51 цикла одно- читать работает секвенирования; N = 100), а также понижение разрешения ни переулков, чтобы SimulatE 50%, 33% и 25% читает на полосу (2, 3, и 4 образца мультиплексирования на полосу, соответственно; п = 3). Как число просмотров за образца уменьшается с коэффициентом мультиплексирования, количество CpGs охваченных при минимальном освещении 10x и освещения в CpG уменьшается, а (рис 5 и в таблице 8). Средние курсы пересчета сайтов без CpG ожидаемые являются 99,85% ± 0,04% (п = 400). Коэффициенты пересчета ниже, чем 99% может свидетельствовать о менее оптимальном бисульфита преобразования, которые могут привести к высокому уровню ложных уровнях метилирования.
Данные из библиотеки ERRBS, приготовленной из представительного человека геномной ДНК анализировали в R 2.15.2 45 с помощью methylKit пакет 26 (см Справочная файл кода 1 для деталей команд). Эти данные могут быть визуализированы в обычно используемых геномных браузерах (рис 6А). Данные цитозин метилирование в равной степени получены из обеих лент (рис 6б) и колеблется в цельныеСпектр возможных уровней цитозин метилирования (6С). Анализ технических повторов с представителем отдела урожайности образец ДНК высокой согласованности между результатами данных (рис 6D) и охватывает CpGs в широком спектре локусов генома (рис 6E и F и, как описано ранее 26). В то время как технические реплики будут высоким выходом R 2 значения (больше 97%), биологические реплики даст R 2 значения в диапазоне от 0,92 до 0,96 26, и сравнение различных типов клеток человека даст R 2 значения ниже, чем 0,86 (данные не показаны).
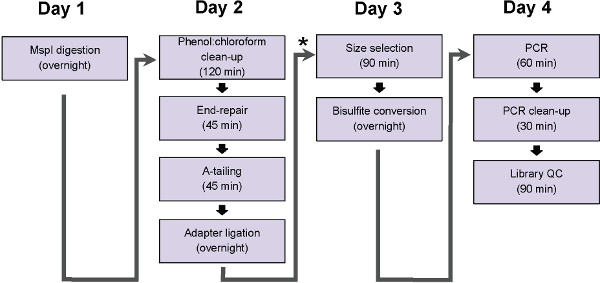
Рисунок 1: Блок-схема шагов протокола ERRBS. График представляет шаги, которые могут быть завершены в традиционном рабочего дня. * Указывает на потенциальную точку паузы (сразу следовать числе перевязка очистки и перед выбором размера, шаг протокол 5), в котором образцы могут быть заморожены при температуре -20 ° C, прежде чем приступить с длительностью протокола.
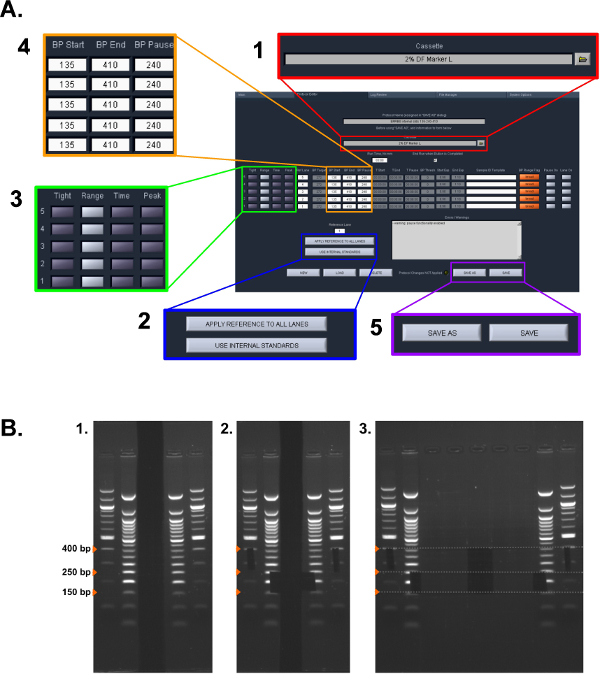
Рисунок 2: Выбор протокола размера. () Снимок экрана настройки, используемые в протоколе ERRBS Пиппин Prep (см раздел протокола 5.1.2 - 5.1.6): (1) Выберите тип кассеты. (2) Выберите стандарт будет использоваться. (3) Выберите режим сбора для каждой полосы. (4) Введите диапазоны коллекция ВР. (5) Сохранить протокол (B) Этапы ручной добычи гель, используемый в разделе протокола 5.2:. (1) Визуализация лестницы гель. (2) Помечено размеров для выбора размера, используя лезвие бритвы. (3) Изображение подакцизных образцов (нижней фракции: 150-250 б.п. и высшего фракции: 250-400 б.п.)."> Нажмите здесь, чтобы посмотреть большую версию этой цифры.
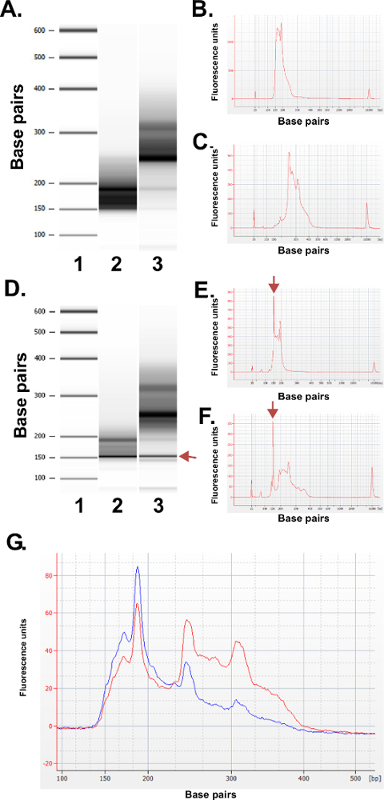
Рисунок 3: Качество контроля результатов репрезентативных ERRBS библиотек, полученных из образцов ДНК человека с помощью Bioanalyzer машину. (А) гелеобразный изображение, показывающее стандартный лестнице (1), ниже, библиотека фракцию (135-240 п.н. фракцию из Pippin Prep); 2) и выше, библиотека фракцию (240-410 п.н. фракцию из Pippin Prep); . 3) (В) Bioanalyzer electropherogram ожидаемого нижней фракции библиотеки (С) Bioanalyzer electropherogram ожидаемого более высокой доли библиотеки D -.. F) Характерные данные из плохого качества библиотеки преп. Гелеобразный изображения (D) в стандартных лестницы (1), меньше доля библиотеки (2) и выше, фракцию библиотеки (3). Полоса 150 б.п., отмеченные АрроW обозначает чрезмерное количество адаптера. Electropherogram из нижней (E) и высшего библиотечно фракции (F) с превышением адаптера пиков на 150 б.п. (отмечен со стрелками). (G) Bioanalyzer electropherogram с пулом библиотеки ERRBS для секвенирования. Красный след представляет собой высококачественный объединенному библиотека с равным представительством высших и низших фракций. Синий след представляет обобщенный библиотеку не подходит для секвенирования из-за отсутствия равного представительства высших и низших фракций. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этой цифры.
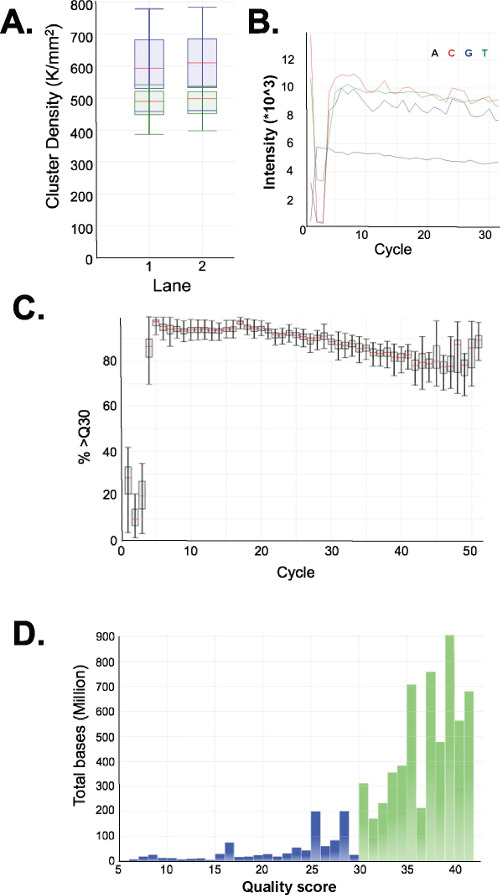
Рисунок 4: Последовательность карт для представителя ERRBS 51 цикла одного чтения секвенирования работать на HiSeq 2500 секвенсор в высокой производительностиРежим. () Плотности кластера (K / мм 2 = 1000 кластеров на миллиметр в квадрате; синий). И плотности кластера, проходящие фильтр (зеленый), в две полосы с ERRBS библиотек (B) Типичные интенсивности видели в первых 30 циклов в переулке с библиотека ERRBS. Обратите внимание на CGG подпись МГПИ Пищеварение в интенсивностей первых трех циклов. (C) в процентах баз с показателем качества 30 или выше (%> Q30) для каждого цикла в одной ERRBS пер. (D) распределение показателя качества для все циклы в одном ERRBS пер. Синий = менее Q30, зеленый = больше или равно Q30. На этой улочке, 84,7% баз были качественные набрали 30 или выше.
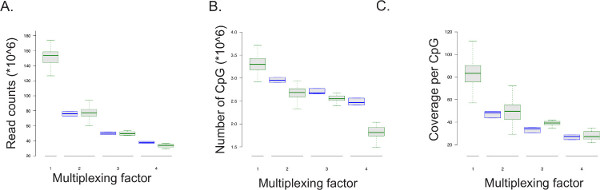
Рисунок 5: Последовательность вывода результатов. Вставка графики экспериментальных данных из мультиплексированных и одного образца на дорожку последовательности ру нс (отображается в виде зеленых коробок) и данных, полученных моделируемой понижающей дискретизации от секвенирования работает трех ERRBS библиотек (отображается в виде синих коробок; выборочные пять раз для каждой секвенирования перспективе) от 51 цикла одного чтения последовательности работает фактор мультиплексирования соответствует. Количество ERRBS библиотек последовательность на полосу. 1 = весь переулок или 100% читает и представляет данные из одной библиотеки ERRBS на дорожку; 2 = 50% полосы движения и представляет данные из двух ERRBS библиотек на одну полосу движения; 3 = 33% переулка и представляет данные из трех ERRBS библиотек на одну полосу движения; и 4 = 25% переулка и представляет данные из четырех ERRBS библиотек на одну полосу движения. () отсчитывает читать, или количество последовательностей анализируемых Пер фактора мультиплексирования. (B) количество КПГ покрыта данных секвенирования в мультиплексирования фактором. (С) средний расход на CpG в мультиплексирования фактор._blank "> Нажмите здесь, чтобы посмотреть большую версию этой цифры.
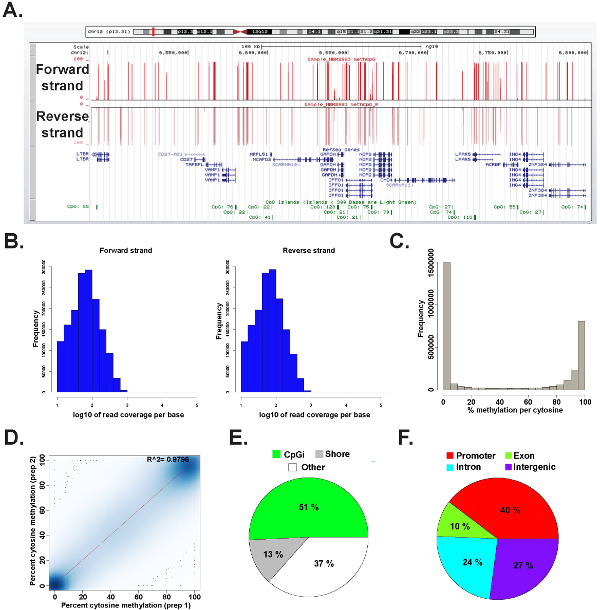
Рисунок 6: Типичные данные из ERRBS библиотеки, полученной из геномной ДНК человека () из Калифорнийского университета в Санта-Круз (УСК) геноме браузеру 43 изображение репрезентативных данных из ERRBS секвенирования переулке.. Шкалы Y-ось представляет 0-100% метилирование цитозина в каждой покрытой минимум в 10 раз. Топ Пользовательские трассы представляет собой вперед прядь и ниже Пользовательские трассы представляет собой обратную прядь. Показана chr12:.. 6,489,523-6,802,422 (hg19) включительно генов RefSeq и CpG островов в рамках этой же области генома (B) гистограммы распределения охвата CpG вдоль прямой и обратной пряди в представительном человек CD34 + костного мозга образца (C) гистограммы распределенияЦГ уровнях метилирования по обеим цепям в представительной человек CD34 + костного мозга образца. (D) Соотношение участка уровней метилирования CpG от представителя технической копия образца ДНК человека. (E) Круговая диаграмма, иллюстрирующая пропорции CpGs указанных в ERRBS, которые примечания, CpG островков (светло-зеленый), CpG берега (серый) и других регионов (кроме белого) в репрезентативной выборке, полученной из геномной ДНК человека. (F) Круговая диаграмма, иллюстрирующая пропорции CpGs указанных в ERRBS которые прилагаемые к промоторов генов (красный ), экзоны (зеленый), интроны (синий) и межгенных (фиолетовый). Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этой цифры.
| Реагент | Объем | Комментарий |
| 10x ДНК-лигазы Т4 реакционного буфера | 10 мкл | |
| Дезоксинуклеотидтрифосфатов (дНТФ) Решение Mix | 4 мкл | Микс из 10 мМ каждого нуклеотида |
| Т4 ДНК-полимеразы | 5 мкл | 3000 ед / мл |
| ДНК-полимераза I Большой (Кленова) Фрагмент | 1 мкл | 5000 единиц / мл |
| Т4 полинуклеотидкиназы | 5 мкл | 10000 единиц / мл |
| ДНКазы свободной воды | 45 мкл |
Таблица 1:. Конечные реакции ремонт реагенты имена реагентов и количеств используемых в реакции, конец ремонта (протокол шагом 2,1).
| Реагент | Объем | Комментарий |
| 10x реакционного буфера | 5 мкл | Например, NEBuffer 2 |
| 1 мМ 2'-дезоксиаденозин 5'-трифосфат (дАТФ) | 10 мкл | |
| Фрагмент Кленова (3 '→ 5' экзо-) | 3 мкл | 5000 единиц / мл |
Таблица 2:. Хвостохранилища реакции реагенты имена реагентов и количеств используемых в реакции, А-хвостохранилища (протокол шагом 3,1).
| Реагент | Объем | Комментарий |
| 15 мкМ отожженных адаптеры в ДНКазы без воды | 3 мкл | PE адаптер 1.0 и PE адаптер 2,0; в таблице 4 последовательностей и ссылки |
| 10x ДНК-лигазы Т4 реакционного буфера | 581, л | |
| ДНК-лигазы Т4 | 1 мкл | 2000000 ед / мл |
| ДНКазы свободной воды | 31 мкл |
Таблица 3: Адаптер лигирования реакции реагенты имена реагентов и количеств используемых в реакции лигирования адаптера (протокол этапе 4.2)..

Таблица 4:. Олигонуклеотиды, используемые в протоколе ERRBS Список олигонуклеотидов, используемых в протоколе ERRBS в реакции лигирования (протокол этап 4) и ПЦР стадий амплификации (протокол шаг 7).
| Реагент | Объем | Комментарий |
| 10x FastStart High Fidelity Реакционный буфер Wiго 18 мМ хлорида магния | 20 мкл | |
| 10 мМ дНТФ Решение Mix | 5 мкл | |
| 25 мкМ ПЦР PE грунтовки 1,0 | 4 мкл | Смотрите таблицу 4 |
| 25 мкМ ПЦР PE грунтовки 2,0 | 4 мкл | Смотрите таблицу 4 |
| FastStart High Fidelity фермента | 2 мкл | 5 единиц / мкл FastStart Taq ДНК-полимеразы |
| ДНКазы свободной воды | 125 мкл |
Таблица 5: ПЦР реакции реагенты имена реагентов и количеств используемых в реакции амплификации ПЦР (протокол этапе 7.1)..
| Шаг протокол | Реагент / подробно протокол | Вход сумма ДНК | ||
| 5-10 нг | 25 нг | 50 нг | ||
| 1 | MspI фермент | 1 мкл | 2 мкл | 2 мкл |
| MspI дайджест реакционный объем | 50 | 100 | 100 | |
| 4 | Адаптеры в реакции лигирования | 1 мкл | 2 мкл | 3 мкл |
| Лигирование реакционный объем | 20 мкл | 25 мкл | 50 мкл | |
| 5 | Выбор протокола Размер | Руководство гель только | Пиппин Prep или ручной гель | Пиппин Prep или ручной гель |
| 7 | Концентрация ПЦР-праймер, | 25 мкМ | 25 мкМ | 10 мкМ в течение 14 циклов; 25 мкм для 18 циклов |
| Количество циклов ПЦР | 18 | 18 | 14-18 | |
Таблица 6:. Протокол шаг модификации для ввода количества материала, начиная от 5-50 нг несколько шагов в течение протокол требует модификации реагентов величин, используемых для получения высоких библиотеки качества от различных количеств исходных материалов. Изменения в ключевых реагентов количествах включены здесь. Отрегулируйте буфер и объемов воды в реакциях соответственно.
| Chr | База | Прядь | Охват | freqC | freqT |
| CHR1 | 10564 | R | 366 | 85.52 | 14.48 |
| CHR1 | 10571 | F | 423 | 91.25 | 8,75 |
| CHR1 | 10542 | F | 432 | 91,2 | 8,8 |
| CHR1 | 10563 | F | 429 | 94.64 | 5.36 |
| CHR1 | 10572 | R | 366 | 96.99 | 3.01 |
| CHR1 | 10590 | R | 370 | 88.11 | 11.89 |
| CHR1 | 10526 | R | 350 | 92 | 8 |
| CHR1 | 10543 | R | 368 | 92.93 | 7,07 |
| CHR1 | 10525 | F | 433 | 91.92 | 8,08 |
| CHR1 | 10497 | F | 435 | 88.74 | 11.26 |
Таблица 7: Типичные данные ERRBS. После выравнивания данных и определения метилирования цитозина, получается база данных пара для каждого CpG покрыты, протокол выравнивание, как описано будет определять геномной координаты (столбцы: CHR = хромосома, базы и Strand)., Уровень охвата конкретного локуса (охват ), а скорость цитозина обнаружения по сравнению с тимидина в процентах (freqC и freqT соответственно).
| Количество ERRBS библиотек на одну полосу движения | Среднее число однозначно согласованы читает | Среднее число CpGs покрыта | Средняя расход на CpG |
| 1 | 152231184 ± 13189678 | 3183594 ± 713547 | 85 ± 16 |
| 2 | 77680837 ± 7657058 | 2674823± 153 494 | 49 ± 9 |
| 3 | 49938156 ± 2436865 | 2552186 ± - 76624 | 39 ± 2 |
| 4 | 34457208 ± 4441686 | 1814461 ± 144339 | 28 ± 4 |
Таблица 8:. Типичные параметры из секвенирования одно- и уплотненные библиотеки ERRBS Показана данные на полосу от 51-цикла одно- читать работает секвенирования: среднее и стандартные отклонения однозначно согласованы читает, количество CpGs покрыты и расход на CpG сайта полученного в результате секвенирования одного ERRBS библиотеки в полосе (N = 100), два ERRBS библиотеки в полосе (N = 128), три ERRBS библиотеки на дорожку (п = 11), и четыре ERRBS библиотеки в полосе (N = 11).
Обсуждение
Протокол, представленные данные с разрешением дает пар оснований цитозина метилирования в биологически соответствующих областей генома. Протокол, как написано оптимизирован для 50 нг исходного материала, однако, он может быть выполнен с возможностью обрабатывать диапазон исходного материала (5 нг или больше) 26. Это потребует корректировки некоторых из шагов протокола, как показано в таблице 6. В ERRBS библиотеки поддаются паре конец последовательности и дальнейшего геномной покрытия также может быть осуществлено путем секвенирования читает больше, чем 51 циклов. Multiplexed последовательности будет предлагать более низкую протокол стоить в образце, однако, это приведет к снижению охвата в CpG сайт, представленных в данных (рис 5 и таблица 8), и не даст достаточной глубины охвата для выполнения анализов, которые требуют высокого уровня охвата за Сайт CpG (например, как описано Landan и др. 33). И, наконец, этот протокол (или любой бисульфита основе Protocoл) не может отличить метил-цитозина и гидроксиметил-цитозин 46,47. Тем не менее, данные, полученные могут быть интегрированы с другими протокола о результатах 48,49 очертить различные модификации, и другие модификации цитозина недавно сообщила 50, они должны быть интересны.
Высокие библиотеки качество будет выглядеть, как показано на фиг.3А-С, и один раз собирали для секвенирования дает след, как показано на рисунке 3G (красный след), представляющий равные мол рные вклады от обоих библиотеки фракций. Библиотека недостаточности препарат может возникнуть в результате любой стадии во время процедуры. Если деградации ДНК обрабатывают это приведет к библиотекам, которые не обогащенных MspI фрагментов и, следовательно, к низкой базы CpG, используя параметры секвенирования, описанных в данном протоколе. Если фермент не функционирует или непреднамеренно исключены из одной из реакций, протокол не дают ожидаемого библиотеку. Если лигирования Риие неэффективно, адаптеры при более высокой концентрации, чем ожидалось, и / или концентрация праймеров использовали является ограничивающим реагентом для конечных стадий амплификации, отказ библиотека может произойти. Избыток адаптеры (рассматривается как пики при ~ 150 б.п. в результатах Bioanalyzer, рисунок 3D-F) в библиотеке также вмешиваться в последовательности из-за неразборчивого кластеризации и в библиотеке и избыточных адаптеров. Хотя такая библиотека может упорядочить, по-видимому, как правило, значительная часть читает будет просто адаптер последовательности. Если избыточные адаптеры наблюдаются в библиотеке, то лучше повторить библиотеки препарата, если материал доступен использовании оптимальной исходного материала к адаптеру количественных соотношений. Наконец, для обеспечения эффективной ПЦР-амплификации библиотеки, низшие и высшие библиотечные фракции поддерживается в отдельных образцах по всему бисульфита преобразования и ПЦР шагов по обогащению. Невыполнение этого требования приводит к дифференциальному эффективность усиления при РCR реакция высших и низших фракций (как показано на рис 3G синяя линия) и потенциальная неравного представительства соответствующего локусов генома, покрытой каждой фракции библиотеки во время секвенирования. Пользователь может выбрать, чтобы включать количественные стадии ПЦР Сразу после бисульфита преобразования для дальнейшего титрования оптимальных циклов ПЦР, необходимых для усиления библиотеки генерируется.
Протокол синтеза библиотека ERRBS имеет несколько ключевых шагов, в которых рекомендованы конкретные реагенты. На этапе конечного ремонт, использование четырех нуклеотидов дНТФ смеси позволяет конечному ремонт любых продуктов, не содержащих выступ CG, такие как те, в результате МГПИ ферментативная звезд активности и стригли фрагментов ДНК, присутствующих в исходном образце ДНК. Это приводит к улучшению представления CpG в результатах. На этапе лигирования очень важно использовать высокую концентрацию лигазы (2000000 ед / мл) и метилированных адаптеры, чтобы гарантировать, что лигирование Reactiна является эффективным и, что бисульфит преобразования не влияет адаптера последовательности, необходимые для точного выравнивания данных. На этапе ПЦР, с использованием полимеразы, способной усиливать бисульфита обработанные GC-богатых фрагментов ДНК необходимо для высокой специфичностью. Наконец, чтобы обеспечить удаление избыточных адаптеров и праймеров, SPRI очистки шарик (например: Ажанкур AMPure XP) рекомендуется, а не анализы, основанные столбцов для перевязки и выделений продукта ПЦР.
Для того, чтобы генерировать высококачественные данные, важно обеспечить эффективное преобразование бисульфита. Управления представлены предлагает пользователю возможность, чтобы определить эффективность преобразования до секвенирования. В качестве альтернативы, не-ДНК человека, таких как лямбда-ДНК может быть использован в качестве внутреннего контроля (шип-в). Из-за различий в виду, этот тип управления может быть непосредственно включена в выходной последовательности (например, используется Yu и др. 34). Однако, если шип в Iы использовали, он не может быть использован, чтобы определить эффективность преобразования до библиотеки последовательности, если однозначно не усиливается и независимо друг от друга последовательность до библиотеки секвенирования. Обменные курсы определяются на основе статуса метилирования в-сайты, не CpG. Это не может быть подходящим для использования в контексте высокой цитозина метилирования в не-CpG контексте (например эмбриональные стволовые клетки) и параллельных образцов или других средств для оценки эффективности преобразования могут быть использованы для этой цели.
Есть несколько предостережений в адрес, которые являются уникальными для секвенирования ERRBS библиотек. Первые три основы библиотеки фракций секвенированы почти равномерно неслучайное из-за MspI распознавания вырезом сайта (C ^ CGG; рис 4, б). Это приводит к потенциал для значительной потери данных из-за низкого качества чтения в связи с неудовлетворительной локализации кластера, несмотря на кажущуюся высокой плотности кластера во время секвенирования. Чтобы преодолеть этот барьер,включают в себя высокую библиотеку сложности в (контроля PhiX или другой библиотеки) независимой переулке, на специальной контрольной полосе. Высокие библиотеки комплексность концы, содержащие сбалансированное представительство A, C, T и G в течение первых четырех баз секвенированных. Подходящие полосы контроля включают библиотеки, такие как РНК-SEQ, чип-Seq, всего секвенирования генома, или контроля предлагаемой производителем последовательности устройства (например, v3 PhiX управления). При обозначены в качестве контрольной полосе для соответствующего секвенирования перспективе, он может служить в качестве основы для генерации матрицы, которое используется в течение первых четырех оснований последовательности, чтобы обнаружить кластера позиции. Высокое качество чтения захватили поднимет среднюю Расход на сайте CpG на 5,2 (N = 4). С другой стороны, это техническая сложность также могут быть преодолены с помощью темного подход секвенирования, как описано ранее 23. Другие критерии секвенирования соблюдать стандартные операционные процедуры в протоколах изготовителя. Наконец, расход на CpG сHosen для анализа данных будет руководствоваться пользователя и частично на биологические вопросы, представляющие интерес. Порог охват 10x дает подход анализа с высоким уровнем охвата, однако этот порог может быть снижен это должно быть интересны.
Полное обсуждение анализа данных ERRBS выходит за рамки данной статьи, однако, по-разному метилированные цитозины и регионы могут быть определены с помощью с открытым исходным кодом инструменты 31,51-53. Дополнительные соображения и подходы анализа были хорошо описаны 54,55, и читателю предлагается искать литературу для инструментов наиболее подходящих для анализа запланированных.
По сравнению с другими известными методами, ERRBS предлагает четыре дня протокол, который когда проводили, как описано урожайность высокие темпы воспроизводимости. Это была утверждена по сравнению с золотым стандартом MassARRAY EpiTYPER 26, является экономически эффективным для данных высокого охвата и адаптируется для различных входных материаловсуммы (благоприятные для клинической обработке образцов и другие мобильные типов низкой частоты) и секвенирования подходы. Он предлагает разрешение пар оснований в биологически соответствующей локусов и могут быть использованы в интегративных анализ с другими методами профилирования генома связывания транскрипционных факторов, ремоделирование хроматина, эпигенетические и другие цитозин изменения интересов. Использование данных ERRBS в таких исследованиях может способствовать всестороннему молекулярной подхода и позволяют высокой размерной анализа в исследовании биологических моделей и болезни человека.
Раскрытие информации
Авторы не имеют конфликта интересов раскрывать.
Благодарности
We thank all the authors of the original ERRBS report. We thank Mame Fall for technical assistance. We acknowledge the Weill Cornell Medical College Epigenomics Core for technical services and assistance. The work was supported by a Sass Foundation Judah Folkman Fellowship, an NCI K08CA169055 and ASH-AMFDP12005 to FGB, NIH R01HG006798 and R01NS076465, funding from the Irma T. Hirschl and Monique Weill-Caulier Charitable Trusts and STARR Consortium (I7-A765) to CEM, and an LLS SCORE grant (7006-13) to AMM.
Материалы
| Name | Company | Catalog Number | Comments |
| MspI | New England Biolabs | R0106M | 100,000 units/ml |
| NEBuffer 2 | New England Biolabs | B7002S | Reaction buffer for MspI enzyme; protocol step 1.2 |
| Phenol solution | Sigma-Aldrich | P4557 | Equilibrated with 10 mM Tris HCl, pH 8.0; see safety and handling instructions at http://www.sigmaaldrich.com/catalog/product/sigma/p4557 |
| Chloroform | Sigma-Aldrich | C2432 | See safety and handling instructions at http://www.sigmaaldrich.com/catalog/product/sial/c2432 |
| Glycogen | Sigma-Aldrich | G1767 | 19-22 mg/ml |
| NaOAc | Sigma-Aldrich | S7899 | 3 M, pH 5.2 |
| Ethanol | Sigma-Aldrich | E7023 | 200 proof, for molecular biology |
| Buffer EB | Qiagen | 19086 | 10 mM Tris-Cl, pH 8.5 |
| tris(hydroxymethyl)aminomethane (Tris) | Sigma-Aldrich | T1503 | prepare a 1 M, pH 8.5 solution |
| Tris- Ethylenediaminetetraacetic acid (TE) | Sigma-Aldrich | T9285 | Dilute to 1x buffer solution per manufacturer's recommendations |
| T4 DNA Ligase Reaction Buffer | New England Biolabs | B0202S | 10x concentration |
| Deoxynucleotide triphosphate (dNTP) Solution Mix | New England Biolabs | N0447L | 10 mM each nucleotide |
| T4 DNA Polymerase | New England Biolabs | M0203L | 3,000 units/ml |
| DNA Polymerase I, Large (Klenow) Fragment | New England Biolabs | M0210L | 5,000 units/ml |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | 10,000 units/ml |
| QIAquick PCR Purification Kit | Qiagen | 28104 | Used for DNA product purification in protocol step 2.3 |
| 2'-deoxyadenosine 5'-triphosphate (dATP) | Promega | U1201 | 100 mM |
| Klenow Fragment (3'→5' exo-) | New England Biolabs | M0212L | 5,000 units/ml |
| MinElute PCR Purification Kit | Qiagen | 28004 | Used for DNA product purification in protocol step 3.3 |
| T4 DNA Ligase | New England Biolabs | M0202M | 2,000,000 units/ml |
| Methylation Adapter Oligo Kit | Illumina | ME-100-0010 | |
| Agencourt AMPure XP | Beckman Coulter | A63881 | Used in protocol sections that implement magnetic bead purification steps (steps 4.3 and 8.2). Equilibrate to room temperature before use. |
| Pippin Prep Gel Cassettes, 2% Agarose, dye-free | Sage Science | CDF2010 | with internal standards |
| Certified Low Range Ultra Agarose | Bio-Rad | 161-3106 | |
| Tris-Borate-EDTA (TBE) buffer | Sigma-Aldrich | T4415 | |
| Ethidium bromide solution | Sigma-Aldrich | E1510 | 10 mg/ml |
| 50 bp DNA Ladder | NEB | N3236S | |
| 100 bp DNA Ladder | NEB | N3231S | |
| Gel Loading Dye, Orange (6x) | NEB | B7022S | |
| Scalpel Blade No. 11 | Fisher Scientific | 3120030 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| EZ DNA Methylation Kit | Zymo Research | D5001 | Used in protocol step 6.2 |
| EZ DNA Methylation-Lightning Kit | Zymo Research | D5030 | Alternative for step 6.2 |
| Universal Methylated Human DNA Standard | Zymo Research | D5011 | Used as bisulfite conversion control |
| FastStart High Fidelity PCR System | Roche | 03553426001 | |
| Qubit dsDNA High Sensitivity Assay Kit | Life Technologies | Q32854 | A fluorescence-based DNA quantitation assay; used in protocol steps 1.1, 9.1 and 10.1 |
| DynaMag-2 Magnet | Life Technologies | 12321D | |
| High Sensitivity DNA Kit | Agilent Technologies | 5067-4626 | |
| 2100 Bioanalyzer | Agilent Technologies | ||
| PhiX Control v3 | Illumina | FC-110-3001 | |
| HiSeq 2500 | Illumina | ||
| Pippin Prep | Sage Science | ||
| Qubit 2.0 Fluorometer | Life Technologies | Q32872 | |
| TruSeq SR Cluster Kit v3-cBot-HS | Illumina | GD-401-3001 | |
| TruSeq SBS Kit v3-HS | Illumina | FC-401-3002 | |
| TruSeq RNA Sample prep | Illumina | RS-122-2001 | Barcoded adapters used for multiplexing libraries; See Supplemental file for multiplexing protocol. |
| Microcentrifuge | |||
| Vortex Mixer | |||
| Dry Block Heater | |||
| Thermal Cycler | |||
| Water Bath | |||
| Gel electrophoresis system | |||
| Electrophoresis power supply | |||
| Gel doc | |||
| UV or blue light transilluminator |
Ссылки
- Jones, P. A. Functions of DNA methylation: islands, start sites, gene bodies and beyond. Nat Rev Genet. 13 (7), 484-492 (2012).
- Barlow, D. P. Genomic imprinting: a mammalian epigenetic discovery model. Annual Review Of Genetics. 45, 379-403 (2011).
- Thiagarajan, R. D., Morey, R., Laurent, L. C. The epigenome in pluripotency and differentiation. Epigenomics. 6 (1), 121-137 (2014).
- Reik, W. Stability and flexibility of epigenetic gene regulation in mammalian development. Nature. 447 (7143), 425-432 (2007).
- Hartnett, L., Egan, L. J. Inflammation, DNA methylation and colitis-associated cancer. Carcinogenesis. 33 (4), 723-731 (2012).
- Smith, Z. D., Meissner, A. DNA methylation: roles in mammalian development. Nat Rev Genet. 14 (3), 204-220 (2013).
- Li, E., Bestor, T. H., Jaenisch, R. Targeted mutation of the DNA methyltransferase gene results in embryonic lethality. Cell. 69 (6), 915-926 (1992).
- Okano, M., Bell, D. W., Haber, D. A., Li, E. DNA methyltransferases Dnmt3a and Dnmt3b are essential for de novo methylation and mammalian development. Cell. 99 (3), 247-257 (1999).
- Feinberg, A. P. Phenotypic plasticity and the epigenetics of human disease. Nature. 447 (7143), 433-440 (2007).
- Bock, C. Epigenetic biomarker development. Epigenomics. 1 (1), 99-110 (2009).
- Laird, P. W. The power and the promise of DNA methylation markers. Nat Rev Cancer. 3 (4), 253-266 (2003).
- How Kit, A., Nielsen, H. M., Tost, J. DNA methylation based biomarkers: practical considerations and applications. Biochimie. 94 (11), 2314-2337 (2012).
- Mikeska, T., Bock, C., Do, H., Dobrovic, A. DNA methylation biomarkers in cancer: progress towards clinical implementation. Expert Review Of Molecular Diagnostics. 12 (5), 473-487 (2012).
- Gyparaki, M. T., Basdra, E. K., Papavassiliou, A. G. DNA methylation biomarkers as diagnostic and prognostic tools in colorectal cancer. Journal of Molecular Medicine. 91 (11), 1249-1256 (2013).
- Figueroa, M. E., et al. DNA methylation signatures identify biologically distinct subtypes in acute myeloid leukemia. Cancer Cell. 17 (1), 13-27 (2010).
- Heyn, H., Mendez-Gonzalez, J., Esteller, M. Epigenetic profiling joins personalized cancer medicine. Expert review of Molecular Diagnostics. 13 (5), 473-479 (2013).
- Kulis, M., Esteller, M. DNA methylation and cancer. Advances in Genetics. 70, 27-56 (2010).
- Xiong, Z., Laird, P. W. COBRA: a sensitive and quantitative DNA methylation assay. Nucleic Acids Res. 25 (12), 2532-2534 (1997).
- Meissner, A., et al. Reduced representation bisulfite sequencing for comparative high-resolution DNA methylation analysis. Nucleic Acids Res. 33 (18), 5868-5877 (2005).
- Gu, H., et al. Preparation of reduced representation bisulfite sequencing libraries for genome-scale DNA methylation profiling. Nat Protoc. 6 (4), 468-481 (2011).
- Bock, C., et al. Quantitative comparison of genome-wide DNA methylation mapping technologies. Nat Biotechnol. 28 (10), 1106-1114 (2010).
- Harris, R. A., et al. Comparison of sequencing-based methods to profile DNA methylation and identification of monoallelic epigenetic modifications. Nat Biotechnol. 28 (10), 1097-1105 (2010).
- Boyle, P., et al. Gel-free multiplexed reduced representation bisulfite sequencing for large-scale DNA methylation profiling. Genome Biol. 13 (10), R92(2012).
- Chatterjee, A., Rodger, E. J., Stockwell, P. A., Weeks, R. J., Morison, I. M. Technical considerations for reduced representation bisulfite sequencing with multiplexed libraries. Journal of Biomedicine & Biotechnology. 2012, 741542(2012).
- Lee, Y. K., et al. Improved reduced representation bisulfite sequencing for epigenomic profiling of clinical samples. Biological Procedures Online. 16 (1), 1(2014).
- Akalin, A., et al. Base-pair resolution DNA methylation sequencing reveals profoundly divergent epigenetic landscapes in acute myeloid leukemia. PLoS Genet. 8 (6), e1002781(2012).
- Hatzi, K., et al. A Hybrid Mechanism of Action for BCL6 in B Cells Defined by Formation of Functionally Distinct Complexes at Enhancers and Promoters. Cell Reports. 4 (3), 578-588 (2013).
- Will, B., et al. Satb1 regulates the self-renewal of hematopoietic stem cells by promoting quiescence and repressing differentiation commitment. Nature Immunology. 14 (5), 437-445 (2013).
- Lu, C., et al. Induction of sarcomas by mutant IDH2. Genes Dev. 27 (18), 1986-1998 (2013).
- Kumar, R., et al. AID stabilizes stem-cell phenotype by removing epigenetic memory of pluripotency genes. Nature. 500 (7460), 89-92 (2013).
- Li, S., et al. An optimized algorithm for detecting and annotating regional differential methylation. BMC Bioinformatics. 14, Suppl 5. S10(2013).
- Patterson, K., Molloy, L., Qu, W., Clark, S. DNA methylation: bisulphite modification and analysis. Journal of Visualized Experiments. (56), 3170(2011).
- Landan, G., et al. Epigenetic polymorphism and the stochastic formation of differentially methylated regions in normal and cancerous tissues. Nat Genet. 44 (11), 1207-1214 (2012).
- Yu, M., et al. Tet-assisted bisulfite sequencing of 5-hydroxymethylcytosine. Nat Protoc. 7 (12), 2159-2170 (2012).
- Goecks, J., Nekrutenko, A., Taylor, J., Galaxy, T. Galaxy: a comprehensive approach for supporting accessible, reproducible, and transparent computational research in the life sciences. Genome Biol. 11 (8), R86(2010).
- Dorff, K. C., et al. GobyWeb: simplified management and analysis of gene expression and DNA methylation sequencing data. PLoS One. 8 (7), e69666(2013).
- Roehr, J. T., Dodt, M., Ahmed, R., Dieterich, C. Flexbar − flexible barcode and adapter processing for next-generation sequencing platforms. MDPI Biology. 1 (3), 895-905 (2012).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal, North America. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Needleman, S. B., Wunsch, C. D. A general method applicable to the search for similarities in the amino acid sequence of two proteins. J Mol Biol. 48 (3), 443-453 (1970).
- Krueger, F., Andrews, S. R. Bismark: a flexible aligner and methylation caller for Bisulfite-Seq applications. Bioinformatics. 27 (11), 1571-1572 (2011).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Kent, W. J., et al. The human genome browser at UCSC. Genome Res. 12 (6), 996-1006 (2002).
- Thorvaldsdottir, H., Robinson, J. T., Mesirov, J. P. Integrative Genomics Viewer (IGV): high-performance genomics data visualization and exploration. Briefings in Bioinformatics. 14 (2), 178-192 (2013).
- Team, R. C. R. A language and environment for statistical computing. R Foundation for Statistical Computing. Vienna, Austria. ISBN 3-900051-07-0, http://www.R-project.org (2012).
- Nestor, C., Ruzov, A., Meehan, R., Dunican, D. Enzymatic approaches and bisulfite sequencing cannot distinguish between 5-methylcytosine and 5-hydroxymethylcytosine in DNA. BioTechniques. 48 (4), 317-319 (2010).
- Huang, Y., et al. The behaviour of 5-hydroxymethylcytosine in bisulfite sequencing. PLoS One. 5 (1), e8888(2010).
- Yu, M., et al. Base-resolution analysis of 5-hydroxymethylcytosine in the mammalian genome. Cell. 149 (6), 1368-1380 (2012).
- Song, C. X., et al. Genome-wide profiling of 5-formylcytosine reveals its roles in epigenetic priming. Cell. 153 (3), 678-691 (2013).
- Ito, S., et al. Tet proteins can convert 5-methylcytosine to 5-formylcytosine and 5-carboxylcytosine. Science. 333 (6047), 1300-1303 (2011).
- Akalin, A., et al. methylKit: a comprehensive R package for the analysis of genome-wide DNA methylation profiles. Genome Biol. 13 (10), R87-1186 (2012).
- Stockwell, P. A., Chatterjee, A., Rodger, E. J., Morison, I. M. DMAP: Differential Methylation Analysis Package for RRBS and WGBS data. Bioinformatics. 30 (13), 1814-1822 (2014).
- Sun, D., et al. MOABS: model based analysis of bisulfite sequencing data. Genome Biol. 15 (2), R38(2014).
- Bock, C. Analysing and interpreting DNA methylation data. Nat Rev Genet. 13 (10), 705-719 (2012).
- Rivera, C. M., Ren, B. Mapping human epigenomes. Cell. 155 (1), 39-55 (2013).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены