A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
הדור של Fusions חלבון פלורסנט
In This Article
Summary
PCR בתיווך שינוי גנטי יכול לשמש כדי ליצור תערובות חלבון פלואורסצנטי במיני קנדידה, המאפשר הדמיה לכמת בתאי שמרים וחלבונים. בזאת, אנו מציגים אסטרטגיה לבניית שילוב חלבון פלואורסצנטי (Eno1-FP) ב קנדידה parapsilosis.
Abstract
מיני קנדידה, הקולוניאליסטים רווחים של קטעי מעיים ו מין ושתן, הן הגורם של רוב הזיהומים פטרייתיים פולשניים בבני אדם. לכן, כלים מולקולריים וגנטיים נדרשים כדי להקל על המחקר של המנגנונים בפתוגנזה שלהם. PCR בתיווך שינוי גנטי הוא גישה פשוטה ומהירה ליצור חלבונים מתויג epitope כדי להקל הגילוי שלהן. בפרט, חלבון פלואורסצנטי (FP) התכה הם כלים רבי עוצמה המאפשרים הדמיה לכמת הן בתאי שמרים וחלבונים על ידי מיקרוסקופ פלואורסצנטי ו immunoblotting, בהתאמה. פלסמידים המכילים רצפי קידוד FP, יחד עם גני סמן תזונתיים המסייעים טרנספורמציה של מיני קנדידה, נוצרו לצורך בניית הביטוי FP ב קנדידה. בזאת, אנו מציגים אסטרטגיה לבניית היתוך FP בתוך מיני קנדידה. פלסמידים המכילים את עמיד nourseothricinגן סמן טרנספורמציה NCE (NAT1) וכן רצפים לאף ירוק, צהוב, או דובדבן FPS (GFP, YFP, mCherry) משמש יחד עם פריימרים הכוללים רצפים ספציפי גני תגובת שרשרת פולימראז (PCR) כדי ליצור קלטת FP . יש קלטת גן ספציפי זה יש את היכולת להשתלב סוף 3'של מוקד הגן המקביל באמצעות רקומבינציה הומולוגיים. היתוך בתוך המסגרת מוצלחת של רצף FP לתוך מוקד הגן של עניין מאומת גנטי, ואחריו ניתוח של ביטוי חלבון היתוך על ידי מיקרוסקופ ו / או שיטות איתור חיסוני. בנוסף, במקרה של חלבונים מבוטאים בכמות גבוהה, יכולים להיות מוקרנים בשילובים מוצלחים בעיקר על ידי שיטות הדמית קרינה.
Introduction
מיני קנדידה הם פטריות commensal כי ליישב את שטחי מעי מין ושתן של כל בני האדם. בתנאים של כשל חיסוני, כגון המתרחשות עם לידה מוקדמת או תופעות מדכאות חיסון מן הטיפולים בסרטן, מיני הקנדידה יכולים להיות פתוגנים אופורטוניסטים. של מיני קנדידה, קנדידה אלביקנס הקולוניאליסט פטרייתי הנפוץ ביותר וגורם רוב הזיהומים פטרייתיים פולשניים. מינים אחרים קנדידה כגון ג glabrata, ג parapsilosis, tropicalis ג, ו- C. kruseii גם לגרום לזיהומים רציניים בחולים מדוכאי חיסון, עם קצת התנגדות פנימית מפגין נפוצים אנטיביוטיקה אנטי פטרייתי כגון fluconazole ו amphotericin B. לפיכך, זיהומים עם כמה מינים אלה הנצפים בתדירות גבוהה יותר, במיוחד בקרב חולי המטופלים מניעתי עם תרופות אנטי פטרייתי. אפילו עם הולם במועדNTI-פטרייתי לטיפול, זיהומי קנדידה פולשניים להמשיך להיות מזוהה עם תחלואה משמעותית ותמותה 1. בגלל המשמעות של מינים הקנדידה לבריאות האדם, יש צורך בכלים מולקולריים זמינים המאפשרים לימוד ובירור מהותה של מנגנוני בפתוגנזה שלהם.
כלי אחד חשוב המאפשר לחוקרים לחזות ולכמת תאים מיקרוביאליים החלבונים שהם מביעים היא טכנולוגיית היתוך FP. תגובת שרשרת פולימראז (PCR) שינוי גנטי בתיווך, כמתואר במאמר זה, מאפשרת בנייה של התכה, בין רצפי FP לבין חלבון קנדידה קידוד רצף של ריבית מוקד הגנומי שלו. שילוב יציב של המבנה מאפשר ניתוח של ביטוי חלבון כמו גם דינמיקת לוקליזציה חלבון. פלסמידים המכילים רצפי FP, אופטימיזציה עבור ביטוי קנדידה אלביקנס וכי ניתן להשתמש ב- G בתיווך PCRאנרגית אסטרטגית שינוי, נבנתה בעבר 2, 3, 4, 5. פלסמידים המכילים FP טרנספורמציה "קלטות": רצף FP צמוד גן סמן תזונתי המאפשר השינוי של ג אלביקנס ו- C. parapsilosis 2, 3, 4, 5, 6, 7. נכון לעכשיו פלסמידים זמינים כוללים מגוון של גני סמן תזונתיים לבחירה (URA3, HIS1, ARG4) לחידוש זני auxotrophic גם כסמן עמידות לתרופות דומיננטיות (NAT1), המאפשר שינוי של זנים קליניים חסרי auxotrophies. בנוסף, פלסמידים המכילים אפשרויות עבור עד ארבעה רצפים FP שונים (ירוק [GFP], Yellow [YFP], ציאן [CFP], ודובדבן [mCherry]) ואו רצף סיום ADH1 לבנייה של התכה חלבון carboxy הסופית-, או רצף האמרגן לבנייה של התכה חלבון אמינו הסופית-. Primers נועדו עם הומולוגיה ל- DNA פלסמיד סביב קלטת FP. בנוסף, פריימרים גם כוללים סדרות 5'-רחבת נושאות הומולוגיה לגן שמרי עניין להיות מתויג, המאפשר אינטגרציה של הקלטת לתוך מוקד גנומית באמצעות רקומבינציה הומולוגי (איור 1). גן ספציפי קלטות FP מופקות על ידי PCR ואז להפוך לתאי קנדידה עשו מוסמכת לספיגה של ה- DNA על ידי טיפול עם אצטט ליתיום.

איור 1: תרשים של איך בשילובי רצף FP נוצרים מיני קנדידה. (א) includ DNA פלסמידes רצף FP וכן קידוד רצף nourseothricin התנגדות (NAT1). מיקומים יחסיים של קדימה (FWD) primers ההפוכה (REV) נראים לעין, עם חלקים שחורים של פריימרים המציינים באזור של הומולוגיה לרצף פלסמיד ואת המנות הסגולות המציינות באזור הומולוגית הגן ספציפי או הארכה יחל. (B) קלטות FP הופכות קנדידה ולשלב בתוך מוקד גנומית ENO1 באמצעות רקומבינציה הומולוגי (קווים מקווקווים). (ג) כתוצאה רצף היתוך FP בבית 3'end של ENO1. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
בזאת, אנו מציגים דוגמא של היתוך חלבון (Eno1-FP) מבנים במיני קנדידה. אנו משתמשים תיוג פלסמידים המכילים את הגן סמן טרנספורמציה NAT1 וכן רצפי קידוד GFP, YFP, אוmCherry (איור 2). פלסמידים אלה משמשים יחד עם פריימרים ב PCR כדי ליצור קלטות גן ספציפי המאפשרים שילוב של FPS כדי 3'-סוף ENO1, וכתוצאה מכך ביטוי Eno1 התמזגו fps ב carboxy הסופית- שלה.
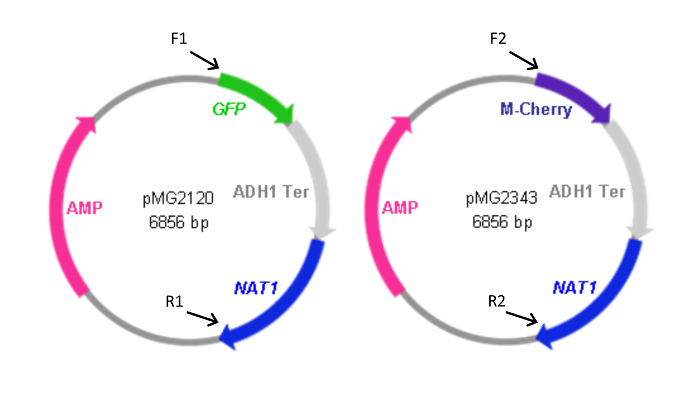
איור 2: מפות של פלסמידים קלטת המכילה FP. קדמי (F) הפוך (R) פריימרים השתמשו כדי ליצור את הקלטות מן פלסמידים מסומנים יחד עם המיקום היחסי של ההומולוגיה שלהם פלסמידים. רצפים פריימר הם כמפורט בטבלה 1. F1 ו- R1 שימשו גם כדי ליצור את קלטת NAT1 pYFP-. הפלסמיד המכיל את הקלטת NAT1 YFP- (pMG2263) זהה pMG2120 למעט YFP במקום של רצף ה- GFP. בגדלים קלטת: GFP-NAT1, 3.7 KBP; mCherry- NAT1, 3.2 KBP; YFP- NAT1, 3.KBP 7. נתון זה יש הבדל בין Gerami-נג'אד, et al. 4 אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
Protocol
1. פלסמידים תבנית לבודד מן החיידק
- לגדול החיידק המכיל את הפלסמיד תבנית לילה 10 מ"ל lysogeny מרק (LB) + 200 מ"ג / L אמפיצילין (AMP) על 37 מעלות צלזיוס עם רעד.
- קציר תאים על ידי צנטריפוגה ב 6000 XG במשך 2 דקות.
- למזוג נוזל, לבודד ולטהר דנ"א E. coli תא על ידי שיטה סטנדרטית כפי שתואר לעיל Ausubel et al. 8.
- Resuspend DNA טריס-EDTA (TE; 10 מ"מ טריס, pH 8.0, 1 mM EDTA, pH 8.0) בריכוז העבודה של 50-100 ng / ml.
2. Primers עיצוב
| תֶחֶל | רצף פריימר |
| F1 | 5 ' GAGAATTGAAGAAGAGTTGGGAGACAATGCTATCTATGCTGGTAAGGACTTCCACAATGCTCAAACTTTG GGTGGTTCTAAAGGTGAAGAATTATT 3 ' |
| R1 | 5 'GAGCGTTTGCACCAACAGGCCATCATTTGTGACGAGAGAAGACCTGACGTCATTAGATTGGCACCTTTGC GTAAAACGACGGCCAGTGAATTC 3 ' |
| F2 | 5 'GAGAATTGAAGAAGAGTTGGGAGACAATGCTATCTATGCTGGTAAGGACTTCCACAATGCTCAAACTTTG GGTGGTGTTTCAAAAGGTGAAGAAGATAAT 3 ' |
| R2 | 5 'GAGCGTTTGCACCAACAGGCCATCATTTGTGACGAGAGAAGACCTGACGTCATTAGATTGGCACCTTTGC ACTGGATGGCGGCGTTAGTATC 3 ' |
טבלה 1: רצפים פריימר השתמשו במחקר זה. טקסט נטוי מודגש מציין הומולוגיה אל מוקד ENO1 גנומית, אזורי גופן נורמלים הם הומולוגיים פלסמיד דנ"א.
- פריימרים עיצוב להיות הומולוגיים פלסמיד רצפים הגובלים קלטת להיות מוגבר וכן 3'-end של הגן היעד של עניין (למשל ENO1) כדי להקל על רקומבינציה לתוך מוקד הגנומי של הגן (איור 2 ולוח 1) .
- ודא כי הרצפים פריימר קדימה להתאים 70 זוגות הבסיסים האחרונים (נ"ב) של הגן של עניין, 5'- 3 ', מינוס קודון העצירה, כדי לשמור על מסגרת הקידוד, בתוספת BP כ -30 הראשונים של רצף פלסמיד להיות מוגבר. בתור הערה, את GGTGGTGGTT בכל פריימר הוא מקשר גליצין-פולי ללא הומולוגיה FP. מן ראוי לציין כי, בהעדר מקשר, לא יכול להיות היתוך ישיר של תחומים פונקציונליים, אשר באופן תיאורטי יכול להוביל misfolding חלבון, תשואה נמוכה בייצור חלבון, או את פעילות ביולוגית של לקוי.
- ודא כי הרצפים פריימר ההפוכים הם 70 נ"ב רק במורד זרם של גן, 3'- 5 ', לא כולל כל רצף גן, בתוספת appro האחרוןximately 30 נ"ב של הסמן התזונתי או סמי התנגדות המשמש את הפלסמיד.
- השתמש F1 ו- R1 כדי ליצור GFP- NAT1 ו YFP- NAT1 קלטות עם pMG2120 ו pMG2263, בהתאמה ו F2 ו R2 כדי ליצור mCherry- NAT1 עם pMG2343.
3. צור FP קלטות ידי PCR (יום 1)
- כן ריאגנטים עבור PCR. הכן תערובת מאסטר (500 נפח סופי μl) על ידי הוספת כמויות וריכוזים הבאים אל צינור 1.5 מ"ל: 50 μl חיץ PCR (אשלגן כלורי 500 מ"מ, 100 מ"מ טריס pH 8.0 במים), 20 deoxynucleotides μl (dNTPs; תערובת המניות של נוקלאוטידים ב- 10 מ"מ כל אחד), 40 מגנזיום כלוריד μl 25 מ"מ, 20 μl מטוהרים פלסמיד (מ ~ 50-100 ng / פתרון מניות μl), 10 μl כל קדימה לאחור תחל (מ -10 פתרונות מניות מ"מ), 30 תקי μl פולימראז (גנריות, 5,000 יחידות / מ"ל), ו 320 מים μl.
- Aliquot 50 μl של תמהיל מאסטר לתוך כל אחד 10 PCR-compatible 0.5 מ"ל צינורות.
- מניחים צינורות PCR ב thermocycler ולהפעיל את השלבים הבאים: 1 מחזור של 5 דק 'ב 94 ° C עד לפגל dsDNA; 40 מחזורים ברצף של 45 שניות על 94 מעלות צלזיוס, 30 שניות על 55 מעלות צלזיוס, כדי לאפשר פריימרים כדי לחשל את תבנית ה- DNA פלסמיד, ו -4 דקות ב 68 מעלות צלזיוס למשך הארכת מוצרי DNA; ו 1 מחזור רחב סופי של 15 דקות ב 72 מעלות צלזיוס.
הערה: מיקס מאסטר PCR ופרמטרים אופניים ייתכן שיהיה צורך לשנות מבוסס על פולימראז תקי מסוים בשימוש. - ברכה כל המוצרים מ -10 תגובות PCR צינור 1.5 מיליליטר.
- נושא 5 μl של מוצר ה- PCR נקווה כדי ג'ל אלקטרופורזה agarose לאמת גודל amplicon ולקבל אומדן של ריכוז המוצר, המבוסס על השוואה סולם DNA. באופן כללי, השתמשו ~ 250 מיקרוגרם של ה- DNA קלטת בכל תמהיל שינוי שלאחר מכן.
- המשקע DNA על ידי הוספת 50 μl 3 נתרן אצטט M ואחריו 750 μl 95% אתנול למוצרים דגירה לפחות 30 דק 'ב -20 ° C.
- קציר את מוצרי ה- PCR על ידי צנטריפוגה את הצינור ב XG 16,000 למשך 10 דקות. מוציאים בזהירות וזורקים supernatant ומייבשים גלולה לילה. Resuspend גלולה קלטת DNA מיובש 40 μl TE pH 8.0 ולאחסן בטמפרטורת החדר עד לשימוש.
4. משני תאי קנדידה עם FP DNA קלטות
- ביום 1, לשחזר זן שמרים להפוך ממורי גליצרול 15% קפוא (-80 ° C) מניות על ידי מפוספס על כמה גבישים מגורדים על דקסטרוז שמרים peptone עם אדנין (YPAD) אגרתי לדגור על 30 מעלות צלזיוס. לאחר ההתאוששות של צמיחת מושבה, לחסן מושבה אחת לתוך מדיום YPAD נוזל 2 מיליליטר צינור תרבות זכוכית עם מכסה לנשימת דגירת הלילה בשעת 30 ° C עם תסיסה.
- ביום 2, לדלל 300 μl של תרבית שמרים הלילה לתוך 50 מ"ל YPAD טריים (עד OD הסופי 600 של ~ 0.2) בבקבוק Erlenmeyer 125 מ"ל עם מכסה לנשימה. לנער על 30 מעלות צלזיוסעבור ~ 3 שעות (עד OD הסופי 600 ~ 0.6-0.8).
- יוצקים את תרבות הלילה לתוך צינור חרוטי 50 מ"ל ו גלולה התאים על ידי ספינינג במשך 5 דקות ב 1 XG 500 בצנטריפוגה בראש הטבלה.
- יוצקים מעל כראוי וזורקים supernatant. Resuspend התא גלולה במים מ"ל 5. Re-גלולת התאים על ידי צנטריפוגה שוב במשך 5 דקות ב 1500 XG בצנטריפוגה בראש הטבלה.
- יוצקים מעל כראוי וזורקים supernatant. Resuspend התאים 500 μl TELiAc (אצטט ליתיום TE: 10 מ"מ טריס, pH 8.0, 1 mM EDTA, pH 8.0, 0.1 מ 'אצטט ליתיום) בעת העברת לצינור 1.5 מ"ל. צנטריפוגה הצינור במשך 2 דקות ב 3000 XG ב microcentrifuge.
- תאים Resuspend ב 250 TELiAc μl. ההיקף הכולל כולל הגלולה צריך להיות ~ 300 μl.
- כדי נקי (שפופרת microfuge שונה מאשר 4.2.4), להוסיף 5 DNA המוביל μl (10 מ"ג / מ"ל) ו -150 μl של תאים מוכנים קנדידה (מ 4.2.4). זהו בקרה שלילית עבור transformation.
- לצינור microfuge השני נקי, להוסיף 5 μl של ה- DNA המוביל מפוגל (כי כבר מבושל על 90 מעלות צלזיוס למשך 10 דקות ו מקורר 4 ° C), כל 40 μl של מוצר ה- PCR מוכן (מ 3.3.1) ו -150 μl של תאי קנדידה מוכנים (מ 4.2.4).
- דגירה תערובות טרנספורמציה שני למשך 30 דקות בטמפרטורת החדר.
- צינור אחד לערבב טרנספורמציה, להוסיף 700 תמהיל צלחת μl (10 מ"מ טריס, pH 8.0, 1 mM EDTA, pH 8.0, 0.1 מ 'אצטט ליתיום פוליאתילן גליקול 50% 3350). להפוך את הצינורות כדי לערבב דגירה אותם בן לילה ב RT.
- ביום 3, דגירה טרנספורמציה מתערבב על 42 מעלות צלזיוס במשך שעה 1. (הלם חום).
- צנטריפוגה טרנספורמציה מתערבב במשך 30 שניות XG 16,000 ב microcentrifuge. הסר כראוי וזורקים supernatant. Resuspend כל של כדורי תא מים 150 μl ידי pipetting בעדינות מעלה ומטה כדי שלא לגרום נזק לתאים.
- עבור טרנספורמציות utilizing גנים סמן auxotrophic (למשל URA3), צלחת כל התערובת כולה על ידי pipetting הפתרונות על אגר התקשורת סלקטיבית המתאימה (uridine למשל חסר) ולהפיץ את התערובת באופן שווה באמצעות חרוזי זכוכית סטרילית.
- עבור טרנספורמציות ניצול גן סמן התנגדות nourseothricin (NAT1), כפי שמתואר כאן, שינוי צלחת המערבב הראשון על אגרת YPAD הלא סלקטיבי לדגור על 30 מעלות צלזיוס למשך 6-12 שעות. עזרי צעד זה ב התאוששות תא, הלם חום פוסט, לפני nourseothricin לחץ מוחל.
- לאחר התאוששות הצמיחה חלקית, צלחת העתק התאים קנדידה על YPAD המכיל 400 מיקרוגרם / מ"ל nourseothricin. עבור טרנספורמציות ניצול גנים סמן תזונתיים (למשל URA3), צעד ציפוי ביניים זו אינה נחוצה ותאי יכול להיות מצופה ישירות על גבי מדיה שמרים סלקטיבית (uridine למשל YPAD חסר) כמתואר 4.4.2.
הערה: אם השינוי הוא מוצלח, colonies אמור להופיע, בתוך שלושה ימים (פוטנציאל עד חמישה ימים של תולדה לבחירה על אגרת nourseothricin המכיל). אין מושבות אמורות להופיע על צלחות להתפשט עם תערובות טרנספורמציה המכילות מוביל DNA לבד (שליטה שלילית).
- לבחירת סמן auxotrophic ו nourseothricin, transformants המשוערת פס כמו מושבות אחת צלחות אגרו תקשורת סלקטיבית טריות לדגור על 30 מעלות צלזיוס כדי להפיץ תאים שמרים כי יכול להיות מוקרן לבנייה מוצלחת של התכת FP.
- Transformants מסך לשילוב נכון של קלטת תיוג (ראה נציג תוצאות עבור דוגמה מפורטת). אם הגן של עניין מתבטא בסכומים מספיק, הקרינה המושבה כולה עלולה להתרחש כך אפשר לזהות integrants מועמד פוטנציאלי באמצעות מערכת הדמיה צלחת עם יכולת גילוי הקרינה.
- בדוק integrants המשוערת על ידי PCR באמצעות הומולוגיים פריימרים רצפים outsidדואר של האזור של אינטגרציה לאשר היתוך גן היעד.
- בנוסף, שקול ניתוח כתם המערבי על מנת לקבוע את הביטוי וגודל של החלבון היתוך, כמו גם על ידי מיקרוסקופ פלואורסצנטי של תאים בודדים עבור אישור חזותי של לוקליזציה חלבון, אם הוא ידוע.
תוצאות
כדוגמא, השתמשנו הפרוטוקול שתואר לעיל לבנות בשילובי GFP ו mCherry כדי Eno1 ב זן מעבדת parapsilosis ג. כל transformant המשוערת בתחילה restreaked לצמיחה. בדוגמא זו, שכן חלבון ההיתוך כתוצאה מבוטא בכמות גבוהה (אנולאז) ואת FPS בהיר, הצלחנו להקרין transformants ידי מיקרוסקופ פלואורסצ?...
Discussion
בניית epitope מתויגת רצפים במיני קנדידה באמצעות אסטרטגיית שינוי הגנטית בתיווך PCR שתוארה לעיל ניתן לסכם כמו תהליך בן שלושה שלבים. ראשית, קלטת נעשית על ידי PCR מקודד הוא הרצף רצוי לשילוב ואזורים הומולוגי אל המוקד להכנסה לתוך הגנום שמרים. שנית, תאי שהמרים חייב להשתנות עש...
Disclosures
החוקרים אין לי מה לחשוף.
Acknowledgements
אנו מודים נ דין למתן את רצף FP mCherry המקורי, מ 'Gerami-נג'אד לבנייה של פלסמידים, ב לארסון לקבלת סיוע טכני, וט Heisel עבור עצות מועילות במהלך הפיתוח של הפרויקט הזה. JB נתמכה על ידי בפרס מתקדם המועצה האירופית למחקר 340,087 (RAPLODAPT). מערכות מיקרוסקופיה הדמיה נמסרו על ידי אוניברסיטת קרן Pediatrics מינסוטה ואוניברסיטת מרכז הדמיה מינסוטה.
Materials
| Name | Company | Catalog Number | Comments |
| 100 W mercury lamp | CHIU Technical Corporation | M-100T | |
| 95% Ethanol | Any | NA | |
| Adenine | Any | NA | |
| Ampicillin | Any | NA | |
| Carrier DNA | Ambion | AM9680 | Sheared Salmon Sperm DNA 10 mg/ml |
| CCD Camera | Photometrics | CoolSNAP HQ | |
| Conical Tube | Corning | 430828 | 50 ml |
| Culture Tube Rotator | New Brunswick | 2013923 | TC-8, or Any Culture Tube Rotator |
| Deoxynucleotides (dNTP) PCR Grade | Any | NA | |
| Eppendorf Tubes | Eppendorf | 022363719, 022363212 | 0.5 ml, 1.5 ml |
| Erlenmeyer Flask | Fisher Scientific | 7250089 | 125 ml |
| Ethylenediaminetetraacetic Acid (EDTA) | Any | NA | |
| Freezer (-80 °C) | Thermo Electron Corporation | ULT-1386-9-V | Revco Ultima II |
| GFP, YFP and Texas Red Filter Sets | Chroma Technology Corporation | 49002, 86004v2, 49008 | |
| Glass culture tubes | Fisher Scientific | 1496126 | 75 mm |
| HRP goat anti-mouse antibody | Santa Cruz Biotechnology | SC-2005 | |
| HRP goat anti-rabbit antibody | Santa Cruz Biotechnology | SC-2301 | |
| Incubator (30 °C) | Any | NA | |
| Lithium Acetate | Any | NA | |
| Lysogeny Broth (LB) Media | Any | NA | |
| Magnesium Chloride | Any | NA | |
| Microcentrifuge | Eppendorf | 5415 D | |
| Microscope | Nikon | E600 | Nikon Eclipse E600 |
| Microscope Image Analysis Software | Universal Imaging Corporation | 6.3r7 | MetaMorph Software Series 6.3r7 |
| Mouse anti-GFP antibody | Roche | 11814460001 | |
| Nourseothricin | Fisher Scientific | 50997939 | |
| PCR Thermocycler | Applied Biosystems | 9700 | GeneAmp PCR System |
| PCR tubes | BioExpress, GeneMate | T-3035-1 | 0.2 ml |
| Polyethylene Glycol 3350 | Any | NA | |
| Potassium Chloride | Any | NA | |
| Rabbit anti-mCherry antibody | BioVision | 5993-100 | |
| Refrigerator (4 °C) | Any | NA | |
| Sodium Acetate | Any | NA | |
| Stereomicroscope | Nikon | SMZ1500 | |
| Table Top Centrifuge | Labnet | Z 400 | Hermle Z 400 |
| Taq DNA Polymerase | Any | NA | |
| Tris(hydroxymethyl)aminomethane (Tris) | Any | NA | |
| Vortex Mixer | Scientific Industries | SI-0236 | Vortex Genie 2 |
| Yeast Extract Peptone Dextrose (YPD) Media | Any | NA |
References
- Bendel, C. M. Colonization and epithelial adhesion in the pathogenesis of neonatal candidiasis. Semin. Perinatol. 27 (5), 357-364 (2003).
- Gerami-Nejad, M., Berman, J., Gale, C. A. Cassettes for PCR-mediated construction of green, yellow, and cyan fluorescent protein fusions in Candida albicans. Yeast. 18 (9), 859-864 (2001).
- Gerami-Nejad, M., Dulmage, K., Berman, J. Additional cassettes for epitope and fluorescent fusion proteins in Candida albicans. Yeast. 26 (7), 399-406 (2009).
- Gerami-Nejad, M., Forche, A., McClellan, M., Berman, J. Analysis of protein function in clinical C. albicans isolates. Yeast. 29 (8), 303-309 (2012).
- Gerami-Nejad, M., Hausauer, D., McClellan, M., Berman, J., Gale, C. Cassettes for the PCR-mediated construction of regulatable alleles in Candida albicans. Yeast. 21 (5), 429-436 (2004).
- Gonia, S., Larson, B., Gale, C. A. PCR-mediated gene modification strategy for construction of fluorescent protein fusions in Candida parapsilosis. Yeast. 33 (2), 63-69 (2016).
- Milne, S. W., Cheetham, J., Lloyd, D., Aves, S., Bates, S. Cassettes for PCR- mediated gene tagging in Candida albicans utilizing nourseothricin resistance. Yeast. 28 (12), 833-841 (2011).
- Ausubel, F. M., et al. . Current Protocols in Molecular Biology. , (1995).
- Wilson, R. B., Davis, D., Mitchell, A. P. Rapid hypothesis testing with Candida albicans through gene disruption with short homology regions. J. Bacteriol. 181 (6), 1868-1874 (1999).
- Pulver, R., et al. Rsr1 focuses Cdc42 activity at hyphal tips and promotes maintenance of hyphal development in Candida albicans. Eukaryotic Cell. 12 (4), 482-495 (2013).
- Falgier, C., et al. Candida species differ in their interactions with immature human gastrointestinal epithelial cells. Pediatr. Res. 69 (5), 384-389 (2011).
- Nosek, J., et al. Genetic manipulation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 42 (1), 27-35 (2002).
- Zemanova, J., Nosek, J., Tomaska, L. High-efficiency transformation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 45 (3), 183-186 (2004).
- Benjamin, D. K., et al. Neonatal candidiasis among extremely low birth weight infants: risk factors, mortality rates, and neurodevelopmental outcomes at 18 to 22 months. Pediatrics. 117 (1), 84-92 (2006).
- Kullberg, B. J., Arendrup, M. C. Invasive Candidiasis. N Engl J Med. 373 (15), 1445-1456 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved