Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Génération de fusions de protéines fluorescentes dans
Dans cet article
Résumé
La modification du gène à médiation par PCR peut être utilisée pour générer des fusions de protéines fluorescentes dans des espèces de Candida, ce qui facilite la visualisation et la quantification des cellules de levure et les protéines. Ici, nous présentons une stratégie pour la construction d' une protéine de fusion fluorescente (ENO1-FP) dans Candida parapsilosis.
Résumé
Les espèces de Candida, colonisateurs répandues des voies intestinales et génito, sont la cause de la majorité des infections fongiques invasives chez les humains. Ainsi, les outils moléculaires et génétiques sont nécessaires pour faciliter l'étude de leurs mécanismes de pathogénicité. modification génique par PCR est une approche simple et rapide pour générer des protéines de marquage épitopique pour faciliter leur détection. En particulier, la protéine fluorescente (PF), les fusions sont des outils puissants qui permettent la visualisation et la quantification des deux cellules de levure et des protéines par microscopie à fluorescence et immunotransfert, respectivement. Les plasmides contenant des séquences codant pour la PF, ainsi que des gènes marqueurs nutritionnels qui facilitent la transformation des espèces de Candida, ont été produites à des fins de construction et d' expression dans la PF Candida. Ici, nous présentons une stratégie pour la construction d' une fusion FP dans une espèce de Candida. Les plasmides contenant l'antitache nourseothricinence gène marqueur de transformation (en NAT1) , ainsi que des séquences soit pour le vert, le jaune, ou la cerise vps (GFP, YFP, mCherry) sont utilisées conjointement avec des amorces qui comprennent des séquences spécifiques d' un gène dans une réaction en chaîne par polymérase (PCR) pour générer une cassette FP . Cette cassette spécifique du gène a la possibilité d'intégrer dans l'extrémité 3 'du locus du gène correspondant par l'intermédiaire d'une recombinaison homologue. Réussi fusion en cadre de la séquence PF dans le locus du gène d'intérêt est contrôlée génétiquement, suivie d'une analyse de l'expression de la protéine de fusion par microscopie et / ou des méthodes d'immuno-détection. En outre, pour le cas des protéines hautement exprimés, des fusions avec succès peuvent être criblés principalement par les techniques d'imagerie par fluorescence.
Introduction
Les espèces de Candida sont des champignons commensaux qui colonisent le tractus intestinal et génito de tous les humains. Dans des conditions d'immunodéficience, telles que celle se produisent avec une naissance prématurée ou d' effets immunosuppresseurs de traitements pour le cancer, les espèces de Candida peuvent devenir pathogènes opportunistes. Parmi les espèces de Candida, Candida albicans est le colonisateur fongique la plus répandue et provoque la majorité des infections fongiques invasives. D' autres espèces de Candida telles que C. glabrata, C. parapsilosis, C. tropicalis et C. kruseii aussi causer des infections graves chez les patients immunodéprimés, avec une certaine résistance intrinsèque présentant aux antibiotiques couramment utilisés anti-fongiques comme le fluconazole et l' amphotéricine B. Par conséquent, infections avec certaines de ces espèces sont observées plus fréquemment, en particulier chez les patients traités à titre prophylactique avec des agents anti-fongiques. Même avec un approprié et opportuntraitement nti-fongiques, les infections à Candida invasives continuent d'être associée à une morbidité et une mortalité significatives. En raison de l'importance des espèces de Candida dans la santé humaine, il y a un besoin d'outils moléculaires facilement disponibles qui permettent l'étude et l' élucidation de leurs mécanismes de pathogénicité.
Un outil important qui permet aux chercheurs de visualiser et de quantifier les cellules microbiennes et les protéines qu'ils expriment est la technologie de fusion FP. Réaction en chaîne par polymérase (PCR) de modification de gène médiée, tel que décrit dans le présent document, permet la construction de fusions, entre les séquences de PF et une séquence codante d'intérêt à son locus génomique protéine Candida. Une intégration stable de la construction facilite l'analyse de l'expression des protéines ainsi que la dynamique des protéines de localisation. Les plasmides contenant des séquences de PF, optimisés pour l' expression dans Candida albicans et que peuvent être utilisés dans la g médiée par PCRstratégie de modification ène ont été préalablement construits 2, 3, 4, 5. Plasmides contiennent PF transformation «cassettes»: une séquence de FP lié à un gène marqueur nutritionnel qui favorise la transformation de C. albicans et C. parapsilosis 2, 3, 4, 5, 6, 7. À l' heure actuelle plasmides disponibles contiennent une variété de gènes sélectionnables marqueurs nutritionnels (URA3 His1, ARG4) pour la transformation des souches auxotrophes, ainsi qu'un marqueur de résistance aux médicaments dominante (NAT1), ce qui facilite la transformation des souches cliniques dépourvues auxotrophies. En outre, des plasmides contiennent des options pour jusqu'à quatre séquences différentes de PF (vert [GFP], yellow [YFP], cyan [PCP] et de cerise [mCherry]) et soit une séquence de terminaison ADH1 pour la construction d' un carboxy-terminale des protéines de fusion, ou une séquence de promoteur pour la construction d'amino-terminale des protéines de fusion. Les amorces sont conçues avec une homologie avec l'ADN du plasmide entourant la cassette FP. En outre, les amorces contiennent également des séquences 5 ' d' extension portant une homologie avec le gène de levure d'intérêt à marquer, ce qui facilite l' intégration de la cassette dans le locus génomique par recombinaison homologue (figure 1). Spécifique du gène FP cassettes sont générés par PCR et ensuite transformés en cellules de Candida rendues compétentes pour l' absorption de l' ADN par un traitement avec de l' acétate de lithium.

Figure 1: Schéma de la façon dont les fusions de séquences PF sont générées dans les espèces de Candida. (A) L' ADN de plasmide includes une séquence de PF et une séquence codant pour la résistance nourseothricine (NAT1). emplacements relatifs de l'avant (FWD) et inverse (REV) les amorces sont représentées, avec des parties noires des amorces indiquant la région d'homologie à la séquence plasmidique et les portions violet dénotant la région d'homologie spécifique d'un gène ou d'une extension d'amorce. Cassettes (B) PF sont transformées en Candida et à intégrer au sein du locus génomique de ENO1 par recombinaison homologue (lignes en pointillés). (C) résultant séquence de fusion FP à l'extrémité 3 du ENO1. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Ici, nous présentons un exemple de la protéine de fusion (ENO1-FP) constructions dans les espèces de Candida. Nous utilisons le marquage des plasmides contenant le gène marqueur de transformation NAT1 avec des séquences codant pour la GFP, YFP, oumCherry (figure 2). Ces plasmides sont utilisés conjointement avec des amorces de PCR pour générer des cassettes spécifiques de gènes qui facilitent la fusion de PCR à l'extrémité 3 'de ENO1, ayant pour résultat l' expression de ENO1 fusionnée à son extrémité carboxy à MF-terminale.
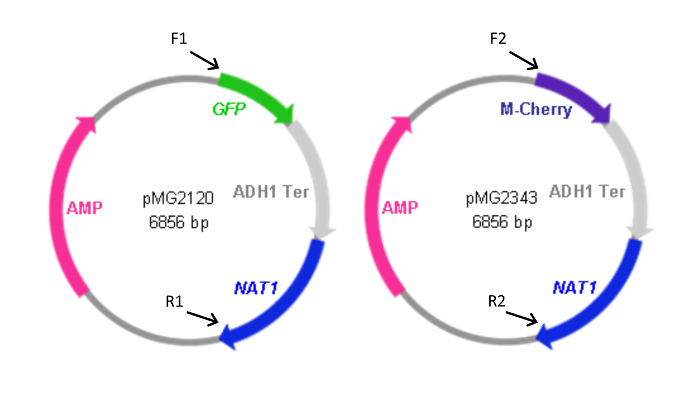
Figure 2: Carte de PF plasmides contenant la cassette. Vers l'avant (F) et arrière (R) des amorces utilisées pour générer les cassettes à partir des plasmides sont indiqués ainsi que la position relative de leur homologie avec les plasmides. Les séquences des amorces sont énumérées dans le tableau 1. F1 et R1 ont également été utilisés pour générer la cassette pYFP- NAT1. Le plasmide contenant la cassette YFP- NAT1 (pMG2263) est identique à pMG2120 à l'exception de la YFP à la place de la séquence de la GFP. Tailles de cassettes: GFP-Nat1, 3,7 kbp; mCherry- NAT1, 3,2 kbp; YFP- NAT1, 3.7 kpb. Ce chiffre a été modifié depuis Gerami-Nejad, et al. 4 S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Isoler Template Plasmides de E. coli
- Cultiver E. coli contenant le plasmide de matrice pendant une nuit dans 10 ml de bouillon lysogénie (LB) + 200 mg / L d'ampicilline (AMP) à 37 ° C sous agitation.
- Récolte des cellules par centrifugation à 6000 g pendant 2 min.
- Décanter le liquide, isoler et purifier l' ADN de cellules de E. coli par un procédé standard tel que décrit précédemment dans Ausubel et al. 8.
- ADN remettre en suspension dans du Tris-EDTA (TE, Tris 10 mM, pH 8,0, EDTA 1 mM, pH 8,0) à une concentration de travail de 50-100 ng / ml.
2. Conception Amorces
| Apprêt | séquence d' amorce |
| F1 | 5 ' GAGAATTGAAGAAGAGTTGGGAGACAATGCTATCTATGCTGGTAAGGACTTCCACAATGCTCAAACTTTG GGTGGTTCTAAAGGTGAAGAATTATT 3 ' |
| R1 | 5 'GAGCGTTTGCACCAACAGGCCATCATTTGTGACGAGAGAAGACCTGACGTCATTAGATTGGCACCTTTGC GTAAAACGACGGCCAGTGAATTC 3 ' |
| F2 | 5 'GAGAATTGAAGAAGAGTTGGGAGACAATGCTATCTATGCTGGTAAGGACTTCCACAATGCTCAAACTTTG GGTGGTGTTTCAAAAGGTGAAGAAGATAAT 3 ' |
| R2 | 5 'GAGCGTTTGCACCAACAGGCCATCATTTGTGACGAGAGAAGACCTGACGTCATTAGATTGGCACCTTTGC ACTGGATGGCGGCGTTAGTATC 3 ' |
Tableau 1: séquences des amorces utilisées dans cette étude. Le texte en italique gras indique une homologie avec le locus génomique de ENO1, les régions de police normales sont homologues à l' ADN plasmidique.
- Conception des amorces pour être homologue au plasmide les séquences bordant la cassette à amplifier ainsi qu'à l'extrémité 3 'du gène cible d'intérêt (par exemple ENO1) pour faciliter la recombinaison dans locus génomique du gène (figure 2 et tableau 1) .
- Faire en sorte que les séquences d'amorce avant correspondent aux 70 dernières paires de bases (pb) du gène d'intérêt, 5'- 3 ', moins le codon d'arrêt, afin de maintenir la trame de codage, ainsi que la première approximativement 30 pb de la séquence du plasmide à être amplifié. Comme une note, l'GGTGGTGGTT dans chaque amorce est un lieur de poly-glycine avec aucune homologie FP. Il convient de noter qu'en l'absence d'une liaison, il peut y avoir fusion directe des domaines fonctionnels, qui peuvent théoriquement conduire à un mauvais repliement des protéines, avec un faible rendement dans la production de protéines ou de troubles de la bioactivité.
- Veiller à ce que les séquences d'amorces inverses sont 70 pb juste en aval du gène, 3'- 5 ', ne comprenant pas la séquence du gène, ainsi que le dernier approximately 30 pb du marqueur nutritionnel ou la résistance aux médicaments utilisés dans le plasmide.
- Utilisez F1 et R1 pour générer GFP Nat1 et YFP- Nat1 cassettes avec pMG2120 et pMG2263, respectivement , et F2 et R2 pour générer mCherry- NAT1 avec pMG2343.
3. Générer FP Cassettes par PCR (Jour 1)
- Préparer des réactifs pour la PCR. Faites un mélange maître (500 volume final ul) en ajoutant les volumes et les concentrations suivantes à un tube de 1,5 ml: tampon de 50 ul PCR (500 mM de chlorure de potassium, 100 mM Tris pH 8,0 dans l'eau), désoxynucléotides 20 pi (dNTP; mélange des stocks de nucléotides à 10 mM chacun), le chlorure de magnésium 40 ul de 25 mM, 20 pl purifiées plasmidique (de ~ 50-100 ng / pl solution mère), 10 ul chacun avant et primaire (à partir de solutions mères 10 mM inverse), 30 Taq ul polymérase (générique, 5000 unités / ml) et 320 ul d'eau.
- Aliquoter 50 pi de mélange maître dans chacune des 10 PCR-CompatibLe tubes de 0,5 ml.
- Placer les tubes PCR en thermocycleur et exécuter les étapes suivantes: 1 cycle de 5 min à 94 ° C pour dénaturer dsDNA; 40 cycles de 45 secondes de manière séquentielle à 94 ° C, 30 s à 55 ° C pour permettre aux amorces hybrident avec l'ADN matrice du plasmide, et 4 min à 68 ° C pour l'extension des produits d'ADN; et 1 cycle d'extension finale de 15 min à 72 ° C.
peuvent avoir besoin d'être modifié en fonction de la polymérase Taq particulier utilisé Master Mix PCR et cyclistes paramètres: NOTE. - Piscine tous les produits des réactions 10 PCR dans un tube de 1,5 ml.
- Sous réserve 5 pi de produit de PCR mis en commun à une électrophorèse sur gel d'agarose pour vérifier la taille de l'amplicon et obtenir une estimation de la concentration du produit, sur la base de la comparaison à une échelle de l'ADN. En règle générale, utiliser ~ 250 pg d'ADN de cassette dans chaque mélange de transformation ultérieure.
- Précipiter l'ADN par addition d'acétate 50 ul de sodium 3 M, suivi par 750 ul d'éthanol à 95% pour les produits et incuber pendant au moins 30 minà -20 ° C
- Récolter les produits de la PCR par centrifugation du tube à 16 000 xg pendant 10 min. Retirez délicatement et jeter le surnageant et sécher le culot du jour au lendemain. Resuspendre la cassette culot d'ADN séché dans 40 pi de TE pH 8,0 et stocker à température ambiante jusqu'à utilisation.
4. Transformer des cellules de Candida avec FP DNA Cassettes
- Le jour 1, récupérer la souche de levure à transformer à partir d'un glycérol de 15% congelé (-80 ° C) disponible en striant quelques cristaux grattées SUR DES levure peptone dextrose avec adénine (YPAD) agar et incuber à 30 ° C. Après rétablissement de la croissance des colonies, inoculer une colonie unique dans 2 ml de milieu YPAD liquide dans un tube de culture en verre avec un bouchon perméable à l'air et laisser incuber pendant une nuit à 30 ° C sous agitation.
- Le jour 2, diluer 300 pi de culture de levure pendant la nuit dans 50 ml YPAD frais (à une DO finale 600 de ~ 0,2) dans un Erlenmeyer de 125 ml avec un bouchon perméable à l' air. Agiter à 30 ° Cpour ~ 3 h (à une DO finale 600 ~ 0,6-0,8).
- Verser la culture de la nuit dans un tube conique de 50 ml et sédimenter les cellules par centrifugation pendant 5 minutes à 1 500 xg dans une centrifugeuse de table.
- Décanter et correctement jeter le surnageant. Remettre en suspension le culot cellulaire dans 5 de l'eau. Re-sédimenter les cellules par centrifugation à nouveau pendant 5 min à 1500 x g dans une centrifugeuse de dessus de table.
- Décanter et correctement jeter le surnageant. Remettre en suspension les cellules dans 500 ul TELiAc (TE acétate de lithium: Tris 10 mM, pH 8,0, EDTA 1 mM, pH 8,0, 0,1 M d'acétate de lithium) pendant le transfert dans un tube de 1,5 ml. Centrifuger le tube pendant 2 min à 3000 xg dans une microcentrifugeuse.
- Resuspendre les cellules dans 250 TELiAc ul. Le volume total y compris le culot devrait être ~ 300 pi.
- À un (microtube différent que dans 4.2.4) propre, ajouter 5 ul d' ADN de support (10 mg / ml) et 150 ul de cellules de Candida préparées ( à partir 4.2.4). Ceci est le contrôle négatif pour transformation.
- Un second tube microfuge propre, ajouter 5 pl d'ADN porteur dénaturé (qui a été bouilli à 90 ° C pendant 10 minutes et refroidi à 4 ° C), les 40 ul du produit de PCR préparé (de 3.3.1) et 150 ul de cellules de Candida préparés (de 4.2.4).
- Incuber les deux mélanges de transformation pendant 30 min à température ambiante.
- A chaque tube de mélange de transformation, ajouter 700 pi de mélange de plaque (Tris 10 mM, pH 8,0, EDTA 1 mM, pH 8,0, 0,1 M d'acétate de lithium dans 50% de polyéthylèneglycol 3350). Inversez les tubes à mélanger et à les incuber pendant une nuit à température ambiante.
- Le Jour 3, incuber la transformation se mélange à 42 ° C pendant 1 heure (de choc thermique).
- Centrifugeuse la transformation se mélange pendant 30 secondes à 16.000 xg dans une microcentrifugeuse. Retirez et jetez-le surnageant. La remise en suspension de chacune des pastilles de cellules dans 150 ul d'eau en pipettant doucement vers le haut et vers le bas de manière à ne pas endommager les cellules.
- Pour les transformations utilizing gènes marqueurs auxotrophes (par exemple URA3), plaque chaque mélange ensemble par pipetage des solutions sur la gélose de milieu sélectif approprié (par exemple manquant d' uridine) et étaler le mélange uniformément à l' aide de billes de verre stériles.
- Pour les transformations utilisant le gène marqueur de résistance à nourseothricine (de NAT1), comme décrit ici, la transformation de la plaque premier mélange sur de la gélose YPAD non sélectif et incuber à 30 ° C pendant 6 à 12 heures. Cette étape facilite la récupération des cellules, le choc thermique post, avant nourseothricine le stress est appliqué.
- Après reprise de la croissance partielle, plaque réplique les cellules de Candida YPAD SUR DES contenant 400 ug / ml nourseothricine. Pour des transformations utilisant des gènes marqueurs nutritionnels (par exemple , URA3), cette étape de métallisation intermédiaire est pas nécessaire et les cellules peuvent être directement plaquées sur des milieux sélectifs de levure (par exemple , de YPAD manquant d' uridine) comme décrit dans 4.4.2.
NOTE: Si la transformation est réussie, colOnies devrait apparaître dans un à trois jours (potentiellement jusqu'à cinq jours de l'excroissance pour la sélection sur gélose nourseothricine contenant). Aucune colonie doivent apparaître sur des plaques réparties avec des mélanges de transformation contenant de l'ADN de support seul (témoin négatif).
- Pour la sélection du marqueur d'auxotrophie et nourseothricine, streak plantes transformées putatives comme des colonies isolées sur des plaques d'agar de milieu sélectif frais et on incube à 30 ° C pour propager des cellules de levure qui peuvent être criblés pour la construction réussie de fusion PF.
- Transformants d'écran pour l' intégration correcte de la cassette de marquage (voir résultats représentatifs pour un exemple détaillé). Si le gène d'intérêt est exprimé en des quantités suffisantes, la fluorescence de colonie entière peut se produire de telle sorte qu'il est possible de détecter des intégrants candidats potentiels en utilisant un système d'imagerie de la plaque avec la capacité de détection de fluorescence.
- Vérifiez intégrants putatifs par PCR en utilisant des amorces homologues aux séquences outside de la région d'intégration pour confirmer la fusion du gène cible.
- En outre, envisager une analyse par transfert de Western pour déterminer l'expression et la taille de la protéine de fusion, ainsi que par microscopie à fluorescence de cellules uniques pour la confirmation visuelle de la localisation de la protéine, si elle est connue.
Access restricted. Please log in or start a trial to view this content.
Résultats
A titre d'exemple, nous avons utilisé le protocole décrit ci - dessus pour construire GFP et mCherry fusions à ENO1 dans une souche de laboratoire de parapsilosis C.. Chaque transformant putatif a été initialement réensemencer en stries pour la croissance. Dans cet exemple, étant donné que la protéine de fusion résultante est fortement exprimée (énolase) et les médecins de famille sont lumineuses, nous avons été en mesure de dépister les transformants par mic...
Access restricted. Please log in or start a trial to view this content.
Discussion
Construction d'épitope tagged séquences dans les espèces de Candida en utilisant la stratégie de modification génique par PCR décrit ci - dessus peuvent être résumées comme un processus en trois étapes. Tout d'abord, la cassette est réalisée par PCR qui code à la fois la séquence souhaitée pour l'intégration et des régions homologues au locus d'insertion dans le génome de la levure. D'autre part, les cellules de levure à transformer chimiquement compétentes sont effectué...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n'ont rien à dévoiler.
Remerciements
Nous remercions N. Dean pour fournir la séquence mCherry FP d'origine, M. Gerami-Nejad pour la construction de plasmides, B. Larson pour l'assistance technique, et T. Heisel pour obtenir des conseils utiles lors de l'élaboration de ce projet. JB a été soutenu par le Prix du Conseil européen de la recherche avancée 340087 (RAPLODAPT). systèmes de microscopie et d'imagerie ont été fournis par la Fondation Université de Minnesota Pediatrics et l'Université du Minnesota Imaging Center.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 100 W mercury lamp | CHIU Technical Corporation | M-100T | |
| 95% Ethanol | Any | NA | |
| Adenine | Any | NA | |
| Ampicillin | Any | NA | |
| Carrier DNA | Ambion | AM9680 | Sheared Salmon Sperm DNA 10 mg/ml |
| CCD Camera | Photometrics | CoolSNAP HQ | |
| Conical Tube | Corning | 430828 | 50 ml |
| Culture Tube Rotator | New Brunswick | 2013923 | TC-8, or Any Culture Tube Rotator |
| Deoxynucleotides (dNTP) PCR Grade | Any | NA | |
| Eppendorf Tubes | Eppendorf | 022363719, 022363212 | 0.5 ml, 1.5 ml |
| Erlenmeyer Flask | Fisher Scientific | 7250089 | 125 ml |
| Ethylenediaminetetraacetic Acid (EDTA) | Any | NA | |
| Freezer (-80 °C) | Thermo Electron Corporation | ULT-1386-9-V | Revco Ultima II |
| GFP, YFP and Texas Red Filter Sets | Chroma Technology Corporation | 49002, 86004v2, 49008 | |
| Glass culture tubes | Fisher Scientific | 1496126 | 75 mm |
| HRP goat anti-mouse antibody | Santa Cruz Biotechnology | SC-2005 | |
| HRP goat anti-rabbit antibody | Santa Cruz Biotechnology | SC-2301 | |
| Incubator (30 °C) | Any | NA | |
| Lithium Acetate | Any | NA | |
| Lysogeny Broth (LB) Media | Any | NA | |
| Magnesium Chloride | Any | NA | |
| Microcentrifuge | Eppendorf | 5415 D | |
| Microscope | Nikon | E600 | Nikon Eclipse E600 |
| Microscope Image Analysis Software | Universal Imaging Corporation | 6.3r7 | MetaMorph Software Series 6.3r7 |
| Mouse anti-GFP antibody | Roche | 11814460001 | |
| Nourseothricin | Fisher Scientific | 50997939 | |
| PCR Thermocycler | Applied Biosystems | 9700 | GeneAmp PCR System |
| PCR tubes | BioExpress, GeneMate | T-3035-1 | 0.2 ml |
| Polyethylene Glycol 3350 | Any | NA | |
| Potassium Chloride | Any | NA | |
| Rabbit anti-mCherry antibody | BioVision | 5993-100 | |
| Refrigerator (4 °C) | Any | NA | |
| Sodium Acetate | Any | NA | |
| Stereomicroscope | Nikon | SMZ1500 | |
| Table Top Centrifuge | Labnet | Z 400 | Hermle Z 400 |
| Taq DNA Polymerase | Any | NA | |
| Tris(hydroxymethyl)aminomethane (Tris) | Any | NA | |
| Vortex Mixer | Scientific Industries | SI-0236 | Vortex Genie 2 |
| Yeast Extract Peptone Dextrose (YPD) Media | Any | NA |
Références
- Bendel, C. M. Colonization and epithelial adhesion in the pathogenesis of neonatal candidiasis. Semin. Perinatol. 27 (5), 357-364 (2003).
- Gerami-Nejad, M., Berman, J., Gale, C. A. Cassettes for PCR-mediated construction of green, yellow, and cyan fluorescent protein fusions in Candida albicans. Yeast. 18 (9), 859-864 (2001).
- Gerami-Nejad, M., Dulmage, K., Berman, J. Additional cassettes for epitope and fluorescent fusion proteins in Candida albicans. Yeast. 26 (7), 399-406 (2009).
- Gerami-Nejad, M., Forche, A., McClellan, M., Berman, J. Analysis of protein function in clinical C. albicans isolates. Yeast. 29 (8), 303-309 (2012).
- Gerami-Nejad, M., Hausauer, D., McClellan, M., Berman, J., Gale, C. Cassettes for the PCR-mediated construction of regulatable alleles in Candida albicans. Yeast. 21 (5), 429-436 (2004).
- Gonia, S., Larson, B., Gale, C. A. PCR-mediated gene modification strategy for construction of fluorescent protein fusions in Candida parapsilosis. Yeast. 33 (2), 63-69 (2016).
- Milne, S. W., Cheetham, J., Lloyd, D., Aves, S., Bates, S. Cassettes for PCR- mediated gene tagging in Candida albicans utilizing nourseothricin resistance. Yeast. 28 (12), 833-841 (2011).
- Ausubel, F. M., et al. Current Protocols in Molecular Biology. , Wiley. New York. (1995).
- Wilson, R. B., Davis, D., Mitchell, A. P. Rapid hypothesis testing with Candida albicans through gene disruption with short homology regions. J. Bacteriol. 181 (6), 1868-1874 (1999).
- Pulver, R., et al. Rsr1 focuses Cdc42 activity at hyphal tips and promotes maintenance of hyphal development in Candida albicans. Eukaryotic Cell. 12 (4), 482-495 (2013).
- Falgier, C., et al. Candida species differ in their interactions with immature human gastrointestinal epithelial cells. Pediatr. Res. 69 (5), 384-389 (2011).
- Nosek, J., et al. Genetic manipulation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 42 (1), 27-35 (2002).
- Zemanova, J., Nosek, J., Tomaska, L. High-efficiency transformation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 45 (3), 183-186 (2004).
- Benjamin, D. K., et al. Neonatal candidiasis among extremely low birth weight infants: risk factors, mortality rates, and neurodevelopmental outcomes at 18 to 22 months. Pediatrics. 117 (1), 84-92 (2006).
- Kullberg, B. J., Arendrup, M. C. Invasive Candidiasis. N Engl J Med. 373 (15), 1445-1456 (2015).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon