Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Generación de fusiones de proteínas fluorescentes en
En este artículo
Resumen
La modificación genética mediada por PCR se puede utilizar para generar fusiones de proteínas fluorescentes en especies de Candida, lo que facilita la visualización y cuantificación de células de levadura y las proteínas. Aquí, se presenta una estrategia para la construcción de una fusión proteína fluorescente (Eno1-FP) en Candida parapsilosis.
Resumen
Las especies de Candida, colonizadores frecuentes de los tractos intestinal y genitourinario, son la causa de la mayoría de las infecciones fúngicas invasivas en seres humanos. Por lo tanto, se necesitan herramientas moleculares y genéticos para facilitar el estudio de sus mecanismos de la patogénesis. la modificación genética mediada por PCR es un método sencillo y rápido para generar proteínas etiquetadas con epítopo para facilitar su detección. En particular, la proteína fluorescente (FP) fusiones son herramientas poderosas que permiten la visualización y cuantificación de ambas células de levadura y las proteínas por microscopía de fluorescencia y la inmunotransferencia, respectivamente. Los plásmidos que contienen secuencias de codificación de PF, junto con los genes marcadores nutricionales que facilitan la transformación de las especies Candida, se han generado con el propósito de la construcción FP y expresión en Candida. Aquí, se presenta una estrategia para la construcción de una fusión FP en una especie de Candida. Los plásmidos que contienen el sold nourseothricinna vez que el gen marcador de transformación (NAT1) junto con secuencias para cualquiera verde, amarillo, o de cerezas FPS (GFP, YFP, mCherry) se utilizan junto con los cebadores que incluyen secuencias de genes específicos en una reacción en cadena de la polimerasa (PCR) para generar un módulo de FP . Este casete de genes específicos tiene la capacidad de integrar en el extremo 3 'del locus del gen correspondiente a través de recombinación homóloga. El éxito de fusión en el marco de la secuencia de FP en el locus de gen de interés se verifica genéticamente, seguido por el análisis de la expresión de proteína de fusión por microscopía y / o métodos de inmunodetección. Además, para el caso de proteínas altamente expresados, fusiones exitosas pueden ser examinados para principalmente por técnicas de imagen de fluorescencia.
Introducción
Las especies de Candida son comensales hongos que colonizan el tracto intestinal y genitourinario de todos los seres humanos. En condiciones de inmunodeficiencia, tales como que se producen con el nacimiento prematuro o efectos inmunosupresores de los tratamientos para el cáncer, las especies de Candida pueden llegar a ser patógenos oportunistas. De las especies de Candida, Candida albicans es el colonizador fúngica más frecuente y causa la mayoría de las infecciones fúngicas invasivas. Otras especies de Candida, como C. glabrata, C. parapsilosis, C. tropicalis y C. También kruseii causan infecciones graves en pacientes inmunocomprometidos, con alguna resistencia intrínseca que presenta a los antibióticos comúnmente utilizados antifúngicos como fluconazol y anfotericina B. Por lo tanto, las infecciones con algunas de estas especies están siendo observados con mayor frecuencia, especialmente en pacientes que están siendo tratados profilácticamente con agentes anti-hongos. Incluso con una apropiada y oportunanti tratamiento de las infecciones fúngicas, infecciones invasivas por Candida continúan siendo asociado con morbilidad y mortalidad significativa 1. Debido a la importancia de especies de Candida en la salud humana, existe una necesidad de herramientas moleculares fácilmente disponibles que permiten el estudio y la elucidación de sus mecanismos de la patogénesis.
Una herramienta importante que permite a los investigadores visualizar y cuantificar las células microbianas y las proteínas que se expresan es la tecnología de fusión FP. Reacción en cadena de la polimerasa (PCR), la modificación genética mediada, tal como se describe en este documento, permite la construcción de fusiones, entre las secuencias de PF y una proteína de Candida secuencia codificante de interés en su locus genómico. La integración estable de la construcción facilita el análisis de la expresión de proteínas, así como la dinámica de localización de proteínas. Los plásmidos que contienen secuencias de PF, optimizados para la expresión en Candida albicans y que se pueden utilizar en la g mediada por PCRestrategia de modificación eno, se han construido previamente 2, 3, 4, 5. Los plásmidos contienen FP transformación "casetes": una secuencia FP ligado a un gen marcador nutricional que facilita la transformación de C. albicans y C. parapsilosis 2, 3, 4, 5, 6, 7. Actualmente plásmidos disponibles contienen una variedad de genes marcadores seleccionables nutricionales (URA3, His1, ARG4) para la transformación de las cepas auxotróficas, así como un marcador de resistencia dominante fármaco (NAT1), que facilita la transformación de cepas clínicas que carecen de auxotrophies. Además, los plásmidos que contienen opciones para hasta cuatro secuencias diferentes (FP verde [GFP], yellow [YFP], cian [PPC], y cereza [mCherry]) y, o bien una secuencia de terminación de ADH1 para la construcción de fusiones de proteínas carboxi-terminal, o una secuencia promotora para la construcción de fusiones de proteína amino-terminal. Los cebadores están diseñados con homología con el plásmido de ADN que rodea a la casete de FP. Además, los cebadores también contienen secuencias 5 'de extensión que llevan homología con el gen de levadura de interés para ser marcados, lo que facilita la integración del casete en el locus genómico mediante recombinación homóloga (Figura 1). Cassettes de PF de genes específicos son generados por PCR y luego se transformaron en células de Candida hechas competentes para la captación de ADN por tratamiento con acetato de litio.

Figura 1: Diagrama de cómo FP fusiones de secuencia se generan en las especies de Candida. (A) El ADN del plásmido INCLUYENDOes una secuencia FP y una secuencia que codifica resistencia nourseothricin (NAT1). ubicaciones relativas de Forward (FWD) y atrás primers (REV) se muestran, con partes negras de los cebadores que indica la región de homología con la secuencia del plásmido y las porciones de color púrpura que denotan la región de homología con el gen específico o extensión del cebador. Casetes (B) de PF se transforman en Candida y se integran dentro del locus genómico ENO1 mediante recombinación homóloga (líneas de puntos). (C) resultante secuencia de fusión FP al extremo 3 'de ENO1. Haga clic aquí para ver una versión más grande de esta figura.
Aquí, se presenta un ejemplo de construcciones de proteínas de fusión (Eno1-FP) en especies de Candida. Nos utilizan referencia de plásmidos que contienen el gen marcador de la transformación NAT1 junto con secuencias que codifican GFP, YFP, omCherry (Figura 2). Estos plásmidos se utilizan junto con cebadores en PCR para generar casetes de genes específicos que facilitan la fusión de los programas marco para el extremo 3 'de ENO1, lo que resulta en la expresión de Eno1 fusionado a programas marco en su extremo carboxi-terminal.
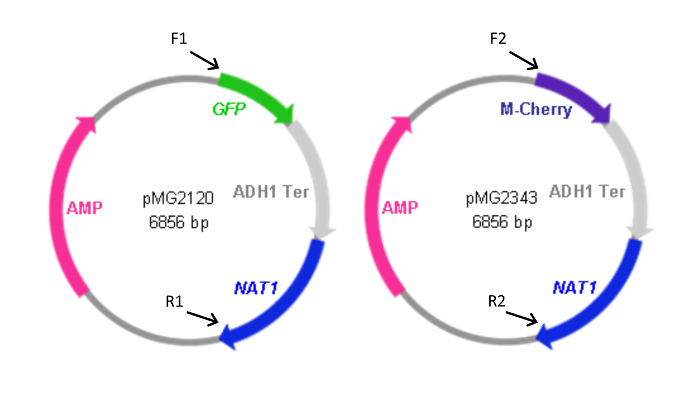
Figura 2: Mapas de los plásmidos que contienen cassettes-PF. Forward (F) y atrás primers (R) utilizados para generar los casetes de los plásmidos se indican junto con la ubicación relativa de su homología con los plásmidos. Las secuencias de cebador son como se indica en la Tabla 1. F1 y R1 también se utilizaron para generar el casete pYFP- NAT1. El plásmido que contiene el casete YFP- NAT1 (pMG2263) es idéntica a pMG2120 con la excepción de YFP en lugar de la secuencia de GFP. Los tamaños de casete: GFP-NAT1, 3,7 kbp; mCherry- NAT1, 3,2 kpb; YFP- NAT1, 3.7 kpb. Esta cifra ha sido modificado a partir de Gerami-Nejad, et al. 4 Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
1. Aislar Plantilla plásmidos de E. coli
- Crecer E. coli que contiene el plásmido de plantilla durante la noche en 10 ml de caldo de lysogeny (LB) + 200 mg / l de ampicilina (AMP) a 37 ° C con agitación.
- Cosecha células por centrifugación a 6.000 xg durante 2 min.
- Decantar líquido, aislar y purificar el ADN de células de E. coli por un método estándar como se describe anteriormente en Ausubel et al. 8.
- ADN resuspender en Tris-EDTA (TE; Tris 10 mM, pH 8,0, EDTA 1 mM, pH 8,0) a una concentración de trabajo de 50-100 ng / ml.
2. diseñar cebadores
| Cebador | Secuencia Primer |
| F1 | 5 ' GAGAATTGAAGAAGAGTTGGGAGACAATGCTATCTATGCTGGTAAGGACTTCCACAATGCTCAAACTTTG GGTGGTTCTAAAGGTGAAGAATTATT 3 ' |
| R1 | 5 'GAGCGTTTGCACCAACAGGCCATCATTTGTGACGAGAGAAGACCTGACGTCATTAGATTGGCACCTTTGC GTAAAACGACGGCCAGTGAATTC 3 ' |
| F2 | 5 'GAGAATTGAAGAAGAGTTGGGAGACAATGCTATCTATGCTGGTAAGGACTTCCACAATGCTCAAACTTTG GGTGGTGTTTCAAAAGGTGAAGAAGATAAT 3 ' |
| R2 | 5 'GAGCGTTTGCACCAACAGGCCATCATTTGTGACGAGAGAAGACCTGACGTCATTAGATTGGCACCTTTGC ACTGGATGGCGGCGTTAGTATC 3 ' |
Tabla 1: Primer secuencias utilizadas en este estudio. Negrita texto en cursiva indica homología con el locus genómico ENO1, las regiones de fuente normal son homólogas al plásmido ADN.
- cebadores diseño sea homóloga al plásmido secuencias que bordean el casete a amplificar, así como para el extremo 3 'del gen diana de interés (por ejemplo, ENO1) para facilitar la recombinación en el locus genómico del gen (Figura 2 y Tabla 1) .
- Asegúrese de que las secuencias de cebador directo se ajustan a los últimos 70 pares de bases (pb) del gen de interés, 5'-3 ', menos el codón de parada, para mantener el marco de codificación, además de la primera de aproximadamente 30 pb de la secuencia del plásmido sean amplificado. Como nota, la GGTGGTGGTT en cada cebador es un enlazador de poli-glicina con ninguna homología FP. Es de destacar que, en ausencia de un enlazador, no puede haber fusión directa de los dominios funcionales, que pueden conducir teóricamente a un mal plegamiento de proteínas, bajo rendimiento en la producción de proteínas, o alteración de la bioactividad.
- Asegúrese de que las secuencias de los cebadores inversos son 70 pb justo aguas abajo del gen, 3'-5 ', sin incluir cualquier secuencia de genes, además de la última aproximadamente 30 pb del marcador nutricional o resistencia a los medicamentos utilizados en el plásmido.
- Use F1 y R1 para generar casetes GFP NAT1 y YFP- NAT1 con pMG2120 y pMG2263, respectivamente, y F2 y R2 para generar mCherry- NAT1 con pMG2343.
3. Generar FP Cassettes por PCR (Día 1)
- Preparar los reactivos para la PCR. Hacer una mezcla maestra (500 l de volumen final) mediante la adición de los siguientes volúmenes y concentraciones a un tubo de 1.5 ml: 50 l de tampón de PCR (mM cloruro 500 de potasio, 100 mM Tris pH 8,0 en agua), desoxinucleótidos 20 l (dNTPs; mezcla madre de nucleótidos en 10 mM de cada uno), cloruro de magnesio 40 l 25 mM, 20 l purificados plásmido (de ~ 50-100 ng solución madre / l), 10 l cada uno hacia adelante y el cebador (de 10 mM de soluciones madre reversa), 30 l de Taq la polimerasa (genérico, 5.000 unidades / ml), y 320 l de agua.
- Alícuota de 50 l de mezcla maestra en cada uno de 10 PCR-compatiLe tubos de 0,5 ml.
- Coloque los tubos de PCR en el termociclador y ejecute los siguientes pasos: 1 ciclo de 5 min a 94 ° C para desnaturalizar ADN de doble cadena; 40 ciclos secuencialmente de 45 segundos a 94 ° C, 30 seg a 55 ° C para permitir que los cebadores hibriden a la plantilla de ADN plásmido, y 4 min a 68 ° C para la extensión de los productos de ADN; y 1 ciclo de extensión final de 15 min a 72 ° C.
pueden necesitar ser modificado en base a la polimerasa Taq particular, se utiliza la mezcla maestra de PCR y ciclismo parámetros: NOTA. - Piscina todos los productos de las 10 reacciones de PCR en un tubo de 1,5 ml.
- Asunto 5 l de producto de PCR agrupada a electroforesis en gel de agarosa para verificar tamaño de amplificación y obtener una estimación de la concentración del producto, basada en la comparación a una escalera de ADN. Generalmente, se utiliza ~ 250 g de ADN de casete en cada mezcla de transformación posterior.
- Precipitado de ADN mediante la adición de acetato de 50 l de sodio 3 M, seguido de 750 l 95% de etanol a los productos y se incuba al menos 30 mina -20 ° C.
- Cosecha de los productos de PCR mediante la centrifugación el tubo a 16.000 xg durante 10 min. Con cuidado, retirar y desechar el sobrenadante y secar el pellet durante la noche. Resuspender el precipitado casete de ADN se secó en 40 l de TE pH 8,0 y se almacena a temperatura ambiente hasta su uso.
4. transformar células de Candida con FP ADN Casetes
- En el Día 1, recuperar cepa de levadura que se transformó de un 15% de glicerol congelado (-80 ° C) de stock de rayas unos pocos cristales raspadas Onto dextrosa de levadura peptona agar con adenina (YPAD) y se incuba a 30 ° C. Después de la recuperación de crecimiento de la colonia, inocular una única colonia en 2 ml de medio YPAD líquido en un tubo de cultivo de vidrio con una tapa transpirable e incubar durante la noche a 30 ° C con agitación.
- En el Día 2, diluir a 300 l de cultivo de levadura durante la noche en 50 ml YPAD fresco (a una DO final 600 de ~ 0,2) en un matraz Erlenmeyer de 125 ml con una tapa transpirable. Se agita a 30 ° Cpara ~ 3 horas (a una DO final 600 ~ 0,6-0,8).
- Verter el cultivo de una noche en un tubo cónico de 50 ml y sedimentar las células por centrifugación durante 5 min a 1 500 xg en una centrífuga de mesa.
- Retirar y desechar adecuadamente el sobrenadante. Resuspender el sedimento celular en 5 ml de agua. Re-sedimentar las células por centrifugación de nuevo durante 5 min a 1.500 xg en una centrífuga de mesa.
- Retirar y desechar adecuadamente el sobrenadante. Resuspender las células en 500 l TELiAc (TE acetato de litio: 10 mM Tris, pH 8,0, EDTA 1 mM, pH 8,0, 0,1 M de acetato de litio) durante la transferencia a un tubo de 1,5 ml. Centrifugar el tubo durante 2 min a 3.000 xg en una microcentrífuga.
- Resuspender las células en 250 l TELiAc. El volumen total incluyendo el sedimento debe ser ~ 300 l.
- Para un (diferente tubo de microcentrífuga que en 4.2.4) limpio, añadir 5 l de ADN portador (10 mg / ml) y 150 ml de células de Candida preparados (desde 4.2.4). Este es el control negativo para transformación.
- A un segundo tubo de microcentrífuga limpio, añadir 5 l de ADN portador desnaturalizado (que ha sido hervida a 90 ° C durante 10 minutos y se enfrió a 4 ° C), los 40 l de producto de PCR (preparado a partir de 3.3.1) y 150 l de células de Candida preparados (de 4.2.4).
- Se incuban las dos mezclas de transformación para 30 min a temperatura ambiente.
- A cada tubo mezcla de transformación, añadir 700 l de la mezcla de placa (Tris 10 mM, pH 8,0, EDTA 1 mM, pH 8,0, 0,1 M de acetato de litio en 50% de polietilenglicol 3350). Invertir los tubos para mezclar e incubar durante la noche a TA.
- En el día 3, se incuba la transformación se mezcla a 42 ° C durante 1 hora (choque térmico).
- Se centrifuga la transformación se mezcla durante 30 segundos a 16.000 xg en una microcentrífuga. Retire y deseche adecuadamente el sobrenadante. Resuspender cada uno de los sedimentos de células en 150 l de agua pipeteando suavemente arriba y abajo con el fin de no dañar las células.
- Para las transformaciones utilizing genes marcadores auxotróficos (por ejemplo URA3), la placa de cada mezcla todo con la pipeta las soluciones sobre el agar medios selectivos apropiados (por ejemplo, carece de uridina) y extender la mezcla uniformemente con perlas de vidrio estériles.
- Para las transformaciones que utilizan el gen marcador de resistencia a nourseothricin (NAT1), como se describe aquí, la transformación placa se mezcla primero en agar YPAD no selectivo y se incuba a 30 ° C durante 6-12 h. Este paso permite la recuperación celular, choque térmico posterior, antes de nourseothricin se aplica esfuerzo.
- Después de la recuperación parcial del crecimiento, réplica de la placa las células de Candida Onto YPAD que contiene 400 mg / ml nourseothricin. Para las transformaciones que utilizan genes marcadores nutricionales (por ejemplo, URA3), este paso de recubrimiento intermedio no es necesario y las células puede ser plateado directamente sobre medios selectivos de levadura (por ejemplo YPAD que carece de uridina) como se describe en 4.4.2.
NOTA: Si la transformación se realiza correctamente, colOnies debería aparecer dentro de uno a tres días (potencialmente hasta cinco días de derivación para la selección en agar que contiene nourseothricin). No hay colonias deben aparecer en placas untadas con mezclas de transformación que contienen ADN portador solo (control negativo).
- Para la selección de marcador auxotrófico y nourseothricin, racha transformantes putativos como colonias individuales a placas de agar selectivo de los medios frescos y se incuba a 30 ° C para propagar células de levadura que pueden ser seleccionados para la construcción con éxito de fusiones de PF.
- Transformantes de pantalla para la correcta integración de la casete de marcado (ver resultados representativos para un ejemplo detallado). Si el gen de interés se expresa en cantidades suficientes, toda la fluorescencia colonia se puede producir de tal manera que es posible detectar potenciales integrantes candidatos utilizando un sistema de imagen de placa con capacidad de detección de fluorescencia.
- Compruebe integrantes putativos mediante PCR utilizando cebadores homólogos a las secuencias outside de la región de integración para confirmar la fusión al gen diana.
- Además, tener en cuenta el análisis de transferencia Western para determinar la expresión y el tamaño de la proteína de fusión, así como mediante microscopía de fluorescencia de células individuales para la confirmación visual de la localización de proteínas, si se conoce.
Resultados
Como ejemplo, se utilizó el protocolo descrito anteriormente para la construcción de GFP y mCherry fusiones a Eno1 en una cepa de C. parapsilosis laboratorio. Cada transformante putativo se volvieron a sembrar inicialmente para el crecimiento. En este ejemplo, ya que la proteína de fusión resultante es altamente expresado (enolasa) y los programas marco son brillantes, hemos sido capaces de detectar transformantes por microscopía de fluorescencia antes de realizar el diagn?...
Discusión
Construcción de epítopo etiquetado secuencias en las especies de Candida utilizando la estrategia de modificación del gen mediada por PCR descrito anteriormente se pueden resumir como un proceso de tres pasos. En primer lugar, un casete se hace por PCR que codifica tanto la secuencia deseada para la integración y regiones homólogas al locus de la inserción en el genoma de la levadura. En segundo lugar, las células de levadura para ser transformadas se hacen químicamente competente con acetato d...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a N. Dean para proporcionar la secuencia original mCherry FP, M. Gerami-Nejad para la construcción de plásmidos, B. Larson para la asistencia técnica, y T. Heisel útil para el asesoramiento durante el desarrollo de este proyecto. JB fue apoyado por el Premio del Consejo Europeo de Investigación Avanzada 340087 (RAPLODAPT). sistemas de microscopía e imagen fueron proporcionados por la Fundación Universidad de Minnesota Pediatría y la Universidad de Minnesota Centro de Imagen.
Materiales
| Name | Company | Catalog Number | Comments |
| 100 W mercury lamp | CHIU Technical Corporation | M-100T | |
| 95% Ethanol | Any | NA | |
| Adenine | Any | NA | |
| Ampicillin | Any | NA | |
| Carrier DNA | Ambion | AM9680 | Sheared Salmon Sperm DNA 10 mg/ml |
| CCD Camera | Photometrics | CoolSNAP HQ | |
| Conical Tube | Corning | 430828 | 50 ml |
| Culture Tube Rotator | New Brunswick | 2013923 | TC-8, or Any Culture Tube Rotator |
| Deoxynucleotides (dNTP) PCR Grade | Any | NA | |
| Eppendorf Tubes | Eppendorf | 022363719, 022363212 | 0.5 ml, 1.5 ml |
| Erlenmeyer Flask | Fisher Scientific | 7250089 | 125 ml |
| Ethylenediaminetetraacetic Acid (EDTA) | Any | NA | |
| Freezer (-80 °C) | Thermo Electron Corporation | ULT-1386-9-V | Revco Ultima II |
| GFP, YFP and Texas Red Filter Sets | Chroma Technology Corporation | 49002, 86004v2, 49008 | |
| Glass culture tubes | Fisher Scientific | 1496126 | 75 mm |
| HRP goat anti-mouse antibody | Santa Cruz Biotechnology | SC-2005 | |
| HRP goat anti-rabbit antibody | Santa Cruz Biotechnology | SC-2301 | |
| Incubator (30 °C) | Any | NA | |
| Lithium Acetate | Any | NA | |
| Lysogeny Broth (LB) Media | Any | NA | |
| Magnesium Chloride | Any | NA | |
| Microcentrifuge | Eppendorf | 5415 D | |
| Microscope | Nikon | E600 | Nikon Eclipse E600 |
| Microscope Image Analysis Software | Universal Imaging Corporation | 6.3r7 | MetaMorph Software Series 6.3r7 |
| Mouse anti-GFP antibody | Roche | 11814460001 | |
| Nourseothricin | Fisher Scientific | 50997939 | |
| PCR Thermocycler | Applied Biosystems | 9700 | GeneAmp PCR System |
| PCR tubes | BioExpress, GeneMate | T-3035-1 | 0.2 ml |
| Polyethylene Glycol 3350 | Any | NA | |
| Potassium Chloride | Any | NA | |
| Rabbit anti-mCherry antibody | BioVision | 5993-100 | |
| Refrigerator (4 °C) | Any | NA | |
| Sodium Acetate | Any | NA | |
| Stereomicroscope | Nikon | SMZ1500 | |
| Table Top Centrifuge | Labnet | Z 400 | Hermle Z 400 |
| Taq DNA Polymerase | Any | NA | |
| Tris(hydroxymethyl)aminomethane (Tris) | Any | NA | |
| Vortex Mixer | Scientific Industries | SI-0236 | Vortex Genie 2 |
| Yeast Extract Peptone Dextrose (YPD) Media | Any | NA |
Referencias
- Bendel, C. M. Colonization and epithelial adhesion in the pathogenesis of neonatal candidiasis. Semin. Perinatol. 27 (5), 357-364 (2003).
- Gerami-Nejad, M., Berman, J., Gale, C. A. Cassettes for PCR-mediated construction of green, yellow, and cyan fluorescent protein fusions in Candida albicans. Yeast. 18 (9), 859-864 (2001).
- Gerami-Nejad, M., Dulmage, K., Berman, J. Additional cassettes for epitope and fluorescent fusion proteins in Candida albicans. Yeast. 26 (7), 399-406 (2009).
- Gerami-Nejad, M., Forche, A., McClellan, M., Berman, J. Analysis of protein function in clinical C. albicans isolates. Yeast. 29 (8), 303-309 (2012).
- Gerami-Nejad, M., Hausauer, D., McClellan, M., Berman, J., Gale, C. Cassettes for the PCR-mediated construction of regulatable alleles in Candida albicans. Yeast. 21 (5), 429-436 (2004).
- Gonia, S., Larson, B., Gale, C. A. PCR-mediated gene modification strategy for construction of fluorescent protein fusions in Candida parapsilosis. Yeast. 33 (2), 63-69 (2016).
- Milne, S. W., Cheetham, J., Lloyd, D., Aves, S., Bates, S. Cassettes for PCR- mediated gene tagging in Candida albicans utilizing nourseothricin resistance. Yeast. 28 (12), 833-841 (2011).
- Ausubel, F. M., et al. . Current Protocols in Molecular Biology. , (1995).
- Wilson, R. B., Davis, D., Mitchell, A. P. Rapid hypothesis testing with Candida albicans through gene disruption with short homology regions. J. Bacteriol. 181 (6), 1868-1874 (1999).
- Pulver, R., et al. Rsr1 focuses Cdc42 activity at hyphal tips and promotes maintenance of hyphal development in Candida albicans. Eukaryotic Cell. 12 (4), 482-495 (2013).
- Falgier, C., et al. Candida species differ in their interactions with immature human gastrointestinal epithelial cells. Pediatr. Res. 69 (5), 384-389 (2011).
- Nosek, J., et al. Genetic manipulation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 42 (1), 27-35 (2002).
- Zemanova, J., Nosek, J., Tomaska, L. High-efficiency transformation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 45 (3), 183-186 (2004).
- Benjamin, D. K., et al. Neonatal candidiasis among extremely low birth weight infants: risk factors, mortality rates, and neurodevelopmental outcomes at 18 to 22 months. Pediatrics. 117 (1), 84-92 (2006).
- Kullberg, B. J., Arendrup, M. C. Invasive Candidiasis. N Engl J Med. 373 (15), 1445-1456 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados