É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Produção de fusões de proteína fluorescente em
Neste Artigo
Resumo
Modificação do gene mediada por PCR pode ser usado para gerar fusões de proteínas fluorescentes em espécies de Candida, o que facilita a visualização e quantificação das células de levedura e proteínas. Aqui, nós apresentamos uma estratégia para a construção de uma fusão proteína fluorescente (ENO1-FP) em Candida parapsilosis.
Resumo
Espécies de Candida, colonizadores prevalentes das vias intestinais e genito-urinário, são a causa da maioria das infecções fúngicas invasivas em humanos. Assim, ferramentas moleculares e genéticos são necessários para facilitar o estudo de seus mecanismos de patogénese. modificação do gene mediada por PCR é uma abordagem simples e rápida para gerar proteínas marcadas no epitopo para facilitar a sua detecção. Em particular, a proteína fluorescente (FP) fusões são ferramentas poderosas que permitem a visualização e quantificação de ambas as células de levedura e proteínas por microscopia de fluorescência e imunotransferência, respectivamente. Os plasmídeos contendo as sequências de codificação de FP, juntamente com genes marcadores nutricionais que facilitam a transformação de espécies de Cândida, foram geradas com a finalidade de construção e expressão em FP Candida. Aqui, nós apresentamos uma estratégia para a construção de uma fusão FP em uma espécie de Candida. Os plasmídeos contendo o trabalho a prova nourseothricingene transformação marcador NCE (NAT1), juntamente com sequências, quer para verde, amarelo, ou cereja PQ (GFP, YFP, mCherry) são utilizados juntamente com iniciadores que incluem sequências específicas de gene numa reacção em cadeia da polimerase (PCR) para gerar uma cassete de FP . Esta cassete de gene-específico tem a capacidade de se integrar na extremidade 3 'do locus do gene correspondente através de recombinação homóloga. Bem sucedida de fusão em-quadro da sequência de FP no locus do gene de interesse é verificada geneticamente, seguido por análise de expressão de proteína de fusão por microscopia e / ou métodos de detecção de imuno. Além disso, para o caso de proteínas altamente expressas, fusões bem sucedidas pode ser rastreada para principalmente por técnicas de imagem por fluorescência.
Introdução
Espécies de Candida são fungos comensais que colonizam as vias intestinais e genito-urinário de todos os seres humanos. Sob condições de imunodeficiência, tal como o que ocorre com o nascimento prematuro ou efeitos imunossupressores de tratamentos para o cancro, espécies de Cândida pode tornar-se agentes patogénicos oportunistas. Das espécies de Candida, Candida albicans é o colonizador fúngica mais prevalente e faz com que a maioria das infecções fúngicas invasivas. Outras espécies de Candida, como C. glabrata, C. parapsilosis, C. tropicalis e C. kruseii também causar infecções graves em pacientes imunocomprometidos, com algumas apresentando resistência intrínseca aos comumente utilizados antibióticos anti-fúngicas, como fluconazol e anfotericina B. Assim, infecções com algumas destas espécies estão sendo observados com maior frequência, especialmente em pacientes que estão sendo tratados profilaticamente com agentes anti-fúngicos. Mesmo com uma adequada e atempadatratamento nti-fúngica, infecções por Candida invasivos continuem a ser associado com uma morbidade e mortalidade significativas. Devido à importância de espécies de Candida em saúde humana, existe uma necessidade para ferramentas moleculares prontamente disponíveis que permitem o estudo e a elucidação dos seus mecanismos de patogénese.
Uma importante ferramenta que permite que os pesquisadores para visualizar e quantificar células microbianas e as proteínas que eles expressam é a tecnologia de fusão FP. Reacção em cadeia da polimerase (PCR) do gene mediada por modificação, tal como descrito no presente documento, permite a construção de fusões, entre as sequências de FP e uma proteína Candida sequência codificante de interesse no seu locus genómico. A integração estável da construção facilita a análise da expressão da proteína, bem como dinâmica localização de proteínas. Os plasmídeos contendo as sequências de PF, optimizado para a expressão em Cândida albicans e que pode ser utilizado no g mediada por PCRestratégia de modificação eno, foram previamente construídas 2, 3, 4, 5. Os plasmídeos contêm FP transformação de "cassetes": uma sequência de FP ligado a um gene marcador nutricional que facilita a transformação de C. albicans e C. parapsilosis 2, 3, 4, 5, 6, 7. Actualmente plasmídeos disponíveis contêm uma variedade de genes de selecção marcadores nutricionais (URA3, His1, ARG4) para a transformação de estirpes auxotróf iças, bem como um marcador de resistência a droga dominante (NAT1), o que facilita a transformação de estirpes clínicas falta auxotrophies. Além disso, os plasmídeos contêm opções para até quatro sequências FP diferentes (verde [GFP], yelloW [YFP], ciano [PCP], e cereja [mCherry]) e, ou uma sequência de terminação de ADH1 para construção de fusões de proteína carboxi-terminal, ou uma sequência promotora para a construção de proteínas de fusão do terminal amino. Os iniciadores foram concebidos com homologia com o ADN de plasmídeo em torno da cassete de FP. Além disso, os iniciadores contêm também sequências 5'-extensão portadoras de homologia para o gene de levedura de interesse a ser marcado, o que facilita a integração da cassete no locus genómico através de recombinação homóloga (Figura 1). Cassetes de genes específicos de FP são gerados por PCR e, em seguida, transformados em células de Candida tornadas competentes para a absorção de ADN por meio do tratamento com acetato de lítio.

Figura 1: Diagrama de como fusões de sequências de FP são geradas em espécies de Candida. (A) inclu O DNA de plasmídeoes uma sequência FP e uma sequência que codifica nourseothricin resistência (NAT1). localizações relativas da frente (FWD) e inverso (REV) iniciadores são mostrados, com porções pretas dos iniciadores que indicam a região de homologia com a sequência de plasmídeo e as porções roxo que denotam a região de homologia específico para o gene ou a extensão do iniciador. Cassetes (B) FP são transformados em Candida e integrar dentro do locus genómico ENO1 através de recombinação homóloga (linhas pontilhadas). (C) resultante sequência de fusão de FP na extremidade 3 'de ENO1. Por favor clique aqui para ver uma versão maior desta figura.
Aqui, apresentamos um exemplo de proteína de fusão (ENO1-FP) construções em espécies de Candida. Nós utilizam a marcação de plasmídeos contendo o gene NAT1 transformação marcador juntamente com as sequências que codificam GFP, YFP, oumCherry (Figura 2). Estes plasmídeos são usados juntamente com iniciadores de PCR para gerar cassetes de gene-específicos que facilitam a fusão de PQ para a extremidade 3 'de ENO1, resultando em expressão de ENO1 fundido com PQ no seu terminal carboxi.
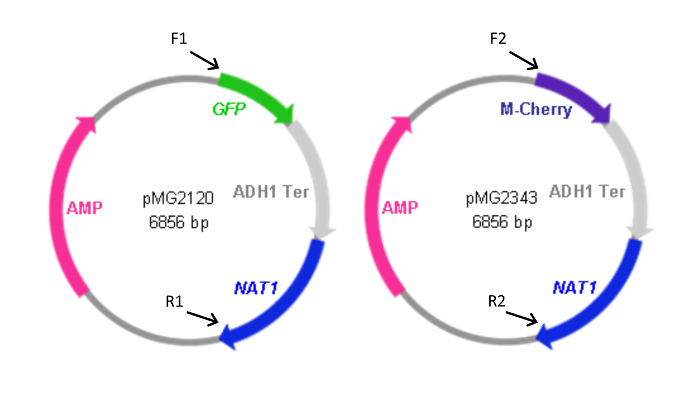
Figura 2: Mapas de FP plasmídeos contendo cassetes. Para a frente (F) e inverso (R) iniciadores utilizados para gerar as cassetes dos plasmídeos estão indicados juntamente com a localização relativa da sua homologia com os plasmídeos. As sequências dos iniciadores são como listados na Tabela 1. F1 e R1 também foram utilizadas para gerar a cassete pYFP- NAT1. O plasmídeo contendo a cassete de YFP- NAT1 (pMG2263) é idêntico ao pMG2120 com excepção da YFP no lugar da sequência de GFP. Formatos de cassete: GFP-NAT1, 3,7 kbp; mCherry- NAT1, 3,2 kpb; YFP- NAT1, 3.7 kpb. Este valor foi modificado a partir Gerami-Nejad, et al. 4 Por favor clique aqui para ver uma versão maior desta figura.
Protocolo
1. Modelo Isolar os plasmídeos a partir de E. coli
- Crescer E. coli contendo o plasmídeo molde durante a noite em 10 ml de caldo de lisogenia (LB) com 200 mg / L de ampicilina (AMP), a 37 ° C com agitação.
- Células colheita por centrifugação a 6000 xg durante 2 min.
- Decantar líquido, isolar e purificar DNA a partir de células de E. coli por um método padrão tal como previamente descrito em Ausubel et al. 8.
- ADN Ressuspender em Tris-EDTA (TE; Tris 10 mM, pH 8,0, EDTA 1 mM, pH 8,0) a uma concentração de trabalho de 50-100 ng / ml.
2. Projeto Primers
| cartilha | sequência iniciadora |
| F1 | 5 ' GAGAATTGAAGAAGAGTTGGGAGACAATGCTATCTATGCTGGTAAGGACTTCCACAATGCTCAAACTTTG GGTGGTTCTAAAGGTGAAGAATTATT 3 ' |
| R1 | 5 'GAGCGTTTGCACCAACAGGCCATCATTTGTGACGAGAGAAGACCTGACGTCATTAGATTGGCACCTTTGC GTAAAACGACGGCCAGTGAATTC 3 ' |
| F2 | 5 'GAGAATTGAAGAAGAGTTGGGAGACAATGCTATCTATGCTGGTAAGGACTTCCACAATGCTCAAACTTTG GGTGGTGTTTCAAAAGGTGAAGAAGATAAT 3 ' |
| R2 | 5 'GAGCGTTTGCACCAACAGGCCATCATTTGTGACGAGAGAAGACCTGACGTCATTAGATTGGCACCTTTGC ACTGGATGGCGGCGTTAGTATC 3 ' |
Tabela 1: As sequências dos iniciadores utilizados neste estudo. Texto em itálico a negrito indica homologia com o locus genómico ENO1, as regiões de fonte normais são homólogas ao plasmídeo de ADN.
- conceber iniciadores para ser homólogo ao plasmídeo sequências limítrofes da cassete a ser amplificado, bem como para a extremidade 3 'do gene alvo de interesse (por exemplo, ENO1) para facilitar a recombinação em local genómico do gene (Figura 2 e Tabela 1) .
- Assegure-se que as sequências de iniciador de sentido directo corresponder aos últimos 70 pares de bases (pb) do gene de interesse, 5'-3 ', menos o codão de paragem, para manter a grelha de codificação, além do primeiro, aproximadamente 30 pb da sequência de plasmídeo a ser amplificado. Como uma nota, o GGTGGTGGTT em cada iniciador é um ligante de poli-glicina com nenhuma homologia de FP. É de notar, na ausência de um agente de ligação, pode haver fusão directa de domínios funcionais, o que pode teoricamente conduzir a enrolamento incorrecto da proteína, baixo rendimento na produção de proteínas, ou bioactividade prejudicada.
- Certifique-se que as sequências de primer reverso são 70 bp a jusante do gene, 3'-5 ', não incluindo qualquer sequência do gene, mais a última approdamente 30 pb do marcador nutricional ou resistência a drogas usadas no plasmídeo.
- Uso F1 e R1 a gerar cassetes GFP NAT1 e YFP- NAT1 com pMG2120 e pMG2263, respectivamente e F2 e R2 para gerar mCherry- NAT1 com pMG2343.
3. Gerar FP Cassetes por PCR (Dia 1)
- Prepare os reagentes para a PCR. Adicione uma mistura principal (500 volume final ul), adicionando os seguintes volumes e concentrações para um tubo de 1,5 ml: 50 ul de tampão de PCR (cloreto de potássio 500 mM, Tris 100 mM, pH 8,0 em água), desoxinucleótidos 20 ul (dNTPs; mistura estoque de nucleótidos em 10 mM de cada), cloreto de magnésio 40 ul de 25 mM, 20 ul do plasmídeo (~ 50-100 ng de solução de estoque / ul purificada), 10 ul de cada iniciador directo e (a partir de soluções de estoque 10 mM reverso), 30 ul de Taq polimerase (genérico, 5.000 unidades / ml), e 320 ul de água.
- Alíquota de 50 ul da mistura principal em cada um dos 10-PCR compatible 0,5 ml de tubos.
- Colocar os tubos de PCR no termociclador e executar os passos seguintes: 1 ciclo de 5 minutos a 94 ° C para desnaturar ADNcd; 40 ciclos de 45 s sequencialmente a 94 ° C, 30 seg a 55 ° C para permitir que os iniciadores para hibridar com o ADN molde de plasmídeo, e 4 min a 68 ° C para extensão de produtos de ADN; e um ciclo de extensão final de 15 min a 72 ° C.
NOTA: A PCR Master Mix e ciclismo parâmetros podem precisar de ser modificado com base na polimerase Taq particular usado. - Piscina todos os produtos das reações 10 de PCR em um tubo de 1,5 ml.
- Sujeito 5 ul de produtos de PCR reunidos para electroforese em gel de agarose para verificar a dimensão do amplicon e obter uma estimativa da concentração do produto, com base em comparação com uma escada de ADN. Geralmente, utilizar ~ 250 ug de ADN em cada cassete de mistura de transformação subsequente.
- Precipitar o DNA por adição de acetato de 50 ul de sódio 3M seguida por 750 ul de etanol a 95% para os produtos e incubar pelo menos 30 mina -20 ° C.
- Colher os produtos de PCR por centrifugação do tubo a 16.000 xg durante 10 min. Remova cuidadosamente e elimine o sobrenadante e secar o sedimento durante a noite. Ressuspender o sedimento cassete de ADN secou-se em 40 ul de TE, pH 8,0 e armazenar a temperatura ambiente até à sua utilização.
4. transformar células de Candida com FP DNA Cassetes
- No Dia 1, recuperar estirpe de levedura a ser transformada a partir de uma glicerol 15% congelado (-80 ° C) da riscando algumas cristais raspados Onto dextrose de levedura peptona de agar com adenina (YPAD) e incubar a 30 ° C. Após a recuperação do crescimento de colónias, inocular uma única colónia em 2 ml de meio YPAD líquido num tubo de cultura de vidro com uma tampa respirável e incubar durante a noite a 30 ° C com agitação.
- No Dia 2, dilui-se a 300 ul de cultura de levedura durante a noite em 50 ml YPAD fresco (para uma DO final de 600 ~ 0,2) num balão de Erlenmeyer de 125 ml com uma tampa respirável. Agitar a 30 ° Cpara ~ 3 horas (a um OD final de 600 ~ 0,6-0,8).
- Verter a cultura durante a noite num tubo cónico de 50 ml e sedimentar as células por centrifugação durante 5 min a 1 500 xg numa centrifugadora de bancada.
- Deitar fora e descartar adequadamente o sobrenadante. Ressuspender o sedimento de células em 5 ml de água. Re-sedimentar as células por centrifugação novamente durante 5 min a 1500 xg numa centrifugadora de bancada.
- Deitar fora e descartar adequadamente o sobrenadante. Ressuspender as células em 500 ul TELiAc (acetato de lítio TE: Tris 10 mM, pH 8,0, EDTA 1 mM, pH 8,0, acetato de lítio 0,1 M) durante a transferência para um tubo de 1,5 ml. Centrifugar o tubo durante 2 min a 3000 x g numa microcentrífuga.
- Ressuspender as células em 250 ul TELiAc. O volume total, incluindo o sedimento deve ser ~ 300 uL.
- Para um (tubo de microcentrífuga diferente do que em 4.2.4) limpo, adicionar 5 uL de ADN transportador (10 mg / ml) e 150 ul de células de Candida preparada (a partir de 4.2.4). Este é o controlo negativo para o transformation.
- Para um segundo tubo de microcentrífuga limpo, adicionam-se 5 ul de ADN de transporte (desnaturado que foi fervida a 90 ° C durante 10 min e arrefeceu-se para 4 ° C), todos os 40 ul do produto de PCR preparado (a partir de 3.3.1) e 150 ul de células de Candida preparado (a partir de 4.2.4).
- Incubam-se as duas misturas de transformação durante 30 min à temperatura ambiente.
- Para cada tubo de mistura de transformação, adicionar 700 ul de mistura placa (Tris 10 mM, pH 8,0, EDTA 1 mM, pH 8,0, acetato de lítio 0,1 M em 50% de polietileno glicol 3350). Inverta os tubos para misturar e incubar durante a noite à RT.
- No Dia 3, incubar a transformação mistura a 42 ° C durante 1 h (choque térmico).
- Centrifugar a transformação mistura durante 30 segundos a 16000 xg numa microcentrífuga. Retirar e descartar adequadamente o sobrenadante. Ressuspender cada um dos sedimentos celulares em 150 ul de água por suavemente pipetando para cima e para baixo de modo a não danificar as células.
- Para transformações UTilizing genes marcadores auxotróf icos (por exemplo, URA3), cada placa totalidade da mistura pipetando as soluções para o meio de agar selectivo apropriado (por exemplo, uridina falta) e espalhar a mistura uniformemente com esferas de vidro estéreis.
- Para transformações utilizando o gene marcador de resistência nourseothricin (NAT1), como descrito aqui, a transformação placa primeira mistura em agar YPAD não selectivo e incuba-se a 30 ° C durante 6-12 h. Este passo ajuda na recuperação das células, choque pós calor, antes nourseothricin estresse é aplicado.
- Após a recuperação parcial de crescimento, placa replica as células de Candida Onto YPAD contendo 400 ug / ml nourseothricin. Para transformações utilizando genes marcadores nutricionais (por exemplo, URA3), este passo intermediário chapeamento não é necessária e as células podem ser directamente plaqueadas em meios selectivos de levedura (por exemplo, falta de uridina YPAD) tal como descrito em 4.4.2.
NOTA: Se a transformação for bem sucedida, colonies deve aparecer dentro de um a três dias (potencialmente até cinco dias de crescimento para a seleção no agar contendo nourseothricin-). Há colônias devem aparecer em placas espalhadas com misturas de transformação contendo DNA transportador sozinho (controle negativo).
- Para a selecção do marcador auxotrófico e nourseothricin, raia transformantes putativos como colónias individuais para placas frescas de agar de meio selectivo e incuba-se a 30 ° C, para propagar as células de levedura que podem ser rastreados para a construção de fusões bem sucedidas FP.
- Transformantes de tela para a integração correta da cassete de marcação (ver resultados representativos para um exemplo detalhado). Se o gene de interesse é expresso em quantidades suficientes, toda colónia de fluorescência pode ocorrer de tal modo que é possível detectar integrantes candidatos potenciais, utilizando um sistema de imagem da placa com capacidade de detecção de fluorescência.
- Verificar integrantes putativos por PCR utilizando iniciadores homólogos às sequências Outside da região de integração para confirmar a fusão do gene alvo.
- Além disso, considerar a análise de transferência de Western para determinar a expressão e o tamanho da proteína de fusão, bem como por microscopia de fluorescência de células individuais para a confirmação visual da localização da proteína, se for conhecida.
Resultados
Como um exemplo, utilizou-se o protocolo descrito acima para GFP e mCherry fusões de ENO1 numa estirpe de laboratório C. parapsilosis. Cada transformante putativo foi inicialmente novamente riscados para o crescimento. Neste exemplo, uma vez que a proteína de fusão resultante é altamente expresso (enolase) e PQ são brilhantes, fomos capazes de rastrear os transformantes por microscopia de fluorescência, antes de realizar PCR de diagnóstico (Figura 3)
Discussão
Construção de sequências de epitopo etiquetado em espécies de Candida, utilizando a estratégia de modificação de genes mediada por PCR acima descrito pode ser resumido como um processo em três etapas. Em primeiro lugar, a cassete é feita por PCR que codifica a sequência desejada, tanto por integração e regiões homólogas para o locus da inserção no genoma da levedura. Em segundo lugar, as células de levedura a ser transformadas são feitas quimicamente competente com acetato de lítio e co-incub...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Agradecemos N. Dean para fornecer a sequência mCherry FP original, M. Gerami-Nejad para a construção de plasmídeos, B. Larson para a assistência técnica, e T. Heisel para o conselho útil durante o desenvolvimento deste projeto. JB foi apoiado pelo Prémio Europeu Conselho de Pesquisa Avançada 340.087 (RAPLODAPT). sistemas de microscopia e de imagem foram fornecidos pela Universidade de Minnesota Pediatria Foundation e da Universidade de Minnesota Imaging Center.
Materiais
| Name | Company | Catalog Number | Comments |
| 100 W mercury lamp | CHIU Technical Corporation | M-100T | |
| 95% Ethanol | Any | NA | |
| Adenine | Any | NA | |
| Ampicillin | Any | NA | |
| Carrier DNA | Ambion | AM9680 | Sheared Salmon Sperm DNA 10 mg/ml |
| CCD Camera | Photometrics | CoolSNAP HQ | |
| Conical Tube | Corning | 430828 | 50 ml |
| Culture Tube Rotator | New Brunswick | 2013923 | TC-8, or Any Culture Tube Rotator |
| Deoxynucleotides (dNTP) PCR Grade | Any | NA | |
| Eppendorf Tubes | Eppendorf | 022363719, 022363212 | 0.5 ml, 1.5 ml |
| Erlenmeyer Flask | Fisher Scientific | 7250089 | 125 ml |
| Ethylenediaminetetraacetic Acid (EDTA) | Any | NA | |
| Freezer (-80 °C) | Thermo Electron Corporation | ULT-1386-9-V | Revco Ultima II |
| GFP, YFP and Texas Red Filter Sets | Chroma Technology Corporation | 49002, 86004v2, 49008 | |
| Glass culture tubes | Fisher Scientific | 1496126 | 75 mm |
| HRP goat anti-mouse antibody | Santa Cruz Biotechnology | SC-2005 | |
| HRP goat anti-rabbit antibody | Santa Cruz Biotechnology | SC-2301 | |
| Incubator (30 °C) | Any | NA | |
| Lithium Acetate | Any | NA | |
| Lysogeny Broth (LB) Media | Any | NA | |
| Magnesium Chloride | Any | NA | |
| Microcentrifuge | Eppendorf | 5415 D | |
| Microscope | Nikon | E600 | Nikon Eclipse E600 |
| Microscope Image Analysis Software | Universal Imaging Corporation | 6.3r7 | MetaMorph Software Series 6.3r7 |
| Mouse anti-GFP antibody | Roche | 11814460001 | |
| Nourseothricin | Fisher Scientific | 50997939 | |
| PCR Thermocycler | Applied Biosystems | 9700 | GeneAmp PCR System |
| PCR tubes | BioExpress, GeneMate | T-3035-1 | 0.2 ml |
| Polyethylene Glycol 3350 | Any | NA | |
| Potassium Chloride | Any | NA | |
| Rabbit anti-mCherry antibody | BioVision | 5993-100 | |
| Refrigerator (4 °C) | Any | NA | |
| Sodium Acetate | Any | NA | |
| Stereomicroscope | Nikon | SMZ1500 | |
| Table Top Centrifuge | Labnet | Z 400 | Hermle Z 400 |
| Taq DNA Polymerase | Any | NA | |
| Tris(hydroxymethyl)aminomethane (Tris) | Any | NA | |
| Vortex Mixer | Scientific Industries | SI-0236 | Vortex Genie 2 |
| Yeast Extract Peptone Dextrose (YPD) Media | Any | NA |
Referências
- Bendel, C. M. Colonization and epithelial adhesion in the pathogenesis of neonatal candidiasis. Semin. Perinatol. 27 (5), 357-364 (2003).
- Gerami-Nejad, M., Berman, J., Gale, C. A. Cassettes for PCR-mediated construction of green, yellow, and cyan fluorescent protein fusions in Candida albicans. Yeast. 18 (9), 859-864 (2001).
- Gerami-Nejad, M., Dulmage, K., Berman, J. Additional cassettes for epitope and fluorescent fusion proteins in Candida albicans. Yeast. 26 (7), 399-406 (2009).
- Gerami-Nejad, M., Forche, A., McClellan, M., Berman, J. Analysis of protein function in clinical C. albicans isolates. Yeast. 29 (8), 303-309 (2012).
- Gerami-Nejad, M., Hausauer, D., McClellan, M., Berman, J., Gale, C. Cassettes for the PCR-mediated construction of regulatable alleles in Candida albicans. Yeast. 21 (5), 429-436 (2004).
- Gonia, S., Larson, B., Gale, C. A. PCR-mediated gene modification strategy for construction of fluorescent protein fusions in Candida parapsilosis. Yeast. 33 (2), 63-69 (2016).
- Milne, S. W., Cheetham, J., Lloyd, D., Aves, S., Bates, S. Cassettes for PCR- mediated gene tagging in Candida albicans utilizing nourseothricin resistance. Yeast. 28 (12), 833-841 (2011).
- Ausubel, F. M., et al. . Current Protocols in Molecular Biology. , (1995).
- Wilson, R. B., Davis, D., Mitchell, A. P. Rapid hypothesis testing with Candida albicans through gene disruption with short homology regions. J. Bacteriol. 181 (6), 1868-1874 (1999).
- Pulver, R., et al. Rsr1 focuses Cdc42 activity at hyphal tips and promotes maintenance of hyphal development in Candida albicans. Eukaryotic Cell. 12 (4), 482-495 (2013).
- Falgier, C., et al. Candida species differ in their interactions with immature human gastrointestinal epithelial cells. Pediatr. Res. 69 (5), 384-389 (2011).
- Nosek, J., et al. Genetic manipulation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 42 (1), 27-35 (2002).
- Zemanova, J., Nosek, J., Tomaska, L. High-efficiency transformation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 45 (3), 183-186 (2004).
- Benjamin, D. K., et al. Neonatal candidiasis among extremely low birth weight infants: risk factors, mortality rates, and neurodevelopmental outcomes at 18 to 22 months. Pediatrics. 117 (1), 84-92 (2006).
- Kullberg, B. J., Arendrup, M. C. Invasive Candidiasis. N Engl J Med. 373 (15), 1445-1456 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados