JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
에 형광 단백질 융합의 시대
요약
PCR 매개 유전자 변형은 시각화 및 효모 세포 및 단백질의 정량을 용이 칸디다 종의 형광 단백질 융합체를 생성하기 위해 사용될 수있다. 여기서, 우리는 칸디다에서 형광 단백질 융합 (Eno1-FP)을 구축하기위한 전략을 제시한다.
초록
칸디다 종, 장 및 비뇨 생식기 책자의 유행 식민지 개척자는, 인간의 침습성 진균 감염의 대부분의 원인이다. 따라서, 분자 유전 도구들은 발병 메커니즘에 대한 연구를 촉진하기 위해 필요하다. PCR 매개 유전자 변형은 검출을 용이하게하기 위해 에피토프 태그 된 단백질을 생성하는 간단하고 빠른 방법이다. 특히, 형광 단백질 (FP) 융합은 각각 형광 현미경 및 면역에 의해 모두 효모 세포와 단백질의 시각화 및 정량 분석을 할 수있는 강력한 도구입니다. 칸디다 종의 형질 전환을 용이하게 영양 마커 유전자와 함께 FP 인코딩 서열을 포함하는 플라스미드, 칸디다 FP의 구축 및 발현을 목적으로 생성되었다. 여기서, 우리는 칸디다 종의 FP 융합을 구성하기위한 전략을 제시한다. nourseothricin resista를 포함하는 플라스미드어느 녹색, 황색, 또는 체리 FPS (GFP, YFP, mCherry)는 폴리머 라제 연쇄 반응 (PCR)의 유전자 - 특이 서열을 포함하는 프라이머와 함께 사용되는 FP 카세트를 생성하기위한 서열과 함께 NCE 변환 마커 유전자 (NAT1) . 이 유전자 특이 카세트를 상동 재조합을 통해 대응 유전자좌의 3'- 말단에 통합 할 수있는 능력을 갖는다. 관심의 유전자 유전자좌에 FP 서열의 성공적인 프레임 내 융합 현미경 및 / 또는 면역 검출 방법에 의한 융합 단백질의 발현을 분석 한 후, 유전자가 확인되어있다. 또한, 고도로 발현 된 단백질의 경우에 성공적인 융합체 형광 이미징 기술에 의해 주로 대해 스크리닝 할 수있다.
서문
칸디다 종은 모든 인간의 장 및 비뇨 생식기 책자를 식민지 공생 균이다. 그러한 즉 조산 또는 암 치료에서 면역 억제 효과 발생과 같은 면역 결핍의 조건에서, 칸디다 종은 기회 병원균이 될 수 있습니다. 칸디다 종, 칸디다 알비 칸스 (Candida albicans)가 가장 널리 진균 식민이며 침습성 진균 감염의 대부분을 야기한다. 이러한 C. 글라 브라, C.의 실로 시스, C.의 tropicalis 및 C와 같은 다른 칸디다 종, 일부 나타내는 고유 저항, 면역 저하 환자에서 심각한 감염을 일으킬 kruseii 일반적 따라서 이러한 플루코나졸과 암포 테리 신 B와 항진균 항생제를 사용하는 이 종의 일부와 감염은 특히 항 곰팡이 제와 예방 치료를 받고있는 환자에서 더 자주 관찰되고있다. 심지어 적절하고 적시에NTI 곰팡이 처리, 침략 칸디다 감염은 상당한 이환율과 사망률 1과 연관 될 것을 계속한다. 이 때문에 인체의 칸디다 종의 중요성, 그 병인 기전 연구 및 해명 있도록 쉽게 구할 분자 도구에 대한 요구가있다.
연구진은 시각화하고 미생물 세포와 그들이 표현하는 단백질을 정량화 할 수 있습니다 하나의 중요한 도구는 FP 융합 기술입니다. 중합 효소 연쇄 반응 (PCR) - 매개 유전자 변형이 논문에 기재된 바와 같이, FP 서열 및 게놈 유전자좌 관심 서열을 코딩 칸디다 단백질 간의 융합의 구성을 허용한다. 구조물의 안정적 통합은 단백질 발현의 분석뿐만 아니라 단백질 현지화 역학을 용이하게한다. FP 서열을 함유하는 플라스미드는, 칸디다 알비 칸스의 발현을 최적화하고는 PCR 매개 g 사용될 수있다엔 변형 전략은 이전에 2, 3, 4, 5를 구성 하였다. C. 알비 칸스 및 C. 실로 시스 2, 3, 4, 5, 6, 7의 변환을 용이하게하는 영양 마커 유전자에 연결된 FP 서열 플라스미드 FP 변환 "카세트"을 포함한다. 현재 사용 가능한 플라스미드를 선택 영양 영양 요 구성 균주의 변환을위한 마커 유전자 (URA3, HIS1, ARG4)뿐만 아니라 auxotrophies이 결여 된 임상 균주의 변환을 용이하게하는 지배적 인 약물 내성 마커 (NAT1)의 다양한 포함되어 있습니다. 또한, 플라스미드 여보 쇼 최대 네 개의 다른 FP 시퀀스 (녹색 [GFP]에 대한 옵션을 포함[YFP] w, 시안 [CFP], 체리 [mCherry]) 및 카르복시 말단 단백질 융합의 건설, 또는 아미노 말단 단백질 융합의 건설을위한 프로모터 서열에 대한 ADH1 종결 서열 중 하나. 프라이머는 FP 카세트를 둘러싼 플라스미드 DNA에 동성으로 설계되었습니다. 또한, 프라이머는 상동 재조합 (그림 1)를 통해 게놈 궤적에 카세트의 통합을 용이하게 태그 할 관심있는 효모 유전자에 상 동성을 베어링 5'-확장 시퀀스가 포함되어 있습니다. 유전자 특이 FP 카세트를 PCR에 의해 생성 된 리튬 아세테이트로 처리하여 DNA의 흡수를위한 제작 능력 칸디다 세포로 형질 전환된다.

그림 1 : FP 시퀀스 융합은 칸디다 종에서 생성하는 방법의 다이어그램. (A) 플라스미드 DNA를 포함하여에스 FP 순서 및 저항 (NAT1) nourseothricin 시퀀스 인코딩. 앞으로 (FWD) 및 역방향 (REV) 프라이머의 상대 위치는 플라스미드 서열 상 동성의 영역을 나타내는 프라이머 및 유전자 고유 동성 영역 또는 프라이머 확장을 나타내는 보라색 부분의 검은 부분으로 표시됩니다. (B) FP 카세트는 칸디다로 변환 및 상동 재조합 (점선)를 통해 ENO1 게놈 궤적 내에 통합되어있다. ENO1의 말단에 FP 융합 시퀀스를 생성 (C). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
여기서, 우리는 칸디다 단백질 융합 (Eno1-FP) 구조의 일례를 제시한다. 우리는 GFP, YFP를 코딩하는 서열과 함께 NAT1 변환 마커 유전자를 포함하는 플라스미드에 태그 또는 사용mCherry (그림 2). 이러한 플라스미드의 카르복시 말단에 융합 FPS Eno1 발현 결과 ENO1의 3'- 말단에 FPS의 융합을 촉진 유전자 특이 카세트를 생성하기 위해 PCR에서 프라이머와 함께 사용된다.
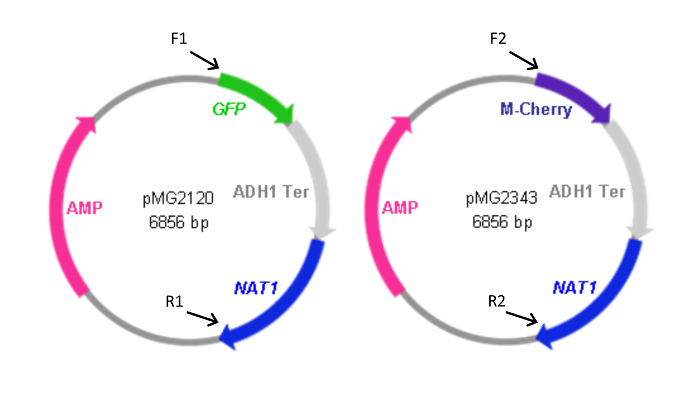
그림 2 : FP 카세트 함유 플라스미드의지도. 앞으로 (F) 및 플라스미드에서 카세트를 생성하는 데 사용 역 (R) 프라이머는 플라스미드에 자신의 동성의 상대 위치와 함께 표시됩니다. 표 1에 열거 된 프라이머 서열이다. F1과 R1은 또한 pYFP- NAT1 카세트를 생성 하였다. YFP- NAT1 카세트 (pMG2263)를 함유하는 플라스미드 GFP 서열 대신 YFP 제외한 pMG2120 동일하다. 카세트 크기 : GFP-NAT1, 3.7 KBP; mCherry- NAT1, 3.2 KBP; YFP- NAT1, 3.7 KBP. 이 그림은 Gerami-Nejad, 등에서 수정되었습니다. 4 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Access restricted. Please log in or start a trial to view this content.
프로토콜
대장균 1. 격리 템플릿 플라스미드
- 진탕 37 ° C에서 10 ml의 원성 국물 (LB) + 200 ㎎ / ℓ의 암피실린 (AMP)에서 하룻밤 템플릿 플라스미드를 포함하는 대장균 성장.
- 2 분 동안 6,000 XG에 원심 분리하여 수확 세포.
- 액체를 가만히 따르다 분리 및 문헌 [Ausubel 등 이전에 설명 된대로 E. 표준 방법에 의해 세포를 대장균에서 DNA를 정화. 8.
- 50 ~ 100 ng를 / ㎖의 작업 농도, 트리스 - EDTA (10 mM 트리스, 산도 8.0, 1 mM의 EDTA, pH가 8.0 TE)에 재현 탁의 DNA.
2. 디자인 프라이머
| 뇌관 | 프라이머 시퀀스 |
| F1 | 5 ' GAGAATTGAAGAAGAGTTGGGAGACAATGCTATCTATGCTGGTAAGGACTTCCACAATGCTCAAACTTTG GGTGGTTCTAAAGGTGAAGAATTATT 3 ' |
| R1 | 5 'GAGCGTTTGCACCAACAGGCCATCATTTGTGACGAGAGAAGACCTGACGTCATTAGATTGGCACCTTTGC GTAAAACGACGGCCAGTGAATTC 3 ' |
| F2 | 5 'GAGAATTGAAGAAGAGTTGGGAGACAATGCTATCTATGCTGGTAAGGACTTCCACAATGCTCAAACTTTG GGTGGTGTTTCAAAAGGTGAAGAAGATAAT 3 ' |
| R2 | 5 'GAGCGTTTGCACCAACAGGCCATCATTTGTGACGAGAGAAGACCTGACGTCATTAGATTGGCACCTTTGC ACTGGATGGCGGCGTTAGTATC 3 ' |
표 1 : 본 연구에 사용 된 프라이머 서열. 굵은 이탤릭체 텍스트가 ENO1 게놈 유전자좌에 상 동성을 나타내고, 정상 폰트 영역 DNA 플라스미드 상동이다.
- <리> 설계 프라이머 카세트 경계 서열이 유전자의 게놈 유전자좌로 재결합을 용이하게하기 위해 관심있는 표적 유전자 (예 ENO1)의 3'- 말단뿐만 아니라 증폭하는 플라스미드 상동 될 (도 2 및 표 1) .
- 정방향 프라이머 서열은 지난 70 염기쌍 관심 유전자 (BP), 5'-3 '을 뺀 종결 코돈과 일치하는 부호화 프레임을 유지하기 위해, 플러스 플라스미드 서열의 처음 약 30 염기쌍이되고 있는지 확인 증폭. 참고로, 각 프라이머의 GGTGGTGGTT없이 FP 상 동성을 가진 폴리 글리신 링커입니다. 참고로, 링커가없는 경우에, 이론적으로 단백질 미스 폴딩 단백질 생산 수율이 낮거나 손상된 생체 활성을 초래할 수 기능적 도메인을 직접 융합있을 수있다.
- 역방향 프라이머 서열이 유전자의 70 bp의 하류 있는지 확인 3'- 5 ', 어떤 유전자 서열, 플러스 마지막 아프로을 포함하지 않는ximately 플라스미드에서 사용되는 영양 또는 약물 내성 마커의 30 염기쌍.
- 사용 F1과 R1은 pMG2343와 mCherry- NAT1를 생성하는 pMG2120 및 pMG2263, 각각 및 F2와 R2와 GFP- NAT1 및 YFP- NAT1 카세트를 생성합니다.
3. PCR에 의해 FP 카세트를 생성 (1 일)
- PCR 용 시약을 준비합니다. 1.5 ㎖의 튜브에 다음의 양과 농도를 첨가하여 마스터 믹스 (500 ㎕의 최종 부피) 확인 : 50 ㎕의 PCR 완충액 (500 mM의 염화칼륨, 물 100 mM 트리스 pH를 8.0) 20 μL의 데 옥시 뉴클레오티드 (dNTPs를 단계; 스톡 혼합물 10 mM의 각), 40 ㎕의 25 mM의 염화 마그네슘에서 뉴클레오티드의 20 μL는) 10 μl를 앞으로 각각 10 mM의 스톡 솔루션에서 프라이머 (역방향), 30 ㎕의 DNA 형성 촉매를 ~ 50 ~ 100 NG / μL 원액에서 플라스미드 (정제 중합 효소 (일반, 5,000 단위 / ㎖) 320 ㎕를 물.
- 10 PCR-compatib 각각에 마스터 믹스의 분취 액 50 μL제작 : 0.5 ml의 튜브.
- 다음 단계를 열 순환기에 PCR 튜브를 놓고 실행 : 1주기 분 (5)의 94 ° C에서 dsDNA를 변성; 94 ° C에서 45 초 40주기를 순차적으로, 55 ° C에서 30 초 프라이머 플라스미드 템플릿 DNA에 어닐링 할 수 있도록하고, DNA 제품의 확장을위한 68 ° C에서 4 분하는 단계; 72 ° C에서 15 분의 1 최종 확장 사이클.
참고 : PCR 마스터 믹스와 자전거 매개 변수가 사용되는 특정 Taq 중합 효소에 따라 수정해야 할 수도 있습니다. - 풀 1.5 ML 튜브에서 10 PCR 반응에서 모든 제품.
- 아가 로스 겔 전기 풀링 된 PCR 산물의 제목 5 μL은 DNA 사다리 비교에 기초하여, 앰플 리콘의 크기를 확인하고 제품 농도의 추정치를 얻었다. 일반적으로, 각각의 후속 변환 믹스에 카세트 DNA의 ~ 250 μg의를 사용합니다.
- 제품을 750 ㎕의 95 % 에탄올에 이어 50 ㎕의 3 M 아세트산 나트륨을 첨가하여 DNA를 침전 적어도 30 분 부화-20 ℃에서
- 10 분 동안 16,000 XG에서 튜브를 원심 분리하여 PCR 산물 수확. 조심스럽게 제거하고 상층 액을 버리고 밤새 펠렛을 건조. 40 ㎕의 TE의 pH 8.0에서 건조 DNA 카세트 펠렛을 재현 탁하고 사용할 때까지 실온에서 저장합니다.
4. FP DNA 카세트와 칸디다 셀 변환
- 1 일에, 효모 균주를 복구하는 아데닌 (YPAD) 한천과 효모 펩톤 덱 스트로스 상에 약간의 긁힌 결정을 질주하고 30 ° C에서 부화하여 (-80 ° C) 주식 냉동 15 % 글리세롤에서 변환 할 수 있습니다. 콜로니의 성장을 회수 한 후, 통기성 캡 유리 배양 관에 2 ㎖의 액체 YPAD 매체에 단일 콜로니를 접종하고 교반하면서 30 ℃에서 밤새 배양한다.
- 제 2 일에, 통기성 캡 125 ㎖의 삼각 플라스크에 50 ㎖에 (최종 OD 600 ~ 0.2) 신선한 YPAD 밤새 효모 배양 300 μl를 희석. 30 ° C에서 흔들어(최종 OD ~ 0.6-0.8 600) ~ 3 시간 동안.
- 50 ML 원뿔 튜브에 하룻밤 문화를 붓고 테이블 위에 원심 분리기 1 500 XG에서 5 분 동안 회전시켜 세포 펠렛.
- 붓고 제대로 상층 액을 버린다. 5 ㎖의 물에서 세포 펠렛을 재현 탁. 테이블 탑 원심 분리기에서 1,500 XG에서 5 분간 다시 원심 분리하여 세포를 다시하는 것은-펠렛.
- 붓고 제대로 상층 액을 버린다. 1.5 ㎖의 튜브에 전송하는 동안 500 μL TELiAc (10 mM 트리스, pH가 8.0, 1 mM의 EDTA, pH가 8.0, 0.1 M 아세트산 리튬 TE 리튬 아세테이트)에 세포를 재현 탁. 미세 원심에서 3,000 XG에서 2 분 동안 튜브를 원심 분리기.
- 250 μL TELiAc에 재현 탁 세포. 펠렛을 포함하여 총 부피는 ~ 300 ㎕를해야한다.
- 클린 (4.2.4에서 다른 미세 원심 분리 튜브)에 5 μL 캐리어 DNA (10 ㎎ / ㎖)과 (4.2.4)에서 제조 된 칸디다 세포 150 μl를 추가합니다. 이 t에 대한 부정적인 컨트롤입니다ransformation.
- 제 깨끗한 미세 원심 분리 관에 변성 캐리어 DNA의 5 μL (즉, 10 분간 90 ℃에서 비등하고 4 ℃로 냉각되었다) (3.3.1)에서 준비된 PCR 산물의 모든 40 μL, 150을 추가 (4.2.4)에서 제조 칸디다 셀 μL.
- 실온에서 30 분 동안 두 변환 믹스 부화.
- 각 변환 믹스 튜브에 700 μL PLATE 믹스 (10 mM 트리스, pH가 8.0, 1 mM의 EDTA, pH가 8.0, 50 % 폴리에틸렌 글리콜 3350 0.1 M 리튬 아세테이트)를 추가한다. 혼합 RT에서 하룻밤을 배양하기 위해 튜브를 반전.
- 3 일에, 변환 1 시간 동안 42 ° C (열 충격)에서 혼합 배양한다.
- 원심 분리기 변환은 마이크로 원심에서 16,000 XG에 30 초 동안 혼합합니다. 제거하고 제대로 상층 액을 버린다. 세포를 손상시키지 않도록하기 위해서 아래로 부드럽게 피펫 팅하여 150 ㎕의 물에서 세포 펠렛을 각각 재현 탁하고.
- 변환의 유타에 대한영양 요 구성 마커 유전자 (예를 들어 URA3)을 ilizing 적절한 선택 배지에 한천 용액을 피펫 팅 (예를 들어 결여 딘)에 의해 각 전 혼합 접시에 균일하게 멸균 유리 비드를 사용하여 혼합물을 분산.
- 여기에 설명 된 바와 같이 nourseothricin 저항 마커 유전자 (NAT1)를 이용한 변환의 경우, 변환 판은 비 선택적 YPAD 한천 상에 제 혼합물 및 6-12 시간 동안 30 ℃에서 배양한다. 스트레스를 nourseothricin 전에 세포 복구, 후 열 충격이 단계 에이즈가 적용된다.
- 부분적인 성장 복구 후 YPAD 상에 복제 판 칸디다 세포는 400 μg의가 / ㎖ nourseothricin 포함. 영양 마커 유전자 (예를 들어 URA3)를 이용한 변환의 경우,이 중간 도금 단계가 필요하지 않은 세포를 직접적 4.4.2에 기술 된 바와 같이 선택적 효모 매체 (예 YPAD 결여 딘) 상에 도금 될 수있다.
주 : 변환이 성공적이면, COLonies은 1 ~ 3 일 내에 표시 (잠재적 nourseothricin 함유 한천에 선택에 대한 파생물 5 일까지)한다. 어떤 식민지 단독 (대조군)을 캐리어 DNA를 포함하는 변환 믹스와 확산 판에 표시되지 않아야합니다.
- 영양 요 구성 및 nourseothricin 마커 선택, 신선한 선택적 매체 한천 플레이트에 하나의 식민지로 행진 추정 형질 전환 및 들어 FP 융합의 성공적인 건설을위한 검사를 할 수 있습니다 효모 세포를 전파하기 위해 30 ° C에서 품어.
- 화면 형질 전환 체는 태그 카세트의 올바른 통합 (자세한 예를 들어 대표 결과를 참조). 관심의 유전자를 충분한 양으로 발현되는 경우, 콜로니 전체의 형광은 형광 검출 능력 판 촬상 시스템을 이용하여 잠재적 인 후보 통합 체를 검출 할 수 있도록 발생할 수있다.
- PCR은 outsid 시퀀스 프라이머 동성을 사용하여 추정 통합 체를 확인통합의 영역의 전자는 표적 유전자에 융합을 확인한다.
- 또한, 웨스턴 융합 단백질의 발현 및 크기를 결정하기 위해 오점 분석뿐만 아니라 알려진 경우 단백질 지역화 시각적 확인을 위해 단 전지의 형광 현미경을 고려한다.
Access restricted. Please log in or start a trial to view this content.
결과
예를 들어, 우리는 C. 실로 시스 실험실 균주에 Eno1하는 GFP 및 mCherry 융합을 구성하기 위해 위에서 설명한 프로토콜을 사용했다. 각각의 추정 형질 전환 초기 성장 restreaked했다. 생성 된 융합 단백질이 높은 (에 놀라 제)를 표현하고, FPS 밝은 때문에이 예에서, 우리는 (도 3) 종래의 진단 PCR 수행을 형광 현미경에 의해 형질 전환 체를 선별 할 수 있었다
Access restricted. Please log in or start a trial to view this content.
토론
에피토프의 구조는 상술 한 PCR 매개 유전자 변형 전략을 사용하는 칸디다 종의 서열은 세 단계로 요약 될 수있다 태그. 먼저, 카세트 모두 효모 게놈 내로 삽입 궤적 상동 통합 영역에 대해 원하는 서열을 코딩하는 PCR에 의해 이루어진다. 둘째로, 변환 될 수있는 효모 세포 리튬 아세테이트 카세트가 공동 인큐베이션 화학적 능력 만들어진다. 셋째, 세포 형질 전환 체를 복구하는 선택 배지 ?...
Access restricted. Please log in or start a trial to view this content.
공개
저자는 공개 아무것도 없어.
감사의 말
우리는 원래 mCherry FP 순서를 제공 N. 딘 감사, M. Gerami-Nejad 플라스미드의 건설이 프로젝트의 개발 기간 동안 도움이되는 조언을 기술 지원 B. 라슨, 그리고 T. Heisel. JB는 유럽 연구위원회 고급 상 340087 (RAPLODAPT)에 의해 지원되었다. 현미경 및 이미징 시스템은 미네소타 소아 과학 재단의 대학과 미네소타 이미징 센터의 대학에 의해 제공되었다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| 100 W mercury lamp | CHIU Technical Corporation | M-100T | |
| 95% Ethanol | Any | NA | |
| Adenine | Any | NA | |
| Ampicillin | Any | NA | |
| Carrier DNA | Ambion | AM9680 | Sheared Salmon Sperm DNA 10 mg/ml |
| CCD Camera | Photometrics | CoolSNAP HQ | |
| Conical Tube | Corning | 430828 | 50 ml |
| Culture Tube Rotator | New Brunswick | 2013923 | TC-8, or Any Culture Tube Rotator |
| Deoxynucleotides (dNTP) PCR Grade | Any | NA | |
| Eppendorf Tubes | Eppendorf | 022363719, 022363212 | 0.5 ml, 1.5 ml |
| Erlenmeyer Flask | Fisher Scientific | 7250089 | 125 ml |
| Ethylenediaminetetraacetic Acid (EDTA) | Any | NA | |
| Freezer (-80 °C) | Thermo Electron Corporation | ULT-1386-9-V | Revco Ultima II |
| GFP, YFP and Texas Red Filter Sets | Chroma Technology Corporation | 49002, 86004v2, 49008 | |
| Glass culture tubes | Fisher Scientific | 1496126 | 75 mm |
| HRP goat anti-mouse antibody | Santa Cruz Biotechnology | SC-2005 | |
| HRP goat anti-rabbit antibody | Santa Cruz Biotechnology | SC-2301 | |
| Incubator (30 °C) | Any | NA | |
| Lithium Acetate | Any | NA | |
| Lysogeny Broth (LB) Media | Any | NA | |
| Magnesium Chloride | Any | NA | |
| Microcentrifuge | Eppendorf | 5415 D | |
| Microscope | Nikon | E600 | Nikon Eclipse E600 |
| Microscope Image Analysis Software | Universal Imaging Corporation | 6.3r7 | MetaMorph Software Series 6.3r7 |
| Mouse anti-GFP antibody | Roche | 11814460001 | |
| Nourseothricin | Fisher Scientific | 50997939 | |
| PCR Thermocycler | Applied Biosystems | 9700 | GeneAmp PCR System |
| PCR tubes | BioExpress, GeneMate | T-3035-1 | 0.2 ml |
| Polyethylene Glycol 3350 | Any | NA | |
| Potassium Chloride | Any | NA | |
| Rabbit anti-mCherry antibody | BioVision | 5993-100 | |
| Refrigerator (4 °C) | Any | NA | |
| Sodium Acetate | Any | NA | |
| Stereomicroscope | Nikon | SMZ1500 | |
| Table Top Centrifuge | Labnet | Z 400 | Hermle Z 400 |
| Taq DNA Polymerase | Any | NA | |
| Tris(hydroxymethyl)aminomethane (Tris) | Any | NA | |
| Vortex Mixer | Scientific Industries | SI-0236 | Vortex Genie 2 |
| Yeast Extract Peptone Dextrose (YPD) Media | Any | NA |
참고문헌
- Bendel, C. M. Colonization and epithelial adhesion in the pathogenesis of neonatal candidiasis. Semin. Perinatol. 27 (5), 357-364 (2003).
- Gerami-Nejad, M., Berman, J., Gale, C. A. Cassettes for PCR-mediated construction of green, yellow, and cyan fluorescent protein fusions in Candida albicans. Yeast. 18 (9), 859-864 (2001).
- Gerami-Nejad, M., Dulmage, K., Berman, J. Additional cassettes for epitope and fluorescent fusion proteins in Candida albicans. Yeast. 26 (7), 399-406 (2009).
- Gerami-Nejad, M., Forche, A., McClellan, M., Berman, J. Analysis of protein function in clinical C. albicans isolates. Yeast. 29 (8), 303-309 (2012).
- Gerami-Nejad, M., Hausauer, D., McClellan, M., Berman, J., Gale, C. Cassettes for the PCR-mediated construction of regulatable alleles in Candida albicans. Yeast. 21 (5), 429-436 (2004).
- Gonia, S., Larson, B., Gale, C. A. PCR-mediated gene modification strategy for construction of fluorescent protein fusions in Candida parapsilosis. Yeast. 33 (2), 63-69 (2016).
- Milne, S. W., Cheetham, J., Lloyd, D., Aves, S., Bates, S. Cassettes for PCR- mediated gene tagging in Candida albicans utilizing nourseothricin resistance. Yeast. 28 (12), 833-841 (2011).
- Ausubel, F. M., et al. Current Protocols in Molecular Biology. , Wiley. New York. (1995).
- Wilson, R. B., Davis, D., Mitchell, A. P. Rapid hypothesis testing with Candida albicans through gene disruption with short homology regions. J. Bacteriol. 181 (6), 1868-1874 (1999).
- Pulver, R., et al. Rsr1 focuses Cdc42 activity at hyphal tips and promotes maintenance of hyphal development in Candida albicans. Eukaryotic Cell. 12 (4), 482-495 (2013).
- Falgier, C., et al. Candida species differ in their interactions with immature human gastrointestinal epithelial cells. Pediatr. Res. 69 (5), 384-389 (2011).
- Nosek, J., et al. Genetic manipulation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 42 (1), 27-35 (2002).
- Zemanova, J., Nosek, J., Tomaska, L. High-efficiency transformation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 45 (3), 183-186 (2004).
- Benjamin, D. K., et al. Neonatal candidiasis among extremely low birth weight infants: risk factors, mortality rates, and neurodevelopmental outcomes at 18 to 22 months. Pediatrics. 117 (1), 84-92 (2006).
- Kullberg, B. J., Arendrup, M. C. Invasive Candidiasis. N Engl J Med. 373 (15), 1445-1456 (2015).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유