このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
蛍光タンパク質融合物中の生成
要約
PCRによる遺伝子改変は、酵母細胞およびタンパク質の可視化および定量化を容易にする、 カンジダ種で蛍光タンパク質融合体を生成するために使用することができます。ここで、我々は、 カンジダ・パラプシロシスに蛍光タンパク質融合(ENO1-FP)を構築するための方策を提示します。
要約
カンジダ種、腸管および尿生殖路の流行入植は、ヒトにおける侵襲性真菌感染症の大部分の原因となっています。このように、分子および遺伝子ツールは、それらの病因メカニズムの研究を促進するために必要とされています。 PCRによる遺伝子改変は、それらの検出を容易にするために、エピトープタグ融合タンパク質を生成する簡単かつ迅速なアプローチです。具体的には、蛍光タンパク質(FP)の融合体は、それぞれ、蛍光顕微鏡および免疫ブロッティングにより酵母細胞およびタンパク質の両方の可視化および定量化を可能にする強力なツールです。 FPをコードする配列を含むプラスミドは、 カンジダ種の形質転換を促進する栄養マーカー遺伝子と共に、 カンジダにおけるFPの構造および発現のために生成されています。ここで、我々は、 カンジダ種にFP融合を構築するための方策を提示します。ヌーセオスリシンresistaを含むプラスミドNCEの形質転換マーカー遺伝子(NAT1)、緑、黄、またはチェリーのFP(GFP、YFP、mCherryを)のいずれかのための配列は、FPカセットを生成するために、ポリメラーゼ連鎖反応(PCR)で、遺伝子特異的な配列を含むプライマーと一緒に使用されるとともに。この遺伝子特異的カセットは、相同組換えを介して、対応する遺伝子座の3 '末端に統合する能力を有しています。関心対象の遺伝子座にFPシーケンスが正常にインフレーム融合は、顕微鏡および/または免疫検出法により、融合タンパク質発現の分析に続いて、遺伝的に検証されます。また、高度に発現されたタンパク質の場合について、成功した融合体は、主に蛍光イメージング技術によってスクリーニングすることができます。
概要
カンジダ種は、すべての人間の腸や尿生殖路を植民地化共生菌で。その結果として免疫不全の条件、早産または癌の治療の免疫抑制作用で発生する下では、 カンジダ種は、日和見病原体になることができます。 カンジダ種のうち、 カンジダ・アルビカンスは、最も一般的な真菌の植民あり、侵襲性真菌感染症の大部分の原因となります。このようなC.グラブラタ 、C。パラプシローシス 、C。トロピカリス 、Cのような他のカンジダ種は、また、いくつかの展示固有抵抗で、免疫不全患者で重篤な感染症を引き起こすkruseii一般的にしたがって、このようなフルコナゾールやアムホテリシンBのような抗真菌性抗生物質を使用しますこれらの種のいくつかの感染は、特に抗真菌剤で予防的に治療を受けている患者では、より頻繁に観察されています。でも適切かつタイムリーなAとNTI-真菌治療は、侵襲性カンジダ感染症はかなりの罹患率と死亡率1と関連することを続けています。しかし、人の健康におけるカンジダ種の意義、その病因機構の研究と解明を可能にする、容易に入手可能な分子ツールの必要性があります。
研究者が視覚化し、微生物細胞とそれらが発現するタンパク質を定量化することを可能にする一つの重要なツールは、FP融合技術です。ポリメラーゼ連鎖反応(PCR)媒介遺伝子改変は、この論文に記載のように、FP配列、そのゲノム遺伝子座に目的の配列をコードカンジダタンパク質との間の融合物の構築を可能にします。構築物の安定な組込みは、タンパク質発現の解析だけでなく、タンパク質の局在のダイナミクスを容易にします。 FP配列を含むプラスミドは、 カンジダ・アルビカンスにおける発現のために最適化され、それは、PCR媒介Gで使用することができエン修飾戦略は、以前に2、3、4、5が構築されています。 C.アルビカンス及びC.パラプシローシス 2、3、4、5、6、7の変換を促進する栄養マーカー遺伝子に連結されたFPの配列:プラスミドはFP変換「カセット」を含みます。現在利用可能なプラスミドは選択可能な栄養栄養要求株の形質転換のためのマーカー遺伝子(URA3、HIS1、ARG4)だけでなく、栄養要求性を欠く臨床株の形質転換を容易に支配的な薬剤耐性マーカー(NAT1)、さまざまな含まれています。また、プラスミドは、最大4つの異なるFPシーケンス(緑[GFP]、YELLOのオプションが含まれています【YFP]ワット、シアン[CFP]、および桜[mCherryを])およびカルボキシ末端タンパク質融合物の建設、またはアミノ末端タンパク質融合体の構築のためのプロモーター配列のためのADH1終結配列のいずれか。プライマーは、FPカセットを囲むプラスミドDNAの相同性を有するように設計されています。また、プライマーはまた、相同組換え( 図1)を介してゲノム座にカセットの組込みを容易にタグ付けされるべき関心の酵母遺伝子に相同性をもつ5'-延長配列を含みます。遺伝子特異的FPカセットは、PCRによって生成され、酢酸リチウムで処理することによりDNAの取り込みのためにコンピテントカンジダ細胞に形質転換されます。

図1:FPシーケンス融合がカンジダ種にどのように生成されるかの図。 (A)プラスミドDNAはこちらよりエスFP配列および抵抗ヌーセオスリシンをコードする配列(NAT1)。フォワード(FWD)およびリバース(REV)プライマーの相対的位置は、プラスミド配列と相同性の領域を示すプライマーおよび遺伝子特異的相同領域またはプライマー伸長を示す紫色の部分の黒い部分で示されています。 (B)FPカセットは、 カンジダに形質転換し、相同組換え(点線)を介してENO1ゲノム遺伝子座の中に統合されています。 ENO1の3 '末端にFP融合配列を得られた(C)。 この図の拡大版をご覧になるにはこちらをクリックしてください。
ここで、我々は、 カンジダ種におけるタンパク質融合(ENO1-FP)の構造の例を提示します。私たちは、GFP、YFPをコードする配列とともにNAT1の形質転換マーカー遺伝子を含むタグ付けするプラスミドを使用しますか、mCherryを( 図2)。これらのプラスミドは、そのカルボキシ末端でのFPに融合ENO1の発現をもたらす、ENO1の3 '末端へのFPの融合を促進する遺伝子特異的カセットを生成するために、PCRにおけるプライマーと一緒に使用されています。
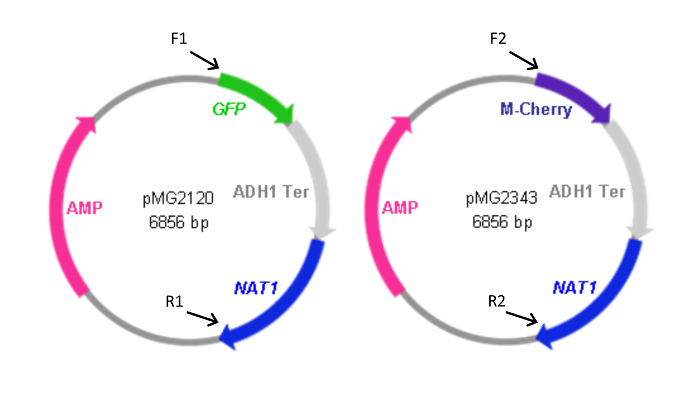
図2:FPカセットを含むプラスミドのマップ。フォワード(F)および逆方向(R)プラスミドからカセットを生成するために使用されるプライマーは、プラスミドに対する相同性の相対的な位置と共に示されています。 表1に記載されたプライマー配列です。 F1およびR1もpYFP- NAT1カセットを生成するために使用しました。 YFP- NAT1カセット (pMG2263)を含有するプラスミドは、GFP配列の代わりにYFPを除いてpMG2120と同じです。カセットサイズ:GFP-NAT1、3.7 kbpの。 mCherry- NAT1、3.2 kbpの。 YFP- NAT1、3。7 kbpの。この図は、Gerami-Nejad、 らから変更されています。 4 この図の拡大版をご覧になるにはこちらをクリックしてください。
Access restricted. Please log in or start a trial to view this content.
プロトコル
大腸菌から1アイソテンプレートプラスミド
- 振盪しながら37℃で10 mlのLB培地(LB)+ 200 mg / Lのアンピシリン(AMP)で一晩鋳型プラスミドを含む大腸菌を成長させます。
- 2分間6000×gで遠心分離によって細胞を回収します。
- 液体をデカントし、のAusubelに前述したようにらの標準的な方法により、 大腸菌細胞からDNAを単離および精製します。 8。
- トリスEDTA中で再懸濁したDNA(TE、10mMトリス、pH8.0の、1mMのEDTA、pH8.0)で50〜100 ng / mlでの作業濃度で。
2.デザインプライマー
| プライマー | プライマー配列 |
| F1 | 5 ' GAGAATTGAAGAAGAGTTGGGAGACAATGCTATCTATGCTGGTAAGGACTTCCACAATGCTCAAACTTTG GGTGGTTCTAAAGGTGAAGAATTATT 3 ' |
| R1 | 5 'GAGCGTTTGCACCAACAGGCCATCATTTGTGACGAGAGAAGACCTGACGTCATTAGATTGGCACCTTTGC GTAAAACGACGGCCAGTGAATTC 3 ' |
| F2 | 5 'GAGAATTGAAGAAGAGTTGGGAGACAATGCTATCTATGCTGGTAAGGACTTCCACAATGCTCAAACTTTG GGTGGTGTTTCAAAAGGTGAAGAAGATAAT 3 ' |
| R2 | 5 'GAGCGTTTGCACCAACAGGCCATCATTTGTGACGAGAGAAGACCTGACGTCATTAGATTGGCACCTTTGC ACTGGATGGCGGCGTTAGTATC 3 ' |
表1:本研究で用いたプライマー配列。太字イタリック体のテキストは、通常のフォント領域はプラスミドDNAに相同である、ENO1ゲノム遺伝子座と相同性を示しています。
- <李>デザインプライマーは、遺伝子のゲノム遺伝子座への組換えを容易にするために、目的の標的遺伝子の3 '末端( 例えば ENO1)に同様に増幅されるカセットに隣接する配列をプラスミドに相同であることにする( 図2、 表1) 。
- なるように符号化フレームに加え、プラスミド配列の最初の約30塩基対を維持するために、フォワードプライマーの配列は、最後の目的の遺伝子の70塩基対(bp)、5'-3 '、マイナス停止コドンと一致していることを確認増幅されました。注意点としては、各プライマーでGGTGGTGGTTなしFP相同性を有するポリグリシンリンカーです。注目すべきは、リンカーの非存在下では、理論的には、タンパク質ミスフォールディング、タンパク質産生の低い収率、又は障害の生物活性をもたらすことができ、機能的ドメインの直接的な融合が存在することができます。
- リバースプライマー配列は、遺伝子の70 bpのすぐ下流にあることを確認してください3 '、5'、任意の遺伝子配列に加え、最後のアプロを含みませんximatelyプラスミドで使用される栄養または薬剤耐性マーカーの30bpの。
- 利用F1およびR1はpMG2343でmCherry- NAT1を生成するpMG2120とpMG2263、それぞれとF2とR2とのGFP-NAT1とYFP- NAT1カセットを生成します。
3. PCRによりFPカセットを生成します(1日目)
- PCR用試薬を準備します。 1.5ミリリットルチューブに以下の体積および濃度を追加することによって、マスターミックス(500μlの最終容量)を行います。50μlのPCR緩衝液(500mMの塩化カリウム、水中の100 mMトリスpHは8.0)、20μlのデオキシヌクレオチド(dNTP類;原料混合物各10mMのヌクレオチド)、40μlの25 mMの〜50-100 ngの/μlのストック溶液から塩化マグネシウム、20μlの精製されたプラスミド()、10μlの各フォワードのと、()10mMストック溶液から30μlのTaqポリメラーゼを逆方向プライマーポリメラーゼ(ジェネリック、5000単位/ ml)、および320μlの水。
- 10 PCR-の互換性がのそれぞれにマスターミックスのアリコート50μlのル0.5ミリリットルチューブ。
- 次の手順をサーモサイクラーにPCRチューブを配置し、実行:1サイクル分5を94℃で二本鎖DNAを変性させ; 94℃で45秒の40サイクルを順次、55℃で30秒のプライマーは、プラスミド鋳型DNAにアニールすることを可能にし、DNA産物の伸長のために68℃で4分間します。 72℃で15分の1の最終伸長サイクル。
注:PCRマスターミックス、サイクリングパラメータは、使用される特定のTaqポリメラーゼに基づいて変更する必要があります。 - プール1.5mlチューブ内の10のPCR反応からのすべての製品。
- アガロースゲル電気泳動にプールされたPCR産物の主題5μlのDNAラダーとの比較に基づいて、アンプリコンのサイズを確認し、生成物濃度の推定値を得ます。一般的に、後続の各形質転換混合物にカセットDNAの〜250μgのを使用します。
- 生成物を750μlの95%エタノール、続いて50μlの3M酢酸ナトリウムを添加することによってDNAを沈殿させ、少なくとも30分間インキュベート-20℃で
- 10分間16,000×gでチューブを遠心分離することにより、PCR産物を収穫。慎重に除去し、上清を捨て、一晩ペレットを乾燥させます。 40μlのTE pH8.0の中で乾燥させたDNAカセットペレットを再懸濁し、使用するまで室温で保管してください。
4. FP DNAカセットでカンジダ細胞を形質転換
- 1日目に、いくつかのストリーキングによって凍結15%グリセロール(-80°C)在庫から変換される回復する酵母株はアデニン(YPAD)寒天で酵母ペプトンデキストロース上に結晶を掻き取り、30℃でインキュベートします。コロニー増殖の回復後、通気キャップを有するガラス培養管2mlの液体YPAD培地に単一コロニーを接種し、攪拌しながら30℃で一晩インキュベートします。
- 2日目、通気性キャップと125ミリリットルの三角フラスコに(0.2〜の最終OD 600)新鮮なYPAD 50ミリリットルに一晩の酵母培養物の300μlの希釈します。 30℃で振ります(最終OD 600〜0.6から0.8へ)〜3時間。
- 50ミリリットルコニカルチューブに一晩培養物を注ぎ、テーブルトップ遠心機で1 500×gで5分間回転させることによって細胞をペレット化。
- 捨て、適切に上清を捨てます。 5ミリリットルの水で細胞ペレットを再懸濁します。テーブルトップ遠心機で1,500×gで5分間再度遠心分離して細胞を再ことペレット。
- 捨て、適切に上清を捨てます。 1.5mlチューブに転送中:500μlのTELiAc(10 mMトリス、pH8.0,1mMのEDTA、pHが8.0、0.1 M酢酸リチウムTEの酢酸リチウム)で細胞を再懸濁します。微量で3000×gで2分間遠心操作を。
- 250μlのTELiAcで細胞を再懸濁し。ペレットを含む総体積は〜300μlのでなければなりません。
- クリーン(4.2.4とは異なるマイクロチューブ)に、5μlのキャリアDNA(10mg / ml)および(4.2.4)から調製したカンジダ細胞の150μlのを追加します。これは、トンの陰性対照でありますransformation。
- 第二の清浄なマイクロチューブに、変性キャリアDNA5μlの(すなわち、10分間90℃で煮沸し、4℃に冷却されている)、(3.3.1)から調製されたPCR産物の全てを40μlおよび150を追加(4.2.4)から調製したカンジダ細胞 μlの。
- 室温で30分のための2つの形質転換混合物をインキュベートします。
- 各形質転換混合管に、700μlのプレートミックス(10mMのトリス、pH8.0の、1mMのEDTA、pH8.0の、50%のポリエチレングリコール3350 0.1 M酢酸リチウム)を加えます。 RTで一晩、それらを混合し、インキュベートするチューブを反転。
- 3日目に、変換は1時間42°C(ヒートショック)でミックスインキュベートします。
- 遠心分離機変換は微量で16,000×gで30秒間混合します。削除し、適切に上清を捨てます。細胞に損傷を与えないようにダウンして静かにピペッティングにより150μlの水で細胞ペレットのそれぞれを再懸濁します。
- 変換utのための栄養要求性マーカー遺伝子( 例えば 、URA3)を ilizing、適切な選択培地寒天( 例えばウリジンを欠く)の上にソリューションをピペッティングすることにより、各混合物全体をメッキし、滅菌ガラスビーズを用いて均一に混合物を広げました。
- ここに記載されるようにヌーセオスリシン耐性マーカー遺伝子(NAT1)を利用して変換するために、プレートの変換は、非選択YPAD寒天上に最初に混合し、6-12時間30℃でインキュベートします。ストレスをヌーセオスリシン前の細胞の回復、ポストヒートショックでこのステップ助剤は、適用されます。
- 部分的な成長の回復後、YPAD上にレプリカプレートカンジダ細胞が400μgの/ mlのヌーセオスリシン含みます。栄養マーカー遺伝子( 例えば、URA3) を利用して変換するために、この中間のめっき工程が不要であり、4.4.2に記載のように細胞を直接選択酵母媒体( 例えば YPAD欠くウリジン)上にプレーティングすることができます。
注:変換が成功した場合、列oniesは(ヌーセオスリシン含む寒天上での選択のための成長の潜在的に5日まで)1〜3日以内に表示されます。いいえコロニーは、単独でキャリアDNA(ネガティブコントロール)を含有する形質転換ミックスで拡散板に表示されないはずです。
- 栄養要求性とヌーセオスリシンマーカー選択のために、新鮮な選択培地寒天プレートに単一コロニーとしてスジ推定される形質転換体とFP融合の成功の建設のためにスクリーニングすることができる酵母細胞を増殖させるために30℃でインキュベートします。
- タギングカセットの正しい統合のための画面の形質転換体(詳しい例えば代表的な結果を参照してください)。目的の遺伝子は、十分な量で発現される場合、全体のコロニーの蛍光は、蛍光検出能力のプレートイメージングシステムを使用して、潜在的な候補の組込みを検出することができるように、発生する可能性があります。
- outsid配列とプライマーの相同性を用いたPCRによる推定の組込みを確認します積分領域の電子は、標的遺伝子への融合を確認しました。
- 既知の場合に加えて、タンパク質の局在を視覚的に確認のための単一細胞の蛍光顕微鏡検査によって、ならびに、融合タンパク質の発現およびサイズを決定するために、ウェスタンブロット分析を考えます。
Access restricted. Please log in or start a trial to view this content.
結果
一例として、我々は、プロトコルを使用するC.パラプシローシスの実験室株でENO1するGFPとmCherryを融合物を構築するために、上述。各推定形質転換体は、最初は成長のために再ストリークしました。得られた融合タンパク質が高度に発現されるので、この例では(エノラーゼ)とFPを明るく、我々は前の診断PCR( 図 3)6を
Access restricted. Please log in or start a trial to view this content.
ディスカッション
エピトープの構造は、上述したPCRによる遺伝子改変戦略を使用してカンジダ種における配列は3段階のプロセスとして要約することができるタグを付けました。まず、カセットは、両方の酵母ゲノムへの挿入の遺伝子座に相同な統合および領域に対して所望の配列をコードするPCRによって行われます。次に、形質転換される酵母細胞は、酢酸リチウムと化学的にコンピテントにされ、カ...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者らは、開示することは何もありません。
謝辞
私たちは、元のmCherryをFPシーケンスを提供するためのN.ディーンに感謝、M. Gerami-Nejadプラスミドの構築のために、このプロジェクトの開発中に有益な助言のための技術支援のためのB.ラーソン、およびT. Heisel。 JBは、欧州研究評議会上級賞340087(RAPLODAPT)によってサポートされていました。顕微鏡およびイメージングシステムは、ミネソタ小児科財団の大学とミネソタイメージングセンターの大学によって提供されました。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| 100 W mercury lamp | CHIU Technical Corporation | M-100T | |
| 95% Ethanol | Any | NA | |
| Adenine | Any | NA | |
| Ampicillin | Any | NA | |
| Carrier DNA | Ambion | AM9680 | Sheared Salmon Sperm DNA 10 mg/ml |
| CCD Camera | Photometrics | CoolSNAP HQ | |
| Conical Tube | Corning | 430828 | 50 ml |
| Culture Tube Rotator | New Brunswick | 2013923 | TC-8, or Any Culture Tube Rotator |
| Deoxynucleotides (dNTP) PCR Grade | Any | NA | |
| Eppendorf Tubes | Eppendorf | 022363719, 022363212 | 0.5 ml, 1.5 ml |
| Erlenmeyer Flask | Fisher Scientific | 7250089 | 125 ml |
| Ethylenediaminetetraacetic Acid (EDTA) | Any | NA | |
| Freezer (-80 °C) | Thermo Electron Corporation | ULT-1386-9-V | Revco Ultima II |
| GFP, YFP and Texas Red Filter Sets | Chroma Technology Corporation | 49002, 86004v2, 49008 | |
| Glass culture tubes | Fisher Scientific | 1496126 | 75 mm |
| HRP goat anti-mouse antibody | Santa Cruz Biotechnology | SC-2005 | |
| HRP goat anti-rabbit antibody | Santa Cruz Biotechnology | SC-2301 | |
| Incubator (30 °C) | Any | NA | |
| Lithium Acetate | Any | NA | |
| Lysogeny Broth (LB) Media | Any | NA | |
| Magnesium Chloride | Any | NA | |
| Microcentrifuge | Eppendorf | 5415 D | |
| Microscope | Nikon | E600 | Nikon Eclipse E600 |
| Microscope Image Analysis Software | Universal Imaging Corporation | 6.3r7 | MetaMorph Software Series 6.3r7 |
| Mouse anti-GFP antibody | Roche | 11814460001 | |
| Nourseothricin | Fisher Scientific | 50997939 | |
| PCR Thermocycler | Applied Biosystems | 9700 | GeneAmp PCR System |
| PCR tubes | BioExpress, GeneMate | T-3035-1 | 0.2 ml |
| Polyethylene Glycol 3350 | Any | NA | |
| Potassium Chloride | Any | NA | |
| Rabbit anti-mCherry antibody | BioVision | 5993-100 | |
| Refrigerator (4 °C) | Any | NA | |
| Sodium Acetate | Any | NA | |
| Stereomicroscope | Nikon | SMZ1500 | |
| Table Top Centrifuge | Labnet | Z 400 | Hermle Z 400 |
| Taq DNA Polymerase | Any | NA | |
| Tris(hydroxymethyl)aminomethane (Tris) | Any | NA | |
| Vortex Mixer | Scientific Industries | SI-0236 | Vortex Genie 2 |
| Yeast Extract Peptone Dextrose (YPD) Media | Any | NA |
参考文献
- Bendel, C. M. Colonization and epithelial adhesion in the pathogenesis of neonatal candidiasis. Semin. Perinatol. 27 (5), 357-364 (2003).
- Gerami-Nejad, M., Berman, J., Gale, C. A. Cassettes for PCR-mediated construction of green, yellow, and cyan fluorescent protein fusions in Candida albicans. Yeast. 18 (9), 859-864 (2001).
- Gerami-Nejad, M., Dulmage, K., Berman, J. Additional cassettes for epitope and fluorescent fusion proteins in Candida albicans. Yeast. 26 (7), 399-406 (2009).
- Gerami-Nejad, M., Forche, A., McClellan, M., Berman, J. Analysis of protein function in clinical C. albicans isolates. Yeast. 29 (8), 303-309 (2012).
- Gerami-Nejad, M., Hausauer, D., McClellan, M., Berman, J., Gale, C. Cassettes for the PCR-mediated construction of regulatable alleles in Candida albicans. Yeast. 21 (5), 429-436 (2004).
- Gonia, S., Larson, B., Gale, C. A. PCR-mediated gene modification strategy for construction of fluorescent protein fusions in Candida parapsilosis. Yeast. 33 (2), 63-69 (2016).
- Milne, S. W., Cheetham, J., Lloyd, D., Aves, S., Bates, S. Cassettes for PCR- mediated gene tagging in Candida albicans utilizing nourseothricin resistance. Yeast. 28 (12), 833-841 (2011).
- Ausubel, F. M., et al. Current Protocols in Molecular Biology. , Wiley. New York. (1995).
- Wilson, R. B., Davis, D., Mitchell, A. P. Rapid hypothesis testing with Candida albicans through gene disruption with short homology regions. J. Bacteriol. 181 (6), 1868-1874 (1999).
- Pulver, R., et al. Rsr1 focuses Cdc42 activity at hyphal tips and promotes maintenance of hyphal development in Candida albicans. Eukaryotic Cell. 12 (4), 482-495 (2013).
- Falgier, C., et al. Candida species differ in their interactions with immature human gastrointestinal epithelial cells. Pediatr. Res. 69 (5), 384-389 (2011).
- Nosek, J., et al. Genetic manipulation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 42 (1), 27-35 (2002).
- Zemanova, J., Nosek, J., Tomaska, L. High-efficiency transformation of the pathogenic yeast Candida parapsilosis. Curr. Genet. 45 (3), 183-186 (2004).
- Benjamin, D. K., et al. Neonatal candidiasis among extremely low birth weight infants: risk factors, mortality rates, and neurodevelopmental outcomes at 18 to 22 months. Pediatrics. 117 (1), 84-92 (2006).
- Kullberg, B. J., Arendrup, M. C. Invasive Candidiasis. N Engl J Med. 373 (15), 1445-1456 (2015).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved