A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
איתור דנ"א ללא תאים בדגימות פלזמה בדם של חולי סרטן
In This Article
Summary
במאמר זה אנו מציגים פרוטוקול מפורט עבור טכניקת ביופסיה נוזלית לא פולשנית, כולל איסוף דם, פלזמה והפרדת מעיל באפי, cfDNA ומיצוי DNA נבט, כימות של CFDNA או DNA נבט, וניתוח העשרת שבר cfDNA.
Abstract
זיהוי מוטציות בגידולים של חולי סרטן הוא צעד חשוב מאוד בניהול מחלות. מוטציות אלה משמשות סמנים ביולוגיים לאבחון הגידול, כמו גם לבחירת הטיפול ותגובתו בחולי סרטן. השיטה הנוכחית של תקן הזהב לגילוי מוטציות בגידול כרוכה בבדיקה גנטית של דנ"א גידולי באמצעות ביופסיות גידול. עם זאת, שיטה פולשנית זו קשה להתבצע שוב ושוב כבדיקת מעקב של רפרטואר מוטציה הגידול. ביופסיה נוזלית היא טכניקה חדשה ומתפתחת לגילוי מוטציות בגידולים כגישה ביופסיה קלה לשימוש ולא פולשנית.
תאים סרטניים מתרבים במהירות. במקביל, תאים סרטניים רבים עוברים אפופטוזיס. פסולת מתאים אלה משתחררים לתוך מערכת הדם של המטופל, יחד עם חתיכות DNA מקוטע דק, הנקרא DNA ללא תאים (cfDNA) שברים, אשר נושאים מוטציות DNA הגידול. לכן, לזיהוי סמנים ביולוגיים מבוססי cfDNA באמצעות טכניקת ביופסיה נוזלית, דגימות דם נאספות מחולי הסרטן, ואחריו הפרדת פלזמה ומעיל באפי. לאחר מכן, פלזמה מעובדת לבידוד של cfDNA, ואת מעיל באפי בהתאמה מעובד לבידוד של ה-DNA הגנומי של המטופל. שתי דגימות חומצת גרעין נבדקות לאחר מכן עבור כמות ואיכותן; ונותחו עבור מוטציות באמצעות טכניקות רצף הדור הבא (NGS).
בכתב יד זה, אנו מציגים פרוטוקול מפורט לביופסיה נוזלית, כולל איסוף דם, פלזמה והפרדת מעיל באפי, הפקת דנ"א cfDNA ונבט, כימות של CFDNA או DNA נבט, וניתוח העשרת שבר cfDNA.
Introduction
ההתקדמות הטכנולוגית הובילה לריצוף של מאות גנומים וסרטן ותעתיקים1. זה תרם להבנת נופים של שינויים מולקולריים על פני סוגי סרטן שונים2. מחקרים נוספים על נופים אלה סייעו לאפיין את השינויים הסומטיים הרציפים והיתוךגנים-גנים 3 המעורבים בהתקדמות סרטן או גידול, על ידי שיבוש סדרתי של מסלולי אפופטוזיס4. לכן, מוטציות סומטיות והיתוך גנים-גנים יכולים לספק מידע על גידולים על ידי לשמש סמנים ביולוגיים בחולים בודדים עבור סוג גידול מסוים5, זיהוי גידולים ראשוניים קיימים פרוגנוזה6, סיווג גידולים משניים המבוססים על שינויים מולקולריים7, וזיהוי מטרות גידול תרופתי8. מידע כזה עשוי להקל בבחירת טיפול מותאם אישית לחולי סרטן ובקביעת תגובות טיפול חיוביות ושליליות9. עם זאת, קבלת חומר הגידול לזיהוי פרופיל גנומי של רקמת הגידול הוא הליך פולשני10. יתר על כן, ביופסיה גידול מורכב רק חלק קטן של גידול הטרוגניים; ולכן, לא להיות מייצג את הפרופיל המולקולרי של הגידול כולו11. ניטור סדרתי ו genotyping הגידול דורשים אוסף חוזר ונשנה של רקמות הגידול, אשר, בדרך כלל, אינו ריאלי בשל פולשנות של הליך ביופסיה הגידול ואת בעיות הבטיחות הנובעות הליכים כאלה12.
טכניקת הביופסיה הנוזלית, לעומת זאת, צברה תשומת לב עצומה באונקולוגיה מדויקת בעשור האחרון13,14. זה בעיקר בשל אי פולשניות של טכניקה זו, ואת האפשרות של זה חוזר על עצמו בנקודות זמן מרובות, ובכך מאפשר טכניקה קלה לשימוש ניטור בטוח עבור קורסי המחלה15,16. ביופסיה נוזלית מבוססת על תופעה שתאי הגידול מתרבים במהירות, ובמקביל רבים מהם עוברים אפופטוזיס ונמק. זה מוביל לשחרור פסולת תא אפופטוטי לדם של המטופלים, יחד עם שברי ה- DNA שנחתכים בגדלים מדויקים במהלך אפופטוזיס17. האפופטוזיס של תאים לא סרטניים מוביל גם לשחרור הפסולת התאית שלו לדם, עם זאת, שיעור האפופטוזיס בתאים אלה נמוך יחסית לתאי הגידול18. הרציונל של טכניקת הביופסיה הנוזלית הוא ללכוד מולקולות הקשורות לגידולים כגון DNA, RNA, חלבונים ותאי גידול14,19 אשר מסתובבים ברציפות בדם. טכניקות שונות20 ניתן להשתמש לניתוח של מולקולות אלה כולל הדור הבא רצף (NGS), תגובת שרשרת פולימראז טיפה דיגיטלית (ddPCR), PCR בזמן אמת, ואנזים מקושר immunosorbent assay (ELISA). טכניקת ביופסיה נוזלית מאפשרת זיהוי סמנים ביולוגיים שהם מאפיינים של תאים סרטניים. מולקולות סמן ביולוגי אלה אינן משתחררות רק מחלקים מסוימים של גידול, אלא מכל חלקי הגידול21. לפיכך, סמנים שזוהו בביופסיה נוזלית מייצגים את הפרופיל המולקולרי של גידול הטרוגנטי שלם, בנוסף לגידולים אחרים בגוף, ובכך, יש יתרונות על טכניקה מבוססת ביופסיה רקמה22.
cfDNA יש זמן מחצית חיים קצר בדם במחזור הנע בין כמה דקות 1-2 שעות23. עם זאת, זמן מחצית החיים הקצר של cfDNA מקל על ניתוחים בזמן אמת על ידי הערכת תגובת הטיפול והערכות גידול דינמי. רמות cfDNA שמקורן בגידול מצביעות על פרוגנוסטיזציה של שלב הגידול / גודל העיד על ידי מספר מחקרים, אשר הראו קשר בין רמות cfDNA ותוצאות ההישרדות24. יתר על כן, מחקרים הוכיחו כי cfDNA יש יכולת חיזוי טובה יותר מאשר סמני הגידול הקיימים25. הפרוגנוסטיזציה של cfDNA בולטת עוד יותר לאחר טיפול בסרטן, רמות גבוהות יותר של cfDNA בעקבות הטיפול בקורלציה טובה עם שיעור הישרדות מופחת, ועמידות לטיפול. בעוד, רמות נמוכות יותר של cfDNA בעקבות טיפול בדרך כלל מתאים עם תגובה טיפולית חיובית. בנוסף, cfDNA מאפשר גילוי מוקדם של תגובת טיפול מאשר שיטות הגילוי המסורתיות.
CFDNA מגביר את האפשרות של גילוי מוקדם של מוטציות הקשורות לסרטן: במהלך מחלה בשלב מוקדם15, תחילת הסימפטומים26 ולפני אבחון סרטן עד 2 שנים27. כמו cfDNA משתחרר מאזורי גידול מרובים או מוקדים, הניתוח שלה מספק תצוגה מקיפה של הגנום הגידול הוא מייצג28. לכן, cfDNA מאפשר לזהות מוטציות סומטיות שאולי הוחמצו בדגימות הרקמה29. כמו הטרוגניות תוך גידול מוטציות תת שבטיות ניתן לזהות על ידי רצף עמוק של אזורים גנומיים פורש אלפי בסיסים, ומכאן הניתוח של cfDNA מאפשר לחשוף תת סוגים מולקולריים ספציפיים עם חתימות גנומיות ברורות13. כדי להשיג רמה דומה של מידע באמצעות דגימת רקמה ביופסיות מוצקות רבות היה צורך.
יתר על כן, רמות cfDNA בחולים עם מחלה מקומית כגון המעי הגס, השחלות, וסרטן הריאות לאחר טיפול כירורגי ו / או כימותרפיה, הוכיח להיות סמן פרוגנוסטי רב עוצמה עבור הישנות סרטן ותוצאות הטיפול20. יתר על כן, בחולים עם סרטן המעי הגס, השד והריאות, ניתוחים של cfDNA מהדם יכול לזהות בהצלחה את השינויים ספציפיים לגידול, מה שהוביל לחיזוי מדויק של הישנות מספר חודשים מראש13. יתר על כן, סמני עמידות לטיפול, כגון מוטציות KRAS בחולים עם CRC קבלת טיפול נגד EGFR30; VAFs עבור גנים כגון PIK3CA, MED1 או EGFR בחולים עם סרטן השד לאחר הטיפול עם טיפולים שונים31; מוטציית התנגדות EGFR T790M בחולי סרטן ריאות שטופלו TKIs ממוקד EGFR32 ניתן לזהות גם על ידי ניתוח cfDNA.
לסיכום, ניתוח cfDNA יכול לשמש לזיהוי סמנים ביולוגיים מדויקים בתחום האונקולוגיה13,33. בפרוטוקול זה, דגימות דם של 3 חולי גליומה ו 3 פקדים בריאים עובדו כדי לקבל DNA גנומי מ WBCs ו cfDNA מהפלזמה. בסרטן גליומה, מוטציות IDH, TERT, ATRX, EGFR, ו TP53 משמש אבחון, כמו גם סמנים פרוגנוסטיים שעשויים לעזור באבחון מוקדם של גידולים גליומה, סיווג סוגים שונים של גידולים גליומה, המנחה את הטיפול המדויק עבור המטופל הפרט והבנת התגובההטיפולית 34,35. מצב מוטציה של גנים אלה ניתן לזהות באמצעות cfDNA שמקורו בדם. בכתב יד זה, אנו מציגים פרוטוקול מפורט של cfDNA שמקורו בפלזמה ששימש לחקר שינויים מוטציה בסרטן גליומה12. כזה cfDNA מבוססי פרוטוקול ביופסיה נוזלית הסביר במאמר זה יכול לשמש לחקר שינויים מוטציה בסוגים רבים אחרים של סרטן. יתר על כן, מחקר שנערך לאחרונה הראה כי ביופסיה נוזלית מבוססת cfDNA יכולה לזהות 50 סוגים שונים של סרטן36.
איסוף דגימת דם, אחסון, ומשלוח הם צעדים מכריעים בפרוטוקול זה, כמו טמפרטורה בלתי מבוקרת במהלך שלבים אלה גורם תמוגה של WBCs, המוביל לשחרור של DNA גנומי מן WBC לתוך הפלזמה ולגרום לזיהום של מדגם cfDNA, אשר משפיע על שאר ההליך37. המוליזה עקב טמפרטורה בלתי מבוקרת עלולה לפגוע בתהליכי הכנת מדגם במורד הזרם של cfDNA, כגון שלבים PCR38. הסרום מכיל שיעור גבוה של נבט cfDNA ולא פלזמה, למרות שהוא מציג רעש רקע גדול עבור cfDNA הקשורים לגידול39. לכן, עבור בידוד cfDNA הקשורים לגידול, פלזמה היא מדגם מתאים39. דם נמשך צינור איסוף דם המכיל קרישה צריך להיות צנטריפוגה מיד או בתוך שעתיים, כדי להפריד את הפלזמה כדי למנוע זיהום cfDNA. בפרוטוקול זה, צינורות ייעודיים לשימור cfDNA דם שימור משמשים (ראה טבלה של חומרים), המהווים חלופה נוגדי קרישה המכילים צינורות איסוף דם. אלה צינורות איסוף דם ייעודי לשמר cfDNA ו cfRNA, ומונע תמוגה של WBCs עד 30 ימים בטמפרטורת הסביבה, ועד 8 ימים ב 37 °C (69 °F). זה מקל על שמירה על הטמפרטורה המתאימה במהלך משלוח דגימת דם ועד הפלזמה ו- WBC מופרדים40.
ישנם שלושה סוגים של מתודולוגיות מיצוי cfDNA זמין כיום: בידוד פאזה, טור ספין מבוסס סיליקון קרום, ובידוד מבוסס חרוז מגנטי41. שיטת עמודת ספין מבוססת ממברנת סיליקון הניבה כמות גבוהה של cfDNA עם שלמות גבוהה בהשוואה לשיטות חילוץ cfDNA אחרות42.
ההערכה הכמותית של ה- DNA היא דרישה בסיסית בביופסיה נוזלית, יש צורך לפתח הליך פשוט, סביר ומתוקנן ליישומם הקל ולשימוש רחב. שלוש שיטות נפוצות לכימות cfDNA הן ספקטרופוטומטריות, פלואורימטריות ו- qPCR. השיטה הפלואורימטרית מוכחת טוב יותר על פני שיטות אחרות לגבי הדיוק, העלות וקלות ההולכה43.
השלמות והטוהר של cfDNA ניתן להעריך על ידי אלקטרופורזה agarose או אלקטרופורזה נימי. אלקטרופורזה אגרוז לא מראה רגישות בריכוז נמוך של cfDNA וגם אין רזולוציה גבוהה כדי להראות גודל שבר מדויק של cfDNA. מצד שני, אלקטרופורזה נימי יש יתרון על אלקטרופורזה agarose על ידי התגברות על האתגרים הקשורים, ולכן, בשימוש נרחב על ידי החוקרים לניתוח גודל שבר cfDNA. בפרוטוקול זה, התפלגות גודל הקטע של cfDNA מבודד הוערך באמצעות מכשיר אלקטרופורזה נימי אוטומטי (ראה טבלה של חומרים).
Protocol
לפני איסוף הדם, נדרשת הסכמה מדעת של הנבדקים המשתתפים במחקר ויש להשיגה. המחקר המתואר בכתב יד זה בוצע בהתאם לעמידה במרכז הרפואי רבין, בוועדת האתיקה הישראלית (קוד אתי: 0039-17-RMC) ובפקולטה לרפואה דר כריסטיאן-אלברכטס-אוניברסיטת זו קיל, ועדת האתיקה של גרמניה (קוד אתי: ד' 405/14).
1. איסוף ואחסון דגימת דם בצינורות משמרים cfDNA או cfRNA
- תווית נכונה של צינורות השימור
- לאסוף ~ 8 מ"ל של דם לתוך צינור שימור CF-DNA (ראה טבלה של חומרים),באמצעות ערכת איסוף דם ומחזיק, לפי הפרוטוקול המוסדי הסטנדרטי עבור venipuncture כמתואר להלן.
הערה: השימוש בערכת איסוף דם עשוי למנוע זרימה אחורית אפשרית של הדם מהצינור.- ליישר את המטופל עם הזרוע בתנוחה כלפי מטה.
- החזק את הצינור זקוף, עם הכובע פונה כלפי מעלה, תוך הבטחת כי תכולת הצינור לא לגעת כובע או קצה מחט.
- כשהדם מתחיל לזרום לתוך הצינור, שחררו את חוסם העורקים לאט.
- מיד לאחר הצינור מלא בדם (קיבולת מקסימלית: 8.4 מ"ל של דם שלם), בעדינות להפוך את הצינור (להפוך את פרק כף היד של הזרוע המחזיקה את הצינור על ידי 180 ° כלפי מטה ובחזרה) 5 פעמים כדי לייצב את המדגם.
הערה: היפוך מבטיח שהחומר המשמר מעורבב באופן אחיד עם הדגימה. עם זאת, לא לנער את התוכן שוב, עוד לפני הכנת פלזמה. ערבוב לא מספיק של חומרים משמרים עם דגימת הדם מוביל לחוסר יציבות של התוכן והיווצרות של קרישי מיקרו או המוליסיס. בשלב זה, הפרוטוקול יכול להימשך מיד עבור הפרדת פלזמה או צינורות מלאים בדם יכול לחכות עד 30 ימים בטמפרטורת הסביבה (15-25 מעלות צלזיוס), ועד 8 ימים ב 37 מעלות צלזיוס.
2. הפרדת מעיל פלזמה ומעיל באפי ואחסון
- צנטריפוגה צינור שימור מלא בדם ב 425 x g במשך 20 דקות בטמפרטורת החדר כדי להפריד פלזמה.
הערה: שלבים 2.2 ו- 2.3 צריכים להתבצע בארון biosafety. - בזהירות פיפטה החוצה את שכבת הפלזמה העליונה לצינור טרי ב 1 aliquots מ"ל, מבלי להפריע את השכבות התחתונות.
- בזהירות להעביר את השכבה הבאה של מעיל באפי לצינור טרי (השכבה מופיעה כטבעת מעל כדורי RBC), תוך הימנעות RBCs בשכבה התחתונה.
- המשך לשלב 3 עם פלזמה וצעד 4 עם מעיל באפי. במידת הצורך לאחסן את התוכן המופרד ב -80 °C (69 °F).
3. טיהור של cfDNA במחזור מ 1 מ"ל של פלזמה
הערה: שלב זה מבוצע עם ערכה מסחרית (ראה טבלת חומרים). כל המאגרים מסופקים עם הערכה.
- הכנת מאגרים ריאגנטים
התראה: אין להוסיף פתרונות חומציים או להלבין ישירות לפסולת ההכנה לדוגמה. מלחי גואנידין הנמצאים במאגר תמוגה, חיץ כריכה ו-Wash Buffer-1 בשילוב עם אקונומיקה או חומצות יכולים לייצר תרכובות תגובתיות מאוד.- מאגר איגוד: לערבב 300 מ"ל של רכז מאגר איגוד עם 200 מ"ל של 100% isopropanol כדי להפוך 500 מ"ל של מאגר איגוד עובד. יש לאחסן בטמפרטורת החדר.
הערה: חיץ כריכה מאפשר את הכריכה האופטימלית של חומצות הגרעין במחזור לקרום סיליקה. 500 מ"ל של מאגר הכריכה מספיק לעיבוד 276, 138, 92, 69 או 55 דגימות של 1, 2, 3, 4 או 5 מ"ל של פלזמה בהתאמה והוא יציב במשך שנה בטמפרטורת החדר. - Wash Buffer-1: יש לערבב 19 מ"ל של תרכיז Wash Buffer-1 עם 25 מ"ל של 96-100% אתנול כדי ליצור 44 מ"ל של Wash Buffer-1 עובד. יש לאחסן בטמפרטורת החדר.
הערה: Wash Buffer-1 מבטל את המזהמים הקשורים לקרום סיליקה. 44 מ"ל של עבודה לשטוף Buffer-1 מספיק לעיבוד 73 דגימות של 1/2/3/4/5 מ"ל של פלזמה והוא יציב במשך שנה אחת בטמפרטורת החדר. - שטיפת חיץ-2: מערבבים היטב 13 מ"ל לשטוף חוצץ-2 להתרכז עם 30 מ"ל של 96-100% אתנול לעשות 43 מ"ל של עבודה לשטוף Buffer-2. יש לאחסן בטמפרטורת החדר.
הערה: Wash Buffer-2 מבטל את המזהמים הקשורים לקרום סיליקה. 43 מ"ל של עבודה לשטוף Buffer-2 מספיק לעיבוד ~ 56 דגימות של 1/2/3/4/5 מ"ל של פלזמה והוא יציב במשך שנה אחת בטמפרטורת החדר. - לצינור המכיל RNA נושאת 310 מיקרוגרם lyophilized, להוסיף 1,550 μL של מאגר Elution, כדי להכין פתרון RNA המוביל של 0.2 מיקרוגרם / μL. לאחר המסה יסודית של ה-RNA המוביל, מחלקים את הפתרון לאליקוטים מתאימים ומאחסנים ב-30°C עד 15 °C (60 °F) עד 15 °C (60 °F). אין להקפיא-להפשיר את ה aliquots אלה יותר מ 3 פעמים. למאגר הליסיס, כפי שמוצג בטבלה S1, הוסף את RNA המוביל מחדש מומס במאגר Elution.
הערה: מכיוון ש-RNA של המוביל אינו מתמוסס ישירות במאגר הליסיס, יש להמיס אותו תחילה במאגר Elution ולאחר מכן במאגר תמוגה. ראשית, חומצות קרום-גרעין סיליקה מחייב משופר כאשר יש מעט מאוד מולקולות היעד נוכח המדגם. שנית, הסיכון של השפלה RNA מצטמצם בגלל נוכחות של כמויות גדולות של RNA נושאת.
- מאגר איגוד: לערבב 300 מ"ל של רכז מאגר איגוד עם 200 מ"ל של 100% isopropanol כדי להפוך 500 מ"ל של מאגר איגוד עובד. יש לאחסן בטמפרטורת החדר.
- לפני תחילת הבידוד, הביאו את העמודות והדגימות לטמפרטורת החדר והתאימו את נפחי הדגימה ל- 1 מ"ל עם מלוחים סטריליים עם אגירת פוספט (PBS), במידת הצורך. מחממים מראש 2 אמבטיות מים או בלוקי חימום המכילים צינורות צנטריפוגה 50 מ"ל וצינורות איסוף 2 מ"ל ל 60 °C (60 °F) ו 56 °C (66 °F), בהתאמה.
- לצינור צנטריפוגה של 50 מ"ל, הוסיפו 100 μL של Proteinase K, פלזמה 1 מ"ל ו-0.8 מ"ל של מאגר תמוגה המכיל 1.0 מיקרוגרם של RNA נושא (מוכן בשלב 3.1.4). סגור את צינור הצנטריפוגה עם כובע ומערבבים את התוכן על ידי דופק מערבולת במשך 30 s, תוך הבטחת מערבולת גלויה בצינור. ערבוב יסודי של התוכן חשוב עבור תמוגה יעילה.
הערה: מיד לאחר המערבולת, המשך לשלב 3.4, ללא דיחוי. - דגירה את הפתרון ב 60 מעלות צלזיוס במשך 30 דקות.
- מוציאים את המכסה, מוסיפים 1.8 מ"ל של מאגר הכריכה לצינור, ומערבבים היטב עם מערבולת דופק למשך 15-30 שניות לאחר הנחת המכסה.
- דגירה את התערובת וכתוצאה מכך במשך 5 דקות על קרח ולהכניס את עמוד קרום סיליקה לתוך מנגנון ואקום המחובר למשאבת ואקום. לאחר מכן, הכנס בחוזקה מרחיב צינור 20 מ"ל לתוך העמודה הפתוחה כדי למנוע דליפת מדגם.
- יוצקים בזהירות את תערובת הדגירה לתוך מרחיב הצינור של העמודה ומדליקים את משאבת הוואקום. אחרי כל תערובת lysate פועל לחלוטין דרך העמודים, לכבות את משאבת ואקום, לשחרר את הלחץ 0 mbar, ולהסיר ולהשליך את מרחיב הצינור.
הערה: הימנעות זיהום צולב, מאריך הצינור צריך להיות מושלך בזהירות, כדי למנוע את התפשטותו על פני עמודים סמוכים. - הסר את העמודה ממנגנון ואקום, להכניס לתוך צינור האיסוף, וצנטריפוגה ב 11,000 x g עבור 30 s בטמפרטורת החדר, כדי להסיר כל lysate שיורית. השלך את הזרימה.
- הוסף 600 μL של חוצץ לשטוף-1 לתוך העמודה, צנטריפוגה ב 11,000 x g במשך 1 דקות בטמפרטורת החדר, להשליך את הזרימה דרך.
- הוסף 750 μL של חוצץ לשטוף-2 לעמודה, צנטריפוגה ב 11,000 x g במשך 1 דקות בטמפרטורת החדר, ולהשליך את הזרימה דרך.
- הוסף 750 μL של אתנול (96–100%) לעמודה, צנטריפוגה ב 11,000 x g במשך 1 דקות בטמפרטורת החדר ולהשליך את הזרימה דרך.
- צנטריפוגה העמודה ב 20,000 x g במשך 3 דקות, על ידי הצבת אותו בצינור אוסף נקי 2 מ"ל.
- יבש את הרכבת עמוד הממברנה לחלוטין על ידי הצבתו לתוך צינור אוסף חדש 2 מ"ל עם המכסה פתוח דגירה ב 56 מעלות צלזיוס במשך 10 דקות.
- מניחים את העמוד בצינור elution נקי 1.5 מ"ל. למרכז ממברנת העמוד, יש למרוח 20-150 μL של חיץ Elution ותדגיר בטמפרטורת החדר למשך 3 דקות כאשר המכסה סגור.
הערה: ודאו שמאגר Elution משוויון לטמפרטורת החדר. במקרה של שימוש במאגר elution פחות מ 50 μL, ודא כי הוא מחולק בזהירות על מרכז הממברנה. זה עוזר עם התרוממות רוח מלאה של ה-DNA כבול. עם זאת, אמצעי האחסון elution אינו קבוע וניתן לשנות לפי היישומים במורד הזרם. eluate התאושש יכול להיות עד 5 μL ובוודאי פחות נפח elution להחיל על העמודה. - צנטריפוגה eluate התאושש ב microcentrifuge ב 20,000 x g במשך 1 דקות כדי elute חומצות גרעין, ולאחסן ב -20 °C (69 °F).
4. טיהור דנ"א גנומי ממעיל באפי
הערה: ערכה מסחרית המשמשת בפרוטוקול זה מוזכרת בטבלת החומרים. מאגרים ריאגנטים המוזכרים בפרוטוקול הבא כלומר, מאגר תמוגה A, מאגר תמוגה B, חוצץ לשטוף X, לשטוף חוצץ Y, מאגר פרוטאינאז, מאגר Elution, ו Proteinase K הם חלק ערכה מסחרית זו.
- הכנת המאגרים והריאגנטים
התראה: אין להוסיף פתרונות חומציים או להלבין ישירות לפסולת ההכנה לדוגמה. מלחי גואנידין הנמצאים במאגר Lysis B ו- Wash Buffer X בשילוב עם אקונומיקה או חומצות, יכולים לייצר תרכובות תגובתיות מאוד.- שטיפת חוצץ Y: לערבב היטב 12 מ"ל של רכז Y חוצץ לשטוף עם 48 מ"ל אתנול (96–100%) כדי לקבל 60 מ"ל של עבודה לשטוף מאגר Y. חנות בטמפרטורת החדר.
הערה: 60 מ"ל של עבודה לשטוף חוצץ Y מספיק לעיבוד 100 דגימות מעיל באפי והוא יציב במשך 1 שנה. - Proteinase K: הכינו את תמיסת Proteinase K על ידי המסת 30 מ"ג חלבון K ליאופילי ל-1.35 מ"ל של מאגר פרוטאיניאז.
הערה: פתרון העבודה הכולל של Proteinase K מספיק לעיבוד 52 דגימות מעיל באפי. ניתן לאחסן את פתרון העבודה Proteinase K למשך 6 חודשים לפחות ב-20 °C (60 °F).
- שטיפת חוצץ Y: לערבב היטב 12 מ"ל של רכז Y חוצץ לשטוף עם 48 מ"ל אתנול (96–100%) כדי לקבל 60 מ"ל של עבודה לשטוף מאגר Y. חנות בטמפרטורת החדר.
- שלבים לפני תחילת ההליך
- השווה את המעיל הבאפי לטמפרטורת החדר.
- הגדר את בלוק החום או אמבט מים ב 56 מעלות צלזיוס.
- להשעות מעיל באפי במאגר Lysis A כדי לקבל נפח סופי של 200 μL. לאחר מכן הוסיפו 25 μL של תמיסת Proteinase K, ו-200 μL של מאגר Lysis B. ערבבו על ידי מערבולת וגרירה ב-70°C למשך 10-15 דקות. ודא שהדגימות מכוסות לחלוטין בתמיסת התמוגה.
הערה: לעיבוד סדרה של דגימות, Proteinase K ומאגר Lysis A עשוי להיות premixed 10-15 דקות לפני ההליך, אבל כבר לא לפני זה, כמו Proteinase K עיכול עצמי במאגר Lysis A ללא מצע. - הוסף 210 μL של 96–100% אתנול לתערובת לעיל ומערבולת במרץ.
הערה: תוספת של אתנול עשויה ליצור משקע מחרוזת; עם זאת, זה לא ישפיע על בידוד ה-DNA. הקפד לטעון את המשקעים גם בעמודה, כפי שמוצג בשלבים הבאים. - טען את כל הדגימה על עמוד הסיליקה הממוקם בצינור איסוף. צנטריפוגה למשך דקה ב-11,000 x גרם. מקם את העמודה בצינור אוסף חדש והשליך את הצינור הקודם יחד עם זרימה דרך.
הערה: חזור על שלב הצנטריפוגה אם המדגם אינו נמשך לחלוטין דרך המטריצה. - הוסף 500 μL של מאגר לשטוף X, צנטריפוגה במשך 1 דקות ב 11,000 x g, ולהשליך את הזרימה דרך.
- הנח את העמודה בצינור האיסוף, הוסף 600 μL של Wash Buffer Y על העמודה, צנטריפוגה למשך דקה אחת ב- 11,000 x g והשליך את הזרימה.
- שוב, למקם את העמודה לתוך צינור האיסוף, וצנטריפוגה העמודה במשך 1 דקה ב 20,000 x g לייבש את קרום סיליקה.
- דגירה את העמוד בטמפרטורת החדר במשך 1 דקות, ממוקם לתוך צינור microcentrifuge 1.5 מ"ל, ולאחר מכן להוסיף 100 μL של מאגר Elution. לאחר מכן, לגמיש את ה-DNA על ידי צנטריפוגה במשך 1 דקות ב 11,000 x g ולאחסן ב -20 מעלות צלזיוס.
5. כימות של cfDNA ודנ"א גנומי באמצעות פלואורומטר
- לפני הפעלת הפרוטוקול, בצע את השלבים הבאים.
- לדלל 2 μL של DNA גנומי eluted (משלב 4.9) בפרופורציות 1:10 עם מים ללא גרעין אולטרה-דק. בשל ריכוזים נמוכים צפויים, לא לדלל דגימות cfDNA משלב 3.15.
- השווה את #1 הסטנדרטיים של מבחני ההסתעפות ו- assay Standard #2 לטמפרטורת החדר.
- הכינו בסך הכל 6 צינורות צלולים דקי קירות בגודל 0.5 מ"ל.
הערה: הפרוטוקול המוצג הוא לכימות של 2 cfDNA ו 2 דגימות DNA גנומי, ולכן, 4 צינורות עבור 4 דגימות ו assay זה דורש 2 סטנדרטים. - תייג את העפעפיים של הצינור.
הערה: תיוג בצד הצינור עלול להפריע לקריאה. בנוסף, צינורות תקן הבדיקה מסומנים בקפידה, שכן כיול של פלואורומטר דורש כי הסטנדרטים הם בסדר הנכון. - לדלל את ריאגנט ההסמכה ב 1:200 עם מאגר Assay כדי להכין את פתרון העבודה. עבור 4 דגימות ו 2 תקנים, להשתמש 6 μL של מגיב assay בתוספת 1,194 μL של מאגר Assay לעשות 1,200 μL (200 μL בכל צינור) של פתרון עבודה.
הערה: אין להשתמש במיכל זכוכית, במקום זאת להשתמש בצינור פלסטיק נקי. כל צינור חייב להכיל כ 200 μL של הנפח הסופי (צינור תקן מבחני חייב להכיל 190 μL של פתרון העבודה, ואת הצינור מדגם חייב להכיל 180-199 μL של פתרון העבודה). פתרון עבודה מספיק חייב להיות מוכן כדי להתאים לכל הסטנדרטים והדגימות של מבחנים. - בצינורות הסטנדרטיים של מבחני העבודה, הוסיפו 190 μL של פתרון עבודה ותקן 10 μL assay וערבבו את הפתרון על ידי מערבולת למשך 2-3 שניות. הימנע היווצרות של בועות בתוך הפתרון.
- בצינורות המדגם, להוסיף 198 μL פתרון עבודה ו 2 μL של cfDNA או DNA גנומי. מערבבים את הפתרון על ידי מערבולת במשך 2-3 s, ולשמור אותו דגירה בטמפרטורת החדר במשך 2 דקות.
- במסך 'Home' של מכשיר הפלואורומטר, לחץ על 'DNA' ובחר 'dsDNA רגישות גבוהה Assay', כדי להציג את המסך 'סטנדרטים', ולאחר מכן הקש 'כן' על מסך' סטנדרטים' פלואורומטר כדי לקרוא את הסטנדרטים.
- בתא הדגימה, הכנס את השינור הסטנדרטי #1, סגור את המכסה ולחץ על 'קרא'. הסר את הצינור לאחר השלמת הקריאה (כ 3 s) ולחזור על אותו שלב עבור #2 רגיל.
- מסך לדוגמה מוצג לאחר השלמת תהליך הכיול ולאחר מכן הכנס צינור לדוגמה וחזור על שלב '5.8'. "מסך המדגם" יציג לאחר מכן ערך התואם את ריכוז המדגם לאחר דילול בצינור המדגם.
- עבור כל שלב חזרה לדוגמה '5.9', עד לקריאת כל הדגימות.
- השתמש במשוואה הבאה כדי לחשב את הריכוז הממשי של המדגם.
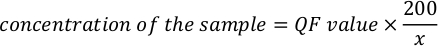
הערה: ערכי ההסתעפות נמצאים ב- ng/mL ומתאימים לריכוז לאחר דילול בצינור התסעפות. משוואה שהוזכרה לעיל שלב 5.11, ערך QF הוא הערך שניתן על ידי מכשיר פלואורומטר, ו- x הוא מספר microliters של מדגם הוסיף צינור הבדיקה. היחידות עבור ערכי QF הנוצרים על-ידי המשוואה דומות לערך שמספק הפלואורומטר. לדוגמה, אם הערך של הפלואורומטר הוא ב- ng/mL, היחידות עבור הריכוז המחושב על-ידי המשוואה הן ng/mL.
6. התפלגות גודל שבר DNA של cfDNA על ידי מנתח שבר
- השלב לפני ייזום ההליך: לאכוף את תרכיז צבע DNA ומטריצת ג'ל DNA לטמפרטורת החדר במשך 30 דקות.
- הכנת תערובת צבע הג'ל
התראה: טפל בפתרונות בזהירות שכן DMSO ידוע כמקל על כניסת מולקולות אורגניות לרקמות.- ביסודיות להפשיר את DMSO על ידי מערבולת בקבוקון רכז צבע DNA במשך 10 s. פיפטה החוצה 15 μL של תרכיז זה לתוך בקבוקון מטריצת DNA ג'ל ולאחסן ב 4 מעלות צלזיוס בחושך.
- שוב, מערבולת הבקבוקון כתרים במשך 10 s עד ערבוב של ג'ל וצבע הוא דמיין.
- יוצקים את התערובת על מסנן ספין לכלי הקיבול העליון.
- מיקרוצנטריפוגה מסנן ספין ב 2,240 x גרם ± 20% במשך 10 דקות בטמפרטורת החדר.
- סמן את צבע הג'ל המוכן בצינור והשליך את המסנן, בהתאם לשיטות מעבדה טובות. תייג את הצינור והקלט את תאריך ההכנה.
הערה: השלך את הסינון לפי שיטות מעבדה טובות. תערובת צבע הג'ל יכולה לשמש עבור 5 שבבי DNA רגישות גבוהה (HS). אם לא נעשה שימוש במשך יותר משעה אחת, יש לאחסן ב-4 מעלות צלזיוס. אחסון בחושך אפשרי עד 6 שבועות.
- כדי לטעון את תערובת צבע הג'ל, ודאו את מיקום לוחית הבסיס של תחנת הפרימינג של השבב והתאימו את הקליפ במיקום הנמוך ביותר.
- משווים את תערובת צבע הג'ל לטמפרטורת החדר למשך 30 דקות, תוך ניטור החשיפה לאור.
- קח שבב DNA HS חדש משקית אטומה ומניחים אותו על תחנת priming שבב, ולאחר מכן להסיר 9.0 μL של תערובת צבע ג'ל ולחלק אותו בחלק התחתון של השבב היטב, מסומן כמו "G".
הערה: צייר את תערובת צבע הג'ל, הימנעות חלקיקים שעלולים להצטבר בתחתית הבקבוקון. תוך חלוקת תערובת צבע הג'ל לתוך שבב ה-DNA HS היטב, להכניס את קצה פיפטה לחלוטין, כדי למנוע היווצרות של בועות אוויר גדולות. יתר על כן, נגיעה פיפטה בשולי הבאר יפיק תוצאות גרועות. - מקם את הוכנה ב 1 מ"ל ולסגור את תחנת priming שבב. ודא את המנעול של לחיצות תפס ולהגדיר את שעון העצר ל 60 s, ולאחר מכן לחץ על הכוכנה למטה עד שהוא מוחזק על ידי קליפ, ובדיוק אחרי 60 s, לשחרר את הכוכנה עם מנגנון שחרור קליפ.
- כאשר הבוכנה נסוגה לפחות לסימן 0.3 מ"ל, לחכות 5 s, ולאחר מכן לאט למשוך בחזרה למצב 1 מ"ל, ואז לפתוח את תחנת priming שבב ושוב להסיר 9.0 μL של תערובת צבע ג'ל לוותר בחלק התחתון של שבב ה-DNA HS היטב, מסומן כמו "G".
- כדי לטעון את סמן ה- DNA, לוותר על 5 μL של סמן ה-DNA לתוך הבאר, מסומן בסמל הסולם. חזור על ההליך עבור כל 11 בארות מדגם.
- כדי לטעון את הסולם ואת הדגימות, לוותר 1 μL של סולם ה-DNA בבאר, מסומן בסמל הסולם ולאחר מכן להוסיף 1 μL של מדגם (בארות משומשות) או 1 μL של סמן (בארות שאינן בשימוש) בכל 11 בארות מדגם.
- וורטקס שבב ה-DNA HS עבור 60 s ב 2,400 סל"ד על ידי הצבת השבב אופקית במתאם. ודא שהבליטה שמתקן את שבב ה-DNA של HS אינה פגומה במהלך המערבולת.
- כדי להכניס את שבב ה- DNA של HS למכשיר מנתח הקטעים, פתח את המכסה וודא שמחסנית האלקטרודה מוכנסת כראוי, ובוחר השבבים ממוקם ל- 'DNA רגישות dsHigh' במכשיר מנתח הקטעים.
- בזהירות להרכיב את שבב ה-DNA HS לתוך כלי הקיבול, אשר מתאים לכיוון אחד בלבד, ואז לאבד את המכסה על ידי הבטחת מחסנית אלקטרודה מתאים בדיוק לתוך בארות של שבב ה-DNA HS.
- התצוגה במסך התוכנה של מנתח הקטעים מציינת את שבב ה- DNA של HS שהוכנס ואת המכסה הסגור, דרך סמל השבב בחלק השמאלי העליון של המסך.
- כדי ליזום את הפעלת שבב ה- DNA של HS, בחר את בדיקת הרגישות הגבוהה של dsDNA מתפריט 'Assay' במסך המכשיר, ולאחר מכן מלא כראוי את טבלת השמות לדוגמה על-ידי הזנת מידע כגון שמות לדוגמה והערות והתחל את הפעלת השבב על-ידי לחיצה על לחצן'התחל' בפינה השמאלית העליונה של המסך.
- ניקוי אלקטרודה לאחר ריצת שבב DNA HS: הסר מיד את שבב ה- DNA המשמש של HS, ברגע שהסייחה תושלם וייפטר ממנה בהתאם לנוהלי מעבדה טובים. בצע את ההליך הבא כדי להבטיח שהאלקטרודות נקיות, ללא שאריות מהסימן הקודם.
- למלא לאט 350 μL של מים כיתה ניתוח deionized לתוך אחת בארות מנקה אלקטרודה ומניחים את מנקה אלקטרודה במכשיר מנתח שבר על ידי פתיחת המכסה ולאחר מכן לסגור את המכסה ולחכות כ 10 s.
- הסר את מנקה האלקטרודה על ידי פתיחת המכסה ולחכות עוד 10 s, עבור המים על האלקטרודות להתאדות לפני סגירת המכסה.
תוצאות
הפרדת פלזמה
8.5-9 דם מ"ל שנאסף cfDNA או cfRNA צינורות משמרים מניב סביב ~ 4 פלזמה מ"ל בנפח. נפח הפלזמה המופרדת מדם שנאסף בצינורות EDTA עשוי להשתנות בהתאם לטמפרטורה. חשיפה של צינורות EDTA המכילים דם בטמפרטורה גבוהה מ 37 מעלות צלזיוס מוביל לירידה בתפוקת נפחהפלזמה 44.
Discussion
איסוף הדם של המטופל בצינור, משלוח ואחסון הם צעדים ראשוניים חיוניים בביופסיה נוזלית. טיפול לא נכון יכול לפגוע באיכות הפלזמה, ולכן, יכול להפריע לתוצאות הביופסיה הנוזלית47. אם דגימת דם נאספת בצינור דם EDTA, הפלזמה חייבת להיות מופרדת בתוך שעתיים של איסוף הדם, כדי למנוע תמוגה של WBCs ושח...
Disclosures
המחברים מצהירים כי אין להם אינטרסים כלכליים מתחרים.
Acknowledgements
המחברים מבקשים להודות לחברי המעבדה לגנומיקה של סרטן וביו-מחשב של מחלות מורכבות על תשומות התצפית החדות שלהם והשתתפותם בדיונים מרובים בשלבים שונים של פרויקט זה. התמיכה במימון כוללת את האגודה למלחמה בסרטן (מענק ICA ל-M.F-M 2017-2019) ואת מענק קמין מרשות החדשנות (עבור מ.פ.מ).
Materials
| Name | Company | Catalog Number | Comments |
| 2100 Bioanalyzer Instrument | Agilent Technologies, Inc. | G2939BA | The 2100 Bioanalyzer system is an established automated electrophoresis tool for the sample quality control of biomolecules. |
| Adjustable Clip for Priming Station | Agilent Technologies, Inc. | 5042-1398 | Used in combination with syringe to apply defined pressure for chip priming. |
| Agilent High Sensitivity DNA Kit | Agilent Technologies, Inc. | 5067-4626 | The High Sensitivity DNA assays are often used for sample quality control for next-generation sequencing libraries |
| cf-DNA/cf-RNA Preservative Tubes | Norgen Biotek Corp. | 63950 | Norgen's cf-DNA/cf-RNA Preservative Tubes are closed, evacuated plastic tubes for the collection and the preservation of cf-DNA, circulating tumor DNA, cf-RNA and circulating tumor cells in human whole blood samples during storage and shipping |
| Chip Priming Station | Agilent Technologies, Inc. | 5065-4401 | Used to load gel matrix into a chip with a syringe provided with each assay kit— used for RNA, DNA, and protein assays. Includes priming station, stop watch, and 1 syringe clip |
| Electrode Cleaner Kit | Agilent Technologies, Inc. | 5065-9951 | Prevents cross-contamination. Removes bacterial or protein contaminants from electrodes. |
| Filters for Gel Matrix | Agilent Technologies, Inc. | 185-5990 | Used for proper mixing of DNA dye concentrate and DNA gel matrix |
| IKA Basic Chip Vortex | IKA-Werke GmbH & Co. KG | MS-3-S36 | Used for proper mixing of DNA ladder and DNA sample on Bioanalyzer assay chips |
| NucleoSpin Tissue kit | MACHEREY-NAGEL | 740952.5 | With the NucleoSpin Tissue kit, genomic DNA can be prepared from tissue, cells (e.g., bacteria), and many other sources. |
| QIAamp circulating nucleic acid kit | Qiagen | 55114 | The QIAamp Circulating Nucleic Acid Kit enables efficient purification of these circulating nucleic acids from human plasma or serum and other cell-free body fluids. |
| QIAvac 24 Plus vacuum manifold | Qiagen | 19413 | The QIAvac 24 Plus vacuum manifold is designed for vacuum processing of QIAGEN columns in parallel. |
| QIAvac Connecting System | Qiagen | 19419 | In combination with the QIAvac Connecting System, the QIAvac 24 Plus vacuum manifold can be used as a flow-through system. The sample flow-through, containing possibly infectious material, is collected in a separate waste bottle. |
| Qubit 2.0 fluorometer | Invitrogen | Q32866 | The Qubit 2.0 Fluorometer is an easy-to-use, analytical instrument designed to work with the Qubit assays for DNA, RNA, and protein quantitation. |
| Qubit assay tubes | Thermo Fisher Scientific | Q32856 | Qubit assay tubes are 500 µL thin-walled polypropylene tubes for use with the Qubit Fluorometer. |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific | Q32851 | The Qubit dsDNA HS (High Sensitivity) Assay Kit is designed specifically for use with the Qubit Fluorometer. The assay is highly selective for double-stranded DNA (dsDNA) over RNA and is designed to be accurate for initial sample concentrations from 10 pg/µL to 100 ng/µL. |
| Vacuum Pump | Qiagen | 84010 | used for vacuum processing of QIAGEN columns |
| Miscellaneous | |||
| 50 ml centrifuge tubes | |||
| Crushed ice | |||
| Ethanol (96–100%) | |||
| Heating block or similar at 56 °C (capable of holding 2 ml collection tubes) | |||
| Isopropanol (100%) | |||
| Microcentrifuge | |||
| Phosphate-buffered saline (PBS) | |||
| Pipettes (adjustable) | |||
| Sterile pipette tips (pipette tips with aerosol barriers are recommended to help prevent cross-contamination) | |||
| Water bath or heating block capable of holding 50 mL centrifuge tubes at 60 °C |
References
- Campbell, P. J., et al. Pan-cancer analysis of whole genomes. Nature. 578 (7793), 82-93 (2020).
- Liotta, L., Petricoin, E. Molecular profiling of human cancer. Nature Reviews Genetics. 1 (1), 48-56 (2000).
- Balamurali, D., et al. ChiTaRS 5.0: the comprehensive database of chimeric transcripts matched with druggable fusions and 3D chromatin maps. Nucleic Acids Research. 48 (1), 825-834 (2019).
- Trédan, O., et al. Molecular screening program to select molecular-based recommended therapies for metastatic cancer patients: Analysis from the ProfiLER trial. Annals of Oncology. 30 (5), 757-765 (2019).
- Oliveira, K. C. S., et al. Current perspectives on circulating tumor DNA, precision medicine, and personalized clinical management of cancer. Molecular Cancer Research. 18 (4), 517-528 (2020).
- Siegal, T. Clinical impact of molecular biomarkers in gliomas. Journal of Clinical Neuroscience. 22 (3), 437-444 (2015).
- Komori, T. The 2016 WHO Classification of Tumours of the Central Nervous System: The Major Points of Revision. Neurologia medico-chirurgica. 57 (7), 301-311 (2017).
- Duffy, M. J., O'Donovan, N., Crown, J. Use of molecular markers for predicting therapy response in cancer patients. Cancer Treatment Reviews. 37 (2), 151-159 (2011).
- Saenz-Antoñanzas, A., et al. Liquid Biopsy in Glioblastoma: Opportunities, Applications and Challenges. Cancers. 11 (7), 950 (2019).
- Marrugo-Ramírez, J., Mir, M., Samitier, J. Blood-Based Cancer Biomarkers in Liquid Biopsy: A Promising Non-Invasive Alternative to Tissue Biopsy. International Journal of Molecular Sciences. 19 (10), 2877 (2018).
- Pantel, K., Alix-Panabières, C. Liquid biopsy in 2016: Circulating tumour cells and cell-free DNA in gastrointestinal cancer. Nature Reviews Gastroenterology & Hepatology. 14 (2), 73-74 (2017).
- Bronkhorst, A. J., et al. The emerging role of cell-free DNA as a molecular marker for cancer management. Biomolecular Detection and Quantification. 17, 100087 (2019).
- Cescon, D. W., Bratman, S. V., Chan, S. M., Siu, L. L. Circulating tumor DNA and liquid biopsy in oncology. Nature Cancer. 1 (3), 276-290 (2020).
- Palmirotta, R., et al. Liquid biopsy of cancer: a multimodal diagnostic tool in clinical oncology. Therapeutic Advances in Medical Oncology. 10, 175883591879463 (2018).
- Cohen, J. D. J., et al. Detection and localization of surgically resectable cancers with a multi-analyte blood test. Science. 359 (6378), 926-930 (2018).
- Wan, J. C. M., et al. Liquid biopsies come of age: towards implementation of circulating tumour DNA. Nature Reviews Cancer. 17 (4), 223-238 (2017).
- Heitzer, E., Haque, I. S., Roberts, C. E. S., Speicher, M. R. Current and future perspectives of liquid biopsies in genomics-driven oncology. Nature Reviews Genetics. 20 (2), 71-88 (2018).
- Thierry, A. R., et al. Origins, structures, and functions of circulating DNA in oncology. Cancer and Metastasis Reviews. 35 (3), 347-376 (2016).
- Buscail, E., et al. High Clinical Value of Liquid Biopsy to Detect Circulating Tumor Cells and Tumor Exosomes in Pancreatic Ductal Adenocarcinoma Patients Eligible for Up-Front Surgery. Cancers. 11 (11), 1656 (2019).
- Heitzer, E., Ulz, P., Geigl, J. B. Circulating Tumor DNA as a Liquid Biopsy for Cancer. Clinical Chemistry. 61 (1), 112-123 (2015).
- Kustanovich, A., Schwartz, R., Peretz, T., Grinshpun, A. Life and death of circulating cell-free DNA. Cancer Biology and Therapy. 20 (8), 1057-1067 (2019).
- Crowley, E., Di Nicolantonio, F., Loupakis, F., Bardelli, A. Liquid biopsy: monitoring cancer-genetics in the blood. Nature Reviews Clinical Oncology. 10 (8), 472-484 (2013).
- Celec, P., et al. Cell-free DNA: the role in pathophysiology and as a biomarker in kidney diseases. Expert Reviews in Molecular Medicine. 20, (2018).
- Gautschi, O., et al. Origin and prognostic value of circulating KRAS mutations in lung cancer patients. Cancer Letters. 254 (2), 265-273 (2007).
- Bidard, F., et al. Detection rate and prognostic value of circulating tumor cells and circulating tumor DNA in metastatic uveal melanoma. International Journal of Cancer. 134 (5), 1207-1213 (2013).
- Chan, K. C. A., et al. Analysis of Plasma Epstein–Barr Virus DNA to Screen for Nasopharyngeal Cancer. New England Journal of Medicine. 377 (6), 513-522 (2017).
- Mao, L., et al. Detection of Oncogene Mutations in Sputum Precedes Diagnosis of Lung Cancer. Cancer Research. 54 (7), 1634-1637 (1994).
- De Mattos-Arruda, L., et al. Cerebrospinal fluid-derived circulating tumour DNA better represents the genomic alterations of brain tumours than plasma. Nature communications. 6 (1), 8839 (2015).
- Khier, S., Lohan, L. Kinetics of circulating cell-free DNA for biomedical applications: critical appraisal of the literature. Future science OA. 4 (4), 295 (2018).
- Misale, S., et al. Resistance to Anti-EGFR therapy in colorectal cancer: From heterogeneity to convergent evolution. Cancer Discovery. 4 (11), 1269-1280 (2014).
- Beddowes, E., Sammut, S. J., Gao, M., Caldas, C. Predicting treatment resistance and relapse through circulating DNA. Breast. 34, 31-35 (2017).
- Sacher, A. G., et al. Prospective Validation of Rapid Plasma Genotyping for the Detection of EGFR and KRAS Mutations in Advanced Lung Cancer. JAMA oncology. 2 (8), 1014-1022 (2016).
- Vaidyanathan, R., et al. Cancer diagnosis: from tumor to liquid biopsy and beyond. Lab on a Chip. 19 (1), 11-34 (2019).
- Kelly, P. Gliomas: Survival, origin and early detection. Surgical Neurology International. 1 (1), 96 (2010).
- Faria, G., Silva, E., Da Fonseca, C., Quirico-Santos, T. Circulating Cell-Free DNA as a Prognostic and Molecular Marker for Patients with Brain Tumors under Perillyl Alcohol-Based Therapy. International Journal of Molecular Sciences. 19 (6), 1610 (2018).
- Liu, M. C., et al. Sensitive and specific multi-cancer detection and localization using methylation signatures in cell-free DNA. Annals of Oncology. 31 (6), 745-759 (2020).
- Enko, D., Halwachs-Baumann, G., Kriegshäuser, G. Plasma free DNA: Evaluation of temperature-associated storage effects observed for roche cell-free DNA collection tubes. Biochemia Medica. 29 (1), 153-156 (2019).
- Streleckiene, G., et al. Effects of Quantification Methods Isolation Kits, Plasma Biobanking, and Hemolysis on Cell-Free DNA Analysis in Plasma. Biopreservation and Biobanking. 17 (6), 553-561 (2019).
- Thress, K. S., et al. Acquired EGFR C797S mutation mediates resistance to AZD9291 in non-small cell lung cancer harboring EGFR T790M. Nature Medicine. 21 (6), 560-562 (2015).
- Ward Gahlawat, A., et al. Evaluation of Storage Tubes for Combined Analysis of Circulating Nucleic Acids in Liquid Biopsies. International Journal of Molecular Sciences. 20 (3), 704 (2019).
- Lu, J. L., Liang, Z. Y. Circulating free DNA in the era of precision oncology: Pre- and post-analytical concerns. Chronic Diseases and Translational Medicine. 2 (4), 223-230 (2016).
- Iyapparaj, P., et al. Optimization of bacteriocin production by Lactobacillus sp. MSU3IR against shrimp bacterial pathogens. Aquatic Biosystems. 9 (1), 12 (2013).
- Ponti, G., et al. The value of fluorimetry (Qubit) and spectrophotometry (NanoDrop) in the quantification of cell-free DNA (cfDNA) in malignant melanoma and prostate cancer patients. Clinica Chimica Acta. 479, 14-19 (2018).
- Medina Diaz, I., et al. Performance of Streck cfDNA blood collection tubes for liquid biopsy testing. PLoS One. 11 (11), 0166354 (2016).
- Perkins, G., et al. Multi-Purpose Utility of Circulating Plasma DNA Testing in Patients with Advanced Cancers. PLoS One. 7 (11), 47020 (2012).
- Mouliere, F., et al. Multi-marker analysis of circulating cell-free DNA toward personalized medicine for colorectal cancer. Molecular Oncology. 8 (5), 927-941 (2014).
- Trigg, R. M., Martinson, L. J., Parpart-Li, S., Shaw, J. A. Factors that influence quality and yield of circulating-free DNA: A systematic review of the methodology literature. Heliyon. 4 (7), 00699 (2018).
- Risberg, B., et al. Effects of Collection and Processing Procedures on Plasma Circulating Cell-Free DNA from Cancer Patients. Journal of Molecular Diagnostics. 20 (6), 883-892 (2018).
- Markus, H., et al. Evaluation of pre-analytical factors affecting plasma DNA analysis. Scientific Reports. 8 (1), 7375 (2018).
- Chen, Z., et al. Comprehensive Evaluation of the Factors Affecting Plasma Circulating Cell-Free DNA Levels and Their Application in Diagnosing Nonsmall Cell Lung Cancer. Genetic Testing and Molecular Biomarkers. 23 (4), 270-276 (2019).
- Schwarzenbach, H., Hoon, D. S. B., Pantel, K. Cell-free nucleic acids as biomarkers in cancer patients. Nature Reviews Cancer. 11 (6), 426-437 (2011).
- Malyuchenko, N. V., et al. PARP1 Inhibitors: antitumor drug design. Acta Naturae. 7 (3), 27-37 (2015).
- Fleischhacker, M., Schmidt, B. Circulating nucleic acids (CNAs) and cancer-A survey. Biochimica et Biophysica Acta (BBA) - Reviews on Cancer. 1775 (1), 181-232 (2007).
- Jung, K., Fleischhacker, M., Rabien, A. Cell-free DNA in the blood as a solid tumor biomarker—A critical appraisal of the literature. Clinica Chimica Acta. 411 (21-22), 1611-1624 (2010).
- Frenkel-Morgenstern, M., et al. ChiTaRS: a database of human, mouse and fruit fly chimeric transcripts and RNA-sequencing data. Nucleic Acids Research. 41, 142-151 (2013).
- Frenkel-Morgenstern, M., et al. ChiTaRS 2.1--an improved database of the chimeric transcripts and RNA-seq data with novel sense-antisense chimeric RNA transcripts. Nucleic Acids Research. 43, 68-75 (2015).
- Gorohovski, A., et al. ChiTaRS-3.1-the enhanced chimeric transcripts and RNA-seq database matched with protein-protein interactions. Nucleic Acids Research. 45 (1), 790-795 (2017).
- Tate, J. G., et al. COSMIC: the Catalogue Of Somatic Mutations In Cancer. Nucleic Acids Research. 47 (1), 941-947 (2019).
- Brennan, C. W., et al. The somatic genomic landscape of glioblastoma. Cell. 155 (2), 462-477 (2013).
- Szopa, W., Burley, T. A., Kramer-Marek, G., Kaspera, W. Diagnostic and Therapeutic Biomarkers in Glioblastoma: Current Status and Future Perspectives. BioMed Research International. 2017, 1-13 (2017).
- Salesse, S., Verfaillie, C. M. BCR/ABL: from molecular mechanisms of leukemia induction to treatment of chronic myelogenous leukemia. Oncogene. 21 (56), 8547-8559 (2002).
- Frenkel-Morgenstern, M., Valencia, A. Novel domain combinations in proteins encoded by chimeric transcripts. Bioinformatics. 28 (12), 67-74 (2012).
- Frenkel-Morgenstern, M., et al. Chimeras taking shape: Potential functions of proteins encoded by chimeric RNA transcripts. Genome Research. 22 (7), 1231-1242 (2012).
- Simon, M., et al. TERT promoter mutations: a novel independent prognostic factor in primary glioblastomas. Neuro-Oncology. 17 (1), 45-52 (2015).
- Waitkus, M. S., Diplas, B. H., Yan, H. Isocitrate dehydrogenase mutations in gliomas. Neuro-Oncology. 18 (1), 16-26 (2016).
- Kindler, T., Meyer, R. G., Fischer, T. BCR-ABL as a target for novel therapeutic interventions. Expert Opinion on Therapeutic Targets. 6 (1), 85-101 (2002).
- Overman, M. J., et al. Use of research biopsies in clinical trials: are risks and benefits adequately discussed. Journal of Clinical Oncology Official Journal of the American Society of Clinical Oncology. 31 (1), 17-22 (2013).
- Vanderlaan, P. A., et al. Success and failure rates of tumor genotyping techniques in routine pathological samples with non-small-cell lung cancer. Lung Cancer. 84 (1), 39-44 (2014).
- Stewart, C. M., Tsui, D. W. Y. Circulating cell-free DNA for non-invasive cancer management. Cancer Genetics. 228-229, 169-179 (2018).
- Normanno, N., et al. The liquid biopsy in the management of colorectal cancer patients: Current applications and future scenarios. Cancer Treatment Reviews. 70, 1-8 (2018).
- Hufnagl, C., et al. Evaluation of circulating cell-free DNA as a molecular monitoring tool in patients with metastatic cancer. Oncology Letters. 19 (2), 1551-1558 (2020).
- Petit, J., et al. Cell-Free DNA as a Diagnostic Blood-Based Biomarker for Colorectal Cancer: A Systematic Review. The Journal of Surgical Research. 236, 184-197 (2019).
- Poulet, G., Massias, J., Taly, V. Liquid Biopsy: General Concepts. Acta Cytologica. 63 (6), 449-455 (2019).
- Alix-Panabières, C. The future of liquid biopsy. Nature. 579 (7800), 9 (2020).
- Eisenstein, M. Could liquid biopsies help deliver better treatment. Nature. 579 (7800), 6-8 (2020).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved