È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Rilevamento di DNA privo di cellule in campioni di plasma sanguigno di pazienti oncologici
In questo articolo
Riepilogo
In questo articolo presentiamo un protocollo dettagliato per la tecnica della biopsia liquida non invasiva, tra cui la raccolta del sangue, la separazione del mantello plasmatico e buffy, l'estrazione del DNA cfDNA e germinale, la quantificazione del DNA cfDNA o germinale e l'analisi dell'arricchimento dei frammenti cfDNA.
Abstract
Identificare le mutazioni nei tumori dei pazienti oncologici è un passo molto importante nella gestione delle malattie. Queste mutazioni fungono da biomarcatori per la diagnosi del tumore, nonché per la selezione del trattamento e la sua risposta nei pazienti oncologici. L'attuale metodo gold standard per rilevare le mutazioni tumorali comporta un test genetico del DNA tumorale attraverso biopsie tumorali. Tuttavia, questo metodo invasivo è difficile da eseguire ripetutamente come test di follow-up del repertorio mutazionale tumorale. La biopsia liquida è una tecnica nuova ed emergente per rilevare le mutazioni tumorali come approccio di biopsia facile da usare e non invasivo.
Le cellule tumorali si moltiplicano rapidamente. In parallelo, numerose cellule tumorali subiscono l'apoptosi. I detriti di queste cellule vengono rilasciati nel sistema circolatorio di un paziente, insieme a pezzi di DNA finemente frammentati, chiamati frammenti di DNA senza cellule (cfDNA), che portano mutazioni del DNA tumorale. Pertanto, per identificare i biomarcatori a base di CFDNA utilizzando la tecnica della biopsia liquida, i campioni di sangue vengono raccolti dai pazienti oncologici, seguiti dalla separazione del plasma e del mantello di buffy. Successivamente, il plasma viene elaborato per l'isolamento del cfDNA e il rispettivo strato di buffy viene elaborato per l'isolamento del DNA genomico di un paziente. Entrambi i campioni di acido nucleico vengono quindi controllati per verificarne la quantità e la qualità; e analizzato per le mutazioni utilizzando tecniche di sequenziamento di nuova generazione (NGS).
In questo manoscritto, presentiamo un protocollo dettagliato per la biopsia liquida, tra cui la raccolta del sangue, la separazione del plasma e del mantello di buffy, l'estrazione del DNA cfDNA e germinale, la quantificazione del DNA cfDNA o germinale e l'analisi dell'arricchimento dei frammenti cfDNA.
Introduzione
I progressi tecnologici hanno portato al sequenziamento di centinaia di genomi e trascrizioni tumorali1. Ciò ha contribuito a comprendere paesaggi di cambiamenti molecolari in diversi tipi dicancro 2. Ulteriori studi su questi paesaggi hanno contribuito a caratterizzare le alterazioni somatiche sequenziali e le fusioni gene-geniche3 che sono coinvolte nella progressione del cancro o del tumore, interrompendo serialmente le vie di apoptosi4. Pertanto, mutazioni somatiche e fusioni gene-geniche possono fornire informazioni sui tumori servendo come biomarcatori nei singoli pazienti per un particolare tumore ditipo 5,identificando la prognosi dei tumori primariesistenti 6,categorizzando i tumori secondari in base aicambiamenti molecolari 7e identificando i target tumorali farmacogable8. Tali informazioni possono facilitare la selezione di trattamenti personalizzati per i pazienti oncologici e la determinazione delle risposte positive e negative al trattamento9. Tuttavia, ottenere materiale tumorale per identificare la profilazione genomica del tessuto tumorale è una procedurainvasiva 10. Inoltre, una biopsia tumorale comprende solo una piccola parte di un tumore eterogeneo; e può, quindi, non essere rappresentativo per il profilo molecolare dell'intero tumore11. Il monitoraggio seriale e la genotipizzazione tumorale richiedono una raccolta ripetuta di tessuti tumorali, che, di solito, non è fattibile a causa dell'invasività della procedura di biopsia tumorale e dei problemi di sicurezza che derivano da taliprocedure 12.
La tecnica della biopsia liquida, d'altra parte, ha guadagnato un'enorme attenzione nell'oncologia diprecisione nell'ultimo decennio 13,14. Ciò è dovuto principalmente alla non invasività di questa tecnica e alla possibilità che venga ripetuta in più punti di tempo, consentendo così una tecnica di monitoraggio facile da usare e sicura per i corsidi malattia 15,16. La biopsia liquida si basa su un fenomeno che le cellule tumorali si moltiplicano rapidamente, e contemporaneamente molte di esse subiscono apoptosi e necrosi. Ciò porta al rilascio di detriti cellulari apoptotici nel sangue dei pazienti, insieme ai frammenti di DNA che vengono tagliati a dimensioni precise durante l'apoptosi17. L'apoptosi delle cellule non cancerose porta anche al rilascio dei suoi detriti cellulari nel sangue, tuttavia, il tasso di apoptosi in queste cellule è relativamente molto inferiore alle cellule tumorali18. Il razionale della tecnica della biopsia liquida è quello di catturare molecole associate al tumore come DNA, RNA, proteine e cellule tumorali14,19 che circolano continuamente nel sangue. Varie tecniche20 possono essere utilizzate per l'analisi di queste molecole tra cui il sequenziamento di nuova generazione (NGS), la reazione a catena della polimerasi a goccia digitale (ddPCR), la PCR in tempo reale e il saggio immunoassorbente legato all'enzima (ELISA). La tecnica della biopsia liquida consente di identificare i biomarcatori che sono caratteristiche delle cellule tumorali. Queste molecole di biomarcatore non vengono rilasciate solo da parti specifiche di un tumore, ma piuttosto da tutte le parti del tumore21. Pertanto, i marcatori identificati nella biopsia liquida rappresentano il profilo molecolare di un intero tumore eterogeneo, oltre ad altri tumori nel corpo, avendo così vantaggi rispetto alla tecnica a base di biopsia tissutale22.
Il cfDNA ha un breve periodo di emidità nel sangue circolante che va da pochi minuti a 1-2 ore23. Tuttavia, il breve periodo di emidità del cfDNA facilita le analisi in tempo reale valutando la risposta al trattamento e le valutazioni dinamiche del tumore. I livelli di CFDNA derivati dal tumore indicano la prognosticazione dello stadio/dimensione tumorale evidenziata da diversi studi, che hanno mostrato una relazione tra i livelli di CFDNA e gli esiti disopravvivenza 24. Inoltre, gli studi hanno dimostrato che il cfDNA ha una capacità di previsione migliore rispetto ai marcatori tumoraliesistenti 25. La prognosticazione del cfDNA è ancora più pronunciata dopo il trattamento del cancro, livelli più elevati di cfDNA dopo il trattamento sono ben correlati con un tasso ridotto di sopravvivenza e resistenza al trattamento. Mentre, livelli più bassi di cfDNA dopo la terapia corrispondono generalmente a una risposta positiva al trattamento. Inoltre, il cfDNA facilita la diagnosi precoce della risposta al trattamento rispetto ai metodi di rilevamento tradizionali.
Il cfDNA aumenta la possibilità di individuazione precoce delle mutazioni associate al cancro: durante la malattia in fase iniziale15, l'insorgenzadei sintomi 26 e prima della diagnosi di cancro fino a 2anni 27. Poiché cfDNA viene rilasciato da più regioni tumorali o focolai, la sua analisi fornisce una visione completa del genoma tumorale che rappresenta28. Pertanto, il cfDNA consente di rilevare mutazioni somatiche che potrebbero essere mancate nei campioni di tessuto29. Poiché l'eterogeneità intra-tumorale e le mutazioni subclonali possono essere rilevate sequenziando in profondità regioni genomiche che coprono migliaia di basi, quindi l'analisi del cfDNA consente di scoprire specifici sottotipi molecolari con distinte firme genomiche13. Per ottenere un livello simile di informazioni attraverso il campione di tessuto sarebbero state necessarie molte biopsie solide.
Inoltre, i livelli di cfDNA nei pazienti con una malattia localizzata come il cancro al colon, all'ovaio e al polmone dopo un trattamento chirurgico e / o chemioterapia, hanno dimostrato di essere un potente marcatore prognostico per la recidiva del cancro e gli esiti deltrattamento 20. Inoltre, nei pazienti con cancro al colon, al seno e al polmone, le analisi del cfDNA dal sangue potrebbero rilevare con successo i cambiamenti specifici del tumore, che hanno portato alla previsione precisa della recidiva con diversi mesi dianticipo 13. Inoltre, i marcatori di resistenza al trattamento, come le mutazioni KRAS in pazienti con CRC che ricevono terapia anti-EGFR30; VAF per geni come PIK3CA, MED1 o EGFR in pazienti con tumore al seno dopo il trattamento con varie terapie31; e la mutazione della resistenza EGFR T790M nei pazienti con cancro ai polmoni trattati con TKI32 mirati all'EGFR può anche essere identificata dall'analisi cfDNA.
In sintesi, l'analisi cfDNA può essere utilizzata per identificare biomarcatori precisi nel campo dell'oncologia13,33. In questo protocollo, campioni di sangue di 3 pazienti con glioma e 3 controlli sani sono stati elaborati per ottenere DNA genomico da WBC e cfDNA dal plasma. Nel cancro al glioma, le mutazioni in IDH , TERT, ATRX, EGFR e TP53 servono come marcatori diagnostici e prognostici che possono aiutare nella diagnosi precoce dei tumori del glioma, classificando diversi tipi di tumori del glioma, guidando il trattamento accurato per il singolo paziente e comprendendo la risposta altrattamento 34,35. Lo stato mutazionale di questi geni può essere identificato utilizzando cfDNA derivato dal sangue. In questo manoscritto, presentiamo un protocollo dettagliato di cfDNA derivato dal plasma che è stato utilizzato per studiare i cambiamenti mutazionali nel cancro al glioma12. Tale protocollo di biopsia liquida a base di CFDNA spiegato in questo articolo può essere utilizzato per studiare i cambiamenti mutazionali in molti altri tipi di tumori. Inoltre, un recente studio ha dimostrato che la biopsia liquida a base di CFDNA può rilevare 50 diversi tipi di cancro36.
La raccolta, lo stoccaggio e la spedizione dei campioni di sangue sono passaggi cruciali in questo protocollo, poiché la temperatura incontrollata durante questi passaggi causa lisi dei WBC, portando al rilascio di DNA genomico dal WBC nel plasma e causando la contaminazione del campione cfDNA, che influisce sul resto della procedura37. L'emolisi dovuta alla temperatura incontrollata può compromettere i processi di preparazione del campione a valle di cfDNA, come i passaggiPCR 38. Il siero contiene un'alta percentuale di cfDNA germinale piuttosto che plasma, sebbene presenti un grande rumore di fondo per cfDNA39associato al tumore . Pertanto, per isolare il cfDNA associato al tumore, il plasma è un campioneadatto 39. Il sangue prelevato in un antico coagulante contenente un tubo di raccolta del sangue deve essere centrifugato immediatamente o entro un massimo di due ore, per separare il plasma ed evitare la contaminazione da CFDNA. In questo protocollo vengono utilizzati tubi di raccolta del sangue di conservazione cfDNA commerciali dedicati (vedi Tabella dei materiali), che sono un'alternativa all'anticoagulante contenente tubi di raccolta del sangue. Questi tubi dedicati per la raccolta del sangue preservano cfDNA e cfRNA e prevengono la llisi dei WBC per un massimo di 30 giorni a temperatura ambiente e fino a 8 giorni a 37 °C. Ciò facilita il mantenimento della temperatura appropriata durante una spedizione del campione di sangue e fino a quando il plasma e il WBC nonsono separati 40.
Esistono attualmente tre tipi di metodologie di estrazione cfDNA: isolamento di fase, colonna di spin a base di membrana di silicio e isolamento magnetico basato su perline41. Il metodo della colonna di spin a base di membrana di silicio ha prodotto un'elevata quantità di cfDNA ad alta integrità rispetto ad altri metodi di estrazione cfDNA42.
La valutazione quantitativa del DNA è un requisito fondamentale nella biopsia liquida, è necessario sviluppare una procedura semplice, conveniente e standardizzata per la loro facile implementazione e ampio utilizzo. Tre metodi comunemente usati per la quantificazione cfDNA sono spettrofotometrici, fluorimetrici e qPCR. Il metodo fluorimetrico si è dimostrato migliore rispetto agli altri metodi riguardanti l'accuratezza, il costo e la facilità dicondotta 43.
L'integrità e la purezza del cfDNA possono essere stimate mediante elettroforesi di agarosio o elettroforesi capillare. L'elettroforesi dell'agarosio non mostra sensibilità a bassa concentrazione di cfDNA né ha un'alta risoluzione per mostrare una dimensione precisa del frammento di cfDNA. D'altra parte, l'elettroforesi capillare ha un vantaggio rispetto all'elettroforesi dell'agarosio superando le sfide associate e, quindi, ampiamente utilizzata dai ricercatori per l'analisi delle dimensioni dei frammenti cfDNA. In questo protocollo, la distribuzione delle dimensioni dei frammenti del cfDNA isolato è stata stimata utilizzando uno strumento automatizzato di elettroforesi capillare (vedi Tabella dei materiali).
Protocollo
Prima della raccolta del sangue, è richiesto e deve essere ottenuto il consenso informato dei soggetti che partecipano alla ricerca. La ricerca descritta in questo manoscritto è stata eseguita in conformità con il Rabin Medical Center, israele etico committee (codice etico: 0039-17-RMC) e la Facoltà di Medicina Der Christian-Albrechts-Universität zu Kiel, Comitato etico tedesco (codice etico: D 405/14).
1. Raccolta e conservazione di campioni di sangue in tubi conservanti cfDNA o cfRNA
- Etichettare correttamente i tubi di conservazione
- Raccogliere ~8 ml di sangue nel tubo di conservazione cf-DNA (vedi Tabella dei materiali), utilizzando un set di raccolta del sangue e un supporto, secondo il protocollo istituzionale standard per la venipunctura come descritto di seguito.
NOTA: L'uso di un set di raccolta del sangue può impedire un possibile riflusso del sangue dal tubo.- Allineare il paziente con il braccio in posizione verso il basso.
- Tenere il tubo in posizione verticale, con il cappuccio affrontato verso l'alto, assicurando al contempo che il contenuto del tubo non tocchi il cappuccio o la punta dell'ago.
- Quando il sangue inizia a fluire nel tubo, rilasciare lentamente il laccio emostatico.
- Immediatamente dopo che il tubo è pieno di sangue (capacità massima: 8,4 ml di sangue intero), invertire delicatamente il tubo (girare il polso del braccio che tiene il tubo di 180 ° verso il basso e indietro) 5 volte per stabilizzare il campione.
NOTA: L'inversione assicura che il conservante sia miscelato uniformemente con il campione. Tuttavia, non scuotere di nuovo il contenuto, anche prima della preparazione del plasma. L'insufficiente miscelazione dei conservanti con il campione di sangue porta alla destabilizzazione del contenuto e alla formazione di micro coaguli o emolisi. In questa fase, il protocollo può essere continuato immediatamente per la separazione del plasma o i tubi pieni di sangue possono attendere fino a 30 giorni a temperatura ambiente (15-25 °C) e fino a 8 giorni a 37 °C.
2. Separazione e conservazione del mantello al plasma e buffy
- Centrifugare il tubo di conservazione pieno di sangue a 425 x g per 20 minuti a temperatura ambiente per separare il plasma.
NOTA: I passaggi 2.2 e 2.3 devono essere eseguiti in un armadio di biosicurezza. - Pipettare con cura lo strato superiore di plasma su un tubo fresco in aliquote da 1 ml, senza disturbare gli strati inferiori.
- Trasferire con cura il successivo strato di pelo di buffy su un tubo fresco (lo strato appare come un anello sopra i pellet RBC), evitando gli RBC nello strato inferiore.
- Procedere al passaggio 3 con plasma e al passaggio 4 con il cappotto buffy. Se necessario conservare il contenuto separato a -80 °C.
3. Purificazione della cfDNA circolante da 1 mL di plasma
NOTA: Questo passaggio viene eseguito con un kit commerciale (vedi Tabella dei materiali). Tutti i buffer sono dotati del kit.
- Preparazione di tamponi e reagenti
ATTENZIONE: Non aggiungere soluzioni acide o candeggina direttamente ai rifiuti di preparazione del campione. I sali di guanidina presenti nel tampone di Lysis, nel tampone di legame e nel Wash Buffer-1 se combinati con candeggina o acidi possono produrre composti altamente reattivi.- Buffer di legame: mescolare 300 mL di concentrato tampone di legame con 200 mL di isopropanolo al 100% per fare 500 mL di buffer di binding funzionante. Conservare a temperatura ambiente.
NOTA: Il tampone legante consente il legame ottimale degli acidi nucleici circolanti alla membrana silicea. 500 mL del tampone di legame sono sufficienti per la lavorazione rispettivamente di 276, 138, 92, 69 o 55 campioni di 1, 2, 3, 4 o 5 mL di plasma ed è stabile per 1 anno a temperatura ambiente. - Tampone di lavaggio-1: Mescolare 19 mL di concentrato Wash Buffer-1 con 25 mL di etanolo 96-100% per fare 44 mL di Wash Buffer-1 funzionante. Conservare a temperatura ambiente.
NOTA: Wash Buffer-1 elimina i contaminanti legati alla membrana silicea. 44 mL di tampone di lavaggio di lavoro-1 è sufficiente per la lavorazione di 73 campioni di 1/2/3/4/5 mL di plasma ed è stabile per 1 anno a temperatura ambiente. - Tampone di lavaggio-2: Mescolare bene 13 mL Wash Buffer-2 concentrato con 30 mL di etanolo 96-100% per fare 43 mL di wash buffer-2 funzionante. Conservare a temperatura ambiente.
NOTA: Wash Buffer-2 elimina i contaminanti legati alla membrana silicea. 43 mL di tampone di lavaggio di lavoro-2 sono sufficienti per la lavorazione di ~56 campioni di 1/2/3/4/5 mL di plasma ed è stabile per 1 anno a temperatura ambiente. - Ad un tubo contenente 310 μg di RNA portatore liofilizzato, aggiungere 1.550 μL di tampone di eluizione, per preparare una soluzione di RNA portante di 0,2 μg/μL. Dopo aver completamente dissolto l'RNA portante, dividere la soluzione in aliquote adatte e conservare a -30 °C a -15 °C. Non congelare-scongelare queste aliquote più di 3 volte. Al buffer di Lysis, come mostrato nella tabella S1, aggiungere l'RNA portante ricostituito sciolto nel buffer di eluizione.
NOTA: Poiché l'RNA portante non si dissolve direttamente nel buffer di lisi, deve essere sciolto prima in un buffer di eluizione e poi in Lysis Buffer. In primo luogo, il legame silice membrana-acidi nucleici viene migliorato quando ci sono pochissime molecole bersaglio presenti nel campione. In secondo luogo, il rischio di degradazione dell'RNA è ridotto a causa della presenza di grandi quantità di RNA vettore.
- Buffer di legame: mescolare 300 mL di concentrato tampone di legame con 200 mL di isopropanolo al 100% per fare 500 mL di buffer di binding funzionante. Conservare a temperatura ambiente.
- Prima di iniziare l'isolamento, portare le colonne e i campioni a temperatura ambiente e regolare i volumi del campione a 1 mL con soluzione salina sterile tamponata da fosfati (PBS), se necessario. Preriscaldare 2 bagni d'acqua o blocchi riscaldanti che contengono tubi di centrifuga da 50 ml e 2 tubi di raccolta mL rispettivamente a 60 °C e 56 °C.
- A un tubo di centrifuga da 50 ml aggiungere 100 μL di Proteinasi K, 1 mL plasma e 0,8 ml di tampone di lisi contenente 1,0 μg di RNA portante (preparato nel passaggio 3.1.4). Chiudere il tubo di centrifuga con un cappuccio e mescolare il contenuto a impulsi-vortice per 30 s, garantendo al contempo un vortice visibile nel tubo. Un'accurata miscelazione dei contenuti è importante per una lysis efficiente.
NOTA: Subito dopo il vortice, procedere al passaggio 3.4, senza indugio. - Incubare la soluzione a 60 °C per 30 min.
- Rimuovere il cappuccio, aggiungere 1,8 ml del tampone di legame al tubo e mescolare accuratamente con il vortice di impulso per 15-30 s dopo aver posizionato il cappuccio.
- Incubare la miscela risultante per 5 minuti sul ghiaccio e inserire la colonna della membrana di silice nell'apparato del vuoto collegato alla pompa per vuoto. Quindi, inserire saldamente un'estensione del tubo da 20 ml nella colonna aperta per evitare perdite di campioni.
- Versare con cura la miscela incubata nell'estensore del tubo della colonna e accendere la pompa per vuoto. Dopo che tutta la miscela di lisato attraversa completamente le colonne, spegnere la pompa del vuoto, rilasciare la pressione a 0 mbar e rimuovere e scartare l'estensore del tubo.
NOTA: Evitando la contaminazione incrociata, l'estensore del tubo deve essere scartato con attenzione, per impedirne la diffusione su colonne adiacenti. - Rimuovere la colonna dall'apparato aspirapolvere, inserirlo nel tubo di raccolta e centrifugare a 11.000 x g per 30 s a temperatura ambiente, per rimuovere eventuali lysate residuo. Scartare il flusso.
- Aggiungere 600 μL di Wash Buffer-1 nella colonna, centrifugare a 11.000 x g per 1 min a temperatura ambiente, scartare il flusso.
- Aggiungere 750 μL di Wash Buffer-2 alla colonna, centrifugare a 11.000 x g per 1 min a temperatura ambiente e scartare il flusso.
- Aggiungere 750 μL di etanolo (96-100%) alla colonna, centrifugare a 11.000 x g per 1 min a temperatura ambiente e scartare il flusso.
- Centrifugare la colonna a 20.000 x g per 3 minuti, posizionandolo in un tubo di raccolta pulito da 2 ml.
- Asciugare completamente l'assemblaggio della colonna di membrana posizionandolo in un nuovo tubo di raccolta da 2 ml con il coperchio aperto e incubando a 56 °C per 10 minuti.
- Posizionare la colonna in un tubo di eluizione pulito da 1,5 ml. Al centro della membrana della colonna, applicare 20-150 μL di tampone di eluizione e incubare a temperatura ambiente per 3 minuti con il coperchio chiuso.
NOTA: Assicurarsi che il tampone di eluizione sia equilibrato a temperatura ambiente. In caso di utilizzo di tampone di eluizione inferiore a 50 μL, assicurarsi che sia erogato con cura al centro della membrana. Questo aiuta con la completa eluizione del DNA legato. Tuttavia, il volume di eluizione non è fisso e può essere modificato in base alle applicazioni downstream. L'eluito recuperato può essere fino a 5 μL e certamente inferiore al volume di eluizione applicato alla colonna. - Centrifugare l'eluito recuperato in un microcentrifugo a 20.000 x g per 1 minuto per eludere gli acidi nucleici e conservare a -20 °C.
4. Purificazione del DNA genomico dal mantello di buffy
NOTA: Il kit commerciale utilizzato in questo protocollo è menzionato nella tabella dei materiali. Tamponi e reagenti menzionati nel protocollo seguente, ad esempio tampone di lisi A, tampone di lisi B, tampone di lavaggio X, tampone di lavaggio Y, tampone proteico, tampone di eluizione e proteinasi K fanno parte di questo kit commerciale.
- Preparazione dei tamponi e dei reagenti
ATTENZIONE: Non aggiungere soluzioni acide o candeggina direttamente ai rifiuti di preparazione del campione. I sali di guanidina presenti nel tampone B di Lysis e nel tampone di lavaggio X se combinati con candeggina o acidi, possono produrre composti altamente reattivi.- Tampone di lavaggio Y: Mescolare bene 12 mL di concentrato Wash Buffer Y con 48 mL di etanolo (96-100%) per ottenere 60 mL di wash buffer di lavoro Y. Conservare a temperatura ambiente.
NOTA: 60 mL di tampone di lavaggio Y funzionante è sufficiente per la lavorazione di 100 campioni di mantello buffy ed è stabile per 1 anno. - Proteinasi K: Preparare la soluzione di Proteinasi K sciogliendo 30 mg di proteinasi K lyophilized in 1,35 mL di Proteinase Buffer.
NOTA: La soluzione di lavoro totale di Proteinase K è sufficiente per la lavorazione di 52 campioni di mantello di buffy. La soluzione di lavoro proteinasi K può essere conservata per almeno 6 mesi a -20 °C.
- Tampone di lavaggio Y: Mescolare bene 12 mL di concentrato Wash Buffer Y con 48 mL di etanolo (96-100%) per ottenere 60 mL di wash buffer di lavoro Y. Conservare a temperatura ambiente.
- Passaggi prima dell'avvio della procedura
- Equilibrare il mantello buffy a temperatura ambiente.
- Impostare il blocco termico o il bagno d'acqua a 56 °C.
- Sospendere il mantello di buffy nel tampone di Lysis A per ottenere un volume finale di 200 μL. Quindi aggiungere 25 μL di soluzione proteinasi K e 200 μL di tampone di lisi B. Mescolare con vortici e incubare a 70 °C per 10-15 min. Assicurarsi che i campioni siano completamente coperti con la soluzione dilisi.
NOTA: Per la lavorazione di serie di campioni, proteinasi K e tampone di lysis A possono essere premiscelati 10-15 minuti prima della procedura, ma non più prima, poiché proteinasi K auto-digerisce nel tampone di Lysis A senza substrato. - Aggiungere 210 μL di etanolo al 96-100% alla miscela e al vortice di cui sopra vigorosamente.
NOTA: L'aggiunta di etanolo può formare un precipitato filante; tuttavia, ciò non influirà sull'isolamento del DNA. Assicurarsi di caricare il precipitato anche sulla colonna, come illustrato nei passaggi seguenti. - Caricare l'intero campione sulla colonna di silice posta in un tubo di raccolta. Centrifuga per 1 min a 11.000 x g. Posizionare la colonna in un nuovo tubo di raccolta e scartare il tubo precedente insieme al flusso.
NOTA: Ripetere il passaggio di centrifugazione se il campione non viene disegnato completamente attraverso la matrice. - Aggiungere 500 μL di tampone di lavaggio X, centrifugare per 1 min a 11.000 x g e scartare il flusso.
- Posizionare la colonna nel tubo di raccolta, aggiungere 600 μL di Wash Buffer Y sulla colonna, centrifugare per 1 min a 11.000 x g e scartare il flusso.
- Ancora una volta, posizionare la colonna nel tubo di raccolta e centrifugare la colonna per 1 minuto a 20.000 x g per asciugare la membrana di silice.
- Incubare la colonna a temperatura ambiente per 1 minuto, posta in un tubo di microcentrifugo da 1,5 ml, quindi aggiungere 100 μL di tampone di eluizione. Quindi, elutare il DNA centrifugando per 1 minuto a 11.000 x g e conservare a -20 °C.
5. Quantificazione del CFDNA e del DNA genomico mediante fluorometro
- Prima di avviare il protocollo, eseguire la procedura seguente.
- Diluire 2 μL di DNA genomico eluita (dal passaggio 4.9) in proporzioni 1:10 con acqua ultrapura priva di nucleasi. A causa delle basse concentrazioni previste, non diluire i campioni di CFDNA dal passaggio 3.15.
- Equilibrare il saggio Standard #1 e saggio Standard #2 a temperatura ambiente.
- Preparare un totale di 6 tubi chiari a parete sottile di dimensioni di 0,5 ml.
NOTA: Il protocollo presentato è per la quantificazione di 2 cfDNA e 2 campioni di DNA genomico, quindi 4 tubi per 4 campioni e questo test richiede 2 standard. - Etichettare i coperchi del tubo.
NOTA: L'etichettatura sul lato del tubo potrebbe interferire con la lettura. Inoltre, i tubi standard di dosaggio sono etichettati con cura, poiché la calibrazione del fluorometro richiede che gli standard siano nell'ordine corretto. - Diluire il reagente del saggio in 1:200 con il tampone di dosaggio per preparare la soluzione di lavoro. Per 4 campioni e 2 standard, utilizzare 6 μL di reagente di dosaggio più 1.194 μL di tampone di dosaggio per fare 1.200 μL (200 μL in ogni tubo) di soluzione di lavoro.
NOTA: Non utilizzare un contenitore di vetro, ma utilizzare un tubo di plastica pulito. Ogni tubo deve contenere circa 200 μL del volume finale (un tubo standard di dosaggio deve contenere 190 μL della soluzione di lavoro e il tubo campione deve contenere 180-199 μL della soluzione di lavoro). Deve essere preparata una soluzione di lavoro sufficiente per soddisfare tutti gli standard e i campioni di dosaggio. - Nei tubi standard di dosaggio di lavoro, aggiungere 190 μL di soluzione di lavoro e 10 μL standard di dosaggio e mescolare la soluzione vortice per 2-3 s. Evitare la formazione di bolle all'interno della soluzione.
- Nei tubi campione aggiungere 198 μL di soluzione di lavoro e 2 μL di CFDNA o DNA genomico. Mescolare la soluzione vortice per 2-3 s e mantenerla incubata a temperatura ambiente per 2 minuti.
- Nella schermata 'Home' dello strumento fluorometro, premere 'DNA' e selezionare 'dsDNA High Sensitivity Assay', per visualizzare lo schermo 'Standards', quindi premere 'Yes' sullo schermo del fluorometro 'Standards' per leggere gli standard.
- Nella camera campione, inserire il saggio Standard #1 tubo, chiudere il coperchio e premere 'Leggi'. Rimuovere il tubo una volta completata la lettura (circa 3 s) e ripetere lo stesso passaggio per le #2.
- Una schermata campione viene visualizzata dopo il completamento del processo di calibrazione, quindi inserire un tubo campione e ripetere il passaggio '5.8'. La "schermata campione" mostrerà quindi un valore che corrisponde alla concentrazione del campione dopo la diluizione nel tubo campione.
- Per ogni campione ripetere il passaggio '5.9', fino a quando tutti i campioni non vengono letti.
- Utilizzare la seguente equazione per calcolare la concentrazione effettiva del campione.
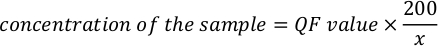
NOTA: I valori di saggio sono in ng/mL e corrispondono alla concentrazione dopo la diluizione nel tubo di dosaggio. Equazione menzionata nel passaggio 5.11 precedente, valore QF è il valore dato dallo strumento fluorometro, e x è il numero di microlitri di campione aggiunti al tubo di dosaggio. Le unità per i valori QF generate dall'equazione sono simili al valore fornito dal fluorometro. Ad esempio, se il valore del fluorometro è in ng/mL, le unità per la concentrazione calcolata dall'equazione sono ng/mL.
6. Distribuzione delle dimensioni dei frammenti di DNA del cfDNA per analizzatore di frammenti
- Il passaggio prima di iniziare la procedura: Equilibrare il concentrato di colorante del DNA e la matrice di gel di DNA a temperatura ambiente per 30 minuti.
- Preparazione del mix gel-colorante
ATTENZIONE: Maneggiare le soluzioni con cautela in quanto DMSO è noto per facilitare l'ingresso di molecole organiche nei tessuti.- Scongelare accuratamente il DMSO vortice la fiala di concentrato di colorante al DNA per 10 s. Pipettare 15 μL di questo concentrato in una fiala a matrice di gel di DNA e conservare a 4 °C al buio.
- Ancora una volta, vortice la fiala limitata per 10 s fino a quando non viene visualizzato la miscelazione del gel e del colorante.
- Versare la miscela su un filtro di rotazione sul recipiente superiore.
- Microcentrifugare il filtro di rotazione a 2.240 x g ± 20% per 10 min a temperatura ambiente.
- Etichettare il gel-colorante preparato nel tubo e scartare il filtro, secondo buone pratiche di laboratorio. Etichettare il tubo e registrare la data di preparazione.
NOTA: Scartare il filtrato secondo buone pratiche di laboratorio. Il mix gel-colorante può essere utilizzato per 5 chip di DNA ad alta sensibilità (HS). Se non utilizzato per più di 1 h, conservare a 4 °C. Lo stoccaggio al buio è possibile per un massimo di 6 settimane.
- Per caricare la miscela gel-colorante, assicurarsi la posizione della piastra di base della stazione di adescamento del truciolo e regolare la clip nella posizione più bassa.
- Equilibrare il mix gel-colorante a temperatura ambiente per 30 minuti, monitorando al contempo l'esposizione alla luce.
- Prendere un nuovo chip HS DNA da un sacchetto sigillato e posizionarlo sulla stazione di adescamento del truciolo, quindi rimuovere 9,0 μL della miscela gel-colorante e erogarlo nella parte inferiore del chip bene, contrassegnato come "G".
NOTA: Disegnare il mix gel-colorante, evitando particelle che possono accumularsi nella parte inferiore del flaconcino. Durante l'erogazione del mix gel-colorante nel chip HS DNA bene, inserire completamente la punta della pipetta, per evitare la formazione di grandi bolle d'aria. Inoltre, toccare la pipetta ai bordi del pozzo produrrà scarsi risultati. - Posizionare lo stantuffo a 1 mL e chiudere la stazione di adescamento del truciolo. Assicurarsi che il blocco del fermo clic e impostare il timer su 60 s, quindi premere lo stantuffo verso il basso fino a quando non viene tenuto dalla clip, e esattamente dopo 60 s, rilasciare lo stantuffo con il meccanismo di rilascio della clip.
- Quando lo stantuffo si ritira almeno al segno di 0,3 mL, attendere 5 s, quindi tirare lentamente indietro nella posizione di 1 mL, quindi aprire la stazione di adescamento del truciolo e rimuovere nuovamente 9,0 μL della miscela gel-colorante e erogare nella parte inferiore del chip HS DNA bene, contrassegnato come 'G'.
- Per caricare il marcatore del DNA, distribuire 5 μL del marcatore di DNA nel pozzo, contrassegnato con il simbolo della scala. Ripetere la procedura per tutti gli 11 pozzi campione.
- Per caricare la scala e i campioni, erogare 1 μL della scala del DNA nel pozzo, contrassegnata con il simbolo della scala e quindi aggiungere 1 μL di campione (pozzi usati) o 1 μL di marcatore (pozzi inutilizzati) in tutti gli 11 pozzi campione.
- Vortice il chip HS DNA per 60 s a 2.400 giri/min posizionando il chip orizzontalmente nell'adattatore. Assicurarsi che il rigonfiamento che fissa il chip HS DNA non sia danneggiato durante il vortice.
- Per inserire il chip HS DNA nello strumento dell'analizzatore di frammenti, aprire il coperchio e assicurarsi che la cartuccia dell'elettrodo sia inserita correttamente e che il selettore del chip sia posizionato su 'DNA ad alta sensibilità' nello strumento dell'analizzatore di frammenti.
- Montare con cura il chip HS DNA nel recipiente, che si adatta in un solo modo, quindi perdere il coperchio assicurando che la cartuccia dell'elettrodo si adatti esattamente ai pozzi del chip HS DNA.
- Il display sullo schermo del software dell'analizzatore di frammenti indica il chip HS DNA inserito e il coperchio chiuso, attraverso l'icona del chip in alto a sinistra dello schermo.
- Per avviare l'esecuzione del chip HS DNA, selezionare il saggio dsDNA ad alta sensibilità dal menu 'Assay' sullo schermo dello strumento, quindi riempire correttamente la tabella dei nomi dei campioni alimentando informazioni come nomi di campioni e commenti e avviare l'esecuzione del chip facendo clic sul pulsante 'Start ' in alto a destra dello schermo.
- Pulizia degli elettrodi dopo l'esecuzione di un chip HS DNA: rimuovere immediatamente il chip HS DNA utilizzato, non appena il test è completato e smaltirlo secondo buone pratiche di laboratorio. Eseguire la seguente procedura per assicurarsi che gli elettrodi siano puliti, senza residui rimanenti del saggio precedente.
- Riempire lentamente 350 μL di acqua deionizzata di grado di analisi in uno dei pozzi di pulizia degli elettrodi e posizionare il pulitore dell'elettrodo nello strumento dell'analizzatore di frammenti aprendo il coperchio e quindi chiudere il coperchio e attendere circa 10 s.
- Rimuovere il pulitore dell'elettrodo aprendo il coperchio e attendere altri 10 s, affinché l'acqua sugli elettrodi evapori prima di chiudere il coperchio.
Risultati
Separazione al plasma
8,5-9 ml di sangue raccolto in tubi conservanti cfDNA o cfRNA produce circa ~ 4 ml di plasma in volume. Il volume di plasma separato dal sangue raccolto nei tubi EDTA può variare a seconda della temperatura. L'esposizione di tubi EDTA contenenti sangue ad una temperatura superiore a 37 °C porta a una diminuzione della resa del volumeplasmatico 44.
Risultati saggio fluorometro
La concentrazione di cfDNA nel ...
Discussione
La raccolta del sangue di un paziente in un tubo, la spedizione e lo stoccaggio sono i primi passi cruciali nella biopsia liquida. Una manipolazione impropria può compromettere la qualità del plasma e, pertanto, può interferire con i risultati della biopsialiquida 47. Se un campione di sangue viene raccolto in un tubo sanguigno EDTA, il plasma deve essere separato entro due ore dalla raccolta del sangue per evitare la lisi dei WBC e il rilascio del suo DNA genomico nel plasma4...
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti.
Riconoscimenti
Gli autori ringraziano i membri del Laboratorio di Genomica del Cancro e Biocalcolamento delle Malattie Complesse per i loro acumesi input osservazionali e la loro partecipazione a molteplici discussioni in diverse fasi di questo progetto. Il sostegno finanziario include Israel Cancer Association (sovvenzione ICA per M.F-M 2017-2019) e sovvenzione Kamin dell'Israel Innovation Authority (per M.F-M.).
Materiali
| Name | Company | Catalog Number | Comments |
| 2100 Bioanalyzer Instrument | Agilent Technologies, Inc. | G2939BA | The 2100 Bioanalyzer system is an established automated electrophoresis tool for the sample quality control of biomolecules. |
| Adjustable Clip for Priming Station | Agilent Technologies, Inc. | 5042-1398 | Used in combination with syringe to apply defined pressure for chip priming. |
| Agilent High Sensitivity DNA Kit | Agilent Technologies, Inc. | 5067-4626 | The High Sensitivity DNA assays are often used for sample quality control for next-generation sequencing libraries |
| cf-DNA/cf-RNA Preservative Tubes | Norgen Biotek Corp. | 63950 | Norgen's cf-DNA/cf-RNA Preservative Tubes are closed, evacuated plastic tubes for the collection and the preservation of cf-DNA, circulating tumor DNA, cf-RNA and circulating tumor cells in human whole blood samples during storage and shipping |
| Chip Priming Station | Agilent Technologies, Inc. | 5065-4401 | Used to load gel matrix into a chip with a syringe provided with each assay kit— used for RNA, DNA, and protein assays. Includes priming station, stop watch, and 1 syringe clip |
| Electrode Cleaner Kit | Agilent Technologies, Inc. | 5065-9951 | Prevents cross-contamination. Removes bacterial or protein contaminants from electrodes. |
| Filters for Gel Matrix | Agilent Technologies, Inc. | 185-5990 | Used for proper mixing of DNA dye concentrate and DNA gel matrix |
| IKA Basic Chip Vortex | IKA-Werke GmbH & Co. KG | MS-3-S36 | Used for proper mixing of DNA ladder and DNA sample on Bioanalyzer assay chips |
| NucleoSpin Tissue kit | MACHEREY-NAGEL | 740952.5 | With the NucleoSpin Tissue kit, genomic DNA can be prepared from tissue, cells (e.g., bacteria), and many other sources. |
| QIAamp circulating nucleic acid kit | Qiagen | 55114 | The QIAamp Circulating Nucleic Acid Kit enables efficient purification of these circulating nucleic acids from human plasma or serum and other cell-free body fluids. |
| QIAvac 24 Plus vacuum manifold | Qiagen | 19413 | The QIAvac 24 Plus vacuum manifold is designed for vacuum processing of QIAGEN columns in parallel. |
| QIAvac Connecting System | Qiagen | 19419 | In combination with the QIAvac Connecting System, the QIAvac 24 Plus vacuum manifold can be used as a flow-through system. The sample flow-through, containing possibly infectious material, is collected in a separate waste bottle. |
| Qubit 2.0 fluorometer | Invitrogen | Q32866 | The Qubit 2.0 Fluorometer is an easy-to-use, analytical instrument designed to work with the Qubit assays for DNA, RNA, and protein quantitation. |
| Qubit assay tubes | Thermo Fisher Scientific | Q32856 | Qubit assay tubes are 500 µL thin-walled polypropylene tubes for use with the Qubit Fluorometer. |
| Qubit dsDNA HS Assay Kit | Thermo Fisher Scientific | Q32851 | The Qubit dsDNA HS (High Sensitivity) Assay Kit is designed specifically for use with the Qubit Fluorometer. The assay is highly selective for double-stranded DNA (dsDNA) over RNA and is designed to be accurate for initial sample concentrations from 10 pg/µL to 100 ng/µL. |
| Vacuum Pump | Qiagen | 84010 | used for vacuum processing of QIAGEN columns |
| Miscellaneous | |||
| 50 ml centrifuge tubes | |||
| Crushed ice | |||
| Ethanol (96–100%) | |||
| Heating block or similar at 56 °C (capable of holding 2 ml collection tubes) | |||
| Isopropanol (100%) | |||
| Microcentrifuge | |||
| Phosphate-buffered saline (PBS) | |||
| Pipettes (adjustable) | |||
| Sterile pipette tips (pipette tips with aerosol barriers are recommended to help prevent cross-contamination) | |||
| Water bath or heating block capable of holding 50 mL centrifuge tubes at 60 °C |
Riferimenti
- Campbell, P. J., et al. Pan-cancer analysis of whole genomes. Nature. 578 (7793), 82-93 (2020).
- Liotta, L., Petricoin, E. Molecular profiling of human cancer. Nature Reviews Genetics. 1 (1), 48-56 (2000).
- Balamurali, D., et al. ChiTaRS 5.0: the comprehensive database of chimeric transcripts matched with druggable fusions and 3D chromatin maps. Nucleic Acids Research. 48 (1), 825-834 (2019).
- Trédan, O., et al. Molecular screening program to select molecular-based recommended therapies for metastatic cancer patients: Analysis from the ProfiLER trial. Annals of Oncology. 30 (5), 757-765 (2019).
- Oliveira, K. C. S., et al. Current perspectives on circulating tumor DNA, precision medicine, and personalized clinical management of cancer. Molecular Cancer Research. 18 (4), 517-528 (2020).
- Siegal, T. Clinical impact of molecular biomarkers in gliomas. Journal of Clinical Neuroscience. 22 (3), 437-444 (2015).
- Komori, T. The 2016 WHO Classification of Tumours of the Central Nervous System: The Major Points of Revision. Neurologia medico-chirurgica. 57 (7), 301-311 (2017).
- Duffy, M. J., O'Donovan, N., Crown, J. Use of molecular markers for predicting therapy response in cancer patients. Cancer Treatment Reviews. 37 (2), 151-159 (2011).
- Saenz-Antoñanzas, A., et al. Liquid Biopsy in Glioblastoma: Opportunities, Applications and Challenges. Cancers. 11 (7), 950 (2019).
- Marrugo-Ramírez, J., Mir, M., Samitier, J. Blood-Based Cancer Biomarkers in Liquid Biopsy: A Promising Non-Invasive Alternative to Tissue Biopsy. International Journal of Molecular Sciences. 19 (10), 2877 (2018).
- Pantel, K., Alix-Panabières, C. Liquid biopsy in 2016: Circulating tumour cells and cell-free DNA in gastrointestinal cancer. Nature Reviews Gastroenterology & Hepatology. 14 (2), 73-74 (2017).
- Bronkhorst, A. J., et al. The emerging role of cell-free DNA as a molecular marker for cancer management. Biomolecular Detection and Quantification. 17, 100087 (2019).
- Cescon, D. W., Bratman, S. V., Chan, S. M., Siu, L. L. Circulating tumor DNA and liquid biopsy in oncology. Nature Cancer. 1 (3), 276-290 (2020).
- Palmirotta, R., et al. Liquid biopsy of cancer: a multimodal diagnostic tool in clinical oncology. Therapeutic Advances in Medical Oncology. 10, 175883591879463 (2018).
- Cohen, J. D. J., et al. Detection and localization of surgically resectable cancers with a multi-analyte blood test. Science. 359 (6378), 926-930 (2018).
- Wan, J. C. M., et al. Liquid biopsies come of age: towards implementation of circulating tumour DNA. Nature Reviews Cancer. 17 (4), 223-238 (2017).
- Heitzer, E., Haque, I. S., Roberts, C. E. S., Speicher, M. R. Current and future perspectives of liquid biopsies in genomics-driven oncology. Nature Reviews Genetics. 20 (2), 71-88 (2018).
- Thierry, A. R., et al. Origins, structures, and functions of circulating DNA in oncology. Cancer and Metastasis Reviews. 35 (3), 347-376 (2016).
- Buscail, E., et al. High Clinical Value of Liquid Biopsy to Detect Circulating Tumor Cells and Tumor Exosomes in Pancreatic Ductal Adenocarcinoma Patients Eligible for Up-Front Surgery. Cancers. 11 (11), 1656 (2019).
- Heitzer, E., Ulz, P., Geigl, J. B. Circulating Tumor DNA as a Liquid Biopsy for Cancer. Clinical Chemistry. 61 (1), 112-123 (2015).
- Kustanovich, A., Schwartz, R., Peretz, T., Grinshpun, A. Life and death of circulating cell-free DNA. Cancer Biology and Therapy. 20 (8), 1057-1067 (2019).
- Crowley, E., Di Nicolantonio, F., Loupakis, F., Bardelli, A. Liquid biopsy: monitoring cancer-genetics in the blood. Nature Reviews Clinical Oncology. 10 (8), 472-484 (2013).
- Celec, P., et al. Cell-free DNA: the role in pathophysiology and as a biomarker in kidney diseases. Expert Reviews in Molecular Medicine. 20, (2018).
- Gautschi, O., et al. Origin and prognostic value of circulating KRAS mutations in lung cancer patients. Cancer Letters. 254 (2), 265-273 (2007).
- Bidard, F., et al. Detection rate and prognostic value of circulating tumor cells and circulating tumor DNA in metastatic uveal melanoma. International Journal of Cancer. 134 (5), 1207-1213 (2013).
- Chan, K. C. A., et al. Analysis of Plasma Epstein–Barr Virus DNA to Screen for Nasopharyngeal Cancer. New England Journal of Medicine. 377 (6), 513-522 (2017).
- Mao, L., et al. Detection of Oncogene Mutations in Sputum Precedes Diagnosis of Lung Cancer. Cancer Research. 54 (7), 1634-1637 (1994).
- De Mattos-Arruda, L., et al. Cerebrospinal fluid-derived circulating tumour DNA better represents the genomic alterations of brain tumours than plasma. Nature communications. 6 (1), 8839 (2015).
- Khier, S., Lohan, L. Kinetics of circulating cell-free DNA for biomedical applications: critical appraisal of the literature. Future science OA. 4 (4), 295 (2018).
- Misale, S., et al. Resistance to Anti-EGFR therapy in colorectal cancer: From heterogeneity to convergent evolution. Cancer Discovery. 4 (11), 1269-1280 (2014).
- Beddowes, E., Sammut, S. J., Gao, M., Caldas, C. Predicting treatment resistance and relapse through circulating DNA. Breast. 34, 31-35 (2017).
- Sacher, A. G., et al. Prospective Validation of Rapid Plasma Genotyping for the Detection of EGFR and KRAS Mutations in Advanced Lung Cancer. JAMA oncology. 2 (8), 1014-1022 (2016).
- Vaidyanathan, R., et al. Cancer diagnosis: from tumor to liquid biopsy and beyond. Lab on a Chip. 19 (1), 11-34 (2019).
- Kelly, P. Gliomas: Survival, origin and early detection. Surgical Neurology International. 1 (1), 96 (2010).
- Faria, G., Silva, E., Da Fonseca, C., Quirico-Santos, T. Circulating Cell-Free DNA as a Prognostic and Molecular Marker for Patients with Brain Tumors under Perillyl Alcohol-Based Therapy. International Journal of Molecular Sciences. 19 (6), 1610 (2018).
- Liu, M. C., et al. Sensitive and specific multi-cancer detection and localization using methylation signatures in cell-free DNA. Annals of Oncology. 31 (6), 745-759 (2020).
- Enko, D., Halwachs-Baumann, G., Kriegshäuser, G. Plasma free DNA: Evaluation of temperature-associated storage effects observed for roche cell-free DNA collection tubes. Biochemia Medica. 29 (1), 153-156 (2019).
- Streleckiene, G., et al. Effects of Quantification Methods Isolation Kits, Plasma Biobanking, and Hemolysis on Cell-Free DNA Analysis in Plasma. Biopreservation and Biobanking. 17 (6), 553-561 (2019).
- Thress, K. S., et al. Acquired EGFR C797S mutation mediates resistance to AZD9291 in non-small cell lung cancer harboring EGFR T790M. Nature Medicine. 21 (6), 560-562 (2015).
- Ward Gahlawat, A., et al. Evaluation of Storage Tubes for Combined Analysis of Circulating Nucleic Acids in Liquid Biopsies. International Journal of Molecular Sciences. 20 (3), 704 (2019).
- Lu, J. L., Liang, Z. Y. Circulating free DNA in the era of precision oncology: Pre- and post-analytical concerns. Chronic Diseases and Translational Medicine. 2 (4), 223-230 (2016).
- Iyapparaj, P., et al. Optimization of bacteriocin production by Lactobacillus sp. MSU3IR against shrimp bacterial pathogens. Aquatic Biosystems. 9 (1), 12 (2013).
- Ponti, G., et al. The value of fluorimetry (Qubit) and spectrophotometry (NanoDrop) in the quantification of cell-free DNA (cfDNA) in malignant melanoma and prostate cancer patients. Clinica Chimica Acta. 479, 14-19 (2018).
- Medina Diaz, I., et al. Performance of Streck cfDNA blood collection tubes for liquid biopsy testing. PLoS One. 11 (11), 0166354 (2016).
- Perkins, G., et al. Multi-Purpose Utility of Circulating Plasma DNA Testing in Patients with Advanced Cancers. PLoS One. 7 (11), 47020 (2012).
- Mouliere, F., et al. Multi-marker analysis of circulating cell-free DNA toward personalized medicine for colorectal cancer. Molecular Oncology. 8 (5), 927-941 (2014).
- Trigg, R. M., Martinson, L. J., Parpart-Li, S., Shaw, J. A. Factors that influence quality and yield of circulating-free DNA: A systematic review of the methodology literature. Heliyon. 4 (7), 00699 (2018).
- Risberg, B., et al. Effects of Collection and Processing Procedures on Plasma Circulating Cell-Free DNA from Cancer Patients. Journal of Molecular Diagnostics. 20 (6), 883-892 (2018).
- Markus, H., et al. Evaluation of pre-analytical factors affecting plasma DNA analysis. Scientific Reports. 8 (1), 7375 (2018).
- Chen, Z., et al. Comprehensive Evaluation of the Factors Affecting Plasma Circulating Cell-Free DNA Levels and Their Application in Diagnosing Nonsmall Cell Lung Cancer. Genetic Testing and Molecular Biomarkers. 23 (4), 270-276 (2019).
- Schwarzenbach, H., Hoon, D. S. B., Pantel, K. Cell-free nucleic acids as biomarkers in cancer patients. Nature Reviews Cancer. 11 (6), 426-437 (2011).
- Malyuchenko, N. V., et al. PARP1 Inhibitors: antitumor drug design. Acta Naturae. 7 (3), 27-37 (2015).
- Fleischhacker, M., Schmidt, B. Circulating nucleic acids (CNAs) and cancer-A survey. Biochimica et Biophysica Acta (BBA) - Reviews on Cancer. 1775 (1), 181-232 (2007).
- Jung, K., Fleischhacker, M., Rabien, A. Cell-free DNA in the blood as a solid tumor biomarker—A critical appraisal of the literature. Clinica Chimica Acta. 411 (21-22), 1611-1624 (2010).
- Frenkel-Morgenstern, M., et al. ChiTaRS: a database of human, mouse and fruit fly chimeric transcripts and RNA-sequencing data. Nucleic Acids Research. 41, 142-151 (2013).
- Frenkel-Morgenstern, M., et al. ChiTaRS 2.1--an improved database of the chimeric transcripts and RNA-seq data with novel sense-antisense chimeric RNA transcripts. Nucleic Acids Research. 43, 68-75 (2015).
- Gorohovski, A., et al. ChiTaRS-3.1-the enhanced chimeric transcripts and RNA-seq database matched with protein-protein interactions. Nucleic Acids Research. 45 (1), 790-795 (2017).
- Tate, J. G., et al. COSMIC: the Catalogue Of Somatic Mutations In Cancer. Nucleic Acids Research. 47 (1), 941-947 (2019).
- Brennan, C. W., et al. The somatic genomic landscape of glioblastoma. Cell. 155 (2), 462-477 (2013).
- Szopa, W., Burley, T. A., Kramer-Marek, G., Kaspera, W. Diagnostic and Therapeutic Biomarkers in Glioblastoma: Current Status and Future Perspectives. BioMed Research International. 2017, 1-13 (2017).
- Salesse, S., Verfaillie, C. M. BCR/ABL: from molecular mechanisms of leukemia induction to treatment of chronic myelogenous leukemia. Oncogene. 21 (56), 8547-8559 (2002).
- Frenkel-Morgenstern, M., Valencia, A. Novel domain combinations in proteins encoded by chimeric transcripts. Bioinformatics. 28 (12), 67-74 (2012).
- Frenkel-Morgenstern, M., et al. Chimeras taking shape: Potential functions of proteins encoded by chimeric RNA transcripts. Genome Research. 22 (7), 1231-1242 (2012).
- Simon, M., et al. TERT promoter mutations: a novel independent prognostic factor in primary glioblastomas. Neuro-Oncology. 17 (1), 45-52 (2015).
- Waitkus, M. S., Diplas, B. H., Yan, H. Isocitrate dehydrogenase mutations in gliomas. Neuro-Oncology. 18 (1), 16-26 (2016).
- Kindler, T., Meyer, R. G., Fischer, T. BCR-ABL as a target for novel therapeutic interventions. Expert Opinion on Therapeutic Targets. 6 (1), 85-101 (2002).
- Overman, M. J., et al. Use of research biopsies in clinical trials: are risks and benefits adequately discussed. Journal of Clinical Oncology Official Journal of the American Society of Clinical Oncology. 31 (1), 17-22 (2013).
- Vanderlaan, P. A., et al. Success and failure rates of tumor genotyping techniques in routine pathological samples with non-small-cell lung cancer. Lung Cancer. 84 (1), 39-44 (2014).
- Stewart, C. M., Tsui, D. W. Y. Circulating cell-free DNA for non-invasive cancer management. Cancer Genetics. 228-229, 169-179 (2018).
- Normanno, N., et al. The liquid biopsy in the management of colorectal cancer patients: Current applications and future scenarios. Cancer Treatment Reviews. 70, 1-8 (2018).
- Hufnagl, C., et al. Evaluation of circulating cell-free DNA as a molecular monitoring tool in patients with metastatic cancer. Oncology Letters. 19 (2), 1551-1558 (2020).
- Petit, J., et al. Cell-Free DNA as a Diagnostic Blood-Based Biomarker for Colorectal Cancer: A Systematic Review. The Journal of Surgical Research. 236, 184-197 (2019).
- Poulet, G., Massias, J., Taly, V. Liquid Biopsy: General Concepts. Acta Cytologica. 63 (6), 449-455 (2019).
- Alix-Panabières, C. The future of liquid biopsy. Nature. 579 (7800), 9 (2020).
- Eisenstein, M. Could liquid biopsies help deliver better treatment. Nature. 579 (7800), 6-8 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon